服务
文章信息
- 宋书杰, 熊智强, 王勇
- Song Shujie, Xiong Zhiqiang, Wang Yong
- 利用合成调控RNA技术提高大肠杆菌异源合成红霉素前体6-脱氧红霉内酯B产量
- Enhancing erythromycin precursor 6-dEB production by using synthetic small regulatory RNAs in Escherichia coli
- 生物工程学报, 2015, 31(7): 1039-1049
- Chin J Biotech, 2015, 31(7): 1039-1049
- 10.13345/j.cjb.140581
-
文章历史
- Received: December 15, 2014
- Accepted: January 30, 2015
2. 上海海洋大学 食品学院海洋药物教研室,上海 200306
2. Marine Medicine Laboratory, College of Food Science & technology, Shanghai Ocean University, Shanghai 200306, China
红霉素是目前聚酮类化合物生物合成研究最为清楚的模式化合物,其生物合成途径的建立和机制的解析,为异源生物合成获得红霉素或其衍生物奠定了基础。在过去的十年中,大肠杆菌以其良好的异源基因表达特性和遗传特性,越来越成为聚酮异源合成的主流通用宿主之一。通过合成生物学的设计,目前已经解决了ACP修饰、前体供应、红霉内酯环的后修饰等关键问题[1, 2],实现了在大肠杆菌中的异源合成。其高效合成体系的设计,也做了诸多的尝试:如为了提高质粒稳定性,2003年Murli等[3]用pccB和pccA基因整合到Escherichia coli编码甲基丙二酰辅酶A脱羧酶ygfG位点,同时用RSF1010的复制起始位点替换pET28a来源于pBR322的起始位点,将pBP144改造为pKOS207-129,获得工程菌株K207-3/pKOS207-129/pBP130在摇瓶中可以获得22.5 mg/L的6-dEB。Lau等[4]利用该菌株,在5 L生物反应器中,以分批补料的方法进行高密度培养,得到了1.1 g/L的6-dEB,这是到目前为止,6-dEB异源生物合成的最高纪录。为了提高宿主本身的稳定性,Wang等[5]将红霉素聚酮合酶基因eryAI、eryAII和eryAIII通过染色体重组Red/ET方法整合入大肠杆菌染色体上,获得了染色体修饰的稳定菌株,与多个质粒共表达相比,该菌株可稳定的合成红霉素中间体6-dEB。Wang等[6]在早期的研究中发现,在红霉糖多胞菌中过量表达S-腺苷甲硫氨酸合成酶基因对红霉素的生物合成有促进作用。通过在大肠杆菌中过量表达该基因,将大肠杆菌中6-dEB的产量提高了一倍[7]。此外,通过前体代谢工程手段,对大肠杆菌异源合成聚酮类化合物做了一系列的定向合成尝试[8]。
尽管以红霉素为代表的聚酮异源生物合成获得了巨大的进展,但是仍然存在着诸多问题。比如,目前无抗菌活性的红霉素中间体6-dEB在高密度培养大肠杆菌中的效价约为 1.1 g/L,而红霉素的活性产物红霉素A或红霉素C在大肠杆菌中的效价却低于1 mg/L。尽管有着发酵周期、遗传操作等诸多优势,这一水平与红霉素的原产株红霉糖多孢菌相比,仍然有较大的差距 (工业规模中,红霉糖多胞菌产红霉素A效价可达5−10 g/L)。本课题组通过基于全局代谢网络模型的in silico模拟计算发现[9],在目前以大肠杆菌为宿主的红霉素异源合成中,即使给予充足的丙酸前体,其中仅有约5%−10%的前体直接参与了聚酮的合成,而其他的前体即使供应充足,也并不参与聚酮的生物合成。计算还发现,目前大肠杆菌合成聚酮化合物的实际产量仅到了理论产量的1/10左右。这说明复杂天然产物在底盘细胞中的异源合成仍有巨大的提升空间,绝不仅仅是引入新的合成途径这样简单。我们需要对这一复杂代谢网络的本质和作用规律有更深层次的认识,以此指导设计更高效的异源合成系统。
近年来,基因组尺度代谢模型 (Genome- scale metabolic model,GSMM) 的提出[10]使菌种改造研究提升到系统的高度。随着组学数据的日益丰富和完善,大肠杆菌、酿酒酵母等多个物种的全基因组代谢网络模型得以重建。结合经典的代谢网络通量分析方法 (如代谢通量分析 (Metabolic flux analysis,MFA)[11, 12]、通量平衡分析 (Flux balance analysis,FBA)、最小代谢调整分析 (Minimization of metabolic adjustment,MOMA)[13]等),人们可对宿主细胞的代谢表型进行模拟和预测,从而发现一些可能影响目标化合物产量的关键节点;通过对这些节点实施改造可改变细胞代谢流的分布状况,使得更多的碳架底物通向目标产物的合成,从而改善目标产物的产量。在前期研究中,我们针对 红霉素合成关键中间体6dEB异源合成产率低的问题 (这也是目前异源合成普遍存在的问题),分析了其在不同异源宿主中的最大理论产率、影响理论产率的关键因素以及为提高产率而可能需要改造的潜在基因位点。
本研究中,我们基于Fiest等[14]构建的 E. coli基因组尺度代谢模型iAF1260,利用in silico分析挖掘出了若干需要弱化的关键靶点基因。采用sRNA (Synthetic small regulatory RNAs) 技术,对拟下调的关键靶点进行了实验验证 (即在翻译水平上通过sRNA干扰调控靶点基因的表达[15],利用sRNA与靶基因的mRNA结合,从而阻遏靶基因的mRNA与核糖体的结合,继而抑制靶基因的蛋白表达),并取得了良好的结果。
1 材料与方法 1.1 菌株、质粒及相关试剂菌株E. coli DH10B作为克隆宿主,菌株 E. coli WG (pZG07/pZG08) 为聚酮类化合物红霉素母核6-dEB的生产菌 (表1)。分子克隆相关的酶、DNA片段与质粒抽提纯化试剂盒分别由NEB公司、TaKaRa公司及Axygen公司提供。培养基、抗生素及其他相关试剂购自Oxiod、国药集团与上海生工生物工程技术服务有限公司。引物由南京金斯瑞生物科技公司合成。
| Strain and plasmid | Relevant characteristics | Source |
| WG | F-ompT hsdSB (rB-mB-) gal dcm (DE3) DprpRBCDHT7prom-sfp, T7prom-prpE | Stored in our lab |
| DH10B | F-endA1 recA1 galE15 galK16 nupG rpsL ΔlacX74 Φ80lacZΔM15 araD139 Δ (ara, leu)7697 mcrA Δ (mrr-hsdRMS-mcrBC), λ- | Invitrogen |
| BL21 (DE3) | F-ompT gal dcm lon hsdSB (rB- mB-) λ (DE3 [lacI lacUV5-T7 gene 1 ind1 sam7 nin5]) | Invitrogen |
| pZG07 | pET21c derived, T7prom-DEBS2-ribosome binding site-DEBS3-T7term, ampicillin | Stored in our lab |
| pZG08 | pET28a derived, T7prom-pccB-rbs-accA1-T7prom-DEBS1-T7term, kanamycin | Stored in our lab |
| pJF650 | pACYCDuent-1 derived, whose T7 promoter site was replaced with deo-snRNA sequences in-frame, chloramphenicol | This study |
| pSJ02 | pJF650 derived, carrying Pr Promoter-tdcD binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ07 | pJF650 derived, carrying Pr Promoter-pflD binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ08 | pJF650 derived, carrying Pr Promoter-paaF binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ09 | pJF650 derived, carrying Pr Promoter-fadJ binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ10 | pJF650 derived, carrying Pr Promoter-fadB binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ11 | pJF650 derived, carrying Pr Promoter-ackA binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ10 | pJF650 derived, carrying Pr Promoter-pta binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ13 | pJF650 derived, carrying Pr Promoter-leuD binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ14 | pJF650 derived, carrying Pr Promoter-leuC binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ20 | pJF651 derived, carrying Pr Promoter-ptsH binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ21 | pJF652 derived, carrying Pr Promoter-ptsI binding site-micC-Pr terminator, chloramphenicol | This study |
| pSJ53 | pJF653 derived, carrying Pr Promoter-lsrC binding site-micC-Pr terminator, chloramphenicol | This study |
本研究采用sRNAs调控大肠杆菌染色体基因。质粒构建方法如下[16]:弱化靶点deoB (编码磷酸戊糖变位酶)的sRNAs基因序列片段deoB-sRNA (表2) (包括Pr启动子、deoB靶基因结合位点和TE终止子,并在两端分别引入 NdeⅠ和Hind Ⅲ酶切位点),由上海捷瑞生物工程有限公司直接化学合成。通过引物pACYC-F和pACYC-R,以pACYCDunet-1为模板,克隆得到只含氯霉素抗性和p15A复制子的载体片段,回收后采用NdeⅠ和Hind Ⅲ双酶切;同时采用NdeⅠ和Hind Ⅲ双酶切上述化学合成的deoB-sRNA序列片段。清洁回收双酶切后的载体片段和deoB-sRNA片段通过T4DNA连接酶连接,得到模板质粒pJF650。
| Primer name | Primer sequence (5′-3′) |
| tdcD-sRNA-F | CAAAACAACCGGAAATTCATTCATTTTCTGTTGGGCCATTGCATTGCC |
| tdcD-sRNA-R | ATGAATGAATTTCCGGTTGTTTTGGCAACCATTATCACCGCCAGAGGTA |
| pflD-sRNA-F | GAGGCGAGAGATACGATTCGTCATTTTCTGTTGGGCCATTGCATTGCC |
| pflD-sRNA-R | ATGACGAATCGTATCTCTCGCCTCGCAACCATTATCACCGCCAGAGGTA |
| paaF-sRNA-F | ACGGCTGACGATCAGTTCGCTCATTTTCTGTTGGGCCATTGCATTGCC |
| paaF-sRNA-R | ATGAGCGAACTGATCGTCAGCCGtGCAACCATTATCACCGCCAGAGGTA |
| fadJ-sRNA-F | GGTAAACGCTGATGTCATTTCCATTTTCTGTTGGGCCATTGCATTGCC |
| fadJ-sRNA-R | ATGGAAATGACATCAGCGTTTACCGCAACCATTATCACCGCCAGAGGTA |
| fadB-sRNA-F | CAGGGTGTCGCCTTTGTAAAGCATTTTCTGTTGGGCCATTGCATTGCC |
| fadB-sRNA-R | ATGCTTTACAAAGGCGACACCCTGGCAACCATTATCACCGCCAGAGGTA |
| ackA-sRNA-F | AACCAGTACTAACTTACTCGACATTTTCTGTTGGGCCATTGCATTGCC |
| ackA-sRNA-R | ATGTCGAGTAAGTTAGTACTGGTTGCAACCATTATCACCGCCAGAGGTA |
| pta-sRNA-F | GATCAGCATAATAATACGGGACACTTTCTGTTGGGCCATTGCATTGCC |
| pta-sRNA-R | GTGTCCCGTATTATTATGCTGATCGCAACCATTATCACCGCCAGAGGTA |
| leuD-sRNA-F | GTGTTTGATAAATTTCTCTGCCATTTTCTGTTGGGCCATTGCATTGCC |
| leuD-sRNA-R | ATGGCAGAGAAATTTATCAAACACGCAACCATTATCACCGCCAGAGGTA |
| leuC-sRNA-F | TTTTTCGTATAACGTCTTAGCCATTTTCTGTTGGGCCATTGCATTGCC |
| leuC-sRNA-R | ATGGCTAAGACGTTATACGAAAAAGCAACCATTATCACCGCCAGAGGTA |
| ptsH-sRNA-F | AATGGTAACTTCTTGCTGGAACATTTTCTGTTGGGCCATTGCATTGCC |
| ptsH-sRNA-R | ATGTTCCAGCAAGAAGTTACCATTGCAACCATTATCACCGCCAGAGGTA |
| ptsI-sRNA-F | GGATGCTAAAATGCCTGAAATCATTTTCTGTTGGGCCATTGCATTGCC |
| ptsI-sRNA-R | ATGATTTCAGGCATTTTAGCATCCGCAACCATTATCACCGCCAGAGGTA |
| lsrC-sRNA-F | GTTGTTCTGAATAAACTTCAGCATTTTCTGTTGGGCCATTGCATTGCC |
| lsrC-sRNA-R | ATGCTGAAGTTTATTCAGAACAACGCAACCATTATCACCGCCAGAGGTA |
| pACYC-F | CCCAAGCTTCTGAAACCTCAGGCATTTGA |
| pACYC-R | CGGGATCCGCGCAACGCAATTAATGTAA |
| deoB-sRNA |
CATATGGGATCCTAACACCGTGCGTGTTGACTATTTTACCTCTGGCGGTGATAATGGTTGCCACCA TAATAAATGCACGTTTCATTTTCTGTTGGGCCATTGCATTGCCACTGATTTTCCAACATATAAAAA GACAAGCCCGAACAGTCGTCCGGGCTTTTTTTCTCGAGCTCGAGCCAGGCATCAAATAAAACGA AAGGCTCAGTCGAAAGACTGGGCCTTTCGTTTTATCTGTTTTTGTCGGTGAACGCTCTCTACTAG AGTCACACTGGCTCACCTTCGGGTGGGCCTTTCTGCGTTTATAACTAGTAGATCTAAGCTT |
以出发质粒pJF650为模板,采用25 μL的PCR体系:2×GC 缓冲液I 12.5 μL,上下游引物各0.25 μL,dNTPs 2.5 μL,模板质粒0.25 μL,LA Taq酶0.25 μL,ddH2O将反应体系补足至25 μL;利用表2中的引物,通过定点突变PCR扩增直接获得能够弱化大肠杆菌中不同靶点基因表达的sRNAs质粒。以构建弱化tdcD靶基因的sRNA质粒为例,采用表2中的tdcD-sRNA-F和tdcD-sRNA-R引物及上述PCR条件,以模板质粒pJF650为模板,通过定点突变PCR直接将pJF650质粒骨架deoB靶基因结合位点突变成tdcD靶基因结合位点,得到能够弱化tdcD基因的质粒pSJ02。
1.3 In silico模型的构建与靶点预测通量分布比较分析典(Flux distribution comparison analysis,FDCA) 是建立在通量平衡分析 (FBA) 基础上的一种代谢通量分析方法,即是从代谢反应的通量变化出发,上溯到相应的基因,是一种自下而上 (Bottom-up) 的算法。本研究采用大肠杆菌基因组尺度代谢模型iAF1260为计算模型,分别以生物量和最佳红霉素母核6-dEB合成速率为目标,进行通量比较分析,计算相应的代谢通量分布,找出差异显著的节点 (反应),这些节点将成为影响红霉素母核6-dEB合成的潜在改造靶点。
1.4 菌株的培养与发酵 1.4.1 菌株的培养将sRNAs质粒 (以pACYCDuet-1作为对照) 转化到6-dEB产生菌E. coli WG (pZG07/pZG08)中,挑取单菌落到含有氨苄青霉素100 μg/L、卡那霉素50 μg/L和氯霉素34 μg/L抗性的LB培养基 (NaCl 10 g/L,蛋白胨10 g/L,酵母提取物5 g/L,甘油15 g/L) 中,37 ℃、250 r/min过夜培养,作为种子液备用。
1.4.2 菌株的发酵发酵采用摇瓶培养:在100 mL三角摇瓶中加入10 mL发酵培养基[17] (NaCl 10 g/L,蛋白胨10 g/L,酵母提取物5 g/L,甘油15 g/L,100 mmol/L HEPES,pH 7.6,含有氨苄青霉素100 μg/L,卡那霉素50 μg/L,氯霉素34 μg/L,诱导剂异丙基-β-D-硫代吡喃半乳糖苷 (IPTG) 24 μg/mL,前体丙酸钠20 μmmol/L)。将1%的种子液接种于发酵培养基中,置于22 ℃、 250 r/min的摇床上发酵5 d,每个样品3次平行。
1.5 分析检测菌体的生长状况使用比浊法测定,即用UV2000紫外分光光度计在波长λ=600 nm时测定菌液的吸光值A (OD600)。
发酵产物6-dEB利用HPLC-ELSD进行分析检测[18],测定条件如下:色谱柱为TSK-100V (5 μm,4.6 mm×50 mm),流速1 mL/min,流动相为乙腈和水,采用50%乙腈等度洗脱。ELSD检测器条件:漂移管温度50 ℃,气体流速 1.6 L/min,增益值8。
2 结果与分析 2.1 In silico预测关键基因靶点FDCA方法是本课题组开发的基于全基因组代谢模型新算法,依据本研究中FDCA方法描述,我们首先设定最大生物量和最大红霉素母核6-dEB生物合成速率 (预期菌株) 作为两个独立的目标,进行通量平衡分析,再对两个设定目标的基因尺度通量分布进行比较,挖掘出大量潜在的代谢靶点 (反应)[9]。基于6-dEB合成途径的前体供应代谢网络,筛选出12个潜在需要弱化的基因靶点进行实验验证 (表3)。
| Gene ID | Genes | Reaction (enzyme) |
| b3846 | fadB | Fatty acid oxidation complex |
| b1514 | lsrC | Autoinducer-2 ABC transporter |
| b2415 | ptsH | The phosphoenolpyruvate: sugar phosphotransferase system HPr |
| b2416 | ptsI | Phosphoenolpyruvate-protein phosphotransferase PtsI |
| b2296 | ackA | Acetatekinase |
| b2297 | pta | Phosphateacetyltransferase/phosphatepropionyltransferase |
| b0071 | leuD | Isopropylmalateisomerase LeuD |
| b0072 | leuC | Isopropylmalateisomerase LeuC |
| b2341 | fadJ | FadJ component of anaerobic fatty acid oxidation complex |
| b3115 | tdcD | Propionatekinase |
| b1393 | paaF | Predicted 2,3-dehydroadipyl-CoA hydratase |
| b3951 | pflD | Formate acetyltransferase 2 |
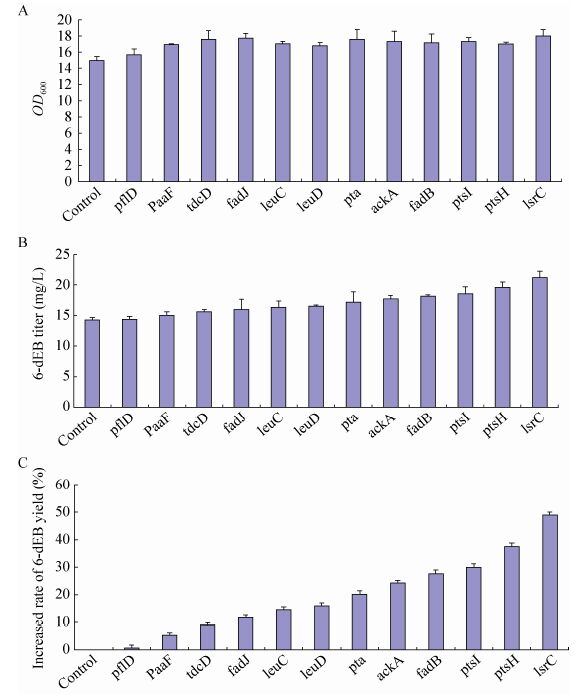
sRNAs技术[16]是一种新的细菌基因调控方法,主要在翻译水平上利用sRNA干扰可以有效地弱化靶点基因的表达。本研究预测靶点的弱化验证是利用sRNAs技术来实施,由图1A可知,与对照空质粒pACYCDuent-1相比,单独弱化12个靶点基因对细胞生长都有轻微的促进作用;例如弱化lsrC基因的菌株OD600达到18 (对照菌OD600为15)。总体而言,弱化筛选的靶点对菌株生长没有显著影响。
|
| 图1 sRNAs 弱化调控对E. coli合成6-dEB的影响 Fig.1 Influence of heterologous 6-dEB production in E. coli by using sRNAs. (A) Cell growth. (B) 6dEB titer. (C) The increased rate of 6dEB yield. |
单独弱化12个靶点基因对宿主异源合成6-dEB的影响却有明显差异 (图1B和1C)。实验表明,弱化lsrC、ptsH、ptsI、fadB、ackA和pta基因后宿主菌的6-dEB产量分别为21.3、19.6、18.6、18.2、17.8和17.2 mg/L,而对照菌仅为14.3 mg/L。6-dEB产量提高率分别为49%、37%、30%、28%、24%和20%。弱化leuD、leuC和fadJ基因位点,6-dEB产量分别为16.6、16.4和16.0 mg/L,产量提高为10%-20%。而弱化pfID、paaF和tdcD产量提高率小于10%。因此,大部分弱化靶点能够提高6-dEB异源合成。由于弱化单个基因位点有效,我们推测组合弱化或同时弱化多个基因位点可能进一步提高6-dEB异源合成。
2.3 组合弱化调控基因位点为了验证组合弱化调控能够进一步提高6-dEB异源合成,我们对lsrC与fadB、ackA、pta、ptsH、ptsI进行了组合弱化实验。由图2可知,同时弱化lsrC+ackA、lsrC+fadB、lsrC+ptsI双基因位点,6-dEB产量达到22.8、21.49和 21.47 mg/L,其产量提高率为59.9%、50.4%和50.2%;而弱化lsrC+ptsH和lsrC+pta组合,6-dEB产量为14.08和20.98 mg/L,提高率为46.7%和1.3%。从比产率考察,弱化双基因位点对比产率提高率都在20%以上,其中最高组合为lsrC +ptsI,比产率提高率为54%。与单独弱化lsrC基因位点产量相比,组合弱化lsrC+ackA、lsrC+fadB和lsrC+ptsI能够进一步提高生产菌的6-dEB异源合成能力。

|
| 图2 组合弱化对异源合成6-dEB的影响 Fig.2 Effect of co-repression of the target genes on 6dEB production. (A) 6dEB titer. (B) The increased rate of 6dEB yield and productivity. |
大肠杆菌是目前生产红霉素等聚酮类化合物最具潜力的底盘细胞[9],相关的研究也取得了一系列进展[2, 5, 7, 8, 17, 18, 19, 20, 21, 22]。但仅依靠单一靶点基因的改造或传统的途径工程改造等方法很难获得更高效的红霉素生产菌。随着全基因尺度的代谢模型的发展,基于大肠杆菌全基因尺度调控来改善红霉素异源合成成为可能。E. coli基因组尺度代谢模型[14]和不同算法[23, 24]为挖掘更多的靶点以改善目标产物合成提供了更多的机会。FDCA方法是基于FBA基础上而发展成的通量比较分析方法[9]。本课题组[25]曾利用FDCA方法对大肠杆菌异源合成萜类化合物前体供应进行计算分析和实验验证,得到51个关键基因位点,改造后的菌株成功地使萜类化合物在大肠杆菌中的产量提高174%。其中约20%的靶点是首次发现的新靶点。本研究基于E. coli基因组尺度代谢模型iAF1260进行FDCA分析,成功地预测出提高6-dEB异源合成的靶点基因。证明了这一方法在预测新靶点方面的可靠性和可行性。
本研究通过sRNAs技术弱化改造宿主的染色体基因,成功改善了6-dEB的合成。研究结果表明,弱化一些靶点可显著提高宿主合成6-dEB的能力。其中lsrC (编码LsrABC转运蛋白)、ptsH (编码磷酸烯醇式丙酮酸-糖磷酸转移酶系统HPr蛋白) 和ptsI (编码磷酸烯醇式丙酮酸-糖磷酸转移酶系统PTSI蛋白) 是E. coli中重要的转运蛋白组分[26];而靶点fadB、ackA和pta分别编码脂肪酸氧化反应复合体的α-单元、乙酸激酶和磷酸转乙酰酶。由于这些靶点并不处于6-dEB合成通路之中,其功能似与异源6dEB合成并无直接关联,这些靶点的弱化可能间接影响了6dEB合成相关的前体物质或辅因子供给,其提高6dEB合成的机理有待进一步研究。
sRNAs技术是最近发展的一种细菌基因弱化方法[16],在翻译水平上利用sRNAs与靶基因的mRNA结合,阻遏靶基因的mRNA与核糖体的结合,最终抑制靶基因的蛋白表达。相比基因敲除方法,它可以更加简单地对基因组中各基因进行调控,也可以快速地、较高通量地同时对多个基因靶点进行弱化,这也是这一方法的优越性所在。与in silico的模型预测相结合,这一方法展示出巨大的优越性。Lee课题组[15, 16]通过设计构建表达sRNAs质粒,将sRNA技术应用到酪氨酸和尸胺cadaverine生产中,高通量筛选出有效改造靶点,使酪氨酸产量达到2 g/L以上,cadaverine生产菌产量提高了55%。因此,采用sRNAs技术可以有效的对调控靶点进行表达弱化。随着更多的细菌调控RNA发现和功能解析,相信sRNAs技术在细菌代谢工程和合成生物学上必将有着广泛的应用。
综上所述,本研究基于in silico的方法预测新靶点基因,利用sRNAs技术最终发现和确认了6个有效的调控靶点,成功改善了6-dEB在大肠杆菌中的异源合成。这一策略和方法的成功,也为进一步改善其他代谢产物的异源合成提供了研究思路。
| [1] | Pfeifer BA, Admiraal SJ, Gramajo H, et al. Biosynthesis of complex polyketides in a metabolically engineered strain of E. coli. Science, 2001, 291(5509): 1790-1792. |
| [2] | Zhang H, Wang Y, Wu J, et al. Complete biosynthesis of erythromycin A and designed analogs using E. coli as a heterologous host. Chem Biol, 2010, 17(11): 1232-1240. |
| [3] | Murli S, Kennedy J, Dayem LC, et al. Metabolic engineering of Escherichia coli for improved 6-deoxyerythronolide B production. J Ind Microbiol Biotechnol, 2003, 30(8): 500-509. |
| [4] | Lau J, Tran C, Licari P, et al. Development of a high cell-density fed-batch bioprocess for the heterologous production of 6-deoxyerythronolide B in Escherichia coli. J Biotechnol, 2004, 110(1): 95-103. |
| [5] | Wang Y, Pfeifer BA. 6-Deoxyerythronolide B production through chromosomal localization of the deoxyerythronolide B synthase genes in E. coli. Metab Eng, 2008, 10(1): 33-38. |
| [6] | Wang Y, Wang Y, Chu J, et al. Improved production of erythromycin A by expression of a heterologous gene encoding S-adenosylmethionine synthetase. Appl Microbiol Biotechnol, 2007, 75(4): 837-842. |
| [7] | Wang Y, Boghigian BA, Pfeifer BA. Improving heterologous polyketide production in Escherichia coli by overexpression of an S-adenosylmethionine synthetase gene. Appl Microbiol Biotechnol, 2007, 77(2): 367-373. |
| [8] | Zhang H, Wang Y, Boghigian B, et al. Probing the heterologous metabolism supporting 6-deoxyerythronolide B biosynthesis in Escherichia coli. Microb Biotechnol, 2009, 2(3): 390-394. |
| [9] | Meng H, Lu Z, Wang Y, et al. In silico improvement of heterologous biosynthesis of erythromycin precursor 6-deoxyerythronolide B in Escherichia coli. Biotechnol Bioprocess Eng, 2011, 16(3): 445-456. |
| [10] | Edwards JS, Palsson BO. The Escherichia coli MG1655 in silico metabolic genotype: its definition, characteristics, and capabilities. Proc Natl Acad Sci USA, 2000, 97(10): 5528-5533. |
| [11] | Varmar A, Palsson BO. Metabolic capabilities of Escherichia coli: I. synthesis of biosynthetic precursors and cofactors. J Theor Biol, 1993, 165(4): 477-502. |
| [12] | Varmar A, Palsson BO. Metabolic capabilities of Escherichia coli: 2. optimal-growth patterns. J Theor Biol, 1993, 165(4): 503-522. |
| [13] | Segre D, Vitkup D, Church GM. Analysis of optimality in natural and perturbed metabolic networks. Proc Natl Acad Sci USA, 2002, 99(23): 15112-15117. |
| [14] | Feist AM, Henry CS, Reed JL, et al. A genome-scale metabolic reconstruction for Escherichia coli K-12 MG1655 that accounts for 1260 ORFs and thermodynamic information. Mol Syst Biol, 2007, 3(1): 121. |
| [15] | Na D, Yoo SM, Chung H, et al. Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs. Nat Biotechnol, 2013, 31(2): 170-174. |
| [16] | Yoo SM, Na D, Lee SY. Design and use of synthetic regulatory small RNAs to control gene expression in Escherichia coli. Nat Protoc, 2013, 8(9): 1694-1707. |
| [17] | Pfeifer B, Hu Z, Licari P, et al. Process and metabolic strategies for improved production of Escherichia coli derived 6-deoxyerythronolide B. Appl Environ Microbiol, 2002, 68(7): 3287-3292. |
| [18] | Pistorino M, Pfeifer BA. Efficient experimental design and micro-scale medium enhancement of 6-deoxyerythronolide B production through Escherichia coli. Biotechnol Prog, 2009, 25(5): 1364-1371. |
| [19] | Dayem LC, Carney JR, Santi DV, et al. Metab Eng of a methylmalonyl-CoA Mutase-Epimerase pathway for complex polyketide biosynthesis in Escherichia coli. Biochemistry, 2002, 41(16): 5193-5201. |
| [20] | Zhang H, Boghigian BA, Pfeifer BA. Investigating the role of native propionyl-CoA and methylmalonyl-CoA metabolism on heterologous polyketide production in Escherichia coli. Biotechnol Bioeng, 2010, 105(3): 567-573. |
| [21] | Wang JF, Xiong ZQ, Li SY, et al. Enhancing isoprenoid production through systematically assembling and modulating efflux pumps in Escherichia coli. Appl Microbiol Biotechnol, 2013, 97(18): 8057-8067. |
| [22] | Wang Y, Pfeifer BA. 6-Deoxyerythronolide B production through chromosomal localization of the deoxyerythronolide B synthase genes in E. coli. Metab Eng, 2008, 10(1): 33-38. |
| [23] | Lper H, Jin YS, Moxley J, et al. Identifying gene targets for the Metab Eng of lycopene biosynthesis in Escherichia coli. Metab Eng, 2005, 7(3): 155-164. |
| [24] | Xu P, Ranganathan S, Fowler ZL, et al. Genome-scale metabolic network modeling results in minimal interventions that cooperatively force carbon flux towards malonyl-CoA. Metab Eng, 2011, 13(5): 578-587. |
| [25] | Wang JF, Meng HL, Xiong ZQ, et al. Identification of novel knockout and up-regulated targets for improving isoprenoid production in E. coli. Biotechnol Lett, 2014, 36(5): 1021-1027. |
| [26] | Bologna FP, Bermudez VA, Saavedra DD, et al. Characterization of Escherichia coli EutD: a phosphotransacetylase of the ethanolamine operon. J Microbiol, 2010, 48(5): 629-636. |
 2015, Vol. 31
2015, Vol. 31