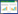服务
文章信息
- 普鸿丽, 吕波, 赵东旭, 李春
- Pu Hongli, Lü Bo, Zhao Zhao, Li Chun
- 重组β-葡萄糖醛酸苷酶键选择性的半理性改造
- Semi-rational modification for improving bond selectivity of recombinant β-glucuronidase
- 生物工程学报, 2015, 31(7): 1119-1128
- Chin J Biotech, 2015, 31(7): 1119-1128
- 10.13345/j.cjb.140595
-
文章历史
- Received: December 2, 2014
- Accepted: January 21, 2015
甘草酸 (Glycyrrhizin,GL) 又称甘草甜素[1],是一种三萜皂苷类化合物,由五环三萜皂苷通过β-1,2糖苷键和β-1,3糖苷键与两个葡萄糖醛酸相连构成。它具有抗炎、抗肿瘤、抗病毒等药理效应,同时作为甜味剂、增香剂、稳定剂广泛应用于化妆品、食品等行业中[2, 3, 4, 5]。但由于其极性较强,溶解性不好,不能有效地穿透细胞膜发挥药效,使其应用受到了很大的限制。与GL的相比,GL衍生物 (单葡萄糖醛酸基甘草次酸(Glycyrrhetinic acid monoglucuronide,GAMG)是一种甜度高、热量低、安全性好的甜味剂,甜 度是甘草酸的5倍多,蔗糖的941倍[6, 7],而且GAMG具有极性适中、溶解性好、易为人体吸收等特性,成为GL最理想的替代品。
β-葡萄糖醛酸苷酶 (EC 3.2.1.31) 是一类能水解含有β-葡萄糖醛酸基的糖苷类化合物,同时释放出β-葡萄糖醛酸 (GlcA) 和相应的苷元的酶,主要分布于糖苷酶水解酶家族1 (GH1)、家族2 (GH2) 和家族79 (GH79),同属于糖苷酶氏簇A (GH-A)[8, 9],目前报道过的不同微生物来源的β-葡萄糖醛酸苷酶性质有所差异,但普遍存在酶表达效率不高、酶活性偏低、稳定性差、存在副反应等特点[10, 11],因此,如何获得可以定向催化GL生成GAMG的β-葡萄糖醛酸苷酶成为甘草酸生物转化的瓶颈。本实验室前期在大肠杆菌原核系统中高效表达了来源于产紫青霉Penicillium purpurogenum Li-3的β-葡萄糖醛酸苷酶 (PGUS)[12, 13],但重组β-葡萄糖醛酸苷酶 (PGUS-E) 的键选择性相对于PGUS发生了改变,在水解GL的β-1,2糖苷键生成产物GAMG的同时亦会水解GAMG的β-1,3糖苷键生成副产物甘草次酸 (GA),造成产物GAMG专一性降低 (图1)。因此,利用蛋白质改造技术研究并改造PGUS-E的键选择性及产物专一性使其更好地适应GAMG的工业化生产具有重要的应用价值。相比较非理性设计中突变文库大、筛选难度大等缺点[14, 15],通过蛋白结构模拟和分析研究,将随机突变限制在特定的区域上的半理性设计,可在较小的突变库中获得性质改善的突变体[16],近年来被广泛的应用于蛋白的定向改造。
|
| 图1 β-葡萄糖醛酸苷酶 (PGUS-E) 催化甘草酸示意图 Fig.1 Biotransformation of GL catalyzed by β-glucuronidase (PGUS-E) |
本文以利用Pymol软件对PGUS-E酶的模拟结构进行分析,理性预测活性中心氨基酸位点对蛋白质的结构和功能的关系的影响,最终选定3个突变位点R329、T369和N467进行饱和突变,产生多样化突变酶,通过薄层层析结合高效液相色谱筛选方法获得键选择性提高的突变酶R329K和T369V。对结果进行分析讨论,
阐述了PGUS-E 酶的结构和功能的关系,为酶分子的蛋白质工程改造提供理论基础和试验依据,也为后续酶分子修饰及相关研究提供性质更为优良的酶源。
1 材料与方法 1.1 材料1) 质粒和菌株: 克隆及表达宿主Escherichia coli TOP10由北京理工大学生命学院生物转化与微生态课题组保存。含有目标基因pgus (GenBank登录号:EU095019) 的组成型热诱导质粒pHCE-gus由本实验室构建。
2) 试剂:PCR反应相关试剂Fastpfu DNA 聚合酶、dNTPs等购自购自TaKaRa公司;质粒DNA小量提取试剂盒购自北京博迈德科技发展有限公司;DpnⅠ内切酶购自Fermentas公司;DNA marker购自宝生物工程 (大连) 有限公司;本实验所用其他化学试剂均为分析纯;PCR引物合成以及DNA测序由上海生工生物工程技术服务有限公司完成。
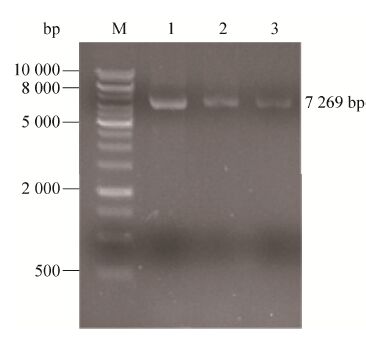
1.2 饱和突变法构建突变体文库根据Statagene公司Quit-Change Kit原理,针对R329、T369、N467三位点设计了6条简并引物 (表1)。以质粒pHCE-gus为模板,扩增出含有突变的质粒。其中PCR的反应体系组成为: 10×Fast Pfu PCR缓冲液2.5 μL,4种 dNTPs混合溶液2 μL,上下游引物各1 μL,pHCE-gus模板DNA 1 μL,5 U/μL的FastPfu DNA聚合酶及无菌水适量,反应总体积为25 μL。PCR反应条件:95 ℃预变性2 min;95 ℃变性30 s,65 ℃退火30 s,72 ℃延伸 3 min 50 s,25个循环;72 ℃下延伸10 min。1% 的琼脂糖凝胶电泳检测PCR反应产物,目标条带为7 269 bp。PCR产物中加入DpnⅠ限制性内切酶,在37 ℃酶解3 h以彻底消化甲基化原始模板,酶解产物采用热击法转化E. coli Top10感受态细胞,转化液涂布于LB平板上 (含有100 μg/mL氨苄青霉素),得到饱和突变文库。
| Primer name | Primer sequence (5'−3') |
| 329-F | GGGGGCCAACTCCTTCNNKACCTCACACTACCCATACG |
| 329-R | GAAGGAGTTGGCCCCCATCCAGTGCAGCAG |
| 369-F | CGGAGCGGGAGC CCAGNNKTC AACCCGCCGGCTACATTC |
| 369-R | CTGGGCTCCCGCTCCGATGGAGAATGCTAGACCG |
| 467-F | GATGTGCTTTGCTTANNKCGGTATTTTGGTTGGTACACGC |
| 467-R | GAGCTCAGCCGTTTGCGTGTACCAACCAAAATACCG |
将LB固体培养板上的单菌落逐个挑到每孔含有1 mL LB液体培养基 (含100 μg/mL氨苄青霉素;0.5 g/L底物GL) 的Ep管中,30 ℃、200 r/min转速的摇床中培养表达24 h后迅速转至40 ℃,热诱导裂解细胞释放PGUS-E到培养基中,与培养基中底物GL反应24 h后,加10 μL的NaOH (1 mol/L) 终止反应。反应液中的底物GL和产物GAMG,GA通过TLC法进行定性初筛,判断标准如下:在GL转化率相同的条件下 (≥90%),GAMG的积累量越高,则酶的键选择性越高,对GAMG积累量高于PGUS-E酶的优势突变酶,用HPLC进一步进行定量复筛。
1.4 PGUS-E酶液制备及活性分析将获得的突变株接种于新鲜的LB培养基 (含100 μg/mL氨苄青霉素) 中30 ℃培养过夜,1% 接种量转接于200 mL LB培养基,220 r/min 培养16 h后,将发酵液冷冻离心 (4 ℃,5 000 r/min) 10 min,用醋酸缓冲液洗涤,重悬细胞,在冰浴中超声破碎细胞 (300 W,工作2 s,间歇2 s,全程工作时间20 min)。破碎液于 12 000 r/min离心20 min收集上清,即为粗酶液。采用镍亲和层析对所得的粗酶液进行分离纯化。首先用pH 8.0的 Tris-HCl的缓冲液平衡镍柱,通过上样将粗酶液装载到镍柱上,然后清洗杂蛋白将基线A280 降至最低并冲平。最后分别用50和200 mmol/L的咪唑溶液梯度洗脱目的蛋白,最后将洗脱液透析除盐即得到纯酶。
取100 μL酶液加200 μL含有1 g/L甘草酸 (pH=5.0的醋酸-醋酸钠缓冲液),40 ℃反应 60 min后,添加 10 μL NaOH (1 mol/L) 终止反应。取100 μL反应液添加900 μL甲醇,混合均匀,用HPLC检测GL、GAMG及GA的含量。β-葡萄糖醛酸苷酶的活力定义为:在上述条件下每分钟消耗1 nmol甘草酸所需要的β-葡萄糖醛酸苷酶量为一个活力单位 (U)。
1.5 最适温度和 pH的测定按上述方法测定突变酶在不同pH (3.0−8.0) 和温度 (25−60 ℃) 下的酶活力,原始酶与突变酶分别设定最高酶活为100%,以相对酶活力和 pH,温度关系作图,确定酶的最适pH和温度。
1.6 PGUS-E突变酶的键选择分析采用高效液相色谱 (HPLC) 对反应混合物中底物GL及产物GAMG、GA进行定量分析。仪器主要参数:岛津LC-10A,色谱柱:Shim-pack,VP-ODS (150 mm,4.6 mm),检测器:SPD,检测波长:254 nm,流动相:甲醇:0.6%醋酸=81:19,进样量:10 μL,流速:1 mL/min,柱温箱:40 ℃,工作站:LC solution。GL、GAMG、GA 的保留时间分别为5.2 min、11.7 min、 21.3 min。
数据处理计算公式如下:
转化率 (CGL) =(SO-ST)/ SO×100%;
化学键选择性(Scb)=PGAMG/(PGAMG+PGA) × 100%。
SO、ST分别代表底物GL的初始浓度和终浓度;PGAMG、PGA 分别代表产物GAMG和GA的终浓度。
1.7 PGUS-E酶和突变酶的空间结构模拟PGUS-E酶的三维结构通过SWISS-MODEL 蛋白模拟在线服务器 (http://www.expasy.ch/ swissmod/SWISS-MODEL.html) 进行同源模拟获得,模板为E. coli来源的β-葡萄糖醛酸苷酶 (EGUS) 晶体结构 (PDB登录号:3K46),氨基酸序列比对结果显示相似度达到54.34%。采用 Pymol软件理性分析和预测了活性中心氨基酸对PGUS-E键选择性的影响,选取了突变位点。并且应用该软件虚拟氨基酸突变,通过分析突变前后氨基酸极性、疏水作用、氢键等的变化,分析结构-键选择性关系。阐述了PGUS-E的结构和功能的关系,为进一步的酶特性改造提供理论基础。
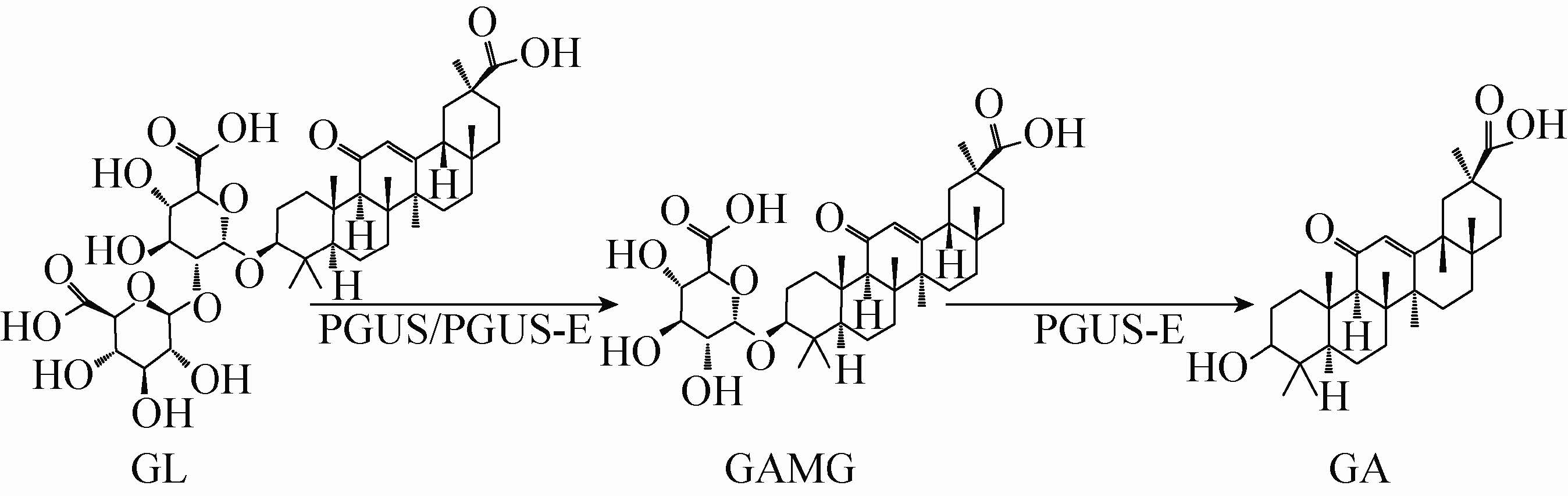
2 结果与讨论 2.1 PGUS-E酶键选择性特异位点分析PGUS-E 酶同源模拟获得的理论结构说明它包含一个 (α/β)8 桶状催化结构域,属于(α/β)8 水解酶亚组。(α/β)8类酶的活性部位均处于一个非常相似的位置,即位于连接 β折叠链C端与α螺旋N端的环构成的漏斗形口袋底部,参与底物结合和催化作用氨基酸在这个口袋中[17, 18]。PGUS-E的糖苷底物分子GL和GAMG的糖基部分均为葡萄糖醛酸,配基则不同 (图2),说明PGUS-E对GL和GAMG的末端糖苷键的糖基识别较强,对配基结构识别性较低,可能导致键选择性低。由此做出以下假设:糖苷键两端结构识别决定酶的键选择性,通过与GL末端糖苷键的配基结构识别相关的氨基酸突变可以提高PGUS-E酶键选择性,最终使GAMG的产量提高。
|
| 图2 PGUS-E酶底物分子的结构示意图 Fig.2 Substrate schematic representationof PGUS-E. GL (left) GAMG (right) |
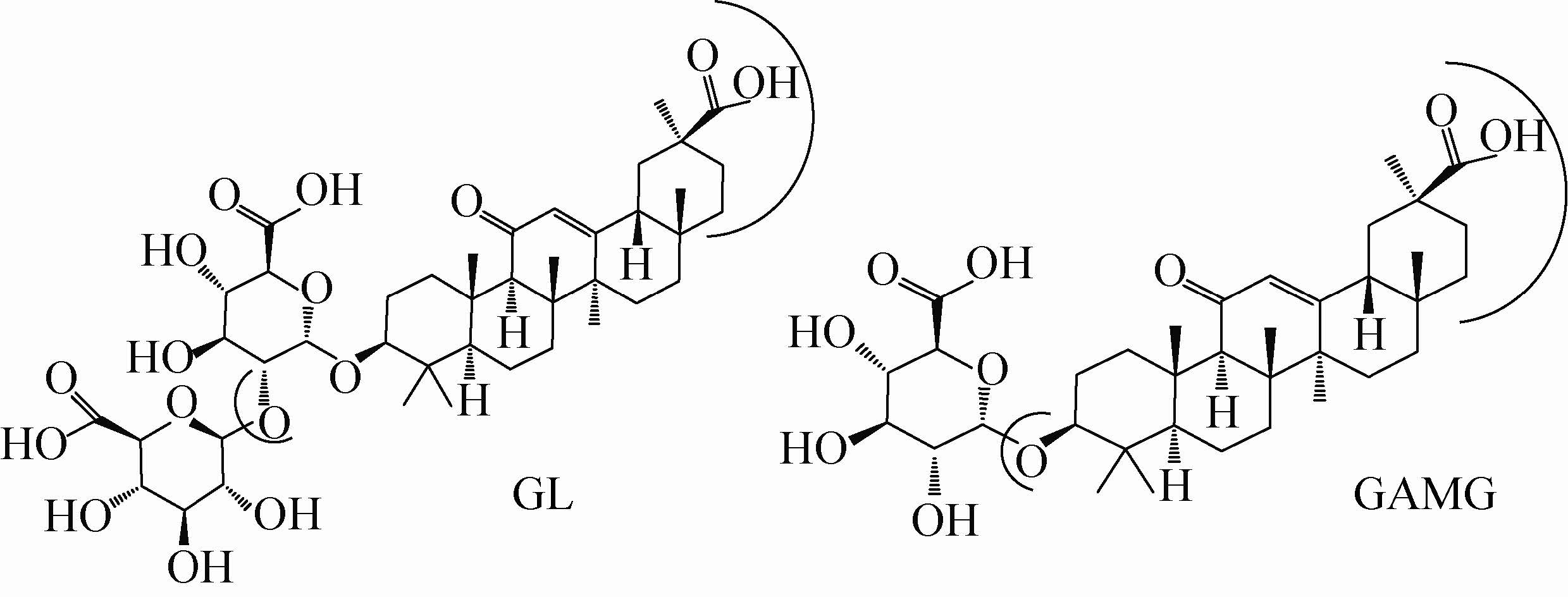
大肠杆菌的β-葡萄糖醛酸苷酶 (EGUS-E) 与PGUS-E同属于GH2,其结构与功能关系为 PGUS-E的研究提供了有利参考。利用Pymol软件对PGUS-E的模拟结构与EGUS-E及葡萄糖醛酸复合物的晶体结构 (PDB登录号:3K4D) 进行比对,分析发现PGUS-E酶三维结构中Arg329、His332、Thr369、Asp414、Asn467、Asp505、Trp550、Arg563、Lys567、Asn569 (图3) 位于底物结合口袋中,说明这些氨基酸与底物分子的糖基或者配基识别结合相关。依据文献报道,β-葡萄糖醛酸苷酶中,Asp414、Asp505为催化残基[19],参与糖苷键的断裂,His332、Arg563、Lys567、Asn569位于糖基结合位点,与糖基识别相关[20, 21]。在PGUS-E酶结构中显示,Trp550位于底物结合口袋的底部,是强疏水性氨基酸,保证催化反应的疏水环境,Arg329、Thr369、Asn467位于糖基结合位点的对立面,可能与底物分子配基部分的识别相关。综上分析,为了确保选择位点的准确性,最终选定与配基识别相关的3个位点 (Arg329、Thr369、Asn467) 进行饱和突变研究,以提高PGUS-E酶键的选择性。
|
| 图3 PGUS-E (绿 ) 与 EGUS复合物 (3K4D粉橙色 ) 的结构比对示意图 Fig.3 Stereo view of the superimposition of the PGUS-E (green) and EGUS (3K4D,salmon). Substrate binding residues are depicted as stick |
利用带有简并密码子的引物,以pHCE-gus 质粒为模板,通过PCR扩增对R329、T369、N467进行饱和突变,PCR扩增质粒结果如图4所示。将PCR产物经过DpnⅠ酶切消化转化 E. coli TOP10中,构建了R329、T369、N467的饱和突变文库。通过TLC定性初筛和HPLC定量复筛相结合的方法对突变文库筛选,通过基因测序及序列分析获得了优势突变酶R329K和T369V (图5)。HPLC检测结果计算获得,原始的PGUS-E的转化率达到90.1%时,键选择性达到8.5%,T369V的转化率达到90.3%时,键选择性达到42.8%,与PGUS-E比较提升了34.3%;R329K的转化率达到90.8%,键选择性达到35.5%,与PGUS-E比较提升了26.9%。但 N467位点饱和突变获得的20种突变酶键选择性并没有提高,且催化GL的酶活力基本丧失。另外,对得到的R329K、T369V优势突变进行组合,发现突变酶M3 (R329 L/T369V) 的键选择性并没有进一步与提高。
|
| 图4 饱和突变 PCR扩增 DNA电泳图 Fig.4 DNA electrophoresis of saturation mutagenesis reactions. M: DNA marker; 1−3: PCR products corresponding to R329,T369 and N467 |

|
| 图5 PGUS-E突变酶筛选结果 Fig.5 Screening results of mutant PGUS-E. (A) The primary screening of TLC. 1−5: corresponding to T369V,PGUS-E,GL,R329K,GAMG. (B) The re-screening of HPLC |
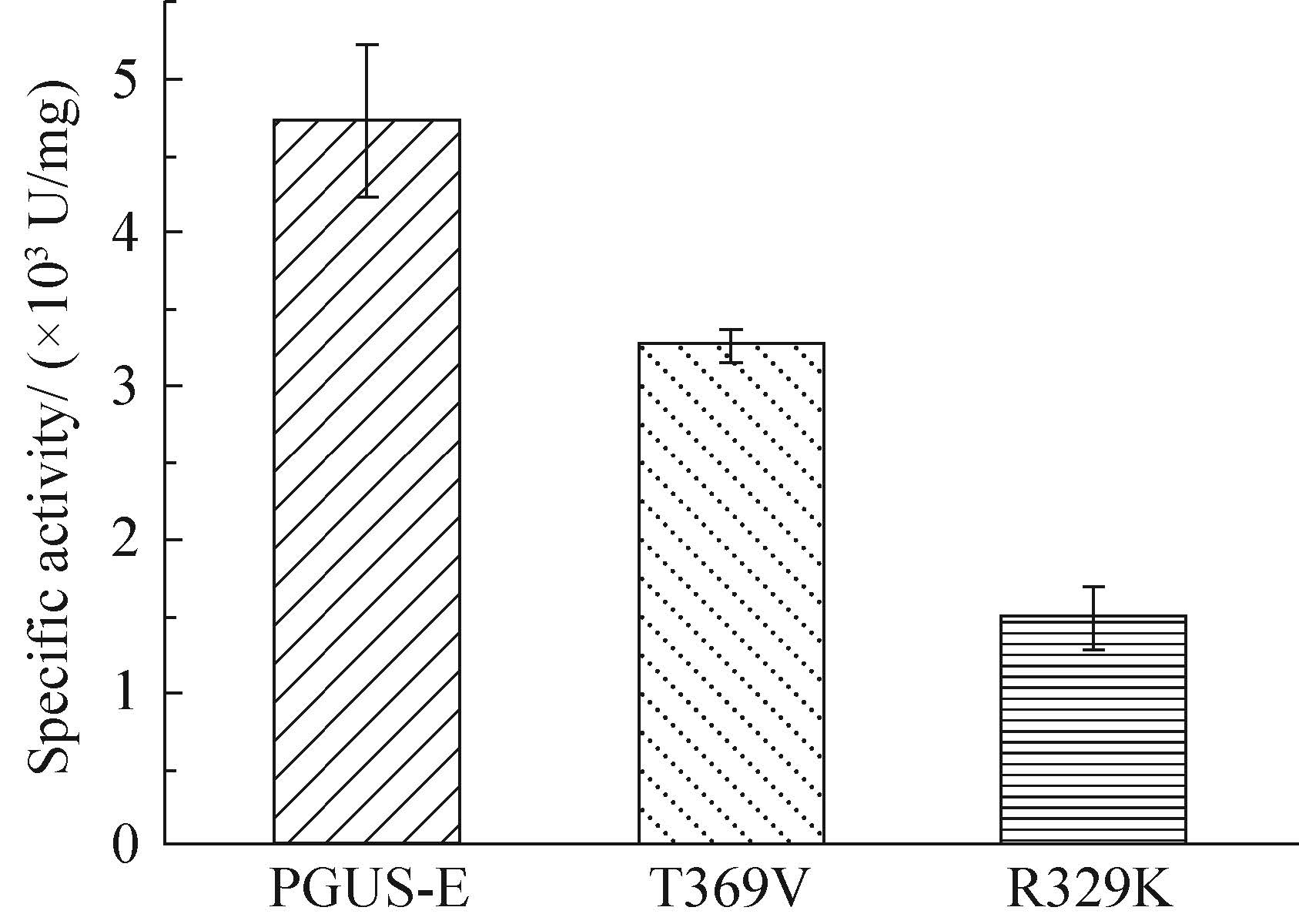
对PGUS-E突变酶的比酶活进行分析,发现与PGUS-E比较,突变酶R329K、T369V催化GL水解能力下降。如图6所示,突变酶 T369V (3273 U/mg) 的比酶活为PGUS-E (4726 U/mg) 的69.3%,突变酶R329K (1492 U/mg) 比酶活仅为PGUS-E的31.6%。
|
| 图6 PGUS-E酶及突变酶的比酶活 Fig.6 Specific activity of PGUS-E and variants |
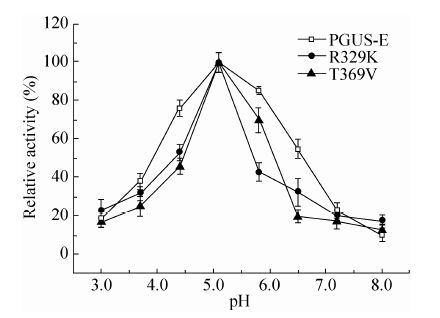
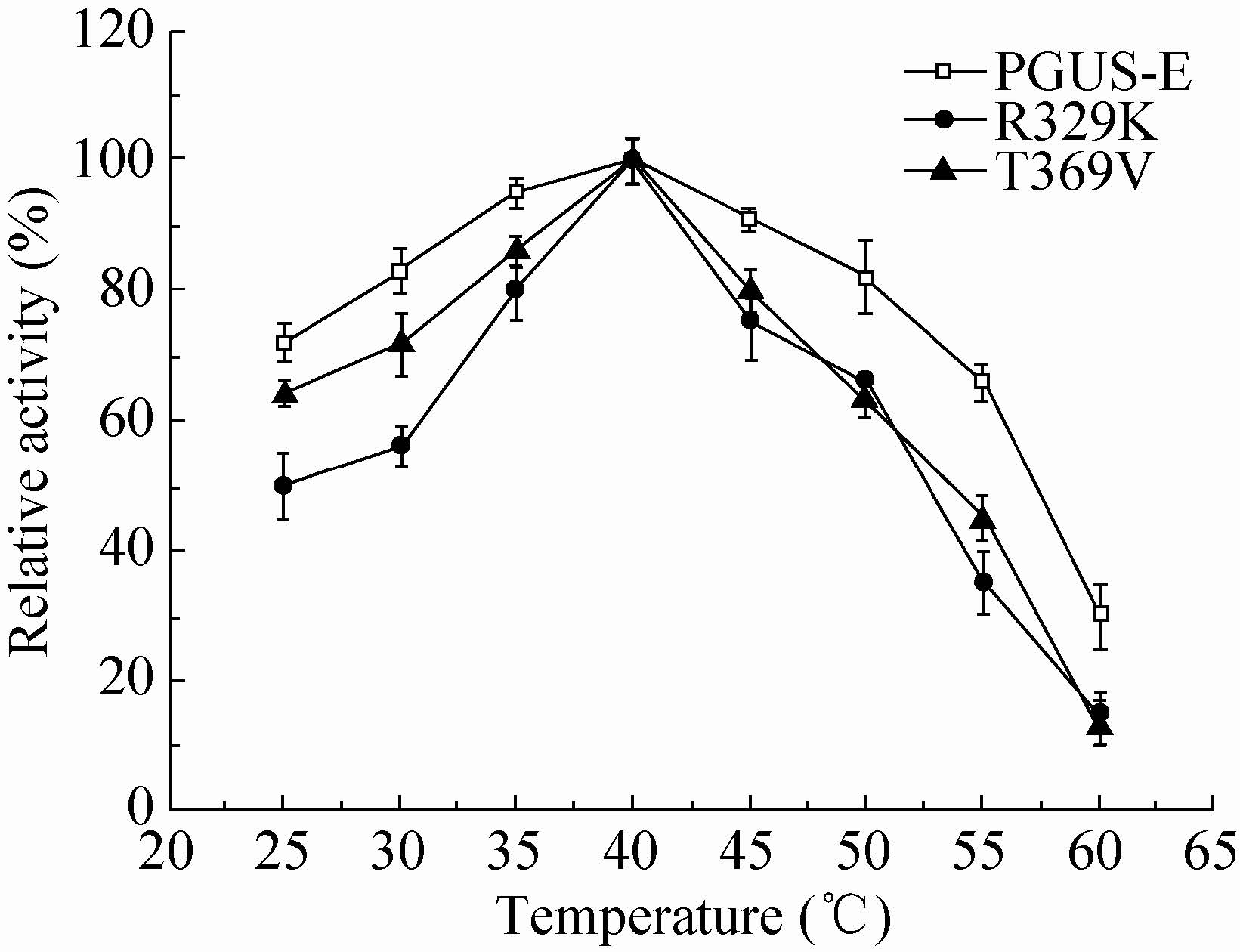
为了确定酶反应的最佳条件,考察了突变酶催化GL的最适pH和温度,结果如图7、图8所示,突变酶T369V、R329K与PGUS-E的最适pH为5.0,最适温度为40 ℃,说明突变氨基酸的引入并没有影响酶的最适pH和最适 温度。
|
| 图7 pH对 PGUS-E及突变酶活力的影响 Fig.7 Effect of pH on the PGUS-E and variants |
|
| 图8 温度对 PGUS-E及突变酶活力的影响 Fig.8 Effect of temperature on the PGUS-E and variants |
为了进一步确定突变酶的作用机制,采用 Autodock软件对酶与底物GL进行对接模拟。图9A−D依此为PGUS-E和T369V、R329K、N467D突变酶。

|
| 图9 PGUS-E和突变酶与 GL作用的模拟结构图 Fig.9 The model structure of PGUS-E and mutants with a GL substrate. (A) PGUS-E. (B) T369V; (C) R329K; (D) N467D |
图9A和图9B比对显示,突变酶T369V的第369位氨基酸由亲水性苏氨酸突变为疏水性缬氨酸,极性变化影响369位氨基酸所在的整个Loop环构象朝向疏水中心移动,增加了酶与GL的契合度,进而增强了酶的键选择性。但是酶与底物GL之间的范德华力的增强,使得催化反应中结构重排的自由能垒变大,影响了酶活性残基的释放,减缓了新一轮底物进入活性中心速率,最终降低了酶催化效率。
图9A和图9C比对显示,R329与E353和T504之间各存在一个氢键的相互作用。突变酶 R329K的第329位氨基酸由精氨酸变为赖氨酸,两者具有相似的极性及电荷性质,但侧链变短,破坏了与E353和T504之间的氢键,引起 E505 位谷氨酸的位移,从而提高了酶的键选择性。但是突变增加了催化位点与GL之间的距离,最终影响其催化效率。
图9A和图9D比对显示,突变前N467分别与 E505 (催化残基) 及T504之间形成一个氢键,且与两者之间的距离很近,分别为1.3 Å和2.1 Å。故微小的变化影响催化残基E505。这里以Asn长度和性质变化最小的Asp进行突变模拟,PGUS-E酶第467位天冬酰胺突变后,破坏了N467与E505之间的氢键,催化残基E505与糖苷键结合构象发生变化,严重影响PGUS-E的催化效率。结果分析表明N467对维持酶功能有重要作用。
3 结论本实验通过SWISS-MODEL软件模拟了PGUS-E酶的三维结构,通结构比对,利用Pymol软件分析了底物分子与酶之间的相互作用,初步确定影响其键选择性的部分位点。通过构建饱和突变文库的研究,发现R329K和T369V突变使PGUS-E酶的键选择性分别提高了26.9%,34.3%,突变酶催化GL生成的GAMG的产率和底物特异性增加。通过突变酶与底物GL作用的结构模拟,进一步分析了R329K和T369V键选择性提高的机理,为进一步键选择性改善提供了理论依据。
本研究对于PGUS-E酶的结构和功能关系的分析和预测进行了验证和完善,为进一步阐明PGUS-E酶的键选择性与结构关系奠定了基础,同时为构建具有理想键选择性和产物特异性的PGUS-E酶突变体提供了新的出发点。
| [1] | Hennell JR, Lee S, Khoo CS, et al. The determination of glycyrrhizic acid in Glycyrrhiza uralensis Fisch. ex DC. (Zhi Gan Cao) root and the dried aqueous extract by LC-DAD. J Pharm Biomed Anal, 2008, 47(3): 494-500. |
| [2] | Liu Y, Huangfu J, Qi F, et al. Effects of a non-conservative sequence on the properties of β-glucuronidase from Aspergillus terreus Li-20. PLoS ONE, 2012, 7(2): 309-318. |
| [3] | Cinatl J, Morgenstern B, Bauer G, et al. Glycyrrhizin, an active component of liquorice roots and replication of SARS-associated coronavirus. Lancet, 2003, 361(9374): 2045-2046. |
| [4] | Lu L, Zhao Y, Yu H, et al. Preparation of glycyrrhetinic acid monoglucuronide by selective hydrolysis of glycyrrhizic acid via biotransformation. Chin Herbal Med, 2012, 4(4): 324-328. |
| [5] |
Chen GL, Zhao QY, Duan RZ. Study selective hydrolysis of glycyrrhizin and the molecular complex of glycyrrhiin and sialic acid. J Lanzhou Univ, 2012, 38(1): 29-34 (in Chinese). 陈桂玲, 赵全义, 段瑞哲. 甘草酸的选择水解及其与唾液酸形成分子复合物的初步研究. 兰州大学学报, 2012, 38(1): 29-34. |
| [6] | Mizutani K, Kambara T, Masuda H, et al. Glycyrrhetic acid monogluronide (MGGR): biological activities. Int Congr Ser, 1998, 43(2): 225-235. |
| [7] | Mizutani K, Kuramoto T, Tamura Y, et al. Sweetness of glycyrrhetic acid 3-O-beta -D-monoglucuronide and the related glycosides. Biosci Biotechol Biochem, 1994, 58(3): 554-555. |
| [8] | Henrissat B, Bairoch A. Updating the sequence-based classification of glycosyl hydrolases. Biochem J, 1996, 316(Pt 2): 695-696. |
| [9] | Geddie ML, Matsumura I. Rapid evolution of β-glucuronidase specificity by saturation mutagenesis of an active site loop. J Bio Chem, 2004, 279(25): 26462-26468. |
| [10] | Akao T, Hattori M, Kobashi K, et al. Hydrolysis of glycyrrhizin to glycyrrhetyl monoglucuronide by lysosomal β-D-glucuronidase of animal livers. Biochem Pharmacol, 1991, 41(6): 1025-1029. |
| [11] | Akao T. Hasty effect on the metabolism of glycyrrhizin by Eubacterium sp. GLH with Ruminococcus sp. PO1-3 and Clostridium innocuum ES24-06 of human intestinal bacteria. Bio Pharm Bull, 2000, 23(1): 6-11. |
| [12] | Feng S, Li C, Xu X, et al. Screening strains for directed biosynthesis of β-D-mono-glucuronide- glycyr rhizin and kinetics of enzyme production. J Mol Catal B Enzym, 2006, 43(1): 63-67. |
| [13] | Song ZK, Wang XY, Li C, et al. Cloning and prokaryotic expression of β-glucuronidase from Penicillium purpurogenum Li-3. J Chem Ind Eng, 2008, 59(12): 3101-3106. |
| [14] |
Xiao ZZ, Qu YB, Wang TH, et al. Advance in study on directed evolution of proteins. Prog Biotech, 2001, 21(6): 31-33 (in Chinese). 肖志壮, 曲音波, 汪天虹, 等. 蛋白质定向进化的研究进展. 生物工程进展, 2001, 21(6): 31-33. |
| [15] | Chen K, Arnold FH. Tuning the activity of an enzyme for unusual environments: sequential random mutagenesis of subtilisin E for catalysis in dimethylformamide. Proc Natl Acad Sci USA, 1993, 90(12): 5618-5622. |
| [16] | Reetz MT, Carballeira JD. Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes. Nat Protoc, 2007, 2(4): 891-903. |
| [17] | Gerlt JA, Raushel FM. Evolution of function in (α/β)8-barrel enzymes. Curr Opin Chem Biol, 2003, 7(2): 252-264. |
| [18] | Bourne Y, Henrissat B. Glycoside hydrolases and glycosyltransferases: families and functional module. Curr Opin Struc Biol, 2001, 11(5): 593-600. |
| [19] | Arul L, Benita G, Balasubramanian P. Functional insight for β-glucuronidase in Escherichia coli and Staphylococcus sp. RLH1. Bioinformation, 2008, 2(8): 339-343. |
| [20] | Michikawa M, Ichinose H, Momma M, et al. Structural and biochemical characterization of glycoside hydrolase family 79-glucuronidase from Acidobacterium capsulatum. J Biol Chem, 2012, 287(17): 14069-14077. |
| [21] | Geddie ML, Matsumura I. Rapid evolution of β-glucuronidase specificity by saturation mutagenesis of an active site loop. J Biol Chem, 2004, 279(25): 26462-26468. |
 2015, Vol. 31
2015, Vol. 31