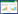服务
文章信息
- 王心哲, 张光亚
- Xinzhe Wang, Guangya Zhang
- 蛋白质环化的研究进展
- Advances in protein cyclization
- 生物工程学报, 2016, 32(4): 430-439
- Chin J Biotech, 2016, 32(4): 430-439
- 10.13345/j.cjb.150284
-
文章历史
- Received: June 19, 2015
- Accepted: October 26, 2015
众所周知,蛋白质 (酶) 在生物体内等温和条件下能够较好地发挥其生理作用。但随着工业生产中酶的广泛应用,酶在工业条件 (如高压、高温、高机械强度、金属离子、有机溶剂) 下,极易发生构象改变从而失去活性的缺点也逐渐暴露。如何构建既具有稳定结构又具有较高活性的酶成为研究者关注的焦点。较早前有研究者发现了大量天然合成的环肽[1-2]。这种环化结构使得天然环肽类物质具有较高的热稳定性和结构稳定性,同时具有抗水解、在化学变性剂条件下能保持完整生物活性的特性[3]。随着蛋白质构效关系研究的深入,环肽结构的稳定性为线性蛋白的改造以提高其稳定性的研究奠定了基础。有研究者建立了研究其序列结构与功能间关系的数据库CyBase[4],蛋白质环化也随之成为了以分子生物手段改造酶结构的热点之一。
蛋白质稳定性与蛋白质结构中的折叠状态 (Folded state) 息息相关,研究表明,环化能够降低蛋白折叠域与非折叠域的构象熵,进而增加蛋白质稳定性,同时可增加蛋白质的受体亲和力,其功能和活性几乎不受影响[5-7]。蛋白质骨架环化是一种将线性肽的C-端和N-端通过酰胺键进行首尾环合形成环状分子的过程。随着对分子生物学及蛋白质工程学的深入研究,已逐步建立起多种经生物法获得环状蛋白的方法,其中较为常用的方法包括蛋白质反式剪接 (PTS)、内含肽介导的蛋白表达连接 (EPL)、蛋白酶催化的转肽作用等[8]。
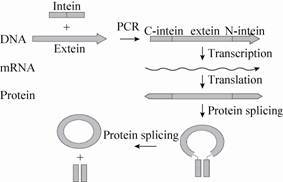
1 内含肽介导的蛋白质剪接作用 (Protein trans-splicing,PTS)内含肽是一种介导蛋白质剪接的作用元件,蛋白质内含肽 (Protein-intein) 是指前体蛋白中的一段插入序列。根据自身结构特征可将其分为3类:大型内含肽 (Maxi-inteins)、小型内含肽 (Minimal or mini-inteins) 和反式剪接内含肽 (Trans-splicing inteins)。内含肽既能够作用于氨基末端也能作用于羧基末端,它在蛋白质翻译后的成熟过程中能够进行自我催化,使自身从前体蛋白中切除,并将其两侧的多肽片段以肽键进行连接,形成成熟的蛋白质[9],其连接过程见图 1[10]。蛋白质剪接 (Protein splicing)是由蛋白质内含子介导的在蛋白质水平上翻译后的加工过程。内含肽在N-末端和C-末端剪接位点催化高度协调的反应。该环化过程是一种分子内程序,不需要其他蛋白和任何ATP等新陈代谢能源类物质。
蛋白质剪接的典型机制包括4个步骤:1) N-S或N-O酰基重排代替上游带有酯键的肽键;2) N-外显肽发生酰基转移作用,重新定位到下游剪接位点,形成结合N-外显肽的C-外显肽酯键;3) 内含肽C-末端的天冬酰胺残基打破内含肽和C-外显肽之间的键进行环化;4) S-N或O-N酰基通过在下游剪接位点重排,通过肽键替换连接N-外显肽的C-外显肽之间的酯键[11-13]。在外显肽的全部氨基酸残基中,只有与C-末端外显肽相连的第一个残基参与整个剪接过程[14-15]。
Gottfried[16]研究小组对以内含肽介导的末端环化的DnaB与天然线性的DnaB进行一系列蛋白结构、蛋白折叠率、构象熵等参数的测定和比较。结果表明,与线性蛋白质相比较,环化蛋白质在变性条件下具有更强的稳定性,并能够通过环化来降低蛋白质的解折叠率及蛋白解折叠域的构象熵以增加其稳定性。环化后的蛋白质其解链温度也相应地有所提升。蛋白质骨架环化带来的构象熵稳定性可以显著提高蛋白稳定性,蛋白解链温度的提升则取决于野生型蛋白非折叠域熵的大小以及蛋白质解折叠时的热容改变量,解链温度的提升程度与该野生酶解折叠域熵的大小成反比。经表征,环化的蛋白质同时具有抗氨基和羧肽酶活[17-18]。
随着对内含肽的深入研究,陆续有研究者发现了受光、温度、pH、氧化还原状态等外部因素调节控制的内含肽。最近,加州大学Huiwang Ai实验小组[19]将光控的半胱氨酸残基用基因方法引入到念珠藻DnaE结构域中,再将这种光激发的内含肽插入到RFP和人酪氨酸激酶中,形成一种不活跃的嵌合蛋白。经长波 (约为250-370 nm) 紫外光照射,发生光引发的光化学反应,可以激发内含肽,继而引发蛋白剪接。这种由基因编码的光控内含肽是一种对蛋白质初级结构和功能研究有效的技术途径。
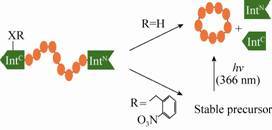
Henning D. Mootz研究团队[20]也通过对蛋白骨架结构和侧链的人工合成构建了一种光激发型内含肽,这种新型由基因编码的光依赖型内含肽通过在其结构内非催化结合位点引入邻硝基苄基酪氨酸(ONBY)——一种酪氨酸衍生物来获得。该带有“光控开关”的光敏感型内含肽ONBY75-intein在波长为366 nm的紫外光照射4 min后即可诱发蛋白剪接作用 的发生,继续照射即可达到约70%的反应率,并可通过His6标记来分离纯化内含肽前体。其分离过程见图 2,在第一次实现了对多肽环化过程中内含肽前体纯化的同时,也实现了从成分复杂的细胞提取物中有效分离纯化目标多肽的目标。实验结果表明,此类由光催化的内含肽引发的蛋白剪接作用既可在大肠杆菌也可在哺乳动物中发生,为其在生物体内的应用奠定了基础[21-22]。
内含肽对蛋白的剪接作用通常是一种不可控的过程,但这种新型的光诱导的内含肽剪接作用具有时间和空间上的灵活性,将为环化提供可以根据需要来进行反应的有利条件。同时,光激活内含肽对蛋白质的结构信息和生化过程没有过多的限制,可以直接从氨基酸序列水平对蛋白质进行调控,实现了对完全由基因编码蛋白质的诱导。这一突破性成果将给蛋白质初级结构和功能的研究带来新的希望[19, 23]。尽管如此,目前研究表明,还没有发现任何一种能够在所有的位点结合序列中均能起到催化PTS作用过程的内含肽。由于PTS在作用过程中通常需要在结合位点附近存在几个或多个突变结合部位,这一特性制约了PTS方法的通用性。若在PTS作用过程中能够找到有特定序列的内含肽,或在反应过程中对内含肽序列的位点选择具有更大的随机性的情况下,均能实现PTS在多种结合位点的有效连接,将使PTS的应用具有更广泛的前景[9]。
2 表达蛋白连接 (Expressed protein ligation,EPL)EPL是一种以内含肽为基础的化学选择性蛋白质半合成方法,最早被报道于1998年,该方法通过改变裂解条件以及对内含肽进行适当修饰,利用蛋白剪接机制生成目标蛋白重组体α-硫酯衍生物,再将N-末端自由的半胱氨酸残基与C-末端α-硫酯片段进行连接,这些片段可进行共价修饰,在硫酯和半胱氨酸之间形成肽键,从而将两种蛋白质连接起来用于氨基酸位点特异性多肽的制备。后续也有研究者从温度、pH、反应时间、反应结构域比率等因素对EPL反应发生的条件进行探索。实验证明,在反应域比率较低情况下,适当添加少量尿素有助于提高连接效率,反应域比率较高的情况下,连接效率自发提高,添加尿素失去了优势作用[24]。EPL可作为连接蛋白产物的同位素标签,被广泛应用于生物物理学探针特异性位点的引入、非天然氨基酸的合成以及翻译后修饰途径[25-28]。不仅如此,通过EPL还实现了对金属蛋白中单个氨基酸功能的全面分析和蛋白质功能的完善,这些金属蛋白包含用来接受高度特异性金属离子的结合位点,借以维持正常的生理功能。同时,可通过EPL法改变金属离子的配位层 (Coordination sphere),以微调所需蛋白突变体的性质[27]。
EPL相对于NCL (天然化学连接) 具有化学选择性、区域选择性以及合成的特异性。EPL合成法克服了DNA重组和异源蛋白表达等方法对蛋白大小和氨基酸数量的限制,将小型蛋白或片段进行聚合,可以实现结合非编码氨基酸的目标,可更高效地用于研究蛋白质结构和功能的关系[25, 29]。目前,EPL已经逐渐发展成为一种较为稳定的化学连接方法,但在研究开展前期,由于在蛋白表达阶段细胞内就会发生目标蛋白与内含肽间的自剪接作用,以及通过EPL方法得到最终蛋白连接产物步骤复杂,使影响蛋白产率的因素增多,以致目标蛋白表达量较低[30]。研究表明较高的α-硫酯衍生物产率是得到高蛋白连接率的关键。Zhao等[27]通过对人载脂蛋白 (apoE) 的研究,使aopE以三联结构 (Triple-labeled) 进行连接反应,实现了对连接程序的简化和连接率的提升。
3 转肽酶A (Sortase A,SrtA) 催化的转肽作用1999 年,Mazmanian等[31]首次对分离自金黄色葡萄球菌的SrtA进行了定义,SrtA是一种来源于革兰氏阳性菌,能够催化酰胺键合成的,能够将细胞表面蛋白共价结合到细菌细胞壁的转肽酶类。其在不同的细菌中具有不同的识别序列 (Motif),如在金黄色葡萄球菌中可识别LPXTG序列,在酿脓链球菌中则能够有效识别LPXTG/LPXTAA序列。SrtA可对LPXTG序列间的Thr和Gly进行切割,形成酰基酶中间化合物,进而与N-末端Gly残基进行结合形成酰胺键。SrtA具有作用条件温和,能够与细胞表面结合的特点,在蛋白质连接方面应用潜力巨大[32]。
2004 年,Mao等研究人员首次成功应用SrtA实现蛋白连接。Proft将重组C-末端带有LPETG-His6标签的绿色荧光蛋白 (GFP) 与多肽进行连接,并证实转肽酶能够催化天然多肽、非天然多肽及非多肽分子的连接。反应时间为 6 h,连接效率约为50%,反应时间大于24 h后连接效率达90%以上[33]。
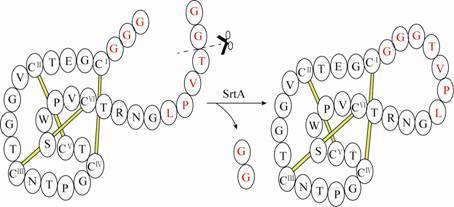
富硫环肽类物质具有酶稳定性、热稳定、抗化学攻击等性能,因此在药物设计方面具有较为广泛应用。Craik团队[34]利用SrtA成功环化了多种小型富硫化物多肽,如Kalata B1 (图 3)、α-conotoxin Vc1.1 (α-芋螺毒素) 以及SFTI-1 (向日葵胰蛋白酶抑制剂1) 等至少包含一个二硫键的多肽。其中SFTI-1是已知最小、最有效的蛋白酶抑制剂。研究表明,这种利用SrtA催化的环化反应对目标蛋白没有结构选择性,即目标蛋白的天然结构既可以是环状也可以是线性。Wu等[35]成功利用SrtA催化了糖基磷酸酰肌醇 (GPI) 与绿色荧光蛋白 (GFP) 的连接。GPI锚定蛋白在许多生化途径中均起重要作用,这一研究成果为合成大型复杂锚定蛋白提供了新途径,同时也为GPI锚定位点和锚定蛋白的生物学研究提供了有效途径。
Bolscher等[36]在对可促进再上皮化和伤口愈合组胺素的研究中,利用SrtA成功对其进行环化,相对于传统化学方法约3%的组胺素产率,转肽酶介导的环化可以使产物得率达到90%左右。且环化多肽摩尔分子活性 (Molar activity) 相对于线性多肽提高了近1 000倍。
此外,SrtA还可催化具有双官能团的分子内多肽和糖肽的从头合成。相对于化学酶合成法,这种方法只要求目标蛋白在N-、C-末端分别存在一个Gly残基和一个短的信号肽。但这种方法的局限性在于多肽中必须包含LPETGG及GG序列,从而能够在转肽酶作用下进行反应。经证实所有的连接产物中都包含有LPXTG序列,而这个序列对蛋白结构和功能是否存在影响也是一个需要探明的重要问题[37]。
4 基于超强分子粘合剂SpyTag/SpyCatcher的蛋白环化随着环肽类物质对天然产物以及生物活性物质结构功能研究的重要作用被证实,通过内含肽来介导蛋白质剪接,以转肽酶催化蛋白进行环化等非天然多肽的合成途径对蛋白类物质的改造研究取得极大进展。随着环化蛋白与其线性蛋白相比具有更高的稳定性、膜通透性等优势的显露。找到更高效、更加具有突破性的方法手段以更好地实现环肽类物质的从头合成逐渐成为研究热点,而新近发现的超强分子粘合剂则为蛋白的环化提供了全新的思路和研究方向。
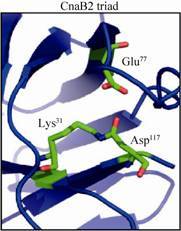
Mark Howarth研究团队[38]在2009年首次在革兰氏阳性菌酿脓链球菌Streptococcus pyogenes 菌毛蛋白 (pilin) Spy0128中分离了带有Lys残基的Pilin-C和携带Asn的异构肽键,实验表明Lys能够和Asn侧链自发地进行共价结合。在后续的研究中,该实验团队从酿脓链球菌纤连蛋白CnaB2中分离了N-末端带有Lys31、C-末端带有Asp117的能够进行自发共价结合的SpyTag/SpyCatcher,其结构示意图见图 4[39]。该反应能够在短时间内完成,并且不需要任何特异性物质和离子进行催化,他们对这种自发共价结合反应的条件进行了进一步验证,结果显示,在一定范围内的不同的pH、温度、缓冲液以及去垢剂的条件下,反应均能发生并具有优良的重组表达效果[40-41]。为验证这种共价结合反应是否较容易发生逆反应,该实验小组将SpyCatcher与SpyTag-MBP进行共价结合反应后,向反应环境中加入大量的Cna从而捕获自由的SpyCatcher,进行过夜培养。实验结果表明,SpyTag-MBP结构没有减少,也没有Cna多肽-SpyCatcher复合物的产生。经实验表征,共价结合的SpyTag/SpyCatcher异构肽键一经形成即具有极强的化学稳定性和耐高温等特性。这种能够在多种环境条件下均能自发反应形成的异构肽键将为蛋白质结构的研究提供更稳定可靠的研究模板。
为了探究这种超强分子粘合剂SpyTag/ SpyCatcher的共价结合对蛋白质的结构、功能、热稳定性及蛋白生物活性的影响,该实验团队通过基因融合,将含13个氨基酸的SpyTag与β-内酰胺酶N-末端连接,将分子量约为15 kDa的SpyCatcher与C-末端连接,使β-内酰胺酶自发环化,其环化过程见图 5[42]。
通常情况下该野生型酶在约37 ℃的条件下发生聚集失活,环化后的β-内酰胺酶热稳定性得到较大提升。为验证该方法是否适用于其他酶类,后续对通过该方法环化的二氢叶酸还原酶 (DHFR) 进行了热稳定性的测定,结果表明,在100 ℃的条件该酶仍然具有催化活性且反应收率很高。通过SpyTag/SpyCatcher共价结合的方法较点突变及其他环化途径对酶热稳定性具有较大幅度提高。SpyTag/SpyCatcher对酶进行环化以期改造其热稳定性,为酶的改造、提高其生物活性提供了新思路和方法,但该方法通用与否仍亟待深入研究[42]。
本课题组近年来一直致力于SpyTag/SpyCatcher的研究,成功构建了基于超强分子粘合剂SpyTag/SpyCatcher构建“Y型”拓扑结构的双酶,并以ELPs为纯化标签实现双功能酶分离纯化及固定化的集成;利用SpyTag/SpyCatcher成功构建了三臂ELPs,其性状与线性ELPs有较大差异,有望应用于一些新的领域。最近,本课题组成功在地衣多糖酶两端分别融合SpyTag和SpyCatcher,在温和条件下能自发共价结合实现酶分子环化。经实验证实,与线性地衣多糖酶相比,环化后的地衣多糖酶的温度稳定性有显著提高,其最适反应温度由55 ℃提高到了60 ℃左右,且最适反应温度范围有了大幅提高。在100 ℃煮沸10 min后,环化的地衣多糖酶酶活回收率较线性相比提高了将近4倍。经一系列酶活性、聚合温度以及高温冷却后酶活力恢复情况的测定,证实利用SpyTag/SpyCatcher能够高效实现地衣多糖酶环化,在高温等条件下,也保有相对较高的酶活力。该方法简单易行,能够自发反应,且共价结合较稳定。然而,在该过程中引入了新的基团,它是否会影响酶与底物的亲和力?在热稳定大幅度提高的同时,酶学性质及活力是否受到不利影响?其影响机制以及如何高效构建拓扑结构合理的环化酶等问题仍有待深入研究,本课题组将会持续探究。
5 总结与展望随着对环状多肽/蛋白结构和功能研究的深入,越来越多的重要生物学功能被发现,用来改造蛋白质结构的有效分子生物学方法不断地被优化以提高蛋白质的稳定性、酶学活性等。这些新的蛋白合成途径成功地替代了化学合成方法,为分子改造提供了新的改造手段。研究表明利用内含肽介导的蛋白剪切作用,表达蛋白连接作用以及蛋白酶催化的转肽作用都能够有效地实现蛋白的环化以提高其热稳定性以及对化学物质、变性条件的耐受性。在过去几十年里,研究者们探索和改进了多种蛋白环化方法。上述方法也都具有各自的优缺点,在实际应用时,需要根据目标蛋白的特性选择合适的环化方法。表 1列举了几种蛋白环化方法的应用实例及各种方法的特性。
| Method | Advantages | Deficiencies | Protein/peptide | Reference |
| PTS | Intramolecular self-catalytic process,require no other proteins or external sources,photoactivatable | Demand of specific binding sites | DnaB,DnaE,RFP | [14,19] |
| EPL | Stability,site specific,without the limit of size | Low protein expression | AopE | [27] |
| Sortase A | High yield,high ligation efficiency | Structure and function of the sequence “LPXTG” unknown | GFP,Kalata B1,SFTI-1 | [33-34] |
| SpyTag/SpyCatcher | Efficiency,spontaneous | Uncontrollable process,potential steric hindrance | BLA,DHFR | [42] |
与此同时,一些更有效、更简便的方法也逐渐走入研究者的视野。SpyTag/SpyCatcher在酶环化中的应用,其反应的自发性、高效性给蛋白环化带来了新突破,值得深入研究。
此外,最近两年,一种相对于传统内含肽剪接方法具有时间及空间灵活性的光催化内含肽作用也受到了蛋白质环化领域的极大关注。该过程可以直接从氨基酸序列水平对蛋白质进行调控,进而实现光对完全由基因编码蛋白质的诱导,由于在大多数蛋白中均存在Cys,因此该方法具有普遍有效性且高效。与传统方法相比,该方法不仅可以规避对细胞内组分的干扰,而且能有效抑制产物毒性。这种能够直接从氨基酸序列水平对蛋白进行调控的方法为蛋白质环化的研究提供了新方向。接下来蛋白环化研究的重点应该围绕增强环化蛋白的稳定性,找
到更快速、简单且对蛋白本身结构和功能影响较小的环化途径来开展。随着对蛋白结构和功能研究的深入以及酶活性和稳定性的研究的进行,相信在不久的将来,一定能够克服目前几种蛋白环化方法的缺陷和不足,使环化蛋白的结构稳定性和生物活性更接近于天然环肽。环化酶在工业、医药等领域的应用一定会取得更多突破性进展。
| [1] | Aboye TL, Camarero JA. Biological synthesis of circular polypeptides. J Biol Chem, 2012, 287 (32) : 27026–27032 (in Chinese). |
| [2] | Cascales L, Craik DJ. Naturally occurring circular proteins: distribution, biosynthesis and evolution. Org Biomol Chem, 2010, 8 (22) : 5035–5047 (in Chinese). |
| [3] | Jagadish K, Borra R, Lacey V, et al. Expression of fluorescent cyclotides using protein trans-splicing for easy monitoring of cyclotide-protein interactions. Angew Chem Int Ed, 2013, 52 (11) : 3126–3131 (in Chinese). |
| [4] | Mulvenna JP, Wang C, Craik DJ. CyBase: a database of cyclic protein sequence and structure. Nucleic Acids Res, 2006, 34 (S1) : D192–D194 (in Chinese). |
| [5] | Mulvenna JP, Mylne JS, Bharathi R, et al. Discovery of cyclotide-like protein sequences in graminaceous crop plants: ancestral precursors of circular proteins?. Plant Cell, 2006, 18 (9) : 2134–2144 (in Chinese). |
| [6] | Volkmann G, Murphy PW, Rowland EE, et al. Intein-mediated cyclization of bacterial acyl carrier protein stabilizes its folded conformation but does not abolish function. J Biol Chem, 2010, 285 (12) : 8605–8614 (in Chinese). |
| [7] | Zhou HX. Effect of backbone cyclization on protein folding stability: chain entropies of both the unfolded and the folded states are restricted. J Mol Biol, 2003, 332 (1) : 257–264 (in Chinese). |
| [8] | Kimura RH, Tran AT, Camarero JA. Biosynthesis of the cyclotide Kalata B1 by using protein splicing. Angew Chem Int Ed, 2006, 45 (6) : 973–976 (in Chinese). |
| [9] | Minato Y, Ueda T, Machiyama A, et al. Segmental isotopic labeling of a 140 kDa dimeric multi-domain protein CheA from Escherichia coli by expressed protein ligation and protein trans-splicing. J Biomol NMR, 2012, 53 (3) : 191–207 (in Chinese). |
| [10] | van Lieshout JFT, Pérez Gutiérrez ON, Vroom W, et al. Thermal stabilization of an endoglucanase by cyclization. Appl Biochem Biotech, 2012, 167 (7) : 2039–2053 (in Chinese). |
| [11] | Evans TC, Martin D, Kolly R, et al. Protein trans-splicing and cyclization by a naturally split intein from the DnaE gene of Synechocystis species PCC6803. J Biol Chem, 2000, 275 (13) : 9091–9094 (in Chinese). |
| [12] | Mujika JI, Lopez X, Mulholland AJ. Mechanism of C-terminal intein cleavage in protein splicing from QM/MM molecular dynamics simulations. Org Biomol Chem, 2012, 10 (6) : 1207–1218 (in Chinese). |
| [13] | Qi XM, Meng Q, Liu XQ. Spontaneous C-cleavage of a mini-intein without its conserved N-terminal motif A. FEBS Lett, 2011, 585 (15) : 2513–2518 (in Chinese). |
| [14] | Ding Y, Xu MQ, Ghosh I, et al. Crystal structure of a mini-intein reveals a conserved catalytic module involved in side chain cyclization of asparagine during protein splicing. J Biol Chem, 2003, 278 (40) : 39133–39142 (in Chinese). |
| [15] | Thiel IV, Volkmann G, Pietrokovski S, et al. An atypical naturally split intein engineered for highly efficient protein labeling. Angew Chem Int Ed, 2014, 53 (5) : 1306–1310 (in Chinese). |
| [16] | Williams NK, Liepinsh E, Watt SJ, et al. Stabilization of native protein fold by intein-mediated covalent cyclization. J Mol Biol, 2005, 346 (4) : 1095–1108 (in Chinese). |
| [17] | Williams NK, Prosselkov P, Liepinsh E, et al. In vivo protein cyclization promoted by a circularly permuted Synechocystis sp. PCC6803 DnaB mini-intein. J Biol Chem, 2002, 277 (10) : 7790–7798 (in Chinese). |
| [18] | Siebold C, Erni B. Intein-mediated cyclization of a soluble and a membrane protein in vivo: function and stability. Biophys Chem, 2002, 96 (2/3) : 163–171 (in Chinese). |
| [19] | Ren W, Ji A, Ai HW. Light activation of protein splicing with a photocaged fast intein. J Am Chem Soc, 2015, 137 (6) : 2155–2158 (in Chinese). |
| [20] | B?cker JK, Friedel K, Matern JCJ, et al. Generation of a genetically encoded, photoactivatable intein for the controlled production of cyclic peptides. Angew Chem Int Ed, 2015, 54 (7) : 2116–2120 (in Chinese). |
| [21] | Binschik J, Zettler J, Mootz HD. Photocontrol of protein activity mediated by the cleavage reaction of a split intein. Angew Chem Int Ed, 2011, 50 (14) : 3249–3252 (in Chinese). |
| [22] | Gautier A, Nguyen DP, Lusic H, et al. Genetically encoded photocontrol of protein localization in mammalian cells. J Am Chem Soc, 2010, 132 (12) : 4086–4088 (in Chinese). |
| [23] | Nguyen DP, Mahesh M, Els?sser SJ, et al. Genetic encoding of photocaged cysteine allows photoactivation of TEV protease in live mammalian cells. J Am Chem Soc, 2014, 136 (6) : 2240–2243 (in Chinese). |
| [24] | Clerico EM, Zhuravleva A, Smock RG, et al. Segmental isotopic labeling of the Hsp70 molecular chaperone DnaK using expressed protein ligation. Biopolymers, 2010, 94 (6) : 742–752 (in Chinese). |
| [25] | Berrade L, Camarero JA. Expressed protein ligation: a resourceful tool to study protein structure and function. Cell Mol Life Sci, 2009, 66 (24) : 3909–3922 (in Chinese). |
| [26] | De Rosa L, Cortajarena AL, Romanelli A, et al. Site-specific protein double labeling by expressed protein ligation: applications to repeat proteins. Org Biomol Chem, 2012, 10 (2) : 273–280 (in Chinese). |
| [27] | Zhao WT, Zhang YH, Cui CX, et al. An efficient on-column expressed protein ligation strategy: application to segmental triple labeling of human apolipoprotein E3. Protein Sci, 2008, 17 (4) : 736–747 (in Chinese). |
| [28] | Clark KM, van der Donk WA, Lu Y. Expressed protein ligation for metalloprotein design and engineering. Methods Enzymol, 2009 : 97–115 (in Chinese). |
| [29] | Kamei A, Hauser PS, Beckstead JA, et al. Expressed protein ligation-mediated template protein extension. Protein Expr Purif, 2012, 83 (2) : 113–116 (in Chinese). |
| [30] | Cui CX, Zhao WT, Chen JL, et al. Elimination of in vivo cleavage between target protein and intein in the intein-mediated protein purification systems. Protein Exp Purif, 2006, 50 (1) : 74–81 (in Chinese). |
| [31] | Mazmanian SK, Liu G, Ton-That H, et al. Staphylococcus aureus sortase, an enzyme that anchors surface proteins to the cell wall. Science, 1999, 285 (5428) : 760–763 (in Chinese). |
| [32] | Strijbis K, Spooner E, Ploegh HL. Protein ligation in living cells using sortase. Traffic, 2012, 13 (6) : 780–789 (in Chinese). |
| [33] | Proft T. Sortase-mediated protein ligation: an emerging biotechnology tool for protein modification and immobilisation. Biotechnol Lett, 2010, 32 (1) : 1–10 (in Chinese). |
| [34] | Jia XY, Kwon S, Wang CIA, et al. Semienzymatic cyclization of disulfide-rich peptides using sortase A. J Biol Chem, 2014, 289 (10) : 6627–6638 (in Chinese). |
| [35] | Wu ZM, Guo XQ, Gao J, et al. Sortase A-mediated chemoenzymatic synthesis of complex glycosylphosphatidylinositol-anchored protein. Chem Commun, 2013, 49 (99) : 11689–11691 (in Chinese). |
| [36] | Bolscher JGM, Oudhoff MJ, Nazmi K, et al. Sortase A as a tool for high-yield histatin cyclization. FASEB J, 2011, 25 (8) : 2650–2658 (in Chinese). |
| [37] | Wu ZM, Guo XQ, Guo ZW. Sortase A-catalyzed peptide cyclization for the synthesis of macrocyclic peptides and glycopeptides. Chem Commun, 2011, 47 (32) : 9218–9220 (in Chinese). |
| [38] | Li L, Fierer JO, Rapoport TA, et al. Structural analysis and optimization of the covalent association between SpyCatcher and a peptide tag. J Mol Biol, 2014, 426 (2) : 309–317 (in Chinese). |
| [39] | Zakeri B, Fierer JO, Celik E, et al. Peptide tag forming a rapid covalent bond to a protein, through engineering a bacterial adhesin. Proc Natl Acad Sci USA, 2012, 109 (12) : E690–E697 (in Chinese). |
| [40] | Wikoff WR, Liljas L, Duda RL, et al. Topologically linked protein rings in the bacteriophage HK97 capsid. Science, 2000, 289 (5487) : 2129–2133 (in Chinese). |
| [41] | Zakeri B, Howarth M. Spontaneous intermolecular amide bond formation between side chains for irreversible peptide targeting. J Am Chem Soc, 2010, 132 (13) : 4526–4527 (in Chinese). |
| [42] | Schoene C, Fierer JO, Bennett SP, et al. SpyTag/SpyCatcher cyclization confers resilience to boiling on a mesophilic enzyme. Angew Chem Int Ed, 2014, 53 (24) : 6101–6104 (in Chinese). |
 2016, Vol. 32
2016, Vol. 32