中国科学院微生物研究所、中国微生物学会主办
文章信息
- 孟曦男, 周恒达, 徐素宏
- Meng Xi'nan, Zhou Hengda, Xu Suhong
- CRISPR-Cas9基因编辑技术在秀丽线虫中的应用
- CRISPR-Cas9 mediated genome editing in Caenorhabditis elegans
- 生物工程学报, 2017, 33(10): 1693-1699
- Chinese Journal of Biotechnology, 2017, 33(10): 1693-1699
- 10.13345/j.cjb.170177
-
文章历史
- Received: May 2, 2017
- Accepted: July 31, 2017
生物学研究的一个重要目标是精确剖析基因在生物体生长发育和疾病中的功能。常用的技术手段是改变基因的表达以观察其对生物体的影响,并进一步研究其发挥作用的分子细胞学机制。几十年来,多种技术手段被用于制造基因功能缺失,例如基因敲降技术RNAi,基因敲除技术(包括时空特异性和组织特异性敲除),以及多种化学诱变剂诱导基因突变的手段等。这些技术构成了正向遗传和反向遗传学解决生物学问题的基础。另一种研究基因功能的方法是观察基因表达产物蛋白质在细胞内的动态运动,例如利用抗体染色或内源性荧光蛋白标记技术。这两种研究基因功能的策略是现代生物学研究的常规策略,在多种模式生物中的研究取得了巨大的成功。
秀丽线虫(Caenorhabditis elegans, 以下简称线虫)是一种经典的遗传学模式生物,具有生命周期短、基因组小、基因数目多、便于遗传操作、通体透明易于显微观察等优势,被广泛用于发育生物学和神经生物学的研究。制造线虫基因突变的方法有化学诱变剂甲磺酸乙酯(Ethyl methane sulfonate,EMS)引起的基因突变,物理辐射引起的基因突变等,以及利用转座子Tc1和Mos1插入的方法改变基因组等多种手段[1-3]。近年来,锌指核糖核酸酶(Zinc finger nucleases,ZFNs)和转录激活样效应蛋白核酸酶(Transcription activator-like effector nucleases,TALENs)也被用于改造线虫的基因组[4-5]。但由于ZFNs和TALENs前期工具质粒构建较为繁琐和昂贵,并没有在线虫中被广泛应用。自2013年以来,从细菌免疫现象中发现和发展的CRISPR-Cas9基因编辑技术由于其构造简单、易于操控和改造以及价格低廉等优势,迅速成为基因编辑领域的主要研究技术[6-7]。该方法几乎在所有模式生物中得到了广泛应用,大力推动了反向遗传学解决生物学问题的研究[8]。关于CRISPR-Cas9在线虫中的应用,国内外期刊已有多篇详尽的综述论文[9-11]。此篇论文主要针对中文读者以及线虫领域的一线研究人员,介绍CRISPR-Cas9基因编辑技术在线虫中的最新发展和应用。
1 基因编辑的原理基因组在进行自我复制以及遭受环境压力下均会发生突变,其中大多数情况下能得到精确修复。整条基因组双链DNA断裂(Double strand break,DSB)的情况下,生物体内源性的修复机制启动并修复基因组序列。目前已知存在两种机制参与修复双链DNA断裂[6],一种为非同源性末端连接(Non-homologous end joining,NHEJ),修复结束后可能会造成DNA上碱基的随机缺失和插入,引起基因开放阅读框的突变;另一种为同源序列指导修复(Homology-directed repair,HDR),即基于模板存在情况下的同源重组,可以利用外源提供的同源序列实现精确修复。无论是ZFNs、TALENs还是CRISPR-Cas9系统,都以这两种修复机制为基础实现对特定基因的靶向编辑。
2 CRISPR-Cas9系统CRISPR-Cas9系统最先于细菌中发现,是细菌抵御外界病毒感染的一种有效方式。CRISPR重复序列间有不同的居间序列,表达出引导RNA,可以通过碱基互补配对的方式指导Cas蛋白家族(现在主要是SpCas9) 特异地在病毒的基因组上进行切割从而阻断病毒基因组的复制从而达到抑制病毒生长的目的。这一工作模式最早被Jennifer Doudna、Emmanuelle Charpentier和张锋等科学家设计用来人工控制特异性地切割外源和内源DNA[12-13]。CRISPR-Cas9系统的核心组分为识别靶向DNA的引导RNA (sgRNA)和核酸酶Cas9。这种简易而且高效的基因编辑方法迅速成为现代生物学研究重要的工具。
CRISPR-Cas9基因编辑手段的优点在于其设计方便,利用碱基互补配对的原则理论上可以设计出任何基因特异性的sgRNA,结合Cas9对特定位点处DNA造成双链断裂[7]。一直以来,在线虫中,外源DNA可以通过显微注射的方法注射到生殖腺中获得转基因[14],对于CRISPR-Cas9系统,显微注射也是最方便和快捷的方式[8]。研究人员发现,注射体外纯化的Cas9核酸酶或表达Cas9核酸酶的质粒混合sgRNA质粒或体外转录的sgRNA都可以获得相应的突变体[15-18](图 1A),这为后期改良系统奠定了基础。在考虑到经济成本和工作量的情况下,如今大多数线虫实验室都通过直接注射编码Cas9和sgRNA的质粒的方法来改造基因组(图 1)。现今CRISPR-Cas9基因编辑技术一个最为关键的问题是其脱靶率较高,但是通过全基因组测序及脱靶基因预测,该编辑技术在线虫中至今鲜有脱靶现象,这可能和线虫相对简单的基因组有关[8]。
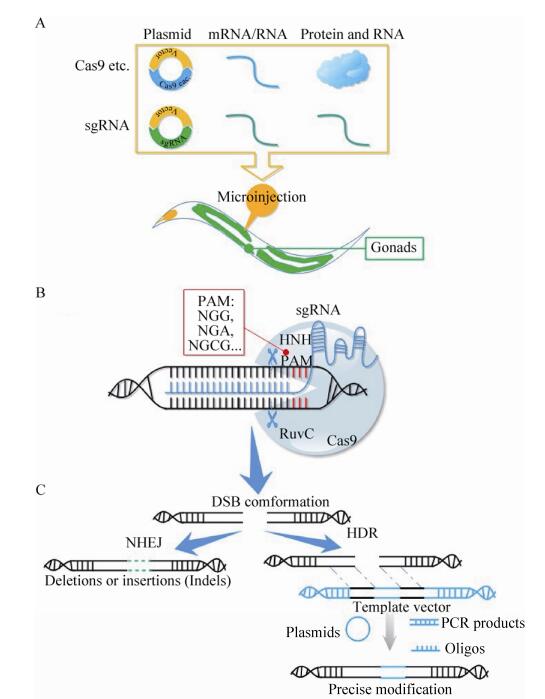
|
| 图 1 CRIPSR/Cas9在线虫中进行基因编辑的工作原理和流程图. (A)共注射质粒或者mRNA或者蛋白到线虫的生殖腺中,线虫的卵母细胞中发生CRISPR/Cas9的基因编辑. (B) Cas9核酸酶的两个活性域负责切割双链DNA,其切割位置由PAM序列决定。一般来说,Cas9识别的PAM序列为NGG序列,一些Cas9突变型可以识别以NGA和NGCG结尾的sgRNA. (C) DSB形成后基因组以NHEJ的方式修复发生各类突变或以寡核苷酸链、PCR产物或质粒为模板启动HDR修复进行特定转基因等编辑 Figure 1 Diagram of the application of CRISPR-Cas9 mediated genome editing in C. elegans. (A) Cas9 (blue) and guided RNA (green) can be expressed in C. elegans germline by microinjection either the mixture of plasmids, mRNA or the combination of Cas9 protein and sgRNA mRNA. (B) Cas9 and sgRNA will be assembly as a complex at its dsDNA target site. Two nuclease domains RuvC and HNH cleave the DNA close to 5′ of PAM (Red) sequence and generate DNA double-strand breaks (DSBs). Many PAM sequences have been found due to different type of Cas9 variants used. (C) The double stand break induced by the Cas9/sgRNA complex can be repaired by non-homologous end joining (NHEJ) or homology-directed repair (HDR). NHEJ can lead to the introduction of insertion/deletion mutations (indels) (green) of various lengths and HDR-mediated repair can introduce specific point mutation or insert/delete desired sequences (blue) with exogenously donor templates. In C. elegans, plasmid DNA, PCR products and synthesized oligos have all been used as HDR repair template. |
| |
为进一步提高CRISPR-Cas9基因编辑的效率,减少后续突变体筛选上的工作量以及力求更经济便捷的手段,研究人员利用线虫的遗传学优势,对CRISPR-Cas9在线虫上的应用做了诸多改良。CRISPR-Cas9系统原理简单,关键影响因素取决于Cas9蛋白、sgRNA和模板DNA,研究人员通过对以下5个方面的改良,力求更高效地获得所需要的突变体。
2.1 Cas9核酸酶的优化现在大多实验室利用的都是CRISPR-Cas9系统进行基因编辑。通常情况下Cas9特异性识别的PAM序列是5′-NGG-3′序列。在基因组上,NGG序列出现的概率较高,但是在特定情况下,例如需要做定点突变,可能很难寻找到合适的sgRNA。最近,Andrew Fire实验室针对性地将Cas9的PAM识别区域进行定点突变,发现可以改变Cas9对PAM序列的识别[19]。新型的Cas9突变(VQR和VRER突变体Cas9) 在线虫基因组上可以分别识别PAM序列5′-NGA-3′和5′-NGCG-3′,为研究人员提供了更多的Cas9可编辑位点的选择(图 1B)[19]。
2.2 sgRNA的优化CRISPR-Cas9基因编辑系统最为关键的是寻找到合适的sgRNA,一方面需要sgRNA非常特异,另一方面需要sgRNA和靶向DNA可以稳定结合。通过对众多已发表的sgRNA所作用的效率进行比较,研究人员发现3′端为GG的sgRNA的基因编辑效果显著高于3′端非GG的sgRNA,即在紧邻PAM序列的5'端还存在另一GG序列,即5′-GGNGG-3′类型序列[20]。实验统计数据也表明这一sgRNA的设计可以显著提高基因编辑的成功率[20-21]。另外sgRNA改良型sgRNA (F+E),其上延伸出一段缺少PolIII终止子与Cas9的结合域,亦能提高CRISPR-Cas9系统基因编辑的成功率[22-23]。此外,由于生物体内大量重复基因的存在,根据DNA序列的保守性,单条sgRNA即可在线虫基因组中实现多个同源基因的编辑[24]。
2.3 修复模板的载体优化在外源模板存在的情况下,断裂的双链DNA可以通过同源重组的方式进行精确修复。这一DNA修复的机制在基因编辑上得到了极大的应用,例如用荧光蛋白标记目标蛋白质以观察其在细胞内的动态变化等。线虫里可以直接注射外源质粒DNA作为模板,同源序列长度大于500 bp的质粒DNA都能发生有效的同源重组,但是同源重组的效率不同的基因有所不同[18]。最近的研究也发现,DNA的PCR产物片段或者单链DNA也可以被用作同源重组的模板,并且同源臂的长度可短至30个核苷酸,且同源重组的效率并不受显著的影响[25-26]。由此所进行的修复模板改良,给以线虫为模型的研究人员提供了更多选择,同时节省了时间和成本,加速了实验的进程。
2.4 筛选手段的优化线虫突变体传统的筛选方法是借助于表型观察和基于PCR方法的基因型鉴定。由于大多数基因的突变不会造成非常明显的表型,因此,PCR鉴定成为了最重要的检测方法。为了减少PCR检测的工作量,利用线虫自身的遗传学优势,一些前期的筛选方法很快被改造出来,例如co-CRISPR和co-Conversion的方法[27-29],其原理主要是筛选Cas9核酸酶有活性的突变体。此外,还有直接的筛选方法如药物筛选一些抗药基因,荧光观察直接插入荧光标记或缺失的基因等[18, 24]。其中药物筛选虽然有效,但也引入了更多的外源基因,为除去这些外源不必要的插入序列,一种基于Cre/LoxP或Flp/FRT重组酶系统的自我消除元件(Self-excising cassette,SEC)被设计出来[30]。这些筛选方法有效地减少了PCR筛选的工作量并提高了基因编辑的效率。
2.5 组织特异性敲除基因表达的时序性和组织特异性为解析基因功能的多效性带来了很大难度,解决这一问题的方法主要是条件性突变或基因失活。目前,在线虫里已经有一些策略被用于条件性基因失活,例如基于定点重组酶系统的Cre/LoxP和Flp/FRT系统,组织、时空或温度特异性的RNA干扰系统和条件性TALENs系统等[5, 18, 31-32]。这些方法极大地方便了对基因在不同组织中功能的解析,特别是对于一些早期胚胎致死基因的研究有非常大的贡献。清华大学欧光朔课题组首先利用CRISRP-Cas9系统开发出了更为简单而高效的条件性敲除系统。他们利用细胞和时空特异性表达的启动子表达Cas9核酸酶以及细胞核定位的sgRNA,可以实现在特定细胞或特定时期敲除基因,确定其在不同生理条件下不同细胞以及不同时期的功能[11]。这一方法为研究发育过程中必需基因的功能创造了条件[33]。
3 总结与展望现今主要运用于秀丽线虫中的基因编辑技术多依赖于CRISPR原理的Cas核酸酶系统,其强大的定点基因编辑能力进一步完善了在线虫中的相关研究手段,使得对线虫基因组的编辑在效率上得到极大提升。对CRISPR的基因编辑系统的不断改良也是科学研究的一个重要方向。比如全部核酸酶功能失活的Cas9,即dCas9现在也被用于结合特定位点参与基因调控。且除了Cas9之外,还有许多新型Cas核酸酶仍在不断被发现和修饰,如Cpf1,不需要tracrRNA即可发生识别和剪切[34];新从地下水和土壤中发现的CasX和CasY,质量更小[35]。虽然没有在线虫中采用过,但是其应用前景广泛。
从线虫的基因编辑技术的发展历程来看,起初利用化学诱变剂、物理高能辐射或者是自然转座随机性地制造点突变、基因插入或缺失,得到了很多突变体,为科学研究提供了大量的材料,帮助科学家解析这些基因的正常生理功能。现在利用CRISPR-Cas9高效并多能的基因编辑系统,科学家可以实现对特定基因进行靶向编辑或标记,并能对单核苷酸进行精准编辑,大大有利于现代生物学对基因及其表达产物在生命体中生理功能的研究。线虫中CRISPR-Cas9系统的优化和改造,也为这一编辑技术在其他物种中的应用提供参考和借鉴。
| [1] | Robert V, Bessereau JL. Targeted engineering of the Caenorhabditis elegans genome following Mos1-triggered chromosomal breaks. EMBO J, 2007, 26(1): 170–183. DOI: 10.1038/sj.emboj.7601463 |
| [2] | Plasterk RHA, Groenen JTM. Targeted alterations of the Caenorhabditis elegans genome by transgene instructed DNA double strand break repair following TcL excision. EMBO J, 1992, 11(1): 287–290. |
| [3] | Gengyo-Ando K, Mitani S. Characterization of mutations induced by ethyl methanesulfonate, UV, and trimethylpsoralen in the nematode Caenorhabditis elegans. Biochem Biophys Res Commun, 2000, 269(1): 64–69. DOI: 10.1006/bbrc.2000.2260 |
| [4] | Wood AJ, Lo TW, Zeitler B, et al. Targeted genome editing across species using ZFNs and TALENs. Science, 2011, 333(6040): 307. DOI: 10.1126/science.1207773 |
| [5] | Cheng Z, Yi PS, Wang XM, et al. Conditional targeted genome editing using somatically expressed TALENs in C. elegans. Nat Biotechnol, 2013, 31(10): 934–937. DOI: 10.1038/nbt.2674 |
| [6] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262–1278. DOI: 10.1016/j.cell.2014.05.010 |
| [7] | Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096. DOI: 10.1126/science.1258096 |
| [8] | Friedland AE, Tzur YB, Esvelt KM, et al. Heritable genome editing in C. elegans via a CRISPR-Cas9 system. Nat Methods, 2013, 10(8): 741–743. DOI: 10.1038/nmeth.2532 |
| [9] | Dickinson DJ, Goldstein B. CRISPR-based methods for Caenorhabditis elegans genome engineering. Genetics, 2016, 202(3): 885–901. DOI: 10.1534/genetics.115.182162 |
| [10] | Xu SH. The application of CRISPR-Cas9 genome editing in Caenorhabditis elegans. J Genet Genom, 2015, 42(8): 413–421. DOI: 10.1016/j.jgg.2015.06.005 |
| [11] | Li W, Ou GS. The application of somatic CRISPR-Cas9 to conditional genome editing in Caenorhabditis elegans. Genesis, 2016, 54(4): 170–181. DOI: 10.1002/dvg.22932 |
| [12] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819–823. DOI: 10.1126/science.1231143 |
| [13] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816–821. DOI: 10.1126/science.1225829 |
| [14] | Berkowitz LA, Knight AL, Caldwell GA, et al. Generation of stable transgenic C. elegans using microinjection. J Vis Exp, 2008(18): 833. |
| [15] | Waaijers S, Portegijs V, Kerver J, et al. CRISPR/Cas9-targeted mutagenesis in Caenorhabditis elegans. Genetics, 2013, 195(3): 1187–1191. DOI: 10.1534/genetics.113.156299 |
| [16] | Tzur YB, Friedland AE, Nadarajan S, et al. Heritable custom genomic modifications in Caenorhabditis elegans via a CRISPR-Cas9 system. Genetics, 2013, 195(3): 1181–1185. DOI: 10.1534/genetics.113.156075 |
| [17] | Katic I, Grosshans H. Targeted heritable mutation and gene conversion by Cas9-CRISPR in Caenorhabditis elegans. Genetics, 2013, 195(3): 1173–1176. DOI: 10.1534/genetics.113.155754 |
| [18] | Dickinson DJ, Ward JD, Reiner DJ, et al. Engineering the Caenorhabditis elegans genome using Cas9-triggered homologous recombination. Nat Meth, 2013, 10(10): 1028–1034. DOI: 10.1038/nmeth.2641 |
| [19] | Bell RT, Fu BX, Fire AZ, et al. Cas9 variants expand the target repertoire in Caenorhabditis elegans. Genetics, 2016, 202(2): 381–388. DOI: 10.1534/genetics.115.185041 |
| [20] | Farboud B, Meyer BJ. Dramatic enhancement of genome editing by CRISPR/Cas9 through improved guide RNA design. Genetics, 2015, 199(4): 959–971. DOI: 10.1534/genetics.115.175166 |
| [21] | Schwartz ML, Jorgensen EM. SapTrap, a toolkit for high-throughput CRISPR/Cas9 gene modification in Caenorhabditis elegans. Genetics, 2016, 202(4): 1277–1288. DOI: 10.1534/genetics.115.184275 |
| [22] | Chen BH, Gilbert LA, Cimini BA, et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell, 2013, 155(7): 1479–1491. DOI: 10.1016/j.cell.2013.12.001 |
| [23] | Zhao P, Zhang Z, Lü XY, et al. One-step homozygosity in precise gene editing by an improved CRISPR/Cas9 system. Cell Res, 2016, 26(5): 633–636. DOI: 10.1038/cr.2016.46 |
| [24] | Xu S, Wang Z, Kim KW, et al. Targeted mutagenesis of duplicated genes in Caenorhabditis elegans using CRISPR-Cas9. J Genet Genomics, 2016, 43(2): 103–106. DOI: 10.1016/j.jgg.2015.11.004 |
| [25] | Paix A, Wang YM, Smith HE, et al. Scalable and versatile genome editing using linear DNAs with microhomology to Cas9 sites in Caenorhabditis elegans. Genetics, 2014, 198(4): 1347–1356. DOI: 10.1534/genetics.114.170423 |
| [26] | Paix A, Folkmann A, Seydoux G. Precision genome editing using CRISPR-Cas9 and linear repair templates in C. elegans. Methods, 2017, 121-122: 86–93. DOI: 10.1016/j.ymeth.2017.03.023 |
| [27] | Ward JD. Rapid and precise engineering of the Caenorhabditis elegans genome with lethal mutation co-conversion and inactivation of NHEJ repair. Genetics, 2015, 199(2): 363–377. DOI: 10.1534/genetics.114.172361 |
| [28] | Kim H, Ishidate T, Ghanta KS, et al. A co-CRISPR strategy for efficient genome editing in Caenorhabditis elegans. Genetics, 2014, 197(4): 1069–1680. DOI: 10.1534/genetics.114.166389 |
| [29] | Arribere JA, Bell RT, Fu BX, et al. Efficient marker-free recovery of custom genetic modifications with CRISPR/Cas9 in Caenorhabditis elegans. Genetics, 2014, 198(3): 837–846. DOI: 10.1534/genetics.114.169730 |
| [30] | Dickinson DJ, Pani AM, Heppert JK, et al. Streamlined genome engineering with a self-excising drug selection cassette. Genetics, 2015, 200(4): 1035–1049. DOI: 10.1534/genetics.115.178335 |
| [31] | Voutev R, Hubbard EJA. A " FLP-Out" system for controlled gene expression in Caenorhabditis elegans. Genetics, 2008, 180(1): 103–119. DOI: 10.1534/genetics.108.090274 |
| [32] | Flavell SW, Pokala N, Macosko EZ, et al. Serotonin and the neuropeptide PDF initiate and extend opposing behavioral states in C. elegans. Cell, 2013, 154(5): 1023–1035. DOI: 10.1016/j.cell.2013.08.001 |
| [33] | Shen ZF, Zhang XL, Chai YP, et al. Conditional knockouts generated by engineered CRISPR-Cas9 endonuclease reveal the roles of coronin in C. elegans neural development. Dev Cell, 2014, 30(5): 625–636. DOI: 10.1016/j.devcel.2014.07.017 |
| [34] | Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759–771. DOI: 10.1016/j.cell.2015.09.038 |
| [35] | Burstein D, Harrington LB, Strutt SC, et al. New CRISPR-cas systems from uncultivated microbes. Nature, 2017, 542(7640): 237–241. |
 2017, Vol. 33
2017, Vol. 33




