中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王春, 王克剑
- Wang Chun, Wang Kejian
- CRISPR-Cas系统在植物基因组编辑中的研究进展
- Advances in CRISPR-Cas-mediated genome editing system in plants
- 生物工程学报, 2017, 33(10): 1712-1722
- Chinese Journal of Biotechnology, 2017, 33(10): 1712-1722
- 10.13345/j.cjb.170170
-
文章历史
- Received: April 24, 2017
- Accepted: June 27, 2017
随着测序技术的进步和测序成本的降低,越来越多物种的全基因组已经被测序,如何解析基因功能并利用这些研究结果改造生物体已成为下一步的研究目标。基因组编辑技术是实现以上目标的重要研究手段,它突破了之前特定突变体生物材料获得困难的限制,并尽可能小地降低了对受体基因组背景的影响,对生物领域的发展与应用有着重要意义。
锌指核酸酶(Zinc-finger nucleases,ZFNs)和类转录激活因子效应物核酸酶(Transcription activator-like effector nucleases,TALENs)最先被应用于基因组定点编辑[1-3]。这两个核酸酶由识别特异DNA序列的蛋白与非特异性的核酸内切酶融合组成,可以发挥基因组定点切割的功能。但由于这两项技术DNA结合结构域的改造组装复杂且成本高,不利于广泛推广应用。最近,科学家们发现了一种新的CRISPR-Cas (Clustered regularly interspaced short palindromic repeats and CRISPR-associated proteins)基因组编辑技术,该技术通过一段向导RNA和配套的核酸酶对特定的基因组序列进行定点编辑,具有简单高效的优点,在短短几年时间内,已被广泛地用于基础科学研究、人类基因治疗与作物遗传育种等领域。
本文结合部分在动物及人类中的研究结果,主要介绍在植物中最常用的CRISPR-Cas9系统的研究和应用,CRISPR-Cas各种改造和升级版本在植物基因功能研究上的拓展应用,以及基因组编辑技术在作物育种上的应用成果,为开展这一领域的研究工作提供参考。
1 CRISPR-Cas9系统的研究历史及在植物中的应用日本科学家最早在细菌中发现一段由长度为29 bp的重复片段(Repeats)和32–33 bp (Spacers)的非重复片段间隔相连的重复序列[4]。后来,科学家们发现这种重复序列广泛存在于细菌和古细菌基因组中,并将其命名为规律成簇间隔短回文重复(Clustered regularly interspaced short palindromic repeats,CRISPR)。之后的研究表明,CRISPR位点表达与入侵病毒基因组序列相匹配的小分子RNA,通过互补序列结合病毒基因组,引导相关核酸酶复合体切割病毒DNA,阻止病毒入侵,为原核生物提供了对抗噬菌体等外源基因的获得性免疫能力[5-6]。
2012年,Jinek等[7]发现Ⅱ型CRISPR系统组成较为简单,只需依赖1个Cas9核酸酶及2个RNA (crRNA,tracrRNA)就可以切割目的双链DNA,并发现把tracrRNA和crRNA人工改造整合为一个RNA转录本时,也能引导Cas9蛋白识别靶序列并引起DNA序列双链断裂(Double-strand breaks,DSBs),为此后RNA介导的基因编辑奠定了基础。随后,基于Ⅱ型CRISPR-Cas9系统,Church实验室在多种人类细胞系中[8],张峰实验室在人类及小鼠细胞中[9],成功获得了预期的非同源末端连接(Non-homologous end joining,NHEJ)或同源重组介导的修复(Homology-directed repair,HDR)方式产生的基因定点突变。CRISPR-Cas9基因组定点编辑技术的开发,使得多个基因的敲除及敲入变得更为简单高效,很快就形成了研究热潮。
经过改造的CRISPR-Cas9系统也迅速地被应用到植物基因组编辑研究中。2013年8月8日,Nature Biotechnology同时报道了3个不同实验室在重要农作物及模式植物中的研究进展:Shan等[10]通过基因枪的方式传递CRISPR-Cas9系统,在水稻和小麦中进行了定点修饰;Li等[11]报道了在拟南芥、烟草等物种中成功应用CRISPR-Cas9进行基因修饰;Nekrasov等[12]利用CRISPR-Cas9系统在模式植物本生烟中实现了基因组的定点突变。此外,Feng等[13]报道在模式植物拟南芥和水稻中利用CRISPR-Cas9系统实现了定点突变;Miao等[14]通过密码子优化Cas9蛋白在水稻中高效获得了叶绿素合成基因CAO1和分蘖夹角控制基因LAZY1突变的植株。
随后,多个实验室对CRISPR-Cas9系统在植物中的应用进行了进一步完善。Ma等[15]采用Gibson或Golden Gate Cloning方法将多个gRNA及Cas9序列组装至一个双元载体,构建了适用于单子叶和双子叶植物的多重基因组编辑CRISPR-Cas9载体系统。Xie等[16]将tRNA和gRNA嵌合在一起,利用tRNA的切割在植物体内游离出多个gRNA,开发了一套从一个多顺反子基因生成大量gRNA的通用策略,在水稻中完成了多基因的同时编辑。本课题组[17]基于同尾酶的特性,设计了一套只包含两个质粒的gRNA拼接系统,通过传统的酶切连接方式,实现多个gRNA元件的快速组装,该系统理论上可以实现无限个gRNA的组装,具有简单、稳定的优点;在此基础上,我们构建了一个双元载体,对8个水稻农艺性状相关基因同时进行了编辑,T0代就获得了8个基因同时敲除的突变体[18]。
转化拟南芥通常采用农杆菌介导的沾花法,胚囊是转基因插入的主要目标,而普遍采用的CaMV 35S启动子在生殖细胞中活性极低,因此,由于转化方式的不同,用CaMV 35S驱动的Cas9在拟南芥基因组的编辑效率明显低于水稻,并且绝大多数T1植株只是体细胞突变,并不能遗传到T2代。Yan等[19]利用在胚、胚囊、胚乳以及花粉中有很高表达量的YAO启动子,Wang等[20]利用卵细胞特异的EC1.2启动子,Mao等[21]利用配子体时期表达的SPL启动子来驱动Cas9在胚时期特异表达,大大提高了拟南芥在T1代的基因突变频率,有效解决了CRISPR/Cas9在拟南芥的转基因T1代株系基因突变频率低、诱导得到的主要是嵌合体的难题。
到目前为止,包括拟南芥、烟草、水稻、玉米、小麦等模式植物及一些作物在内,均已报道成功获得了较高的突变率及可稳定遗传的基因组编辑植株[22]。
2 改造开发CRISPR-Cas系统扩大基因组编辑范围的研究 2.1 Cas9变体除了需要gRNA对目标序列进行20 bp左右的特异匹配,CRISPR-Cas9系统在基因组上的精准切割还需要识别一段靶DNA附近、称作前间区序列邻近基序(Protospacer adjacent motif,PAM)的特异核苷酸序列。最常用的Cas9来自于产脓链球菌Streptococcus pyogenes,被称为SpCas9。SpCas9能够识别的PAM序列主要为NGG,这限制了CRISPR-Cas9系统在基因组上的可编辑范围。为突破这个限制,Kleinstiver等[23]建立了一个工程系统,使得能够快速地演化SpCas9识别不同PAM序列的能力。通过收集随机突变的SpCas9变体,筛选到了使得SpCas9能够识别新PAM序列的突变组合。
根据细菌中的研究结果,本课题组在水稻中,通过定点突变的方法对SpCas9蛋白进行改造,获得两种SpCas9变体(VQR和VRER)[24]。通过一系列的转基因实验证明,VQR和VRER这两种变体在水稻中分别可以识别NGA以及NGCG两个PAM序列。生物信息学分析表明Cas9变体的创制将使得CRISPR-Cas9系统在水稻中可编辑范围拓展到现有的2倍以上。此外,两种变体也可以同时对多基因进行有效的编辑,表明其与野生型Cas9一样具备巨大的应用潜力,为在水稻中进行更广泛的基因组编辑提供了新的可选工具。
2.2 CRISPR-Cpf1系统Zetsche等[25]通过生物信息学手段发现了新的Ⅴ型CRISPR基因编辑系统CRISPR-Cpf1 (CRISPR from Prevotella and Francisella 1)。与CRISPR-Cas9系统相比,CRISPR-Cpf1具有以下优点:1) Cpf1是一个比Cas9更小、更简单的核酸内切酶,仅通过一段较短的crRNA识别目的DNA底物,更易于操作;2) CRISPR-Cpf1识别的PAM序列是连续的2个或3个胸腺嘧啶(T),这将大大扩展基因组编辑范围;3) CRISPR-Cpf1剪切目的DNA造成的是粘性末端,预计有助于目标序列的精确插入和同源重组替换,能够更有效及精确地整合一段DNA;4) Cpf1不仅切割DNA,而且也切割RNA,表现出双重切割活性,利于多基因敲除的载体构建[26-27]。
在植物中,科学家同样检测了CRISPR-Cpf1的基因编辑功能。本课题组[28]对两种Cpf1,即来源于毛螺科菌ND2006 Lachnospiraceae bacterium ND2006的Cpf1 (LbCpf1) 和氨基酸球菌属BV3L6 Acidaminococcus sp. BV3L6的Cpf1 (AsCpf1) 进行了密码子优化,同时配套以适于Cpf1作用的crRNA表达系统,构建载体对水稻基因组中的多个位点进行靶向切割。实验结果显示LbCpf1能够完成对水稻基因组的有效编辑,但未检测到AsCpf1的基因编辑能力。Xu等[29]和Tang等[30]同样报道了LbCpf1在水稻中的基因编辑能力。Endo等[31]报道在水稻和烟草用弗朗西斯菌U112 Francisella novicida U112 Cpf1 (FnCpf1) 进行了基因敲除。Kim等[32]将LbCpf1或AsCpf1和crRNA在体外组装完成后导入大豆和烟草的原生质体中,成功对目标基因进行了编辑。最近,Wang等[33]发现FnCpf1和LbCpf1只需一条非常短的20–21 bp的直接重复序列(Direct repeats,DR)加上22–24 bp的靶位点识别序列(Guide)即可实现单基因敲除;把多个DR-guide单元直接串联,只需要一个启动子驱动即可简单高效地实现多基因敲除。该系统在载体构建上比CRISPR-Cas9系统更加简单易行。短期内大量的研究结果表明,CRISPR-Cpf1在植物基因组编辑上具有广阔的应用前景。
2.3 其他CRISPR-Cas9系统除了SpCas9,还有一些不同来源的CRISPR-Cas系统,也具有基因编辑能力。通过比较基因组,科学家们在600个Cas9同源序列中找到了一个较小的Cas9蛋白——一种来自金黄色葡萄球菌Staphylococcus aureus的Cas9核酸酶(SaCas9),它比SpCas9小25%,且和SpCas9具有相当的DNA靶向精确度[34]。SaCas9与其配套的gRNA识别的PAM序列为NNGRRT。此外,还有一个来自嗜热乳链球菌Streptococcus thermophilus的Cas9 (St1Cas9),识别的PAM序列为NNAGAA[35-36]。这些SpCas9同源蛋白的发现,为基因编辑及调控提供了更多选择,同时也扩大了基因组编辑范围。Steinert等[37]报道经密码子优化的SaCas9和St1Cas9在拟南芥中具有较高的基因编辑效率。
3 Cas9切口酶和失活酶的拓展应用Cas9蛋白有RuvC和HNH两个核酸切割结构域,将这两个结构域中的任一关键残基转换成丙氨酸(D10A或H840A),就获得只对DNA单链进行切割的切口酶nickase Cas9 (nCas9);将这两个催化活性位点都突变,就可得到仍能与sgRNA结合识别特异靶位点但不会对目标序列进行切割的失活Cas9蛋白dead Cas9 (dCas9)。
Ran等[38]在人类细胞中,运用2条相近的gRNA引导一对nCas9分别在DNA两条链上形成缺口,造成DNA双链断裂从而产生突变,大大降低了单个Cas9造成的脱靶率。Chen等[39]将绿色荧光蛋白(GFP)与dCas9融合,利用CRISPR-Cas9系统的基因定位功能来研究基因组特定区段的染色体结构。dCas9通过融合具有转录调控功能的转录激活因子或转录抑制因子,可以作为调控基因表达水平的控制元件,形成研究基因表达调控的有效工具[40],在植物中同样有效[41]。此外,还可以融合诸如乙酰化酶、甲基化酶等功能元件,开发出许多基因调控及修饰的工具[42-44]。
4 特异性单碱基突变编辑系统的研究核苷酸点突变是作物许多重要农艺性状发生变异的遗传基础。大多数时候科学家希望通过引入单碱基突变造成氨基酸的变化达到基因功能的增强或减弱,而不是单纯地敲除基因功能。由于目前定向同源修复(HDR)的编辑效率低,特异性单碱基突变编辑系统的研究就十分必要。
Komor等[45]报道,将nCas9 (nCas9-D10A)融合大鼠胞嘧啶脱氨酶(rAPOBEC1) 和尿嘧啶糖基化酶抑制剂(UGI),构成了高效的单碱基编辑系统BE3 (APOBEC-XTEN-nCas9 (D10A)-UGI)。在人细胞系和小鼠细胞系中,利用该系统将碱基胞嘧啶(C)转化为碱基尿嘧啶(U),随后在一轮DNA复制中,这种尿嘧啶(U)被转化为胸腺嘧啶(T),最终实现PAM窗口前–11 bp至–17 bp靶序列内C至T的替换(图 1)。此外,Nishida等[46]报道,通过将nCas9-D10A与七鳃鳗(Sea lamprey)的胞苷脱氨酶(Activation-induced cytidine deaminase,AID)融合表达,也同样实现PAM窗口前–19 bp至–16 bp靶序列内C至T的替换。Kim等[47]通过融合Cas9变体或SaCas9,增加了CRISPR基因靶向的范围,而且将编辑框从5个核苷酸缩小到1–2个核苷酸,增加了碱基编辑的精确度。
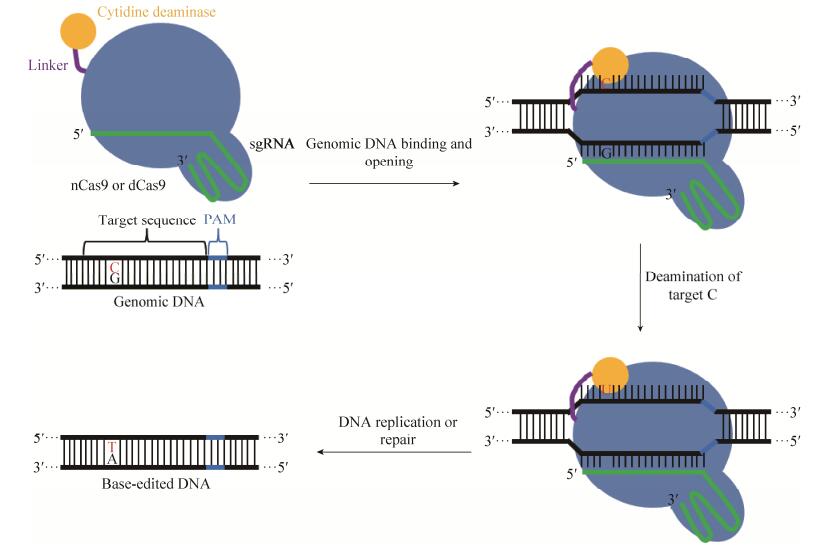
|
| 图 1 特异性单碱基突变编辑示意图 Figure 1 The schematic diagram of precise base editing. |
| |
借鉴哺乳动物单碱基编辑方法,Li[48]和Lu[49]率先报道在水稻中利用BE3系统成功实现了单碱基编辑。同样利用该系统,Ren等[50]在水稻中,Chen等[51]在拟南芥中,Zong等[52]在三大重要农作物(小麦、水稻和玉米)基因组中都实现了高效、精确的单碱基定点突变。Shimatani等[53]则利用AID系统在番茄和水稻中实现了单碱基编辑。单碱基编辑系统的成功建立和应用,为高效和大规模创制单碱基突变体提供了一个可靠方案,为作物遗传改良和新品种培育提供了重要技术支撑。但是该编辑系统不仅受到PAM和编辑窗口的限制,还受到只能由C替换为A的限制,目前还不能做到基因组上任意位点的单碱基精准编辑。
5 无外源DNA污染的植物基因编辑技术应用常规植物基因组编辑手段多通过农杆菌或基因枪的方法将CRISPR-Cas9 DNA表达框转入并整合到植物基因组中,进而发挥功能对目的基因进行编辑。尽管通过后代分离方可获得无CRISPR-Cas9 DNA的植物突变体,但依然存在外源DNA污染的风险。Woo等[54]尝试将Cas9蛋白和gRNA在体外组装成核糖核蛋白复合体(RNP),再通过PEG转化法将RNP转入水稻、生菜、烟草等植物原生质体,成功对目的基因进行了编辑,并且通过原生质体再生,T0代就获得了无任何外源DNA残留的定点编辑成功的生菜植株,为如何避免转基因成分残留提供了新思路。
同样,Liang等[55]利用基因枪法将CRISPR-Cas9 RNP转入小麦细胞中,在两个六倍体小麦品种中分别对两个不同基因tagw2和tagasr7进行了定点编辑,成功地在小麦中建立了全程无外源DNA的基因组编辑体系。与此同时,CRISPR-Cas9的RNP可以明显降低脱靶效应。这种DNA-free的基因组编辑方法具有精准、特异、简单易行、成本低廉的优势,并且成功避免了外源DNA片段整合到基因组中的潜在风险。这一利用CRISPR-Cas9 RNP实现小麦基因组编辑的方法有助于最大程度地减少监管,建立起精准、生物安全的新一代育种技术体系,加快作物基因组编辑育种产业化进程。
6 基因组编辑技术在作物育种上的应用植物基因组编辑技术已经为我们创造了一些之前很难获得的新品种。例如,在异源六倍体小麦中,存在3个白粉病感病基因TaMLO拷贝,通过传统的育种手段,几乎拿不到这3个拷贝同时突变的抗病材料。Wang等[56]利用基因组编辑技术,首次在六倍体小麦中对TaMLO基因的3个拷贝同时进行了突变,获得了对白粉病具有广谱抗性的小麦材料,在小麦农业生产上具有重大意义。Zhou等[57]在水稻中通过CRISPR-Cas9系统敲除温敏型雄性不育(TGMS)基因,开发了11个新的雄性不育品系,仅在一年内就可以应用于两个水稻亚种的杂交育种,不仅显著加快了不育系的培育过程,而且也有利于杂种优势的利用。Shi等[58]在玉米中利用CRISPR-Cas9系统改造乙烯反应的负调控因子ARGOS8基因的启动子区域,获得了新的抗旱玉米品种。
宾夕法尼亚大学的杨亦农实验室利用CRISPR-Cas9技术,在白蘑菇中将容易引起褐变的多酚氧化酶(PPO)的编码基因敲除了1个,将该酶活性降低了30%,从而获得了不易褐变的白蘑菇。美国杜邦先锋公司通过CRISPR-Cas9技术敲除控制直链淀粉合成的Waxy1基因获得了糯玉米新品种。美国农业部于2016年4月批文,表示无需对基因编辑技术改造的这两例农作物进行监管,意味着得到美国政府上市许可的基因组编辑生物可以直接用于种植和销售,无需额外的监管程序,为基因编辑技术用于农产品的商业生产带来了光明前景[59]。
与此同时,在作物育种上应用基因编辑技术也需要严格的实验。本课题组对水稻产量数量性状基因(Quantitative trait locus,QTL)进行定点编辑研究时,发现在不同遗传背景的水稻中进行QTL编辑会产生不同的产量变化[60]。该研究结果显示,对特定性状进行编辑时,在不同的遗传背景下可能会得到不一样的结果。
7 结语与展望基因组编辑技术是继转基因技术之后在生物遗传操作领域的又一革命性技术。短短几年时间的发展,基于CRISPR-Cas9系统的基因编辑技术已经在植物基础生物学研究及作物育种上显现出了巨大的应用潜力,解决了许多之前由于无法获得定点突变材料造成的难题,也已经创造了一些优良品种。与转基因品种不同,通过基因组编辑技术得到的品种不引入外源基因,具有与常规诱变品种无异的优点,因此在作物改良的生产应用上更为安全。可以预见,基因组编辑技术在作物育种上具有十分广阔的应用前景。
与此同时,还存在多个急需克服的难题。目前基因组编辑技术主要是产生内源基因功能缺失的突变体,对植物内源基因进行更为精确的修饰,如基因定点替换以及基因的定点插入等,仍然具有极大的挑战性。对基因定点替换及基因的定点插入在植物中也有研究报道:Li等[61]在水稻中利用非同源末端连接修复方式在水稻中建立了基于CRISPR-Cas9技术的基因替换以及基因定点插入体系,对5-烯醇丙酮酰莽草酸-3-磷酸合成酶基因(OsEPSPS)进行了定点替换,获得了抗草甘膦除草剂的水稻;Sun等[62]通过提供外源DNA供体利用同源重组修复对乙酰乳酸合酶基因(OsALS)进行了定点替换,获得了抗嘧啶羧酸类除草剂的水稻;Wang等[63]利用双生病毒在植物体内的复制扩增DNA的能力,结合CRISPR-Cas9系统,在水稻中将带有潮霉素抗性的一段序列插入了特定的位置。然而,在植物中进行插入或替换的工作仍然受限于获得抗除草剂或其他抗性性状,目前尚无法做到在基因组内任一位点上高效地替换或插入任意序列,这限制了基因组编辑技术在植物基因组学研究和农作物分子设计育种中的应用。我们相信在不久的将来,随着基因组编辑技术的不断发展,这些问题会逐步被解决,将有助于人们更好地了解植物基因的功能并创造新的优良品种。
| [1] | Bibikova M, Beumer K, Trautman JK, et al. Enhancing gene targeting with designed zinc finger nucleases. Science, 2003, 300(5620): 764. DOI: 10.1126/science.1079512 |
| [2] | Hockemeyer D, Wang HY, Kiani S, et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat Biotechnol, 2011, 29(8): 731–734. DOI: 10.1038/nbt.1927 |
| [3] | Huang P, Xiao A, Zhou MG, et al. Heritable gene targeting in zebrafish using customized TALENs. Nat Biotechnol, 2011, 29(8): 699–700. DOI: 10.1038/nbt.1939 |
| [4] | Nakata A, Amemura M, Makino K. Unusual nucleotide arrangement with repeated sequences in the Escherichia coli K-12 chromosome. J Bacteriol, 1989, 171(6): 3553–3556. DOI: 10.1128/jb.171.6.3553-3556.1989 |
| [5] | Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709–1712. DOI: 10.1126/science.1138140 |
| [6] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA. Science, 2008, 322(5909): 1843–1845. DOI: 10.1126/science.1165771 |
| [7] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816–821. DOI: 10.1126/science.1225829 |
| [8] | Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823–826. DOI: 10.1126/science.1232033 |
| [9] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819–823. DOI: 10.1126/science.1231143 |
| [10] | Shan Q, Wang YP, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 686–688. DOI: 10.1038/nbt.2650 |
| [11] | Li JF, Norville JE, Aach J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9. Nat Biotechnol, 2013, 31(8): 688–691. DOI: 10.1038/nbt.2654 |
| [12] | Nekrasov V, Staskawicz B, Weigel D, et al. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nat Biotechnol, 2013, 31(8): 691–693. DOI: 10.1038/nbt.2655 |
| [13] | Feng ZY, Zhang BT, Ding WN, et al. Efficient genome editing in plants using a CRISPR/Cas system. Cell Res, 2013, 23(10): 1229–1232. DOI: 10.1038/cr.2013.114 |
| [14] | Miao J, Guo DS, Zhang JZ, et al. Targeted mutagenesis in rice using CRISPR-Cas system. Cell Res, 2013, 23(10): 1233–1236. DOI: 10.1038/cr.2013.123 |
| [15] | Ma XL, Zhang QY, Zhu QL, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants. Mol Plant, 2015, 8(8): 1274–1284. DOI: 10.1016/j.molp.2015.04.007 |
| [16] | Xie K, Minkenberg B, Yang Y. Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system. Proc Natl Acad Sci USA, 2015, 112(11): 3570–3575. DOI: 10.1073/pnas.1420294112 |
| [17] | Wang C, Shen L, Fu YP, et al. A simple CRISPR/Cas9 system for multiplex genome editing in rice. J Genet Genomics, 2015, 42(12): 703–706. DOI: 10.1016/j.jgg.2015.09.011 |
| [18] | Shen L, Hua YF, Fu YP, et al. Rapid generation of genetic diversity by multiplex CRISPR/Cas9 genome editing in rice. Sci China Life Sci, 2017, 60(5): 506–515. DOI: 10.1007/s11427-017-9008-8 |
| [19] | Yan LH, Wei SW, Wu YR, et al. High-efficiency genome editing in Arabidopsis using YAO promoter-driven CRISPR/Cas9 system. Mol Plant, 2015, 8(12): 1820–1823. DOI: 10.1016/j.molp.2015.10.004 |
| [20] | Wang ZP, Xing HL, Dong L, et al. Egg cell-specific promoter-controlled CRISPR/Cas9 efficiently generates homozygous mutants for multiple target genes in Arabidopsis in a single generation. Genome Biol, 2015, 16: 144. DOI: 10.1186/s13059-015-0715-0 |
| [21] | Mao YF, Zhang ZJ, Feng ZY, et al. Development of germ-line-specific CRISPR-Cas9 systems to improve the production of heritable gene modifications in Arabidopsis. Plant Biotechnol J, 2016, 14(2): 519–532. DOI: 10.1111/pbi.2016.14.issue-2 |
| [22] | Hilscher J, Bürstmayr H, Stoger E. Targeted modification of plant genomes for precision crop breeding. Biotechnol J, 2017, 12(1): 1600173. DOI: 10.1002/biot.201600173 |
| [23] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 2015, 523(7561): 481–485. DOI: 10.1038/nature14592 |
| [24] | Hu XX, Wang C, Fu YP, et al. Expanding the range of CRISPR/Cas9 genome editing in rice. Mol Plant, 2016, 9(6): 943–945. DOI: 10.1016/j.molp.2016.03.003 |
| [25] | Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759–771. DOI: 10.1016/j.cell.2015.09.038 |
| [26] | Fonfara I, Richter H, Bratovič M, et al. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature, 2016, 532(7600): 517–521. DOI: 10.1038/nature17945 |
| [27] | Zetsche B, Heidenreich M, Mohanraju P, et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nat Biotechnol, 2016, 35(1): 31–34. DOI: 10.1038/nbt.3737 |
| [28] | Hu XX, Wang C, Liu Q, et al. Targeted mutagenesis in rice using CRISPR-Cpf1 system. J Genet Genomics, 2017, 44(1): 71–73. DOI: 10.1016/j.jgg.2016.12.001 |
| [29] | Xu RF, Qin RY, Li H, et al. Generation of targeted mutant rice using a CRISPR-Cpf1 system. Plant Biotechnol J, 2017, 15(5): 713–717. DOI: 10.1111/pbi.12669 |
| [30] | Tang X, Lowder LG, Zhang T, et al. A CRISPR-Cpf1 system for efficient genome editing and transcriptional repression in plants. Nat Plants, 2017, 3: 17018. DOI: 10.1038/nplants.2017.18 |
| [31] | Endo A, Masafumi M, Kaya H, et al. Efficient targeted mutagenesis of rice and tobacco genomes using Cpf1 from Francisella novicida. Sci Rep, 2016, 6: 38169. DOI: 10.1038/srep38169 |
| [32] | Kim H, Kim ST, Ryu J, et al. CRISPR/Cpf1-mediated DNA-free plant genome editing. Nat Commun, 2017, 8: 14406. DOI: 10.1038/ncomms14406 |
| [33] | Wang MG, Mao YF, Lu YM, et al. Multiplex gene editing in rice using the CRISPR-Cpf1 system. Mol Plant, 2017. DOI: 10.1016/j.molp.2017.03.001 |
| [34] | Ran FA, Cong L, Yan WX, et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature, 2015, 520(7546): 186–191. DOI: 10.1038/nature14299 |
| [35] | Deveau H, Barrangou R, Garneau JE, et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. J Bacteriol, 2008, 190(4): 1390–1400. DOI: 10.1128/JB.01412-07 |
| [36] | Horvath P, Romero DA, Coté-Monvoisin AC, et al. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus. J Bacteriol, 2008, 190(4): 1401–1412. DOI: 10.1128/JB.01415-07 |
| [37] | Steinert J, Schiml S, Fauser F, et al. Highly efficient heritable plant genome engineering using Cas9 orthologues from Streptococcus thermophilus and Staphylococcus aureus. Plant J, 2015, 84(6): 1295–1305. DOI: 10.1111/tpj.13078 |
| [38] | Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380–1389. DOI: 10.1016/j.cell.2013.08.021 |
| [39] | Chen BH, Gilbert LA, Cimini BA, et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell, 2013, 155(7): 1479–1491. DOI: 10.1016/j.cell.2013.12.001 |
| [40] | Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442–451. DOI: 10.1016/j.cell.2013.06.044 |
| [41] | Piatek A, Ali Z, Baazim H, et al. RNA-guided transcriptional regulation in planta via synthetic dCas9-based transcription factors. Plant Biotechnol J, 2015, 13(4): 578–589. DOI: 10.1111/pbi.12284 |
| [42] | Kearns NA, Pham H, Tabak B, et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods, 2015, 12(5): 401–403. DOI: 10.1038/nmeth.3325 |
| [43] | Hilton IB, D'Ippolito AM, Vockley CM, et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol, 2015, 33(5): 510–517. DOI: 10.1038/nbt.3199 |
| [44] | Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol, 2014, 32(4): 347–355. DOI: 10.1038/nbt.2842 |
| [45] | Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420–424. DOI: 10.1038/nature17946 |
| [46] | Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems. Science, 2016, 353(6305): aaf8729. DOI: 10.1126/science.aaf8729 |
| [47] | Kim YB, Komor AC, Levy JM, et al. Increasing the genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions. Nat Biotechnol, 2017, 35(4): 371–376. DOI: 10.1038/nbt.3803 |
| [48] | Li JY, Sun YW, Du JL, et al. Generation of targeted point mutations in rice by a modified CRISPR/Cas9 system. Mol Plant, 2017, 10(3): 526–529. DOI: 10.1016/j.molp.2016.12.001 |
| [49] | Lu YM, Zhu JK. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system. Mol Plant, 2017, 10(3): 523–525. DOI: 10.1016/j.molp.2016.11.013 |
| [50] | Ren B, Yan F, Kuang YJ, et al. A CRISPR/Cas9 toolkit for efficient targeted base editing to induce genetic variations in rice. Sci China Life Sci, 2017, 60(5): 516–519. DOI: 10.1007/s11427-016-0406-x |
| [51] | Chen YY, Wang ZP, Ni HW, et al. CRISPR/Cas9-mediated base-editing system efficiently generates gain-of-function mutations in Arabidopsis. Sci China Life Sci, 2017, 60(5): 520–523. DOI: 10.1007/s11427-017-9021-5 |
| [52] | Zong Y, Wang YP, Li C, et al. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion. Nat Biotechnol, 2017, 35(5): 438–440. DOI: 10.1038/nbt.3811 |
| [53] | Shimatani Z, Kashojiya S, Takayama M, et al. Targeted base editing in rice and tomato using a CRISPR-Cas9 cytidine deaminase fusion. Nat Biotechnol, 2017, 35(5): 441–443. DOI: 10.1038/nbt.3833 |
| [54] | Woo J W, Kim J, Kwon SI, et al. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins. Nat Biotechnol, 2015, 33(11): 1162–1164. DOI: 10.1038/nbt.3389 |
| [55] | Liang Z, Chen KL, Li TD, et al. Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes. Nat Commun, 2017, 8: 14261. DOI: 10.1038/ncomms14261 |
| [56] | Wang YP, Cheng X, Shan QW, et al. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nat Biotechnol, 2014, 32(9): 947–951. DOI: 10.1038/nbt.2969 |
| [57] | Zhou H, He M, Li J, et al. Development of commercial thermo-sensitive genic male sterile rice accelerates hybrid rice breeding using the CRISPR/Cas9-mediated TMS5 editing system. Sci Rep, 2016, 6: 37395. DOI: 10.1038/srep37395 |
| [58] | Shi JR, Gao HR, Wang HY, et al. ARGOS8 variants generated by CRISPR-Cas9 improve maize grain yield under field drought stress conditions. Plant Biotechnol J, 2017, 15(2): 207–216. DOI: 10.1111/pbi.2017.15.issue-2 |
| [59] | Waltz E. Gene-edited CRISPR mushroom escapes US regulation. Nature, 2016, 532(7599): 293. DOI: 10.1038/nature.2016.19754 |
| [60] | Shen L, Wang C, Fu YP, et al. QTL editing confers opposing yield performance in different rice varieties. J Integr Plant Biol, 2016. DOI: 10.1111/jipb.12501 |
| [61] | Li J, Meng XB, Zong Y, et al. Gene replacements and insertions in rice by intron targeting using CRISPR-Cas9. Nat Plants, 2016, 2: 16139. DOI: 10.1038/nplants.2016.139 |
| [62] | Sun YW, Zhang X, Wu CY, et al. Engineering herbicide-resistant rice plants through CRISPR/ Cas9-Mediated homologous recombination of acetolactate synthase. Mol Plant, 2016, 9(4): 628–631. DOI: 10.1016/j.molp.2016.01.001 |
| [63] | Wang MG, Lu YM, Botella JR, et al. Gene targeting by homology-directed repair in rice using a geminivirus-based CRISPR/Cas9 system. Mol Plant, 2017. DOI: 10.1016/j.molp.2017.03.002 |
 2017, Vol. 33
2017, Vol. 33




