中国科学院微生物研究所、中国微生物学会主办
文章信息
- 金芸, 张涛, 郭惠珊
- Jin Yun, Zhang Tao, Guo Huishan
- 基因沉默技术在抗真菌病害中的应用和展望
- Application of host induced gene silencing in crop protection against fungal diseases
- 生物工程学报, 2017, 33(2): 161-169
- Chin J Biotech, 2017, 33(2): 161-169
- 10.13345/j.cjb.160403
-
文章历史
- Received: October 27, 2016
- Accepted: December 1, 2016
作物真菌病害通常会造成植物组织的坏死、腐烂或萎蔫等症状,严重威胁农作物的生长发育。其中水稻稻瘟病、大豆锈病、小麦锈病、玉米丝黑穗病和马铃薯晚疫病已经使得五大粮食作物的产量在全球范围内每年减少1.25亿t,成为全球性粮食安全问题。据专家推算,假如在极端情况下上述五大粮食作物在一年中同时出现真菌病害大流行,则当年全球粮食减产可能高达9亿t,这将导致42亿人粮食短缺,出现全球饥荒[1]。除此以外,其他一些农作物的真菌病害,比如棉花黄萎病,亦会导致棉花大面积减产甚至绝收,并影响棉纤维的品质,在农业和工业等领域造成巨大的经济损失。
传统的作物抗病技术包括施用农药和培育抗病品种等手段。但是,农药的施用不仅造成生态环境的破坏,而且大量使用会导致病原物产生抗药性,更重要的是,对于一些土传真菌病害,农药的使用效果并不佳,难以满足植物保护的需求;另一方面,虽然常规育种在获得抗病、农艺性状优良的新品种方面已取得了很大成就,大大促进了作物生产,但由于育种周期长,所以仅依靠常规育种很难适应目前作物生产对抗病性的要求;且有些农艺性状优良的主栽品种缺乏抗源种质。因此,探寻新的防治手段势在必行。近年来,基因工程快速发展,使得运用生物技术对多种病虫害进行防控变得有效可行。利用新型基因调控技术培育具有抗性的农作物新品种,是现代农业生产利用高科技提高农作物产量、保护生态环境和促进农业可持续发展的关键措施之一,也是目前世界各国的重点攻关方向。
2 RNA介导的基因沉默原理及真菌中的RNA沉默研究进展RNA介导的基因沉默即RNA干扰(RNA interference,RNAi),是双链RNA或能形成发夹结构的单链RNA,被Dicer酶识别并切割成小分子RNA,进而与Agonaute蛋白等形成沉默复合物,以同源序列配对的形式靶向目标基因,在转录水平或转录后水平下调基因表达。基因沉默现象的机制和功能在动植物中都有广泛深入的研究。但真菌中的RNA沉默途径研究远远落后于动植物,只在几种模式真菌中有较深入的报道。例如,在模式丝状真菌——粗糙脉胞菌Neurospora crassa中首先发现了外源转基因的沉默,被称为quelling现象[2],进而发现了影响quelling发生的一系列qde突变体(Quelling deficient mutants) 并克隆了相关基因[3-6]。在N. crassa中还相继发现了受DNA损伤诱导的qiRNA (QDE-2-interacting sRNA)[7],以及类似microRNA (miRNA-like) 的小RNA (milRNA),且仅有的4个主要milRNAs的合成途径各不相同,功能也未知[8]。另一种模式真菌——卷枝毛霉菌Mucor circinelloides,对其编码的DCL (Dicer-like)、RdRP (RNA dependent RNA polymerase) 及AGO等RNA沉默途径相关蛋白都进行了相应的功能研究[9-14]。除此以外,其他被报道存在RNA沉默途径的真菌,尤其是植物致病真菌,大多集中在小RNA测序及数据分析的层面,未有深入的机制报道。究其原因可能是因为真菌中小RNA合成途径的多样性,以及RNA沉默途径的分化[15-16],导致真菌中的RNA沉默途径研究进展缓慢。
3 基因沉默技术在非真菌病害及真菌病害中的应用基因沉默技术已广泛应用于植物、动物及微生物的功能基因组学中。随着对植物和动物病毒的研究进展,RNAi被发现是动植物抵抗病毒的一种先天适应性免疫抗性[17],并被广泛地拓展到植物抗病毒的应用中[18]。近年来,研究者发现一些害虫能够被外源施加的靶向虫体必需基因的RNAi片段所杀死,进一步为运用RNAi技术进行植物保护提供了可能。其实,早在RNA沉默被认同的10年前,Sanford和Johnson就将病原基因片段转入植物或者动物寄主中并获得对相应病原的抗性,因此提出了parasite/pathogen-derived resistance (PDR) 的概念[19]。随后人们发现,转入双链的病原基因片段能够获得更有效的抗病效果,这就是目前应用最广泛的被称为HD-RNAi (Host-delivered RNAi) 或宿主诱导的基因沉默(Host induced gene silencing,HIGS) 的策略。其基本原理就是在宿主中表达靶向害虫或者病原菌致病基因的RNAi载体,沉默病原靶标基因,从而使宿主获得对病原的抗性。后来的研究证实了这种方法不仅可以有效地用于抵御病毒入侵,在害虫防治以及对抗细菌和真菌的侵染中也同样有效[20]。
显而易见,大部分病毒因其本身在宿主细胞质中复制,有利于宿主利用RNAi对其进行沉默;线虫、果蝇等也可以通过咀嚼实现宿主诱导的基因沉默;但由于真菌的侵染过程及侵染结构的复杂多变,使得在真菌中的应用难度剧增[21]。直到2010年,Nowara将表达靶向真菌葡聚糖转移酶基因的双链RNA转入大麦中,成功地增强了大麦对白粉病的抗性[22],才正式开启了基因沉默技术在真菌病害中的应用大门。2011年,研究者们利用大麦条纹花叶病毒系统(Barley stripe mosaic virus,BSMV) 将小麦条锈菌(Puccinia striiformis f. sp.tritici) 的基因片段转入寄主小麦细胞中,在锈菌吸器中表达较高的靶标基因会被下调,意味着寄主诱导的RNA干扰系统在获得高抗条锈病的小麦中有着巨大的应用潜力[23];2013年,运用农杆菌介导的瞬时表达系统将含有发夹结构的叶锈菌Puccinia triticina基因PtMAPK1、PtCYC1和PtCNB转入小麦叶片中,产生特异靶向锈菌基因的siRNA,观察发现真菌的生物量及孢子产生减少,并且小麦叶片病症减轻,在感病小麦中的锈菌靶基因的表达下调[24],同时利用病毒介导的RNAi系统传递叶锈菌基因片段至小麦中,也能提高小麦对锈病的抗性[25];同年研究者们利用HIGS在镰刀菌引起的抗枯萎病中也取得成功,他们在拟南芥和大麦中表达细胞色素P450羊毛甾醇C14α-脱甲基酶基因CYP51的RNAi结构,增强了寄主对镰刀菌属的抗性[26];紧接着2014年和2015年,研究者们分别在香蕉和拟南芥中表达靶向镰刀菌基因的RNAi也有效地增强了对枯萎病的抗性[27-28];2016年,在拟南芥和番茄中利用HIGS沉默灰霉菌Botrytis cinerea的DCL基因,降低了灰霉菌的生长和致病性[29]。
虽然宿主诱导的基因沉默在作物抗病方面已经被证实是有效的,但是没有明确的分子实验证据和理论依据。对于抵抗病原真菌,尤其是难以防治的土壤传播的真菌病害,其应用研究才刚刚开始,科学家们正在更多病原真菌中进行实践探索。同时,对于这种宿主来源的小RNA进入病原菌中的作用机制还未有明确的阐述,这一领域正吸引着国内外的大量科学家投身其中进行深入的研究。下面将就近期本研究室利用HIGS抵抗棉花黄萎病,在基础理论和实践应用两方面所取得的重大进展,对HIGS在棉花抵抗土传病害的有效性进行详述[30]。
4 利用基因沉默技术抵御棉花黄萎病的最新研究进展 4.1 棉花黄萎病简介棉花黄萎病是一种土传真菌病害[31-33],广泛分布于世界各地,是棉花的主要维管束病害。1914年首先在美国弗吉尼亚州的陆地棉上发现,目前已遍及美国、巴西、西班牙、澳大利亚、俄罗斯以及中国等几十个国家[34]。黄萎病已经成为棉花生产上第一大病害且是最具毁灭性的病害之一,其具有分布广、危害重、传播途径多、存活时间久、化学农药难于防治、生理小种变异多和防治难度大等特点[35-36],被称为棉花的癌症[37]。我国棉花年产量600万t−800万t,约占世界总产量的30%,居全球首位,受黄萎病危害最为严重。作为中国最大的优质棉生产基地,新疆棉花种植面积2015年就达230万hm2,但黄萎病发病面积超过50%。因此棉花黄萎病已经成为当前制约我国棉花生产的突出问题,对棉花生产构成极大的威胁。
4.2 棉花黄萎病主要致病菌——大丽轮枝菌及其侵染过程简介棉花黄萎病是由轮枝属真菌引起的。在我国,大丽轮枝菌(Verticillium dahlia Kleb.) 是棉花黄萎病的主要致病菌。大丽轮枝菌主要以孢子、菌丝和微菌核等形式存在于土壤或植物病残体中[38],但是在逆境的生长条件下,微菌核是其主要存在方式,这种休眠结构可抵抗土壤中包括化学的、物理的各种不利环境条件,存活达15年之久[39],而在合适的环境或者有寄主根的分泌物刺激的条件下会开始萌发进入新一轮的生命周期。
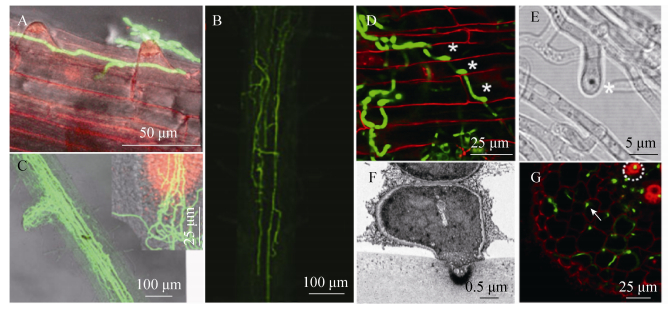
微菌核萌发形成侵染菌丝穿透植物根部,并在木质部维管束生长,产生大量菌丝和孢子,这些孢子会随着寄主植物的蒸腾作用向顶运动,不断侵入寄主的茎及叶片,最终侵染植物的整个地上部分[40],导致感病植株产生萎蔫、褪绿、黄化、组织坏死、生长迟缓和导管褐化等黄萎病的发病症状[41]。本研究室利用激光共聚焦扫描电镜技术观察了大丽轮枝菌侵染拟南芥的过程,发现V. dahliae侵染初期,孢子随机附着于根表面,随后孢子萌发并产生菌丝缠绕于根尖,此阶段(2-days post inoculation,2 dpi) 仅极少数紧贴根表面萌发的菌丝能够进入表皮细胞(图 1A)。随后,通过皮层组织进入维管组织的少数菌丝快速生长,在维管中渐渐形成网络状菌丝复合体(5 dpi) (图 1B)。此后菌丝在维管组织中双向快速生长,一方面到达地上组织,造成病变,另一方面沿着维管向根尖生长。在感染晚期,到达根尖的菌丝导致根冠组织的崩溃(图 1C)。实验表明,虽然V. dahliae菌丝侵入根表皮组织细胞比率极低,但是由于其在维管组织中强大的增殖能力,少量成功侵染维管组织的菌丝,也足以引起维管束阻塞,导致棉花黄萎病的发生[42]。最近,本研究室进一步发现,大丽轮枝菌在侵染植物根时,少量的菌丝分化出侵染结构——附着枝(Hyphopodium) (图 1D、E星号所示),附着枝特异地产生活性氧(ROS) 上调钙离子浓度,激活下游转录因子(Crz1),进而诱导其自身的极性生长,在基部形成侵染钉(Penetration peg) (图 1F) 起始对宿主棉花的侵染,通过对棉花根部横切同样观察到在维管组织大量繁殖的菌丝(图 1G)[43]。
|
| 图 1 大丽轮枝菌侵染过程及侵染结构——附着枝和侵入钉 Figure 1 Observation of root colonization process and the specific infection structure——hyphopodium and penetration peg of Verticillium dahliae. |
| |
由于土壤中含有大量大丽轮枝菌微菌核,一旦接触棉花的根便开始萌发,并起始侵染,因此棉花和病原的相互作用持续于棉花的整个生长期,利用HIGS进行抗棉花黄萎病的探索充满了神秘和挑战。棉花产生的小RNA是否能够跨界进入真菌?大丽轮枝菌是否存在RNAi机制并能利用外来的小RNA诱导基因沉默?为了探讨这些问题,利用以上对大丽轮枝菌侵染机制和过程的认识,本研究室建立了从感病棉花中分离菌丝和RNA等技术平台;从感病棉花中分离的大丽轮枝菌中,检测到很多来源于棉花的内源小RNA (miRNAs),并调控真菌靶基因的下调[44]。这是国际上首次证明了植物内源miRNA的跨界调控。研究同时发现敲除miRNA在真菌中的靶标基因大大减弱大丽轮枝菌的致病性,证明该靶基因是致病基因,表明跨界RNAi是棉花和大丽轮枝菌的互作过程中自然存在的抗病途径;也证明了大丽轮枝菌中存在RNAi途径,为利用HIGS开展棉花抗黄萎病研究提供了重要的理论依据。
因此,本研究室挑选了一个潜在的靶标——大丽轮枝菌的疏水蛋白基因Hydrophobin1 (VdH1),进行了HIGS在棉花抗大丽轮枝菌中的应用效果和机制的探索。首先,敲除VdH1基因的突变体(VdaΔvdh1) 不产生微菌核,能够侵染棉花并在棉花中扩繁增殖,但其引起的黄萎病的症状大幅减轻,证明它是一个致病相关基因。进而将构建的靶向VdH1基因RNAi (VdH1i) 转入棉花中,得到了能够稳定表达来源于VdH1i的siRNA的转基因棉花。抗病检测显示,相比于野生型,转基因棉花无论在实验室条件下,还是大田病圃中都表现出高效的抗黄萎病的抗性。本研究室的结果表明宿主棉花能够利用HIGS沉默大丽轮枝菌中的致病相关基因从而降低黄萎病的发病率。从大丽轮枝菌侵染后的VdH1i转基因棉花分离得到的菌丝中,检测到VdH1基因的降解以及VdH1-siRNA的积累,进一步证明宿主植物能够传输siRNA到真菌细胞中并且诱导目标基因的沉默。这也是国际上首次从分子水平揭示了宿主来源的小RNA跨界诱导病原基因沉默的基本原理[30]。
5 基因沉默技术在作物真菌病害中的应用前景分析及展望根据目前的研究进展,基因沉默技术在实验室环境下的抗病虫害效果已经得到广泛的证实,本研究室的发现也证明了HIGS用于对抗难以防治的土传真菌病害的有效性。由于传统的化学控制方法对根部侵染的病原菌几乎难以奏效,因此拓展HIGS的方法到其他根部侵染的病原菌的防治中非常值得尝试。同时,得益于真菌基因组的测序,基因功能的解析和对侵染过程及宿主抗病过程的深入了解,研究人员将可能挑选合适有效的靶标基因进行特定靶向沉默。因此,基因沉默技术具有非常光明的应用前景及切实可行的操作性。
当然,在实际应用于田间作物抗真菌病害中,尚面临着诸多的困难。首先,在自然条件下,植物面临的环境更为复杂,土壤中存在的多种微生物通常构成混合侵染。结合微生物组学等的研究,设计靶向多种病原的目标基因,或者选择高度保守的目标基因进行攻击,或可能实现更强的抗性。其次,宿主诱导基因沉默的机制的解析问题。RNA作为中心法则的中间一环,目前发现的功能及重要性远大于之前科学家的预测和想象,对于RNA介导的沉默,虽然有大量的研究人员在模式生物中进行研究,但对于RNAi的跨界调控,即RNA分子在2个互作物种之间的运输和诱导沉默的机制都有待阐明。再者,靶向的病菌是否存在RNA沉默途径也因病原菌而异。关键的分子机理的解析必将推动HIGS的抗病应用;这些或许尚待时日,但我们应该理论研究和应用探索齐头并进。
综上所述,针对化学农药难以控制的土传真菌病害,基因沉默技术的应用是一个从零到一的突破;是对传统应用抗性基因策略的一个有效的补充,尤其对于没有抗性资源的物种,HIGS更显其得天独厚的优势。因此,基因沉默技术在作物土传真菌病害中的应用将是未来抗病领域的重点攻关方向。
| [1] | Fisher MC, Henk DA, Briggs CJ, et al. Emerging fungal threats to animal, plant and ecosystem health. Nature, 2012, 484(7393): 186–194. DOI: 10.1038/nature10947 |
| [2] | Cogoni C, Irelan JT, Schumacher M, et al. Transgene silencing of the al-1 gene in vegetative cells of Neurospora is mediated by a cytoplasmic effector and does not depend on DNA-DNA interactions or DNA methylation. EMBO J, 1996, 15(12): 3153–3163. |
| [3] | Cogoni C, Macino G. Isolation of quelling-defective (qde) mutants impaired in posttranscriptional transgene-induced gene silencing in Neurospora crassa. Proc Natl Acad Sci USA, 1997, 94(19): 10233–10238. DOI: 10.1073/pnas.94.19.10233 |
| [4] | Cogoni C, Macino G. Gene silencing in Neurospora crassa requires a protein homologous to RNA-dependent RNA polymerase. Nature, 1999, 399(6732): 166–169. DOI: 10.1038/20215 |
| [5] | Cogoni C, Macino G. Posttranscriptional gene silencing in Neurospora by a RecQ DNA helicase. Science, 1999, 286(5448): 2342–2344. DOI: 10.1126/science.286.5448.2342 |
| [6] | Catalanotto C, Azzalin G, Macino G, et al. Involvement of small RNAs and role of the qde genes in the gene silencing pathway in Neurospora. Genes Dev, 2002, 16(7): 790–795. DOI: 10.1101/gad.222402 |
| [7] | Lee HC, Chang SS, Choudhary S, et al. qiRNA is a new type of small interfering RNA induced by DNA damage. Nature, 2009, 459(7244): 274–277. DOI: 10.1038/nature08041 |
| [8] | Lee HC, Li LD, Gu WF, et al. Diverse pathways generate MicroRNA-like RNAs and dicer-independent small interfering RNAs in fungi. Mol Cell, 2010, 38(6): 803–814. DOI: 10.1016/j.molcel.2010.04.005 |
| [9] | Nicolás FE, de Haro JP, Torres-Martínez S, et al. Mutants defective in a Mucor circinelloides dicer-like gene are not compromised in siRNA silencing but display developmental defects. Fungal Genet Biol, 2007, 44(6): 504–516. DOI: 10.1016/j.fgb.2006.09.003 |
| [10] | de Haro JP, Calo S, Cervantes M, et al. A single dicer Gene is required for efficient gene silencing associated with two classes of small antisense RNAs in Mucor circinelloides. Eukaryot Cell, 2009, 8(10): 1486–1497. DOI: 10.1128/EC.00191-09 |
| [11] | Nicolas FE, Moxon S, de Haro JP, et al. Endogenous short RNAs generated by Dicer 2 and RNA-dependent RNA polymerase 1 regulate mRNAs in the basal fungus Mucor circinelloides. Nucleic Acids Res, 2010, 38(16): 5535–5541. DOI: 10.1093/nar/gkq301 |
| [12] | Calo S, Nicolás FE, Vila A, et al. Two distinct RNA-dependent RNA polymerases are required for initiation and amplification of RNA silencing in the basal fungus Mucor circinelloides. Mol Microbiol, 2012, 83(2): 379–394. DOI: 10.1111/mmi.2012.83.issue-2 |
| [13] | Cervantes M, Vila A, Nicolás FE, et al. A single argonaute gene participates in exogenous and endogenous RNAi and controls cellular functions in the basal fungus Mucor circinelloides. PLoS ONE, 2013, 8(7): e69283. DOI: 10.1371/journal.pone.0069283 |
| [14] | Torres-Martínez S, Ruiz-Vázquez RM. RNAi pathways in Mucor: a tale of proteins, small RNAs and functional diversity. Fungal Genet Biol, 2016, 90: 44–52. DOI: 10.1016/j.fgb.2015.11.006 |
| [15] | Nicolás FE, Ruiz-Vázquez RM. Functional diversity of RNAi-associated sRNAs in fungi. Int J Mol Sci, 2013, 14(8): 15348–15360. DOI: 10.3390/ijms140815348 |
| [16] | Chang SS, Zhang ZY, Liu Y. RNA interference pathways in fungi: mechanisms and functions. Annu Rev Microbiol, 2012, 66(1): 305–323. DOI: 10.1146/annurev-micro-092611-150138 |
| [17] | Ding SW. RNA-based antiviral immunity. Nat Rev Immunol, 2010, 10(9): 632–644. DOI: 10.1038/nri2824 |
| [18] | Duan CG, Wang CH, Guo HS. Application of RNA silencing to plant disease resistance. Silence, 2012, 3(1): 5. DOI: 10.1186/1758-907X-3-5 |
| [19] | Sanford JC, Johnston SA. The concept of parasite-derived resistance-deriving resistance genes from the parasite's own genome. J Theor Biol, 1985, 113(2): 395–405. DOI: 10.1016/S0022-5193(85)80234-4 |
| [20] | Koch A, Kogel KH. New wind in the sails: improving the agronomic value of crop plants through RNAi-mediated gene silencing. Plant Biotechnol J, 2014, 12(7): 821–831. DOI: 10.1111/pbi.2014.12.issue-7 |
| [21] | Bailey AM. A silenced spring. Mol Plant Pathol, 2014, 15(7): 647–649. DOI: 10.1111/mpp.2014.15.issue-7 |
| [22] | Nowara D, Gay A, Lacomme C, et al. HIGS: host-induced gene silencing in the obligate biotrophic fungal pathogen Blumeria graminis. Plant Cell, 2010, 22(9): 3130–3141. DOI: 10.1105/tpc.110.077040 |
| [23] | Yin CT, Jurgenson JE, Hulbert SH. Development of a host-induced RNAi system in the wheat stripe rust fungus Puccinia striiformis f. sp. tritici. Mol Plant-Microbe Interact2011, 2011, 24(5): 554–561. DOI: 10.1094/MPMI-10-10-0229 |
| [24] | Panwar V, McCallum B, Bakkeren G. Endogenous silencing of Puccinia triticina pathogenicity genes through in planta-expressed sequences leads to the suppression of rust diseases on wheat. Plant J, 2013, 73(3): 521–532. DOI: 10.1111/tpj.12047 |
| [25] | Panwar V, McCallum B, Bakkeren G. Host-induced gene silencing of wheat leaf rust fungus Puccinia triticina pathogenicity genes mediated by the Barley stripe mosaic virus. Plant Mol Biol, 2013, 81(6): 595–608. DOI: 10.1007/s11103-013-0022-7 |
| [26] | Koch A, Kumar N, Weber L, et al. Host-induced gene silencing of cytochrome P450 lanosterol C14α-demethylase-encoding genes confers strong resistance to Fusarium species. Proc Natl Acad Sci USA, 2013, 110(48): 19324–19329. DOI: 10.1073/pnas.1306373110 |
| [27] | Ghag SB, Shekhawat UKS, Ganapathi TR. Host-induced post-transcriptional hairpin RNA-mediated gene silencing of vital fungal genes confers efficient resistance against Fusarium wilt in banana. Plant Biotechnol J, 2014, 12(5): 541–553. DOI: 10.1111/pbi.12158 |
| [28] | Hu ZL, Parekh U, Maruta N, et al. Down-regulation of Fusarium oxysporum endogenous genes by host-delivered RNA interference enhances disease resistance. Front Chem, 2015, 3: 1. |
| [29] | Wang M, Weiberg A, Lin FM, et al. Bidirectional cross-kingdom RNAi and fungal uptake of external RNAs confer plant protection. Nat Plants, 2016, 2(10): 16151. DOI: 10.1038/nplants.2016.151 |
| [30] | Zhang T, Jin Y, Zhao JH, et al. Host-induced gene silencing of the target gene in fungal cells confers effective resistance to the cotton wilt disease pathogen Verticillium dahliae. Mol Plant, 2016, 9(6): 939–942. DOI: 10.1016/j.molp.2016.02.008 |
| [31] | Klosterman SJ, Atallah ZK, Vallad GE, et al. Diversity, pathogenicity, and management of verticillium species. Annu Rev Phytopathol, 2009, 47(1): 39–62. DOI: 10.1146/annurev-phyto-080508-081748 |
| [32] | Xia ZJ, Achar PN, Gu BK. Vegetative compatibility groupings of Verticillium dahliae from cotton in mainland China. Eur J Plant Pathol, 1998, 104(9): 871–876. DOI: 10.1023/A:1008628209867 |
| [33] | Schnathorst WC, Mathre DE. Host range and differentiation of a severe form of Verticillium albo-atrum in cotton. Phytopathology, 1966, 56(10): 1155–1161. |
| [34] |
Liu XT, Song XX, Guo JC. Studies and advances on cotton Verticillium wilt.
Acta Gossypii Sin, 1998, 10(1): 6–13.
(in Chinese). 刘学堂, 宋晓轩, 郭金城. 棉花黄萎病菌的研究及最新进展. 棉花学报, 1998, 10(1): 6-13. |
| [35] | Latorre BA, Lolas M, Marholz G. Verticillium wilt, a limiting factor for tobacco production in chile. Plant Dis, 1989, 73(8): 664–666. DOI: 10.1094/PD-73-0664 |
| [36] | Johnson WM, Johnson EK, Brinkerhoff LA. Symptomatology and formation of microsclerotia in weeds inoculated with Verticillium dahliae from cotton. Phytopathology, 1980, 70(1): 31–35. DOI: 10.1094/Phyto-70-31 |
| [37] |
Chen JY, Dai XF. Research advance on the resistant mechanism of cotton against Verticillium wilt.
Mol Plant Breed, 2005, 3(3): 427–435.
(in Chinese). 陈捷胤, 戴小枫. 棉花对黄萎病的抗病机制研究进展. 分子植物育种, 2005, 3(3): 427-435. |
| [38] | Gutierrez AP, DeVay JE, Pullman GS, et al. A model of Verticillium wilt in relation to cotton growth and development. Phytopathology, 1983, 73(1): 89–95. DOI: 10.1094/Phyto-73-89 |
| [39] | Wilhelm S. Longevity of the Verticillium wilt fungus in the laboratory and field. Phytopathology, 1955, 45(3): 180–181. |
| [40] | Pegg G, Brady BL. Verticillium wilts. UK: CABI, 2002. |
| [41] | Fradin EF, Thomma BP. Physiology and molecular aspects of Verticillium wilt diseases caused by V. dahliae and V. albo-atrum. Mol Plant Pathol, 2006, 7(2): 71–86. DOI: 10.1111/mpp.2006.7.issue-2 |
| [42] | Zhao P, Zhao YL, Jin Y, et al. Colonization process of Arabidopsis thaliana roots by a green fluorescent protein-tagged isolate of Verticillium dahliae. Protein Cell, 2014, 5(2): 94–98. DOI: 10.1007/s13238-013-0009-9 |
| [43] | Zhao YL, Zhou TT, Guo HS. Hyphopodium-specific VdNoxB/VdPls1-dependent ROS-Ca2+ signaling is required for plant infection by Verticillium dahliae. PLoS Pathog, 2016, 12(7): e1005793. DOI: 10.1371/journal.ppat.1005793 |
| [44] | Zhang T, Zhao YL, Zhao JH, et al. Cotton plants export microRNAs to inhibit virulence gene expression in a fungal pathogen. Nat Plants, 2016, 2(4): 16153. |
 2017, Vol. 33
2017, Vol. 33