中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘立中, 白阳, 郑海, 傅雄飞, 刘陈立
- Lizhong Liu, Yang Bai, Hai Zheng, Xiongfei Fu, Chenli Liu
- 合成生物学在基础生命科学研究中的应用
- Fundamental aspects of synthetic biology
- 生物工程学报, 2017, 33(3): 315-323
- Chin J Biotech, 2017, 33(3): 315-323
- 10.13345/j.cjb.160477
-
文章历史
- Received: December 13, 2016
- Accepted: February 6, 2017
傅雄飞 博士,中科院深圳先进技术研究院合成生物学工程研究中心研究员,博士生导师,中组部“青年千人计划”入选者。主要研究方向是利用合成生物学方法,通过数理模型和定量实验相结合的研究手段,研究生物系统中的基本问题。成果发表在Science、Phys Rev Lett等国际专业期刊
格物致知是中国古代儒家思想的一个重要概念,它提倡通过观察探究事物,来洞察事物的规律。传统的生物学研究是基于类似的方法论。而合成生物学的出现使得人们可以通过一种全新的角度来研究复杂的生命体,即通过重新创造或改造生物系统来理解生命[1]。
合成生物学是一门结合了生物学、数理科学和工程学等多学科高度交叉融合的新兴学科。它通过“自下而上”的理念,由“元件”到“模块”再到“系统”来设计、创造自然界不存在的人工生物系统,或对已有自然生物系统进行改造、重建。这种由人工设计的元件组装而成,以信号传导、基因调控以及细胞代谢等作用方式整合而成的生物功能和系统,较天然的生物系统具有简单可控的特点,这使得合成生物学系统在化工、医药、能源、环保等诸多领域具有广阔应用前景。除此之外,合成生物学更为基础生命科学研究提供了崭新的手段。正如著名物理学家、1965年诺贝尔物理学奖得主理查德·菲利普·费曼的著名感叹“我不能创造的东西,我就不理解”,合成生物学正是可以通过构造人工生物系统来研究生命科学中的基本问题,我们称之为“建物致知”[2]。
2 理解生物基因网络调控在自然生物系统中,某些普遍存在的系统性基因网络结构可能具有进化优势而得以在漫长的自然进化过程中被保留和扩散。这些系统网络结构可以被视为一种生命的“设计原则”。了解这样的“设计原则”不仅有助于人们建造人工生物系统,也有助于生物学家更加深刻地理解生命的本质。
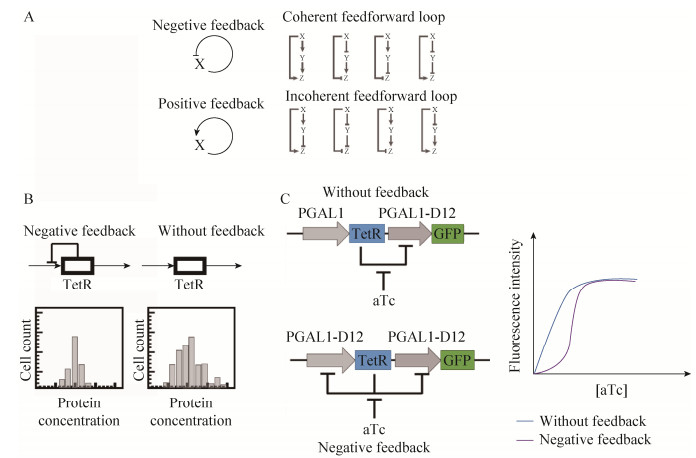
在人造的工程系统中,网络结构单元被广泛运用,例如负反馈、正反馈和前馈回路等 (图 1 A)。以负反馈为例,瓦特在设计蒸汽机之时利用负反馈在蒸汽机中加入调速器部件,其目的是使蒸汽机的速度保持在可控范围内。又如在电子系统中多数元件都具有“输入-输出”非线性的频率范围,往往需要通过引入负反馈来提高元件的线性度。生物学家发现负反馈网络结构单元在原核及真核细胞中广泛存在,40%左右的大肠杆菌转录因子抑制其自身的启动子转录活性[3]。然而,由于缺乏对原生系统各组分的全面认知,传统生物学研究手段无法厘清各网络结构单元的边界,难以对单一变量进行定量化操控,因而对网络结构单元在整个系统中的功能知之不详[4]。近年来,合成生物学研究通过学习人造工程系统的设计,在细胞内构建人工负反馈基因回路并研究其动力学过程,逐步揭示了负反馈这一网络结构单元的重要生物学功能 (图 1B,C)。例如,欧洲分子生物学实验室Becskei和Serrano通过在大肠杆菌中构建四环素抑制子TetR介导的转录负反馈基因回路,发现负反馈可以显著减少由细胞内生化反应随机性导致的基因表达噪声[5]。又如,得克萨斯大学Balázsi等通过在酵母细胞内构建合成基因回路发现负反馈可提高启动子对信号响应的线性程度[6]。另外,以加州理工学院Elowitz为代表的合成生物学家,利用构建合成基因回路,通过创造稳定、快速、可调的“遗传时钟”,验证了一种生命体实现周期调控的机制[7]。北京大学汤超等定量生物学家通过分析不同基因回路的数学模型总结了形成稳定周期回路的基因回路所需要具备的基本条件[8]。这类研究正是以合成生物学手段“建物”,来揭示自然生物系统的运行规律,即“致知”。
|
| 图 1 系统中的网络结构单元 (A:常见的网络结构单元,如负反馈、正反馈、各种前馈回路[1];B:由四环素抑制子TetR以及受TetR抑制的启动子构成的转录负反馈基因回路相比于无反馈基因回路,其基因表达噪声水平显著降低[5];C:负反馈基因回路相比于无反馈基因回路对诱导物的响应具有更好的线性度[6]) Figure 1 Network motif in biological system. (A) Typical network motifs, for example negative feedback, positive feedback and forward feedback loop[1]. (B) The negative feedback consists of tetracycline repressor TetR and TetR-repressive promoter displays significant lower level of gene expression noise, in contrast to non-feedback control[5]. (C) The negative feedback circuit shows better linearity than the non-feedback circuit[6] |
| |
细胞作为生命的最基本单元,其生长速率、周期、大小和功能等都受到严格且定量的调控。一直以来,人们缺乏对研究对象各参数进行定量扰动的技术手段,主要采用基因敲除或过表达等定性或半定量的方式来研究目的基因的功能及其对细胞生理、生化过程的影响。近年来,合成生物学家和定量生物学家通过引入定量可调的基因回路,实现了对单个或多个关键因子的线性调控,进而研究其对细胞全局生理活动的影响。加州大学圣地亚戈分校Hwa实验室借助合成线性化基因操控回路、数理模型以及定量实验相结合的手段,研究大肠杆菌在不同稳态生长条件下的细胞生理状态氮源与碳源的利用,开创性地揭示了大肠杆菌平衡基因表达以及代谢调控等细胞生理活动来维持生长速率的生长定律 (Growth law) 机制[9-14]。
长期以来,细胞是如何设定自身体积,以及如何实现细胞分裂与细胞内染色体复制相协调的机制始终是一个谜。仅靠传统研究手段很难获得清晰可验证的结论。我们通过合成基因线路对大肠杆菌细胞中关键基因的表达量进行线性调控发现,改变mreB或ftsZ基因的表达水平可以在保持细胞生长速率不变的情况下特异性地改变细胞的直径或长度,进而我们分析了在改变直径或长度的情况下,DNA复制和细胞大小之间的协调机制 (图 2)。结果表明,无论是通过改变直径还是长度而实现的细胞大小的改变,细胞内的复制起点的数目与细胞体积呈现线性正相关关系。基于实验结果,我们提出“复制起点对应增量”这一假说来解释决定群体细胞大小稳态的“加量”现象,即细胞两次复制起始事件间细胞体积的增加与起始时细胞内复制起点的数目的比值是一个恒定值[15]。这项研究展现了合成基因回路在研究细胞生理学方面的优势。

|
| 图 2 起点对应增量模型 (将基于Ptet-tetR的负反馈回路构建的mreB (ftsZ) 表达元件插入细胞染色体attB位点,接着将内源的mreB (ftsZ)基因从基因组上移除,从而得到mreB (ftsZ)表达量受诱导剂aTc控制的菌株。通过控制mreB (ftsZ) 的表达量,实现对细胞直径或长度的定量控制。细胞周期相关参数分析表明,长度和直径扰动均影响细胞的D周期;无论是直径还是长度的扰动,细胞大小 (V) 与细胞内复制起点数 (S) 之间的比例关系 (D) 保持恒定不变[15]) Figure 2 Chematic illustration of the experiment and the "adder-per-origin" model. The mreB-or ftsZ-titratable strains were constructed based on Ptet-tetR feedback loop. By the designed genetic circuit, mreB or ftsZexpression levels and cell dimensions were continuously altered. In both case, the D period increased with cell width and length. The C period and doubling time τ remained constant. The perturbed D period sets the average number of origins per cell, which is equal to the scaling factor S because replication initiation triggers cell division. The average number of origins per cell then sets the average cell volume[15] |
| |
自然界中,存在着各种各样的由多细胞组成的生物形态、结构以及行为。这些生物系统中不仅有个体细胞的生理活动,还存在着细胞间的协调与沟通,这大大增加了研究这类问题的复杂度。合成生物学可以通过模拟自然生物系统,制造出具有清晰内涵、外延边界以及各系统参数可定量调控的人工生物系统,为研究多细胞生物结构的形成机制提供全新视角。例如,麻省理工学院Weiss实验室通过将LuxRI群体感应系统中的细胞通信基因元件分别导入信号发送细胞和接收细胞,成功实现了“条带”状的生物图纹。该结果验证了经典的成形素 (Morphogen) 梯度的图案形成机理[16]。杜克大学游凌冲团队通过构建合成基因回路,使大肠杆菌能够形成具有核-环同心结构的生物图案。其基本原理是大肠杆菌菌落能够感应到自身分泌和累积的化学分子AHL,在营养物质的共同作用下,构成了整合反馈回路;另一方面,生物图案的内核区域大小与环形区域的形成速度和持续时间形成了一个非连续反馈回路。作者利用这两种不同的反馈回路研究了累积效应的空间感应机制,并由此阐明了大肠杆菌群落尺寸的自我调节规律[17]。
我们基于群体感应系统构建了携带合成基因回路的大肠杆菌来探索一种新的周期条带结构形成的机理[18]。该工程菌株在半固体琼脂空间生长扩展过程中自发性地形成了周期性条纹图案。基于该系统,我们提出了一个新的密度依赖性运动能力模型。不同于其他反应扩散系统的是,该密度依赖性运动系统在密度波前沿达到阈值时可以形成周期性条纹。此外,根据该模型预测,该系统存在从条纹到非条纹的相变,在两种状态之间可以形成有限数量的条纹 (图 3)。在实验中,我们通过改变单个基因的表达水平成功调控了条纹的数目,以此验证了模型的预测[18-19]。

|
| 图 3 纹图案的形成 (A:基因回路设计图。在高细胞浓度的情况下,AHL的水平升高,导致转录抑制子cI的表达增强。cI通过抑制cheZ的表达导致细菌运动性下降;B:将处于对数生长期的细菌接种在半固体琼脂培养基培养皿上,经过一定时间之后,细菌会在培养皿内形成由高浓度区域组成的条纹 (右图中黑色条纹)。当额外加入由aTc控制的cI表达回路之后,可通过控制培养基中aTc的浓度,产生不同数目的条纹[17]) Figure 3 Ripe pattern formation. (A) Schematic of the circuit. At high cell density, increased AHL level aids to DNA-binding of LuxR, therefore activates the expression of cI. CI suppress the expression of heZ, which is required for bacterial motility. An extra source of CI is regulated by TetR-repressive promoter. aTc as used to induce the expression of the extra cI. (B) Log-phase bacterial cells were inoculated on semi-solid LB agar plate. The cells formed the stripe pattern after a period of time of growth. The number of the stripe can be tuned by varying the aTc oncentration[17] |
| |
传统的生态学研究往往局限于群体数量、分布的观测。随着组学的发展,人们有了更多的手段在分子机制层面研究多物种生态系统。但是,组学研究是依据相关性来推演群体内部的相互作用,缺乏对因果关系的研究。近来,研究人员利用合成生物学的手段,以一种自下而上的方式,构建人工设计的种间相互作用,组建简化的微生态系统,进而在一个人为可控的外部环境条件下,研究物种间的动态相互作用过程。
合作行为是进化学中一个令人困惑的问题。群体中某些个体以损害自身利益的方式为种群内其他成员带来好处。生物学家对此迷惑不解——按照达尔文进化论所认为的适者生存原则,群体内损己利它的个体将因不具备竞争优势而遭淘汰,可事实却并非如此。麻省理工学院Gore实验室通过构建了一个由两株人工改造酵母构成的“合作者”和“欺诈者”二元微生态系统,结合博弈论,为合作行为提供了合理解释[20]。研究显示,如果一个个体能够从合作行为中获取哪怕是微小的利益,那么即使周围的个体均不合作,它也能够生存下去 (图 4)。此外,英国剑桥大学的MacLean实验室借助由瑞典查尔姆斯理工大学人工改造的酵母[21],构建了一个由“自私者”和“互助者”构成的二元微生态,结合动态系统分析,解释了互助行为的合理性。研究发现,尽管“自私者”在均一稳定的环境中拥有竞争优势,但是在周期性变化的资源条件和空间不均一的资源环境下,“互助者”和“自私者”将会长期共存[22]。

|
| 图 4 “合作者”与“欺诈者”实验 (A:实验设计示意图。“合作者”表达蔗糖转化酶,在周质空间内将蔗糖水解成葡萄糖和果糖,但是大部分葡萄糖和果糖扩散进入周围环境被不表达蔗糖转化酶的突变体 (“欺诈者”) 所利用。此外,“合作者”具有HIS3基因缺陷 (his3Δ3),是组氨酸营养缺陷型。研究者可通过调整培养基里组氨酸的浓度来间接调控合作行为的成本[20];B:在蔗糖培养基中,“合作者”和“欺诈者”可以同时存在。实验结果显示,无论起始接种比例如何,随着培养基中的组氨酸浓度下降,“合作者”和“欺诈者”都将达到平衡[21]) Figure 4 Ooperator and cheater. (A) Schematic of the experiment that utilize the sucrose metabolism in yeast. The Cooperator expresses invertase, which hydrolyze sucrose in the periplasmic space. The vast majority of the glucose and fructose created by sucrose hydrolysis diffuse away and can be consumed by a mutant cheater, which does not produce the invertase. The cooperator strain has a defective HIS3 gene, therefore, it is histidine auxotroph. The researchers can tune the cost of cooperation by tuning the concentration of histidine[20]. (B) In sucrose culture, the cooperator and the cheater can coexist. The data suggests that cooperator and cheater reach an equilibrium as the consuming of histidine, regardless of starting fractions[21] |
| |
抗生素耐药是人类面临的一个日益严重的问题。导致抗药性的机理有多种,比如,基因的水平转移、细菌体内药物靶点的自我修饰以及细胞的药物外排泵等等。通常人们认为抗生素增加了细菌彼此之间分享抗药性基因的速率促进耐药性扩散。但是研究人员通过合成生物学方法改造菌株,发现了一个挑战常规的细菌耐药性认识。杜克大学的游凌冲团队通过在一套新开发的高分辨率微流控装置中长时间监测合成生物学改造菌株的抗药性动态变化,发现抗生素对基因的水平转移效率并没有显著影响,该研究结果说明抗生素的添加在耐药性产生过程中的作用可能被高估[23]。
6 从“致知”到“建物”如上所述,合成生物学是一门将工程理念引入生物系统的综合性学科,其目标是按照研究者的意愿设计生物,服务于科学研究和社会民生需求。目前合成生物学研究面临的一大瓶颈是人们缺乏设计一个全新生物系统或生物体的能力。而通过“建物致知”理念开展的合成生物学研究将不断为我们积累关于天然和人造生物系统运作原理的知识,使人们从简单到复杂逐渐掌握设计全新生物系统的能力。合成生物学研究另一个亟待解决的瓶颈是缺乏可用的功能元件。虽然自然生物系统中存在种类繁多的调控和功能基因,但是大部分不能满足实际应用要求,或人们对其缺乏足够了解,因而在实际的合成生物学研究中,可供研究者使用的稳定可靠的分子元件并不多。一个不错的解决策略之一是通过人工定向进化的方式,制造出功能定制型生物元件。最近,由哈佛大学Liu实验室开发的噬菌体辅助连续进化系统可以帮助研究者在极短时间内得到具有所需功能的生物元件[24]。
这些瓶颈的解决将使我们从“建物致知”最终走向“以知建物”。目前,国际同行已经在这方面开展了先驱性工作。例如麻省理工的Lu实验室以及苏黎世联邦理工学院的Fussenegger实验室分别在细菌和哺乳动物细胞中建造了生物计算机系统,使得细胞可以借助由合成基因回路形成的逻辑门对输入的数字或模拟信号进行运算[25-26]。在未来,随着各元件、模块的标准化原则的明确,与之配套的设计方法的建立,操作简易的合成生物学系统设计及仿真软件的出现,大片段DNA合成的通量提高、成本降低,加之自动化实验平台的成熟。合成生物学有望形成一套从电脑辅助设计基因回路,到各元件、模块的合成与拼接,到实验验证基因回路功能、到实验数据拟合数学模型并对各元件、模块的参数进行调整,再到基因回路的进一步优化,这样一个标准化流程。届时,合成生物学将逐步走向成熟,形成类似于今天的机械工程、电子工程的一门新的实用性工程学科。
| [1] | Elowitz M, Lim WA. Build life to understand it. Nature, 2010, 468(7326): 889–890. DOI: 10.1038/468889a |
| [2] | Liu CL, Fu XF, Huang JD. Synthetic biology: a new approach to study biological pattern formation. Quant Biol, 2013, 1(4): 246–252. DOI: 10.1007/s40484-013-0021-3 |
| [3] | Alon U. Network motifs: theory and experimental approaches. Nat Rev Genet, 2007, 8(6): 450–461. DOI: 10.1038/nrg2102 |
| [4] | Lim WA, Lee CM, Tang C. Design principles of regulatory networks: searching for the molecular algorithms of the cell. Mol Cell, 2013, 49(2): 202–212. DOI: 10.1016/j.molcel.2012.12.020 |
| [5] | Becskei A, Serrano L. Engineering stability in gene networks by autoregulation. Nature, 2000, 405(6786): 590–593. DOI: 10.1038/35014651 |
| [6] | Nevozhay D, Adams RM, Murphy KF, et al. Negative autoregulation linearizes the dose-response and suppresses the heterogeneity of gene expression. Proc Natl Acad Sci USA, 2009, 106(13): 5123–5128. DOI: 10.1073/pnas.0809901106 |
| [7] | Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403(6767): 335–338. DOI: 10.1038/35002125 |
| [8] | Ma WZ, Trusina A, El-Samad H, et al. Defining network topologies that can achieve biochemical adaptation. Cell, 2009, 138(4): 760–773. DOI: 10.1016/j.cell.2009.06.013 |
| [9] | Klumpp S, Hwa T. Bacterial growth: global effects on gene expression, growth feedback and proteome partition. Curr Opin Biotechnol, 2014, 28: 96–102. |
| [10] | Klumpp S, Zhang ZG, Hwa T. Growth rate-dependent global effects on gene expression in bacteria. Cell, 2009, 139(7): 1366–1375. DOI: 10.1016/j.cell.2009.12.001 |
| [11] | Scott M, Gunderson CW, Mateescu EM, et al. Interdependence of cell growth and gene expression: origins and consequences. Science, 2010, 330(6007): 1099–1102. DOI: 10.1126/science.1192588 |
| [12] | Scott M, Hwa T. Bacterial growth laws and their applications. Curr Opin Biotechnol, 2011, 22(4): 559–565. DOI: 10.1016/j.copbio.2011.04.014 |
| [13] | You CG, Okano H, Hui S, et al. Coordination of bacterial proteome with metabolism by cyclic AMP signalling. Nature, 2013, 500(7462): 301–306. DOI: 10.1038/nature12446 |
| [14] | Deris JB, Kim M, Zhang ZG, et al. The innate growth bistability and fitness landscapes of antibiotic-resistant bacteria. Science, 2013, 342(6162): 1237435. DOI: 10.1126/science.1237435 |
| [15] | Zheng H, Ho PY, Jiang ML, et al. Interrogating the Escherichia coli cell cycle by cell dimension perturbations. Proc Natl Acad Sci USA, 2016, 113(52): 15000–15005. DOI: 10.1073/pnas.1617932114 |
| [16] | Basu S, Gerchman Y, Collins CH, et al. A synthetic multicellular system for programmed pattern formation. Nature, 2005, 434(7037): 1130–1134. DOI: 10.1038/nature03461 |
| [17] | Cao YXL, Ryser MD, Payne S, et al. Collective space-sensing coordinates pattern scaling in engineered bacteria. Cell, 2016, 165(3): 620–630. DOI: 10.1016/j.cell.2016.03.006 |
| [18] | Liu CL, Fu XF, Liu LZ, et al. Sequential establishment of stripe patterns in an expanding cell population. Science, 2011, 334(6053): 238–241. DOI: 10.1126/science.1209042 |
| [19] | Fu XF, Tang LH, Liu CL, et al. Stripe formation in bacterial systems with density-suppressed motility. Phys Rev Lett, 2012, 108(19): 198102. DOI: 10.1103/PhysRevLett.108.198102 |
| [20] | Gore J, Youk H, Van Oudenaarden A. Snowdrift game dynamics and facultative cheating in yeast. Nature, 2009, 459(7244): 253–256. DOI: 10.1038/nature07921 |
| [21] | Otterstedt K, Larsson C, Bill RM, et al. Switching the mode of metabolism in the yeast Saccharomyces cerevisiae. EMBO Rep, 2004, 5(5): 532–537. DOI: 10.1038/sj.embor.7400132 |
| [22] | MacLean RC, Gudelj I. Resource competition and social conflict in experimental populations of yeast. Nature, 2006, 441(7092): 498–501. DOI: 10.1038/nature04624 |
| [23] | Lopatkin AJ, Huang SQ, Smith RP, et al. Antibiotics as a selective driver for conjugation dynamics. Nat Microbiol, 2016, 1(6): 16044. DOI: 10.1038/nmicrobiol.2016.44 |
| [24] | Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules. Nature, 2011, 472(7344): 499–503. DOI: 10.1038/nature09929 |
| [25] | Farzadfard F, Lu TK. Synthetic biology. Genomically encoded analog memory with precise in vivo DNA writing in living cell populations. Science, 2014, 346(6211): 1256272. DOI: 10.1126/science.1256272 |
| [26] | Ausländer S, Ausländer D, Müller M, et al. Programmable single-cell mammalian biocomputers. Nature, 2012, 487(7405): 123–127. |
 2017, Vol. 33
2017, Vol. 33