中国科学院微生物研究所、中国微生物学会主办
文章信息
- 罗周卿, 戴俊彪
- Zhouqing Luo, Junbiao Dai
- 合成基因组学:设计与合成的艺术
- Synthetic genomics: the art of design and synthesis
- 生物工程学报, 2017, 33(3): 331-342
- Chin J Biotech, 2017, 33(3): 331-342
- 10.13345/j.cjb.160411
-
文章历史
- Received: October 30, 2016
- Accepted: December 9, 2016
“基因组”这一概念最早于1920年由H. Winkler提出,用来代表一个生物体所包含的整套遗传物质。近十年来由于二代和三代测序技术的发展使得测序的速度、精度和深度都有了巨大的提高,成本也发生了显著的下降,越来越多物种的基因组被测序。TALEN以及CRISPR/Cas系统的成功应用极大地提高了对基因组特定位点的编辑能力,基因组的功能逐步被解析。随着基因组阅读能力 (测序) 和修改能力 (基因组编辑) 的提升,人类对基因组的认识达到了一个前所未有的高度。研究者们开始探索:我们能否根据已有的认知全方位改造一个基因组,甚至从头设计与合成一个崭新的基因组?DNA合成和组装技术的发展,特别是基于芯片的DNA合成技术的成功应用,极大降低了DNA的合成成本并同时提高了通量,使得“编写” (化学从头合成) 一个基因组成为可能。合成基因组学作为一个汇集DNA阅读、修改和编写等各方面技术的新学科领域呈现在人们的面前。
本综述将针对合成基因组学所取得的进展,介绍合成基因组学相关的设计理念与技术手段。与此同时,合成基因组学的后期应用以及伦理问题也将在本综述中进行讨论。
1 合成基因组学合成基因组学,指的是通过一系列的技术手段从头化学全合成整个基因组或者是基因组的大部分。它的出现使得同时改变一个生物的遗传物质的多个方面成为可能,从而成为合成生物学领域中对生物体复杂功能体系和全新生物体进行合成的重要方面[1]。
为什么要合成一个基因组?除了对创造一个崭新生命的原始好奇心,还包括了其他许多更加切合实际的目的。首先,尽管最近基因编辑领域的技术进展足以令人称奇,但是仍然需要较长的时间来进行一项基因组水平的改变,比如说从大肠杆菌Escherichia coli的基因组中去除一个密码子[2-3]。而合成基因组学为这类问题提供了另外一种解决方案。并且,通过合成基因组学的相关研究还可以开发和完善相关的基因组编辑技术,从而实现良性循环。其次,通过对合成基因组的设计、合成和检测,可以对一些之前无法或者很难很好地研究的问题提供答案。比如说,占据了人类基因组超过50%的重复序列到底有没有功能?如果有,是什么?再次,我们对基因组的已有认知指导了我们现阶段的合成基因组学实践。反过来,通过合成基因组的过程和对合成的基因组的研究,将有助于进一步加深我们对基因组功能的理解 (Built to understand)。最后,在人类基因组计划初期,人们对于是否值得花费如此多的人力、物力和财力去进行人类基因组测序有着广泛的分歧[4]。而目前合成基因组学也处在这么一个类似的时期,我们并不清楚它最后具体能给我们带来什么,但是至少目前的展望不亚于当年的人类基因组计划。
2 合成基因组学发展简史从寡核苷酸链的化学合成开始,第一个人工合成的基因——丙氨酸tRNA编码基因 (77 bp),于1970年被Khorana组成功合成[5]。2002年,第一个合成基因组——脊髓灰质炎病毒 (Poliovirus) 基因组被成功合成并产生活的病毒颗粒[6],证明了利用已有的基因组序列而不依赖于天然模板从头化学合成基因组的可行性。2008年,自然界中已知的最小原核生物基因组——583 kb的生殖支原体 (Mycoplasma genitalium) 基因组被化学全合成[7]。Gibson等在2010年成功利用人工合成的,长达1.08 Mb的蕈状支原体 (Mycoplasma mycoides) 基因组支持JCVI-syn1.0的存活,诞生了第一个由合成基因组控制的原核生物[8]。从2011年开始,来自世界上多个不同国家的研究者们在Boeke的带领下开展了第一个真核生物基因组合成计划 (Sc2.0),目前已经完成了2、3、5、6、10和12号染色体的合成与组装[9-17]。就在Sc2.0进行的过程中,Boeke于2016年提出了人类基因组编写计划 (HGP-Write)[18],使合成基因组学成为大众的焦点 (图 1)。

|
| 图 1 合成基因组学大事记 Figure 1 A timeline of major events in synthetic genomics |
| |
虽然现在我们可以很方便地进行基因组的测序和编辑,但是我们对基因组的认识还非常有限。以早期完成基因组测序的模式生物为例,E. coli中有大约20%的基因功能未知,酿酒酵母Saccharomyces cerevisiae中有大约一半的基因功能未知[19-20]。根据对基因组的已有认知、对一些基因和代谢通路的合成尝试,人们探索出了一些基因组重编和设计的规则。显而易见,这些规则目前还缺乏系统性。因此,目前合成基因组学主要进行前期技术的储备,对基因组重编的程度都比较小。2017年3月10日Science杂志发表了第1个专门为全基因组技术而编写的软件平台——BioStudio,可以根据研究者的需要按照一定的原则进行全基因范围的重编[13]。相信随着对生命现象了解的深入以及边合成边测试能力的不断提高,这些原则将被更好地定义和理解,使得合成基因组学朝着更加可预测的方向发展。
3.1 对基因组编码区域的重编和设计生物体中的蛋白质主要通过基因组的编码区域进行编码。通过对基因编码区域的重编和设计,可以改变蛋白的表达特性。编码区域的重编和设计主要基于密码子的简并性进行,并根据实验的目的综合考虑不同的因素。这些因素包括密码子的使用频率 (Codon bias)[21]、密码子上下游序列 (Codon context bias)[22]以及mRNA的二级和三级结构等。首先,在不同的生物体中,同义密码子的使用频率通常并不相同。不同的密码子频率可以影响蛋白的翻译速率,从而影响蛋白的表达。因此在合成基因的设计过程中通常会对其密码子进行优化,尤其是在代谢工程领域,使其适应新的表达环境。常用的密码子偏好性计算方法包括Fop、CAI、ENc以及tAI等[23-26]。其次,密码子上下游序列环境在基因的翻译效率调控中也扮演了重要的角色。由于细胞中的tRNA使用完后可以重新加载氨基酸再用于同一mRNA的翻译 (Codon reuse),在同一个mRNA转录本的某一区域通常倾向于使用同一密码子编码某一种氨基酸,以便提高翻译效率。再次,编码区域的重编将有可能改变mRNA的二级和三级结构,从而影响mRNA的稳定性以及翻译效率。基于热力学参数和最小自由能,许多算法被开发出来用于计算RNA的结构,包括UNAFold和ViennaRNA等[27-28]。此外,限制性内切酶识别位点在DNA的分子操作过程中发挥了重要的作用。通过同义重编,可以移除或者是引入相应的酶切位点便于后续的研究操作。其他如序列的GC含量和隐藏的终止密码子等也是重编过程中要考虑的因素。针对这些因素和不同的实验目的,许多软件被开发出来用于编码区域的重编,包括Synthetic Gene Developer、Gene Designer 2.0和Codon Optimization OnLine (COOL) 等[29-31]。目前这些软件都只考虑这些因素中的一种或者几种。考虑的因素越多,对计算能力的要求越大。如何定量预测密码子同义替换对蛋白翻译的影响将是这一领域未来的发展方向之一。
在最早合成的脊髓灰质炎病毒 (Poliovirus) 基因组中,研究者们通过改变总共20个核苷酸序列引入了13个新的酶切位点,去除了一个Pst Ⅰ酶切位点[6]。在Sc2.0项目中,为了区别合成的和野生型的序列,根据同义密码子原则我们引入了一系列的PCR标签,把TAG终止密码子替换成TAA,并且引入或剔除某些酶切位点[9]。通过替换野生型序列和表型测试,我们发现一些基因的同义替换会对其表达和功能造成重要的影响[12, 14-15, 17]。通过同义密码子替换,Church组希望能够构建出一个只含有57个密码子的合成大肠杆菌。通过初期的功能测试,他们发现2 229个基因中有13个基因的重编不能支持大肠杆菌的存活[32]。由这些实践可以看出目前并不能很好地预测重编带来的影响,但是研究者们的这些尝试将为成功预测重编带来的影响提供大量的研究材料。
根据基因的功能进行重编是合成基因组设计的另外一个方面。在合成的Mycoplasma genitalium JCVI-1.0基因组中,MG408基因被抗生素基因破坏,从而去除致病性,并且给合成的基因组提供了一个筛选标记[7]。在Sc2.0计划中,所有tRNA编码基因都将被转移到一条新的染色体上[9]。在合成12号染色体的过程中,我们发现tRNA拷贝数的下降会导致细胞周期在G2/M转化过程中的延迟[17]。对于已有基因组序列的分析显示:在进化过程中具有类似功能或者表达的基因通常倾向于聚集在一起,而且基因的排序也不是随机的[33-34]。利用合成基因组学的手段改变基因的排序将为研究这一问题提供新的材料。
基因的冗余性是基因组的特征之一,冗余基因的敲除通常不会带来大的表型变化。除此之外,基因组中还有许多的非必需基因,这些基因的敲除将有助于简化基因组。最小基因组的构建将有助于优化细胞对目的代谢途径的物质和能量投入。利用改造的Tn5转座子,E. coli K-12的基因组可以被减小200 kb[35]。通过设计、合成、表型测试的循环,Venter组在2016年成功将M. mycoides基因组从1.08 Mb缩小到531 kb。这一基因组比自然界中已知的任何基因组都要小。他们总共删除了428个基因,在剩下的473个基因中仍然还有149个基因功能未知[36]。这一最小基因组将为研究基因组中的核心功能以及探索如何从头合成一个有功能的基因组提供一个通用的平台。
3.2 对基因组其他区域的重编和设计相对于编码区域,基因组其他区域的重编和设计目前还处于比较初级的状态。在合成的M. genitalium基因组中,一些“watermark”被插入到基因组中的基因间区。为了减少对基因组生物功能的可能影响,他们选择了一些耐受转座子插入的位置作为插入位点[7]。类似的策略也被用于Mycoplasma mycoides合成基因组的设计[8]。在Sc2.0计划中,基因组内所有的转座子、长末端重复序列和亚端粒末端重复序列都被删去,以期能获得更加稳定的基因组和探索这些元件在进化上对于基因组的意义[9]。
基因组的非编码区域有很大一部分是基因的启动子和终止子。通过对这些元件的描述 (Characterization) 和模块化 (Modularization),使其拥有更加可控的输出和适配性能,一直是代谢工程和合成生物学的一个重要方面。目前在合成基因组学领域并没有相关的研究报道这部分内容的重编与设计。在Sc2.0计划中,研究者们在每个非必需基因的3′末端引入了一个loxPsym位点,设计了loxP介导的合成染色体重组和修饰进化系统 (Synthetic chromosome recombination and modification by loxP-mediated evolution,简称SCRaMbLE)。通过SCRaMbLE将有可能改变这些基因的启动子和终止子,从而为研究它们的功能提供材料[9, 37-38]。通过对12号染色体的染色体启动SCRaMbLE,我们发现ACE2的3′UTR对于其mRNA的稳定性至关重要 (未发表数据)。
近年来的一些研究逐步发现基因组的三维结构对于基因组的功能和调控起着重要的作用。庄小威组成功利用高分辨成像技术观测了染色体的高级结构域,分辨率可达kbp到Mbp的水平[39-40]。如何将基因组的三维结构和表观遗传状态考虑到基因组的重编和设计中来,将是合成基因组学发展中的重要方向。鉴于目前的合成基因组都是建立在已有基因组的基础上,我们期望能够通过不断的尝试,找到一些普遍的规律,逐步增大重编的程度,最终完全从头设计一个新的基因组。
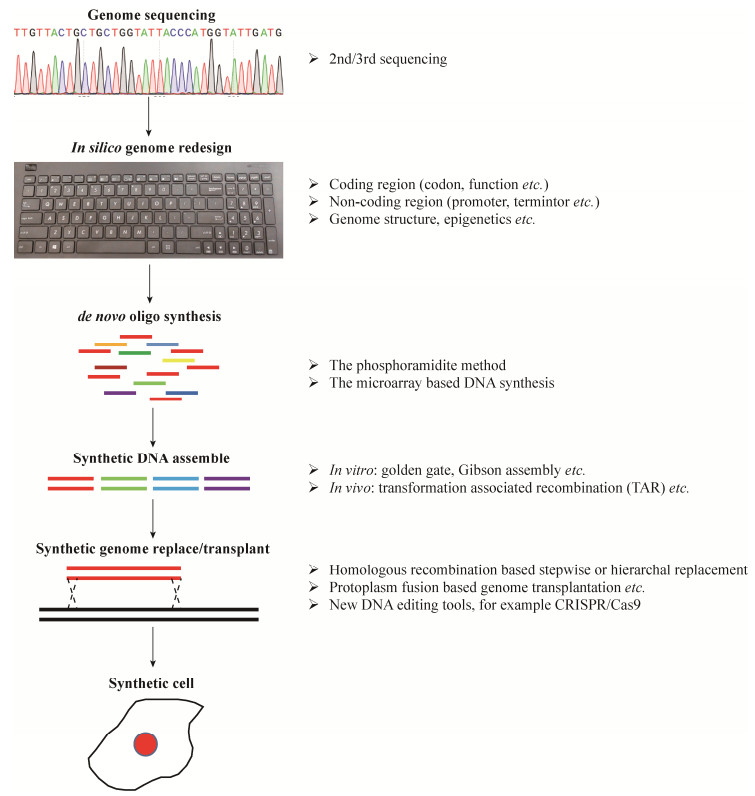
4 基因组的合成、组装与替换DNA合成技术的进展使得在较短的时间内以可承受的价格获得足量的DNA成为可能,这也促使了合成基因组学时代的真正到来。对于计算机设计好的基因组序列,如何快速合成、组装并替换原有的野生型基因组,是合成基因组学在实践过程中必须要解决的关键技术问题。这一部分将结合已有的基因组合成实践,介绍如何从化学合成的寡聚核苷酸链开始,到最后组装成一个有功能的基因组 (图 2)。
|
| 图 2 化学从头合成基因组 Figure 2 A workflow of complete chemical genome synthesis |
| |
目前在寡链核苷酸从头合成中使用最多的是固相亚磷酰胺化学合成法。该法通过去保护、联结、加帽和氧化四步反应的循环在反应柱中逐步合成寡核苷酸链[41]。由于化学反应效率随着寡核苷酸链的增长而降低,DNA合成的完整性、精确度和产量随长度的增长而发生下降,合成的DNA长度一般不超过200 bp[42]。微阵列介导的DNA合成技术的出现极大降低了合成的成本,提高了通量。由于边缘效应、去嘌呤反应等原因,微阵列合成的DNA相较于柱合成的DNA拥有较高的突变率。近年来,微阵列介导的DNA合成技术在合成的质量、效率和自动化程度上都有了显著提高。通过对反应过程的优化,微阵列合成的寡链核苷酸现在也可以达到和柱合成类似的长度 (最长可达200 bp) 和精确度 (错误率在1/600左右) [42-43]。将二代测序与芯片合成有机结合起来,利用二代测序鉴定并回收序列正确的DNA片段,成为提高合成精确度的一个有效手段[44]。
4.2 基因组DNA的组装化学合成的寡核苷酸链长度有限,并不能满足基因和基因组合成的需求,需要进一步组装变成更长的DNA片段。现有的体外酶促DNA拼接技术主要包括限制性内切酶依赖的拼接技术 (BioBrickstTM、BglBricks和Golden gate等) 和同源序列依赖的拼接技术 (In-FusionTM、SLIC和Gibson恒温组装等)。Golden gate拼接利用Ⅱ型限制性内切酶切割位点在识别序列外部的特点,通过设计切割后的4 bp悬挂序列来实现DNA片段的无缝顺序拼接[45]。基于Golden gate的原理,本实验室成功构建了YeastFab和EcoExpress两个系统用于代谢工程的优化和蛋白的表达,DNA拼接的效率可达90%以上[46-47]。Gibson assembly利用5′外切酶、DNA聚合酶和耐热DNA连接酶的混合物实现DNA片段之间的无痕连接[48]。一步恒温反应减少了大片段DNA分子在操作过程中的断裂风险,较高的连接温度 (50 ℃) 也减少了二级结构的形成。但是该方法酶的价格较高,限制了其大规模的应用。
对于芯片合成的DNA,其复杂程度使得整个组装过程更加具有挑战性。利用核苷酸选择性杂交的原则设计“block oligos”,可以将错误的序列排除在最终的组装序列之外[49-50]。在合成酵母12号染色体的过程中,我们通过引入“oligo block”可以成功地将5个左右的1.6 kb片段一次拼接成10 kb的片段[17]。其他的一些方法,比如寡核苷酸库的选择性扩增、芯片分区和基于单链DNA缺口和链替换的原位基因合成,也被开发出来提高芯片合成DNA的组装效率和准确率[51-52]。错配剪切 (Mismatch-cleavage) 等手段也可用来进一步降低最终组装产物的错误率。
宿主体内的同源重组系统也可以用来进行大片段DNA的组装。Itaya等将3.5 Mb的Synechocystis PCC6803基因组通过迭代替换的方法克隆到了Bacillus subtilis的基因组中,变成一个7.7 Mb的融合基因组[53]。S. cerevisiae的同源重组系统非常高效,可以同时组装多个带有重叠序列的DNA片段,是目前利用合成DNA片段组装基因组的最佳选择。Gibson等通过转化辅助的重组技术可以将25个24 kb左右的DNA片段在酵母体内一次性组装成整个Mycoplasma genitalium的基因组[54]。此外,酵母对外源DNA的承载能力很强,目前已报道的最大承载是1.8 Mb的Haemophilus influenza的基因组[55],并且承载的上限并不清楚。需要注意的是,外源基因组中的毒性基因的表达会对酵母的存活产生较大的影响。比如说Acholeplasma laidlawii基因组中的一个内切酶编码基因必须被失活以使得其可以在酵母中克隆[56]。另外,酵母基因组中的复制起始位点通常拥有较低的GC含量,并且每隔大概150 kb就需要一个复制起始位点以保证染色体的稳定。2.7 Mb的Synechococcus elongatus PCC 7942基因组拥有55%的GC含量,在酵母体内克隆大于200 kb的Synechococcus elongatus PCC 7942基因组片段需要插入酵母内源的复制起始位点才能成功。
4.3 基因组DNA的移植和替换将DNA组装成较大的DNA片段甚至是全基因组后,需要用这些合成的序列替换掉原有的野生型序列,并保持原有细胞的存活。对于拥有小型基因组的病毒如Poliovirus,合成的基因组cDNA可通过RNA聚合酶转录成病毒RNA,然后就可以体外在细胞提取液中进行翻译和复制,组装成一个有功能的病毒颗粒[6]。对于基因组较大的原核生物和真核生物,基因组替换的过程要困难许多。对于在酵母中组装完成的基因组 (Mycoplasma genitalium、Mycoplasma mycoides和JCVI-syn3.0最小基因组),可以通过原生质体融合来实现酵母和其他生物间基因组的快速转移,并同时减少机械剪切力对基因组DNA大片段的损伤[7-8, 36]。而对于酵母自身基因组的合成,我们采用了基于同源重组的逐步替换的方法。每条酵母染色体被分为几个到几十个30 kb的片段 (Megachunk),每个megachunk带有一个营养缺陷型LEU2或者URA3标记基因。通过LEU2和URA3的交互循环替代最终将整条染色体变成合成的序列。为了加快整条染色体替换的速度和及早发现存在于合成序列中的可能问题,我们组在合成酵母12号染色体的过程中建立了基于酵母减数分裂的快速组装办法 (Meiotic recombination based assembly,MRA)[17]。对于酵母以外的生物,直接依赖于自身的同源重组系统并不能够满足快速进行基因组替换的需求。基于λ Red噬菌体单链DNA结合蛋白β的MAGE和CAGE的出现,使得快速多位点编辑E. coli的基因组成为可能[2, 57]。CRISPR/Cas9编辑系统的出现,使得对高等生物细胞的编辑变得更加高效[58-60]。这些基因组编辑技术的出现将有助于在不同的生物体内实现像酵母一样的原位合成基因组替换。
5 合成基因组的应用与伦理合成基因组学的发展不仅展示了人类在基因组水平合成生命系统的能力,同时在这过程中所发展的各种技术将从全新的角度,引领生命科学各领域的颠覆性创新。比如大片段DNA分子的组装技术将有助于复杂代谢途径的构建和优化,从而生产全新的药物分子和生物能源物质。又比如合成基因组的过程,通过重编、合成和表型测试的实践提供了一个“自下而上”研究基因组中所有编码和非编码序列功能的崭新的手段。并且,合成的基因组可以引入某些生物学特性,使得合成的生物体有着更加广阔的应用前景。合成的无毒力病毒可用于相关病毒疫苗的研发[61],最小基因组将为代谢工程提供优良的底盘细胞[36],拥有57个密码子的大肠杆菌将和其他细菌产生遗传隔离从而阻断基因的水平转移[32]。在Sc2.0计划中,合成的基因组包含了一个可诱导的自动进化系统SCRaMbLE。通过诱导SCRaMbLE可以产生多种多样的基因组,从而加速菌株的进化,优化代谢工程的底盘细胞[38]。
作为一个崭新的研究领域,合成基因组学对传统生命观念所带来的冲击是巨大的,而且充满了不确定性。就像一把双刃剑,合成基因组学在带来社会效益的同时也有可能带来损害。所有的合成基因组学研究者都必须深刻认识到这里面将可能涉及的伦理和政策问题,包括合成基因组学所带来的知识产权、生物安全以及自我管控能力建设等问题。合成有机体如果进入到自然环境中,将有可能会对环境产生负面的影响,污染自然基因库。因此,一些合成生物控制系统同时被研究出来防止合成生物的逃逸[62-63]。我们所有的研究都必须要以益处最大化和可能带来的风险最小化为准绳,开展有责任的创新。为了管控DNA重组和合成技术可能带来的生物安全问题,NIH制定了相应的指导性原则 (NIH guidelines for research involving recombinant or synthetic nucleic acid molecules),并根据研究进展保持更新。2010年5月,瑞士联邦非人类生物技术伦理委员会 (Federal ethics committee on non-human biotechnology,ECNH) 发布了题为《合成生物学——伦理问题》的报告。同年12月,美国生物伦理研究委员会发表了题为《新方向:合成生物学和新出现技术的伦理》的报告。为了更好地处理这些问题,合成基因组学的研究者们应该加强自我监管,建立开放的跨学科讨论平台,与社会学家和哲学家们合作,在研究的早期阶段公开讨论项目的伦理问题。
6 结语与展望进入21世纪以来,合成基因组学获得了突飞猛进的发展。原核生物基因组被不断成功合成,第一个真核生物基因组已经合成过半,人类基因组的合成也提上了议事日程。但是,目前的合成基因组学还处于起步发展阶段,许多方面都尚未成熟。
一方面,由于目前对基因组各部分序列的功能了解得尚不够清楚,基因组的重编程度仍然较低。如何合成一个功能可预知、可控制的基因组是当前合成基因组学需要解决的一个重大问题。为解决这一问题,需要将基因组的各部分元件标准化、模块化,使其变得更加可控;也需要建立各部分元件的功能的标准化表征体系,用以衡量合成细胞的各个输出;更需要在系统生物学、定量生物学等层面深入理解各元件之间的输入和输出关系,提高对合成基因组的预知能力。通过“设计-合成-测试”这样的循环发展,合成基因组学必将大幅加深我们对基因组的序列与功能对应关系的理解。
另外一方面,基因组合成的成本目前仍然相对较高,DNA的合成、组装和替换技术各方面都有着很大的降价空间。开发新的DNA合成策略,降低合成成本,加快合成速度,实现合成基因组学的工程化、经济化将是未来几年内合成基因组学走向实际应用的必经之路。
| [1] | König H, Frank D, Heil R, et al. Synthetic genomics and synthetic biology applications between hopes and concerns. Curr Genomics, 2013, 14(1): 11–24. |
| [2] | Isaacs FJ, Carr PA, Wang HH, et al. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement. Science, 2011, 333(6040): 348–353. DOI: 10.1126/science.1205822 |
| [3] | Lajoie MJ, Rovner AJ, Goodman DB, et al. Genomically recoded organisms expand biological functions. Science, 2013, 342(6156): 357–360. DOI: 10.1126/science.1241459 |
| [4] | DeLisi C. Meetings that changed the world: Santa Fe 1986: human genome baby-steps. Nature, 2008, 455(7215): 876–877. DOI: 10.1038/455876a |
| [5] | Agarwal KL, Büchi H, Caruthers MH, et al. Total synthesis of the gene for an alanine transfer ribonucleic acid from yeast. Nature, 1970, 227(5253): 27–34. DOI: 10.1038/227027a0 |
| [6] | Cello J, Paul AV, Wimmer E. Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template. Science, 2002, 297(5583): 1016–1018. DOI: 10.1126/science.1072266 |
| [7] | Gibson DG, Benders GA, Andrews-Pfannkoch C, et al. Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome. Science, 2008, 319(5867): 1215–1220. DOI: 10.1126/science.1151721 |
| [8] | Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome. Science, 2010, 329(5987): 52–56. DOI: 10.1126/science.1190719 |
| [9] | Dymond JS, Richardson SM, Coombes CE, et al. Synthetic chromosome arms function in yeast and generate phenotypic diversity by design. Nature, 2011, 477(7365): 471–476. DOI: 10.1038/nature10403 |
| [10] | Annaluru N, Muller H, Mitchell LA, et al. Total synthesis of a functional designer eukaryotic chromosome. Science, 2014, 344(6179): 55–58. DOI: 10.1126/science.1249252 |
| [11] | Mercy G, Mozziconacci J, Scolari VF, et al. 3D organization of synthetic and scrambled chromosomes. Science, 2017, 355(6329): eaaf4597. |
| [12] | Mitchell LA, Wang A, Stracquadanio G, et al. Synthesis, debugging, and effects of synthetic chromosome consolidation: synVI and beyond. Science, 2017, 355(6329): eaaf4831. |
| [13] | Richardson SM, Mitchell LA, Stracquadanio G, et al. Design of a synthetic yeast genome. Science, 2017, 355(6329): 1040–1044. |
| [14] | Shen Y, Wang Y, Chen T, et al. Deep functional analysis of synⅡ, a 770-kilobase synthetic yeast chromosome. Science, 2017, 355(6329): eaaf4791. |
| [15] | Wu Y, Li BZ, Zhao M, et al. Bug mapping and fitness testing of chemically synthesized chromosome Ⅹ. Science, 2017, 355(6329): eaaf4706. |
| [16] | Xie ZX, Li BZ, Mitchell LA, et al. "Perfect" designer chromosome Ⅴ and behavior of a ring derivative. Science, 2017, 355(6329): eaaf4704. |
| [17] | Zhang W, Zhao G, Luo Z, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome. Science, 2017, 355(6329): eaaf3981. |
| [18] | Boeke JD, Church G, Hessel A, et al. The genome project-write. Science, 2016, doi: 10.1126/science.aaf6850. |
| [19] | Keseler IM, Collado-Vides J, Santos-Zavaleta A, et al. EcoCyc: a comprehensive database of Escherichia coli biology. Nucleic Acids Res, 2011, 39(S1): D583–D590. |
| [20] | Peña-Castillo L, Hughes TR. Why are there still over 1000 uncharacterized yeast genes. Genetics, 2007, 176(1): 7–14. DOI: 10.1534/genetics.107.074468 |
| [21] | Gustafsson C, Govindarajan S, Minshull J. Codon bias and heterologous protein expression. Trends Biotechnol, 2004, 22(7): 346–353. DOI: 10.1016/j.tibtech.2004.04.006 |
| [22] | Cannarozzi G, Schraudolph NN, Faty M, et al. A role for codon order in translation dynamics. Cell, 2010, 141(2): 355–367. DOI: 10.1016/j.cell.2010.02.036 |
| [23] | Ikemura T. Correlation between the abundance of Escherichia coli transfer RNAs and the occurrence of the respective codons in its protein genes: a proposal for a synonymous codon choice that is optimal for the E. coli translational system. J Mol Biol, 1981, 151(3): 389–409. DOI: 10.1016/0022-2836(81)90003-6 |
| [24] | dos Reis M, Savva R, Wernisch L. Solving the riddle of codon usage preferences: a test for translational selection. Nucleic Acids Res, 2004, 32(17): 5036–5044. DOI: 10.1093/nar/gkh834 |
| [25] | Sharp PM, Li WH. The codon Adaptation Index-a measure of directional synonymous codon usage bias, and its potential applications. Nucleic Acids Res, 1987, 15(3): 1281–1295. DOI: 10.1093/nar/15.3.1281 |
| [26] | Wright F. The 'effective number of codons' used in a gene. Gene, 1990, 87(1): 23–29. DOI: 10.1016/0378-1119(90)90491-9 |
| [27] | Markham NR, Zuker M. UNAFold: software for nucleic acid folding and hybridization//Keith JM, Ed. Bioinformatics. Clifton: Humana Press, 2008: 3-31. |
| [28] | Schuster P, Fontana W, Stadler PF, et al. From sequences to shapes and back: a case study in RNA secondary structures. Proc Roy Soc B: Biol Sci, 1994, 255(1344): 279–284. DOI: 10.1098/rspb.1994.0040 |
| [29] | Chin JX, Chung BKS, Lee DY. Codon Optimization OnLine (COOL): a web-based multi-objective optimization platform for synthetic gene design. Bioinformatics, 2014, 30(15): 2210–2212. DOI: 10.1093/bioinformatics/btu192 |
| [30] | Villalobos A, Ness JE, Gustafsson C, et al. Gene designer: a synthetic biology tool for constructing artificial DNA segments. BMC Bioinformatics, 2006, 7: 285. DOI: 10.1186/1471-2105-7-285 |
| [31] | Wu G, Bashir-Bello N, Freeland SJ. The Synthetic Gene Designer: a flexible web platform to explore sequence manipulation for heterologous expression. Protein Expr Purif, 2006, 47(2): 441–445. DOI: 10.1016/j.pep.2005.10.020 |
| [32] | Ostrov N, Landon M, Guell M, et al. Design, synthesis, and testing toward a 57-codon genome. Science, 2016, 353(6301): 819–822. DOI: 10.1126/science.aaf3639 |
| [33] | Tamames J. Evolution of gene order conservation in prokaryotes. Genome Biol, 2001, 2(6): research0020. |
| [34] | Hurst LD, Pál C, Lercher MJ. The evolutionary dynamics of eukaryotic gene order. Nat Rev Genet, 2004, 5(4): 299–310. DOI: 10.1038/nrg1319 |
| [35] | Goryshin IY, Naumann TA, Apodaca J, et al. Chromosomal deletion formation system based on Tn5 double transposition: use for making minimal genomes and essential gene analysis. Genome Res, 2003, 13(4): 644–653. DOI: 10.1101/gr.611403 |
| [36] | Hutchison Ⅲ CA, Chuang RY, Noskov VN, et al. Design and synthesis of a minimal bacterial genome. Science, 2016, 351(6280): aad6253. DOI: 10.1126/science.aad6253 |
| [37] | Shen Y, Stracquadanio G, Wang Y, et al. SCRaMbLE generates designed combinatorial stochastic diversity in synthetic chromosomes. Genome Res, 2016, 26(1): 36–49. DOI: 10.1101/gr.193433.115 |
| [38] | Dymond J, Boeke JD. The Saccharomyces cerevisiae SCRaMbLE system and genome minimization. Bioengineered, 2012, 3(3): 170–173. DOI: 10.4161/bbug.19543 |
| [39] | Wang SY, Su JH, Beliveau BJ, et al. Spatial organization of chromatin domains and compartments in single chromosomes. Science, 2016, 353(6299): 598–602. DOI: 10.1126/science.aaf8084 |
| [40] | Boettiger AN, Bintu B, Moffitt JR, et al. Super-resolution imaging reveals distinct chromatin folding for different epigenetic states. Nature, 2016, 529(7586): 418–422. DOI: 10.1038/nature16496 |
| [41] | Caruthers MH, Barone AD, Beaucage SL, et al. Chemical synthesis of deoxyoligonucleotides by the phosphoramidite method. Methods Enzymol, 1987, 154: 287–313. DOI: 10.1016/0076-6879(87)54081-2 |
| [42] | LeProust EM, Peck BJ, Spirin K, et al. Synthesis of high-quality libraries of long (150mer) oligonucleotides by a novel depurination controlled process. Nucleic Acids Res, 2010, 38(8): 2522–2540. DOI: 10.1093/nar/gkq163 |
| [43] | Saaem I, Ma KS, Marchi AN, et al. In-situ synthesis of DNA microarray on functionalized cyclic olefin copolymer substrate. ACS Appl Mater Interfaces, 2010, 2(2): 491–497. DOI: 10.1021/am900884b |
| [44] | Matzas M, Stähler PF, Kefer N, et al. High-fidelity gene synthesis by retrieval of sequence-verified DNA identified using high-throughput pyros equencing. Nat Biotechnol, 2010, 28(12): 1291–1294. DOI: 10.1038/nbt.1710 |
| [45] | Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability. PLoS ONE, 2008, 3(11): e3647. DOI: 10.1371/journal.pone.0003647 |
| [46] | Qin YR, Tan C, Lin JW, et al. EcoExpress-highly efficient construction and expression of multicomponent protein complexes in Escherichia coli. ACS Synth Biol, 2016, 5(11): 1239–1246. |
| [47] | Guo YK, Dong JK, Zhou T, et al. YeastFab: the design and construction of standard biological parts for metabolic engineering in Saccharomyces cerevisiae. Nucleic Acids Res, 2015, 43(13): e88. DOI: 10.1093/nar/gkv464 |
| [48] | Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods, 2009, 6(5): 343–345. DOI: 10.1038/nmeth.1318 |
| [49] | Tian JD, Gong H, Sheng NJ, et al. Accurate multiplex gene synthesis from programmable DNA microchips. Nature, 2004, 432(7020): 1050–1054. DOI: 10.1038/nature03151 |
| [50] | Borovkov AY, Loskutov AV, Robida MD, et al. High-quality gene assembly directly from unpurified mixtures of microarray-synthesized oligonucleotides. Nucleic Acids Res, 2010, 38(19): e180. DOI: 10.1093/nar/gkq677 |
| [51] | Kosuri S, Eroshenko N, LeProust EM, et al. Scalable gene synthesis by selective amplification of DNA pools from high-fidelity microchips. Nat Biotechnol, 2010, 28(12): 1295–1299. DOI: 10.1038/nbt.1716 |
| [52] | Quan JY, Saaem I, Tang N, et al. Parallel on-chip gene synthesis and application to optimization of protein expression. Nat Biotechnol, 2011, 29(5): 449–452. DOI: 10.1038/nbt.1847 |
| [53] | Itaya M, Tsuge K, Koizumi M, et al. Combining two genomes in one cell: stable cloning of the Synechocystis PCC6803 genome in the Bacillus subtilis 168 genome. Proc Natl Acad Sci USA, 2005, 102(44): 15971–15976. DOI: 10.1073/pnas.0503868102 |
| [54] | Gibson DG, Benders GA, Axelrod KC, et al. One-step assembly in yeast of 25 overlapping DNA fragments to form a complete synthetic Mycoplasma genitalium genome. Proc Natl Acad Sci USA, 2008, 105(51): 20404–20409. DOI: 10.1073/pnas.0811011106 |
| [55] | Karas BJ, Jablanovic J, Sun LJ, et al. Direct transfer of whole genomes from bacteria to yeast. Nat Methods, 2013, 10(5): 410–412. DOI: 10.1038/nmeth.2433 |
| [56] | Karas BJ, Tagwerker C, Yonemoto IT, et al. Cloning the Acholeplasma laidlawii PG-8A genome in Saccharomyces cerevisiae as a yeast centromeric plasmid. ACS Synth Biol, 2012, 1(1): 22–28. DOI: 10.1021/sb200013j |
| [57] | Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894–898. DOI: 10.1038/nature08187 |
| [58] | Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc, 2013, 8(11): 2281–2308. DOI: 10.1038/nprot.2013.143 |
| [59] | Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096. DOI: 10.1126/science.1258096 |
| [60] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819–823. DOI: 10.1126/science.1231143 |
| [61] | Coleman JR, Papamichail D, Skiena S, et al. Virus attenuation by genome-scale changes in codon pair bias. Science, 2008, 320(5884): 1784–1787. DOI: 10.1126/science.1155761 |
| [62] | Gallagher RR, Patel JR, Interiano AL, et al. Multilayered genetic safeguards limit growth of microorganisms to defined environments. Nucleic Acids Res, 2015, 43(3): 1945–1954. DOI: 10.1093/nar/gku1378 |
| [63] | Cai YZ, Agmon N, Choi WJ, et al. Intrinsic biocontainment: multiplex genome safeguards combine transcriptional and recombinational control of essential yeast genes. Proc Natl Acad Sci USA, 2015, 112(6): 1803–1808. DOI: 10.1073/pnas.1424704112 |
 2017, Vol. 33
2017, Vol. 33