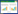中国科学院微生物研究所、中国微生物学会主办
文章信息
- 魏磊, 袁野, 汪小我
- Lei Wei, Ye Yuan, Xiaowo Wang
- 合成基因线路规模化设计面临的挑战
- Challenges in large-scale synthetic gene circuits design
- 生物工程学报, 2017, 33(3): 372-385
- Chin J Biotech, 2017, 33(3): 372-385
- 10.13345/j.cjb.160415
-
文章历史
- Received: November 1, 2016
- Accepted: January 16, 2017
2 清华信息科学与技术国家实验室 (筹) 生物信息学研究部,北京 100084
2 Tsinghua National Laboratory for Information Science and Technology, Beijing 100084, China
合成基因线路 (Synthetic gene circuit) 是指利用合成生物学的相关技术和方法,用DNA等生物分子构建出的在生命体内发挥特定功能的人工生物系统。合成基因线路研究具有重大的生物学意义和实用价值。例如,利用合成基因线路对生命体的代谢系统进行重构,可以使宿主细胞合成特定的化学物质,提高代谢工程的应用范围和产率[1-2];可以使细胞感知特定的生化信号[2],应用于环境监测、疾病诊断等领域[3];可以帮助发现和合成药物,在基因治疗、组织工程等医学应用上也有着非常广阔的前景[4-6];还可以作为研究复杂基因调控网络的重要工具,通过“以建而学”的方式帮助人们研究生命体内复杂的调控机制[5]。
理性设计对合成基因线路的发展具有十分重要的意义。合成基因线路构建的实验周期长,试错成本大,单纯依靠经验进行设计构建难以迅速得到满意的结果;此外,目前人们对基因元件的特性了解依然比较有限,多个元件组合在一起的效果常常难以预测,这制约着合成基因线路实现复杂的功能。理性设计可以通过理论计算、计算机仿真等手段,对合成基因线路的功能进行预测和优化,减少试错的过程,提高构建效率。
早期的合成基因线路设计主要借鉴了电子电路设计中的相关理念。研究者首先构建出基础的功能元件,如双稳态开关[7]、振荡器等[8],然后尝试对这些元件进行组装,实现更为复杂的功能。近年来,合成基因线路的设计和构建取得了一些可喜进展,人们构建出了信息存储器[9-11]、稳定可调的振荡器[12-13]、空间图样生成[14-15]和模拟量计算器[16]等一批功能模块。与此同时,研究者试图在合成基因线路的设计中引入设计标准化和设计自动化的概念,开发了合成生物学开放语言SBOL (Synthetic biology open language)[17],为描述合成基因线路提供了标准方法。基于此类描述语言,研究者开发了一些可以自动化设计合成基因线路的工具。例如MIT的Voigt课题组开发的基因线路自动化设计平台Cello,可以根据用户输入的描述语言自动地对一系列事前经过详细测定的相互绝缘的基础逻辑器件进行排列组合,并基于计算机仿真优化设计结果,最终将其编译为相应的DNA序列[18]。研究者基于这一平台,在大肠杆菌中对60个2输入或3输入逻辑门进行设计构建,实验验证的结果表明,75%的逻辑门的实际输出与设计预期基本相符。
在合成基因线路设计和构建过程中,研究者发现很多因素都会影响基因线路的功能,例如信号分子的绝缘性能、基因线路元器件的非线性特性、生命体内调控机制的复杂性、宿主和环境的异质性等。研究者针对这些因素,不断改进合成基因线路的设计原则,近期亦有相关综述论文对这些问题进行了总结[19-22]。
随着基因线路规模的扩大和应用范围的拓展,传统的合成基因线路设计思路面临着新的挑战。近年来,尽管DNA合成与组装技术取得了巨大进步,合成基因线路规模的增长依然十分缓慢。目前在单个细胞内实现的最大规模合成基因线路,其中的逻辑门不超过10个,启动子的数量也只能达到10个左右[18-19]。其中,基因表达噪声和竞争效应是制约基因线路规模化的重要因素。随着基因线路规模的扩大,一方面基因表达噪声在合成基因线路中传播、积累、甚至放大,其直接影响着合成基因线路的准确性和稳定性;另一方面人工基因线路与生命体内的其他生命过程共同使用宿主细胞内有限的物质、能量资源,这种对有限资源的竞争效应不仅制约着合成基因线路自身的性能,对宿主细胞的生存状况也有不可忽视的影响。因此,本文从基因线路规模化设计的角度出发,重点关注基因表达噪声和竞争效应对合成基因线路造成的干扰,并从理性设计的角度,对解决这些问题的思路进行展望。
2 基因表达噪声对合成基因线路的影响系统在一定的外界扰动和内部波动下仍能正常维持性能的能力被称为鲁棒性。系统鲁棒性是工程设计中的一个重要指标。理想的合成基因线路设计应当能够使其具有较好的鲁棒性[23]。然而在实践中,合成基因线路的性能与设计预期之间经常显示出较大差异,甚至完全不能执行预期的功能。其中,基因表达的随机噪声是导致这种现象的一个重要原因。了解基因表达噪声的来源和性质,有助于我们更好地理解合成基因线路在生命系统中的实际工作情况和性能异常的原因;同时,在设计合成基因线路时规避噪声对基因线路性能的影响,可以提高基因线路的鲁棒性,使基因线路在更多场景下更好地发挥功能。
2.1 基因表达过程中的随机噪声基因表达的过程不可避免地伴随着随机效应与噪声[24-25]。著名物理学家薛定谔在《What is life?》[26]一书中提出
|
| 图 1 分子数对生化反应随机性的影响 Figure 1 Stochastic effects at small molecule numbers. Deterministic model can well describe reaction systems with large molecular number. But stochastic effects are prominent in small biochemical reaction systems with low amounts of molecules. Circles with different colors represent different kinds of molecules in this reaction |
| |
基因表达噪声对生命体的影响具有两面性。一方面基因表达噪声会影响下游基因的功能,增加表型的异质性;但同时,基因表达的随机性在细胞分化、机体发育等过程中发挥着重要作用,适当强度的基因表达噪声可以增强生命体的适应能力[27-29]。
随着流式细胞术、微流控技术和单细胞测序技术的发展,人们开始逐渐了解基因表达噪声的性质和产生原因。基因表达噪声按照其产生机理,可以划分为内源噪声和外源噪声[30]。生化反应中内蕴的随机性会在基因的转录、翻译过程中产生噪声,这部分噪声被称为内源噪声。例如,启动子在开启和关闭状态之间的随机转换会使转录过程间歇进行,这会使不同细胞之间的转录本产生较大差异[31-33]。与之相对的,参与化学反应的其他物质、能量的异质性引起的细胞间的异质性被称为外源噪声[34]。例如,细胞体积[35]、线粒体数量[36]和细胞密度[37]等均会在基因表达的过程中引入噪声。
2.2 噪声对合成基因线路中的影响基因表达噪声会影响合成基因线路的鲁棒性和准确性[38] (图 2)。基因表达噪声会影响逻辑门对信号的处理能力,使基因线路对外界刺激作出错误响应;基因表达噪声还会在基因网络中传播,干扰基因正常发挥功能[39],这种干扰在信号多级传递时尤为显著[40]。例如,噪声对振荡器的鲁棒性有决定性影响。振荡器是最早被设计构建的动态合成基因线路之一。当利用简单的三节点环形结构设计振荡器时,只有40%的细胞显示出了振荡的效果,且其振荡并不规律[8]。近年来人们对振荡器的结构进行了诸多改进[41-44],然而,尽管数学模型的结果预测这些基因线路可以在较大的参数范围内稳定工作,它们在振幅和周期上仍显示出不规律性。噪声无处不在,又难以被完全消除,如何在合成基因线路中控制噪声水平,降低其对合成基因线路性能的影响,是合成基因线路设计中必须考虑的问题。

|
| 图 2 噪声对基因调控准确性的影响 Figure 2 Stochastic noise affects the accuracy of gene regulation. In an ideal deterministic model, gene regulation process will generate accurate responses according to different levels of inputs. In reality, however, the responses are noisy because of the stochasticity of input molecular numbers and regulatory process |
| |
在设计合成基因线路时,研究者往往只对与基因线路直接相关的组分进行建模和分析。然而,很多看似与基因线路无关的组分也会对基因线路的功能产生影响 (图 3)。由于细胞内的能量、资源是有限的,基因线路的元件会与细胞内的其他生物过程产生竞争,对有限的资源进行重新分配,这不仅会影响宿主细胞的状态,也会影响合成基因线路本身的功能。此外,合成基因线路的元件也有可能在内部产生竞争效应,这些竞争效应使看似“绝缘”的模块间产生相互作用,对基因线路本身的影响是不可忽略的。随着基因线路的规模不断增大,竞争效应的影响越来越难以被忽视 (图 4)。因此,在设计合成基因线路时,必须将竞争效应作为约束条件,纳入整体考虑。
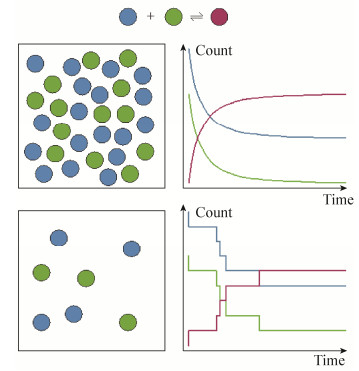
|
| 图 3 分子竞争效应原理示意 Figure 3 Molecular competition effect. A limiting amount of regulator (green triangle) is shared by multiple types of targets (red and yellow circles). When its number increases, target A attracts more regulators, which results in less amount of regulator to bind target B. In this scenario target A can modulate target B's activity even without physical interaction with B |
| |
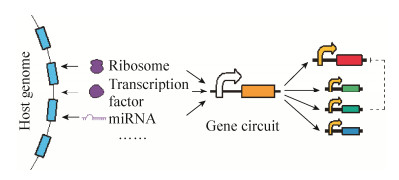
|
| 图 4 合成基因线路与细胞内各组分间的竞争效应 Figure 4 Synthetic genes compete limiting resources with endogenous genes and other synthetic genes in host cells |
| |
竞争效应广泛存在于生命体中。例如ceRNA (Competing endogenous RNA,竞争性内源RNA) 效应[45]就是一种重要的竞争性现象。受到同一microRNA调控的不同靶RNA之间会对microRNA产生竞争,进而引发靶RNA之间的间接调控效应,这一效应被称为ceRNA效应。我们通过数学建模和合成生物学实验研究表明,ceRNA竞争效应的强弱与microRNA和ceRNA的丰度和结合能力有着密切的联系,并且竞争效应会在microRNA调控中引入超敏感性[46]。最近有研究发现,当较多的DNA靶点竞争同一转录因子时,转录因子对基因的调控也会产生明显的竞争效应[47]。合成基因线路来自宿主细胞外,拷贝数和表达量一般较高,其中的竞争效应也尤为显著。此外,面对有限的资源,细胞内形成了复杂的资源分配机制,不同生化过程在各个层面互相竞争,影响细胞在不同外界条件下的基因表达和增殖速率[12, 48-50]。竞争效应在多个层次影响着生命活动,其作用不容忽视。
3.2 竞争效应对基因线路性能的影响目前的绝大部分合成基因线路设计中,基因的表达量和拷贝数会在一定范围内尽可能地高,往往忽略细胞本身资源的限制。然而,在细胞中的能量、核糖体、蛋白质等资源有限的情况下,合成基因线路的功能会受到影响,拷贝数和表达量过高反而会使基因线路出现异常。有研究建立了描述基因表达与细胞资源间关系的定量模型,并基于模型预测了外界负载不同时,振荡器线路的工作情况[12]。当合成基因线路的拷贝数很大时,振荡器的振幅并不会随拷贝数的增大而增大,反而会有一定程度的降低。
当细胞内的资源不足时,对资源的利用就会出现排队现象。例如,在合成基因线路的构建中,蛋白质降解标签被广泛地用于保证基因的表达量快速变化,这一点在具有动态功能的基因线路中尤为重要。然而研究发现,蛋白质降解标签会竞争蛋白酶ClpXP,当有过多的蛋白质被降解标签标记时,蛋白质的降解过程就会出现排队现象,这可能会使动态线路原有的功能发生紊乱,甚至完全丧失[51]。
当一个调控元件调控多个下游元件时,被调控元件之间也会产生竞争,并反过来影响上游元件的调控能力,产生负载效应。例如,在合成基因线路中的非门后设计过多的下游元件,会影响该非门对信号的响应速度。提高该元件的表达量会减轻这一现象[52];然而,过量的外源基因表达会带来细胞毒性,如何把握两者的平衡是应用此类基因线路时必须面对的问题[53]。有研究者在酵母中利用能够快速反应的磷酸转移蛋白设计了驱动负载的模块,通过对上游元件输出信号进行迅速传递和放大减轻负载效应,使基因线路在下游的被调控元件较多时仍能正常发挥功能[54]。
3.3 竞争效应对细胞状态的影响某些基因线路中的调控元件会影响宿主细胞本身的生长状况,甚至对细胞有较大毒副作用[55-56]。比如某些携带类似于核糖体结合位点的小RNA可能会竞争宿主细胞内的核糖体,引起基因表达的不稳定,阻碍宿主细胞的正常生长[57]。当合成基因线路引入的外源基因竞争宿主资源,影响宿主本身RNA和蛋白质的合成时,宿主的生长速度就会明显变慢[58]。当合成基因线路的规模变大时,这种效应会尤为明显[59],甚至会诱发细胞对那些被视作“负担”的合成基因线路的排斥,例如遗传突变率的显著上升,导致合成基因线路难以稳定遗传。如何在合成基因线路稳定高表达和减小对宿主细胞干扰这一矛盾中找到平衡点,是合成基因线路在应用中亟待解决的问题。
4 可能的解决方法和未来的发展方向基因表达噪声和竞争效应二者间有着千丝万缕的联系。要降低基因表达中的噪声往往需要增加基因表达的强度,这会对细胞内有限的资源和能量产生竞争;反之,如果通过减少合成基因线路的元件数和表达量来降低其对资源的占用,则会增加基因线路的噪声水平和表达的不确定性。因此,合成基因线路的设计中必须对这两种因素进行综合考虑,平衡对基因线路的影响。下面我们从理性设计的角度出发,归纳近年来一些新的设计思路和进展,并展望这一领域未来一些可能的发展方向。
4.1 模拟运算设计与布尔逻辑设计在合成生物学发展初期,基因线路的设计思路大多借鉴了在信息科学领域广泛应用的布尔逻辑运算,然而,随着基因线路规模的增加,利用布尔逻辑进行运算的基因线路可能无法良好地发挥功能[60]。例如,如果基于布尔逻辑的思路在细胞内设计加法器,其基因线路的规模会随着计算位数的增加而快速膨胀,对基础元件有限的合成基因线路设计带来了巨大挑战。此外,多逻辑门的基因线路会消耗大量的细胞资源,影响基因线路的正常工作和宿主细胞的状态。而另一方面,生命活动可以看作是一系列有序的化学反应组合,其中相当多的调控作用都是通过浓度这种模拟信号来进行控制,因而模拟量运算在自然界中广泛存在[61-62],在对不同的环境信号进行定量响应时发挥重要作用;类似地,在合成基因线路中,引入模拟量运算可以有效地突破资源和环境的限制,增加基因线路的运算能力,拓展合成基因线路的应用前景。例如,研究人员利用3组基因元件,构建了对数计算模块,并以此为基础,构建了除法、幂律等一系列的模拟运算线路[16]。这些线路具有较大的动态范围,传递函数也可以在一定范围内进行调整。
模拟量运算为系统节约资源、提高效率,而布尔量运算则为系统提升准确率和稳定性。与布尔量运算相比,模拟量运算可以在更少的模块和能耗情况下,解决相同的问题,这可以有效规避合成基因线路对元件数量、资源、能量的限制。然而,模拟量运算元件会引入较大的噪声,在参与传递信号的分子数较低时这种现象尤为明显。在神经系统中,生命体把模拟运算和布尔逻辑预算相结合,在保证系统准确率的同时有效降低了功耗[61, 63]。如何在合成基因线路的设计中将二者结合发挥作用,在减少能量和资源消耗的同时增加计算的准确性,是合成生物学的工程实践中需要解决的重要问题[64]。
4.2 基于网络拓扑的鲁棒性设计基因往往通过组成基因网络来发挥功能。某些具有特定拓扑结构的基因网络出现频率显著高于其他结构,这些拓扑结构被称为基因网络模体 (Network motif)[65]。研究发现,特定的网络结构可以实现较为复杂的功能,如瞬态响应[66]、细胞命运决策[67]、生物振荡[68]和倍数感知[69]等。研究者通过对这些常见结构进行仿真,揭示了这些结构在面对外界信号时的响应模式;并在自然界中存在的基因线路中也观察到了类似的现象,从而阐明了这些拓扑结构的生物学意义[70]。
在自然界中,生命体利用特定的网络结构抵抗噪声,增加系统稳定性[71]。自抑制的基因建立稳态的时间更快,其稳态对短时的信号扰动和自身的表达水平涨落不敏感,因而具有更小的表达噪声[72]。具有某些特定网络模体的合成基因线路可以过滤短时的脉冲信号,而仅对持续时间较长的信号作出响应[70]。视网膜紫质对光子的接收过程利用多步磷酸化反应控制噪声,其对光子的响应的离散程度与磷酸化位点数呈平方根反比关系[73]。
有研究者通过穷举3个基因构成的所有网络拓扑结构,筛选出具有较好适应能力的基因网络拓扑结构[74],并由此在一定程度上解释了具有较好适应能力的网络模体之所以存在的原因。以此为基础,通过对基因线路进行结构设计,研究者可以筛选出那些对参数不敏感的基因网络拓扑,使合成基因线路在外源噪声干扰下依然能稳定工作。对基因网络的拓扑结构进行重构,可以有效地降低表达过程中个体间的差异[75]。此外,有研究指出,通过对简单的生化反应进行组合,即可在一定程度上实现滤波器的功能;基于这一现象提出的滤波器的优化设计理论,可以对系统内部的噪声性质进行大概估计,并可应用于指导合成基因线路的设计,提高基因线路的鲁棒性[76]。
以目前的计算能力,通过穷举多节点拓扑进行结构设计依然是不现实的。利用控制理论对现有的知识进行充分理解,抽象出一定的设计原则[77],同时拓展新的建模方法,可能可以在一定程度上缩小备选解的范围,提高设计效率。
4.3 基因线路设计的信息传递理论生命体中基因调控可以抽象为信息传递的过程:细胞接收上游信号,对这些信号进行计算处理,并依据其结果决策下游的输出信号 (图 5)。信息传递过程可以利用香农信息论定量刻画输入输出分布间的联系。目前,依赖荧光蛋白技术和微流控技术的发展,已有一些工作尝试利用信息论中的互信息和信道容量等概念对基因网络的信号传递能力进行定量刻画[78-80]。例如,研究者利用信息论的相关研究方法,成功揭示了果蝇发育过程中,胚胎如何利用调控基因的表达量信息精确决定位置信息[81-82]。利用信道容量衡量不同信号通路的信息传递能力,研究者刻画出了某些特定信号通路信道容量的大致范围,解析了不同的信号通路结构对信息传递的影响,并借此推断细胞如何提高决策的鲁棒性[83-85]。
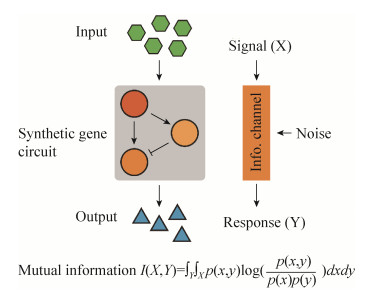
|
| 图 5 合成基因线路的信息传递模型 Figure 5 Information transmission model for synthetic gene circuit. Information theory provides a mathematical framework to describe the fidelity of the synthetic circuit to response input signals |
| |
借助信息论的相关概念,一些工作还揭示了基因网络的某些性质对信息传递性能的影响。例如,负反馈可以降低基因表达的噪声,但在一定条件下也会降低信号通路的信道容量[83]。利用药物对信号通路进行干预,显示某些信号通路结构上的改变可以补偿信号与响应之间的互信息[84]。信息论的相关理论指出,信息在每一步传递的过程中都会丢失一部分,且多个信号组合可以增加信息量,这些都要求在基因网络中两个节点之间的距离应当较短,自然界的基因网络中广泛存在的“小世界网络”恰好具备这一性质[80],这为合成基因线路的设计提出了一些基本的设计原则。
很多合成基因线路的核心功能可以抽象为一个感知决策过程,即基因线路通过感知细胞内基因表达或信号分子的浓度差异,来作出不同的响应 (图 6)。利用互信息、信道容量等概念,我们可以定量刻画基因线路的决策准确度,这些概念与传统方法相比,更贴近这一感知决策过程的本质[78]。将互信息、信道容量等度量引入合成基因线路的设计、构建和测试中,可以在更基础的层面对合成基因线路的性能进行评价。此外,不同的控制目标都有一个理论上的信息量的下界,依据基因线路的功能计算出这一下界,并基于信息传递的原理对生物信道的不同部分的信息传递能力进行刻画,有助于在理论的层面指导研究者在面对不同的设计目标时有针对性地理性设计合成基因线路。

|
| 图 6 合成基因线路的准确感知决策 Figure 6 Accurate decision making of synthetic gene circuits. Synthetic gene circuits are desired to integrate multiple kinds of (noisy) input signals, and compute sophisticated functions to make accurate decision |
| |
生命系统中,各个组分的表达量并不仅仅依靠静态的浓度值发挥调控作用,而是具有非常丰富的动态调控机制[29, 86]。例如,真核细胞中的Ras/Erk信号通路在接收上游信号刺激时会根据上游脉冲信号的持续时间开启不同的下游基因,这解释了为什么单一通路却可以控制多种不同的下游响应[87]。在酵母中,转录因子Crz1展现出类似于频率调制的调控机制[88]。在人类细胞中,p53在受到DNA断裂信号的刺激时,会通过产生不同的动态信号来决定细胞命运[89]。
如果仅以静态的输入输出估算基因调控中的信道容量,单种信号分子承载的信息量一般只有1比特左右[78, 84]。有研究者把调控过程视为一个时序信号,以动态信号作为输入计算,其信道容量显著增大[90],这意味着动态信号可以承载更多的信息量。事实上,利用动态信号传递信息可以为细胞节约有限的资源[91-92]。我们推测,如果在合成基因线路的设计中充分考虑动态特性,可以利用更少的元件和更少的分子数传递同等的信息量,同时减少基因线路对生命体本身的负载。
动态信号还可以降低噪声对合成基因线路的影响。对单个细胞进行时序观测发现,细胞内的基因表达差异小于细胞间的基因表达差异[87]。理论分析和实验结果表明,增加观测的时间点可以大幅增加对外源噪声的推断能力[90]。因此,在合成基因线路中利用动态信号进行运算处理可以在一定程度上抵抗外源噪声带来的不确定性。此外,针对动态信号的特点有针对性地设计基因线路也可以降低基因表达噪声。例如,前述Ras/Erk信号通路表现为一个频带很宽的低通滤波器,这一性质可以阻止持续时间短的脉冲信号向下传递,同时对持续时间较长的信号作出正确响应[87]。通过增大启动子开启和关闭的状态切换频率,利用动态信号调制基因的转录过程,可以降低细胞表型的异质性[93]。
5 结语近年来,随着合成生物学的发展,合成基因线路能够实现的功能越来越丰富。但随着基因线路规模的增加,简单的设计原则已经无法满足日益增长的大规模合成基因线路的设计需要。许多之前被忽略的因素对大规模基因线路的性能造成了巨大影响,基因表达过程中的随机噪声和基因线路对资源、能量、调控元件的竞争效应对合成基因线路的影响愈发凸显。基因表达的随机噪声与表达过量带来的竞争效应呈现此消彼长的关系,如何通过理性设计平衡二者的效应,降低其对基因线路的影响,是合成基因线路设计的重要课题。
合成基因线路的表达量越高,信噪比越高,其理论表现应该越好,但考虑到其会占用更多的细胞资源,恶化宿主的生存状态,所以在设计时应该尽量将外源基因的表达控制在较低水平。这本质上是一个功率有限且具有噪声的信息传递过程。因此,可以采用信息论中的有关理论来指导设计、评价基因线路。同时,信号的传递过程是一个动态的时序过程,利用动态信号传递信息,同时针对动态信号的特点针对性地设计基因线路,可以增加合成基因线路的信道容量,同时降低噪声水平。
传统的合成基因线路设计往往刻意规避外源基因线路与细胞内环境的相互作用,试图保证合成基因线路与宿主细胞之间的绝缘,使其独立发挥功能。然而,宿主细胞内的各种因素对合成基因线路的功能有着无法规避的影响,这可能导致合成基因线路无法正常发挥功能。如果将这些因素充分考虑,将合成基因线路嵌入到细胞内环境中,充分利用自然界中的丰富调控机制,使其真正作为生命体的一部分发挥作用,或许可以在一定程度上解决合成基因线路规模受限、受噪声影响较大、与内源基因相互竞争等一系列问题。例如,最近有研究将竞争效应作为一种调控机制,通过引入外源DNA对合成基因线路的调控元件进行竞争,改善调控元件的响应特性,降低了噪声对基因线路的影响,从而提升环形振荡器的稳定性、适应性和同步性[94]。这提示我们,在基因线路设计中,竞争效应和基因表达噪声并不总是难以同时兼顾的,如果善用竞争效应的调控性质,或许可以在降低噪声等方面改善基因线路的性能。
很多合成基因线路的设计灵感都来自生命体本身。漫长的进化过程对生命体精巧的优化从未停止,更透彻地了解生命体本身多样的控制基因表达差异的机制,并将其借鉴到合成生物学领域,将系统生物学与合成生物学更加紧密地结合,可以为这一领域提供新的设计思路。
随着合成生物学的进一步发展,合成基因线路必定要向着更精准、更鲁棒、更大规模的方向发展,在这个过程中,理性、定量设计将起到关键性作用。综合控制科学、信息论和系统生物学的有关理论,发展更合理、更高效的数理模型对合成基因线路进行刻画,将有助于我们更好面对合成基因线路规模化设计的挑战。
| [1] | Keasling JD. Synthetic biology and the development of tools for metabolic engineering. Metab Eng, 2012, 14(3): 189–195. DOI: 10.1016/j.ymben.2012.01.004 |
| [2] | Khalil AS, Collins JJ. Synthetic biology: applications come of age. Nat Rev Genet, 2010, 11(5): 367–379. DOI: 10.1038/nrg2775 |
| [3] | Slomovic S, Pardee K, Collins JJ. Synthetic biology devices for in vitro and in vivo diagnostics. Proc Natl Acad Sci USA, 2015, 112(47): 14429–14435. DOI: 10.1073/pnas.1508521112 |
| [4] | Ruder WC, Lu T, Collins JJ. Synthetic biology moving into the clinic. Science, 2011, 333(6047): 1248–1252. DOI: 10.1126/science.1206843 |
| [5] | Lienert F, Lohmueller JJ, Garg A, et al. Synthetic biology in mammalian cells: next generation research tools and therapeutics. Nat Rev Mol Cell Biol, 2014, 15(2): 95–107. DOI: 10.1038/nrm3738 |
| [6] | Medema MH, Breitling R, Bovenberg R, et al. Exploiting plug-and-play synthetic biology for drug discovery and production in microorganisms. Nat Rev Microbiol, 2011, 9(2): 131–137. DOI: 10.1038/nrmicro2478 |
| [7] | Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli. Nature, 2000, 403(6767): 339–342. DOI: 10.1038/35002131 |
| [8] | Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403(6767): 335–338. DOI: 10.1038/35002125 |
| [9] | Siuti P, Yazbek J, Lu TK. Synthetic circuits integrating logic and memory in living cells. Nat Biotechnol, 2013, 31(5): 448–452. DOI: 10.1038/nbt.2510 |
| [10] | Bonnet J, Subsoontorn P, Endy D. Rewritable digital data storage in live cells via engineered control of recombination directionality. Proc Natl Acad Sci USA, 2012, 109(23): 8884–8889. DOI: 10.1073/pnas.1202344109 |
| [11] | Yang L, Nielsen AAK, Fernandez-Rodriguez J, et al. Permanent genetic memory with >1-byte capacity. Nat Methods, 2014, 11(12): 1261–1266. DOI: 10.1038/nmeth.3147 |
| [12] | Weiße AY, Oyarzún DA, Danos V, et al. Mechanistic links between cellular trade-offs, gene expression, and growth. Proc Natl Acad Sci USA, 2015, 112(9): E1038–E1047. DOI: 10.1073/pnas.1416533112 |
| [13] | Sowa SW, Gelderman G, Contreras LM. Advances in synthetic dynamic circuits design: using novel synthetic parts to engineer new generations of gene oscillations. Curr Opin Biotechnol, 2015, 36: 161–167. DOI: 10.1016/j.copbio.2015.08.020 |
| [14] | Basu S, Gerchman Y, Collins CH, et al. A synthetic multicellular system for programmed pattern formation. Nature, 2005, 434(7037): 1130–1134. DOI: 10.1038/nature03461 |
| [15] | Liu CL, Fu XF, Liu LZ, et al. Sequential establishment of stripe patterns in an expanding cell population. Science, 2011, 334(6053): 238–241. DOI: 10.1126/science.1209042 |
| [16] | Daniel R, Rubens JR, Sarpeshkar R, et al. Synthetic analog computation in living cells. Nature, 2013, 497(7451): 619–623. DOI: 10.1038/nature12148 |
| [17] | Galdzicki M, Clancy KP, Oberortner E, et al. The synthetic biology open language (SBOL) provides a community standard for communicating designs in synthetic biology. Nat Biotechnol, 2014, 32(6): 545–550. DOI: 10.1038/nbt.2891 |
| [18] | Nielsen AAK, Der BS, Shin J, et al. Genetic circuit design automation. Science, 2016, 352(6281): aac7341. DOI: 10.1126/science.aac7341 |
| [19] | Purnick PEM, Weiss R. The second wave of synthetic biology: from modules to systems. Nat Rev Mol Cell Biol, 2009, 10(6): 410–422. |
| [20] | Cardinale S, Arkin AP. Contextualizing context for synthetic biology-identifying causes of failure of synthetic biological systems. Biotechnol J, 2012, 7(7): 856–866. DOI: 10.1002/biot.201200085 |
| [21] | Brophy JA, Voigt CA. Principles of genetic circuit design. Nat Methods, 2014, 11(5): 508–520. DOI: 10.1038/nmeth.2926 |
| [22] | Way JC, Collins JJ, Keasling JD, et al. Integrating biological redesign: where synthetic biology came from and where it needs to go. Cell, 2014, 157(1): 151–161. DOI: 10.1016/j.cell.2014.02.039 |
| [23] | Lucks JB, Qi L, Whitaker WR, et al. Toward scalable parts families for predictable design of biological circuits. Curr Opin Microbiol, 2008, 11(6): 567–573. DOI: 10.1016/j.mib.2008.10.002 |
| [24] | Rao CV, Wolf DM, Arkin AP. Control, exploitation and tolerance of intracellular noise. Nature, 2002, 420(6912): 231–237. DOI: 10.1038/nature01258 |
| [25] | Kærn M, Elston TC, Blake WJ, et al. Stochasticity in gene expression: from theories to phenotypes. Nat Rev Genet, 2005, 6(6): 451–464. DOI: 10.1038/nrg1615 |
| [26] | Schrödinger E. What is Life?. Cambridge: Cambridge University Press, 1992. |
| [27] | Raser JM, O'Shea EK. Noise in gene expression: origins, consequences, and control. Science, 2005, 309(5743): 2010–2013. DOI: 10.1126/science.1105891 |
| [28] | Losick R, Desplan C. Stochasticity and cell fate. Science, 2008, 320(5872): 65–68. DOI: 10.1126/science.1147888 |
| [29] | Levine JH, Lin YH, Elowitz MB. Functional roles of pulsing in genetic circuits. Science, 2013, 342(6163): 1193–1200. DOI: 10.1126/science.1239999 |
| [30] | Elowitz MB, Levine AJ, Siggia ED, et al. Stochastic gene expression in a single cell. Science, 2002, 297(5584): 1183–1186. DOI: 10.1126/science.1070919 |
| [31] | Golding I, Paulsson J, Zawilski SM, et al. Real-time kinetics of gene activity in individual bacteria. Cell, 2005, 123(6): 1025–1036. DOI: 10.1016/j.cell.2005.09.031 |
| [32] | Suter DM, Molina N, Gatfield D, et al. Mammalian genes are transcribed with widely different bursting kinetics. Science, 2011, 332(6028): 472–474. DOI: 10.1126/science.1198817 |
| [33] | Zenklusen D, Larson DR, Singer RH. Single-RNA counting reveals alternative modes of gene expression in yeast. Nat Struct Mol Biol, 2008, 15(12): 1263–1271. DOI: 10.1038/nsmb.1514 |
| [34] | Swain PS, Elowitz MB, Siggia ED. Intrinsic and extrinsic contributions to stochasticity in gene expression. Proc Natl Acad Sci USA, 2002, 99(20): 12795–12800. DOI: 10.1073/pnas.162041399 |
| [35] | Raser JM, O'Shea EK. Control of stochasticity in eukaryotic gene expression. Science, 2004, 304(5678): 1811–1814. DOI: 10.1126/science.1098641 |
| [36] | das Neves RP, Jones NS, Andreu L, et al. Connecting variability in global transcription rate to mitochondrial variability. PLoS Biol, 2010, 8(12): e1000560. DOI: 10.1371/journal.pbio.1000560 |
| [37] | Battich N, Stoeger T, Pelkmans L. Control of transcript variability in single mammalian cells. Cell, 2015, 163(7): 1596–1610. DOI: 10.1016/j.cell.2015.11.018 |
| [38] | Raj A, van Oudenaarden A. Nature, nurture, or chance: stochastic gene expression and its consequences. Cell, 2008, 135(2): 216–226. DOI: 10.1016/j.cell.2008.09.050 |
| [39] | Pedraza JM, van Oudenaarden A. Noise propagation in gene networks. Science, 2005, 307(5717): 1965–1969. DOI: 10.1126/science.1109090 |
| [40] | Hooshangi S, Thiberge S, Weiss R. Ultrasensitivity and noise propagation in a synthetic transcriptional cascade. Proc Natl Acad Sci USA, 2005, 102(10): 3581–3586. DOI: 10.1073/pnas.0408507102 |
| [41] | Fung E, Wong WW, Suen JK, et al. A synthetic gene-metabolic oscillator. Nature, 2005, 435(7038): 118–122. DOI: 10.1038/nature03508 |
| [42] | Stricker J, Cookson S, Bennett MR, et al. A fast, robust and tunable synthetic gene oscillator. Nature, 2008, 456(7221): 516–519. DOI: 10.1038/nature07389 |
| [43] | Tigges M, Marquez-Lago TT, Stelling J, et al. A tunable synthetic mammalian oscillator. Nature, 2009, 457(7227): 309–312. DOI: 10.1038/nature07616 |
| [44] | Mondragón-Palomino O, Danino T, Selimkhanov J, et al. Entrainment of a population of synthetic genetic oscillators. Science, 2011, 333(6047): 1315–1319. DOI: 10.1126/science.1205369 |
| [45] | Tay Y, Rinn J, Pandolfi PP. The multilayered complexity of ceRNA crosstalk and competition. Nature, 2014, 505(7483): 344–352. DOI: 10.1038/nature12986 |
| [46] | Yuan Y, Liu B, Xie P, et al. Model-guided quantitative analysis of microRNA-mediated regulation on competing endogenous RNAs using a synthetic gene circuit. Proc Natl Acad Sci USA, 2015, 112(10): 3158–3163. DOI: 10.1073/pnas.1413896112 |
| [47] | Brewster RC, Weinert FM, Garcia HG, et al. The transcription factor titration effect dictates level of gene expression. Cell, 2014, 156(6): 1312–1323. DOI: 10.1016/j.cell.2014.02.022 |
| [48] | Maitra A, Dill KA. Bacterial growth laws reflect the evolutionary importance of energy efficiency. Proc Natl Acad Sci USA, 2015, 112(2): 406–411. DOI: 10.1073/pnas.1421138111 |
| [49] | Scott M, Klumpp S, Mateescu EM, et al. Emergence of robust growth laws from optimal regulation of ribosome synthesis. Mol Syst Biol, 2014, 10(8): 747. DOI: 10.15252/msb.20145379 |
| [50] | Hui S, Silverman JM, Chen SS, et al. Quantitative proteomic analysis reveals a simple strategy of global resource allocation in bacteria. Mol Syst Biol, 2015, 11(2): e784. DOI: 10.15252/msb.20145697 |
| [51] | Cookson NA, Mather WH, Danino T, et al. Queueing up for enzymatic processing: correlated signaling through coupled degradation. Mol Syst Biol, 2011, 7: 561. |
| [52] | Del Vecchio D, Ninfa AJ, Sontag ED. Modular cell biology: retroactivity and insulation. Mol Syst Biol, 2008, 4: 161. |
| [53] | Jayanthi S, Nilgiriwala KS, Del Vecchio D. Retroactivity controls the temporal dynamics of gene transcription. ACS Synth Biol, 2013, 2(8): 431–441. DOI: 10.1021/sb300098w |
| [54] | Mishra D, Rivera PM, Lin A, et al. A load driver device for engineering modularity in biological networks. Nat Biotechnol, 2014, 32(12): 1268–1275. DOI: 10.1038/nbt.3044 |
| [55] | Rhodius VA, Segall‐Shapiro TH, Sharon BD, et al. Design of orthogonal genetic switches based on a crosstalk map of σs, anti-σs, and promoters. Mol Syst Biol, 2013, 9: 702. |
| [56] | Stanton BC, Nielsen AA, Tamsir A, et al. Genomic mining of prokaryotic repressors for orthogonal logic gates. Nat Chem Biol, 2014, 10(2): 99–105. |
| [57] | Callura JM, Dwyer DJ, Isaacs FJ, et al. Tracking, tuning, and terminating microbial physiology using synthetic riboregulators. Proc Natl Acad Sci USA, 2010, 107(36): 15898–15903. DOI: 10.1073/pnas.1009747107 |
| [58] | Dong H, Nilsson L, Kurland CG. Gratuitous overexpression of genes in Escherichia coli leads to growth inhibition and ribosome destruction. J Bacteriol, 1995, 177(6): 1497–1504. DOI: 10.1128/jb.177.6.1497-1504.1995 |
| [59] | Arkin AP, Fletcher DA. Fast, cheap and somewhat in control. Genome Biol, 2006, 7(8): 114. DOI: 10.1186/gb-2006-7-8-114 |
| [60] | Sarpeshkar R. Analog synthetic biology. Philos Trans A Math Phys Eng Sci, 2014, 372(2012): 20130110. DOI: 10.1098/rsta.2013.0110 |
| [61] | Clark B, Häusser M. Neural coding: hybrid analog and digital signalling in axons. Curr Biol, 2006, 16(15): R585–R588. DOI: 10.1016/j.cub.2006.07.007 |
| [62] | Giorgetti L, Siggers T, Tiana G, et al. Noncooperative interactions between transcription factors and clustered DNA binding sites enable graded transcriptional responses to environmental inputs. Mol Cell, 2010, 37(3): 418–428. DOI: 10.1016/j.molcel.2010.01.016 |
| [63] | Sarpeshkar R. Analog versus digital: extrapolating from electronics to neurobiology. Neural Comput, 1998, 10(7): 1601–1638. DOI: 10.1162/089976698300017052 |
| [64] | Purcell O, Lu TK. Synthetic analog and digital circuits for cellular computation and memory. Curr Opin Biotechnol, 2014, 29: 146–155. DOI: 10.1016/j.copbio.2014.04.009 |
| [65] | Milo R, Shen-Orr S, Itzkovitz S, et al. Network motifs: simple building blocks of complex networks. Science, 2002, 298(5594): 824–847. DOI: 10.1126/science.298.5594.824 |
| [66] | Shen-Orr SS, Milo R, Mangan S, et al. Network motifs in the transcriptional regulation network of Escherichia coli. Nat Genet, 2002, 31(1): 64–68. DOI: 10.1038/ng881 |
| [67] | Brandman O, Ferrell Jr JE, Li R, et al. Interlinked fast and slow positive feedback loops drive reliable cell decisions. Science, 2005, 310(5747): 496–498. DOI: 10.1126/science.1113834 |
| [68] | Tsai TYC, Choi YS, Ma WZ, et al. Robust, tunable biological oscillations from interlinked positive and negative feedback loops. Science, 2008, 321(5885): 126–129. DOI: 10.1126/science.1156951 |
| [69] | Goentoro L, Shoval O, Kirschner MW, et al. The incoherent feedforward loop can provide fold-change detection in gene regulation. Mol Cell, 2009, 36(5): 894–899. DOI: 10.1016/j.molcel.2009.11.018 |
| [70] | Alon U. Network motifs: theory and experimental approaches. Nat Rev Genet, 2007, 8(6): 450–461. DOI: 10.1038/nrg2102 |
| [71] | Chalancon G, Ravarani CNJ, Balaji S, et al. Interplay between gene expression noise and regulatory network architecture. Trends Genet, 2012, 28(5): 221–232. DOI: 10.1016/j.tig.2012.01.006 |
| [72] | Becskei A, Serrano L. Engineering stability in gene networks by autoregulation. Nature, 2000, 405(6786): 590–593. DOI: 10.1038/35014651 |
| [73] | Doan T, Mendez A, Detwiler PB, et al. Multiple phosphorylation sites confer reproducibility of the rod's single-photon responses. Science, 2006, 313(5786): 530–533. DOI: 10.1126/science.1126612 |
| [74] | Ma WZ, Trusina A, El-Samad H, et al. Defining network topologies that can achieve biochemical adaptation. Cell, 2009, 138(4): 760–73. DOI: 10.1016/j.cell.2009.06.013 |
| [75] | Çağatay T, Turcotte M, Elowitz MB, et al. Architecture-dependent noise discriminates functionally analogous differentiation circuits. Cell, 2009, 139(3): 512–522. DOI: 10.1016/j.cell.2009.07.046 |
| [76] | Zechner C, Seelig G, Rullan M, et al. Molecular circuits for dynamic noise filtering. Proc Natl Acad Sci USA, 2016, 113(17): 4729–4734. DOI: 10.1073/pnas.1517109113 |
| [77] | Del Vecchio D, Dy AJ, Qian Y. Control theory meets synthetic biology. J R Soc Interface, 2016, 13(120): 20160380. DOI: 10.1098/rsif.2016.0380 |
| [78] | Bowsher CG, Swain PS. Environmental sensing, information transfer, and cellular decision-making. Curr Opin Biotechnol, 2014, 28: 149–155. DOI: 10.1016/j.copbio.2014.04.010 |
| [79] | Levchenko A, Nemenman I. Cellular noise and information transmission. Curr Opin Biotechnol, 2014, 28: 156–164. DOI: 10.1016/j.copbio.2014.05.002 |
| [80] | Brennan MD, Cheong R, Levchenko A. Systems biology. How information theory handles cell signaling and uncertainty. Science, 2012, 338(6105): 334–335. DOI: 10.1126/science.1227946 |
| [81] | Gregor T, Tank DW, Wieschaus EF, et al. Probing the limits to positional information. Cell, 2007, 130(1): 153–164. DOI: 10.1016/j.cell.2007.05.025 |
| [82] | Dubuis JO, Tkačik G, Wieschaus EF, et al. Positional information, in bits. Proc Natl Acad Sci USA, 2013, 110(41): 16301–16308. DOI: 10.1073/pnas.1315642110 |
| [83] | Cheong R, Rhee A, Wang CJ, et al. Information transduction capacity of noisy biochemical signaling networks. Science, 2011, 334(6054): 354–358. DOI: 10.1126/science.1204553 |
| [84] | Uda S, Saito TH, Kudo T, et al. Robustness and compensation of information transmission of signaling pathways. Science, 2013, 341(6145): 558–561. DOI: 10.1126/science.1234511 |
| [85] | Voliotis M, Perrett RM, McWilliams C, et al. Information transfer by leaky, heterogeneous, protein kinase signaling systems. Proc Natl Acad Sci USA, 2014, 111(3): E326–E333. DOI: 10.1073/pnas.1314446111 |
| [86] | Locke JCW, Elowitz MB. Using movies to analyse gene circuit dynamics in single cells. Nat Rev Microbiol, 2009, 7(5): 383–392. DOI: 10.1038/nrmicro2056 |
| [87] | Toettcher JE, Weiner OD, Lim WA. Using optogenetics to interrogate the dynamic control of signal transmission by the Ras/Erk module. Cell, 2013, 155(6): 1422–1434. DOI: 10.1016/j.cell.2013.11.004 |
| [88] | Cai L, Dalal CK, Elowitz MB. Frequency-modulated nuclear localization bursts coordinate gene regulation. Nature, 2008, 455(7212): 485–490. DOI: 10.1038/nature07292 |
| [89] | Purvis JE, Karhohs KW, Mock C, et al. p53 dynamics control cell fate. Science, 2012, 336(6087): 1440–1444. DOI: 10.1126/science.1218351 |
| [90] | Selimkhanov J, Taylor B, Yao J, et al. Accurate information transmission through dynamic biochemical signaling networks. Science, 2014, 346(6215): 1370–1373. DOI: 10.1126/science.1254933 |
| [91] | Tu BP, Kudlicki A, Rowicka M, et al. Logic of the yeast metabolic cycle: temporal compartmentalization of cellular processes. Science, 2005, 310(5751): 1152–1158. DOI: 10.1126/science.1120499 |
| [92] | Silverman SJ, Petti AA, Slavov N, et al. Metabolic cycling in single yeast cells from unsynchronized steady-state populations limited on glucose or phosphate. Proc Natl Acad Sci USA, 2010, 107(15): 6946–6951. DOI: 10.1073/pnas.1002422107 |
| [93] | Hung M, Chang E, Hussein R, et al. Modulating the frequency and bias of stochastic switching to control phenotypic variation. Nat Commun, 2014, 5: 4574. |
| [94] | Potvin-Trottier L, Lord ND, Vinnicombe G, et al. Synchronous long-term oscillations in a synthetic gene circuit. Nature, 2016, 538(7626): 514–517. DOI: 10.1038/nature19841 |
 2017, Vol. 33
2017, Vol. 33