中国科学院微生物研究所、中国微生物学会主办
文章信息
- 周文君, 郭日红, 邓明田, 王锋, 张艳丽
- Zhou Wenjun, Guo Rihong, Deng Mingtian, Wang Feng, Zhang Yanli
- RS-1提高CRISPR-Cas9系统介导的人乳铁蛋白基因敲入效率
- RS-1 enhanced the efficiency of CRISPR-Cas9 mediated knock-in of human lactoferrin
- 生物工程学报, 2017, 33(8): 1224-1234
- Chinese Journal of Biotechnology, 2017, 33(8): 1224-1234
- 10.13345/j.cjb.170105
-
文章历史
- Received: March 16, 2017
- Accepted: June 5, 2017
基因编辑技术现已成为生物学和医学研究领域中最热门的技术之一,其中的串联间隔重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)/相关蛋白9 (Cas9) 系统[1]被众多研究者用于基因组定点编辑等多种用途,被Science杂志评为2015年十大科学突破之首。其中,CRISPR-Cas9系统首先被发现于细菌和古细菌的Ⅱ型CRISPR/Cas系统中,其作为“获得性免疫系统”可以抵御外来病毒入侵[2],现被应用于真核生物细胞和个体的基因编辑[3-5]。由sgRNA引导的Cas9核酸内切酶可以在基因组中引入双链断裂(Double-stranded break,DSB),其可以通过非同源末端连接(Nonhomologous end joining,NHEJ)和同源修复(Homology-directed repair,HDR)两种途径修复[6-7]。利用CRISPR-Cas9系统和同源重组模板可以高效敲入外源基因,因此该系统被广泛应用在转基因动物细胞和个体的研究与生产中[8-10]。
羊乳,作为牛乳的一种替代品,有着较之前者更高的乳脂和蛋白含量。β-乳球蛋白(β-lactoglobulin,BLG)是羊乳中主要的乳清蛋白,也是羊乳中引起人体过敏反应的主要过敏原之一[11]。酶水解、加热并不能减少BLG蛋白过敏原性,反而会增加人体的过敏反应,发酵工艺由于会产生新的副产物,也无法有效减少其过敏原性[12-13]。人乳铁蛋白(Human lactoferrin, hLF)是一种多功能糖蛋白,具有促进铁吸收、抗菌、抗病毒、抗癌和抗氧化等多种生物学功能,是一种十分理想的食物添加剂和药物[14-16]。在山羊BLG基因座中敲入hLF基因既可以去除山羊乳中的过敏原,又能增加羊乳中的营养成分,使其更加“人源化”。然而目前利用CRISPR-Cas9技术敲除山羊BLG基因、同时定点整合hLF基因的相关研究鲜见报道。
研究表明,RAD5l是有丝分裂细胞中催化DNA同源重组修复过程中同源配对的唯一蛋白因子,其通过介导DNA链配对和链交换而参与了DNA同源重组修复[17]。RAD51蛋白激活剂(RAD51 stimulatory compound,RS-1) 可激活RAD51蛋白活性且使得RAD51蛋白与DNA的结合更加稳定[18],可让同源重组的效率提升至3倍,然而过高浓度的RS-1具有较强的细胞毒性[19]。目前关于RS-1在山羊体细胞中提高基因编辑效率的研究未见报道。
因此,本试验尝试利用CRIPSR-Cas9技术在山羊BLG位点精确敲入hLF,同时还探讨了不同浓度RS-1对CRISPR-Cas9介导山羊基因组敲入效率的影响,为今后高效利用CRIPSR-Cas9技术获得基因编辑动物提供了参考依据。
1 材料与方法 1.1 材料 1.1.1 质粒、菌种和细胞pX330质粒(Addgene,#42230) 由本实验室留存;pMD-19T克隆载体购自TaKaRa公司;pHA-hLF包括hLF cDNA序列;pBC-hLF包含NEO基因和EGFP基因,宿主菌为埃希氏大肠杆菌DH5α,60日龄雌性羔羊耳成纤维细胞,均由本实验室保存。
1.1.2 引物本试验中所使用到的引物通过NCBI的Primer Blast在线设计,引物由南京金斯瑞生物技术有限公司和南京擎科生物技术有限公司合成,引物序列见表 1。
| Primer name | Primer sequence (5′-3′) | Size (bp) | Purpose |
| BLG-5′HA-F | TCTTGTAGACGCCTCAGT | 1 564 | Amplification of the 5′homology arm |
| BLG-5′HA-R | GCTAGCCGGAATCCTACTTGGCTCATCC | ||
| BLG-3′HA-F | CTCGAGGCCTGGACATCCAGAAGGTT | 1 699 | Amplification of the 3′homology arm |
| BLG-3′HA-R | TCTAGAGCCGTTCTCCCTGAAAGACA | ||
| NEO-IRES-EGFP-F | CTCGAGTAGTTATTAATAGTAATCAA | 3 064 | Amplification of CMV-NEO-IRES2-EGFP |
| NEO-IRES-EGFP-R | CTCGAGTTGGACAAACCACAACTAGA | ||
| T7-BLG-F | ACCTGCCCTTGTCTAAGA | 622 | Identification of sgRNA editing efficiency |
| T7-BLG-R | CTTCCAGCCTCCAGAATG | ||
| 5KI-F | TCTGGATGGTATTCTCTG | 2 409 | Identification of the 5′junction |
| 5KI-R | CGGCATAATAGTGAGTTC | ||
| 3KI-F | GAAGAACGGCATCAAGGT | 2 371 | Identification of the 3′junction |
| 3KI-R | CAAGTGAAGCAGGTGTCA |
FBS (Gibco,26140079)、DMEM (Gibco,11965092)、Trypsin (Gibco,25200056)、G418 (Gibco,11811023) 和NeonTM 100 μL转染试剂盒购自Life Technology公司;各种限制性内切酶、各种分子量marker、高保真PrimerStar DNA聚合酶、LA Taq DNA聚合酶和pMD-19T载体购自宝生物工程(大连)有限公司;T4 DNA连接酶、热敏碱性磷酸酶,BbsⅠ限制性内切酶购自NEB公司;纯化试剂盒购自Qiagen公司;质粒提取试剂盒购自Omega公司;其他未说明试剂均为国产分析纯。
1.2 方法 1.2.1 sgRNA设计与pCas9-sgBLG表达载体构建 1.2.2 pBHA-hLF-NIE打靶载体的构建1 558 bp的5′同源臂和1 687 bp的3′同源臂分别扩增自野生型山羊基因组BLG基因(GenBank登录号Z33881.1) 505-2 062 bp和2 268-3 954 bp区域。在5′同源臂添加NheⅠ酶切位点,3′同源臂添加XhoⅠ和KpnⅠ酶切位点,分别将其连接至pMD-19T载体,得到pBHA-5′和pBHA-3′。然后使用KpnⅠ酶切pBHA-5′和pBHA-3′载体,回收线性化的pBHA-5′载体和3′同源臂,通过T4 DNA连接酶将3′同源臂连接至线性化的pBHA-5′载体中,构建成pBHA-5′-3′载体。CMV-NEO-IRES2-EGFP片段从pBC-hLF质粒上扩增后回收,两端添加XhoⅠ酶切位点。hLF cDNA片段从pHA-hLF质粒通过NheⅠ和XhoⅠ双酶切后回收得到。将3′BLG同源臂、hLF cDNA片段和NEO-IRES2-EGFP片段通过酶切连接依次连接至pBHA-5′-3′载体,最终构建成BLG基因的打靶载体pBHA-hLF-NIE。
1.2.3 CRISPR-Cas9编辑山羊BLG位点活性检测解冻第3代山羊耳成纤维细胞,培养液为DMEM+10% FBS+1%谷氨酰胺,待汇合度达到80%-90%的时候,进行转染。将pCas9-sgBLG表达载体与电转溶液和细胞混合至终浓度为1 μg/105细胞,然后在1.6 kV、10 ms、3 pulses的参数下进行电转染,将5×105细胞接种于6孔板中继续培养。转染72 h后,收集细胞,提取DNA,通过PCR扩增sgBLG靶标区域,产物回收后进行T7E1检测和连接T载体测序。T7E1检测采用400 ng PCR回收产物,退火后使用T7核酸内切酶Ⅰ处理0.5 h,产物通过2.5%的琼脂糖凝胶电泳和凝胶成像系统进行分析。引物分别为T7-BLG-F和T7-BLG-R。
1.2.4 RS-1对CRISPR-Cas9引导的基因敲入效率的影响为进一步检测RS-1对基因敲入效率的影响,将酶消化后悬浮的山羊耳成纤维细胞(1×105)与pCas9-sgBLG表达载体(0.5 μg)和pBHA-hLF-NIE打靶载体(0.5 μg)混合进行电转染,电转染方法参照1.2.3中的转染方法进行。转染后将细胞继续培养在分别含有0、5、10和20 μmol/L RS-1的培养液中72 h,一方面将利用R & D流式细胞仪对进行绿色荧光细胞计数,统计绿色荧光细胞比例;另一方面在RS-1处理后添加G418 (800 μg/mL)进行筛选,每3天更换1次培养液,筛选培养至16-20 d后,挑取EGFP阳性克隆,然后继续用含G418 (400 μg/mL)的完全培养基进行扩大培养。收集细胞,其中1/5继续培养后冻存,剩余的4/5提取DNA并用于PCR扩增打靶区域,所有PCR鉴定后的阳性产物回收后进行TA克隆并测序。PCR中所使用引物为5KI-F/R和3KI-F/R,5KI-F和3KI-R分别位于山羊BLG基因42-59 bp和4 140-4 157 bp区域,5KI-R和3KI-F分别包括部分hLF基因和EGFP基因(图 1)。

|
| 图 1 sgBLG靶标位点和打靶载体示意图 Figure 1 Scheme of sgRNA targeting BLG locus and targeting strategy. BLG: β-lactoglobulin; Ex 1: exon 1 of BLG; Ex 2: exon 2 of BLG; Ex 3: exon 3 of BLG; hLF: human lactoferrin cDNA; NEO: neomycin resistance gene; IRES: internal ribosome entry site; EGFP: enhanced green fluorescent protein; HA: homologous arm; PAM: protospacer adjacent motif. 5KI-F/R: primers used for 5′ junction PCR. 3KI-F/R: primers used for 3′ junction PCR. |
| |
将hLF cDNA片段和NEO-IRES2-EGFP片段连接至含有5′和3′同源臂的载体构建成pBHA-hLF-NIE打靶载体,其结构与打靶位置示意图见图 1。
2.2 Cas9在山羊耳成纤维细胞中活性检验将构建成功的pCas9-BLG电转染至山羊耳成纤维细胞,转染后72 h后收集细胞提取基因组DNA,然后使用引物T7-BLG-F/R扩增得到622 bp的sgBLG基因组靶标区域。PCR产物回收后分别进行T7E1检测和连接T载体测序。T7E1检测结果表明,sgBLG靶标区域存在插入和缺失等突变情况,测序结果显示,sgBLG靶位点存在1-59 bp的缺失,提示sgBLG可以有效引导Cas9蛋白编辑山羊耳成纤维细胞BLG位点(图 2)。进一步对T7E1的酶切结果进行灰度分析发现sgBLG引导的Cas9编辑活性为25%-31%。

|
| 图 2 在GEFs中检测sgBLG编辑活性 Figure 2 Testing of sgBLG targeting activity by transfection of pCas9-sgBLG into GEFs. (A) T7E1 assay of sgBLG targeting activity. M: DNA marker. WT: wild-type. (B) Sequencing result of sgBLG target region. The sequence in blue indicates the guide sequence of sgBLG, and the sequence in red indicates the PAM. |
| |
将pCas9-sgBLG表达载体和pBHA-hLF-NIE打靶载体共转染至山羊耳成纤维细胞,然后分别在细胞培养液中添加0、5、10和20 μmol/L的RS-1进行继续培养。为了减少非整合打靶载体表达的EGFP干扰,本研究将RS-1处理后的细胞传4代后利用流式细胞计数统计绿色荧光细胞比例。结果表明,绿色荧光细胞的比例随着RS-1浓度升高而增加,10 μmol/L和20 μmol/L RS-1处理组的绿色荧光阳性细胞比例显著高于对照组(P < 0.05,图 3C)。该结果表明,RS-1可以显著提高山羊耳成纤维细胞中的外源基因敲入效率,且与浓度呈正相关。

|
| 图 3 RS-1对基因敲入效率的影响 Figure 3 Effect of RS-1 on knock-in efficiency. The goat ear fibroblasts were transfected with pCas9-sgBLG plasmid and pBHA-hLF-NIE and observed under microscopy in white field (A) and with FITC filter (B). (C) Analysis of effect of RS-1 on knock-in efficiency by flow cytometry. GFP-positive rates indicated the knock-in efficiency. The asterisk means a significant difference (P < 0.05) between two groups. |
| |
对共转染后的山羊成纤维细胞分别进行0、5、10和20 μmol/L RS-1处理后,进一步添加G418筛选阳性细胞克隆,分别获得EGFP阳性克隆42、43、46和27个。对获得的阳性细胞进行基因敲入效率检测(图 4和表 2),结果显示,添加RS-1的处理组5′和3′端敲入阳性率均高于未添加的对照组,其中10 μmol/L浓度RS-1处理组的敲入效率最高,5′端连接阳性率从16.67%提升至36.69% (P < 0.05),3′端连接阳性率从11.90%提升至32.61% (P < 0.05)。然而当RS-1浓度达到20 μmol/L时,5′和3′端连接阳性率均出现下降,分别为25.93% (P > 0.05) 和22.22% (P > 0.05)。另外,如表 2所示,本试验中3′端连接PCR阳性克隆的5′端检测结果均为阳性,因此以3′端连接PCR阳性克隆率来表示hLF定点敲入效率。测序结果进一步表明,hLF基因已经精确整合至山羊BLG基因座(图 5)。此外,本研究中细胞经过20 μmol/L RS-1处理后,死亡细胞数目明显增加,衰老细胞克隆数目显著增多(P < 0.05)。

|
| 图 4 部分PCR筛选hLF敲入阳性细胞克隆结果 Figure 4 Partial results of PCR screening for hLF knock-in clones. (A) Partial PCR amplification results of hLF knock-in clones using primer 5KI-F/R. KI: knock-in. (B) Partial PCR amplification results of hLF knock-in clones using primers 3KI-F/R. KI: knock-in. |
| |
| Groups | Drug resistant clones | 5′junction PCR+(%) | 3′junction PCR+(%) | Senescent |
| 0 μmol/L | 42 | 7 (16.67%)a | 5 (11.90%)a | 1a |
| 5 μmol/L | 43 | 6 (14.29%)a | 6 (14.29%)a | 2a |
| 10 μmol/L | 46 | 17 (36.96%)b | 15 (32.61%)b | 1a |
| 20 μmol/L | 27 | 8 (29.63%)a | 6 (22.22%)a | 7b |
| Senescent: cells in the clone did not proliferate after 7 days. Data with different characters indicate a significant difference (P < 0.05) and with the same characters indicate no significant difference (P > 0.05). | ||||
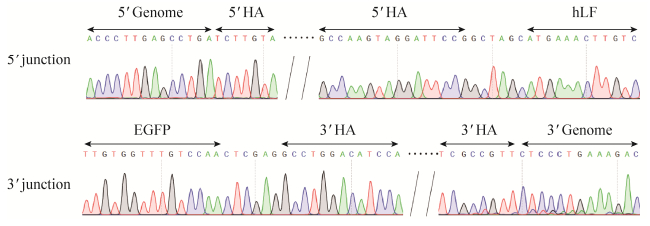
|
| 图 5 部分5′与3′连接PCR阳性细胞克隆测序峰图 Figure 5 Partial sequencing peak diagrams of 5′ and 3′ junction PCR positive clones. |
| |
本试验首次使用CRISPR-Cas9系统在山羊基因组BLG基因位点定点敲入hLF cDNA序列。同时本试验还研究了不同浓度RS-1对Cas9引导的敲入效率的影响。目前利用靶向性的核酸酶基因编辑技术可以快速、高效地获得基因编辑家畜。使用CRISPR-Cas9技术敲除BLG和同时插入hLF,既可以减少山羊乳的过敏原性,又保障了其营养价值,从而使其更加“人源化”[20]。在早期的研究中,通过显微注射和随机插入等方法获得转基因家畜的效率相对较低,存在敲入位点不够精确和拷贝数不确定等问题[21]。随后,研究者尝试使用长同源臂构建打靶载体,可以通过同源重组获得精确基因打靶细胞系或个体。2004年,孙丽新[22]在山羊乳腺β-酪蛋
白基因座定点插入hLF cDNA;Shen等[23]将人组织型纤溶酶原激活剂突变体(ht-PAm)基因插入至山羊胎儿成纤维细胞β-酪蛋白基因座;张学明[24]将胶质细胞源性神经营养因子(GDNF)基因插入至山羊胎儿成纤维细胞β-酪蛋白基因座;孟立[25]在山羊乳腺上皮细胞的β-酪蛋白基因座定点插入hLF cDNA,并且体外激素诱导获得了hLF蛋白。胡林勇[26]在山羊耳成纤维细胞BLG基因位点插入α-乳白蛋白(ALA),并结合体细胞核移植技术获得了转基因后代。然而,以上方法依然存在着试验周期长和整合效率低等问题。
近几年,随着基因编辑技术的飞速进步,ZFN、TALENs和CRISPR-Cas9已被广泛应用于转基因研究和转基因动物的生产。由于CRISPR-Cas9技术较之ZFN和TALENs的便捷性和高效性[27],现已经成为最热门的基因编辑技术之一,而且已经被应用于反刍动物的基因打靶研究中。Jeong等[28]使用CRISPR-Cas9技术在牛β-酪蛋白基因位点插入人胰岛素(hINS)基因。同时文献表明,Cas9较之TALENs有更高的编辑效率[29],而且张锋实验室改良后的Cas9蛋白具有更高的特异性[30]。本试验的阳性细胞克隆鉴定结果表明,未经RS-1处理的对照组,阳性率达到16.67%,显著高于传统的打靶方法[31]。Cui等[32]使用TALENs编辑山羊BLG位点,效率为5%-13%;宋绍征等使用TALENs和3种药物连续筛选的方法提升编辑效率至17%,与本研究结果相近[33]。本试验在体细胞中的实验结果表明,非筛选条件下基因敲入效率最高可达14%,药物筛选后基因敲入效率可进一步提升至32.61%,提示CRISPR-Cas9系统可能是一种优于传统同源重组的打靶方法,适用于家畜体细胞的基因打靶。
目前关于提高同源重组效率的研究表明,通过同步细胞周期,基因沉默和添加不同的化学物质可以提升同源重组效率[34-37]。添加化学物质法作为一种简单便捷的方法被众多研究者所采用,常见的化合物分为两类:一类为NHEJ途径中关键酶抑制剂,如SCR7[19, 34-35];另一种为HDR途径中关键酶的激活剂,如RS-1[19, 36]。研究报道,在转染后的细胞培养基中添加SCR7可以提升CRISPR-Cas9引导的敲入效率,在人和小鼠细胞细胞系上可以提升至4-5倍[34],在小鼠胚胎上可以提高17%-30%[35]。但同时也有文献表明,SCR7并不能显著提高胚胎中CRISPR-Cas9引导的同源重组效率[36]。本试验尝试在转染后的细胞培养基中添加不同浓度的RS-1,以探究其对同源重组效率的影响。流式细胞术分析结果表明,同源重组效率随着RS-1浓度的升高而增加,在20 μmol/L时效率最高,约14%。Pinder等[19]在不同细胞系中使用RS-1来提升CRISPR-Cas9引导的敲入效率,其结果表明,RS-1浓度在0-20 μmol/L时,同源重组效率与浓度正相关,与本结果一致。
在阳性细胞克隆鉴定结果中,RS-1浓度在0-10 μmol/L时,hLF敲入阳性克隆率与RS-1浓度呈正相关。然而,当RS-1浓度升高至20 μmol/L时,hLF敲入阳性克隆率下降至22.22%,与流式检测结果相反。这可能是由于20 μmol/L RS-1对于山羊耳成纤维细胞有着较强的细胞毒性,导致处理后的细胞在G418筛选时的存活率下降。同时本试验还观察到,20 μmol/L RS-1处理组的细胞在RS-1处理72 h后贴壁数明显少于对照组;此外20 μmol/L处理组获得的细胞克隆数最少,这一现象与Pinder等[19]发现的RS-1的浓度≥10 μmol/L时会出现较强的细胞毒性的现象一致。因此,适宜浓度的RS-1可以显著提升CRISPR-Cas9介导的山羊基因组打靶效率,但RS-1浓度过高时会降低打靶效率。
总之,本试验采用CRISPR-Cas9系统实现了在山羊BLG位点定点敲入hLF基因。同时,本试验还首次使用RS-1来促进山羊耳成纤维细胞的同源重组效率,发现其在10 μmol/L时获得的hLF敲入效率最高,但在20 μmol/L时具有较强细胞毒性,反而会降低hLF敲入效率。该研究为今后高效利用CRISPR-Cas9系统获得基因敲入的家畜提供了参考依据。
| [1] | Brouns SJJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science, 2008, 321(5891): 960–964. DOI: 10.1126/science.1159689 |
| [2] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816–821. DOI: 10.1126/science.1225829 |
| [3] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819–823. DOI: 10.1126/science.1231143 |
| [4] | Wang HY, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 2013, 153(4): 910–918. DOI: 10.1016/j.cell.2013.04.025 |
| [5] | Guo RH, Wan YJ, Xu D, et al. Generation and evaluation of Myostatin knock-out rabbits and goats using CRISPR/Cas9 system. Sci Rep, 2016, 6: 29855. DOI: 10.1038/srep29855 |
| [6] | Urnov FD, Rebar EJ, Holmes MC, et al. Genome editing with engineered zinc finger nucleases. Nat Rev Genet, 2010, 11(9): 636–646. DOI: 10.1038/nrg2842 |
| [7] | Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013, 31(9): 827–832. DOI: 10.1038/nbt.2647 |
| [8] | Wang XL, Yu HH, Lei AM, et al. Generation of gene-modified goats targeting MSTN and FGF5 via zygote injection of CRISPR/Cas9 system. Sci Rep, 2015, 5: 13878. DOI: 10.1038/srep13878 |
| [9] | Lv QY, Yuan L, Deng JC, et al. Efficient generation of myostatin gene mutated rabbit by CRISPR/Cas9. Sci Rep, 2016, 6: 25029. DOI: 10.1038/srep25029 |
| [10] | Platt RJ, Chen SD, Zhou Y, et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell, 2014, 159(2): 440–455. DOI: 10.1016/j.cell.2014.09.014 |
| [11] | Sharma S, Kumar P, Betzel C, et al. Structure and function of proteins involved in milk allergies. J Chromatogr B Biomed Sci Appl, 2001, 756(1/2): 183–187. |
| [12] | Ehn BM, Allmere T, Telemo E, et al. Modification of IgE binding to β-lactoglobulin by fermentation and proteolysis of cow's milk. J Agr Food Chem, 2005, 53(9): 3743–3748. DOI: 10.1021/jf048121w |
| [13] | Ehn BM, Ekstrand B, Bengtsson U, et al. Modification of IgE binding during heat processing of the cow's milk allergen β-lactoglobulin. J Agr Food Chem, 2004, 52(5): 1398–1403. DOI: 10.1021/jf0304371 |
| [14] | Lönnerdal B, Iyer S. Lactoferrin: molecular structure and biological function. Annu Rev Nutr, 1995, 15: 93–110. DOI: 10.1146/annurev.nu.15.070195.000521 |
| [15] | González-Chávez SA, Arévalo-Gallegos S, Rascón-Cruz Q. Lactoferrin: structure, function and applications. Int J Antimicrob Ag, 2009, 33(4): 301.e1–301.e8. |
| [16] | van der Strate BWA, Beljaars L, Molema G, et al. Antiviral activities of lactoferrin. Antivir Res, 2001, 52(3): 225–239. DOI: 10.1016/S0166-3542(01)00195-4 |
| [17] | Baumann P, West SC. Role of the human RAD51 protein in homologous recombination and double-stranded-break repair. Trends Biochem Sci, 1998, 23(7): 247–251. DOI: 10.1016/S0968-0004(98)01232-8 |
| [18] | Jayathilaka K, Sheridan SD, Bold TD, et al. A Chemical compound that stimulates the human homologous recombination protein RAD51. Proc Natl Acad Sci USA, 2008, 105(41): 15848–15853. DOI: 10.1073/pnas.0808046105 |
| [19] | Pinder J, Salsman J, Dellaire G. Nuclear domain 'knock-in' screen for the evaluation and identification of small molecule enhancers of CRISPR-based genome editing. Nucleic Acids Res, 2015, 43(19): 9379–9392. DOI: 10.1093/nar/gkv993 |
| [20] |
Sun BX, Hou WW, Ouyang HS. Transgenic animal bioreactor.
Animal Sci Vet Med, 2000, 17(3): 18–20.
(in Chinese). 孙博兴, 侯万文, 欧阳红生. 转基因动物生物反应器. 动物科学与动物医学, 2000, 17(3): 18-20. |
| [21] | Tan WF, Proudfoot C, Lillico SG, et al. Gene targeting, genome editing: from Dolly to editors. Transgenic Res, 2016, 25(3): 273–287. DOI: 10.1007/s11248-016-9932-x |
| [22] |
Sun LX. The production of gene-targeting at GEF β-casein gene locus with human lactoferrin gene and analysis of gene integrate site[D]. Shanghai: East China Normal University, 2004 (in Chinese). 孙丽新. hLF基因打靶山羊胎儿成纤维细胞株的建立及其整合位点的分析[D]. 上海: 华东师范大学, 2004. |
| [23] | Shen W, Lan GC, Yang XY, et al. Targeting the exogenous htPAm gene on goat somatic cell beta-casein locus for transgenic goat production. Mol Reprod Dev, 2007, 74(4): 428–434. DOI: 10.1002/(ISSN)1098-2795 |
| [24] |
Zhang XM. Human gdnf gene knock-in at beta-casein locus in bovine fetal fibroblast cells and production of gene-targeted blastocysts by SCNT[D]. Hohhot: Inner Mongolia University, 2009 (in Chinese). 张学明. 人gdnf在牛胎儿成纤维细胞β-casein基因座的定位整合及基因打靶克隆囊胚的制备[D]. 呼和浩特: 内蒙古大学, 2009. http://d.wanfangdata.com.cn/Thesis/Y1585476 |
| [25] |
Meng L. Studies on construction of specific gland expressional vector of hLF and modified fibroblast cell line[D]. Nanjing: Nanjing Agricultural University, 2010 (in Chinese). 孟立. 人乳铁蛋白表达载体的构建及转基因阳性细胞株的建立[D]. 南京: 南京农业大学, 2010. http://d.wanfangdata.com.cn/Thesis/Y1763079 |
| [26] |
Hu LY. Gene targeting toward β-lactoglobulin gene in goat[D]. Yangling: Northwest A & F University, 2012 (in Chinese). 胡林勇. 靶向山羊β-乳球蛋白基因的基因打靶研究[D]. 杨凌: 西北农林科技大学, 2012. |
| [27] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262–1278. DOI: 10.1016/j.cell.2014.05.010 |
| [28] | Jeong YH, Kim YJ, Kim EY, et al. Knock-in fibroblasts and transgenic blastocysts for expression of human FGF2 in the bovine β-casein gene locus using CRISPR/Cas9 nuclease-mediated homologous recombination. Zygote, 2015, 24(3): 442–456. |
| [29] |
Liu C. The study on efficiency of CRISPR/Cas9 and TALENs mediated gene targeting at dairy goat β-casein locus[D]. Hohhot: Inner Mongolia University, 2016 (in Chinese). 刘畅. CRISPR/Cas9与TALENs介导奶山羊β-酪蛋白位点基因打靶效率的比较研究[D]. 呼和浩特: 内蒙古大学, 2016. http://d.wanfangdata.com.cn/Thesis/Y3027840 |
| [30] | Slaymaker IM, Gao LY, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity. Science, 2016, 351(6268): 84–88. DOI: 10.1126/science.aad5227 |
| [31] |
Shen W, Yang ZT, Deng JX. Production of mammary gland bioreactor by gene targeting of somatic cells.
Chin J Biotech, 2003, 19(6): 767–770.
(in Chinese). 沈伟, 杨正田, 邓继先. 体细胞基因打靶制备动物乳腺生物反应器的策略与应用. 生物工程学报, 2003, 19(6): 767-770. |
| [32] | Cui CC, Song YJ, Liu J, et al. Gene targeting by TALEN-induced homologous recombination in goats directs production of β-lactoglobulin-free, high-human lactoferrin milk. Sci Rep, 2015, 5: 10482. DOI: 10.1038/srep10482 |
| [33] |
Song SZ, Zhu MM, Yuan YG, et al. BLG gene knockout and hLF gene knock-in at BLG locus in goat by TALENs.
Chin J Biotech, 2016, 32(3): 329–338.
(in Chinese). 宋绍征, 朱孟敏, 袁玉国, 等. 转录激活因子样效应物核酸酶介导的山羊β-乳球蛋白基因敲除和人乳铁蛋白基因定点整合. 生物工程学报, 2016, 32(3): 329-338. |
| [34] | Chu VT, Weber T, Wefers B, et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat Biotechnol, 2015, 33(5): 543–548. DOI: 10.1038/nbt.3198 |
| [35] | Maruyama T, Dougan SK, Truttmann MC, et al. Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining. Nat Biotechnol, 2015, 33(5): 538–542. DOI: 10.1038/nbt.3190 |
| [36] | Song J, Yang DS, Xu J, et al. RS-1 enhances CRISPR/Cas9-and TALEN-mediated knock-in efficiency. Nat Commun, 2016, 7: 10548. DOI: 10.1038/ncomms10548 |
 2017, Vol. 33
2017, Vol. 33




