中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王经纬, 雷喜梅, 覃盼, 赵鹏伟, 王斌, 王逸雯, 李懿婷, 金颢蕊, 李龙, 黄耀伟
- Wang Jingwei, Lei Ximei, Qin Pan, Zhao Pengwei, Wang Bin, Wang Yiwen, Li Yiting, Jin Haorui, Li Long, Huang Yao-Wei
- 猪丁型冠状病毒荧光定量RT-PCR与S1蛋白间接ELISA检测方法的建立及应用
- Development and application of real-time RT-PCR and S1 protein-based indirect ELISA for porcine deltacoronavirus
- 生物工程学报, 2017, 33(8): 1265-1275
- Chinese Journal of Biotechnology, 2017, 33(8): 1265-1275
- 10.13345/j.cjb.170119
-
文章历史
- Received: March 25, 2017
- Accepted: June 9, 2017
2 杭州贝尔塔生物技术有限公司,浙江 杭州 310004
2 Hangzhou Belta-Biotechnology Co., Ltd., Hangzhou 310004, Zhejiang, China
丁型冠状病毒(Deltacoronavirus)属冠状病毒科(Coronaviridae)冠状病毒亚科(Coronavirinae),是近年来新发现的一类冠状病毒[1],能够感染鸟类和哺乳动物。2009年,中国香港学者首次从猪的直肠拭子中分离到猪丁型冠状病毒(Porcine deltacoronavirus, PDCoV)。2014年,美国俄亥俄州出现PDCoV疫情。通过分析得到的PDCoV基因组序列发现美国俄亥俄州分离的毒株与中国香港发现的两个HKU15毒株序列相似度高达99%[2]。随后美国其他17个州相继报道了PDCoV的存在。2014年,韩国首次在本土发现了PDCoV毒株KOR/KNU14-04/2014并进行了全长测序[3]。2015年9月,Song等以及本实验室首次报道了我国内地发现的PDCoV/CHJXNI2/2015和CH/Sichuan/S27/2012毒株全长序列[4-5]。2015年10月,Chen等公布了CH/SXD1/2015的全长序列[6],Dong等也公布了3株中国毒株全长序列并进行了进化分析[7]。值得注意的是,Dong等分离到CHN-AH-2004毒株的样本采集于2004年,说明PDCoV在我国的存在可能早于2004年,限于之前技术条件或者认知局限而未能发现。此后,世界范围内不同国家和地区都有关于PDCoV的报道[8-12]。PDCoV能感染各年龄段的猪群,引起与猪流行性腹泻和猪传染性胃肠炎相似的腹泻、呕吐和脱水等症状[13-14]。PDCoV基因组结构与其他冠状病毒类似,全长约为25.4 kb,从5′-3′端依次编码复制酶ORF1ab、S糖蛋白(S)、小分子膜蛋白(E)、膜蛋白(M)以及核衣壳蛋白(N) [15-16]。
目前,PDCoV受到各国学者的广泛关注,研究多集中于新分离PDCoV毒株基因组序列及其基因功能研究,也有少数针对部分地区PDCoV流行病学检测的报道。Marthaler等先后尝试针对ORF1、S、M基因设计引物采用荧光定量RT-PCR (qRT-PCR)对美国不同州的近300份腹泻粪样和疫病猪场饲料及环境样品进行检测。结果显示,基于M基因建立的检测方法效果最好,利用该方法检测出的PDCoV阳性率高达30%[17]。Song等建立了针对PDCoV N基因的套式PCR检测方法,并对2012年11月到2015年3月从我国江西省采集的356份腹泻猪样本进行检测,发现PDCoV感染率达33.71%,与猪流行性腹泻病毒(PEDV)共感染率为19.66%[4]。Chen等对湖北、广东、山西等省6个猪场收集的64份腹泻猪粪便和小肠样品进行RT-PCR检测,检出样品PDCoV阳性率达23.4%[6]。血清学检测方面,Thachil与本文通讯作者等合作建立了以在哺乳动物细胞表达的截短S蛋白(S1) 为检测抗原的间接ELISA方法,对美国范围内的968份猪血清样本进行检测,PDCoV抗体检出率为8.7%[18]。Su等建立了以N蛋白为包被抗原的间接ELISA方法,并对黑龙江地区319份腹泻猪血清样品进行检测,其中37份样品PDCoV抗体阳性[19]。
本研究优化了一步法qRT-PCR检测方法及以PDCoV S1蛋白为包被抗原的间接ELISA方法,并对来自我国多个省份的病料进行检测,初步掌握了猪群中PDCoV感染和流行情况,为PDCoV的诊断和防控提供了重要参考。
1 材料与方法 1.1 材料和试剂小肠、粪便及血清样本由本实验室从江苏、湖南、河南、浙江、江西、河北、安徽、黑龙江、山东和上海10个省份收集。TRIzol、SuperScript® Ⅲ反转录试剂盒购自Invitrogen公司;质粒抽提试剂盒购自Axygen公司;Q5高保真酶购自NEB公司;核酸电泳凝胶回收试剂盒购自Thermo Fisher Scientific公司;T7 High Efficiency Transcription Kit、TransScript Probe One-Step qRT-PCR SuperMix试剂盒购自北京全式金公司;引物及探针由华大基因生物工程有限公司合成;羊抗猪IgG-Fc-HRP抗体购自Abcam公司;TMB单组分显色液购自北京索莱宝公司,脱脂奶粉为国产分析纯;猪流行性腹泻病毒ELISA试剂盒、猪传染性胃肠炎病毒ELISA试剂盒(内含PEDV和TGEV的阳性标准血清用作以下ELISA实验)购自上海繁旌生物科技有限公司。
1.2 qRT-PCR检测方法的建立 1.2.1 引物的设计及合成优化了Marthaler等针对M基因设计的qRT-PCR检测引物和探针,交由华大基因生物有限公司合成。所设计的特异性引物为:SDCV-M-F:5′-ATCGACCACATGCGCTCAA-3′,SDCV-M-R:5′-CAGCTCTTGCCCATGATCGT C-3′;探针为SDCV-M-PROBE:FAM-5′-CAC ACCAGTCGTTAAGCATGCGAGATT-3′-TAMRA。
1.2.2 标准曲线的构建设计一对引物:PDCoV-TEMPLE-T7-F:5′-TAATACGACTCACTATAGGGATGTCTGACG CAGAAGAGTG-3′;PDCoV-TEMPLE-T7-R:5′-CTACGCTGCTGATT CCTGC-3′ (下划线区为T7启动子序列),在正向引物的5′端引入T7启动子序列,用于扩增体外转录模板。从全长PDCoV cDNA扩增出含M基因的目的片段(1 988 bp)作为体外转录模板。采用全式金T7 High Efficiency Transcription Kit进行体外转录,体外转录结束后经DNaseⅠ作用15 min去除DNA模板后TRIzol法纯化体外转录产物并经过核酸凝胶电泳验证体外转录结果。采用体外转录的方法将模板体外转录成RNA作为阳性标准样品,测量浓度后分装保存。将验证正确的RNA模板进行10倍梯度稀释,采用全式金TransScript Probe One-Step qRT-PCR SuperMix试剂盒进行扩增,以DNA拷贝循环数为横坐标、以Ct值为纵坐标建立标准曲线。同时设置阴性对照。
1.2.3 临床样本检测对我国江苏、湖南和浙江等10个省份2014-2015年间收集的254份小肠或粪便样本进行研磨,重悬后离心,提取上清液核酸后,应用建立的qRT-PCR方法对其进行检测。
1.3 间接ELISA检测方法建立 1.3.1 PDCoV S1蛋白表达和纯化通过PCR扩增截短的S1基因(aa1-571),并在S1基因后加上His tag序列以便于后续纯化工作。将重组基因克隆到pFast-Bac载体上,经测序验证正确后转化至DH10Bac感受态细胞,蓝白斑筛选阳性克隆,并转染Sf9昆虫细胞获得重组的杆状病毒,继续感染Sf9细胞,得到高滴度P2代病毒(108 TCID50/mL),再感染Sf9细胞,72 h后收集细胞上清。用Ni-NTA层析柱纯化S1重组蛋白,利用SDS-PAGE和Western blotting检验纯化效果。
1.3.2 反应条件的优化采用棋盘滴定法,倍比稀释抗原和抗体,以确定最佳的抗原包被浓度、血清稀释度以及二抗稀释度。用碳酸盐包被缓冲液(pH 9.6) 2倍梯度稀释S1蛋白,然后包被不同稀释度的S1蛋白,4 ℃包被过夜,每孔用300 μL PBST洗涤缓冲液(0.01 mol/L PBS,0.05% Tween-20,pH 7.4) 洗涤3次后再用PBST配制的5%脱脂乳37 ℃封闭2 h。将PDCoV阳性猪血清按照1:100、1:200、1:400、1:800、1:1 600稀释后每孔加入100 μL,每组6个重复,并在37 ℃下孵育1 h。PBST洗涤缓冲液洗涤3次。将按1:1 000、1:2 000、1:4 000、1:5 000、1:8 000、1:10 000等6个梯度稀释的HRP标记的山羊抗猪IgG以100 μL/孔的量分别加入上述孔中,37 ℃下孵育30 min。PBST洗涤缓冲液洗涤3次。向每个孔加入100 μL TMB单组分显色液,并于室温孵育15 min。通过加入50 μL/孔的2 mol/L硫酸终止反应,OD450读数。上述实验重复3次以确定最佳反应条件。研究中阳性血清样本由本实验室之前利用Western blotting方法[20]筛选确定,阴性血清是本实验室根据参考文献[18]中已经确定的PDCoV阴性血清作为标准对照,采用上述间接ELISA方法筛选得到。
1.3.3 临床样品的检测应用建立的ELISA检测方法,对收集自江苏、湖南、江西、河南、浙江、黑龙江和山东等地的609份表现腹泻症状的猪血清样品进行检测,用本实验室保存的已确定PDCoV阳性和阴性血清作为对照。
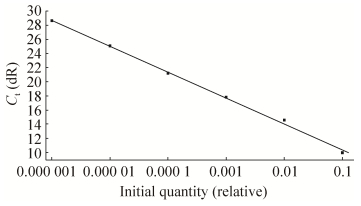
2 结果与分析 2.1 qRT-PCR检测方法的建立体外转录得到的RNA标准样品浓度为40.72 ng/μL,并进行10倍梯度稀释,选取6个稀释梯度,进行qRT-PCR。扩增曲线(图 1)显示,不同稀释度的扩增曲线之间间距均匀。相关系数R2=0.998表明标准曲线具有优良的线性关系;扩增效率为87.3% (图 2)。继续将RNA标准样品进行10倍梯度稀释,最后获得最低检测浓度为4.07×10-8ng/μL,即最低检测拷贝数为1.12×101GE (Genome Equivalent)/μL。阴性对照组和PEDV、TGEV阳性样品均未获得阳性信号(文中未显示),说明该qRT-PCR检测方法特异性好。同时进行了重复性试验,统计结果表明标准阳性样品的组间变异系数为0.20%-0.70%,组间变异系数为0.30%-0.93%。将qRT-PCR阳性产物进行测序,结果显示为预期扩增的PDCoV M基因特异性目的片段(72 bp),说明所建立的qRT-PCR检测方法可靠、准确。

|
| 图 1 标准品的扩增曲线 Figure 1 The kinetic curve of the standard samples. |
| |
|
| 图 2 标准品的标准曲线 Figure 2 The standard curve of the standard samples |
| |
对来自河南、湖南、浙江、江西、安徽、河北、黑龙江、江苏、山东和上海10个省份总共254份小肠或粪便样本进行检测,结果如表 1所示,共检出了11份PDCoV阳性样本(其中粪便样本7份,小肠样本4份),总阳性率为4.33%。从样本来源来看,如表 2所示,检测结果呈阳性的样品主要来自湖南和江苏,其中湖南4份和江苏7份,且全部为保育猪样品。阳性样本Ct值范围在22±5之间,病毒拷贝数范围在2.81×103-1.48×106GE/μL之间。同时对这11份阳性样本进行PEDV检测,结果如表 3所示,发现共有8份样本PEDV阳性,阳性率高达72.73%。
| Samples | Number of samples | Positive samples | Positive rate (%) |
| Fecal | 34 | 7 | 20.59 |
| Small intestine | 220 | 4 | 1.82 |
| Area | Number of samples | Positive samples | Positive rate (%) |
| Henan | 57 | 0 | 0.00 |
| Hunan | 6 | 4 | 66.67 |
| Zhejiang | 109 | 0 | 0.00 |
| Jiangxi | 18 | 0 | 0.00 |
| Anhui | 6 | 0 | 0.00 |
| Hebei | 3 | 0 | 0.00 |
| Heilongjiang | 7 | 0 | 0.00 |
| Jiangsu | 36 | 7 | 19.44 |
| Shandong | 11 | 0 | 0.00 |
| Shanghai | 1 | 0 | 0.00 |
| Total | 254 | 11 | 4.44 |
| Numbering | Area | Age | Type | Results of PEDV | Ct value | Copies (GE/μL) |
| JS-1 | Jiangsu | Nursery | Fecal | Positive | 23.27 | 8.34×103 |
| JS-2 | Jiangsu | Nursery | Fecal | Positive | 22.38 | 1.46×104 |
| JS-3 | Jiangsu | Nursery | Fecal | Positive | 22.85 | 1.08×104 |
| JS-4 | Jiangsu | Nursery | Fecal | Positive | 20.80 | 3.92×104 |
| JS-6 | Jiangsu | Nursery | Fecal | Positive | 22.33 | 1.50×104 |
| JS-7 | Jiangsu | Nursery | Fecal | Positive | 17.08 | 4.06×105 |
| JS-8 | Jiangsu | Nursery | Fecal | Positive | 26.26 | 1.28×103 |
| 1021-1 | Hunan | Nursery | Small intestine | Positive | 22.19 | 1.64×104 |
| 1021-4 | Hunan | Nursery | Small intestine | Negative | 21.50 | 2.53×104 |
| 1021-5 | Hunan | Nursery | Small intestine | Negative | 17.23 | 3.70×105 |
| 1021-6 | Hunan | Nursery | Small intestine | Negative | 20.10 | 6.09×104 |
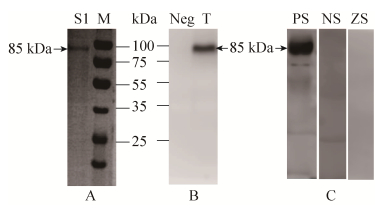
将测序正确的阳性重组质粒转染Sf9细胞,获得重组的杆状病毒,用高滴度的P2病毒再感染Sf9细胞,培养72 h后,对细胞上清进行Ni+柱亲和层析纯化,得到较纯的PDCoV S1蛋白;图 3B是用抗His标签的抗体进行Western blotting分析,可以检测到单一的、分子量与蛋白电泳一致的条带;图 3C是用PDCoV阳性血清和阴性血清作为一抗进行Western blotting分析,阳性血清对照组同样可以检测到相应的条带,而阴性血清对照组则无条带(有部分非特异性反应),ZS是含空载体的重组杆状病毒表达的蛋白作为样品,PDCoV阳性血清作为一抗进行Western blotting分析,无条带显示。利用BCA蛋白试剂盒测得蛋白浓度为0.41 mg/mL。
|
| 图 3 重组S1蛋白纯化结果与Western blotting检测结果 Figure 3 Purification and identification of the recombinant S1 protein expressed in insect cells. (A) SDS-PAGE result of the purified recombinant S1 protein. (B) Detection of purified recombinant S1 using anti-His antibody by Western blotting. (C) Detection of purified recombinant S1 using PDCoV positive serum by Western blotting. M: protein marker (25-100 kDa); S1: purified recombinant S1 protein; Neg: the negative control (normal cell lysates); T: supernatants of infected-cell lysates; PS: the positive control (PDCoV positive serum); NS: the negative control (PDCoV negative serum); ZS: the negative control (The Bacmid without S1 sequences). |
| |
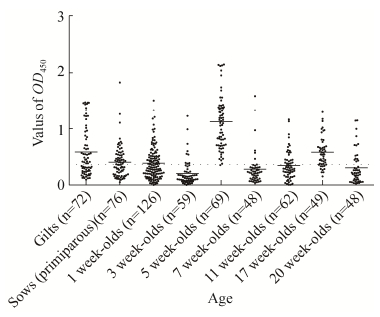
通过棋盘滴定测定抗原的最佳包被量为1.25 ng/孔,通过使用1:100稀释的血清样品和1:5 000稀释的HRP标记的山羊抗猪IgG能获得最佳ELISA结果。应用上述确定的最佳条件检测72份PDCoV抗体阴性血清样品。经生物学统计分析x为0.133,s为0.078,则临界值为x+3s=0.367,即OD450≥0.367判定为阳性。同时,确定可疑区间x+s=0.211,即当OD450值在0.211和0.367之间为疑似阳性,需重测;当重测值仍大于0.211时判为阳性,否则为阴性。本实验室合作者Thachil已证实,PDCoV S1蛋白与PEDV、TGEV等病毒无交叉反应性;同时本实验室利用购买检测猪流行性腹泻病毒和猪传染性胃肠炎病毒的ELISA试剂盒内含的PEDV和TGEV阳性标准血清作为检测一抗检测S1蛋白的交叉反应性,结果OD450均小于0.1,说明S1蛋白与PEDV和TGEV无交叉反应性。对所建立的间接ELISA实验进行敏感性实验,当血清稀释至3 200倍时,显色后根据酶标仪读数OD450=0.371,判定仍为阳性,证明所建立的间接ELISA方法具有较好的敏感性。进行重复性实验统计结果分析发现,批内变异系数为1.46%-4.91%,批间变异系数为2.01%-5.80%,批内和批间重复性试验的变异系数均小于10%,表明本研究建立的间接ELISA检测方法具有较好的重复性。分别比对用昆虫细胞或哺乳动物细胞表达的S1蛋白作为包被抗原一对一随机检测53份临床血清样品,统计结果显示相关性较高(R2=0.9415),说明本研究建立的间接ELISA方法与前面建立的标准方法精准一致(结果未显示),可靠性较高。本实验室利用建立的间接ELISA方法对全国7个省份收集的609份不同日龄的表现腹泻症状的猪血清样品进行检测,结果如表 4所示,共检出269份PDCoV阳性血清,阳性率为44.17%。如图 4所示,经产母猪血清中PDCoV抗体水平高于后备母猪;仔猪出生后1-3周龄,血清PDCoV抗体水平逐渐降低,3周龄猪血清样品OD450平均值为0.206±0.229达到最低,这可能与仔猪体内母源抗体衰减有关。到5周龄时,猪血清样品OD450平均值为1.129±0.520达到最高,表明出生后仔猪感染PDCoV产生体液免疫应答,PDCoV抗体水平达到峰值,随后回复均值,趋于稳定。
| Samples | Number of positive samples | Number of samples | Positive rate (%) | Average value of OD450 |
| Gilts | 38 | 72 | 52.78 | 0.584±0.432 |
| Sows | 35 | 76 | 46.05 | 0.406±0.287 |
| 1 week-olds | 50 | 126 | 39.68 | 0.358±0.290 |
| 3 week-olds | 6 | 59 | 10.17 | 0.206±0.229 |
| 5 week-olds | 67 | 69 | 97.10 | 1.129±0.520 |
| 7 week-olds | 6 | 48 | 12.50 | 0.282±0.301 |
| 11 week-olds | 18 | 62 | 29.03 | 0.344±0.253 |
| 17 week-olds | 35 | 49 | 71.43 | 0.581±0.268 |
| 20 week-olds | 14 | 48 | 29.17 | 0.308±0.301 |
| Total | 269 | 609 | 44.17 | 0.475±0.421 |
|
| 图 4 不同年龄猪血清抗PDCoV抗体的分布情况 Figure 4 Distribution of serum anti-PDCoV antibodies obtained from pigs of different ages. The purified PDCoV S1 protein was used as the coating antigen and the indirect ELISA method was used to detect the OD450 of 609 specific antibodies in porcine sera of different ages, the dotted line represents the cut-off value of ELISA. The graph was generated by GraphPad software. The solid line is the average of the OD450 values for pigs of the same age. |
| |
仔猪腹泻一直是困扰我国乃至世界养猪业的一大难题,对于引起仔猪腹泻病原体的研究一直是一大热点。之前的研究认为,引起猪腹泻主要的致病原是PEDV、TGEV以及其他细菌性病原体。但是很多已经进行上述病原疫苗免疫的猪场依然发生大规模仔猪腹泻。因此,很多专家学者认为,还有其他的未知病原单独感染或与上述病原共感染导致仔猪腹泻,PDCoV的发现也印证了这个猜测。研究发现,PDCoV引起的仔猪临床症状与PEDV、TGEV等病原相似,并且PDCoV常常与PEDV共感染,因此人们常忽视PDCoV的存在及危害。而目前已有的流行病学调查结果显示,PDCoV的流行范围比较广,对养殖行业的威胁不容忽视,而目前尚未有成熟的检测方法用于检测样本中的病毒和抗体,因此从核酸水平以及抗体水平建立高效、灵敏的检测方法对于监视和预防该病的传播和流行有着十分重要的意义。
本研究中优化的一步法qRT-PCR检测方法具有高灵敏、高特异性等特点,并且实验步骤简洁,从而减少了实验操作污染的可能性[21-22]。本研究选用PEDV、TGEV阳性样品进行交叉检测,未发现起峰,说明本检测方法特异性较好。本实验室之前已建立了普通RT-PCR检测方法,相对于本研究建立的qRT-PCR方法,检测结果大致相符,但该方法检测灵敏度很低,难以检出微量病毒的存在,同时操作步骤繁琐,耗时长,加大了样本污染的可能性。
相较Chen等[6] 23.4%和Song等[4] 33.71%的粪拭子或肠道样品PDCoV阳性检出率,本实验室的PDCoV检出率约为4.44%,多见于同一猪场,且检出该病毒的城市大多集中在南方地区(江苏和四川),这可能与该病毒的生存条件和传播途径有关。此外,统计样本量不够大,样品采集的猪场养殖管理和卫生条件状况不同等因素也影响了检出率。从检出的阳性病料中,本实验室成功克隆了1株PDCoV的全长基因组[5]。另一个值得注意的是,本次检出PDCoV阳性的样本中,高达72.73%的样本PEDV检测同样阳性,说明这些猪只同时感染了PEDV和PDCoV,即PDCoV与其他病毒共感染现象比较普遍。
目前仅有少数关于猪血清中PDCoV抗体的血清学检测方法的报道。Su等利用原核表达系统表达PDCoV N蛋白作为包被蛋白检测血清中抗体水平[19];本实验室建立的间接ELISA检测方法是以PDCoV S蛋白的截短部分S1作为包被抗原。S蛋白位于病毒粒子表面[23],介导病毒侵入宿主细胞,含有主要的中和性抗原表位[24],能诱导宿主产生大量针对S蛋白抗原表位的抗体,同时PDCoV S蛋白具有比较高的序列保守性和相较其他冠状病毒结构蛋白的特异性[25],因此本实验室建立的间接ELISA检测方法能够更灵敏更特异性地检测猪血清中的PDCoV抗体水平。Ma等则建立了以PDCoV全病毒为包被抗原的间接ELISA[26],全病毒的纯度直接影响了检测结果的可靠性,而PDCoV病毒粒子纯化的操作要求和费用均较高,该检测方法的敏感性和特异性并没有得到证实。
本实验室合作者Thachil等首次建立了基于截短S1蛋白的间接ELISA检测方法,但其S1蛋白是通过哺乳动物细胞表达,相对于杆状病毒表达系统,其表达步骤复杂、成本高。昆虫-杆状病毒表达系统能高水平分泌表达外源基因,表达的重组蛋白具有较高的生物学活性,且易分离纯化。本实验室参照Thachil等报道的S1蛋白序列截取方案,S1基因重组质粒转染Sf9细胞,获得高滴度的重组杆状病毒,再感染Sf9细胞,对目的蛋白进行纯化,分别用抗His标签的抗体和PDCoV阳性血清2种检测抗体对其进行Western blotting,可以检测到单一的、分子量与蛋白电泳一致的条带,而PDCoV阴性血清对照组则无条带。使用空载体的重组杆状病毒表达的蛋白作为阴性对照,PDCoV阳性血清作为一抗进行Western blotting分析,无条带显示,说明本研究表达的PDCoV S1蛋白是正确无误的,可用于后续研究。
本实验室检测的609份样品来自于我国7个省份,包含不同日龄的猪,覆盖面较广。其中269份血清样品血清学检测呈阳性,阳性率为44.17%。Thachil等报道968份美国血清样本检出PDCoV抗体阳性率为8.7%[18]。Su等对黑龙江省收集的319份血清样品进行检测,阳性率为11.59%[19]。这可能与采样的区域有关,南方区域相较北方的外界环境更有利于病毒的传播。
当前,国内对PDCoV感染后刺激宿主抗体特异性免疫应答所产生的抗体水平监测的报道相对较少。Hu等报道了感染PDCoV的猪血清中PDCoV特异性抗体产生的时间及趋势[27],该研究使用细胞传代的PDCoV人工感染仔猪,不能准确反映临床感染PDCoV的猪只特异性抗体消长规律。本实验室采用建立的间接ELISA检测方法检测全国各省猪场的临床血清样品。仔猪出生后,随着日龄增长,仔猪血清的抗体水平呈现逐渐降低的趋势,到3周龄时,抗体水平消减到最低并趋于稳定。一般来说,母源IgG抗体能够通过胎盘使初生仔猪获得较高的抗PDCoV的母源抗体,随后母源抗体水平逐渐降低。此后,若仔猪再次感染PDCoV,其机体免疫系统能产生特异性免疫应答,从而产生大量PDCoV抗体。本研究检测结果显示,5周龄猪只抗体水平迅速上升,超过平均抗体水平,达到最高。这可能由于猪只在此前已感染PDCoV,从而刺激机体产生大量特异性抗体。随后抗体水平再次逐渐降低稳定在平均水平伴随终生。各个阶段样品阳性率以及平均OD450值显示,PDCoV在各地猪场广泛流行。本试验结果反映了养殖场猪群PDCoV抗体情况和动态变化,但是更准确的相关性关系还需要通过检测更长时间段内更多猪场的样本才能确定。
本研究优化了核酸水平的PDCoV检测方法,建立了PDCoV血清学的检测方法,并对国内多个省份的猪场PDCoV流行情况进行了初步调查分析,为PDCoV的监测和防控打下基础。
| [1] | Woo PC, Lau SK, Lam CS, et al. Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavirus. J Virol, 2012, 86(7): 3995–4008. DOI: 10.1128/JVI.06540-11 |
| [2] | Wang L, Byrum B, Zhang Y. Detection and genetic characterization of deltacoronavirus in pigs, Ohio, USA, 2014. Emerg Infect Dis, 2014, 20(7): 1227–1230. |
| [3] | Lee S, Lee C. Complete genome characterization of Korean porcine deltacoronavirus strain KOR/KNU14-04/2014. Genome Announc, 2014, 2(6): e01191–14. |
| [4] | Song D, Zhou X, Peng Q, et al. Newly emerged porcine deltacoronavirus associated with diarrhoea in swine in China: identification, prevalence and full-length genome sequence analysis. Transbound Emerg Dis, 2015, 62(6): 575–580. DOI: 10.1111/tbed.2015.62.issue-6 |
| [5] | Wang YW, Yue H, Fang WH, et al. Complete genome sequence of porcine deltacoronavirus strain CH/Sichuan/S27/2012 from mainland China. Genome Announc, 2015, 3(5): e00945–15. |
| [6] | Chen FZ, Zhu YX, Wu MZ, et al. Full-Length genome characterization of Chinese porcine deltacoronavirus strain CH/SXD1/2015. Genome Announc, 2015, 3(5): e01284–15. |
| [7] | Dong BQ, Liu W, Fan XH, et al. Detection of a novel and highly divergent coronavirus from Asian leopard cats and Chinese ferret badgers in Southern China. J Virol, 2007, 81(13): 6920–6926. DOI: 10.1128/JVI.00299-07 |
| [8] | Curry SM, Gibson KA, Burrough ER, et al. Nursery pig growth performance and tissue accretion modulation due to porcine epidemic diarrhea virus or porcine deltacoronavirus challenge. J Anim Sci, 2017, 95(1): 173–181. DOI: 10.2527/jas.2016.1000 |
| [9] | Fang PX, Fang LR, Dong N, et al. Research advances in the porcine deltacoronavirus. Chin J Virol, 2016, 32(2): 243–248. |
| [10] | Janetanakit T, Lumyai M, Bunpapong N, et al. Porcine deltacoronavirus, Thailand, 2015. Emerg Infect Dis, 2016, 22(4): 757–759. DOI: 10.3201/eid2204.151852 |
| [11] | Lee JH, Chung HC, Nguyen VG, et al. Detection and phylogenetic analysis of porcine deltacoronavirus in Korean swine farms, 2015. Transbound Emerg Dis, 2016, 63(3): 248–252. DOI: 10.1111/tbed.2016.63.issue-3 |
| [12] | Lorsirigool A, Saeng-Chuto K, Temeeyasen G, et al. The first detection and full-length genome sequence of porcine deltacoronavirus isolated in Lao PDR. Arch Virol, 2016, 161(10): 2909–2911. DOI: 10.1007/s00705-016-2983-8 |
| [13] | Hu H, Jung K, Vlasova AN, et al. Isolation and characterization of porcine deltacoronavirus from pigs with diarrhea in the United States. J Clin Microbiol, 2015, 53(5): 1537–1548. DOI: 10.1128/JCM.00031-15 |
| [14] | Ma Y, Zhang Y, Liang X, et al. Origin, evolution, and virulence of porcine deltacoronaviruses in the United States. MBio, 2015, 6(2): e00064. |
| [15] | Brian DA, Baric RS. Coronavirus genome structure and replication. Curr Top Microbiol Immunol, 2005, 287: 1–30. |
| [16] |
Wang YW, Huang YW. A newly discovered coronavirus genus-deltacoronavirus.
Chem Life, 2016, 36(4): 514–518.
(in Chinese). 王逸雯, 黄耀伟. 新发现的冠状病毒属——丁型冠状病毒. 生命的化学, 2016, 36(4): 514-518. |
| [17] | Marthaler D, Raymond L, Jiang Y, et al. Rapid detection, complete genome sequencing, and phylogenetic analysis of porcine deltacoronavirus. Emerg Infect Dis, 2014, 20(8): 1347–1350. |
| [18] | Thachil A, Gerber PF, Xiao CT, et al. Development and application of an ELISA for the detection of porcine deltacoronavirus IgG antibodies. PLoS ONE, 2015, 10(4): e0124363. DOI: 10.1371/journal.pone.0124363 |
| [19] | Su MJ, Li CQ, Guo DH, et al. A recombinant nucleocapsid protein-based indirect enzyme-linked immunosorbent assay to detect antibodies against porcine deltacoronavirus. J Vet Med Sci, 2016, 78(4): 601–606. DOI: 10.1292/jvms.15-0533 |
| [20] | Huang YW, Harrall KK, Dryman BA, et al. Serological profile of torque teno sus virus species 1 (TTSuV1) in pigs and antigenic relationships between two TTSuV1 genotypes (1a and 1b), between two species (TTSuV1 and-2), and between porcine and human anelloviruses. J Virol, 2012, 86(19): 10628–10639. DOI: 10.1128/JVI.00176-12 |
| [21] | Narayanan K, Huang C, Lokugamage K, et al. Severe acute respiratory syndrome coronavirus nsp1 suppresses host gene expression, including that of type Ⅰ interferon, in infected cells. J Virol, 2008, 82(9): 4471–4479. DOI: 10.1128/JVI.02472-07 |
| [22] | Neuman BW, Joseph JS, Saikatendu KS, et al. Proteomics analysis unravels the functional repertoire of coronavirus nonstructural protein 3. J Virol, 2008, 82(11): 5279–5294. DOI: 10.1128/JVI.02631-07 |
| [23] | Heald-Sargent T, Gallagher T. Ready, set, fuse! The coronavirus spike protein and acquisition of fusion competence. Viruses, 2012, 4(4): 557–580. |
| [24] | Yang JH, Lü J, Wang YY, et al. Replication of murine coronavirus requires multiple cysteines in the endodomain of spike protein. Virology, 2012, 427(2): 98–106. DOI: 10.1016/j.virol.2012.02.015 |
| [25] | Sun DB, Feng L, Shi HY, et al. Spike protein region (aa 636789) of porcine epidemic diarrhea virus is essential for induction of neutralizing antibodies. Acta Virol, 2007, 51(3): 149–156. |
| [26] | Ma YM, Zhang Y, Liang XY, et al. Two-way antigenic cross-reactivity between porcine epidemic diarrhea virus and porcine deltacoronavirus. Vet Microbiol, 2016, 186: 90–96. DOI: 10.1016/j.vetmic.2016.02.004 |
| [27] | Hu H, Jung K, Vlasova AN, et al. Experimental infection of gnotobiotic pigs with the cell-culture-adapted porcine deltacoronavirus strain OH-FD22. Arch Virol, 2016, 161(12): 3421–3434. DOI: 10.1007/s00705-016-3056-8 |
 2017, Vol. 33
2017, Vol. 33




