中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王晓雪, 李红杰, 李永涛, 高冬生, 陈陆, 常洪涛, 刘红英, 王川庆, 赵军
- Wang Xiaoxue, Li Hongjie, Li Yongtao, Gao Dongsheng, Chen Lu, Chang Hongtao, Liu Hongying, Wang Chuanqing, Zhao Jun
- 猪流行性腹泻病毒nsp1对Ⅰ型干扰素应答的影响
- Effect of porcine epidemic diarrhea virus nsp1 on type Ⅰ interferon response
- 生物工程学报, 2017, 33(8): 1325-1334
- Chinese Journal of Biotechnology, 2017, 33(8): 1325-1334
- 10.13345/j.cjb.170068
-
文章历史
- Received: February 24, 2017
- Accepted: May 31, 2017
猪流行性腹泻(Porcine epidemic diarrhea, PED)是由猪流行性腹泻病毒(Porcine epidemic diarrhea virus, PEDV)引起的一种急性、高度传染性的肠道疾病,以感染猪呕吐、脱水和水样腹泻以及仔猪的高死亡率为主要特征[1]。PED在中国的流行于1984年得到证实,随后由于灭活苗的广泛使用使疫情得到有效控制[2-4]。2010年下半年以来,PEDV再次在中国大规模暴发,此次流行对2周龄以内的哺乳仔猪影响较大,死亡率可高达100%,感染仔猪发病急,腹泻3-4 d后严重脱水死亡[5-6]。2013年美国历史上也首次暴发PED,并很快蔓延至其他的北美国家[7-8],PED的流行已经对世界各国的养猪业造成了巨大损失。
PEDV属于冠状病毒科(Coronaviridae) α冠状病毒属(α-CoV)的成员,为单股正链RNA病毒,5′端有一个帽子结构,3′端有一个poly(A)尾。病毒基因组全长28 kb,包含ORF1a、ORF1b、S、ORF3、E、M和N等7个开放阅读框。ORF1a编码复制酶多聚蛋白(Polyprotein) PPla,ORF1b编码的是一个融合蛋白PPla/b,融合蛋白包含16个非结构蛋白(Nsps),从nsp1到nsp16,这些非结构蛋白经病毒蛋白酶酶切后,参与病毒RNA的复制和转录[9-11]。
α-CoV的nsp1编码约110个氨基酸,为PPla蛋白N端的剪切产物,是宿主细胞细胞质中的第一个成熟的病毒蛋白。在冠状病毒的4个种属中,α-CoV和β-CoV编码有nsp1,而γ-CoV和δ-CoV仅编码除nsp1外的15个非结构蛋白(nsp2-nsp16) [12],并且α-CoV和β-CoV两个种属的nsp1也没有非常相似的序列同源性[13],所以nsp1可以被认为是种属特异性基因[12]。有文献报道,鼠肝炎病毒的nsp1截短后,在细胞上的增殖与野生株类似,但是在小鼠体内的增殖明显受到了抑制[14]。另外,将猪传染性胃肠炎病毒的nsp1与nsp2切割位置的碱基突变阻止了nsp1的释放后,病毒的生存能力显著降低[15]。这些结果均提示nsp1可能是病毒的毒力因子之一。近几年,nsp1对宿主基因表达和在病毒逃避宿主天然免疫应答当中的调控作用备受关注。有研究显示,与PEDV nsp1有较高氨基酸序列同源性的人冠状病毒NL63和229E的nsp1,能显著抑制IFN-β、干扰素刺激基因15 (ISG15) 和携带有干扰素刺激反应元件(ISRE)的报告基因的表达[14, 16]。另外,SARS冠状病毒(SARS-CoV)的nsp1能够抑制多种宿主基因mRNA的表达,包括参与天然免疫中的Ⅰ型干扰素的表达[17]。
为了研究PEDV nsp1对宿主天然免疫应答的调控功能,本研究克隆和表达了PEDV nsp1基因,利用Western blotting、间接免疫荧光试验、双荧光素酶报告基因法及猪水泡性口炎病毒(Vesicular stomatitis virus, VSV)复制抑制试验研究nsp1对Ⅰ型IFN应答的影响。
1 材料与方法 1.1 病毒、质粒、菌株、细胞和主要试剂PEDV流行株CH/ZMDZY/11(GenBank登录号:KC196276)、带有绿色荧光标记的猪水泡性口炎病毒(VSV-GFP)、pET-28a(+)原核表达载体、带有HA标签的真核表达载体pCAGGS、含有荧光素酶基因的IFN-β启动子报告质粒(IFN-β-luc),以及含有海肾荧光素酶的pRL-TK内参报告质粒、HEK-293T、A549和Vero细胞,均由本实验室制备和保存。DH5α和BL21(DE3) 菌株感受态细胞购自生工生物工程(上海)股份有限公司。人IFN-β ELISA检测试剂盒购自上海瓦兰生物科技有限公司。
限制性内切酶NcoⅠ、EcoRⅠ和XhoⅠ购自宝生物工程(大连)有限公司。HRP-羊抗鼠IgG、HRP-羊抗兔IgG购自武汉三鹰生物技术有限公司。FITC-羊抗兔IgG和DAPI购自武汉博士德生物工程有限公司。poly(I:C)购自Sigma公司。Dual-Luciferase reporter assay system购自Promega公司。
1.2 基因克隆与载体构建以本实验室分离和保存的PEDV河南流行株CH/ZMDZY/11序列(GenBank登录号:KC196276) 为基础,设计两对nsp1序列特异性引物。以PEDV CH/ZMDZY/11毒株基因组cDNA为模板,PCR扩增nsp1基因,引物对1的上、下游引物分别引入NcoⅠ和XhoⅠ酶切位点,序列如下,上游引物:5′-CATGCCATGGGCGCTAGCA ACCAAGTCACATTGG-3′;下游引物:5′-ATACT CGAGACCACCACGACGACCAAAAG-3′,扩增出的片段经NcoⅠ和XhoⅠ酶切后克隆入经相同酶切的pET-28a(+)载体中。引物对2的上、下游引物分别引入EcoRⅠ和XhoⅠ酶切位点,序列如下,上游引物:5′-AATGAATTCGCTAGCAACCA AGTCACATTGGC-3′;下游引物:5′-ATACTCGA GACCACCACGACGACCAAAAGTG-3′,扩增出的片段经EcoRⅠ和XhoⅠ酶切后克隆入经相同酶切的pCAGGS-HA载体中。最终获得重组质粒pET-28a-nsp1和pCAGGS-nsp1,经酶切和序列测定验证载体序列的正确性。
1.3 Nsp1多克隆抗体的制备及抗体效价的测定将鉴定正确的重组质粒pET-28a-nsp1转化入BL21(DE3) 宿主菌中,经过异丙基硫代半乳糖苷(IPTG)诱导表达后,超声裂解表达菌体,收取上清,参照His标签蛋白镍柱亲和层析纯化说明书进行重组蛋白的纯化,洗脱样品进行SDS-PAGE分析。将纯化后的nsp1用BCA法进行蛋白含量的测定,每只兔子免疫剂量为500 μg,与弗氏佐剂体积比1:1混合乳化后进行背部多点注射免疫。间隔2周分别进行二免和三免,三免10 d后采血,分离血清。用纯化的nsp1原核蛋白包被板子,包被量为0.5 μg/孔,将分离得到的抗nsp1阳性及阴性血清分别按照1:500、1:1 000、1:2 000、1:4 000、1:8 000、1:16 000、1:32 000、1:64 000、1:128 000等倍数进行稀释,以HRP-羊抗兔IgG (1:5 000稀释)作为二抗,间接ELISA方法测定nsp1多克隆抗体效价。
1.4 nsp1真核表达鉴定在六孔细胞板中培养HEK-293T细胞,待细胞长至单层时转染pCAGGS-nsp1和pCAGGS质粒,转染剂量为2 μg/孔,转染24 h后,分别以兔抗nsp1多克隆抗体和鼠抗GAPDH为一抗,以HRP-羊抗兔IgG和HRP-羊抗鼠IgG为二抗,通过Western blotting来验证nsp1和内参蛋白GAPDH的真核表达。
1.5 PEDV感染细胞中nsp1表达鉴定在六孔细胞板中培养Vero细胞,待细胞长至单层时感染PEDV流行株CH/ZMDZY/11,接毒剂量为0.5 MOI (Multiplicity of infection, MOI),待20 h细胞病变完全后,通过常规方法进行间接免疫荧光试验鉴定病毒感染细胞中nsp1的表达:以兔抗nsp1多克隆抗体(1:100稀释)为一抗,以FITC-羊抗兔IgG (1:100稀释)为二抗,在荧光显微镜下观察染色结果。
1.6 双荧光素酶法验证nsp1对IFN-β启动子活性影响HEK-293T接种12孔板,待细胞生长密度达到70%左右时转染pCAGGS-nsp1质粒以及干扰素报告质粒IFN-β-luc和内参基因质粒pRL-TK,3种质粒转染剂量比率为10:10:1,转染总剂量为1.05 μg/孔,同时设立转染空质粒pCAGGS和报告质粒对照。24 h后再次转染poly(I:C) (1 μg/孔),使其作用12 h。然后采用双荧光素酶报告基因检测试剂盒进行酶活性测定,结果取萤火虫荧光素酶信号值与海肾荧光素酶活性值的比值,每个组设3个重复。
1.7 nsp1不同表达剂量对IFN-β启动子活性影响为探究nsp1表达量对Ⅰ型干扰素启动子活性的影响,将HEK-293T接种12孔板,待细胞生长密度达到70%左右时,分别将0.5 μg/孔、1 μg/孔和2 μg/孔3种剂量的pCAGGS-nsp1质粒与等剂量的IFN-β-luc (0.5 μg/孔)和pRL-TK (0.05 μg/孔)共转染,并且同时设立pCAGGS (2 μg/孔)和报告质粒对照组,poly(I:C)刺激12 h后测定萤火虫荧光素酶信号值与海肾荧光素酶活性值的比值,每个组设3个重复。
1.8 ELISA检测IFN-β的表达水平将目的质粒pCAGGS-nsp1和空质粒pCAGGS分别转染HEK-293T细胞(各1 μg/孔),24 h后再转染poly(I:C) (1 μg/孔),使其作用12 h,收取细胞上清,用IFN-β定量ELISA试剂盒于450 nm处测定各孔OD值。根据标准曲线换算出样本中IFN-β含量(pg/mL)。
1.9 VSV病毒复制抑制试验HEK-293T接种12孔板,等细胞生长密度达到70%左右,转染目的质粒pCAGGS-nsp1,同时设立转染空质粒pCAGGS对照。转染质粒24 h后,转染poly(I:C) (1 μg/孔),12 h后将细胞及培养液混合物反复冻融3次收取细胞上清,然后将细胞上清以20、2-1、2-2、2-3、2-4、2-5、2-6稀释后接种于96孔板上培养的A549细胞,每孔100 μL,作用24 h后,接种VSV-GFP病毒(100 TCID50/孔),接毒后16 h,通过荧光显微镜观察荧光表达情况。
1.10 统计学分析以上所有数据均采用双尾T检验进行统计分析,数据由平均值±标准方差表示,P < 0.05表示差异显著,P < 0.01表示差异极为显著。
2 结果与分析 2.1 nsp1基因克隆与载体构建选取GenBank数据库中24株国内外代表性PEDV毒株(包括22株流行毒株和2株疫苗毒株),通过生物信息学预测这些PEDV nsp1基因的开放阅读框。氨基酸序列分析结果显示,不同地域的PEDV流行毒株与疫苗毒株的nsp1非常保守,氨基酸序列同源性最低为97.2%,最高为100% (结果未显示)。利用所设计的PEDV nsp1基因引物,通过PCR扩增出约330 bp的单一片段,与预期大小相符。提取pET-28a-nsp1与pCAGGS-nsp1重组质粒,对pET-28a-nsp1进行NcoⅠ和XhoⅠ双酶切,对pCAGGS-nsp1进行EcoRⅠ和XhoⅠ双酶切,均获得了约330 bp左右的目的条带(图 1)。将酶切鉴定正确的表达载体进行序列测定,测序结果说明nsp1基因在两个载体中的位置插入正确,核酸序列与预期结果一致。

|
| 图 1 PEDV nsp1的克隆与重组质粒的鉴定 Figure 1 Cloning of PEDV nsp1 and identification of recombinant plasmids. M: 15 000 bp ladder DNA marker; 1: pCAGGS-nsp1; 2: pCAGGS-nsp1/EcoRⅠ+ XhoⅠ; 3: pET-28a-nsp1; 4: pET-28a-nsp1/NcoⅠ+ XhoⅠ. |
| |
将获得的重组阳性质粒pET-28a-nsp1转入BL21(DE3) 感受态,IPTG诱导表达后,经SDS-PAGE验证表达,并经镍柱纯化后得到带有His标签的nsp1融合蛋白,相对分子质量大小约为12 kDa (图 2)。用纯化的重组nsp1免疫小鼠,三免10 d后采血,采用间接ELISA测定抗体效价。结果显示,三免后多克隆抗体效价达1:64 000 (表 1)。
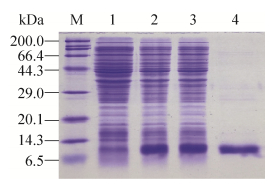
|
| 图 2 重组nsp1的表达及纯化 Figure 2 Expression and purification of recombinant nsp1. M: protein marker; 1: whole cell lysates of pET-28a-nsp1/BL21(DE3) uninduced; 2: whole cell lysates of pET-28a-nsp1/BL21(DE3) induced by IPTG; 3: solution supernatant from whole cell lysates of pET-28a-nsp1/BL21 (DE3) induced by IPTG; 4: purified nsp1. |
| |
| Serum (OD450) | Rabbit serum dilutions | |||||||
| 1:500 | 1:1 000 | 1:2 000 | 1:4 000 | 1:8 000 | 1:16 000 | 1:32 000 | 1:64 000 | |
| Positive serum | 1.579 | 1.402 | 1.285 | 1.212 | 0.963 | 0.732 | 0.531 | 0.391 |
| Negative serum | 0.182 | 0.156 | 0.118 | 0.103 | 0.092 | 0.081 | 0.082 | 0.061 |
将质粒pCAGGS-nsp1转染HEK-293T细胞,转染24 h后通过Western blotting来验证nsp1的真核表达(以GAPDH作为内参蛋白对照)。结果显示,pCAGGS-nsp1转染HEK-293T细胞24 h后能成功检测到nsp1的表达(图 3)。

|
| 图 3 nsp1真核表达鉴定 Figure 3 Identification of nsp1 eukaryotic expression. |
| |
为证明病毒感染细胞中存在所预测的nsp1,将PEDV流行株CH/ZMDZY/11感染Vero细胞,多聚甲醛固定细胞后,分别用制备的nsp1多抗作为一抗,以FITC-羊抗兔IgG作为二抗。间接免疫荧光实验结果显示,病毒在感染细胞过程中确实表达nsp1 (图 4)。

|
| 图 4 病毒感染细胞中nsp1蛋白的表达(100×) Figure 4 nsp1 expressed in PEDV-infected cells (100×). Mock stands for cells uninfected with PEDV. |
| |
为探究nsp1对Ⅰ型干扰素应答的影响,将pCAGGS-nsp1、IFN-β-luc和pRL-TK共转染HEK-293T细胞,同时设立空质粒对照组,24 h后转染poly(I:C)刺激细胞。然后裂解细胞,测定双荧光素酶活性,最终取萤火虫荧光素酶信号值与海肾荧光素酶活性值的比值。结果显示,与转染poly(I:C)组和空载体pCAGGS组相比,nsp1能显著抑制IFN-β启动子的活性(P < 0.01) (图 5)。
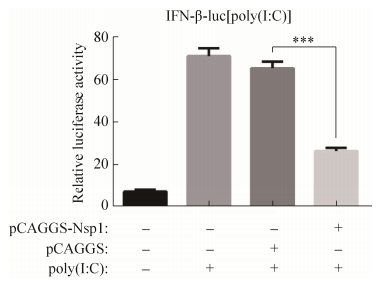
|
| 图 5 报告基因法评价nsp1对IFN-β启动子活性的影响 Figure 5 Effect of nsp1 on the promoter activity of IFN-β confirmed by dual luciferase reporter assay. |
| |
为探究不同nsp1表达水平对Ⅰ型干扰素的应答是否存在差异,将3种不同剂量的pCAGGS-nsp1与IFN-β-luc和pRL-TK共转染HEK-293T细胞,比较双荧光素酶活性的差异。结果显示,与转染poly(I:C)组和空载体pCAGGS组相比,不同转染剂量的nsp1真核表达质粒均能显著抑制IFN-β启动子的活性(P < 0.01),并且存在剂量依赖性,随着pCAGGS-nsp1转染剂量的增加,对Ⅰ型干扰素启动子活性的影响也逐渐增强(图 6)。
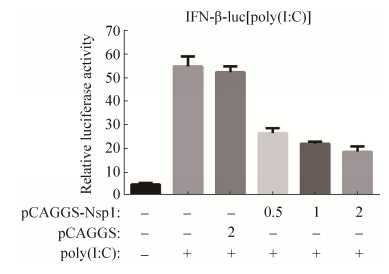
|
| 图 6 nsp1表达水平对IFN-β启动子活性的影响 Figure 6 Effect of nsp1 expression level on the promoter activity of IFN-β. |
| |
将pCAGGS-nsp1和pCAGGS分别转染293T细胞,24 h后再转染poly(I:C),12 h后收取细胞上清,使用ELISA试剂盒检测IFN-β蛋白含量。结果显示,与转染poly(I:C)组和空载体pCAGGS组相比,转染pCAGGS-nsp1组的IFN-β水平明显降低,即nsp1能显著抑制poly(I:C)诱导的IFN-β蛋白水平的表达(P < 0.01) (图 7)。

|
| 图 7 ELISA检测nsp1对IFN-β表达的影响 Figure 7 Effect of nsp1 on IFN-β expression detected by ELISA. |
| |
由于VSV对Ⅰ型IFN高度敏感,本研究利用表达GFP的重组VSV进行复制抑制试验来直观验证nsp1对细胞Ⅰ型IFN应答的影响。结果显示,在未加poly(I:C)对照组细胞裂解上清处理的A549细胞中,VSV-GFP的复制不受抑制;单独添加poly(I:C)刺激组和空载体pCAGGS转染+poly(I:C)刺激组细胞裂解上清处理的A549细胞中,VSV-GFP重组病毒的复制均受到不同程度的抑制,而在pCAGGS-nsp1转染组细胞裂解上清处理的A549细胞中,VSV-GFP重组病毒的复制受抑制程度大大降低,证明nsp1能明显抑制poly(I:C)介导的Ⅰ型IFN的产生和其抗病毒作用(图 8)。

|
| 图 8 VSV病毒复制抑制试验 Figure 8 VSV replication-inhibition bioassay. Mock stands for the group without poly (I:C). |
| |
干扰素和干扰素诱导的细胞抗病毒应答是机体抵抗病原体侵袭的第一道防线[18]。病毒感染期间,为了抵抗干扰素的抗病毒作用,包括冠状病毒在内的许多病毒都进化出了复杂的机制来逃避宿主的天然免疫,从而建立持续的感染。目前,已证实SARS-CoV编码至少8种蛋白可以作为干扰素的拮抗剂:nsp1、PLP、nsp7、nsp15、N、M、ORF3b和ORF6[19-20]。研究表明,同属于冠状病毒科的PEDV感染宿主后也能抑制其天然免疫应答[21]。研究发现PEDV可利用多个蛋白通过不同信号转导途径干扰宿主的Ⅰ型IFN应答。目前已证明PEDV的N蛋白能够与TBK1发生互作,通过阻断TBK1与下游IRF3的结合拮抗细胞的Ⅰ型IFN应答[22]。另外,在PEDV编码的16个nsps中,部分nsps对Ⅰ型IFN的拮抗功能及相关机制已得到解析[23]。其中,PEDV nsp3编码的木瓜样蛋白酶(PLP2) 作为去泛素化酶将RIG-I和STING去泛素化后失活,从而阻断该信号通路[24]。PEDV nsp5编码的3C样蛋白酶(3CLpro)能够通过蛋白水解切割核转录因子NF-κB必须调节蛋白(NEMO),从而拮抗细胞的Ⅰ型IFN应答[25]。
本研究在前期克隆和表达了PEDV中国流行株CH/ZMDZY/11的nsp1并制备了高效价的兔抗nsp1多克隆抗体,为了深入研究PEDV的nsp1对Ⅰ型干扰素的影响,我们构建了真核表达载体pCAGGS-nsp1,并将该载体转染293T细胞。通过Western blotting验证了nsp1能够在293T细胞中高效表达。PEDV流行株CH/ZMDZY/11感染Vero细胞的间接免疫荧光结果显示,PEDV自然感染状态下能够表达nsp1蛋白。我们通过双荧光报告基因法、ELISA试验以及VSV复制抑制试验等,证明了nsp1能显著抑制由poly(I:C)介导的IFN-β的转录和蛋白表达,并抑制Ⅰ型IFN介导的细胞抗病毒应答。
目前预防PEDV的主要手段是免疫母猪而使新生仔猪通过被动免疫获得保护。PEDV现有的商品化疫苗主要是基于CV777毒株的弱毒活疫苗,虽然该疫苗能在一定程度上减缓该病的发生与发展,但其免疫保护效果仍然存在不足[26]。另外,该疫苗对仔猪具有一定致病性。目前,国内外研究者正利用所分离的流行毒株进行传代致弱,以期研制相关疫苗。疫苗毒株的不稳定性使得对病毒致病性和动物机体抗病毒作用的机理的研究尤为重要。本研究证明,PEDV编码的nsp1能通过与IFN通路中的相关接头分子互作来抑制Ⅰ型IFN的表达及其抗病毒效应,从而来逃避天然免疫的识别和清除。本研究结果为PED高效疫苗及抗病毒药物的研发提供了具体的作用靶点,nsp1可作为潜在的可修饰毒力因子,为发现新的抗病毒治疗药物靶标提供了重要理论基础。
| [1] | Tian YF, Yu ZJ, Cheng KH, et al. Molecular characterization and phylogenetic analysis of new variants of the porcine epidemic diarrhea virus in Gansu, China in 2012. Viruses, 2013, 5(8): 1991–2004. DOI: 10.3390/v5081991 |
| [2] | Pensaert MB, de Bouck P. A new coronavirus-like particle associated with diarrhea in swine. Arch Virol, 1978, 58(3): 243–247. DOI: 10.1007/BF01317606 |
| [3] |
Ma SQ, Wang M, Feng L, et al. Development of bi-combined inactivated aluminum hydroxide adjuvanted vaccine against transmissible gastroenteritis virus and porcine epidemic diarrhea virus.
Chin J Anim Poul Infect Dis, 1995(6): 23–27.
(in Chinese). 马思奇, 王明, 冯力, 等. 猪传染性胃肠炎与猪流行性腹泻二联氢氧化铝细胞灭活疫苗的研究. 中国畜禽传染病, 1995(6): 23-27. |
| [4] |
Tong YE, Feng L, Li WJ, et al. Development of bi-combined attenuated vaccine against transmissible gastroenteritis virus and porcine epidemic diarrhea virus.
Chin J Prev Vet Med, 1999, 21(6): 406–410.
(in Chinese). 佟有恩, 冯力, 李伟杰, 等. 猪传染性胃肠炎与猪流行性腹泻二联弱毒疫苗的研究. 中国预防兽医学报, 1999, 21(6): 406-410. |
| [5] | Chen J, Wang C, Shi H, et al. Molecular epidemiology of porcine epidemic diarrhea virus in China. Arch Virol, 2010, 155(9): 1471–1476. DOI: 10.1007/s00705-010-0720-2 |
| [6] | Li WT, Li H, Liu YB, et al. New variants of porcine epidemic diarrhea virus, China, 2011. Emerg Infect Dis, 2012, 18(8): 1350–1353. DOI: 10.3201/eid1803.120002 |
| [7] | Wang LY, Byrum B, Zhang Y. New variant of porcine epidemic diarrhea virus, United States, 2014. Emerg Infect Dis, 2014, 20(5): 917–919. DOI: 10.3201/eid2005.140195 |
| [8] | Cima G. Viral disease affects US pigs: porcine epidemic diarrhea found in at least 11 states. J Am Vet Med Assoc, 2013, 243(1): 30–31. |
| [9] | Bhardwaj K, Guarino L, Kao CC. The severe acute respiratory syndrome coronavirus Nsp15 protein is an endoribonuclease that prefers manganese as a cofactor. J Virol, 2004, 78(22): 12218–12224. DOI: 10.1128/JVI.78.22.12218-12224.2004 |
| [10] | Park SJ, Kim HK, Song DS, et al. Complete genome sequences of a Korean virulent porcine epidemic diarrhea virus and its attenuated counterpart. J Virol, 2012, 86(10): 5964. DOI: 10.1128/JVI.00557-12 |
| [11] | Utiger A, Rosskopf M, Guscetti F, et al. Preliminary characterization of a monoclonal antibody specific for a viral 27 kD glycoprotein family synthesized in porcine epidemic diarrhoea virus infected cells. Coronaviruses Springer US, 1994: 197-202. |
| [12] | Snijder EJ, Bredenbeek PJ, Dobbe JC, et al. Unique and conserved features of genome and proteome of SARS-coronavirus, an early split-off from the coronavirus group 2 lineage. J Mol Biol, 2003, 331(5): 991–1004. DOI: 10.1016/S0022-2836(03)00865-9 |
| [13] | Connor RF, Roper RL. Unique SARS-CoV protein nsp1: bioinformatics, biochemistry and potential effects on virulence. Trends Microbiol, 2007, 15(2): 51–53. DOI: 10.1016/j.tim.2006.12.005 |
| [14] | Züst R, Cervantes-Barragán L, Kuri T, et al. Coronavirus non-structural protein 1 is a major pathogenicity factor: implications for the rational design of coronavirus vaccines. PLoS Pathog, 2007, 3(8): e109. DOI: 10.1371/journal.ppat.0030109 |
| [15] | Galán C, Enjuanes L, Almazán F. A point mutation within the replicase gene differentially affects coronavirus genome versus minigenome replication. J Virol, 2005, 79(24): 15016–15026. DOI: 10.1128/JVI.79.24.15016-15026.2005 |
| [16] | Wang YJ, Shi HL, Rigolet P, et al. Nsp1 proteins of group Ⅰ and SARS coronaviruses share structural and functional similarities. Infect Genet Evol, 2010, 10(7): 919–924. DOI: 10.1016/j.meegid.2010.05.014 |
| [17] | Wathelet MG, Orr M, Frieman MB, et al. Severe acute respiratory syndrome coronavirus evades antiviral signaling: role of nsp1 and rational design of an attenuated strain. J Virol, 2007, 81(21): 11620–11633. DOI: 10.1128/JVI.00702-07 |
| [18] | Rathinam VA, Fitzgerald KA. Cytosolic surveillance and antiviral immunity. Curr Opin Virol, 2011, 1(6): 455–462. DOI: 10.1016/j.coviro.2011.11.004 |
| [19] | Totura AL, Baric RS. SARS coronavirus pathogenesis: host innate immune responses and viral antagonism of interferon. Curr Opin Virol, 2012, 2(3): 264–275. DOI: 10.1016/j.coviro.2012.04.004 |
| [20] | Totura A. SARS coronavirus antagonizes innate immune signaling initiated by RIG-I but is recognized by TLR signaling via the adaptor molecule TRIF[D]. Dissertations & Theses -Gradworks, 2014. |
| [21] | Cao L, Ge X, Gao Y, et al. Porcine epidemic diarrhea virus inhibits dsRNA-induced interferon-β production in porcine intestinal epithelial cells by blockade of the RIG-I-mediated pathway. Virol J, 2015, 12(1): 1–8. DOI: 10.1186/s12985-014-0235-7 |
| [22] | Ding Z, Fang LR, Jing HY, et al. Porcine epidemic diarrhea virus nucleocapsid protein antagonizes beta interferon production by sequestering the interaction between IRF3 and TBK1. J Virol, 2014, 88(16): 8936–8945. DOI: 10.1128/JVI.00700-14 |
| [23] | Zhang QZ, Shi KC, Yoo D. Suppression of type Ⅰ interferon production by porcine epidemic diarrhea virus and degradation of CREB-binding protein by nsp1. Virology, 2016, 489: 252–268. DOI: 10.1016/j.virol.2015.12.010 |
| [24] | Xing YL, Chen JF, Tu J, et al. The papain-like protease of porcine epidemic diarrhea virus negatively regulates type Ⅰ interferon pathway by acting as a viral deubiquitinase. J Gen Virol, 2013, 94(7): 1554–1567. |
| [25] | Wang D, Fang LR, Shi YL, et al. Porcine epidemic diarrhea virus 3C-like protease regulates its interferon antagonism by cleaving NEMO. J Virol, 2016, 90(4): 2090–2101. DOI: 10.1128/JVI.02514-15 |
| [26] | Lee C. Porcine epidemic diarrhea virus: an emerging and re-emerging epizootic swine virus. Virol J, 2015, 12: 193. DOI: 10.1186/s12985-015-0421-2 |
 2017, Vol. 33
2017, Vol. 33




