中国科学院微生物研究所、中国微生物学会主办
文章信息
- 谢周杰, 张昭, 刘力伟, 柳孝晨, 陈义华
- Zhoujie Xie, Zhao Zhang, Liwei Liu, Xiaochen Liu, Yihua Chen
- 变形链球菌中的次级代谢产物及其在口腔生物被膜中的生态功能
- Secondary metabolites from Streptococcus mutans and their ecological roles in dental biofilm
- 生物工程学报, 2017, 33(9): 1547-1554
- Chinese Journal of Biotechnology, 2017, 33(9): 1547-1554
- 10.13345/j.cjb.170046
-
文章历史
- Received: February 15, 2017
- Accepted: April 20, 2017
2 中国科学院大学, 北京 100049;
3 南京农业大学 食品科技学院, 江苏 南京 210095
2 University of Chinese Academy of Sciences, Beijing 100049, China;
3 College of Food Science and Technology, Nanjing Agricultural University, Nanjing 210095, Jiangsu, China
口腔生物被膜(又称牙菌斑),是指位于口腔环境内的复杂微生物群落[1]。牙菌斑具有典型的生物被膜结构和生理功能,与龋病、牙周病等口腔感染性疾病的发生密切相关[2]。现在已经从牙菌斑中分离出来数百种细菌,流行病学、体外模型及动物模型的众多实验数据都支持变形链球菌(Streptococcus mutans,简称变链菌)是引起龋病的主要病原菌[3-5]。变链菌所具有的强黏附能力、代谢产酸和耐酸特性是其引起龋病的主要毒力特征[6]。变链菌UA159 (首个完成全集因组测序的变链菌菌株)的基因组分析为变链菌在口腔环境中的适应性和致龋性提供了很好的解释[7]。首先,变链菌基因组编码种类多样的糖基转移酶和具有黏附功能的细胞表面蛋白。在蔗糖存在的环境中,位于细胞表面的糖基转移酶能够高效合成葡聚糖,葡聚糖的产生极大地促进了细菌-细胞间的聚集和生物被膜方式生长。在没有蔗糖的情况下,细胞表面的黏附蛋白介导细菌与唾液组分及其他细菌细胞的相互作用,促进变链菌生物被膜方式的生长。另外,变链菌贡献了大约15%的遗传物质用于编码碳水化合物代谢和转运的相关蛋白,使得其碳水化合物的利用范围广于任何一个已知的革兰氏阳性细菌。然而变链菌基因组中没有发现有氧呼吸电子传递链的相关组分的编码基因,因此变链菌代谢种类繁多的碳水化合物为其生长提供能量的同时,最终伴随有甲酸、乙酸、乳酸等有机酸的产生,从而导致局部环境pH值的降低。低pH环境在抑制其他酸敏感细菌的同时,还造成牙釉质的持续性损伤,最终导致龋病的发生。变链菌具有F0F1 ATPase编码基因。F0F1 ATPase位于细胞膜上,具有酸稳定性和质子转运功能,能够在酸性环境下维持胞内的中性pH,从而赋予变链菌耐酸性。
与其他口腔细菌相比,变链菌的另一独特之处是具有丰富的次级代谢产物合成能力。细菌素(Bacteriocins)、聚酮(Polyketides,PKs)和非核糖体肽(Non-ribosomal peptides,NRPs)类化合物的生物合成基因簇广泛分布在不同变链菌菌株的基因组中[8]。变链菌中细菌素的研究最早起始于20世纪70年代[9]。近年来研究人员也发现变链菌UA159还具有产生杂合的聚酮/非核糖体肽类天然产物的能力[10]。本文对变型链球菌次级代谢产物及其在口腔生物被膜中的生态功能进行了总结,以期对后续的研究提供指导和借鉴。
1 变链菌产生的细菌素——变链素(Mutacins) 1.1 变链素的种类和活性细菌素(Bacteriocins)是由细菌通过核糖体合成的一类具有抗菌活性的多肽[11]。变链菌产生的细菌素又称为变链素。已知的变链素根据结构不同分为两大类,分别属于羊毛硫细菌素(Lantibiotics)和非羊毛硫细菌素(Non-lantibiotics) [12]。所谓羊毛硫细菌素,是指结构中包含羊毛硫氨酸(Lanthionine)或甲基羊毛硫氨酸(Methyllanthionine)的一类细菌素,这类结构单元的出现是翻译后修饰的产物。非羊毛硫细菌素结构上由蛋白类氨基酸残基组成,这些多肽经核糖体翻译生成,后经特殊的蛋白酶或者转运蛋白进行切割,转运至胞外变成成熟的多肽,不需要进一步的结构修饰。到目前为止,一共发现了10种变链素,包括6种羊毛硫类变链素mutacin Ⅰ、Ⅱ、Ⅲ (1140)、K8、Smb和B-Ny266,以及4种非羊毛硫类变链素mutacin Ⅳ、Ⅴ、Ⅵ和N[12]。尽管每种变链素都具有特异的抗菌谱,但是这两类变链素的抗菌谱则可以通过分明的特征来区分。羊毛硫类变链素一般呈现广谱的抗G+细菌的特征;而非羊毛硫类细菌素抗菌谱较窄,一般只对一些分类上较近的链球菌呈现出抑制活性。
虽然目前已经报道的变链素只有以上几种,Morency等根据抗菌表型筛选结果,将85个变链菌菌株分为24个不同类群[13],这一结果初步表明变链素在变链菌中的多样性。这一特点也进一步得到了遗传上的证实,Ali等通过遗传杂交的方法证实受试变链菌菌株中只有一小部分包含已知的变链素基因型,大部分菌株含有未知类型的变链素基因型[14]。本研究组通过对169株变链菌的基因组进行分析,发现211个mutacin基因簇,其中不乏有新颖前体肽的细菌素基因簇[8]。
1.2 变链素的生态功能变链素能够促进产生菌在口腔生物被膜中的早期定殖和长期生存,这一生态功能已经得到了众多研究报道的证实。体外实验证实,mutacinⅠ和mutacin Ⅳ可以促进变链菌在双种群生物被膜(Two-species biofilm)中占据主导地位[15]。在体内实验中,通过感染mutacin 1140的产生菌,发现变链菌在新宿主口腔中的定殖变得持久和稳定[16]。临床研究也发现,具有高活性细菌素表型的变链菌菌株更容易从母亲传播给儿童[17]。与非羊毛硫类细菌素相比,羊毛硫细菌素具有的广谱抗菌活性,为产生菌提供了强大的竞争优势,一般认为对变链菌在定殖和长期存在于口腔生物被膜的过程中起到了更大的作用。
研究表明在变链菌中,非羊毛硫类变链素的产生在时空上和天然感受态的形成存在着协同调控的关系[18],也就是说,变链素的产生和获取外源DNA的能力往往同时出现。在肺炎链球菌中也有类似的发现,这就暗示了非羊毛硫类变链素在体内的另一重要生态功能,即使得产生菌能够从相近的链球菌获得外源DNA作为营养来源,同时也可用于自身的遗传修复和新性状的获得,并最终赋予产生菌生存竞争优势[19]。另外,研究也发现mutacin Ⅳ和Ⅴ也是产生菌启动自杀途径的重要效应分子[20-21],意味着它们可能在种内群体调控方面发挥着重要作用。关于变链素生态功能的更详细描述,请参见Merritt等的综述[12]。
变链素促进产生菌在口腔中的定殖和生存的生态功能意味着变链素的产生将间接增加变链菌的致龋风险。鉴于此,变链素常被认为是变链菌产生的众多类型的毒力因子之一。然而,变链素与变链菌致龋性直接关系目前还缺乏足够的临床数据支撑。有研究发现龋病来源的变链菌,在变链素Ⅰ、Ⅲ、Ⅳ检出率方面要明显高于无龋来源的菌株[22]。另有研究报道却没有发现变链素编码基因与菌株来源的龋病状态存在相关性[23]。未来还需要更加深入的研究来阐明变链素与变链菌致龋性的关系。
2 变链菌产生的PKs/NRPs类天然产物 2.1 PKs/NRPs类天然产物种类PKs和NRPs是微生物中常见的两大类次级代谢产物,不同于核糖体合成的细菌素,它们分别由聚酮合酶(Polyketide synthases,PKSs)和非核糖体肽合成酶(Non-ribosomal peptide synthetases,NRPSs)催化合成。相对于变链素,变链菌产生的PKs/NRPs类小分子化合物引起人们关注的时间较晚。最近,Wu等发现在S. mutans UA159内存在一个功能性的杂合PKS-NRPS基因簇[10],Braga等首次分离鉴定了该基因簇负责合成的化合物mutanobactin A [23],由该基因簇负责合成的更多类型的mutanobactin衍生物也陆续被发现[24-26]。如图 1所示,mutanobactins在分子结构上是带有脂肪酸链修饰的环六肽或六肽。不同衍生物之间或含有不同的脂肪酸链,或含有不同的氨基酸残基,或没有发生大环环化。Zvanych等系统地研究了变链菌UA159中mutanobactins的生物合成过程,发现该基因簇除了合成mutanobactins外,还可以合成更多类型代谢物[26],包括mutanolins和mutanamide。Mutanolin类化合物含有和mutanobactins类似的大环结构,但包含更多氨基酸残基修饰,因而具有更大的分子量(图 1)。Mutanamide则具有更小的分子量,结构上是N端带有癸烯酸修饰的亮氨酸-丙氨酸二肽(图 1),推测是截短的合成产物。Braga等发现,mutanobactin A能够抑制白色念珠菌从单细胞酵母形态到丝状形态的转变,但对细胞分裂没有影响[23]。此外,Wang等实验证实mutanamide具有和mutanobactin A类似的活性,也能够抑制白色念珠菌菌丝体的形成。同时他们还发现mutanobactin B对激活巨噬细胞释放促炎细胞因子IL-6和IL-12具有显著的促进作用,但明显抑制MCP-1、GM-CSF、G-CSF和TNF-α等其他4个促炎因子的释放[25]。
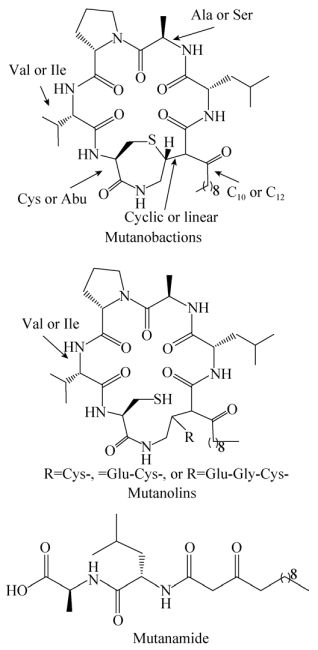
|
| 图 1 Mutanobactins、mutanolins及mutanamide的化学结构 Figure 1 Chemical structures of mutanobactins, mutanolins and mutanamide. |
| |
上述化合物都是由同一个生物合成基因簇负责合成,但是不同类型的NRPS/PKS基因簇还广泛分布在不同的变链菌菌株中。本研究组对169株可获得基因组信息的变链菌菌株进行了生物信息学分析,发现114株菌中共含有143个NRPS/PKS基因簇,它们在基因结构组成上分属于8种不同的类型,这一结果暗示着变链菌中还有更多新颖结构类型的次级代谢产物有待被发现[8]。
2.2 聚酮-非核糖体肽类天然产物可能的生态功能尽管在变链菌中发现了多样的PKS-NRPS基因簇,但是大部分基因簇负责合成的化合物还是未知的。Mutanobactins及其衍生物是目前在变链菌中分离鉴定的唯一一类PKs/NRPs类天然产物。变链菌中PKs/NRPs类天然产物的研究才刚刚开始,这类化合物的体内生态功能尚不明确,有待进一步研究。在与人类宿主的长期共进化过程中,PKS-NRPS基因簇在变链菌中较为普遍地保留下来,我们推测该类化合物可能是参与了变链菌与其他微生物或变链菌与宿主间的相互作用,进而赋予产生菌对口腔生物被膜环境的适应性。这一点可以从mutanobactins的生物活性中初见端倪。Mutanobactin A和mutanamide具有抑制白色念珠菌从单细胞酵母形态到丝状形态转变的活性。白色念珠菌是存在于人体粘膜组织表面的一种共生真菌,也常被发现与变链菌共存于口腔生物被膜之中,并存在着密切的相互作用[27]。与mutanobactin A生物活性一致的是,体外和昆虫模型都证明变链菌UA159能够抑制白色念珠菌生长形态的转变,使其保持单细胞酵母状态生长[28],这一结果暗示mutanobactins可能在变链菌-白色念珠菌相互作用中发挥着重要作用。Mutanobactin B具有的免疫调节活性暗示着它在变链菌-宿主互作中发挥着重要作用。
3 存在的问题及解决策略综上所述,在变链菌中还存在更多新颖结构类型的次级代谢产物生物合成基因簇,它们对产生菌的生理生态功能还不明确。对于某一基因的功能研究,经典的遗传学方法是构建基因突变体,并结合体外培养和动物模型来进行。但是当待研究的基因(或基因簇)在体外培养或动物模型培养条件下不表达时,突变株和野生型之间的表型将没有差异,此时经典的遗传学方法就出现了局限性。我们在对变链菌中的这些基因簇进行功能研究时,就遇到了同样的问题。我们发现这些基因簇在常规培养条件下往往是沉默的(不表达或表达量很低)。这一现象很容易理解,因为次级代谢产物的合成需要消耗很大的能量,这些额外能量消耗对于面对复杂多变的口腔环境(在营养供给、温度、pH和供氧等方面不断地经历着剧烈的变动)的变链菌来说,无疑是个巨大的负担。因此在变链菌中这些次级代谢过程受到了严格的调控,次级代谢产物仅在需要时才得以表达[12]。
微生物天然产物基因组挖掘是近年来天然产物领域的一个研究热点。该领域已经积累了一系列的方法和技术用于沉默基因簇的激活和天然产物的发现[29]。未来对变链菌中次级代谢的研究,可以结合天然产物基因组挖掘中沉默基因簇激活的手段和策略,首先获得基因簇对应的化合物。进一步的,在研究化合物生物活性的基础上推测它们可能的生理生态功能,进而通过体外模型和动物模型进行推测功能的验证。我们相信通过结合天然产物研究领域成熟的技术手段和研究策略,有望突破目前变链菌中次级代谢产物研究所面临的瓶颈。
4 总结与展望变链菌中的次级代谢基因簇具有丰富的多样性。而进行功能描述的次级代谢产物却只占很少一部分,它们包括6种羊毛硫类变链素、4种非羊毛硫类变链素和1种PKs/NRPs类天然产物(mutanobactins及其代谢衍生物)。它们或呈现出不同抗菌谱的抗细菌活性,或具有抑制真菌生长形态转变的活性,或具有免疫调节的活性。从多样的生物活性可知,这些次级代谢产物参与了变链菌与口腔生物被膜中的其他细菌、真菌或宿主间相互作用的过程。体外生物被膜模型和动物实验都不同程度地证实了变链素促进变链菌在口腔生物被膜中的定殖,以及在生物被膜中的长期存活过程。对于NRPs/PKs类天然产物的研究才刚刚起步,mutanobactin生物活性也暗示该类天然产物可能赋予变链菌在口腔生物被膜中的生存优势。变链菌中产生的次级代谢产物与变链菌的毒力关系尚不明确,推测它们可能更多的是赋予变链菌在口腔环境中的适应能力而非引起龋病的毒力。
基因组数据分析显示变链菌中存在类型多样的新细菌素生物合成基因簇和PKS/NRPS基因簇。未来随着更多的临床菌株和基因组数据的增加,预计会有更多的新颖次级代谢基因簇被发现。多样的基因簇暗示着多样的化合物结构、多样的生物活性和生理功能。结合天然产物研究领域的基因组挖掘手段,未来该领域将会不断出现更多有趣的发现。新的研究发现将使我们更加全面地认识变链菌与其他微生物及宿主的相互作用过程,从而为口腔疾病的治疗和预防提供新的策略和手段。同时,变链菌的次级代谢研究对新药研发也具有重要意义。羊毛硫变链素的广谱抗菌活性和mutanobactins的新颖活性,已经引起了人们的广泛兴趣[11, 30]。现有的证据让我们有理由相信更多的具有成药价值的化合物就存在于我们的口腔中,等待我们去发掘。
| [1] |
Zhou XD, Shi WY. Human oral microbial community and dental plaque biofilm.
West China J Stomatol, 2010, 28(2): 115–118.
(in Chinese). 周学东, 施文元. 人体口腔微生物组群与牙菌斑生物膜. 华西口腔医学杂志, 2010, 28(2): 115-118. |
| [2] | Seneviratne CJ, Zhang CF, Samaranayake LP. Dental plaque biofilm in oral health and disease. Chin J Dent Res, 2011, 14(2): 87–94. |
| [3] | Barsamian-Wunsch P, Park JH, Watson MR, et al. Microbiological screening for cariogenic bacteria in children 9 to 36 months of age. Pediatr Dent, 2004, 26(3): 231–239. |
| [4] | Zambon JJ, Kasprzak SA. The microbiology and histopathology of human root caries. Am J Dent, 1995, 8(6): 323–328. |
| [5] | Nobre dos Santos M, Melo dos Santos L, Francisco SB, et al. Relationship among dental plaque composition, daily sugar exposure and caries in the primary dentition. Caries Res, 2002, 36(5): 347–352. DOI: 10.1159/000065959 |
| [6] | Mitchell TJ. The pathogenesis of streptococcal infections:from tooth decay to meningitis. Nat Rev Microbiol, 2003, 1(3): 219–230. DOI: 10.1038/nrmicro771 |
| [7] | Ajdić D, McShan WM, McLaughlin RE, et al. Genome sequence of Streptococcus mutans UA159, a cariogenic dental pathogen. Proc Natl Acad Sci USA, 2002, 99(22): 14434–14439. DOI: 10.1073/pnas.172501299 |
| [8] | Liu LW, Hao TT, Xie ZJ, et al. Genome mining unveils widespread natural product biosynthetic capacity in human oral microbe Streptococcus mutans. Sci Rep, 2016, 6: 37479. DOI: 10.1038/srep37479 |
| [9] | Hamada S, Ooshima T. Inhibitory spectrum of a bacteriocinlike substance (mutacin) produced by some strains of Streptococcus mutans. J Dent Res, 1975, 54(1): 140–145. DOI: 10.1177/00220345750540010801 |
| [10] | Wu CG, Cichewicz R, Li YH, et al. Genomic island TnSmu2 of Streptococcus mutans harbors a nonribosomal peptide synthetase-polyketide synthase gene cluster responsible for the biosynthesis of pigments involved in oxygen and H2O2 tolerance. Appl Environ Microb, 2010, 76(17): 5815–5826. DOI: 10.1128/AEM.03079-09 |
| [11] | Cotter PD, Ross RP, Hill C. Bacteriocins-a viable alternative to antibiotics?. Nat Rev Microbiol, 2012, 11(2): 95–105. DOI: 10.1038/nrmicro2937 |
| [12] | Merritt J, Qi F. The mutacins of Streptococcus mutans:regulation and ecology. Mol Oral Microbiol, 2012, 27(2): 57–69. DOI: 10.1111/omi.2012.27.issue-2 |
| [13] | Morency H, Trahan L, Lavoie MC. Preliminary grouping of mutacins. Can J Microbiol, 1995, 41(9): 826–831. DOI: 10.1139/m95-113 |
| [14] | Ali SBS, Hurtubise Y, Lavoie MC, et al. Diversity of Streptococcusmutans bacteriocins as confirmed by DNA analysis using specific molecular probes. Gene, 2002, 283(1/2): 125–131. |
| [15] | Kreth J, Merritt J, Qi FX. Bacterial and host interactions of oral Streptococci. DNA Cell Biol, 2009, 28(8): 397–403. DOI: 10.1089/dna.2009.0868 |
| [16] | Hillman JD. Genetically modified Streptococcus mutans for the prevention of dental caries. Antonie van Leeuwenhoek, 2003, 82(1/4): 361–366. |
| [17] | Grönroos L, Saarela M, Mättö J, et al. Mutacin production by Streptococcus mutans may promote transmission of bacteria from mother to child. Infect Immun, 1998, 66(6): 2595–2600. |
| [18] | van der Ploeg JR. Regulation of bacteriocin production in Streptococcus mutans by the quorum-sensing system required for development of genetic competence. J Bacteriol, 2005, 187(12): 3980–3989. DOI: 10.1128/JB.187.12.3980-3989.2005 |
| [19] | Wholey WY, Kochan TJ, Storck DN, et al. Coordinated bacteriocin expression and competence in Streptococcus pneumoniae contributes to genetic adaptation through neighbor predation. PLoS Pathog, 2016, 12(2): e1005413. DOI: 10.1371/journal.ppat.1005413 |
| [20] | Perry JA, Jones MB, Peterson SN, et al. Peptide alarmone signalling triggers an auto-active bacteriocin necessary for genetic competence. Mol Microbiol, 2009, 72(4): 905–917. DOI: 10.1111/mmi.2009.72.issue-4 |
| [21] | Xie ZJ, Okinaga T, Niu GQ, et al. Identification of a novel bacteriocin regulatory system in Streptococcus mutans. Mol Microbiol, 2010, 78(6): 1431–1447. DOI: 10.1111/j.1365-2958.2010.07417.x |
| [22] | Kamiya RU, Napimoga MH, Höfling JF, et al. Frequency of four different mutacin genes in Streptococcus mutans genotypes isolated from caries-free and caries-active individuals. J Med Microbiol, 2005, 54(6): 599–604. DOI: 10.1099/jmm.0.45870-0 |
| [23] | Braga MP, Piovesan A, Valarini N, et al. Genotypic diversity and virulence factors of Streptococcus mutans in caries-free and caries-active individuals. Braz Arch Biol Tech, 2013, 56(2): 241–248. DOI: 10.1590/S1516-89132013000200009 |
| [24] | Joyner PM, Liu JM, Zhang ZJ, et al. Mutanobactin A from the human oral pathogen Streptococcus mutans is a cross-kingdom regulator of the yeast-mycelium transition. Org Biomol Chem, 2010, 8(24): 5486–5489. DOI: 10.1039/c0ob00579g |
| [25] | Wang XR, Du L, You JL, et al. Fungal biofilm inhibitors from a human oral microbiome-derived bacterium. Org Biomol Chem, 2012, 10(10): 2044–2050. DOI: 10.1039/c2ob06856g |
| [26] | Zvanych R, Lukenda N, Li X, et al. Systems biosynthesis of secondary metabolic pathways within the oral human microbiome member Streptococcus mutans. Mol Biosyst, 2015, 11(1): 97–104. DOI: 10.1039/C4MB00406J |
| [27] | Metwalli KH, Khan SA, Krom BP, et al. Streptococcus mutans, Candida albicans, and the human mouth:a sticky situation. PLoS Pathog, 2013, 9(10): e1003616. DOI: 10.1371/journal.ppat.1003616 |
| [28] | Barbosa JO, Rossoni RD, Vilela SFG, et al. Streptococcus mutans can modulate biofilm formation and attenuate the virulence of Candida albicans. PLoS ONE, 2016, 11(3): e0150457. DOI: 10.1371/journal.pone.0150457 |
| [29] | Bachmann BO, van Lanen SG, Baltz RH. Microbial genome mining for accelerated natural products discovery:is a renaissance in the making?. J Ind Microbiol Biotechnol, 2014, 41(2): 175–184. DOI: 10.1007/s10295-013-1389-9 |
| [30] | Pierce CG, Lopez-Ribot JL. Candidiasis drug discovery and development:new approaches targeting virulence for discovering and identifying new drugs. Exp Opin Drug Dis, 2013, 8(9): 1117–1126. DOI: 10.1517/17460441.2013.807245 |
 2017, Vol. 33
2017, Vol. 33