中国科学院微生物研究所、中国微生物学会主办
文章信息
- 蔡艳青, 齐显尼, 齐奇, 蔺玉萍, 王正祥, 王钦宏
- Cai Yanqing, Qi Xianni, Qi Qi, Lin Yuping, Wang Zhengxiang, Wang Qinhong
- 敲除MIG1和SNF1基因对酿酒酵母共利用葡萄糖和木糖的影响
- Effect of MIG1 and SNF1 deletion on simultaneous utilization of glucose and xylose by Saccharomyces cerevisiae
- 生物工程学报, 2018, 34(1): 54-67
- Chinese Journal of Biotechnology, 2018, 34(1): 54-67
- 10.13345/j.cjb.170098
-
文章历史
- Received: March 10, 2017
- Accepted: May 27, 2017
2 中国科学院天津工业生物技术研究所 中国科学院系统微生物工程重点实验室,天津 300308;
3 中国科学院大学,北京 100049
2 Key Laboratory of Systems Microbial Biotechnology, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
3 University of Chinese Academy of Sciences, Beijing 100049, China
利用可再生的木质纤维素生物质生产生物燃料和生物基化学品是解决全球可持续发展所面临的资源和环境问题的重要途径之一。木质纤维素主要由纤维素(38%–50%)、半纤维素(23%–32%)和木质素(15%–30%)组成[1]。当前工业条件下,木质纤维素经过预处理技术解离出纤维素和半纤维素,随后被纤维素酶等水解成微生物可发酵的糖类[2],水解液中木糖的含量仅次于葡萄糖。目前,如何实现木糖的高效转化以及与葡萄糖的共利用依然是阻碍木质纤维素类生物质经济性利用的主要关键因素。
自然界中有许多微生物包括细菌、真菌和酵母菌可以利用木糖,而酵母菌由于具有较强的工业应用属性,引起人们的广泛关注。利用木糖的酵母菌主要可以分为两大类:一类是以树干毕赤酵母Pichia stipitis为代表的天然利用木糖的酵母菌,另一类是携带外源木糖代谢途径的酿酒酵母Saccharomyces cerevisiae工程菌。然而,葡萄糖阻遏效应(Glucose repression)在酵母菌糖代谢调控中普遍存在。无论是天然菌[3]还是工程菌[4-5],在利用葡萄糖和木糖的混合糖时都表现出葡萄糖的优先利用现象,也就是说,木糖只有在葡萄糖耗尽后才开始被利用,从而导致发酵过程的总糖转化率和生产强度降低。在混合糖条件下酿酒酵母工程菌不能速效利用木糖的原因之一可能是由于葡萄糖抑制木糖代谢所需基因的表达,葡萄糖对这些基因表达抑制作用的解除或缓解有利于促进混合糖条件下木糖的利用效率。有研究表明,酿酒酵母工程菌的一些糖转运蛋白可以非特异地转运木糖,例如Hxt7,但在葡萄糖存在条件下HXT7的表达受抑制,从而限制混合糖条件下木糖的转运和利用[6]。Wahlbom等[7]通过研究化学诱变获得的木糖代谢能力提高的突变工程菌中基因表达水平的变化,发现该突变株中一些参与糖转运、初始木糖代谢和磷酸戊糖途径的基因表达加强,而且突变株在木糖唯一碳源条件下葡萄糖阻遏效应关键抑制因子MIG1的表达水平也降低,但未明确指出表明MIG1是否直接参与葡萄糖抑制木糖代谢的调控。de Figueiredo Vilela等[8]通过进化工程(Evolutionary engineering)获得木糖代谢效率提升的菌株,与出发菌株相比,该进化菌的HXT2 (编码低亲和力己糖转运蛋白)和TAL1 (编码磷酸戊糖途径非氧化阶段的转醛酶)表达水平均升高。另一方面有研究表明,MIG1的敲除能部分缓解工程菌中葡萄糖对木糖代谢的抑制作用,提高木糖在葡萄糖保持低水平的限碳或限氮连续发酵时的发酵能力[9-10]。另外,呼吸缺陷菌株经过工程改造和进一步的适应性进化后,木糖的发酵能力得到提高,这可能与经由Snf1/Mig1的葡萄糖感应和阻遏调控途径受到扰动有关[11]。虽然以上研究表明葡萄糖阻遏木糖代谢所需基因的表达可能是在混合糖条件下工程菌不能速效利用木糖的原因之一,另一方面也表明解除或扰动葡萄糖阻遏效应的关键因子可能有助于实现葡萄糖和木糖的共利用。然而,尚无研究报道明确指出MIG1和/或SNF1如何影响混合糖条件下木糖代谢所需基因的表达调控。
关于酿酒酵母的葡萄糖阻遏效应已经有大量的研究[12-15]。MIG1和SNF1是葡萄糖阻遏效应的两个关键因子。MIG1基因编码一个含有2个C2H2锌指结构的蛋白,可以结合到一些基因的启动子区域,在葡萄糖存在的条件下抑制这些基因的转录,例如麦芽糖、蔗糖和半乳糖代谢相关的酶基因[16-17]。Mig1受Snf1的负调控。SNF1编码一个蛋白激酶,是碳代谢中的中心调节因子,可用于解除葡萄糖对次级碳源的阻碍作用,如麦芽糖、半乳糖、蔗糖等。在葡萄糖存在的条件下,Snf1的活性受到抑制[18-19]。在没有葡萄糖或葡萄糖浓度很低的条件下,Snf1磷酸化Mig1,使细胞核中的Mig1转移到细胞质中,不能再结合到基因启动子上抑制基因的转录表达,从而导致葡萄糖所抑制基因的表达解抑制[20-21]。因此,葡萄糖阻遏效应可以归结为3个连续的负调控步骤:葡萄糖抑制Snf1的活性,Snf1抑制Mig1转录抑制因子的功能,Mig1抑制靶基因的转录。
本实验利用前期构建的一株可以利用木糖的酿酒酵母工程菌株W32N55a,通过同源重组技术分别对葡萄糖阻遏效应中的关键基因SNF1和MIG1基因进行单敲除和双敲除,然后评价了敲除菌株对葡萄糖和木糖共利用的性能,并利用转录组测序(RNA-seq)技术分析了在低浓度混合糖条件下MIG1和SNF1单敲除和双敲除对全基因组水平上基因表达谱的影响。发现SNF1的敲除以及与MIG1的双敲除有利于葡萄糖和木糖的共利用,初步揭示了Mig1和Snf1在负调控氮分解阻遏基因中的作用,同时,为进一步提高葡萄糖和木糖的共利用提供了依据和线索。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒本文所用菌株和质粒见表 1。
| Strains and plasmids | Characteristics | Source |
| S. cerevisiae strains | ||
| W32N55a | MATa, PsXR, PsXDH, ScXK | Provided by Yin Li lab at Institute of Microbiology, Chinese Academy of Sciences |
| W32N55a (mig1△0) | W32N55a (mig1::ZeoR) | This study |
| W32N55a (snf1△0) | W32N55a (snf1::KanR) | This study |
| W32N55a (mig1 & snf1△0) | W32N55a (mig1::ZeoR, snf1::KanR) | This study |
| Plasmids | ||
| pYES2/CT-ZGO-ZeoR | Plasmid carrying ZeoR gene | This lab |
| pRS313-ZGO-KanR | Plasmid carrying KanR gene | This lab |
| PsXR: xylose reductase from P. stipitis; PsXDH: xylitol dehydrogenase from P. stipitis; ScXK: xylulokinase from S. cerevisiae. | ||
根据Saccharomyces Genome Database (SGD,ID:S000003003)中报道的酿酒酵母中MIG1基因序列,设计两对同源臂片段扩增引物F1、R1和F2、R2;根据质粒pYES2/CT-ZGO-ZeoR中ZeoR表达框,设计引物F3、R3;敲除验证引物F4、R4。根据SGD (ID:S000002885)中报道的酿酒酵母中SNF1基因序列,设计两对同源臂片段扩增引物F5、R5和F6、R6;根据质粒pRS313-ZGO-KanR中KanR表达框,设计引物F7、R7;敲除验证引物F8、R8。引物由生工生物工程(上海)股份有限公司合成。本文所用引物见表 2。
| Primer | Sequence (5′–3′) | Restriction site |
| F1 | GAGAAACTCCAGAGTGGCGT | |
| R1 | CGGGGTACCATCAACGTTAGACACTTGTG | KpnⅠ |
| F2 | CGCGGATCCCCCACACATGGACTGATACG | BamHⅠ |
| R2 | TGACGAGACAACCAACCACC | |
| F3 | CGGGGTACCCATATGCCCACACACCATAG | KpnⅠ |
| R3 | CGCGGATCCGATATCAGCTTGCAAATTA | BamHⅠ |
| F4 | AGTACTCATTAACGAAGAC | |
| R4 | TCCTGCTCCTCGGCCACGAAGT | |
| F5 | CCACATGACGCAAGTAACTT | |
| R5 | CGGGGTACCTTGTTGTTACTGCTCATGTT | KpnⅠ |
| F6 | AAATATGCGGCCGCATGGAATTAGCCGTTAACAG | NotⅠ |
| R6 | TGGCAGCATGATTTGATATC | |
| F7 | CGGGGTACCCCCGGGCGTACGCTGCA | KpnⅠ |
| R7 | AAATATGCGGCCGCCGGCCGATCGATGAATTCG | NotⅠ |
| F8 | TGCGCATTCGTGTCCAAACA | |
| R8 | GCATCAACAATATTTTCACC |
Taq DNA聚合酶、Pfu DNA聚合酶、限制性内切酶、T4 DNA连接酶购自北京全式金生物技术有限公司;蛋白胨、酵母提取物购自英国Oxoid公司;博来霉素、G418购自北京索莱宝公司;葡萄糖、木糖购自北京国药集团有限公司。核酸电泳仪、凝胶成像系统购自美国Bio-Rad公司;PTC100型PCR仪购自美国M J Research公司等。
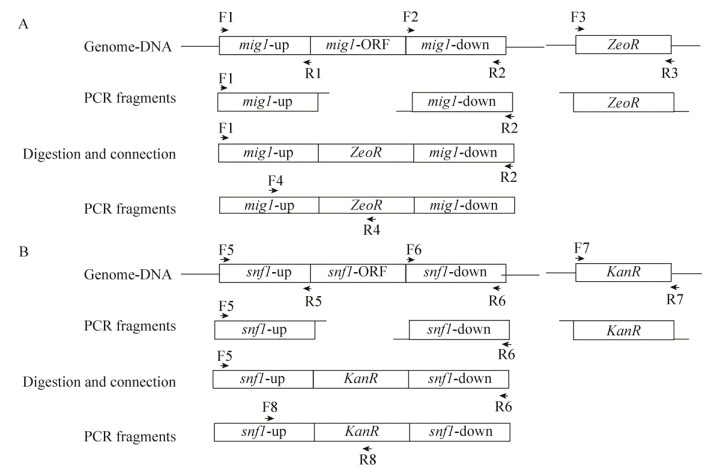
1.2 方法 1.2.1 SNF1和MIG1基因同源重组DNA片段的构建MIG1基因敲除同源片段的构建过程如图 1A所示。以W32N55a菌株基因组为模板,用引物对F1/R1和F2/R2,分别扩增MIG1基因两端同源臂mig1-up和mig1-down,各500 bp左右。以质粒pYES2/CT-ZGO-ZeoR为模板,用引物对F3/R3扩增基因ZeoR表达框,大小为1 200 bp左右。用限制性内切酶KpnⅠ和BamHⅠ将相应片段分别酶切,用T4 DNA连接酶将获得的片段mig1-up、ZeoR、mig1-down连接起来获得MIG1基因敲除同源片段“mig1-up-ZeoR-mig1-down”,并用引物对F1/R2扩增此片段,大小约为2 200 bp。
|
| 图 1 MIG1和SNF1基因敲除同源片段的构建过程 Figure 1 Working flow for MIG1 and SNF1 disruption cassette construction. |
| |
SNF1基因敲除组同源片段的构建过程如图 1B所示。以W32N55a菌株基因组为模板,用引物对F5/R5和F6/R6,分别扩增SNF1基因两端同源臂snf1-up和snf1-down,各500 bp左右。以质粒pRS313-ZGO-KanR为模板,用引物对F7/R7扩增基因KanR表达框,大小为1 500 bp左右。用限制性内切酶KpnⅠ和NotⅠ将相应片段分别酶切,用T4 DNA连接酶将片段snf1-up、KanR、snf1-down接起来获得SNF1基因敲除同源片段“snf1-up-KanR-snf1-down”,并用引物对F5/R6扩增敲除同源片段,大小约为2 500 bp。
敲除同源片段构建过程中各片段的合成都采用高保真的pfu DNA聚合酶。
1.2.2 酵母细胞培养,基因组提取和PCR 1.2.3 SNF1基因和MIG1基因敲除同源片段的转化及克隆子筛选采用LiAc/SS-DNA/PEG高效酵母转化法[25],将MIG1基因敲除同源片段转化至酿酒酵母W32N55a感受态细胞中。转化后的菌液涂布在含100 μg/mL博来霉素的YPD平板(酵母膏2%、蛋白胨2%,葡萄糖2%,琼脂2%)上,30 ℃培养2–5 d单菌落出现。将SNF1基因敲除同源片段转化至酿酒酵母W32N55a感受态细胞中。转化后的菌液涂布在含200 μg/mL G418的YPD (酵母膏1%,蛋白胨2%,葡萄糖2%,琼脂糖2%)平板上,30 ℃培养2–5 d单菌落出现。将SNF1基因敲除同源片段同时转化至已经敲除掉MIG1的酿酒酵母W32N55a (mig1∆0)感受态细胞中,转化后的菌液涂布在含100 μg/mL的博来霉素和200 μg/mL G418的YPD平板上,30℃培养2–5 d单菌落出现。
1.2.4 克隆子验证将平板上出现的单菌落,挑选至YPD液体培养中,提取基因组,用Taq DNA聚合酶进行PCR验证。用引物对F4/R4验证MIG1基因敲除菌,用引物对F8/R8验证SNF1基因敲除菌,用这两对引物验证双敲菌株。
1.2.5 发酵实验将亲本菌株和敲除菌株在相同的条件下进行代谢评价。发酵条件分别为:YP培养基(酵母膏2%、蛋白胨2%),5%混合糖和10%混合糖(葡萄糖:木糖=1:1),30℃,初始接种量为0.5 OD,250 mL三角瓶中发酵体系为80 mL、200 r/min。
1.2.6 转录组测序和数据分析按照5%混合糖发酵条件培养细胞,取9 h处的细胞送至北京金唯智公司进行转录组测序并分析表达差异基因。基因功能注释和途径分析采用SGD的途径数据库(http://pathway.yeastgenome.org/)。YEASTRACT在线数据库用来预测可能参与调控表达差异基因的关键转录因子[26]。
1.2.7 葡萄糖、木糖、乙醇含量的测定发酵液中的葡萄糖、木糖、乙醇含量利用HPLC进行测定。采用装有示差折光检测器的安捷伦HPLC检测仪器,伯乐Aminex HPX-87H分析柱,流动相使用0.005 mol/L硫酸,流速为0.6 mL/min,温度63 ℃。
2 结果与分析 2.1 SNF1和MIG1基因敲除菌的构建 2.1.1 同源重组DNA片段的构建以酿酒酵母W32N55a基因组为模板,用PCR分别扩增MIG1和SNF1基因上、下游同源臂片段,结果如图 2A所示;ZeoR和KanR抗性基因片段结果如图 2B所示;SNF1基因敲除的同源片段“snf1-up-KanR-snf1-down”以及MIG1基因敲除的同源片段“mig1-up-ZeoR-mig1-down”结果如图 2C所示。经过测序结果正确。这样,获得的DNA片段可以用于相应目标基因的敲除。

|
| 图 2 同源重组DNA片段的构建 Figure 2 Construction of homologous recombination DNA fragments. (A) M: DNA marker; 1: snf1-up; 2: snf1-down; 3: mig1-up; 4: mig1-down. (B) M: DNA marker; 1: ZeoR; 2: KanR. (C) M: DNA marker; 1: snf1-up-KanR-snf1-down; 2 200bp; 2: mig1-up-ZeoR-mig1-down. |
| |
用引物对F4/R4进行PCR鉴定筛选出W32N55a (mig1△0)的单克隆,用引物对F8/R8进行PCR鉴定筛选出W32N55a (snf1△0)和W32N55a (mig1 & snf1△0)的单克隆,电泳结果如图 3所示。这些结果表明目标突变菌株被成功构建。

|
| 图 3 阳性克隆子的验证 Figure 3 Screening for positive clones. (A) M: marker; lane 1–2: W32N55a (snf1Δ0), PCR with primer F8 and R8; lane 3–5: W32N55a (mig1Δ0), PCR with primer F4 and R4. (B) M: marker; lane 1–3: W32N55a (mig1 & snf1Δ0), PCR with primer F8 and R8. |
| |
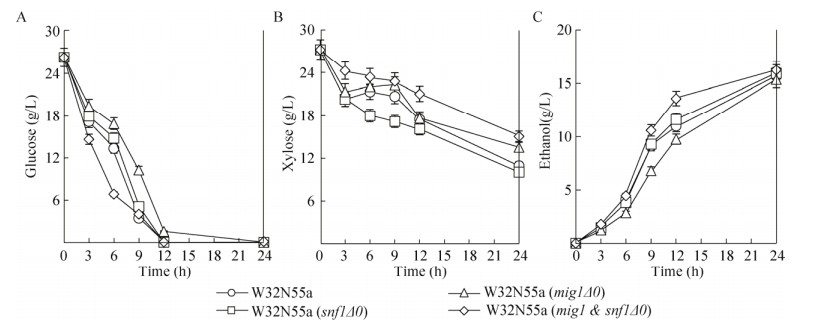
对原始菌W32N55a、单敲除菌株W32N55a (mig1△0)和W32N55a (snf1△0)、双敲除菌株W32N55a (mig1 & snf1△0)进行发酵,并测定发酵过程中发酵液葡萄糖、木糖、乙醇含量的变化。对于5%混合糖中的葡萄糖消耗而言,从图 4A的发酵曲线可以看出,与原始菌相比,MIG1敲除菌降低,SNF1敲除菌基本不变,双敲除菌显著加快。根据表 3中的发酵参数比较,原始菌株的最大葡萄糖消耗速率和最大葡萄糖比消耗速率分别为(3.05±0.33) g/(L·h)和(2.65±0.29) g/(L·h·OD),MIG1敲除菌相应的发酵参数分别降低为原始菌株的0.76倍和0.70倍,而MIG1和SNF1双敲除菌相应的发酵参数分别提高到原始菌株的1.28倍和1.24倍。Klein等曾报道[27],MIG1敲除菌的葡萄糖代谢能力下降,延滞期变长,导致混合糖的综合利用速度变慢。这与我们的结果相似。而且,SNF1的敲除可能在某种程度上消除了MIG1敲除对葡萄糖消耗的负面影响,甚至让葡萄糖代谢更好。
|
| 图 4 原始菌和敲除菌混合糖发酵 Figure 4 Evaluation for mixed xylose and glucose fermentation. (A, B, C) glucose consumption, xylose consumption and ethanol production profiles using 5% mixed sugar. Data represent the mean and standard error of duplicate cultures grown on each source. |
| |
| Conditions | 5% mixed glucose and xylose | 10% mixed glucose and xylose | ||||||
| Strains | WT | mig1∆ | snf1∆ | mig1 & snf1∆ | WT | mig1∆ | snf1∆ | mig1 & snf1∆ |
| Maximum glucose consumption rate, g/(L·h) | 3.05±0.33 | 2.31±0.12 | 2.79±0.29 | 3.89±0.22 | 2.12±0.01 | 2.12±0.02 | 2.12±0.00 | 2.06±0.00 |
| Maximum specific glucose consumption rate, g/(L·h·OD) | 2.65±0.29 | 1.86±0.10 | 2.27±0.24 | 3.30±0.19 | 0.23±0.01 | 0.22±0.00 | 0.21±0.00 | 0.19±0.00 |
| Maximum xylose consumption rate, g/(L·h) | 2.23±0.04 | 1.95±0.19 | 2.31±0.01 | 0.98±0.01 | 0.71±0.01 | 0.80±0.01 | 0.90±0.01 | 0.76±0.01 |
| Maximum specific xylose consumption rate, g/(L·h·OD) | 1.94±0.03 | 1.58±0.15 | 1.88±0.01 | 0.83±0.00 | 0.07±0.00 | 0.08±0.00 | 0.09±0.00 | 0.08±0.00 |
| Ethanol productivity, g/(L·h) | 1.01±0.02 | 0.81±0.01 | 1.03±0.03 | 1.18±0.15 | 1.24±0.01 | 1.28±0.01 | 1.27±0.03 | 1.33±0.01 |
| Specific ethanol productivity, g/(L·h·OD) | 0.46±0.07 | 0.34±0.10 | 0.40±0.07 | 0.51±0.01 | 0.16±0.00 | 0.14±0.00 | 0.16±0.00 | 0.15±0.00 |
| Ethanol yield, g/g | 0.37±0.00 | 0.36±0.00 | 0.37±0.00 | 0.38±0.00 | 0.37±0.00 | 0.36±0.00 | 0.37±0.00 | 0.38±0.00 |
| Data represent the mean and standard error of duplicate cultures grown on each source. | ||||||||
对于5%混合糖中的木糖利用而言,从图 4B的发酵曲线可以看出,在0–3 h内,原始菌可以代谢木糖,似乎没有受到葡萄糖的抑制,当3 h以后,细胞似乎受到葡萄糖抑制效应的影响,不再代谢木糖,直到9 h以后才继续代谢木糖,此时混合糖中残余的葡萄糖只剩下约3 g/L。与原始菌相比,MIG1敲除菌对混合糖中木糖的利用表现类似的代谢趋势,表明MIG1的敲除对解除葡萄糖对木糖的抑制作用没有作用。而SNF1敲除菌的木糖利用在6–9 h没有停滞,表明SNF1的敲除可能有助于解除葡萄糖对木糖代谢的抑制作用。与SNF1敲除菌相似,MIG1和SNF1双敲除菌木糖的利用在6–9 h没有明显的停滞,但木糖利用速率明显变慢,原始菌株的最大木糖消耗速率和最大木糖比消耗速率分别为(2.23±0.04) g/(L·h)和(1.94±0.03) g/(L·h·OD) (表 3),双敲除菌的相应发酵参数降低为原始菌株的0.44倍和0.43倍,进一步说明葡萄糖对木糖抑制作用在某种程度上的解除可能归功于SNF1的敲除而不是MIG1;总体而言,该菌利用木糖的速度与原始菌株相比明显变慢。
对于5%混合糖发酵最终的乙醇生产性能而言(图 4C),MIG1敲除会降低代谢葡萄糖产生乙醇的速率,其最大乙醇生产速率和最大乙醇比生产速率分别降低为原始菌株的0.80倍和0.74倍。曾有研究表明,MIG1敲除提高混合糖利用的能力有限,只有在特定的低水平葡萄糖条件下的限碳或限氮连续发酵中才能实现[9-10]。在我们利用丰富氮源培养的条件下,没有观察到MIG1敲除对木糖代谢和混合糖利用的促进效应,这可能与所采用的发酵条件不同有关。由于葡萄糖和木糖利用的叠加效应,SNF1敲除菌与原始菌相比乙醇的生产速率相当。MIG1和SNF1双敲除菌的乙醇生产速率加快,最大乙醇生产速率和最大乙醇比生产速率分别提高到原始菌株的1.08倍和1.11倍,这可能与SNF1敲除后部分解除了葡萄糖对木糖利用的延滞同时促进了葡萄糖的代谢能力有关,MIG1在其中的作用比较复杂,尚难以分析。另外,对于最终糖醇转化率,即乙醇产率而言(表 3),敲除菌株与原始菌株之间并没有发现明显的差异。因此,MIG1和SNF1的双敲除能够促进葡萄糖和木糖共利用,主要体现在提高混合糖产生乙醇的效率方面。
当将混合糖浓度提到10%时,与5%混合糖的发酵结果相比,敲除菌对发酵产生的影响程度明显降低。从表 3中可以看出,敲除菌和原始菌的大部分发酵参数没有明显的差异,只有MIG1和SNF1双敲除的最大葡萄糖比消耗速率降低为原始菌株的0.82倍,而最大木糖比消耗速率提高到原始菌株的1.23倍。这个结果表明MIG1和SNF1的双敲除对葡萄糖和木糖共利用也受不同糖浓度的影响,具体的机理尚待研究。
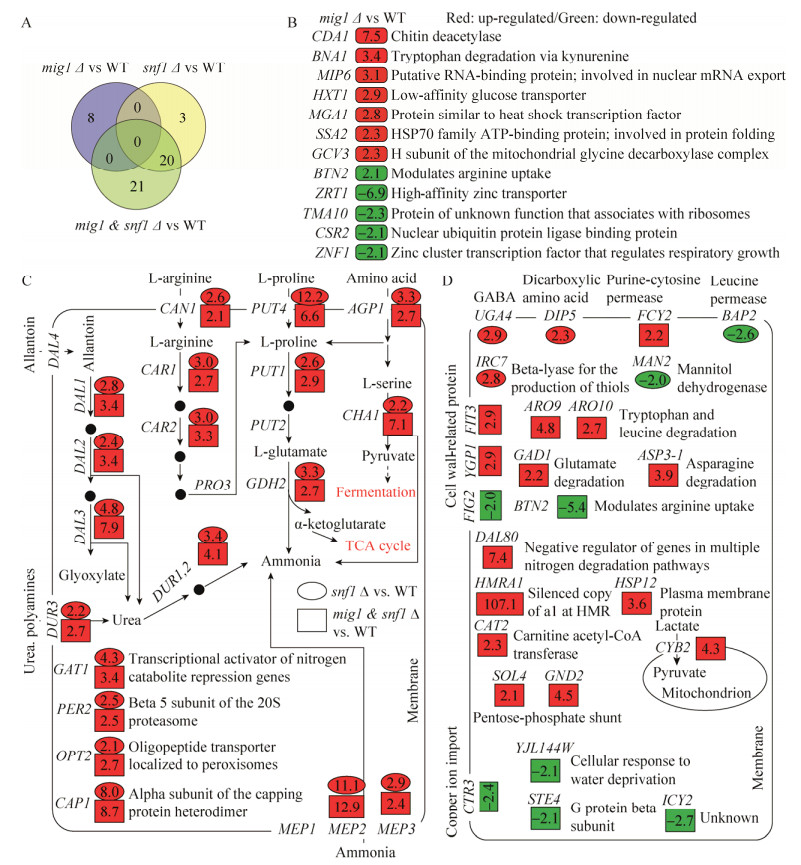
2.3 转录组测序分析敲除菌的基因表达差异为了从全基因组转录水平上解析MIG1和SNF1单敲除和双敲除对酵母共利用葡萄糖和木糖产生影响的可能原因,我们对5%混合糖发酵9 h的酵母细胞进行了转录组测序分析,并以相同发酵条件下的原始菌株W32N55a为对照,分析了敲除菌中发生明显表达差异的基因,并结合SGD数据库的基因功能注释和途径分析(图 5),对这些基因可能影响的细胞代谢途径进行探究。与原始菌株比,敲除菌中发生明显表达差异的基因数量情况如下:MIG1敲除菌有12个基因(上调表达的8个,下调表达的4个);SNF1敲除菌有25个(上调表达的23个,下调表达的2个);MIG1和SNF1双敲菌株有47个(上调表达的41个,下调表达的6个)。其中,MIG1敲除菌与MIG1和SNF1双敲除菌没有共享的表达上调基因,SNF1敲除菌与MIG1和SNF1双敲除菌共享20个表达上调基因,MIG1和SNF1双敲除菌特有21个表达上调基因。整体而言,MIG1和SNF1的敲除导致更多的基因表达发生上调,也就是说这些基因的转录可能是受MIG1或(和) SNF1直接或间接抑制的,敲除MIG1和SNF1则解除了这些抑制,从而使细胞更有利于共利用葡萄糖和木糖。
|
| 图 5 表达差异基因的功能和参与途径示意图 Figure 5 Gene function and pathway mapping of differentially expressed genes. (A) Venn diagram among differentially expressed genes in W32N55a (mig1Δ0), W32N55a (snf1Δ0) and W32N55a (mig1 & snf1Δ0) by comparing with wild type strain W32N55a. (B) Up-and down-regulated genes in W32N55a (mig1Δ0) compared with wild type strain W32N55a. (C) Overlapped up-regulated genes in W32N55a (snf1Δ0) and W32N55a (mig1 & snf1Δ0) compared with wild type strain W32N55a. (D) Unique up-and down-regulated genes in W32N55a (snf1Δ0) and W32N55a (mig1 & snf1Δ0) compared with wild type strain W32N55a. Red: up-regulated; Green: down-regulated. |
| |
通过进一步考察表达差异基因的功能注释和途径分析,可以更清晰地了解在混合糖条件下可能受MIG1和SNF1调控的基因和途径。MIG1敲除菌中上调和下调的基因功能如图 5B所示。上调的基因与细胞壁成分、色氨酸的降解、细胞核mRNA输出、葡萄糖转运、热激转录因子、蛋白折叠、精氨酸吸收以及甘氨酸代谢有关,这些基因可能直接或间接受MIG1的抑制。下调的基因与锌的转运、蛋白的合成和降解以及呼吸生长调控有关,这些基因可能是直接或间接受MIG1的激活。需要注意的是,编码低亲和力高转运性能的葡萄糖转运蛋白基因HXT1在MIG1敲除菌中表达上调。曾有报道指出[28],HXT1在葡萄糖浓度较低时被Rgt1阻遏,Mig1并不是HXT1的调控蛋白,另外,其他葡萄糖转运蛋白基因HXT2/HXT3/HXT4是由Mig1/Mig2和Rgt1共同阻遏调控。由于MIG1敲除菌中只有HXT1表达上调,而其他葡萄糖转运蛋白基因表达未发生明显变化,意味着这一结果可能是由非MIG1敲除的因素造成的。重新审视基因组测序分析的取样点(图 4A,9 h)时,发现MIG1敲除菌的残留葡萄糖浓度为10 g/L左右,明显高于原始菌株的3 g/L左右。因此,与原始菌株相比,高水平的残留葡萄糖浓度可能造成了MIG1敲除菌中HXT1表达的解抑制。另外,高水平的残留葡萄糖浓度还可能造成MIG1敲除对基因表达的影响被高水平残留葡萄糖的调控所掩盖,其具体情况尚待研究。但是从分析混合糖条件下影响木糖代谢变化相关的基因表达差异的角度,我们可以忽略葡萄糖残留浓度的影响,而认为所有样品都是在有葡萄糖的情况下进行比较。
SNF1敲除菌中明显上调表达的基因有23个,其中20个基因在MIG1和SNF1双敲除菌中表达也明显上调(图 5A),而在MIG1敲除菌中表达没有发生明显变化(图 5B)。这一比较结果说明这20个基因可能是受Snf1特异负调控,而不受Mig1的调控。也就是说,只要SNF1被敲除,这20个基因的表达就会被解抑制。非常有趣的是,这20个基因明显富集在与氨基酸(主要包括精氨酸、脯氨酸和丝氨酸)和其他氮源(尿囊素、尿素和氨)的吸收和降解相关的途径(图 5C),而这些基因是氮分解代谢物阻遏(NCR)基因,受氮源的营养水平调控[29-30]。精氨酸可以作为酵母偏好的氮源,在Can1、Car1和Car2以及Pro3的作用下产生脯氨酸;脯氨酸由Put4、Put1和Put2的作用下转化为谷氨酸,再由Gdh2作用降解为氨和α-酮戊二酸,α-酮戊二酸可以经由TCA循环进入碳中心代谢,而氨又可以进入氨基酸代谢。另外,丝氨酸在Agp1和Cha1的作用下降解为氨和丙酮酸,丙酮酸进入碳代谢途径进而生产乙醇。与原始菌株相比,SNF1敲除菌可以更快利用木糖可能与胞内更丰富的氮源有关。综上所述,氮分解代谢物阻遏基因表达上调,可能更有利于细胞吸收更多的氮源,从而使细胞积累更多的氨,更有利于细胞的基础代谢,比如蛋白质合成前体——氨基酸的积累;同时这些氨基酸和氮源降解的产物又与碳中心代谢途径相连,可能影响细胞的糖代谢,其分子机理尚待研究。
在MIG1和SNF1双敲除菌中发现的41个表达上调的基因,除了在SNF1敲除菌中也发现表达上调的20个基因外,还有21个基因。这21个基因在MIG1敲除菌中表达未发生明显变化,说明这21基因,可能是受Snf1和Mig1共同负调控的作用。也就是说,单独敲除MIG1和SNF1中的任何一个,都不会影响这21个基因的表达,当同时敲除这两个转录因子时,这21个基因的表达就会被解抑制。代谢途径分析没有发现明显的富集。不过,在Snf1调控功能缺失的基础上,MIG1的敲除会进一步导致参与氨基酸吸收和降解途径的个别基因表达上调,包括DIP5、BAP2、ARO9、ARO10、GAD1和ASP3-1 (图 5D)。另外,还包括磷酸戊糖途径中的SOL4和GND2以及参与线粒体丙酮酸合成的CYB2,其他上调基因还包括一些与膜结构蛋白相关的基因。以上结果暗示,在SNF1敲除的基础上,MIG1的敲除可能造成细胞对其他氨基酸氮源的偏好,同时影响到磷酸戊糖途径和线粒体丙酮酸代谢,从而造成细胞更倾向于快速利用葡萄糖,同时虽然木糖的利用速度降低,但并不会受到抑制,表明葡萄糖对木糖代谢的抑制作用部分得到缓解,而且最终导致代谢混合糖积累乙醇的速度变快。
2.4 Mig1和Snf1对差异表达基因的层级调控利用YEASTRACT可以分析可能参与表达差异基因调控的转录因子,从而有助于解析在混合糖条件下Mig1和Snf1如何调控氨基酸和氮源代谢进而影响葡萄糖和木糖的共利用(图 6)。结果发现SNF1敲除菌与MIG1和SNF1双敲除菌共有的20个表达上调基因均受Gln3调控(P-value小于0.000 1);另外,GAT1编码调控NCR基因表达的转录激活因子,其转录水平也上调。Gln3和Gat1是NCR基因表达必需的转录激活因子,并且Snf1位于基因表达层级的上游来调控Gln3的活性[31]。之前的诸多研究指出Snf1只在葡萄糖缺乏的条件下才起作用[9, 12]。然而Nicastro等最新的研究表明[32],Snf1可以在有葡萄糖存在的条件下起调控作用,SNF1的敲除会造成细胞吸收和代谢氨基酸的能力加强,而且这种现象在葡萄糖浓度低(2%)的条件下比高浓度(5%)时更为明显。也就是说,即便在葡萄糖存在的条件下,Snf1存在某种活性形式可以参与氨基酸和氮源代谢相关基因的抑制调控,这种相应的Snf1形式我们界定为Snf1in,在原始菌株中,混合糖条件下Snf1in可能抑制Gln3的活性,从而抑制这些NCR基因的表达。相应地,受葡萄糖代谢影响的Snf1形式我们界定为Snf1dp。

|
| 图 6 Mig1和Snf1对表达差异的基因不同层级的调控 Figure 6 Mig1 and Snf1 in a hierarchical regulatory network for regulating differentially expressed genes. Snf1in: Snf1 analog independent of glucose metabolism; Snf1in: Snf1 analog dependent of glucose metabolism. |
| |
另一方面,经过YEASTRACT分析,MIG1和SNF1双敲除菌特有的21个表达上调基因均受Gcn4的调控,Gcn4是参与调控氨基酸合成基因表达的转录激活因子。这21个基因的负调控可能是通过两条路径来实现,一条是经由不受葡萄糖代谢影响的Snf1in形式,在原始菌株中,混合糖条件下Snf1in会抑制Gcn4的活性,从而抑制这21个基因的表达;另一条是通过Mig1对这21个基因的抑制作用,Snf1dp是Mig1上游层级的负调控因子,在葡萄糖存在的条件下不具有活性。基于以上分析,MIG1敲除菌中,由于Snf1in的存在,会抑制Gln3和Gcn4的活性,因此受这两个转录因子激活的基因水平不会发生变化;SNF1敲除菌中,Gln3和Gcn4的活性得到解抑制,因此,受Gln3激活的20个基因表达上调,而受Gcn4激活的21个基因,由于依然受Mig1的抑制,其表达水平未发生明显变化;当在SNF1双敲除菌中继续敲除MIG1时,Mig1对这21个基因的抑制得到解除,从而基因表达发生上调。至于以上层级调控的具体细节以及Snf1in在葡萄糖存在条件下的形式尚待研究。
基于以上分析,在混合糖发酵条件下,一方面不受葡萄糖代谢影响的Snf1in具有活性,通过抑制转录激活因子Gln3和Gcn4的功能,分别经由Snf1in-Gln3和Snf1in-Gcn4途径抑制两部分参与氨基酸吸收和降解等功能基因的表达;另一方面,由于葡萄糖的存在,受葡萄糖代谢影响的Snf1dp处于非活性状态,不能够磷酸化失活Mig1,因此Mig1也可能抑制经由Snf1in-Gcn4途径所抑制的那部分基因的表达,其中包括磷酸戊糖途径氧化阶段的两个基因——SOL4 (6-磷酸葡萄糖酸内酯酶)和GND2 (编码6-磷酸葡萄糖酸脱氢酶) (图 6)。无论是单敲除SNF1还是双敲除SNF1和MIG1,与原始菌株相比,木糖的利用在3–9 h之间都没有明显地被延滞(图 4B),而且只要SNF1被敲除,经由Snf1in-Gln3途径抑制的基因表达发生上调,这表明经由Snf1in-Gln3途径抑制的这些参与氨基酸吸收和降解功能的基因(图 5、图 6),其表达的解抑制是实现葡萄糖和木糖的同步利用所必需的。与单敲除SNF1相比,SNF1和MIG1双敲除菌的葡萄糖代谢速率明显加快(图 4A,表 3),而木糖代谢速率大大降低(图 4B,表 3),与此同时,由于MIG1也被敲除,经由Snf1in-Gcn4途径抑制的基因表达发生上调(图 5、图 6),说明这些基因很可能参与细胞对葡萄糖和木糖代谢流的控制。另外,SNF1和MIG1双敲除能够加快乙醇的积累(图 4C,表 3),从而改善葡萄糖和木糖的共利用,这可能主要归功于葡萄糖代谢能力的大幅度加快。虽然曾有研究表明[7-8],磷酸戊糖途径相关基因表达上调有利于促进木糖代谢,但是在SNF1和MIG1双敲除菌中,表达上调的SOL4和GND2以及其他经由Snf1in-Gcn4途径抑制的基因所起的作用依然有待进一步研究。
3 结论以酿酒酵母工程菌株W32N55a为出发菌株,通过同源重组技术分别对葡萄糖阻遏效应中的关键基因SNF1和MIG1基因进行单敲除和双敲除,评价了敲除菌株共利用葡萄糖和木糖的性能。在低浓度混合糖条件下MIG1单敲除对混合糖的共利用影响不大,SNF1单敲除明显提高了木糖的利用效率,MIG1和SNF1双敲除明显促进葡萄糖的利用效率同时可以以较慢的速度共利用木糖,从而积累乙醇的效率得到提高;当混合糖浓度提高到10%时,MIG1和SNF1的单敲除效应变得不明显,MIG1和SNF1双敲除的效应依然很明显,但影响程度已经明显低于低浓度混合糖的情况。我们利用转录组测序技术分析了在低浓度混合糖条件下MIG1和SNF1单敲除和双敲除对全基因组水平上基因表达谱的影响。SNF1单敲除后,一些氮分解代谢物阻遏基因表达明显上调,这可能是导致SNF1单敲除菌利用木糖效率提高的主要原因。MIG1和SNF1双敲除菌除了上述SNF1单敲除对转录组产生的影响外,还会进一步导致参与氨基酸分解代谢相关的基因表达上调,可能是造成双敲除菌倾向于更快利用葡萄糖但同时可以共利用木糖的主要原因。我们的研究结果暗示了Snf1可能以依赖于葡萄糖代谢和不依赖于葡萄糖代谢这两种形式参与NCR基因的层级调控,这些研究发现将有助于进一步提高酿酒酵母工程菌共利用葡萄糖和木糖的效率。
| [1] | Somerville C, Bauer S, Brininstool G, et al. Toward a systems approach to understanding plant cell walls. Science, 2004, 306(5705): 2206–2211. DOI: 10.1126/science.1102765 |
| [2] | Chundawat SPS, Beckham GT, Himmel ME, et al. Deconstruction of lignocellulosic biomass to fuels and chemicals. Annu Rev Chem Biomol Eng, 2011, 2: 121–145. DOI: 10.1146/annurev-chembioeng-061010-114205 |
| [3] |
Yu JD, Cai JP, Geng XL, et al. Characteristics of mixed sugar fermentation by Candida maltosa.
Biotech World, 2013(1): 2–3.
(in Chinese). 于佳动, 蔡静平, 耿笑林, 等. 麦芽糖假丝酵母菌混合糖代谢的发酵特性. 生物技术世界, 2013(1): 2-3. |
| [4] | Kim SR, Ha SJ, Wei N, et al. Simultaneous co-fermentation of mixed sugars: a promising strategy for producing cellulosic ethanol. Trends Biotechnol, 2012, 30(5): 274–282. DOI: 10.1016/j.tibtech.2012.01.005 |
| [5] | Hsiao HY, Chiang LC, Ueng PP, et al. Sequential utilization of mixed monosaccharides by yeasts. Appl Environ Microbiol, 1982, 43(4): 840–845. |
| [6] | Sedlak M, Ho NWY. Characterization of the effectiveness of hexose transporters for transporting xylose during glucose and xylose co-fermentation by a recombinant Saccharomyces yeast. Yeast, 2004, 21(8): 671–684. DOI: 10.1002/(ISSN)1097-0061 |
| [7] | Wahlbom CF, Cordero Otero RR, van Zyl WH, et al. Molecular analysis of a Saccharomyces cerevisiae mutant with improved ability to utilize xylose shows enhanced expression of proteins involved in transport, initial xylose metabolism, and the pentose phosphate pathway. Appl Environ Microbiol, 2003, 69(2): 740–746. DOI: 10.1128/AEM.69.2.740-746.2003 |
| [8] | de Figueiredo Vilela L, de Araujo VPG, de Sousa Paredes R, et al. Enhanced xylose fermentation and ethanol production by engineered Saccharomyces cerevisiae strain. AMB Express, 2015, 5(1): 16. DOI: 10.1186/s13568-015-0102-y |
| [9] | Roca C, Haack MB, Olsson L. Engineering of carbon catabolite repression in recombinant xylose fermenting Saccharomyces cerevisiae. Appl Microbiol Biotechnol, 2004, 63(5): 578–583. DOI: 10.1007/s00253-003-1408-2 |
| [10] | Thanvanthri Gururajan V, Gorwa-Grauslund MF, Hahn-H gerdal B, et al. A constitutive catabolite repression mutant of a recombinant Saccharomyces cerevisiae strain improves xylose consumption during fermentation. Ann Microbiol, 2007, 57(1): 85–92. DOI: 10.1007/BF03175055 |
| [11] | Shen Y, Hou J, Bao XM. Enhanced xylose fermentation capacity related to an altered glucose sensing and repression network in a recombinant Saccharomyces cerevisiae. Bioengineered, 2013, 4(6): 435–437. DOI: 10.4161/bioe.25542 |
| [12] | Krahulec S, Petschacher B, Wallner M, et al. Fermentation of mixed glucose-xylose substrates by engineered strains of Saccharomyces cerevisiae: role of the coenzyme specificity of xylose reductase, and effect of glucose on xylose utilization. Microb Cell Fact, 2010, 9(1): 16. DOI: 10.1186/1475-2859-9-16 |
| [13] | Zaman S, Lippman SI, Zhao X, et al. How Saccharomyces responds to nutrients. Annu Rev Genet, 2008, 42: 27–81. DOI: 10.1146/annurev.genet.41.110306.130206 |
| [14] | Christensen TS, Oliveira AP, Nielsen J. Reconstruction and logical modeling of glucose repression signaling pathways in Saccharomyces cerevisiae. BMC Syst Biol, 2009, 3(1): 7. |
| [15] | R dk r SV, F rgeman NJ. Glucose-and nitrogen sensing and regulatory mechanisms in Saccharomyces cerevisiae. FEMS Yeast Res, 2014, 14(5): 683–696. DOI: 10.1111/fyr.2014.14.issue-5 |
| [16] | Conrad M, Schothorst J, Kankipati HN, et al. Nutrient sensing and signaling in the yeast Saccharomyces cerevisiae. FEMS Microbiol Rev, 2014, 38(2): 254–299. DOI: 10.1111/1574-6976.12065 |
| [17] | Klein CJL, Olsson L, Nielsen J. Glucose control in Saccharomyces cerevisiae: the role of MIG1 in metabolic functions. Microbiology, 1998, 144(1): 13–24. DOI: 10.1099/00221287-144-1-13 |
| [18] | Gancedo JM. Yeast carbon catabolite repression. Microbiol Mol Biol Rev, 1998, 62(2): 334–361. |
| [19] | García-Salcedo R, Lubitz T, Beltran G, et al. Glucose de-repression by yeast AMP-activated protein kinase SNF1 is controlled via at least two independent steps. FEBS J, 2014, 281(7): 1901–1917. DOI: 10.1111/febs.12753 |
| [20] | Simpson-Lavy KJ, Johnston M. SUMOylation regulates the SNF1 protein kinase. Proc Natl Acad Sci USA, 2013, 110(43): 17432–17437. DOI: 10.1073/pnas.1304839110 |
| [21] | De Vit MJ, Waddle JA, Johnston M. Regulated nuclear translocation of the Mig1 glucose repressor. Mol Biol Cell, 1997, 8(8): 1603–1618. DOI: 10.1091/mbc.8.8.1603 |
| [22] | Treitel MA, Kuchin S, Carlson M. Snf1 protein kinase regulates phosphorylation of the Mig1 repressor in Saccharomyces cerevisiae. Mol Cell Biol, 1998, 18(11): 6273–6280. DOI: 10.1128/MCB.18.11.6273 |
| [23] | Liao XS, Butow RA. RTG1 and RTG2: two yeast genes required for a novel path of communication from mitochondria to the nucleus. Cell, 1993, 72(1): 61–71. DOI: 10.1016/0092-8674(93)90050-Z |
| [24] | Vanderauwera S, Vandenbroucke K, Inzé A, et al. AtWRKY15 perturbation abolishes the mitochondrial stress response that steers osmotic stress tolerance in Arabidopsis. Proc Natl Acad Sci USA, 2012, 109(49): 20113–20118. DOI: 10.1073/pnas.1217516109 |
| [25] | Gietz RD, Schiestl TH. Large-scale high-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nat Protoc, 2007, 2(1): 38–41. DOI: 10.1038/nprot.2007.15 |
| [26] | Teixeira MC, Monteiro PT, Guerreiro JF, et al. The YEASTRACT database: an upgraded information system for the analysis of gene and genomic transcription regulation in Saccharomyces cerevisiae. Nucleic Acids Res, 2014, 42(D1): D161–D166. DOI: 10.1093/nar/gkt1015 |
| [27] | Klein CJL, Rasmussen JJ, Rønnow B, et al. Investigation of the impact of MIG1 and MIG2 on the physiology of Saccharomyces cerevisiae. J Biotechnol, 1999, 68(2/3): 197–212. |
| [28] | Kaniak A, Xue ZX, Macool D, et al. Regulatory network connecting two glucose signal transduction pathways in Saccharomyces cerevisiae. Eukaryotic Cell, 2004, 3(1): 221–231. DOI: 10.1128/EC.3.1.221-231.2004 |
| [29] | Ljungdahl PO, Daignan-Fornier B. Regulation of amino acid, nucleotide, and phosphate metabolism in Saccharomyces cerevisiae. Genetics, 2012, 190(3): 885–929. DOI: 10.1534/genetics.111.133306 |
| [30] | Hofman-Bang J. Nitrogen catabolite repression in Saccharomyces cerevisiae. Mol Biotechnol, 1999, 12(1): 35–74. DOI: 10.1385/MB:12:1 |
| [31] | Bertram PG, Choi JH, Carvalho J, et al. Convergence of TOR-nitrogen and Snf1-glucose signaling pathways onto Gln3. Mol Cell Biol, 2002, 22(4): 1246–1252. DOI: 10.1128/MCB.22.4.1246-1252.2002 |
| [32] | Nicastro R, Tripodi F, Guzzi C, et al. Enhanced amino acid utilization sustains growth of cells lacking Snf1/AMPK. Biochim Biophys Acta, 2015, 1853(7): 1615–1625. DOI: 10.1016/j.bbamcr.2015.03.014 |
 2018, Vol. 34
2018, Vol. 34