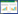中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘海燕
- Liu Haiyan
- 工业酶研究中的计算化学方法
- Computational chemistry approaches in studies on industrial enzymes
- 生物工程学报, 2019, 35(10): 1819-1828
- Chinese Journal of Biotechnology, 2019, 35(10): 1819-1828
- 10.13345/j.cjb.190293
-
文章历史
- Received: July 3, 2019
- Accepted: August 19, 2019
酶的工业应用已有百年历史,酶催化具有高效、高特异性和高选择性、环境友好等优点,在食品、农业、医药、化工等不同行业广泛应用[1-2]。由于工业应用环境远远不同于酶在自然界所处的生命环境,天然酶的性质、催化功能等和其应用环境通常不匹配,或者不是最优的。此时,需要借助酶工程对酶的天然氨基酸序列进行改造,以改进其性能[3]。最常用的酶工程策略是构建突变库进行筛选,即实验室定向进化[4]。有效的定向进化的必要前提之一是:相对于潜在有益突变体在库中所占的比例而言,接受筛选的突变库的库容(即库中包含的突变体数)要足够大。突变体库的库容常常受到筛选方法、可用资源等客观条件的限制。此时要解决的关键问题是如何提高突变库中有效突变体的占比。对酶序列、结构与重要特性的关系的深入了解,有助于发现突变热点,限定突变范围,实现优质突变库设计。计算化学方法是获取这方面认识的重要手段。有研究表明,相对于随机突变库,基于计算设计的蛋白质突变库可以将有效突变体占比提高多个数量级[5]。对一些困难的酶工程或蛋白质工程课题而言,计算可带来的大幅度改进可能足以决定课题的最终成败,而不再仅仅局限于效率的提升。实际上,计算化学、计算生物学方法已成功实现从头设计具有天然酶不具备的催化功能的人工酶。由于本专辑中已有其他综述专门讨论对氨基酸序列进行自动优化设计的方法,本文将主要介绍对给定氨基酸序列的酶进行模拟和分析的计算方法。当然,研究者可以用这些方法分别研究野生型和突变体,再对结果进行比较。
长期以来,对蛋白质特别是酶的研究一直是计算化学研究的重要前沿[6-8]。主要方法包括基于经典分子力学力场的分子动力学模拟(经典MD)[9]、量子力学(QM)[10]以及量子力学/分子力学(QM/MM)结合方法[8, 11-12]、分子间复合物预测即分子对接(Docking)[13]、定量分析静电和溶剂效应的可极化连续介质模型(PCM)如泊松-玻尔兹曼模型(PB)[14],以及一些基于几何性质的模型等。本文将从以下两个角度分别概要地介绍这些方法:一是关于方法本身,包括基本原理、原始计算结果、适用条件和(潜在)优缺点等;二是如何用这些方法获得工程相关信息,如更深入理解催化相关机制、对不同突变体相对于野生型的性质或功能变化进行理论预测或解释,从而指导定向进化所需的优质突变库设计,或者基于对原始计算结果的分析建议特定的突变位点和突变类型等。
1 基于经典分子力学力场(MM)的分子动力学模拟(MD) 1.1 方法简介我们暂不考虑酶催化所涉及的化学变化,仅考虑由于分子热运动引起的酶构象变化、酶与反应物(或产物)之间非共价复合物的形成与解离等过程。在这些过程中,分子的电子状态没有发生变化(例如没有共价键的断裂或生成),适用分子力学力场模型。所谓分子力学力场,是用经验的数学函数来表示分子体系势能对几何构型(即组成分子体系的所有原子的空间坐标)的依赖关系(图 1A)。换言之,如果我们用X代表所有原子的空间坐标,VMM (X)代表分子力场势能,则当分子从一种构象X1变到另一种构象X2后,其势能变化:

|
|
| 图 1 分子力学力场(A)和分子动力学模拟(B) Fig. 1 Molecular mechanics force field (A) and molecular dynamics simulation (B). |
| |
依据热力学理论,分子中的原子一直在进行热运动,即X在随时间不断变化;此外,当我们进行实验观察时,样品总是由大量分子组成(单分子实验例外),不同分子处于不同构象态。所以,从动力学角度,我们需要考虑构象随时间的变化;从热力学角度,我们需要考虑分子具有不同构象的概率分布。分子动力学模拟(MD)是考察这两个方面性质的最直接的模型(图 1B)。在进行MD模拟时,我们从某个初始构象出发,在每个时间点根据当前构象和势能函数计算出作用在每个原子上的力(力是势能函数对原子坐标的负导数),数值积分牛顿运动方程,得到下一个时间点的构象;重复该过程,即得到构象随时间演化的轨迹。
其间,可采用特殊算法模拟环境因素(如温度、压强等)对分子运动的影响。根据热力学原理,当时间间隔足够长,同一分子在不同时间点的构象与热力学平衡态下不同分子的构象这二者的概率分布是相同的(即时间平均等价于系综平均)。因此,如果MD模拟的时间足够长,模拟获得的构象集合可以作为在特定热力学平衡态下分子构象分布的样本。基于此原理,我们可以基于MD得到的时间轨迹分析体系热力学平衡态下的任意可观察性质。
MD提供了一种以原子分辨率全面解析构象变化动力学变化过程和重要微观量热力学分布的强大计算工具,这对阐释酶等复杂生物大分子机器的设计原理和工作机制尤为重要。由于目前大分子结构的实验解析方法还只能提供时空平均的静态结构,MD模拟在相关研究中有不可替代的功能。在此前提下,MD工具本身还在不断改进完善之中。在方法学上,MD的主要局限来源于两个方面:一是分子力场模型的精度问题;二是模拟时间有限难以实现对构象空间的充分采样问题。对于第一个问题,近年来分子力场已有较大改进,对生物大分子特别是蛋白质体系构象平衡的热力学描述准确度提高了,成功模拟了多种蛋白分子从头折叠到天然结构的过程[15-16]。在模拟时间方面,由于计算机软硬件发展,目前可以用常规计算硬件(如课题组用多核服务器)完成对通常大小的体系(如数百个残基的酶分子在水溶液中的体系)微秒量级的模拟。在此时间尺度下,可观察到结构域或环区开合等过程。如果可用计算资源更多,对底物结合/解离等过程的直接模拟也可以实现。如果要研究时间尺度在模拟可及范围之外的过程(如别构蛋白的大尺度功能变化等),可以采用增强采样方法[17],但使用者对MD理论要有较为深入的了解。
目前绝大部分MD模拟应用涵盖的时间尺度为纳秒到微秒,对构象空间的采样多被限制在初始结构附近(对单结构域蛋白而言,通常是均方根位移在3-4 Å 幅度内的结构涨落)。故而需要使用合理的初始结构作为MD的输入,模拟结果才有意义。多数情况下人们使用实验测定的晶体结构或者基于同源蛋白比较建模得到的结构作为MD的初始结构。在模拟酶和底物复合物时,常常需要根据空酶或酶与其他分子复合物的结构对复合物初始结构进行建模,方法包括使用分子对接或用底物直接替换晶体结构中的其他小分子(如抑制剂)。进行MD模拟前还需要构建能刻画体系中所有化学单元的分子力场。当被模拟的体系中包括底物小分子时,常会遇到MD软件包中提供的标准分子力场不覆盖该底物小分子的情况。此时可使用能自动生成小分子力场的工具软件[18-19]。在使用软件自动生成的力场进行长时间MD模拟之前,应人工核对力场文件并用其进行短时间模拟试算。
1.2 在酶研究中的应用从MD模拟得到的信息可以不同方式应用于指导酶工程改造[20]。例如,通过比较常温和高温MD模拟,可以预测酶分子哪些区域的结构稳定性对环境温度可能最为敏感。在这些区域引入脯氨酸点突变、二硫键等,有可能提升酶的耐热性[21-24]。提高稳定性的另一策略是设计能形成更多的表面氢键和盐键的突变体[25-26]。在实验验证这类突变体之前,可平行模拟野生型和突变体,理论上评估突变是否可能达到预期效果[27-28]。除温度外,MD还可以用于分析环境pH、溶剂等的变化对蛋白构象及其稳定性的影响[29-30]。
除稳定性外,MD还被应用于预测有可能显著影响与底物结合/产物释放相关的构象动力学的热点残基,为设计能改变底物选择性、反应选择性、产物释放速率等的突变或突变库提供依据[31-32]。用MD研究底物/反应选择性的方法之一是比较(初始结构)不同的酶-底物复合物的模拟结果,预测亲和力更高(或反应活性更高)的底物或结构状态。计算亲和力(或反应活性)的严格定量方法是自由能计算[33-34]。由于自由能计算计算量大,目前大多数应用中使用了定性方法预测:对相对亲和力的定性判别可以小分子-大分子复合物结构的稳定性、平均分子间相互作用能等为依据;对反应活性的定性判别依据则包括催化和反应官能团之间相对几何构型分布等[35]。这类定性判别结果可用作设计定向进化序列文库的依据。此外,MD模拟也可以用于分析底物结合/产物解离孔道周边的热点残基[36-37]。这类应用涉及对小分子从蛋白质解离的解离路径的模拟,如存在模拟时间尺度不足的困难,可使用增强采样技术来克服[38-39]。
2 量子力学(QM)以及量子力学/分子力学(QM/MM)结合模型 2.1 方法简介要模拟酶催化中的化学步骤,如共价键生成和断裂、电子转移、不同电子态之间的跃迁等,需要使用量子力学(QM)模型。目前计算化学中常用的QM模型分为从头算(ab initio)、密度泛函理论(DFT)、半经验方法等几种类型[40]。其中,半经验方法计算代价最低。但它们是非第一性原理方法,计算结果的可靠性对特定体系和问题的依赖性高。从头算和DFT方法都属于第一性原理方法,具普适性。实际DFT模型中可能比从头算包含了更多具经验性的理论近似,但DFT能以非常高的计算效率来处理电子相关能。此外,对很多化学反应问题,最好的DFT模型对反应程中能量变化等关键参数的计算误差已能小至约1 kcal/mol左右,其结果已经足以作为判别特定催化机制、反应路径化学上是否合理的依据。
给定分子的几何构型后,可用QM计算其能量。这被称为单点计算(即只处理几何构型空间中的一个点)。QM模型更多被用于分子几何构型的优化,即从一个初始构型出发经连续改变后找到一个局部稳定结构(能量比相邻结构低),也可以用于发现连接反应物和产物的最低能量路径和路径上的过渡态。这类计算中要考虑和比较不同的几何构型,一般要进行数十到上千次的单点计算,计算量较大。节省计算量的一种常用策略是先使用效率高、精度有限的QM模型进行大范围的反应路径搜索优化,在搜寻到的最低能量构型/路径附近再使用更高精度的模型完成构型优化,或进行单点计算。
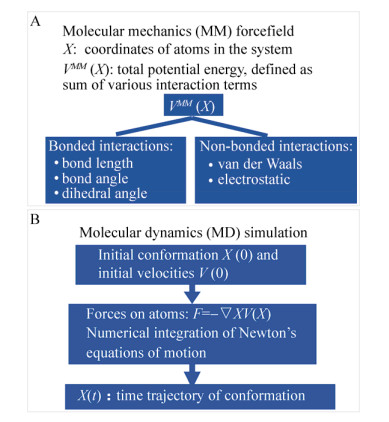
目前,将第一性原理QM方法应用于整个酶分子计算量大,基本仅限于单点计算,还缺乏实用性。对大分子常用的是QM/MM模型(图 2)[11]。在此模型中,分子体系被划分为至少两部分:直接参与化学反应的部分用QM模型处理,其余部分用分子力学(MM)处理。有多种不同的策略可以处理QM-MM间的边界和相互作用[41]。在第一性原理QM/MM模型中,QM计算花费的代价远超MM。因此,对QM区域大多使用构型优化的方法来预测或模拟其几何结构,对MM部分可使用分子动力学模拟采样[42]。这意味着计算结果可能对体系QM区的初始结构比较敏感。此时需要用不同初始结构模型进行计算以获得可靠结果。如果QM部分使用半经验方法[43]或经验价键理论[44-45],则可能通过较长时间的QM/MM MD采样更充分地探索构象空间,降低初始结构的影响。
|
| 图 2 量子力学(QM)/分子力学(MM)模型 Fig. 2 Quantum mechanical (QM)/molecular mechanical (MM) model. |
| |
QM模型[10]和QM/MM模型[41]都被广泛应用于酶催化反应化学机制的理论预测和检验。其结果可以帮助我们判别哪些关键残基参加了化学反应过程,找到反应的限速步骤,建立反应中间物和过渡态的结构模型,分析它们如何与酶环境相互作用等。相对于QM团簇模型,QM/MM模型能更真实地模拟化学反应中心所处的酶环境。QM/MM已被广泛应用于从理论上预测/检验酶催化的化学机制,分析预测环境氨基酸残基对催化过程的可能影响[46]。原则上,这些结果可用于指导以提升催化活力、改变特异性或选择性为目标的定向进化突变库设计。更有挑战性的研究,是基于从QM或QM/MM预测的过渡态结构模型从头设计新的活性中心,得到全新的人工酶[47]。
3 静电连续介质模型 3.1 方法原理酶催化几乎总是在特定溶液环境中完成的。溶剂效应对酶的性质有至关重要的影响。计算化学处理溶剂效应的模型分为两类:一类是显溶剂模型,例如,在分子力学力场或QM模型中,每个溶剂分子和其中的每个原子是被显式地包含在模型中的;另一类是隐溶剂模型或连续介质模型[48],模型中不包含溶剂分子和原子,而是用所谓的“溶剂平均场”来处理溶剂效应。显溶剂模型的优点是能够以完全一致的方式处理溶质和溶剂,真实地模拟溶质-溶剂间氢键、盐键等特异性相互作用。其缺点是溶剂分子数目多,耗费的计算量大。此外,溶剂随机涨落对体系总能量的贡献很大,必须进行长时间的模拟采样平均才能消除涨落的影响。隐溶剂模型刻画的是溶剂的平均效应,溶剂的热力学涨落已经被平均了。
为简化处理,在隐溶剂模型中我们通常把非极性溶剂效应(疏水效应)与极性溶剂效应分开。经验表明,非极性溶质的溶剂化自由能与其溶剂可接近表面积(SASA)成正比。因此,对这部分常使用SASA溶剂化模型。该模型中的参数包括计算SASA所需的原子半径、溶剂分子半径(水分子为1.4 Å ),以及溶剂化自由能正比于SASA的比例常数。这些参数一般是通过拟合小分子溶剂化自由能的实验值来确定的。
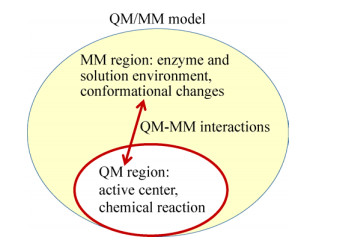
考虑极性溶剂效应最常用的模型把被溶剂占据的区域视为具有特定介电常数(水为78.4)的连续介质,溶质区域则视为被低介电常数(常用值为2-8)或真空(介电常数为1)介质占据(图 3A)。连续介质会被溶质区的电荷分布所产生的静电场极化,由此产生的极化电荷分布又会在溶质区产生静电场,作用于溶质电荷上。极化电荷产生的电场被称作反应场。因此,静电连续介质模型又被称为反应场模型。在溶剂区中无游离离子的连续介质模型中,空间静电势与空间电荷分布之间的关系满足泊松方程。对包含游离离子的溶液环境而言,离子的空间分布会受到空间静电势的影响。考虑这一因素,空间静电势与空间电荷分布之间的关系满足泊松-玻尔兹曼方程(PB方程)。PB方程是关于三维空间中静电势分布与电荷和电介质分布之间关系的偏微分方程,可以用数值方法求解。求解酶等大分子体系PB方程的最常用数值方法是有限差分法(FD),合称为FDPB模型(图 3B)[14]。用FDPB可以基于溶质的空间电荷分布计算三维空间的静电势,进而计算静电自由能等其他性质。在小分子体系的QM计算中,反应场往往用分子表面的面电荷分布产生的电场来等效替代,相应模型称为可极化连续介质(PCM)模型。
|
| 图 3 连续介质静电模型(A)和有限差分泊松-玻尔兹曼(FDPB)方法(B) Fig. 3 Electrostatic continuum model (A) and the finite difference Poisson-Boltzmann (FDPB) method (B). |
| |
MM/PBSA模型[49]是一种把显溶剂模型和隐溶剂模型结合起来的方法:用显溶剂分子力场模型进行分子动力学模拟,对溶质构象空间进行抽样;再对MD得到的溶质构象用隐溶剂模型进行溶剂化自由能计算,用溶质的分子力场能量(MM)和溶剂化自由能(PBSA)之和来评估溶质构象的稳定性。该方法的好处是:用PBSA代替MM的溶剂部分,消除溶剂涨落;对显溶剂MD得到的溶质构象集合进行PBSA计算,避免PBSA结果对单一溶质构象的依赖性。
3.2 方法应用连续介质模型的重要应用之一是研究酶分子中带电氨基酸侧链基团的质子化状态。软件PROPKA通过求解PB方程计算不同质子化状态的静电自由能来预测各可解离基团的pKa[50]。酶分子的表面静电势分布是影响酶的底物选择性的重要因素。给定酶分子的空间结构和质子化状态,用FDPB方法可以计算酶分子的表面静电势分布,也可以预测氨基酸残基突变或环境pH改变、离子浓度变化等对表面静电势的影响[14]。
在用QM簇模型研究酶催化的化学步骤时,常常需要使用PCM模型来模拟环境对反应区的静电影响。如果反应过程涉及电荷分布的重大变化,不使用连续介质的真空QM计算结果是不合理的,甚至可能导致错误的定性结论。在QM/MM模型中,反应中心周围一般包含用MM方式处理的显溶剂分子,一般不需要考虑连续介质反应场。但如果反应前后体系的净电荷发生变化(如氧化还原电势计算),则很可能需要考虑体系边界以外的溶液环境对反应自由能的贡献,此时可使用连续介质模型对QM/MM结果进行修正。
作为一种兼顾效率和精度的方法,MM/PBSA可以用于分析蛋白质-蛋白质、蛋白质-小分子复合物的亲和力[49]。为实现误差抵消,常规的做法是:对复合物进行显溶剂的分子动力学模拟,得到构象集合;对每一个复合物构象,分别计算复合物整体、组成复合物的每个单体的MM/PBSA能量;用整体的MM/PBSA能量与单体MM/PBSA能量之差对全部构象的平均值来近似估计结合自由能。该方法可用于分析影响底物亲和力的热点残基,也可以用于预测突变体的底物选择性变化。
4 其他方法 4.1 分子对接分子对接(Docking)泛指基于单体的结构预测复合物结构(和亲和力)的计算过程。小分子与蛋白质对接是基于结构进行药物虚拟筛选的核心工具,为此已开发了多种算法[13]。这些算法和模型同样可应用于底物-酶复合物的对接。药物虚拟筛选需要考虑大量不同小分子,出于计算效率考虑,分子对接计算中往往不考虑受体的结构变化(或仅仅考虑侧链的结构变化)。与虚拟筛选不同,在底物-酶对接研究中往往只需要考虑一种或数种不同底物,原则上可以更充分考虑酶的结构变化。实现这一点的最直接方法是通过MD等构象采样方法得到多样的酶结构,分别与底物进行对接。在底物-酶对接中,往往还可以利用底物与催化官能团的相对空间排布来筛选/评价对接结果。
4.2 基于几何结构预测小分子孔道大量实验研究发现,一些远离活性中心的突变对酶的催化性能可产生很大影响。其中一些位点可能是通过改变底物结合/产物释放孔道发挥作用,孔道大小、孔道周边残基的物理化学性质等可以改变底物/产物的通过速率,影响底物选择性等。用孔道预测的方法可以找到相关的热点残基,为定向进化库设计提供依据。目前有数种基于几何结构的方法可以预测蛋白质表面凹坑、内部空腔、连接不同区域的孔道等[51-53]。这些方法使用静态空间结构作为输入,多采用几何学和图论方法实现预测,计算效率高。
4.3 活性中心比较方法目前,蛋白质三维结构数据库(PDB)中已积累了大量不同结构类型、不同家族的酶的三维结构数据。如果我们比较不同的酶,会发现其中一些尽管整体结构序列不相似,但活性中心相似程度较高(典型例子如丝氨酸蛋白酶共有的催化三联体活性中心)。活性中心结构比较方法[54-55]能够用于自动检索与当前酶活性中心相似的其他酶的活性中心。把多个相似的活性中心在三维空间中叠合到一起,分析不同活性中心之间的异同能够为突变位点选择提供有价值的信息。
5 总结为表述清晰,我们对上述方法的介绍是分类进行的。在实际应用中,不同类型的方法互相并不排斥。它们可以多种方式结合起来使用,以更好地回答我们所关心的问题。例如,在酶-底物复合物模拟中,分子对接可以用来获得模拟的初始构象;MD模拟得到的构象集合,可以用于孔道预测分析、分子对接、QM/MM模拟等;可以建立刻画QM或QM/MM模型得到的过渡态的MM模型,用于长时间的经典MD模拟,以分析构象涨落对化学过程的影响,或用于模拟大量突变体,实现基于MD模拟的突变体虚拟筛选;我们已经提到的MM/PBSA方法,是MD与连续介质模型的结合等。
用计算化学方法对蛋白质等生物大分子体系的研究已有40多年的历史。这些方法在自身不断发展的同时,在工业酶研究中也越来越广泛地得到使用。我国在计算化学与工业酶工程这两个学科方向的研究队伍均日益扩大,研究能力快速提升。随着这两个学科方向的交叉结合日渐紧密,计算化学在酶工程中的应用将会不断拓宽和深入。蛋白质工程、定向进化等技术曾经对工业酶研究产生了巨大的影响。计算方法的未来发展,特别是新酶设计方法的突破,将有望为进入合成生物学时代的工业酶研究带来新的技术突破。
| [1] |
Choi JM, Han SS, Kim HS. Industrial applications of enzyme biocatalysis: current status and future aspects. Biotechnol Adv, 2015, 33(7): 1443-1454. DOI:10.1016/j.biotechadv.2015.02.014 |
| [2] |
Singh R, Kumar M, Mittal A, et al. Microbial enzymes: industrial progress in 21st century. 3 Biotech, 2016, 6(2): 174. DOI:10.1007/s13205-016-0485-8 |
| [3] |
Rastetter WH. Enzyme engineering. Appl Biochem Biotechnol, 1983, 8(5): 423-436. DOI:10.1007/BF02779915 |
| [4] |
Zeymer C, Hilvert D. Directed evolution of protein catalysts. Annu Rev Biochem, 2018, 87: 131-157. DOI:10.1146/annurev-biochem-062917-012034 |
| [5] |
Sun MG, Seo MH, Nim S, et al. Protein engineering by highly parallel screening of computationally designed variants. Sci Adv, 2016, 2(7): e160069. |
| [6] |
Garcia-Viloca M, Gao JL, Karplus M, et al. How enzymes work: analysis by modern rate theory and computer simulations. Science, 2004, 303(5655): 186-195. DOI:10.1126/science.1088172 |
| [7] |
Caddell Haatveit K, Garcia-Borràs M, Houk KN. Computational protocol to understand P450 mechanisms and design of efficient and selective biocatalysts. Front Chem, 2018, 6: 663. |
| [8] |
Hu H, Yang WT. Free energies of chemical reactions in solution and in enzymes with ab initio quantum mechanics/molecular mechanics methods. Annu Rev Phys Chem, 2008, 59: 573-601. DOI:10.1146/annurev.physchem.59.032607.093618 |
| [9] |
Karplus M. Molecular dynamics simulations of biomolecules. Acc Chem Res, 2002, 35(6): 321-323. DOI:10.1021/ar020082r |
| [10] |
Siegbahn PE, Himo F. Recent developments of the quantum chemical cluster approach for modeling enzyme reactions. J Biol Inorg Chem, 2009, 14(5): 643-651. DOI:10.1007/s00775-009-0511-y |
| [11] |
Warshel A, Levitt M. Theoretical studies of enzymic reactions: dielectric, electrostatic and steric stabilization of the carbonium ion in the reaction of lysozyme. J Mol Biol, 1976, 103(2): 227-249. DOI:10.1016/0022-2836(76)90311-9 |
| [12] |
Gao JL, Truhlar DG. Quantum mechanical methods for enzyme kinetics. Annu Rev Phys Chem, 2002, 53: 467-505. DOI:10.1146/annurev.physchem.53.091301.150114 |
| [13] |
Warren GL, Andrews CW, Capelli AM, et al. A critical assessment of docking programs and scoring functions. J Med Chem, 2006, 49(20): 5912-5931. DOI:10.1021/jm050362n |
| [14] |
Gilson MK, Honig BH. Energetics of charge-charge interactions in proteins. Prot-Struct Funct Genet, 1988, 3(1): 32-52. DOI:10.1002/prot.340030104 |
| [15] |
Robustelli P, Piana S, Shaw DE. Developing a molecular dynamics force field for both folded and disordered protein states. Proc Natl Acad Sci USA, 2018, 115(21): E4758-E4766. DOI:10.1073/pnas.1800690115 |
| [16] |
Shaw DE, Maragakis P, Lindorff-Larsen K, et al. Atomic-level characterization of the structural dynamics of proteins. Science, 2010, 330(6002): 341-346. DOI:10.1126/science.1187409 |
| [17] |
Bernardi RC, Melo MCR, Schulten K. Enhanced sampling techniques in molecular dynamics simulations of biological systems. Biochim Biophys Acta, 2015, 1850(5): 872-877. DOI:10.1016/j.bbagen.2014.10.019 |
| [18] |
Malde AK, Zuo L, Breeze M, et al. An automated force field topology builder (ATB) and repository: version 1.0. J Chem Theory Comput, 2011, 7(12): 4026-4037. DOI:10.1021/ct200196m |
| [19] |
Vanommeslaeghe K, MacKerell AD Jr. Automation of the CHARMM General Force Field (CGenFF) Ⅰ: bond perception and atom typing. J Chem Inf Model, 2012, 52(12): 3144-3154. DOI:10.1021/ci300363c |
| [20] |
Childers MC, Daggett V. Insights from molecular dynamics simulations for computational protein design. Mol Syst Des Eng, 2017, 2(1): 9-33. |
| [21] |
Joo JC, Pack SP, Kim YH, et al. Thermostabilization of Bacillus circulans xylanase: computational optimization of unstable residues based on thermal fluctuation analysis. J Biotechnol, 2011, 151(1): 56-65. DOI:10.1016/j.jbiotec.2010.10.002 |
| [22] |
Watanabe M, Matsuzawa T, Yaoi K. Rational protein design for thermostabilization of glycoside hydrolases based on structural analysis. Appl Microbiol Biotechnol, 2018, 102(20): 8677-8684. DOI:10.1007/s00253-018-9288-7 |
| [23] |
Yu HR, Huang H. Engineering proteins for thermostability through rigidifying flexible sites. Biotechnol Adv, 2014, 32(2): 308-315. DOI:10.1016/j.biotechadv.2013.10.012 |
| [24] |
Pikkemaat MG, Linssen ABM, Berendsen HJC, et al. Molecular dynamics simulations as a tool for improving protein stability. Protein Eng, 2002, 15(3): 185-192. DOI:10.1093/protein/15.3.185 |
| [25] |
Perl D, Mueller U, Heinemann U, et al. Two exposed amino acid residues confer thermostability on a cold shock protein. Nat Struct Biol, 2000, 7(5): 380-383. DOI:10.1038/75151 |
| [26] |
Ladenstein R, Antranikian G. Proteins from hyperthermophiles: stability and enzymatic catalysis close to the boiling point of water//Antranikian G, Ed. Biotechnology of Extremophiles. Berlin, Heidelberg: Springer, 1998, 61: 37-85.
|
| [27] |
Sakuraba S, Kono H. Spotting the difference in molecular dynamics simulations of biomolecules. J Chem Phys, 2016, 145(7): 074116. DOI:10.1063/1.4961227 |
| [28] |
Tang L, Liu HY. A comparative molecular dynamics study of thermophilic and mesophilic ribonuclease HI enzymes. J Biomol Struct Dyn, 2007, 24(4): 379-392. |
| [29] |
Suplatov D, Panin N, Kirilin E, et al. Computational design of a pH stable enzyme: understanding molecular mechanism of penicillin acylase's adaptation to alkaline conditions. PLoS ONE, 2014, 9(6): e100643. DOI:10.1371/journal.pone.0100643 |
| [30] |
Gu W, Wang TT, Zhu J, et al. Molecular dynamics simulation of the unfolding of the human prion protein domain under low pH and high temperature conditions. Biophys Chem, 2003, 104(1): 79-94. |
| [31] |
Ying HX, Wang J, Shi T, et al. Engineering of lysine cyclodeaminase conformational dynamics for relieving substrate and product inhibitions in the biosynthesis of L-pipecolic acid. Catal Sci Technol, 2019, 9(2): 398-405. DOI:10.1039/C8CY02301H |
| [32] |
Dodani SC, Kiss G, Cahn JKB, et al. Discovery of a regioselectivity switch in nitrating P450s guided by molecular dynamics simulations and Markov models. Nat Chem, 2016, 8(5): 419-425. DOI:10.1038/nchem.2474 |
| [33] |
Mikulskis P, Genheden S, Ryde U. A large-scale test of free-energy simulation estimates of protein-ligand binding affinities. J Chem Inf Model, 2014, 54(10): 2794-2806. DOI:10.1021/ci5004027 |
| [34] |
Liu HY, Mark AE, van Gunsteren WF. Estimating the relative free energy of different molecular states with respect to a single reference state. J Phys Chem, 1996, 100(22): 9485-9494. DOI:10.1021/jp9605212 |
| [35] |
Petrovic D, Bokel A, Allan M, et al. Simulation-guided design of cytochrome P450 for chemo- and regioselective macrocyclic oxidation. J Chem Inf Model, 2018, 58(4): 848-858. DOI:10.1021/acs.jcim.8b00043 |
| [36] |
Kingsley LJ, Lill MA. Substrate tunnels in enzymes: structure-function relationships and computational methodology. Proteins, 2015, 83(4): 599-611. |
| [37] |
Li GY, Yao PY, Gong R, et al. Simultaneous engineering of an enzyme's entrance tunnel and active site: the case of monoamine oxidase MAO-N. Chem Sci, 2017, 8(5): 4093-4099. DOI:10.1039/C6SC05381E |
| [38] |
Hu Y, Liu HY. Case study on temperature-accelerated molecular dynamics simulation of ligand dissociation: inducer dissociation from the Lac repressor protein. J Phys Chem A, 2014, 118(39): 9272-9279. DOI:10.1021/jp503856h |
| [39] |
Lüdemann SK, Lounnas V, Wade RC. How do substrates enter and products exit the buried active site of cytochrome P450cam? 1. Random expulsion molecular dynamics investigation of ligand access channels and mechanisms. J Mol Biol, 2000, 303(5): 797-811. DOI:10.1006/jmbi.2000.4154 |
| [40] |
McDouall JJW. Computational Quantum Chemistry—Molecular Structure and Properties in silico. Cambridge, UK: RSC Publishing, 2013.
|
| [41] |
Senn HM, Thiel W. QM/MM methods for biomolecular systems. Angew Chem Int Ed, 2009, 48(7): 1198-1229. DOI:10.1002/anie.200802019 |
| [42] |
Zhang YK, Liu HY, Yang WT. Free energy calculation on enzyme reactions with an efficient iterative procedure to determine minimum energy paths on a combined ab initio QM/MM potential energy surface. J Chem Phys, 2000, 112(8): 3483-3492. DOI:10.1063/1.480503 |
| [43] |
Liu HY, Müller-Plathe F, van Gunsteren WF. A combined quantum/classical molecular dynamics study of the catalytic mechanism of HIV protease. J Mol Biol, 1996, 261(3): 454-469. DOI:10.1006/jmbi.1996.0476 |
| [44] |
Hwang JK, King G, Creighton S, et al. Simulation of free energy relationships and dynamics of SN2 reactions in aqueous solution. J Am Chem Soc, 1988, 110(16): 5297-5311. DOI:10.1021/ja00224a011 |
| [45] |
Amrein BA, Steffen-Munsberg F, Szeler I, et al. CADEE: computer-aided directed evolution of enzymes. IUCrJ, 2017, 4(1): 50-64. DOI:10.1107/S2052252516018017 |
| [46] |
Liu HY, Zhang YK, Yang WT. How is the active site of enolase organized to catalyze two different reaction steps?. J Am Chem Soc, 2000, 122(28): 6560-6570. DOI:10.1021/ja9936619 |
| [47] |
Kiss G, Çelebi-Ölçüm N, Moretti R, et al. Computational enzyme design. Angew Chem Int Ed, 2013, 52(22): 5700-5725. DOI:10.1002/anie.201204077 |
| [48] |
Roux B, Simonson T. Implicit solvent models. Biophys Chem, 1999, 78(1/2): 1-20. |
| [49] |
Genheden S, Ryde U. The MM/PBSA and MM/GBSA methods to estimate ligand-binding affinities. Expert Opin Drug Discov, 2015, 10(5): 449-461. DOI:10.1517/17460441.2015.1032936 |
| [50] |
Olsson MH, Sondergaard CR, Rostkowski M, et al. PROPKA3: consistent treatment of internal and surface residues in empirical pKa predictions. J Chem Theory Comput, 2011, 7(2): 525-537. DOI:10.1021/ct100578z |
| [51] |
Petřek M, Košinová P, Koča J, et al. MOLE: a voronoi diagram-based explorer of molecular channels, pores, and tunnels. Structure, 2007, 15(11): 1357-1363. DOI:10.1016/j.str.2007.10.007 |
| [52] |
Yaffe E, Fishelovitch D, Wolfson HJ, et al. MolAxis: efficient and accurate identification of channels in macromolecules. Proteins, 2008, 73(1): 72-86. |
| [53] |
Chovancova E, Pavelka A, Benes P, et al. CAVER 3.0: a tool for the analysis of transport pathways in dynamic protein structures. PLoS Comput Biol, 2012, 8(10): e1002708. DOI:10.1371/journal.pcbi.1002708 |
| [54] |
Konc J, Miller BT, Stular T, et al. ProBiS-CHARMMing: web interface for prediction and optimization of ligands in protein binding sites. J Chem Inf Model, 2015, 55(11): 2308-2314. DOI:10.1021/acs.jcim.5b00534 |
| [55] |
Ren JY, Xie L, Li WW, et al. SMAP-WS: a parallel web service for structural proteome-wide ligand-binding site comparison. Nucleic Acids Res, 2010, 38(Web Server issue): W441-W444. |
 2019, Vol. 35
2019, Vol. 35