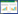中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈琦, 李春秀, 郑高伟, 郁惠蕾, 许建和
- Chen Qi, Li Chunxiu, Zheng Gaowei, Yu Huilei, Xu Jianhe
- 工业蛋白质构效关系的计算生物学解析
- Computational analysis of structure-activity relationship of industrial enzymes
- 生物工程学报, 2019, 35(10): 1829-1842
- Chinese Journal of Biotechnology, 2019, 35(10): 1829-1842
- 10.13345/j.cjb.190329
-
文章历史
- Received: July 23, 2019
- Accepted: September 16, 2019
酶催化为大宗化学品、精细化学品和新型材料等产品提供了一种可持续的绿色合成策略,已经成为现代生物制造产业的核心“芯片”,也是国际知识产权争夺的焦点。天然环境中获得的酶催化性能一般较差,难以满足工业应用对其催化活力、稳定性、溶剂耐受性、底物谱等各个方面的性能要求,需要进行进一步分子改造。因此,不断设计和研发新型、高效的酶催化剂是发展工业生物技术的关键所在。
工业催化剂创新设计的科学基础是对酶与底物的相互作用、结构与功能关系及其调控机制的深入剖析。对蛋白质特定区域或位点特征的计算研究主要集中在:活性位点的构象、动态变化规律、突变效应和反应环境的影响;底物结合和产物释放;底物传递;辅因子结合和释放;催化机制和过渡态途径等。相关的计算手段主要有分子动力学模拟(Molecular dynamics simulation, MD)[1]、量子力学模拟(Quantum mechanics, QM)、混合量子力学和分子力学(Quantum mechanics/molecular mechanics, QM/MM)模拟等[2]。
分子动力学模拟以经典牛顿力学为理论基础,模拟获取体系内所有原子随时间运动的轨迹,通过分析轨迹文件中整个体系的结构或原子之间相互作用变化,来解析整个体系的动力学性质。通过分子动力学模拟或其他采样方法分析酶的构象灵活性,可以阐明活性位点和突变位点对酶催化活性的调控作用。混合量子力学和分子力学用量子力学方法计算生物大分子活性部位,用分子力学、分子动力学或自由能微扰理论计算体系中其他原子,这样就可得到生物大分子活性部位的电子结构,通过能量分析,研究底物的结合模式及酶的催化机理等性质[3]。QM/MM能够在计算能量分布和能垒、获得催化机理信息、反应途径、反应速率等方面提供准确性更高的解析。
除了常规的计算方法,研究者不断开发其他或有针对性的计算策略深入研究蛋白质的结构功能关系。结合计算模拟和实验分析,不仅可以揭示酶分子催化反应背后的结构机理,同时可以指导酶的分子改造,促进其工业应用[4-5]。Baker课题组开发了RosettaDesign和RosettaReModel工具来进行蛋白质的结构和灵活性设计[6]。JANUS通过分析多序列比对的结果来预测结构相关但功能不同的酶改造所需的突变[7]。基于Web服务器的HotSpot Wizard可以用于自动识别酶结构中的“热点”氨基酸,指导针对底物特异性、催化活力或对映选择性的设计改造,以及蛋白质结构的注释[8]。POCKETOPTIMIZER针对活性口袋进行设计,可用于对酶的活性位点残基进行替换,提高其对小分子底物的结合能力[9]。CAVER和POVME2可用于分析通过MD模拟所获得的轨迹中底物口袋和通道的形态和大小,有针对性地指导活性口袋和底物通道的结构改造[10-11]。荷兰Janssen课题组开发的CASCO工具可以用于突变体的设计,通过高通量的MD模拟对设计的突变进行排序,以指导酶的立体选择性改造实验[12]。FRESCO方法通过预测折叠自由能、增加二硫键、剔除不合理突变等步骤来设计小巧型突变体文库,理性指导酶的热稳定性改造[13]。
1 基于计算生物学的酶分子理性设计 1.1 解析酶的催化机理多尺度的分子动力学模拟和QM/MM模拟已被广泛地用于多种类型的酶分子催化反应机理的解析[14-18]。
针对二氢叶酸还原酶ecDHFR催化循环中的关键问题,即Met20环在化学氢化物转移步骤中是否采用闭合构象,Major等使用QM/MM模拟研究了野生和突变型ecDHFR酶的Met20环在反应中的构象。研究表明,尽管Met20环的构象对氢化物转移速率和供体-受体距离的相关性不明显,但它对活性位点水合的有序性具有十分显著的影响[19]。刘永军课题组利用QM/MM探索来源于热带假丝酵母的烯酰基硫酯还原酶Etr1p催化烯酰硫酯为酰基硫酯的催化机制。计算结果表明稳定的共价烯加合物的形成遵循迈克尔加成机理。同时,结晶水分子虽然不参与催化反应,但通过形成氢键网络在氢化物转移和质子转移过程中起关键作用[20]。Loll等解析了典型的二萜合酶CotB2催化反应的化学机制。随着阳离子沿着反应路径的变化,CotB2活性中的三维结构也随之改变,作者确定了活性中心通用的结构特征,可用于其他来源的萜烯合酶生成大环化合物的理性设计[21]。Leys获得了合成单萜的芳樟醇合成酶和1, 8-桉树脑合成酶与氟化底物类似物的复合物晶体结构。模拟表明这些单萜合酶在反应中不会发生显著的构象变化,这样有助于利用基于结构的蛋白质工程手段,将这些酶的催化范围扩展到其他单萜底物[22]。
细胞色素P450酶的底物谱非常广泛,Houk课题组综合使用DFT计算和MD模拟分析酶分子的构象变化和结合机制,解析酶催化机制和过渡态[23]。另外,P450过氧化酶OleTJE使用H2O2作为氧化剂,催化脂肪酸转化为烯烃而无需额外的电子传递。Faponle等同样综合利用DFT和QM/MM研究,针对OLeTJE进行蛋白质工程改造以提高产品的选择性[24]。德国Reetz课题组获得细胞色素P450-BM3单加氧酶可以有效生物催化生成对苯二酚。计算研究证实P450-BM3突变体对苯酚选择性氧化,导致在环氧化物中间体C3=C4双键上产生区域选择性[25]。
Moliner等开展了关于甘氨酸N-甲基转移酶和3种突变体催化SN2反应实现甲基转移的QM / MM理论研究,验证实验观察到的速率恒定还原和反向次级α-3H动力学同位素效应的变化是否应该归因于甲基供体与受体间距离的变化,计算获得的活化自由能和实验数据非常一致[26]。同样的,Damborsky等对卤代烷脱卤素酶DhaA31催化水解1, 2, 3-三氯丙烷所涉及的主要步骤进行了全面的计算研究。研究发现底物从通道进入活性中心并结合是一个相对快速的过程,碳-卤键的裂解为反应中的限速步骤。突变体中可催化的构象数目增加且SN2的反应能垒降低,使得突变体中限速步骤的效率显著增强。最终通过对DhaA31的突变实验和对中间突变体的动力学分析证实了理论观察的结果[27]。
分子氧的活化机制是金属酶研究中的核心问题,Shaik课题组针对非血红素羟乙基膦酸酯双加氧酶HEPD和甲基膦酸合酶MPnS这两种特殊C-H加氧酶进行QM/MM模拟研究,结果显示MPnS中的O2活化是由活性位点中的相邻赖氨酸Lys28介导的,证实了在金属酶的氧活化机制中存在不寻常的质子穿梭机制[28]。Faber等解析了金属依赖型异乳清酸脱羧酶IDCase催化N-芳香杂环化合物区域选择性羧化的反应机制和底物特异性机理。结果表明天然底物5-羧基尿嘧啶的脱羧是通过亲电芳香取代反应进行的,且杂环底物与周围的氨基酸残基形成独特的氢键作用,使得IDCase显示出较高的底物特异性[29]。
1.2 提高酶的催化效率基于催化反应机理的计算解析的催化机理,可以全面揭示蛋白质的结构-功能关系,并针对性地对蛋白质结合口袋和活性位点进行理性设计改造。例如,大位阻的底物对结合位点的构象动力学具有更高的依赖性,通过突变可以调节结合口袋的灵活性(如结合口袋的体积),以形成更适合特定底物识别和稳定的构象状态[30-31]。
通过在活性位点附近的位置引入单突变,孔旭东等将来源于巨大芽孢杆菌的环氧水解酶BmEH底物范围扩大到更具空间要求的环氧化物底物[32]。英国Turner课题组对来自黑曲霉的单胺氧化酶MAO-N野生酶进行研究,设计获得了一系列突变体能够催化各种大位阻的手性胺底物。这些突变位点不仅位于酶的疏水口袋,其中一部分还位于远离酶催化活性中心的远端位置[33-34]。Klinman课题组通过计算手段理性推测得到大豆脂氧合酶SLO-1从溶液到酶活性位点的氧分子通道,结合定点突变方法,最终确定氧通道中两个关键氨基酸的调控机制,并计算出相应突变体的氧分子通道模型[35]。Shin等开展(S)-构型选择性转氨酶OATA的晶体结构及计算模拟分析,从分子机制角度解释了酶催化的反应机理,所获得的双点突变体对3种酮底物的催化活力均有显著提高[36]。Kaila等对FeII/α-酮戊二酸依赖性的双加氧酶AsqJ催化喹诺酮生物碱的多步合成进行研究。发现底物的甲基化对活性位点内π-堆积相互作用进行微调,对AsqJ的催化反应必不可少。为了对AsqJ进行理性设计,作者扩大了活性位点内的相互作用,最终获得的工程酶对非甲基化替代物具有显著增强的催化活性[37]。根据对底物结合模式的分析,Mattevi对5-羟甲基糠醛氧化酶HMFO氧化5-甲酰基-2-呋喃甲酸生成2, 5-呋喃二甲酸进行理性设计改造,两个关键突变增加底物结合亲和力并促进酶与底物醛基以正确结合姿势结合[38]。类似的策略同样被用于设计改造禾谷镰刀菌壳寡糖氧化酶的底物谱,使其对N-乙酰-D-葡糖胺的底物特异性发生改变,成功实现对其他非天然底物的高效催化[39]。
针对来源于光滑假丝酵母Candida glabrata的野生型羰基还原酶CgKR1的底物适用性不够广泛、催化活性不够高的缺陷,郑高伟等通过分子动力学模拟分析发现位于该酶底物通道loop上的92位苯丙氨酸与175位催化酪氨酸形成π-π堆积作用,阻碍底物与催化残基的结合。基于结构分析寻找到了两个位点进行突变验证,最终获得一种活性显著提高的突变体对28种结构多样性的羰基底物的催化活性均得到显著提高,可成功用于多种药物手性中间体的扩大制备(图 1)[40]。除此之外,课题组分析了羧酸酯酶rPPE的野生型和有益突变体中底物结合模式的区别,揭示了突变体酶的催化活力、热稳定性和对映选择性提高的结构机理。综合分析酶的底物结合模式、氨基酸位置及序列保守性来寻找潜在敏感的关键氨基酸,预测候选氨基酸突变对蛋白质稳定性和酶活力的影响程度。虚拟筛选出两个潜在的突变体经试验验证催化活力较母本均有显著提高,大幅度改善了酶促合成药物关键中间体的催化效率[41]。针对胺脱氢酶底物谱较窄的问题,许建和课题组采用计算机辅助的蛋白质工程技术,确定了酶活性口袋中影响酶与大位阻底物结合的关键残基,通过将其突变成分子最小的甘氨酸,实现了对口袋的“裁剪”,拓展了活性口袋的“体积”。将酶催化的底物范围由最长6个碳链的脂肪酮拓展至长达10个碳链的脂肪酮(如2-庚酮、2-辛酮、2-壬酮),显著地拓宽了该酶催化的底物范围。此外,这两个关键氨基酸残基的突变在另外两个同源胺脱氢酶的改造中也具有类似的效果,为其他胺脱氢酶的底物谱拓展提供了有益的参考(图 2)[42]。
具有对映选择性的酶作用于一些前手性底物上,可以精确地生成所需的光学纯的对映体产物。生物催化剂的立体选择性是由底物结合、反应能垒等多种因素组合而产生的。通过对酶分子构象的研究可以发现,酶的底物结合口袋呈现出一种稳定构象,使得其仅适应生成一种特定的对映体。另一方面,可以通过引入突变改变酶的能量图景,使其能更优先形成所需的对映体,实现立体选择性的逆转[43]。
QM/MM和MD模拟可以从分子水平上分析调控酶对映选择性的关键因素[44]。大多数的计算研究都是分析在整个MD模拟过程中,底物分子和关键催化氨基酸之间的距离和角度,从而定量分析底物分子在催化中心形成可催化的pro-(S)或者pro-(R)构象的频率[45-46]。van der Kamp等模拟解析了酮还原酶Ⅱ型聚酮合成酶actKR和两个关键突变体中影响立体选择性的3种潜在因素:利用经典分子动力学评估形成pro-R或pro-S反应姿势的频率;采用MM/PBSA自由能方法比较pro-R或pro-S朝向的结合亲和力;通过QM/MM MD模拟与伞形采样评估它们生成trans-1-decalol反应能垒的差异[47]。柠檬烯环氧水解酶LEH突变体可以催化环戊烯氧化物和环己烯氧化物等环氧化物的水解不对称化,不同突变体具有不同的(R, R)-或(S, S)-立体选择性。Reetz等解析了相关突变体对不同底物的特异选择性和立体选择性催化的分子机制。根据计算出的各种途径的活化能,作者发现了有利反应的潜在立体化学机制。由于活性口袋具有显著的灵活性,亲核水分子和环戊烯氧化物之间无法形成催化构象。相反,在催化环己烯氧化物的立体选择性反应时却容易形成反应构象[48]。相关的研究证明了酶催化中立体选择性的复杂性以及基于原子的模拟方法在分析这类问题中的重要作用,同时研究结果也为立体选择性的理性设计提供了理论基础。
设计具有立体选择性的酶的一种途径是降低它们的活性位点体积,使底物只能以特定的方向结合。亦有研究表明,利用MD模拟进行酶结构灵活性分析可用于识别底物结合口袋附近的关键环结构[49]。通过调节这些环结构的构象动力学也可以实现酶分子对映选择性的逆转[50]。Janssen和Baker通过计算与MD模拟相结合的手段设计了环氧化物水解酶的活性位点,成功地获得对映选择性增强的有益突变[12]。针对环氧水解酶VrEH1和VrEH2在催化对硝基苯乙烯类氧化物的水解过程中立体选择性不够完善,造成环氧对映体汇聚反应没有达到100%的理论收率的问题。郁惠蕾等在解析VrEH2高分辨的酶分子及其与底物复合物晶体结构的基础上,借助于计算机模拟准确锁定了酶分子中控制底物结合构象的关键氨基酸位点。通过建立了一个小巧型突变体酶库成功靶向到调控酶催化不同对映体区域选择性反应的关键位点。并且发现该位点的调控作用具有一定程度的普适性,这为同源环氧水解酶的区域选择性改造提供了新思路和新方向(图 3)[51]。
工业过程所需的酶催化剂需要承受在其自然环境中通常不会遇到的反应条件,例如高温、极端pH、高盐、高底物浓度、存在有机溶剂等。酶的稳定性依赖于通过一定的分子内相互作用使其能维持特定功能结构。提高蛋白质的内部结构稳定性可以提高蛋白质的热稳定性、溶剂耐受性等。
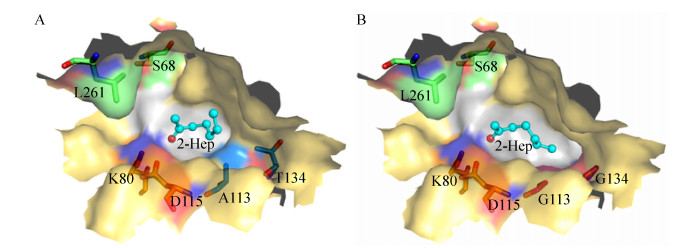
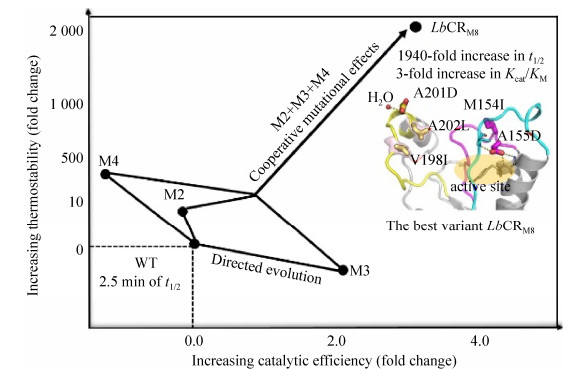
B-FIT方法针对蛋白质晶体结构中B因子较大的灵活氨基酸残基,通过稳定这些灵活的氨基酸来稳定整个蛋白质的结构。利用B-FIT方法,Reetz等获得了来自枯草芽孢杆菌的脂肪酶A (LipA)的突变体,其解链温度Tm增加了45 ℃,进一步研究发现稳定化提高与整个蛋白分子上增加的氢键网络以及新的带电氨基酸残基相关[52]。Bommarius将基于结构的共义性分析方法和B-FIT方法结合起来,成功地将来源于野油菜黄单胞菌的氨基酯水解酶的Tm值提高了8 ℃,kd, obs值提高了13倍[53]。FRESCO方法将可能的单点突变库减少到1 634个,对突变库进行人工检查后,将突变减少到64个进行后续实验筛选。最终获得红色红球菌的柠檬烯环氧化物水解酶的最优突变体在35 ℃时Tm为85 ℃,同时动力学稳定性增加了250倍[13]。许建和课题组利用突变作用的加和性和协同性同时提高了羰基还原酶LbCR的热稳定性和催化活性。突变残基主要位于靠近活性中心的灵活loop环上,热稳定性提高的突变体中引入了新的氢键,并提高了蛋白内部的疏水作用力,从而增强了活性口袋周围灵活loop环的刚性。分子对接结果表明,活性提高的突变点虽然与底物没有直接接触,却导致酶和底物之间氢键作用的增加,从而提高了酶对底物的亲和性,稳定了蛋白与底物结合的作用力。此外,热稳定性提高的突变和活性提高的突变位于两个独立的区域,证实突变作用的加和性为热稳定性和活性的同时提高提供了可能。最终作者成功将反应放大,实现了阿托伐他汀前体6-氰基-3R, 5R-二羟基己酸叔丁酯的规模生物制备(图 4)[54]。
通过定向进化发现的热稳定的酶也具有增加的有机溶剂稳定性,如果糖二磷酸醛缩酶[55]、枯草芽孢杆菌脂肪酶A[56]、葡萄糖脱氢酶等[57]。绘制T50与C50的关系图发现温度稳定性和有机溶剂稳定性之间存在较强的相关性[58]。方柏山等利用基因工程手段构建了甲酸脱氢酶超分子自组装体系,组装的功能片段和融合蛋白形成三维层状结构。将其用于辅酶再生系统时表现出比未组装结构更高的结构稳定性和NAD (H)再循环效率。利用分子动力学模拟研究超分子组装的构型演变,发现FDH的C末端的结构在蛋白质聚合物的形成中起着重要作用[59]。另外一方面,热容变化是解释酶催化反应速率的温度依赖性的重要参数,对酶动力学、热适应性和进化等方面研究都具有重要意义。Arcus等结合计算和实验手段对类固醇异构酶二聚体和α-葡糖苷酶分别进行热容的计算分析,研究发现活性位点周围loop环结构的固定、酶的能量波动的变化以及远离活性位点的远端区域对热容变化都具有重要的影响。相关研究证实可以通过原子水平的分子动力学模拟预测催化反应的活化热容变化,为理性改造酶的最适反应温度提供可行的思路[60]。
1.5 辅酶依赖性反转还原酶的催化需要辅酶NADPH的参与,然而额外添加昂贵的辅酶NADPH,会导致生产成本过高,限制了工业化应用。因此,基于将昂贵NADPH替换成相对廉价NADH的目的,研究者们试图通过蛋白质工程手段改造还原酶的辅酶依赖性。
目前有多种方法被成功用于反转还原酶的辅因子特异性,如:使用结构信息或使用不同辅酶依赖性的酶同源序列进行多序列比对,来寻找有益突变。Turner课题组基于结构和序列比对的方法,在TesADH中成功构建了突变体G198D可以将辅助因子由NADP+到NAD+约1 353倍的反转。最终利用NADH依赖型的最优突变体成功实现了将外消旋仲醇高效地转化生成手性胺[61]。针对辅酶依赖性反转的改造通常会导致酶的催化效率整体下降。2018年诺贝尔化学奖获得者Arnold团队开发了一种计算工具(CSR-SALAD)来寻找辅酶结合关键位点,可用于简化酶的辅酶NAD+或NADP+依赖性逆转的过程,该策略同时可以设计出恢复催化活力的半理性突变库。该方法被成功用于4种结构不同的NADP依赖性的酶(乙醛酸还原酶、肉桂醇脱氢酶、木糖还原酶和含铁醇脱氢酶)的辅酶特异性反转,辅酶依赖性的反转变化达33-4 900倍,证实了该策略的有效性[62]。
针对改变羟甾脱氢酶7β-HSDHs辅酶偏好性的研究,李春秀等提出了一种辅酶特异性反转-小巧突变库的设计策略(CSR-SaSLiD),理性地设计了一个包含5个突变子的小巧定点突变库,快速地筛选到辅酶偏好性反转且活性较高的突变体。相较于母本,突变体实现了辅酶偏好性由NADPH到NADH约953 000倍的反转,活性提高223倍。最后,作者利用分子动力学模拟揭示了辅酶偏好性反转及活性提高的分子机制,并将CSR-SaSLiD策略成功应用于另外两个7β-HSDHs的辅酶偏好性反转和活性提升,表明CSR-SaSLiD是一种有效的改造策略(图 5)[63]。
定量构效关系(QSAR)方法被广泛应用于药物设计领域,目前利用QSAR研究底物结构-功能关系的方法已经逐渐延伸到生物催化领域,成为新的研究热点。酶催化不同底物时将具有不同的催化活力,可以利用定量构效关系方法对酶催化底物谱进行分析,构建合理的酶催化活力的定量构效关系模型,揭示不同底物结构与相应催化活力的关系。
Tomicx课题组提出根据实验数据利用3D-QSAR方法构建描述符,实现了底物专一性常数kcat/Km值的定量预测,并利用这一方法成功实现了来源于洋葱伯克霍尔德菌Bulkholderia cepacia酯酶立体选择性的量化预测[64]。Paier课题组构建3D-QSAR模型分析的环氧化物酶催化反应的立体选择性,分别采用了CoMFA和CoMSIA两种统计算法,两种方法得到的预测结果显示它们都具有较好的预测准确性[65]。Braiuca小组利用实验测得的kcat/Km数据构建回归模型,模型能够准确快速地预测出青霉素酰胺酶的选择性[66]。于洪巍课题组利用分子对接和定量构效关系相结合的方法,构建了具有较高准确性的南极假丝酵母脂肪酶B (CALB)立体选择性的量化预测模型,分别考虑了酰基供体以及5个因素(立体、静电、疏水、氢键供体和受体)对模型的影响,证实了酰基供体的重要性[67]。郁惠蕾等采用3D-QSAR方法构件了羰基还原酶YtbE的定量构效关系模型。通过分子力场轮廓图考察了底物分子取代基团对活力的影响,证实静电作用和疏水作用在分子力场中起到重要作用。进一步地利用该模型对ZINC15小分子数据库进行虚拟筛选,最终成功地筛选到了具有催化活力的化合物,拓展了酶的底物谱[68]。
2.2 基于氨基酸残基网络的研究氨基酸残基网络理论将每个氨基酸定义为一个结点、残基间的互相关系数为边。结合网络拓扑参数、氨基酸相互作用、底物结合能等分析,对酶分子整体结构和动态变化进行分析,可以实现酶分子结构-功能关系的解析。该方法已被成功用于疾病相关蛋白的特异性识别、蛋白质热稳定性等方面的研究。
蒲雪梅等发现单点突变导致β2AR蛋白结构中部分螺旋、loop区及整体结构运动相关性的强弱发生变化,同时也改变了底物结合口袋和信号传导通路的密度,特别是催化氨基酸所在的通路,进而影响药物作用[69]。Osuna课题组对一逆醛缩酶活性位点附近和远离活性中心的氨基酸突变所引起的构象变化进行了系统分析和评估。研究表明,酶的构象状态在不同突变体中发生了显著改变,一些较远距离的氨基酸突变会显著影响酶的催化活力[70]。利用氨基酸残基相互作用网络分析酯酶RhEst1母本和突变体结构稳定性。许建和课题组研究发现突变体中形成更多的氢键、范德华、π-π堆积等相互作用,氨基酸间相互作用的增加稳定了酶的结构的稳定性。基于机理分析,结合氨基酸保守性、氨基酸残基相关性和氨基酸间相互作用,对酶的活力、选择性、热稳定性进行了一系列的理性设计和改造研究[71]。付伟课题组采用动态网络分析方法解析了纤维素酶TmCel12A突变体热稳定显著提高的结构机理,并找到与突变点具有强相关性的关键氨基酸残基,提出了酶热稳定性改造的新策略[72]。
2.3 基于机器学习的方法机器学习技术可以用来识别所有特征变量中的关键特征量,并构建预测模型精确地预测目标参数。研究结果表明机器学习方法能显著提高蛋白质工程中的筛选通量及有益突变的序列空间的质量和多样性[73]。
Umetsu等提出将分子进化与机器学习相结合的蛋白质改造方法。利用第一轮的突变文库构建机器学习模型来指导第二轮的突变。该方法被成功用于绿色荧光蛋白的改造[74]。Arnold课题组利用已发表的GB1结合蛋白的大量数据分析,证实基于机器学习的定性进化方法可以比其他方法发现更优的突变体。利用这一策略,作者成功地对双加氧酶RmaNOD催化新型卡宾Si-H插入反应生成两种对映体产物进行改造,仅通过两轮突变就找到了优势突变体[75]。Tidor等提出了将机器学习与路径采样相结合的新策略来理性寻找影响催化性能的重要区域,并成功地用于酮酸还原异构酶KARI的研究。作者利用计算统计方法,对过渡态采样来模拟反应的动力学,并将其与机器学习方法相结合。确定了与反应性能相关的重要特征并构建了反应轨迹的预测模型。研究发现,利用单一的描述符就可以准确地预测催化活力,准确度超过85%。除此以外,研究发现酶-底物复合物中会存在一个重要区域,当这个区域的构象增多时反应性能大幅增加。相关研究不仅证实了在酶-底物复合物的构象空间中存在能促进反应性能的区域,同时开发了用于鉴定和验证这些特别区域的方法,这将大大提高酶分子设计改造的速率[76]。
2.4 从头设计及人工智能的方法从头设计的思路是针对一些天然酶无法催化的化学反应,设计全新的蛋白质,使之具有特定的空间结构和预期的功能。从头设计的原则是通过计算获得具有特定空间结构中最低自由能状态下的氨基酸序列。
美国华盛顿大学David Baker团队开发的Rosetta算法平台能够从期望的功能反向推测出合适的结构,再计算出最合理的氨基酸序列。利用这一方法,Baker课题组已成功地实现从头创制逆-醛缩酶[77]以及催化Kemp消除反应[78]和Diels-Alder反应的酶[79]。近期该课题组从头设计了荧光蛋白β桶结构[80]、自组装螺旋状蛋白纤维[81]、IL2和IL15受体激动蛋白[82]、抗癌蛋白Neo-2/15[83]、pH环境变化响应蛋白[84]、通过诱导构象改变来调节功能的开关蛋白质[85]等多个复杂的功能蛋白。利用从头设计获得全新的蛋白质为合成生物学和细胞工程的研究提供了新颖的催化元件。
除此之外,谷歌公司DeepMind推出新的人工智能项目AlphaFold,利用经过训练的深度神经网络,可以从基因序列出发预测蛋白质的物理性质和三维结构[86]。基于深度学习的AlphaFold大大缩短了传统计算机模拟计算蛋白质折叠的时间。随着深度学习和人工智能技术的进步,这些技术未来可用于更高效的新酶发掘、更深入的蛋白质结构-功能关系分析及更快速的新药虚拟筛选。
3 总结越来越多的计算分析方法被用于酶的催化反应机理解析,基于酶分子的结构功能关系的深入解析,对结构中特定区域进行理性重构,可以实现高效的定向设计与改造,推动酶的工业应用。理性设计方法用计算替代部分实验室工作,降低了实验的成本和工作量,目的性更强,更加迅速有效。
针对计算本身具有的局限性,发展更快、更高效、更准确的计算方法是未来的技术发展趋势。合理地应用这些计算方法,将有助于提高工业酶的开发效率,推动生物制造产业的发展。
| [1] |
Privett HK, Kiss G, Lee TM, et al. Iterative approach to computational enzyme design. Proc Natl Acad Sci USA, 2012, 109(10): 3790-3795. DOI:10.1073/pnas.1118082108 |
| [2] |
Verma R, Mitchell-Koch K. In silico studies of small molecule interactions with enzymes reveal aspects of catalytic function. Catalysts, 2017, 7(7): 212. DOI:10.3390/catal7070212 |
| [3] |
van der Kamp MW, Mulholland AJ. Combined quantum mechanics/molecular mechanics (QM/MM) methods in computational enzymology. Biochemistry, 2013, 52(16): 2708-2728. DOI:10.1021/bi400215w |
| [4] |
Grajciar L, Heard CJ, Bondarenko AA, et al. Towards operando computational modeling in heterogeneous catalysis. Chem Soc Rev, 2018, 47(22): 8307-8348. DOI:10.1039/C8CS00398J |
| [5] |
Zhu CR, Gao DQ, Ding J, et al. TMD-based highly efficient electrocatalysts developed by combined computational and experimental approaches. Chem Soc Rev, 2018, 47(12): 4332-4356. DOI:10.1039/C7CS00705A |
| [6] |
Huang PS, Ban YEA, Richter F, et al. RosettaRemodel: a generalized framework for flexible backbone protein design. PLoS ONE, 2011, 6(8): e24109. DOI:10.1371/journal.pone.0024109 |
| [7] |
Addington TA, Mertz RW, Siegel JB, et al. Janus: prediction and ranking of mutations required for functional interconversion of enzymes. J Mol Biol, 2013, 425(8): 1378-1389. DOI:10.1016/j.jmb.2013.01.034 |
| [8] |
Bendl J, Stourac J, Sebestova E, et al. HotSpot Wizard 2.0: automated design of site-specific mutations and smart libraries in protein engineering. Nucleic Acids Res, 2016, 44(W1): W479-W487. DOI:10.1093/nar/gkw416 |
| [9] |
Malisi C, Schumann M, Toussaint NC, et al. Binding pocket optimization by computational protein design. PLoS ONE, 2012, 7(12): e52505. DOI:10.1371/journal.pone.0052505 |
| [10] |
Chovancova E, Pavelka A, Benes P, et al. CAVER 3.0: a tool for the analysis of transport pathways in dynamic protein structures. PLoS Comput Biol, 2012, 8(10): e1002708. DOI:10.1371/journal.pcbi.1002708 |
| [11] |
Durrant JD, Votapka L, Sørensen J, et al. POVME 2.0: an enhanced tool for determining pocket shape and volume characteristics. J Chem Theory Comput, 2014, 10(11): 5047-5056. DOI:10.1021/ct500381c |
| [12] |
Wijma HJ, Floor RJ, Bjelic S, et al. Enantioselective enzymes by computational design and in silico screening. Angew Chem Int Ed, 2015, 54(12): 3726-3730. DOI:10.1002/anie.201411415 |
| [13] |
Wijma HJ, Floor RJ, Jekel PA, et al. Computationally designed libraries for rapid enzyme stabilization. Protein Eng Des Sel, 2014, 27(2): 49-58. DOI:10.1093/protein/gzt061 |
| [14] |
Park SB, Bae DW, Clavio NAB, et al. Structural and biochemical characterization of the curcumin-reducing activity of CurA from Vibrio vulnificus. J Agric Food Chem, 2018, 66(40): 10608-10616. DOI:10.1021/acs.jafc.8b03647 |
| [15] |
Ren WW, Pengelly R, Farren-Dai M, et al. Revealing the mechanism for covalent inhibition of glycoside hydrolases by carbasugars at an atomic level. Nat Commun, 2018, 9(1): 3243. |
| [16] |
Fan FF, Chen NH, Wang YH, et al. QM/MM and MM MD simulations on the pyrimidine-specific nucleoside hydrolase: a comprehensive understanding of enzymatic hydrolysis of uridine. J Phys Chem B, 2018, 122(3): 1121-1131. |
| [17] |
Kulkarni YS, Liao QH, Petrović D, et al. Enzyme architecture: modeling the operation of a hydrophobic clamp in catalysis by triosephosphate isomerase. J Am Chem Soc, 2017, 139(30): 10514-10525. DOI:10.1021/jacs.7b05576 |
| [18] |
Schrepfer P, Buettner A, Goerner C, et al. Identification of amino acid networks governing catalysis in the closed complex of class Ⅰ terpene synthases. Proc Natl Acad Sci USA, 2016, 113(8): E958-E967. DOI:10.1073/pnas.1519680113 |
| [19] |
Mhashal AR, Vardi-Kilshtain A, Kohen A, et al. The role of the Met20 loop in the hydride transfer in Escherichia coli dihydrofolate reductase. J Biol Chem, 2017, 292(34): 14229-14239. DOI:10.1074/jbc.M117.777136 |
| [20] |
Ling BP, Li H, Yan LJ, et al. Conversion mechanism of enoyl thioesters into acyl thioesters catalyzed by 2-enoyl-thioester reductases from Candida Tropicalis. Phys Chem Chem Phys, 2019, 21(19): 10105-10113. DOI:10.1039/C9CP00987F |
| [21] |
Driller R, Janke S, Fuchs M, et al. Towards a comprehensive understanding of the structural dynamics of a bacterial diterpene synthase during catalysis. Nat Commun, 2018, 9: 3971. DOI:10.1038/s41467-018-06325-8 |
| [22] |
Karuppiah V, Ranaghan KE, Leferink NGH, et al. Structural basis of catalysis in the bacterial monoterpene synthases linalool synthase and 1, 8-Cineole synthase. ACS Catal, 2017, 7(9): 6268-6282. DOI:10.1021/acscatal.7b01924 |
| [23] |
Haatveit KC, Garcia-Borràs M, Houk KN. Computational protocol to understand P450 mechanisms and design of efficient and selective biocatalysts. Front Chem, 2019, 6: 663. DOI:10.3389/fchem.2018.00663 |
| [24] |
Faponle AS, Quesne MG, De Visser SP. Origin of the regioselective fatty-acid hydroxylation versus decarboxylation by a cytochrome P450 peroxygenase: what drives the reaction to biofuel production?. Chemistry, 2016, 22(16): 5478-5483. DOI:10.1002/chem.201600739 |
| [25] |
Zhou HY, Wang BJ, Wang F, et al. Chemo-and regioselective dihydroxylation of benzene to hydroquinone enabled by engineered cytochrome P450 monooxygenase. Angew Chem Int Ed, 2019, 58(3): 764-768. DOI:10.1002/anie.201812093 |
| [26] |
Świderek K, Tuñón I, Williams IH, et al. Insights on the origin of catalysis on glycine N-methyltransferase from computational modeling. J Am Chem Soc, 2018, 140(12): 4327-4334. DOI:10.1021/jacs.7b13655 |
| [27] |
Marques SM, Dunajova Z, Prokop Z, et al. Catalytic cycle of haloalkane dehalogenases toward unnatural substrates explored by computational modeling. J Chem Inf Model, 2017, 57(8): 1970-1989. DOI:10.1021/acs.jcim.7b00070 |
| [28] |
Wang BJ, Cao ZX, Rovira C, et al. Fenton-derived OH radicals enable the MPnS enzyme to convert 2-hydroxyethylphosphonate to methylphosphonate: insights from Ab initio QM/MM MD simulations. J Am Chem Soc, 2019, 141(23): 9284-9291. DOI:10.1021/jacs.9b02659 |
| [29] |
Sheng X, Plasch K, Payer SE, et al. Reaction mechanism and substrate specificity of Iso-orotate decarboxylase: a combined theoretical and experimental study. Front Chem, 2018, 6: 608. DOI:10.3389/fchem.2018.00608 |
| [30] |
Behiry EM, Ruiz-Pernia JJ, Luk L, et al. Isotope substitution of promiscuous alcohol dehydrogenase reveals the origin of substrate preference in the transition state. Angew Chem Int Ed, 2018, 57(12): 3128-3131. DOI:10.1002/anie.201712826 |
| [31] |
Liu J, Wu SK, Li Z. Recent advances in enzymatic oxidation of alcohols. Curr Opin Chem Biol, 2018, 43: 77-86. DOI:10.1016/j.cbpa.2017.12.001 |
| [32] |
Kong XD, Yuan SG, Li L, et al. Engineering of an epoxide hydrolase for efficient bioresolution of bulky pharmaco substrates. Proc Natl Acad Sci USA, 2014, 111(44): 15717-15722. DOI:10.1073/pnas.1404915111 |
| [33] |
Herter S, Medina F, Wagschal S, et al. Mapping the substrate scope of monoamine oxidase (MAO-N) as a synthetic tool for the enantioselective synthesis of chiral amines. Bioorg Med Chem, 2018, 26(7): 1338-1346. DOI:10.1016/j.bmc.2017.07.023 |
| [34] |
Li GY, Yao PY, Gong R, et al. Simultaneous engineering of an enzyme's entrance tunnel and active site: the case of monoamine oxidase MAO-N. Chem Sci, 2017, 8(5): 4093-4099. DOI:10.1039/C6SC05381E |
| [35] |
Collazo L, Klinman JP. Control of the position of oxygen delivery in soybean lipoxygenase-1 by amino acid side chains within a gas migration channel. J Biol Chem, 2016, 291(17): 9052-9059. DOI:10.1074/jbc.M115.709154 |
| [36] |
Han SW, Kim J, Cho HS, et al. Active site engineering of ω-transaminase guided by docking orientation analysis and virtual activity screening. ACS Catal, 2017, 7(6): 3752-3762. DOI:10.1021/acscatal.6b03242 |
| [37] |
Mader SL, Bräuer A, Groll M, et al. Catalytic mechanism and molecular engineering of quinolone biosynthesis in dioxygenase AsqJ. Nat Commun, 2018, 9(1): 1168. DOI:10.1038/s41467-018-03442-2 |
| [38] |
Dijkman WP, Binda C, Fraaije MW, et al. Structure- based enzyme tailoring of 5-hydroxymethylfurfural oxidase. ACS Catal, 2015, 5(3): 1833-1839. DOI:10.1021/acscatal.5b00031 |
| [39] |
Ferrari AR, Lee M, Fraaije MW. Expanding the substrate scope of chitooligosaccharide oxidase from Fusarium graminearum by structure-inspired mutagenesis. Biotechnol Bioeng, 2015, 112(6): 1074-1080. DOI:10.1002/bit.25532 |
| [40] |
Zheng GW, Liu YY, Chen Q, et al. Preparation of structurally diverse chiral alcohols by engineering ketoreductase CgKR1. ACS Catal, 2017, 7(10): 7174-7181. DOI:10.1021/acscatal.7b01933 |
| [41] |
Chen Q, Luan ZJ, Cheng XL, et al. Molecular dynamics investigation of the substrate binding mechanism in carboxylesterase. Biochemistry, 2015, 54(9): 1841-1848. DOI:10.1021/bi5015612 |
| [42] |
Chen FF, Zheng GW, Liu L, et al. Reshaping the active pocket of amine dehydrogenases for asymmetric synthesis of bulky aliphatic amines. ACS Catal, 2018, 8(3): 2622-2628. DOI:10.1021/acscatal.7b04135 |
| [43] |
Maria-Solano MA, Serrano-Hervás E, Romero-Rivera A, et al. Role of conformational dynamics in the evolution of novel enzyme function. Chem Commun (Camb), 2018, 54(50): 6622-6634. DOI:10.1039/C8CC02426J |
| [44] |
Romero-Rivera A, Garcia-Borràs M, Osuna S. Computational tools for the evaluation of laboratory-engineered biocatalysts. Chem Commun (Camb), 2016, 53(2): 284-297. |
| [45] |
Wijma HJ, Marrink SJ, Janssen DB. Computationally efficient and accurate enantioselectivity modeling by clusters of molecular dynamics simulations. J Chem Inf Model, 2014, 54(7): 2079-2092. DOI:10.1021/ci500126x |
| [46] |
Maria-Solano MA, Romero-Rivera A, Osuna S. Exploring the reversal of enantioselectivity on a zinc-dependent alcohol dehydrogenase. Org Biomol Chem, 2017, 15(19): 4122-4129. DOI:10.1039/C7OB00482F |
| [47] |
Serapian SA, van der Kamp MW. Unpicking the cause of stereoselectivity in actinorhodin ketoreductase variants with atomistic simulations. ACS Catal, 2019, 9(3): 2381-2394. DOI:10.1021/acscatal.8b04846 |
| [48] |
Sun ZT, Salas PT, Siirola E, et al. Exploring productive sequence space in directed evolution using binary patterning versus conventional mutagenesis strategies. Bioresour Bioprocess, 2016, 3: 44. DOI:10.1186/s40643-016-0122-8 |
| [49] |
Sun ZT, Wu L, Bocola M, et al. Structural and computational insight into the catalytic mechanism of limonene epoxide hydrolase mutants in stereoselective transformations. J Am Chem Soc, 2018, 140(1): 310-318. DOI:10.1021/jacs.7b10278 |
| [50] |
Yang B, Wang HJ, Song W, et al. Engineering of the conformational dynamics of lipase to increase enantioselectivity. ACS Catal, 2017, 7(11): 7593-7599. DOI:10.1021/acscatal.7b02404 |
| [51] |
Li FL, Kong XD, Chen Q, et al. Regioselectivity engineering of epoxide hydrolase: near-perfect enantioconvergence through a single site mutation. ACS Catal, 2018, 8(9): 8314-8317. DOI:10.1021/acscatal.8b02622 |
| [52] |
Reetz MT, Soni P, Acevedo JP, et al. Creation of an amino acid network of structurally coupled residues in the directed evolution of a thermostable enzyme. Angew Chem Int Ed Engl, 2009, 48(44): 8268-8272. DOI:10.1002/anie.200904209 |
| [53] |
Blum JK, Ricketts MD, Bommarius AS. Improved thermostability of AEH by combining B-FIT analysis and structure-guided consensus method. J Biotechnol, 2012, 160(3/4): 214-221. |
| [54] |
Gong XM, Qin Z, Li FL, et al. Development of an engineered ketoreductase with simultaneously improved thermostability and activity for making a bulky Atorvastatin precursor. ACS Catal, 2019, 9(1): 147-153. DOI:10.1021/acscatal.8b03382 |
| [55] |
Hao JJ, Berry A. A thermostable variant of fructose bisphosphate aldolase constructed by directed evolution also shows increased stability in organic solvents. Protein Eng Des Sel, 2004, 17(9): 689-697. DOI:10.1093/protein/gzh081 |
| [56] |
Reetz MT, Soni P, Fernández L, et al. Increasing the stability of an enzyme toward hostile organic solvents by directed evolution based on iterative saturation mutagenesis using the B-FIT method. Chem Commun, 2010, 46(45): 8657-8658. DOI:10.1039/c0cc02657c |
| [57] |
Vazquez-Figueroa E, Yeh V, Broering JM, et al. Thermostable variants constructed via the structure-guided consensus method also show increased stability in salts solutions and homogeneous aqueous-organic media. Protein Eng Des Sel, 2008, 21(11): 673-680. DOI:10.1093/protein/gzn048 |
| [58] |
Bommarius AS. Biocatalysis: a status report. Annu Rev Chem Biomol Eng, 2015, 6: 319-345. DOI:10.1146/annurev-chembioeng-061114-123415 |
| [59] |
Jiang W, Yang RN, Lin P, et al. Bioinspired genetic engineering of supramolecular assembled formate dehydrogenase with enhanced biocatalysis activities. J Biotechnol, 2019, 292: 50-56. DOI:10.1016/j.jbiotec.2018.12.017 |
| [60] |
van der Kamp MW, Prentice EJ, Kraakman KL, et al. Dynamical origins of heat capacity changes in enzyme-catalysed reactions. Nat Commun, 2018, 9: 1177. DOI:10.1038/s41467-018-03597-y |
| [61] |
Thompson MP, Turner NJ. Two-enzyme hydrogen-borrowing amination of alcohols enabled by a cofactor-switched alcohol dehydrogenase. ChemCatChem, 2017, 9(20): 3833-3836. DOI:10.1002/cctc.201701092 |
| [62] |
Cahn JKB, Werlang CA, Baumschlager A, et al. A general tool for engineering the NAD/NADP cofactor preference of oxidoreductases. ACS Synth Biol, 2017, 6(2): 326-333. DOI:10.1021/acssynbio.6b00188 |
| [63] |
You ZN, Chen Q, Shi SC, et al. Switching cofactor dependence of 7β-Hydroxysteroid dehydrogenase for cost-effective production of ursodeoxycholic acid. ACS Catal, 2019, 9(1): 466-473. DOI:10.1021/acscatal.8b03561 |
| [64] |
Tomić S, Kojić-Prodić BA. Quantitative model for predicting enzyme enantioselectivity: application to Burkholderia cepacia lipase and 3-(aryloxy)-1, 2-propanediol derivatives. J Mol Graph Model, 2001, 21(3): 241-252. |
| [65] |
Paier J, Stockner T, Steinreiber A, et al. Enantioselectivity of epoxide hydrolase catalysed oxirane ring opening: a 3D QSAR study. J Comput Aided Mol Des, 2003, 17(1): 1-11. |
| [66] |
Braiuca P, Boscarol L, Ebert C, et al. 3D-QSAR applied to the quantitative prediction of penicillin G amidase selectivity. Adv Synth Catal, 2006, 348(6): 773-780. DOI:10.1002/adsc.200505346 |
| [67] |
Gu JL, Liu J, Yu HW. Quantitative prediction of enantioselectivity of Candida antarctica lipase B by combining docking simulations and quantitative structure-activity relationship (QSAR) analysis. J Mol Catal B: Enzym, 2011, 72(3/4): 238-247. |
| [68] |
Cheng Q, Chen Q, Xu JH, et al. A 3D-QSAR assisted activity prediction strategy for expanding substrate spectra of an aldehyde ketone reductase. Mol Catalys, 2018, 455: 224-232. DOI:10.1016/j.mcat.2018.06.013 |
| [69] |
Gao N, Liang T, Yuan Y, et al. Exploring the mechanism of F282L mutation-caused constitutive activity of GPCR by a computational study. Phys Chem Chem Phys, 2016, 18(42): 29412-29422. DOI:10.1039/C6CP03710K |
| [70] |
Romero-Rivera A, Garcia-Borràs M, Osuna S. Role of conformational dynamics in the evolution of retro-aldolase activity. ACS Catal, 2017, 7(12): 8524-8532. DOI:10.1021/acscatal.7b02954 |
| [71] |
Chen Q, Yu HL, Cheng XL, et al. Structural investigation of the enantioselectivity and thermostability mechanisms of esterase RhEst1. J Mol Graph Model, 2018, 85: 182-189. DOI:10.1016/j.jmgm.2018.08.010 |
| [72] |
Lian P, Yuan CM, Xu Q, et al. Thermostability mechanism for the hyperthermophilicity of extremophile cellulase TmCel12A: Implied from molecular dynamics simulation. J Phys Chem B, 2016, 120(30): 7346-7352. DOI:10.1021/acs.jpcb.6b03782 |
| [73] |
Yang KK, Wu Z, Arnold FH. Machine-learning-guided directed evolution for protein engineering. Nat Methods, 2019, 16(8): 687-694. DOI:10.1038/s41592-019-0496-6 |
| [74] |
Saito Y, Oikawa M, Nakazawa H, et al. Machine-learning-guided mutagenesis for directed evolution of fluorescent proteins. ACS Synth Biol, 2018, 7(9): 2014-2022. DOI:10.1021/acssynbio.8b00155 |
| [75] |
Wu Z, Kan SBJ, Lewis RD, et al. Machine learning-assisted directed protein evolution with combinatorial libraries. Proc Natl Acad Sci USA, 2019, 116(18): 8852-8858. DOI:10.1073/pnas.1901979116 |
| [76] |
Bonk BM, Weis JW, Tidor B. Machine learning identifies chemical characteristics that promote enzyme catalysis. J Am Chem Soc, 2019, 141(9): 4108-4118. DOI:10.1021/jacs.8b13879 |
| [77] |
Jiang L, Althoff EA, Clemente FR, et al. De novo computational design of retro-aldol enzymes. Science, 2008, 319(5868): 1387-1391. DOI:10.1126/science.1152692 |
| [78] |
Röthlisberger D, Khersonsky O, Wollacott AM, et al. Kemp elimination catalysts by computational enzyme design. Nature, 2008, 453(7192): 190-195. DOI:10.1038/nature06879 |
| [79] |
Siegel JB, Zanghellini A, Lovick HM, et al. Computational design of an enzyme catalyst for a stereo selective bimolecular Diels-Alder reaction. Science, 2010, 329(5989): 309-313. DOI:10.1126/science.1190239 |
| [80] |
Lu PL, Min DY, DiMaio F, et al. Accurate computational design of multipass transmembrane proteins. Science, 2018, 359(6379): 1042-1046. DOI:10.1126/science.aaq1739 |
| [81] |
Dou JY, Vorobieva AA, Sheffler W, et al. De novo design of a fluorescence-activating β-barrel. Nature, 2018, 561(7724): 485-491. DOI:10.1038/s41586-018-0509-0 |
| [82] |
Shen H, Fallas JA, Lynch E, et al. De novo design of self-assembling helical protein filaments. Science, 2018, 362(6415): 705-709. DOI:10.1126/science.aau3775 |
| [83] |
Silva DA, Yu S, Ulge UY, et al. De novo design of potent and selective mimics of IL-2 and IL-15. Nature, 2019, 565(7738): 186-191. DOI:10.1038/s41586-018-0830-7 |
| [84] |
Boyken SE, Benhaim MA, Busch F, et al. De novo design of tunable, pH-driven conformational changes. Science, 2019, 364(6441): 658-664. DOI:10.1126/science.aav7897 |
| [85] |
Langan RA, Boyken SE, Ng AH, et al. De novo design of bioactive protein switches. Nature, 2019, 572(7768): 205-210. DOI:10.1038/s41586-019-1432-8 |
| [86] |
Evans R, Jumper J, Kirkpatrick J, et al. De novo structure prediction with deep-learning based scoring//Thirteenth Critical Assessment of Techniques for Protein Structure Prediction (Abstracts). Iberostar, Paraiso, 2018.
|
 2019, Vol. 35
2019, Vol. 35