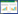中国科学院微生物研究所、中国微生物学会主办
文章信息
- 魏欣蕾, 游淳
- Wei Xinlei, You Chun
- 体外多酶分子机器的现状和最新进展
- In vitro multi-enzyme molecular machines – a review
- 生物工程学报, 2019, 35(10): 1870-1888
- Chinese Journal of Biotechnology, 2019, 35(10): 1870-1888
- 10.13345/j.cjb.190213
-
文章历史
- Received: May 25, 2019
- Accepted: July 15, 2019
体外多酶分子机器是遵循所设计的多酶催化路径,由若干生物酶元件构成,在体外将特定的底物转化为目标化合物的生物催化系统。与目前主流的微生物细胞催化系统相比,体外多酶分子机器具有很多优势,例如副反应少[1]、产品得率高[2-3]、反应速度快[4-5]、产品易分离[6-7]、可耐受有毒的环境[1]、系统可操作性强[1]等,在生物制造领域展现出日益增强的竞争力。
体外多酶分子机器的构建流程通常包括下列步骤:1)确定初始底物和目标产物,设计反应路径,从热力学角度对反应路径进行可行性分析;2)寻找合适的酶元件的来源,对酶元件进行制备和酶学性质表征;3)进行概念实验,检测产物的生成;4)确认反应系统的物质流瓶颈,优化反应条件以解决系统的适配问题。早期的体外多酶分子机器普遍存在酶元件选择少、酶稳定性差、需要添加昂贵辅酶等问题,在多数情况下仅限于高值化学品的生产[8-9]。近年来,随着相关研究的不断发展,上述问题均得到了一定程度的改善,科研人员设计并构建了多种更为复杂的体外多酶分子机器,以经济易得的底物实现了许多大宗化学品的高效合成[3, 10-14]。
随着系统的复杂化,体外多酶分子机器逐渐呈现出模块化的特点[14-17]。每个反应模块由1种或几种酶元件(以及辅酶等非酶元件)构成。元件、模块的自由组合使体外多酶分子机器的催化功能得到了极大的拓展。然而如何在含有大量元件和模块的体外多酶分子机器中实现各组分的有效适配是当前研究面临的关键问题。此外,与细胞工厂不同的是,体外多酶分子机器中的酶元件/模块具有不可再生性,因此对反应系统的稳定性提出了更高的要求。
本文结合国内外已开展的相关研究,介绍体外多酶分子机器中反应模块的设计、构建与应用,并详细阐述各个反应模块的有效适配方法,最后讨论了该分子机器在生物制造领域的发展前景和面临的挑战。
1 反应模块的设计、构建与应用体外多酶分子机器通常包含酶和非酶元件,非酶元件包括辅酶元件和用于构建人工多酶复合体的支架元件、卯榫元件等。在体外多酶分子机器中,完成一些特定功能所需的酶元件或非酶元件的组合就构成了酶的反应模块。根据反应模块对非酶元件的依赖性,反应模块可被分为仅包含酶元件的模块以及由酶元件和非酶元件共同构成的模块两大类。本节将从模块的设计、构建与应用层面分别对这两类反应模块进行介绍。
1.1 仅包含酶元件的反应模块在体外多酶分子机器中,有一类反应模块仅由酶元件构成,无需辅酶等非酶元件的协助即能够独立实现特定的催化功能。例如,淀粉磷酸化模块(Starch phosphorylation module)由α-葡聚糖磷酸化酶(α-glucan phosphorylase,αGP)、异淀粉酶(Isoamylase,IA)以及4-α-糖基转移酶(4-α-glucanotransferase,4GT)这3种酶元件构成,将淀粉和无机磷(Inorganic phosphate,Pi)完全转化为葡萄糖1-磷酸(Glucose 1-phosphate,G1P)并产生少量葡萄糖。在该反应模块的下游添加其他的酶元件或反应模块,能够进一步利用G1P实现氢气[18]、肌醇[19]或昆布二糖[20]等的生产(图 1)。

|
| 图 1 模块化的体外多酶分子机器示例 Fig. 1 Examples of modularized in vitro multi-enzyme molecular machines. IA: isoamylase; 4GT: 4-α-glucanotransferase; αGP: α-glucan phosphorylase; LBP: laminaribiose phosphorylase; PGM: phosphoglucomutase; IPS: inositol 1-phosphate synthase; IMP: inositol monophosphatase; G6PDH: glucose 6-phosphate dehydrogenase; 6PGL: 6-phosphogluconolactonase; 6PGDH: 6-phosphogluconate dehydrogenase; NROR: NADPH rubredoxin oxidoreductase; SHI: soluble hydrogenase I; RPI: ribose 5-phosphate isomerase; RPE: ribulose-phosphate 3-epimerase; TK: transketolase; TAL: transaldolase; TIM: triose phosphate isomerase; ALD: fructose-bisphosphate aldolase; FBP: fructose 1, 6-bisphosphatase; PGI: phosphoglucose isomerase; G1P: glucose 1-phoshpate; Pi: inorganic phosphate; G6P: glucose 6-phosphate; 6-PGL: 6-phosphogluconolactone; 6PG: 6-phosphogluconate; BV: benzyl viologen; Ru5P: ribulose 5-phosphate; R5P: ribose 5-phosphate; Xu5P: xylulose 5-phosphate; G3P: glyceraldehyde 3-phosphate; S7P: sedoheptulose 7-phosphate; E4P: erythrose 4-phosphate; F6P: fructose 6-phosphate; DHAP: dihydroxyacetone phosphate; F1, 6P: fructose 1, 6-bisphosphate. |
| |
在上述以淀粉为底物产氢的体外多酶分子机器中,除了淀粉磷酸化模块之外,另一个仅由酶元件组成的反应模块是将副产物核酮糖5-磷酸(Ribulose 5-phosphate,Ru5P)重新转化为反应体系的中间产物葡萄糖6-磷酸(Glucose 6-phosphate,G6P)的碳重排模块(Carbon rearrangement module) (图 1),其作用是提高底物的利用率。在Cheng等设计的以木糖为底物生产肌醇的体外多酶分子机器中,反应系统的核心也是一个类似的碳重排模块,通过将6分子的木酮糖5-磷酸(Xylulose 5-phosphate,Xu5P)转化为5分子的G6P,完成了从五碳化合物到六碳化合物的转变[21]。碳重排模块也可实现由四碳化合物向六碳化合物的转化。例如,Igor等设计的非氧化糖酵解(Non-oxidative glycolysis)体外多酶分子机器中包含一个将3分子赤藓糖4-磷酸(Erythrose 4-phosphate,E4P)转化为2分子果糖6-磷酸(Fructose 6-phosphate,F6P)的碳重排模块,以实现该反应途径的最终产物——乙酰磷酸(Acetyl phosphate)的化学计量数生成[22]。在这个碳重排模块的基础上,Wei等添加了淀粉磷酸化模块和L-茶氨酸生产元件,构建了以淀粉为底物、以乙酰磷酸为直接能量供体的体外多酶ATP再生底盘系统,实现了ATP的化学计量数再生,并以再生的ATP进行了L-茶氨酸的高效生产[13]。
为了简化反应体系、降低生产成本、提升系统稳定性,研究者们在设计体外多酶分子机器反应路径时会尽量减少所需酶元件的种类,并规避辅酶的使用[16]。例如丙酮酸是糖酵解途径的最终产物,也是重要的平台化合物。然而生物体内利用天然代谢途径将甘油转化为丙酮酸的过程涉及至少8种酶元件以及NAD、ATP等辅酶元件的参与[23-25]。为了简化反应系统,Gao等设计了一个仅需3种热稳酶元件即可将甘油转化为丙酮酸的人工反应模块,并在其下游添加了另外2种酶元件进行(3R)-乙偶姻((3R)-acetoin)的生产,获得了85.5%的高产品得率[26]。该反应模块简单、稳定、高效,为构建以甘油为底物经由丙酮酸生产其他生物化学品的体外多酶分子机器奠定了良好基础。
1.2 由酶元件和非酶元件共同构成的反应模块 1.2.1 NAD(P)辅酶再生模块在多数情况下,体外多酶分子机器反应系统会不可避免地需要辅酶的参与。天然辅酶由于价格昂贵、稳定性差、在高浓度时对某些酶的活性造成抑制等因素,不适合一次性大量加入体外多酶分子机器中进行反应[27-28]。辅酶再生模块能够很好地解决上述问题,是构建可持续运作的辅酶依赖型体外多酶分子机器的重要前提。
天然的烟酰胺类辅酶NAD(P)是氧化还原酶元件最常用的电子中介体,通过在NAD(P)+ (氧化态)和NAD(P)H (还原态)之间相互转化实现电子的传递。常见的NAD(P)+再生模块由NAD(P)H氧化酶(NAD(P)H oxidase)[3, 29]或乳酸脱氢酶(Lactate dehydrogenase)[30]等单一的酶元件与相应的辅酶元件组合而成。而常见的最简单的NAD(P)H再生模块则由醇脱氢酶(Alcohol dehydrogenase,ADH)[30-31]、甲酸脱氢酶(Formate dehydrogenase,FDH)[32-34]、葡萄糖脱氢酶(Glucose dehydrogenase,GDH)[35-37]、亚磷酸脱氢酶(Phosphite dehydrogenase,PTDH)[38-39]或氢酶(Hydrogenase)[40-41]等单一的酶元件与相应的辅酶元件组合而成。
更为复杂的烟酰胺类辅酶再生模块包含多种酶元件。例如,Kim等利用醇脱氢酶、醛脱氢酶(Aldehyde dehydrogenase,ALDH)和甲酸脱氢酶组合成反应模块,使1分子甲醇完全氧化并产生3分子NADH,这些还原力可用于生物产电[42]。该反应模块可作为一个以甲醇为底物的高效NADH再生模块(图 2A)与其他消耗NADH的酶反应模块结合,创建新的体外多酶分子机器。来源于极端嗜热的激烈火球菌Pyrococcus furiosus的可溶性氢酶(Soluble hydrogenase I,SHI)偏好辅酶NADP+而对NAD+的活性极低,可以利用氢气为底物在高温下进行NADPH的再生[43-44]。为了实现基于氢酶的NADH再生,Song等构建了一个包含SHI和具有热稳定性的黄递酶(Diaphorase,DI)的NADH再生模块,利用DI将SHI产生的NADPH转化为NADH (图 2B),并在该反应模块的基础上添加了具有热稳定性的乳酸脱氢酶,在50 ℃的条件下实现了从丙酮酸到乳酸的完全转化[45]。
|
| 图 2 含有多种酶元件的NAD(H)再生模块 Fig. 2 Examples of NAD(H) regeneration modules comprised of more than one enzyme. (A) An NADH regeneration module based on the complete oxidation of methanol. (B) An NADH regeneration module that consumes hydrogen gas. (C) An NAD+ regeneration module that produces NAD+ from its heat-degradation products. ADH: alcohol dehydrogenase; ALDH: aldehyde dehydrogenase; FDH: formate dehydrogenase; SHI: soluble hydrogenase I; DI: diaphorase; ADPRP: ADP-ribose pyrophosphatase; ADK: adenylate kinase; PPK: polyphosphate kinase; RPK: ribose-phosphate pyrophosphokinase; NADase: nicotinamidase; NAPRT: nicotinate phosphoribosyltransferase; NaMAT: nicotinate monomucleotide adenylyltransferase; NADS: NAD+ synthase; polyP: polyphosphate. |
| |
烟酰胺类辅酶再生模块通常进行的是NAD(P)+与NAD(P)(H)之间的转化。这类辅酶再生模块与消耗辅酶的反应模块结合,用以达到理想状态下体外多酶分子机器内部的辅酶平衡。由于多酶反应有时在高温下进行,而NAD(P)+和NAD(P)H易遇热分解,导致在较高的温度下进行反应的体外多酶分子机器在运行过程中辅酶的浓度逐渐降低,影响了催化效率[12, 46]。Honda等发现在高温条件下,NAD+被分解为更稳定的烟酰胺(Nicotinamide)和ADP-核糖(ADP-ribose),因此构建了包含4种热稳酶元件的辅酶再生模块,将NAD+受热分解产生的烟酰胺和ADP-核糖重新合成为NAD+ (图 2C),使反应体系中NAD+的浓度在60 ℃能够保持恒定达15 h[47]。该辅酶再生模块能够在一定程度上缓解辅酶的不稳定性对体外多酶分子机器造成的负面影响。
1.2.2 ATP再生模块三磷酸腺苷(Adenosine triphosphate,ATP)是一种高能磷酸化合物,通过与二磷酸腺苷(Adenosine diphosphate,ADP)的相互转化实现能量的贮存和释放,为生物催化过程提供能量。含有ATP的体外多酶分子机器通常需要加入ATP再生模块,以保证ATP的持续供应。以多聚磷酸激酶(Polyphosphate kinase,PPK)和ADP构成的反应模块是目前应用最为广泛的ATP再生策略[48-51]。该ATP再生模块利用价格低廉的多聚磷酸(Polyphosphate)作为ATP再生的磷酸供体。然而高浓度的多聚磷酸会与体外多酶分子机器反应系统中的二价金属离子螯合,影响酶元件的催化活性[52-53]。因此,研究人员开发出更为复杂的ATP再生模块,以经济易得的不含磷化合物作为能量来源,将ADP和无机磷重新生成为ATP。例如,Kim和Swartz使用含有3种酶元件的反应模块,将丙酮酸经由乙酰辅酶A (Acetyl-CoA)和乙酰磷酸转化为乙酸,同时完成ATP的再生,每分子丙酮酸能再生1分子ATP (图 3)[54]。该ATP再生模块也被称为PANOx系统[55]。为了进一步提高ATP的产量,Wang等向PANOx模块上游添加了糖酵解模块,以葡萄糖为底物生产ATP再生所需的丙酮酸,改进后的ATP再生模块每消耗1分子葡萄糖底物可再生3分子ATP (图 3)[55]。在这个改进后的ATP再生模块中,葡萄糖首先在己糖激酶(Hexokinase,Hex)的催化作用下被磷酸化为G6P,并消耗ATP。为了进一步提高ATP的再生效率,研究者们又使用α-葡聚糖磷酸化酶和磷酸葡萄糖变位酶(Phosphoglucomutase,PGM)取代了己糖激酶,构建了以麦芽糊精和无机磷为底物的ATP再生模块[55]。该模块避免了葡萄糖磷酸化这一消耗ATP的过程,因此每个葡萄糖当量的麦芽糊精总共可再生4分子ATP。然而这些ATP再生模块都需要NAD(H)和辅酶A (Coenzyme A,CoA)等昂贵辅酶的参与。为了规避价格昂贵且不稳定的NAD(P)以及CoA的使用,Wei等构建了另一种基于麦芽糊精的ATP再生模块,在利用α-葡聚糖磷酸化酶和磷酸葡萄糖变位酶将麦芽糊精磷酸化为G6P后,使用磷酸葡萄糖异构酶(Phosphoglucose isomerase,PGI)将G6P转化为F6P,进而利用转酮酶(Phosphoketolase,PKL)将F6P分解为E4P和乙酰磷酸,之后以乙酰磷酸为直接磷酸供体完成ATP的再生(图 3)[13]。在将E4P重新转化为F6P的碳重排模块的协助下,上述ATP再生模块每消耗1个葡萄糖当量的麦芽糊精可再生3分子ATP。

|
| 图 3 以丙酮酸、葡萄糖或麦芽糊精为能量来源的ATP再生模块 Fig. 3 ATP regeneration modules that utilize pyruvate, glucose or maltodextrin as energy source. Hex: hexokinase; PK: pyruvate kinase; αGP: α-glucan phosphorylase; PGM: phosphoglucomutase; PGI: phosphoglucose isomerase; PKL: phosphoketolase; AK: acetate kinase; LDH: lactate dehydrogenase; PDH: pyruvate dehydrogenase; PTA: phosphate acetyltransferase; G6P: glucose 6-phosphate; F1, 6P: fructose 1, 6-bisphosphate; Pi: inorganic phosphate; PEP: phosphoenolpyruvate; F6P: fructose 6-phosphate; E4P: erythrose 4-phosphate. |
| |
对于消耗ATP产生单磷酸腺苷(Adenosine monophosphate,AMP)的酶元件而言,相应的ATP再生模块需要将AMP转化为ATP[56]。Resnick等构建了包含2种酶元件的ATP再生模块,首先以多聚磷酸为磷酸供体,使用多磷酸AMP磷酸转移酶(Polyphosphate:AMP phosphotransferase,PPT)将AMP磷酸化为ADP,之后使用腺苷酸激酶(Adenylate kinase,ADK)将2分子ADP转化为1分子ATP和1分子AMP[57]。为了进一步提高ATP再生的效率,Kameda等构建了PAP-PPK模块,其中包含2种能够利用多聚磷酸为磷酸供体的酶元件,将AMP经由ADP转化为ATP[52]。该ATP再生模块与消耗ATP产生AMP的乙酰辅酶A合成酶(Acetyl-CoA synthase,ACS)联用,实现了以乙酸和辅酶A为底物的乙酰辅酶A的生产,产物基于辅酶A的得率达到99.5%。
1.2.3 人工多酶复合体反应模块除了辅酶元件,常见的非酶元件还包括用于构建人工多酶复合体反应模块的支架元件、卯榫元件等。这些支架元件和卯榫元件能够协助酶元件按照一定的空间结构形成自组装人工多酶复合体,产生底物通道效应(Substrate channeling),减少反应中间产物的扩散,从而展现出高效的催化特性。
基于连接肽这一卯榫元件构建而成的融合酶是一种最常见的人工多酶复合体(图 4A)。Peters等使用长度为12–13个氨基酸残基的连接肽将烯醇还原酶(Enoate reductase)与Baeyer-Villiger单加氧酶(Baeyer-Villiger-monooxygenase)组装成融合酶,并在此基础上添加游离的醇脱氢酶构建成完整的体外多酶分子机器,将不饱和醇转化为内脂(Lactone)[58]。相比以游离酶元件构建而成的多酶分子机器,含有酶复合体的多酶分子机器的产量提高了40%。Fan等使用(GGGGS)3连接肽将NAD依赖型甲醇脱氢酶(Methanol dehydrogenase,Mdh)与6-磷酸己酮糖合成酶(Hexulose-6-phosphate synthase,Hps)及6-磷酸己酮糖异构酶(6-phospho- 3-hexuloisomerase,Phi)改造为Mdh-Hps-Phi融合酶,其消耗甲醇的Vmax约为游离Mdh的5.7倍,以甲醇为底物生产F6P的反应速率约为3种游离酶混合物的1.3倍[59]。
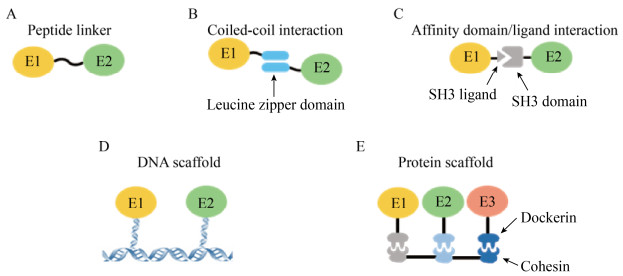
|
| 图 4 人工多酶复合体的组装策略 Fig. 4 Strategies for the construction of artificial multi-enzyme complexes. (A) A multi-enzyme complex using a peptide linker as connecting component. (B) A multi-enzyme complex using the leucine zipper domains as connecting components. (C) A multi-enzyme complex using an affinity domain and its ligand as connecting components. (D) A multi-enzyme complex based on a DNA scaffold. (E) A multi-enzyme complex based on a protein scaffold. E1, E2 and E3 resemble enzyme components. |
| |
卯榫元件的多样性为人工多酶复合体的组装提供了多种策略。例如,亮氨酸拉链结构域(Leucine zipper domain)能够通过coiled-coil相互作用形成二聚体结构[60],可作为一种卯榫元件与酶元件进行融合,从而将多种酶元件组装成复合体(图 4B)。Kim等构建了一个包含3种酶元件的反应模块,利用醇脱氢酶、醛脱氢酶和甲酸脱氢酶的级联反应实现甲醇的完全氧化[42]。每种酶元件的N端都融合了一个亮氨酸拉链结构域,使得这些酶元件能够基于coiled-coil相互作用自组装形成水凝胶结构。与亮氨酸拉链结构域的融合对醛脱氢酶的催化活性几乎没有影响,并提高了醇脱氢酶和甲酸脱氢酶的催化活性。具有亲和作用的特定结构域及其配体也可被作为卯榫元件,介导多酶复合体的组装(图 4C)。例如,Price等将SH3结构域及其配体与Hps-Phi融合酶及甲醇脱氢酶分别融合,通过SH3结构域与其配体的亲和作用组装了三酶复合体,以甲醇和核酮糖5-磷酸为底物生产F6P[61]。与游离酶相比,使用该多酶复合体使F6P的产量提高了近50倍。
为了使人工多酶复合体的组装和催化过程更加可控,研究者们往往将卯榫元件与支架元件联用,使多种酶元件在支架元件上进行距离可控或顺序可控的空间排列。DNA支架元件的稳定性强,且可以通过改变DNA链的长度精准调控酶元件在支架上的排列间距,是研究多酶复合体底物通道机制的重要模型(图 4D)。例如,Wilner等使用单链DNA自组装成内含多个六边形的带状结构,之后通过化学交联的方式将辣根过氧化物酶(Horseradish peroxidase,HRP)、葡萄糖脱氢酶以及辅酶NAD+共固定在DNA支架上,大幅度提高了反应速率[62]。Ngo等在木糖还原酶(Xylose reductase,XR)和木糖醇脱氢酶(Xylitol dehydrogenase,XDH) 2种酶元件上分别融合了不同的DNA结合蛋白(DNA-binding protein)作为卯榫元件,从而将酶元件固定在DNA折纸结构(DNA origami)上形成双酶复合体,以木糖为底物生产木酮糖[63]。酶元件在DNA支架上的距离被认为是影响该双酶复合体催化效率的决定性因素。
与DNA支架相比,蛋白支架的成本更加低廉,更适合被应用于体外多酶分子机器进行生物化学品的生产。最为广泛研究的蛋白支架是由多个黏连蛋白(Cohesin)通过连接肽进行融合制作而成(图 4E)。融合了锚定蛋白(Dockerin)的酶元件可通过黏连蛋白与锚定蛋白之间的特异性亲和作用被安装在蛋白支架上。例如,You等设计了含有3种来源于不同嗜热菌的黏连蛋白的线性支架mini-scaffoldin,将3种热稳酶元件在该蛋白支架上进行了有序排列,使其催化速率相比游离酶提高了1个数量级[64]。该支架蛋白mini-scaffoldin上还携带有纤维素结合域(Cellulose-binding module,CBM),可与纤维素结合,实现多酶复合体的一步组装与纯化[65]。Chen等以同样的mini- scaffoldin蛋白支架将包括氢酶在内的3种热稳酶元件组装成人工多酶复合体,以麦芽糊精为底物进行了氢气的生产[66]。与游离酶相比,该多酶复合体在70 ℃具有相似的稳定性,但能将反应速率提升2.5倍。Liu等将含有3种黏连蛋白的支架展示在酵母细胞表面,将醇脱氢酶、甲醛脱氢酶和甲酸脱氢酶有序地组装成为复合体,使NADH的生产速率提高了5倍[67]。其他具有亲和作用的卯榫元件也可被用于蛋白支架的构建。例如,Yu等设计了包含3个卯榫元件的IPa-IPb-IPc线性蛋白支架,进而通过IPA/IPa、IPB/IPb、IPC/IPc这3组卯榫蛋白元件的相互作用构建了包含3种纤维素酶的复合体,并通过调整3种纤维素酶在蛋白支架上的排列顺序,进一步提高了纤维素分解的效率[68]。
具有平面型空间结构的蛋白支架元件也可以被用于人工多酶复合体的组装。例如,Zhang等利用可自组装成六边形片状结构的肠道沙门氏菌Salmonella enterica壳蛋白(Shell protein)作为支架元件,并将壳蛋白与卯榫元件SpyCatcher融合,继而通过SpyTag/SpyCatcher卯榫元件的共价相互作用构建了双酶复合体,将(S)-2-己醇((S)-2-hexanol)转化为(R)-2-氨基己烷((R)-2- aminohexane)[69]。与游离酶相比,该双酶复合体使反应达到90%底物转化率的时间缩短了50%。Hirakawa等使用具有空心环状结构的增殖细胞核抗原(Proliferating cell nuclear antigen,PCNA)作为蛋白支架,将细菌细胞色素P450 (Cytochrome P450)、铁氧还蛋白(Ferredoxin)以及铁氧还蛋白还原酶(Ferredoxin reductase)分别与异源三聚体PCNA的3个亚基经由连接肽融合,从而基于PCNA蛋白支架的自组装形成了人工三酶复合体,将反应速率提升了2个数量级[70]。
上述研究证明了人工多酶复合体在提升反应效率方面具有很高的应用价值和潜力。在未来的研究中,人工多酶复合体可作为一个独立的反应模块被整合于更为复杂的体外多酶分子机器中,以提升多酶分子机器反应系统的整体效率。
2 体外多酶分子机器各组分的适配构成同一体外多酶分子机器的各生物元件在辅酶偏好性、催化活性、最适温度和pH、热稳定性等方面都可能存在差异。过于显著的元件性质差异将导致反应系统不能很好地适配,从而影响体外多酶分子机器的催化效率。因此,实现体外多酶分子机器各组分之间的适配是提升反应系统运行效率的重要前提。
本节主要从辅酶相关的系统适配、酶元件的催化功能改造与系统适配以及体外多酶分子机器物质流瓶颈的确认与解除这3个方面对目前的研究现状进行详细介绍。
2.1 辅酶相关的系统适配 2.1.1 氧化还原酶的辅酶偏好性改造体外多酶分子机器有时包含多种氧化还原酶元件及相应的辅酶元件。其中,烟酰胺类辅酶NADP(H)和NAD(H)是生命体氧化还原过程最常见的电子中介体。大多数氧化还原酶都具有特定的辅酶偏好性[71]。解决酶元件与辅酶元件的适配问题有助于使整个体外多酶分子机器协调运转,实现高效的生物制造。在1.2.1小节中所介绍的,利用黄递酶将反应体系中的NADP(H)与NAD(H)进行相互转化的方法,是对体外多酶分子机器中酶元件与辅酶元件进行协调适配的一种策略[45]。而对氧化还原酶元件进行辅酶偏好性改造则是一种更为常见的实现酶与辅酶有效适配的方法。
酮醇酸还原异构酶(Keto-acid reductoisomerase,KARI)通常为NADPH依赖型[72],在支链氨基酸(Branched-chain amino acids,BCAAs)等化学品的生产中发挥着重要作用[73-75]。Brinkmann-Chen等以KARI为例,通过蛋白序列比对、蛋白晶体结构分析等手段,总结出转换此类酶的辅酶偏好性的通用方法[76]。经改造后的NADH依赖型KARI能够与产生NADH的反应模块(如常见的糖酵解模块)相结合,实现BCAA的生产。
为了降低生产成本,用于构建体外多酶分子机器的酶元件通常只经由热处理或镍柱亲和纯化等简单的处理即投入使用,导致酶元件中不可避免地含有少量杂质。提升体外多酶分子机器的反应温度有助于减少杂质导致的副反应,提高产品得率,但对元件的稳定性提出了更高的要求。相对于NADP,NAD的价格较低且相对稳定[5, 77-78]。将天然依赖NADP的具有热稳定性的酶元件改造为NAD依赖型,有助于酶元件和整个体外多酶分子机器稳定性的提升。Chen等比较和分析了多种NADP+和NAD+依赖型的6-葡萄糖酸脱氢酶(6-phosphogluconate dehydrogenase,6PGDH)的氨基酸序列,并借助计算机模拟分子对接的技术研究了酶和辅酶之间的相互作用,确认了与NADP+磷酸基团相互作用的氨基酸残基并将其进行理性设计,从而成功将来源于极端嗜热的海栖热袍菌Thermotoga maritima的6PGDH由NADP+依赖型改造成为NAD+依赖型[79]。6PGDH是磷酸戊糖途径(Pentose phosphate pathway,PPP)中的一部分,为了构建一个稳定的NAD依赖型的磷酸戊糖人工反应模块,Kim等又对磷酸戊糖途径中的另一个氧化还原酶——葡萄糖6-磷酸脱氢酶(Glucose 6-phosphate dehydrogenase,G6PDH)进行了辅酶偏好性改造,将来源于T. maritima的G6PDH由NADP+依赖型改造为NAD+依赖型,从而实现了磷酸戊糖人工反应模块中的辅酶适配[80]。该反应模块被应用于以淀粉为能量来源的体外多酶分子机器,能够在80 ℃条件下高效催化水分解产生氢气。
2.1.2 辅酶浓度的智能调控在构建含有辅酶元件的体外多酶分子机器时,常规的策略是通过合理的反应路径设计,达到理想状态下辅酶消耗与再生的化学计量数平衡。然而,体外多酶分子机器的实际运作过程往往伴随一些消耗辅酶的副反应,如NADH的自发性氧化[9]、ATP的自发性水解,以及难以完全去除的ATP水解酶对ATP的消耗[81]等。这些副反应导致了辅酶的实际消耗量大于理论值。因此,在体外多酶分子机器长时间运转后往往会出现体系中辅酶浓度逐渐降低的状况,影响了反应效率。针对ATP降解的问题,Opgenorth等基于变阻器的思路设计了一个ATP调控模块(ATP rheostat) (图 5A)[81]。该调控模块包含2个独立的反应分支,均能将反应的中间产物甘油醛3-磷酸(Glyceraldehyde 3-phosphate,G3P)转化为下游反应所需的3-磷酸甘油酸(3-phosphoglycerate,3PG)。其中一个反应分支仅包含1种不能进行磷酸化的磷酸甘油醛脱氢酶(Nonphosphorylating glyceraldehyde phosphate dehydrogenase,GapN),在将G3P转化为3PG的同时生成下游反应所需的NAPDH,该反应并不产生ATP;另一个反应分支包含可进行磷酸化的磷酸甘油醛脱氢酶(Phosphorylating glyceraldehyde phosphate dehydrogenase,mGapDH)和磷酸甘油酸激酶(Phosphoglycerate kinase,PGK),在这2个酶的作用下,将G3P经由1, 3-二磷酸甘油酸(1, 3- bisphosphoglycerate,1, 3-BPG)转化为3PG并产生NADPH,同时将ADP和无机磷再生为ATP。多酶分子机器中的磷浓度是决定该ATP调控模块运行方式的因素:当磷浓度较低时,GapN支路发挥作用进行NADPH的生产,该反应模块并无ATP生成;当ATP经由副反应水解导致体系中磷浓度升高时,mGapDH/PGK支路被启用,产生额外的ATP以弥补损耗,从而将体外多酶分子机器中的ATP浓度长时间维持在一个恒定的水平,大幅度提高了产品的生成量。

|
| 图 5 能够智能调控辅酶浓度的反应模块 Fig. 5 Reaction modules that facilitate the smart regulation of coenzyme concentrations. (A) An ATP rheostat that responds to changes of phosphate concentration. (B) An NAPDH purge valve that automatically adjusts the NADPH concentration. GapN: nonphosphorylating glyceraldehyde phosphate dehydrogenase; mGapDH: phosphorylating glyceraldehyde phosphate dehydrogenase; PGK: phosphoglycerate kinase; PDH: pyruvate dehydrogenase; NoxE: NADH oxidase; G3P: glyceraldehyde 3-phosphate; Pi: inorganic phosphate; 1, 3-BPG: 1, 3-bisphosphoglycerate; 3PG: 3-phosphoglycerate; PHB: polyhydroxybutyrate. |
| |
在构建体外多酶分子机器的过程中,有时难以通过简单的路径设计达到辅酶消耗与再生的化学计量数平衡。例如在Opgenorth等设计的以丙酮酸为底物的聚羟基丁酸酯(Polyhydroxybutyrate,PHB)合成途径中,丙酮酸脱氢酶(Pyruvate dehydrogenase,PDH)将1分子丙酮酸、1分子辅酶A以及1分子NADP+转化为1分子乙酰辅酶A和1分子NADPH;乙酰辅酶A和NADPH进而被下游的PHB合成模块以2︰1的化学计量数所利用,即每消耗1分子乙酰辅酶A仅再生1/2分子的NADP+,使整个体外多酶分子机器在长时间运行过程中出现NADPH的积累[9]。针对该问题,研究者们设计了一个维持体外多酶分子机器内部NADP+与NADPH平衡的控制阀模块(Purge valve module) (图 5B)。该控制阀模块包含3种酶元件,分别为依赖NADP+的突变型PDH,依赖NAD+的野生型PDH,以及仅能消耗NADH的NADH氧化酶(NADH oxidase,NoxE)。当系统中NADPH/NADP+比值较低的时候,控制阀处于关闭状态,NADP+依赖型的PDH正常发挥功能生成乙酰辅酶A,同时产生NADPH;当系统中NADPH/NADP+比值升高时,控制阀被打开,NADPH依赖型PDH的活性受到高浓度NADPH的抑制,乙酰辅酶A的生产由NADH依赖型PDH完成,而NoxE消耗了所产生的NADH,既保证了系统中NAD+的再生,又避免了无用的NADH的积累。该控制阀模块为体外多酶分子机器的反应路径设计提供了更灵活的思路。
2.1.3 人工仿生辅酶元件的创制及应用人工仿生辅酶(Biomimetic cofactors)元件的创制,有望进一步提高辅酶的稳定性并降低其成本[77, 82],从而为基于酶元件催化的生物制造模式带来更广阔的工业化前景。此外,通过改造特定的酶元件使其偏好人工辅酶,可以将相应的酶催化反应与体外多酶分子机器中其他消耗天然辅酶的酶催化反应分离开来,有利于生物催化过程的精准调控[83]。
烟酰胺类人工仿生辅酶是目前研究的热点。基于天然存在的NAD和NADP (图 6A),目前人们已创制出多种烟酰胺类人工仿生辅酶元件(图 6B),并对酶元件进行了改造,以实现其与人工仿生辅酶的有效适配。Ji等以非天然的氟胞嘧啶(Flucytosine)取代天然辅酶NAD中的腺嘌呤部分,创制出NAD的类似物烟酰胺氟代胞嘧啶二核苷酸(Nicotinamide flucytosine dinucleotide,NFCD),并通过定点饱和突变的方式改造出偏好NFCD的苹果酸脱氢酶(Malate dehydrogenase)和乳酸脱氢酶[83]。这两种突变酶元件的组合形成了1个将苹果酸转化为乳酸,同时实现NFCD循环的简单反应模块。在后续的研究中,Ji等又将NFCD中的氟原子分别以氯原子、溴原子或甲基取代,创制出更多人工仿生辅酶元件,以及偏好这些人工仿生辅酶元件的突变型苹果酸酶(Malic enzyme)[84]。相比由野生型苹果酸酶与NAD组合而成的反应模块,由突变型苹果酸酶与人工仿生辅酶元件组合而成的反应模块具有更高的催化效率(kcat/Km)。Liu等通过半理性设计的方式,改造出偏好另一人工仿生辅酶——烟酰胺胞嘧啶二核苷酸(Nicotinamide cytosine dinucleotide,NCD)的亚磷酸脱氢酶,并解析了突变酶与NCD的结合机制[85]。在对酶元件利用天然辅酶的能力进行改造时,也可能同时获得对人工仿生辅酶具有催化活性的突变体酶元件。例如,Campbell等对来自极端嗜热菌P. furiosus的醇脱氢酶的辅酶结合口袋进行了理性改造,使其对NAD和NADP的催化活性都得到了提升[86],并发现改造后的醇脱氢酶能够利用人工仿生辅酶烟酰胺单核苷酸(Nicotinamide mononucleotide,NMN)进行催化[87]。Maurer等将细胞色素P450的2个氨基酸残基进行突变,使该细胞色素P450从NADPH偏好型转变为NADH偏好型[88]。而Ryan等发现经改造后的该细胞色素P450还获得了利用人工仿生辅酶1-苄基- 1, 4二氢烟酰胺(N-benzyl-1, 4-dihydronicotinamide,BNA)进行催化的能力[89]。

|
| 图 6 天然烟酰胺辅酶与人工仿生烟酰胺辅酶的结构 Fig. 6 Structures of natural nicotinamide-based coenzymes and biomimetic nicotinamide coenzymes. (A) Common natural nicotinamide-based coenzymes. (B) Biomimetic nicotinamide coenzymes. NAD: nicotinamide adenine dinucleotide; NADP: nicotinamide adenine dinucleotide phosphate; NFCD: nicotinamide flucytosine dinucleotide; NMN: nicotinamide mononucleotide; BNA: N-benzyl-1, 4-dihydronicotinamide. |
| |
由于多数人工仿生辅酶与天然辅酶存在较明显的结构差异,对人工仿生辅酶的创制与开发利用仍处于初期研究阶段,面临诸多挑战[87]。未来我们需要创制更多稳定性高、价格低廉的人工辅酶,并对相应的酶元件进行辅酶偏好性改造,进而将人工仿生辅酶与相适配的酶元件应用于更复杂的体外多酶分子机器反应系统中,充分发挥其优势。
2.2 酶元件的催化功能改造与系统适配天然酶元件有时无法高效完成特定的催化反应,因此需要对酶元件的催化功能进行改造,实现其与所设计的体外多酶分子机器的适配。Lu等设计了仅利用3种酶元件即可将甲醛经由糖醛(Glycoaldehyde)和乙酰磷酸最终转化为乙酰辅酶A的全新反应模块[90]。在该反应模块的基础上,可添加上游反应的相应酶元件,实现二氧化碳、甲烷、甲醇等一碳化合物的固定,也可添加下游反应的相应酶元件将乙酰辅酶A进一步转化为蛋白质、糖类等高值化学品。为了获得能够催化模块中第一步反应(将2分子甲醛缩合为1分子糖醛)的酶元件,研究者们根据天然辅酶元件二磷硫酸铵(Thiamine diphosphate,ThDP)协助催化醛类形成二聚物的催化机理创建出theozyme模型,基于模型中ThDP的C2原子与产物糖醛的距离对数据库中ThDP依赖型的天然酶元件进行了筛选,并对其中活性相对最高的酶元件进行了定向进化改造,使其将甲醛转化为糖醛的活性提高了近70倍,进而实现了整个反应模块的顺利运行,为未来以一碳化合物为底物高效生产高值化学品提供了新的思路。
根据热力学分析,将L-阿拉伯糖(L-arabinose)转化为L-核酮糖(L-ribulose)的单酶催化反应是难以进行的。为了实现从L-阿拉伯糖到L-核酮糖的高效转化,Chuaboon等设计了包含2种酶元件的反应模块,将L-阿拉伯糖经由keto-arabinose转化为L-核酮糖[91]。在该反应模块中,糖氧化酶催化第一步反应,过程需要消耗氧气,因此可以通过提高氧气供应的方式推动整个反应模块的运行。使该反应模块顺利运行的关键在于获得一个对L-阿拉伯糖具有较高底物特异性的糖氧化酶。研究者们通过理性设计,将偏好以六碳糖为底物的天然吡喃糖2-氧化酶(Pyranose 2-oxidase,P2O)改造为偏好L-阿拉伯糖的突变体,从而实现了其与下游酶元件的适配。在这个反应模块的基础上,研究者们进一步添加了基于甲酸脱氢酶的NADPH再生模块,以及用于消耗突变体P2O反应副产物的过氧化氢酶(Catalase)元件,组成了一个能够更持久运行的体外多酶分子机器反应系统,实现了从L-阿拉伯糖到L-核酮糖的完全转化。
2.3 物质流瓶颈的确认与解除相对于微生物细胞工厂,体外多酶分子机器的反应路径和组成成分相对简单,因此更易于寻找制约反应效率提升的关键因素。Su等构建了含有7种酶元件的体外多酶分子机器,以蔗糖(Sucrose)为底物生产葡萄糖酸(Glucaric acid,GA)[92]。在确认了该体外多酶分子机器的最适运行温度、pH、底物浓度、缓冲液类型等条件后,研究者们采用了若干实验方法,包括:1)控制单一变量,每次降低1种酶元件的浓度,并检测产物GA的得率;2)测试产物GA对各个酶元件的抑制作用;3)测试各个酶元件的稳定性,从而寻找到该体外多酶分子机器中的限速酶元件,继而通过提高限速酶的浓度以及在反应过程中流加限速酶的手段,解除了物质流瓶颈,使GA的产量得到进一步提升。
随着生物信息学技术的迅速发展,计算机模拟分析(In silico analysis)已成为实现体外多酶分子机器体系适配的重要辅助手段。Zhong等设计了一个包含3种酶元件、将蔗糖转化为纤维二糖(Cellobiose)的体外多酶分子机器,并建立了相应的动力学模型,用于预测3种酶元件之间的最优比例等反应条件,继而在模拟结果的指导下进行了实验[93]。计算机模拟也可以被用于预测更为复杂的体外多酶分子机器反应系统的适配条件,大幅度减少实验工作量。例如,Korman等设计了包含5个反应模块、20余种酶元件、将葡萄糖转化为单萜类化合物(Monoterpenes)的体外多酶分子机器,并使用COPASI软件[94]建立了该多酶分子机器的动力学模型,预测出限制反应效率的关键因素,进而在此基础上进行了实验验证,实现了单萜类化合物的高效生产[95]。
在完善体外多酶分子机器运行效率的过程中,有时需要人为设置限速步骤,以实现各个酶元件的适配。例如,在上述以葡萄糖为底物生产单萜类化合物的体外多酶分子机器中,处于反应路径最上游的己糖激酶(Hexokinase,Hex)和处于相对下游的磷酸果糖激酶(Phosphofructokinase)都需要消耗ATP进行反应[95]。为了防止反应系统上游的Hex反应速度过快将反应系统中的ATP耗尽,研究者们通过限制Hex的添加量,实现了体外多酶分子机器中2种消耗ATP的酶元件的适配。
产物抑制是制约体外多酶分子机器效率的因素之一。Busto等构建了包含3种酶元件的体外多酶分子机器,将酚类衍生物转化为对映异构纯的对羟基苯基乳酸(p-hydroxyphenyl lactic acid)[15]。在概念实验中,产物得率仅为24%。研究者们通过改变底物浓度、改变氧气含量、额外添加最终产物进而分析产品得率的方法,对该体外多酶分子机器的适配性进行了分析,发现反应终产物对多酶分子机器中催化第一步反应的酶元件有抑制作用。为了克服产物抑制,研究者们采用了一锅式分阶段反应,首先向反应体系中加入进行第一步催化反应的酶元件,待其完成反应后再添加下游酶元件,从而将产品得率提升至58%–85%。
除了克服产物抑制,分阶段反应的策略也可被用于解决体外多酶分子机器中某些酶元件稳定性差的问题。Tian等构建了利用4–5种酶元件将蔗糖转化为棉子糖(Raffinose)或水苏糖(Stachyose)的体外多酶分子机器,其中,反应路径最下游的2种酶元件在30 ℃反应36 h后活性显著下降[96]。改进后的体外多酶分子机器仍然采用一锅式反应,但包含2个反应阶段:在第一阶段,反应路径上游几种较为稳定的酶元件首先进行反应,积累大量可被用于后续反应的中间产物;在第二阶段,较不稳定的2种下游酶元件被添加入反应体系中。这种分阶段反应的方法使产物棉子糖的得率提高了4.2倍。Meng等设计的一锅法以纤维多糖(Cellodextrin)为底物生产肌醇(Inositol)的体外多酶分子机器存在各个酶元件的最适反应温度并不一致的问题[97]。反应路径上游的3种酶元件的最适温度为55 ℃,而下游的另外3种热稳酶元件均具有更高的最适温度,其中反应路径下游的肌醇1-磷酸合成酶(Inositol 1-phosphate synthase,IPS)只有在温度超过60 ℃时才有较高酶活。为了提高肌醇的得率,研究者们在55 ℃的起始条件下进行一锅式反应,并对反应中间产物浓度随时间的变化进行了检测,从而确定在反应进行8 h后将温度提升至70 ℃以解除IPS的催化瓶颈,最终达到了接近100%的肌醇得率。分阶段进行反应的方法保证了整个体外多酶分子机器的稳定性与适配性,是一种提高产品得率的有效策略。
纳米材料是提升体外多酶分子机器运行效率和系统稳定性的潜在工具。以固定二氧化碳的体外多酶分子机器为例,该分子机器包含甲酸脱氢酶、甲醛脱氢酶和醇脱氢酶,将二氧化碳经由甲酸和甲醛最终还原为甲醇。热力学性质分析表明,该系统中的甲酸脱氢酶和甲醛脱氢酶所催化的反应并不偏好于生产目标产品,然而研究人员认为将上述酶元件共同固定在具有孔状结构的纳米材质中,有助于提高反应中间产物的局部浓度,从而提升该多酶分子机器的反应效率[98]。Wang等设计了基于纳米硅颗粒和邻苯二酚修饰的明胶的双层微囊结构,使上述3种酶元件依次分布于微囊空腔、微囊内层和微囊外层[99]。与游离酶相比(甲醇基于NADH的得率为35.5%),该微囊结构使甲醇的得率提升至71.6%。在9次循环使用后,基于微囊结构的体外多酶分子机器的甲醇得率仍可达到52.6%,表明该纳米微囊结构的构建可提高体外多酶分子机器的重复利用效率,降低生产成本。Ji等将上述3种酶元件和用于NADH再生的谷氨酸脱氢酶共同包裹在掺杂了聚丙烯胺盐酸盐(Poly(allylamine hydrochloride),PAH)的聚氨酯中空纳米纤维内,并利用PAH与NADH之间的静电作用将NADH稳定地吸附于中空纳米纤维的内壁,同时在纳米纤维外表面固定了碳酸酐酶,从而将甲醇基于NADH的得率提升至103.2%[100]。这个包含酶和辅酶元件的中空纳米纤维结构在10次重复使用后仍保有80%的催化活性。尽管上述基于纳米材料和结构的体外多酶分子机器仅能产生μmol/L浓度级别的甲醇,纳米材料仍不失为一种改进体外多酶分子机器性能的有效手段,有望在未来得到更广泛的应用。
3 总结与展望体外多酶分子机器具有不需维持细胞的生长、反应路径明确、副反应少、无细胞膜的阻碍、生产过程具有高度可调控性等特点,是高效生产目标化合物的重要手段。体外多酶分子机器作为一个强大的生物制造平台,具有广阔的应用前景。例如以淀粉为底物生产肌醇的体外多酶分子机器已被用于工业规模的生产[19],初步展现出这一生物制造平台的工业应用价值。然而,总体来说,体外多酶分子机器的稳定性和系统中元件/模块的适配性仍是目前亟需改善的两大最关键的问题。未来体外多酶分子机器的研究方向和发展趋势是:1)建立可共享的酶元件、非酶元件以及反应模块数据库,实现反应路径的精简化、智能化设计;2)继续新酶元件的发掘和对现有酶元件的改造,以获得催化活性高、热稳定性良好的新酶元件,并创制成本低廉、稳定性高的人工辅酶和蛋白质骨架等非酶元件,提升体外多酶分子机器的稳定性;3)开发多样化的有效手段,解决反应系统中的元件与模块适配问题,加快反应速度和提升产品得率;4)建立并完善目标化合物的大规模生产和产物分离纯化等相关技术,促进体外多酶分子机器的工业化应用。随着相关研究的逐渐深入,上述问题有望在未来得到解决,使体外多酶分子机器这一生物制造平台发挥出更大的潜力,更好地服务于科学研究和工业生产。
| [1] |
Zhang YHP. Production of biocommodities and bioelectricity by cell-free synthetic enzymatic pathway biotransformations: challenges and opportunities. Biotechnol Bioeng, 2010, 105(4): 663-677. |
| [2] |
Rollin JA, Martin del Campo J, Myung S, et al. High-yield hydrogen production from biomass by in vitro metabolic engineering: mixed sugars coutilization and kinetic modeling. Proc Natl Acad Sci USA, 2015, 112(16): 4964-4969. DOI:10.1073/pnas.1417719112 |
| [3] |
Opgenorth PH, Korman TP, Bowie JU. A synthetic biochemistry module for production of bio-based chemicals from glucose. Nat Chem Biol, 2016, 12(6): 393-395. DOI:10.1038/nchembio.2062 |
| [4] |
Zhang YHP, Myung S, You C, et al. Toward low-cost biomanufacturing through in vitro synthetic biology: bottom-up design. J Mater Chem, 2011, 21(47): 18877-18886. DOI:10.1039/c1jm12078f |
| [5] |
Zhang YHP. Production of biofuels and biochemicals by in vitro synthetic biosystems: opportunities and challenges. Biotechnol Adv, 2015, 33(7): 1467-1483. DOI:10.1016/j.biotechadv.2014.10.009 |
| [6] |
Qi P, You C, Zhang YHP. One-pot enzymatic conversion of sucrose to synthetic amylose by using enzyme cascades. ACS Catal, 2014, 4(5): 1311-1317. DOI:10.1021/cs400961a |
| [7] |
Kim JE, Zhang YHP. Biosynthesis of D-xylulose 5-phosphate from D-xylose and polyphosphate through a minimized two-enzyme cascade. Biotechnol Bioeng, 2016, 113(2): 275-282. DOI:10.1002/bit.25718 |
| [8] |
Zhang YHP. Simpler is better: high-yield and potential low-cost biofuels production through cell-free synthetic pathway biotransformation (SyPaB). ACS Catal, 2011, 1(9): 998-1009. |
| [9] |
Opgenorth PH, Korman TP, Bowie JU. A synthetic biochemistry molecular purge valve module that maintains redox balance. Nat Commun, 2014, 5: 4113. DOI:10.1038/ncomms5113 |
| [10] |
Wang YR, Huang WD, Sathitsuksanoh N, et al. Biohydrogenation from biomass sugar mediated by in vitro synthetic enzymatic pathways. Chem Biol, 2011, 18(3): 372-380. DOI:10.1016/j.chembiol.2010.12.019 |
| [11] |
Schultheisz HL, Szymczyna BR, Scott LG, et al. Enzymatic de novo pyrimidine nucleotide synthesis. J Am Chem Soc, 2011, 133(2): 297-304. DOI:10.1021/ja1059685 |
| [12] |
Krutsakorn B, Honda K, Ye XT, et al. In vitro production of n-butanol from glucose. Metab Eng, 2013, 20: 84-91. DOI:10.1016/j.ymben.2013.09.006 |
| [13] |
Wei XL, Xie LP, Zhang YHPJ, et al. Stoichiometric regeneration of ATP by a NAD(P)/CoA-free and phosphate-balanced in vitro synthetic enzymatic biosystem. ChemCatChem, 2018, 10(24): 5597-5601. DOI:10.1002/cctc.201801562 |
| [14] |
Valliere MA, Korman TP, Woodall NB, et al. A cell-free platform for the prenylation of natural products and application to cannabinoid production. Nat Commun, 2019, 10(1): 565. DOI:10.1038/s41467-019-08448-y |
| [15] |
Busto E, Simon RC, Richter N, et al. One-pot, two-module three-step cascade to transform phenol derivatives to enantiomerically pure (R)- or (S)-p-hydroxyphenyl lactic acids. ACS Catal, 2016, 6(4): 2393-2397. DOI:10.1021/acscatal.6b00030 |
| [16] |
Taniguchi H, Okano K, Honda K. Modules for in vitro metabolic engineering: pathway assembly for bio-based production of value-added chemicals. Synth Syst Biotechnol, 2017, 2(2): 65-74. DOI:10.1016/j.synbio.2017.06.002 |
| [17] |
Sperl JM, Sieber V. Multienzyme cascade reactions—status and recent advances. ACS Catal, 2018, 8(3): 2385-2396. DOI:10.1021/acscatal.7b03440 |
| [18] |
Kim JE, Kim EJ, Chen H, et al. Advanced water splitting for green hydrogen gas production through complete oxidation of starch by in vitro metabolic engineering. Metab Eng, 2017, 44: 246-252. DOI:10.1016/j.ymben.2017.09.015 |
| [19] |
You C, Shi T, Li YJ, et al. An in vitro synthetic biology platform for the industrial biomanufacturing of myo-inositol from starch. Biotechnol Bioeng, 2017, 114(8): 1855-1864. DOI:10.1002/bit.26314 |
| [20] |
Sun SS, Wei XL, You C. The Construction of an in vitro synthetic enzymatic biosystem that facilitates laminaribiose biosynthesis from maltodextrin and glucose. Biotechnol J, 2019, 14(4): 1800493. DOI:10.1002/biot.201800493 |
| [21] |
Cheng K, Zheng WM, Chen HG, et al. Upgrade of wood sugar D-xylose to a value-added nutraceutical by in vitro metabolic engineering. Metab Eng, 2019, 52: 1-8. DOI:10.1016/j.ymben.2018.10.007 |
| [22] |
Bogorad IW, Lin TS, Liao JC. Synthetic non-oxidative glycolysis enables complete carbon conservation. Nature, 2013, 502(7473): 693-697. DOI:10.1038/nature12575 |
| [23] |
Chao YP, Patnaik R, Roof WD, et al. Control of gluconeogenic growth by pps and pck in Escherichia coli. J Bacteriol, 1993, 175(21): 6939-6944. DOI:10.1128/jb.175.21.6939-6944.1993 |
| [24] |
Gonzalez R, Murarka A, Dharmadi Y, et al. A new model for the anaerobic fermentation of glycerol in enteric bacteria: trunk and auxiliary pathways in Escherichia coli. Metab Eng, 2008, 10(5): 234-245. DOI:10.1016/j.ymben.2008.05.001 |
| [25] |
Murarka A, Dharmadi Y, Yazdani SS, et al. Fermentative utilization of glycerol by Escherichia coli and its implications for the production of fuels and chemicals. Appl Environ Microbiol, 2008, 74(4): 1124-1135. DOI:10.1128/AEM.02192-07 |
| [26] |
Gao C, Li Z, Zhang LJ, et al. An artificial enzymatic reaction cascade for a cell-free bio-system based on glycerol. Green Chem, 2015, 17(2): 804-807. DOI:10.1039/C4GC01685H |
| [27] |
Iwamoto S, Motomura K, Shinoda Y, et al. Use of an Escherichia coli recombinant producing thermostable polyphosphate kinase as an ATP regenerator to produce fructose 1, 6-diphosphate. Appl Environ Microbiol, 2007, 73(17): 5676-5678. DOI:10.1128/AEM.00278-07 |
| [28] |
Wu H, Tian CY, Song XK, et al. Methods for the regeneration of nicotinamide coenzymes. Green Chem, 2013, 15(7): 1773-1789. DOI:10.1039/c3gc37129h |
| [29] |
Wu X, Kobori H, Orita I, et al. Application of a novel thermostable NAD(P)H oxidase from hyperthermophilic archaeon for the regeneration of both NAD+ and NADP+. Biotechnol Bioeng, 2012, 109(1): 53-62. |
| [30] |
Tong XD, El-Zahab B, Zhao XY, et al. Enzymatic synthesis of L-lactic acid from carbon dioxide and ethanol with an inherent cofactor regeneration cycle. Biotechnol Bioeng, 2011, 108(2): 465-469. DOI:10.1002/bit.22938 |
| [31] |
Fossati E, Polentini F, Carrea G, et al. Exploitation of the alcohol dehydrogenase-acetone NADP- regeneration system for the enzymatic preparative- scale production of 12-ketochenodeoxycholic acid. Biotechnol Bioeng, 2006, 93(6): 1216-1220. DOI:10.1002/bit.20753 |
| [32] |
Wong CH, Whitesides GM. Enzyme-catalyzed organic synthesis: NAD(P)H cofactor regeneration using ethanol/alcohol dehydrogenase/aldehyde dehydrogenase and methanol/alcohol dehydrogenase/ aldehyde dehydrogenase/formate dehydrogenase. J Org Chem, 1982, 47(14): 2816-2818. DOI:10.1021/jo00135a037 |
| [33] |
Božič M, Pricelius S, Guebitz GM, et al. Enzymatic reduction of complex redox dyes using NADH-dependent reductase from Bacillus subtilis coupled with cofactor regeneration. Appl Microbiol Biotechnol, 2010, 85(3): 563-571. DOI:10.1007/s00253-009-2164-8 |
| [34] |
Tao RS, Jiang Y, Zhu FY, et al. A one-pot system for production of L-2-aminobutyric acid from L-threonine by L-threonine deaminase and a NADH-regeneration system based on L-leucine dehydrogenase and formate dehydrogenase. Biotechnol Lett, 2014, 36(4): 835-841. DOI:10.1007/s10529-013-1424-y |
| [35] |
Wong CH, Drueckhammer DG, Sweers HM. Enzymatic vs. fermentative synthesis: thermostable glucose dehydrogenase catalyzed regeneration of NAP(P)H for use in enzymatic synthesis. J Am Chem Soc, 1985, 107(13): 4028-4031. DOI:10.1021/ja00299a044 |
| [36] |
Eguchi T, Kuge Y, Inoue K, et al. NADPH regeneration by glucose dehydrogenase from Gluconobacter scleroides for l-leucovorin synthesis. Biosci Biotech Biochem, 1992, 56(5): 701-703. DOI:10.1271/bbb.56.701 |
| [37] |
Xu ZN, Jing KJ, Liu Y, et al. High-level expression of recombinant glucose dehydrogenase and its application in NADPH regeneration. J Ind Microbiol Biotechnol, 2007, 34(1): 83-90. |
| [38] |
Johannes TW, Woodyer RD, Zhao HM. Directed evolution of a thermostable phosphite dehydrogenase for NAD(P)H regeneration. Appl Environ Microbiol, 2005, 71(10): 5728-5734. DOI:10.1128/AEM.71.10.5728-5734.2005 |
| [39] |
Relyea HA, van der Donk WA. Mechanism and applications of phosphite dehydrogenase. Bioorg Chem, 2005, 33(3): 171-189. DOI:10.1016/j.bioorg.2005.01.003 |
| [40] |
Mertens R, Liese A. Biotechnological applications of hydrogenases. Curr Opin Biotechnol, 2004, 15(4): 343-348. DOI:10.1016/j.copbio.2004.06.010 |
| [41] |
Eberly JO, Ely RL. Thermotolerant hydrogenases: biological diversity, properties, and biotechnological applications. Crit Rev Microbiol, 2008, 34(3/4): 117-130. |
| [42] |
Kim YH, Campbell E, Yu J, et al. Complete oxidation of methanol in biobattery devices using a hydrogel created from three modified dehydrogenases. Angew Chem Int Ed, 2013, 52(5): 1437-1440. DOI:10.1002/anie.201207423 |
| [43] |
Ma K, Weiss R, Adams MWW. Characterization of hydrogenase Ⅱ from the hyperthermophilic archaeon Pyrococcus furiosus and assessment of its role in sulfur reduction. J Bacteriol, 2000, 182(7): 1864-1871. DOI:10.1128/JB.182.7.1864-1871.2000 |
| [44] |
Mertens R, Greiner L, van den Ban ECD, et al. Practical applications of hydrogenase I from Pyrococcus furiosus for NADPH generation and regeneration. J Mol Catal B: Enzym, 2003, 24-25: 39-52. DOI:10.1016/S1381-1177(03)00071-7 |
| [45] |
Song YH, Liu MX, Xie LP, et al. A recombinant 12-His tagged Pyrococcus furiosus soluble. Biotechnol J, 2019, 14(4): 1800301. DOI:10.1002/biot.201800301 |
| [46] |
Ye XT, Honda K, Sakai T, et al. Synthetic metabolic engineering-a novel, simple technology for designing a chimeric metabolic pathway. Microb Cell Fact, 2012, 11: 120. DOI:10.1186/1475-2859-11-120 |
| [47] |
Honda K, Hara N, Cheng M, et al. In vitro metabolic engineering for the salvage synthesis of NAD+. Metab Eng, 2016, 35: 114-120. DOI:10.1016/j.ymben.2016.02.005 |
| [48] |
Liu S, Li Y, Zhu J. Enzymatic production of L-theanine by γ-glutamylmethylamide synthetase coupling with an ATP regeneration system based on polyphosphate kinase. Process Biochem, 2016, 51(10): 1458-1463. DOI:10.1016/j.procbio.2016.06.006 |
| [49] |
Meng QL, Zhang YF, Ju XZ, et al. Production of 5-aminolevulinic acid by cell free multi-enzyme catalysis. J Biotechnol, 2016, 226: 8-13. DOI:10.1016/j.jbiotec.2016.03.024 |
| [50] |
Cao H, Li CC, Zhao J, et al. Enzymatic production of glutathione coupling with an ATP regeneration system based on polyphosphate kinase. Appl Biochem Biotechnol, 2018, 185(2): 385-395. |
| [51] |
Zhang X, Wu H, Huang B, et al. One-pot synthesis of glutathione by a two-enzyme cascade using a thermophilic ATP regeneration system. J Biotechnol, 2017, 241: 163-169. DOI:10.1016/j.jbiotec.2016.11.034 |
| [52] |
Kameda A, Shiba T, Kawazoe Y, et al. A novel ATP regeneration system using polyphosphate-AMP phosphotransferase and polyphosphate kinase. J Biosci Bioeng, 2001, 91(6): 557-563. DOI:10.1016/S1389-1723(01)80173-0 |
| [53] |
Wang W, Liu MX, You C, et al. ATP-free biosynthesis of a high-energy phosphate metabolite fructose 1, 6-diphosphate by in vitro metabolic engineering. Metab Eng, 2017, 42: 168-174. DOI:10.1016/j.ymben.2017.06.006 |
| [54] |
Kim DM, Swartz JR. Regeneration of adenosine triphosphate from glycolytic intermediates for cell-free protein synthesis. Biotechnol Bioeng, 2001, 74(4): 309-316. DOI:10.1002/bit.1121 |
| [55] |
Wang YR, Zhang YHP. Cell-free protein synthesis energized by slowly-metabolized maltodextrin. BMC Biotechnol, 2009, 9: 58. DOI:10.1186/1472-6750-9-58 |
| [56] |
Kitabatake S, Dombou M, Tomioka I, et al. Synthesis of P1, P4-di(adenosine 5'-) tetraphosphate by leucyl-tRNA synthetase, coupled with ATP regeneration. Biochem Biophys Res Commun, 1987, 146(1): 173-178. |
| [57] |
Resnick SM, Zehnder AJB. In vitro ATP regeneration from polyphosphate and AMP by polyphosphate: AMP phosphotransferase and adenylate kinase from Acinetobacter johnsonii 210A. Appl Environ Microbiol, 2000, 66(5): 2045-2051. DOI:10.1128/AEM.66.5.2045-2051.2000 |
| [58] |
Peters C, Rudroff F, Mihovilovic MD, et al. Fusion proteins of an enoate reductase and a Baeyer-Villiger monooxygenase facilitate the synthesis of chiral lactones. Biol Chem, 2017, 398(1): 31-37. DOI:10.1515/hsz-2016-0150 |
| [59] |
Fan LW, Wang Y, Tuyishime P, et al. Engineering artificial fusion proteins for enhanced methanol bioconversion. ChemBioChem, 2018, 19(23): 2465-2471. DOI:10.1002/cbic.201800424 |
| [60] |
Landschulz WH, Johnson PF, McKnight SL. The leucine zipper: a hypothetical structure common to a new class of DNA binding proteins. Science, 1988, 240(4860): 1759-1764. DOI:10.1126/science.3289117 |
| [61] |
Price JV, Chen L, Whitaker WB, et al. Scaffoldless engineered enzyme assembly for enhanced methanol utilization. Proc Natl Acad Sci USA, 2016, 113(45): 12691-12696. DOI:10.1073/pnas.1601797113 |
| [62] |
Wilner OI, Weizmann Y, Gill R, et al. Enzyme cascades activated on topologically programmed DNA scaffolds. Nat Nanotechnol, 2009, 4(4): 249-254. DOI:10.1038/nnano.2009.50 |
| [63] |
Ngo TA, Nakata E, Saimura M, et al. Spatially organized enzymes drive cofactor-coupled cascade reactions. J Am Chem Soc, 2016, 138(9): 3012-3021. DOI:10.1021/jacs.5b10198 |
| [64] |
You C, Myung S, Zhang YHP. Facilitated substrate channeling in a self-assembled trifunctional enzyme complex. Angew Chem Int Ed, 2012, 51(35): 8787-8790. DOI:10.1002/anie.201202441 |
| [65] |
You C, Zhang YHP. Self-assembly of synthetic metabolons through synthetic protein scaffolds: one-step purification, co-immobilization, and substrate channeling. ACS Synth Biol, 2013, 2(2): 102-110. DOI:10.1021/sb300068g |
| [66] |
Chen H, Huang R, Kim EJ, et al. Building a thermostable metabolon for facilitating coenzyme transport and in vitro hydrogen production at elevated temperature. ChemSusChem, 2018, 11(18): 3120-3130. DOI:10.1002/cssc.201801141 |
| [67] |
Liu F, Banta S, Chen W. Functional assembly of a multi-enzyme methanol oxidation cascade on a surface-displayed trifunctional scaffold for enhanced NADH production. Chem Commun, 2013, 49(36): 3766-3768. DOI:10.1039/c3cc40454d |
| [68] |
Yu T, Gao X, Ren YH, et al. Assembly of cellulases with synthetic protein scaffolds in vitro. Bioresour Bioprocess, 2015, 2: 16. DOI:10.1186/s40643-015-0046-8 |
| [69] |
Zhang GQ, Quin MB, Schmidt-Dannert C. Self-assembling protein scaffold system for easy in vitro coimmobilization of biocatalytic cascade enzymes. ACS Catal, 2018, 8(6): 5611-5620. DOI:10.1021/acscatal.8b00986 |
| [70] |
Hirakawa H, Nagamune T. Molecular assembly of P450 with ferredoxin and ferredoxin reductase by fusion to PCNA. ChemBioChem, 2010, 11(11): 1517-1520. DOI:10.1002/cbic.201000226 |
| [71] |
Cahn JKB, Werlang CA, Baumschlager A, et al. A general tool for engineering the NAD/NADP cofactor preference of oxidoreductases. ACS Synth Biol, 2017, 6(2): 326-333. |
| [72] |
Dumas R, Biou V, Halgand F, et al. Enzymology, structure, and dynamics of acetohydroxy acid isomeroreductase. Acc Chem Res, 2001, 34(5): 399-408. DOI:10.1021/ar000082w |
| [73] |
Arfin SM, Umbarger HE. Purification and properties of the acetohydroxy acid isomeroreductase of Salmonella typhimurium. J Biol Chem, 1969, 244(5): 1118-1127. |
| [74] |
Atsumi S, Hanai T, Liao JC. Non-fermentative pathways for synthesis of branched-chain higher alcohols as biofuels. Nature, 2008, 451(7174): 86-89. DOI:10.1038/nature06450 |
| [75] |
Shen CR, Liao JC. Metabolic engineering of Escherichia coli for 1-butanol and 1-propanol production via the keto-acid pathways. Metab Eng, 2008, 10(6): 312-320. DOI:10.1016/j.ymben.2008.08.001 |
| [76] |
Brinkmann-Chen S, Flock T, Cahn JKB, et al. General approach to reversing ketol-acid reductoisomerase cofactor dependence from NADPH to NADH. Proc Natl Acad Sci USA, 2013, 110(27): 10946-10951. DOI:10.1073/pnas.1306073110 |
| [77] |
Paul CE, Arends IWCE, Hollmann F. Is simpler better? Synthetic nicotinamide cofactor analogues for redox chemistry. ACS Catal, 2014, 4(3): 788-797. DOI:10.1021/cs4011056 |
| [78] |
You C, Huang R, Wei XL, et al. Protein engineering of oxidoreductases utilizing nicotinamide-based coenzymes, with applications in synthetic biology. Synth Syst Biotechnol, 2017, 2(3): 208-218. DOI:10.1016/j.synbio.2017.09.002 |
| [79] |
Chen H, Zhu ZG, Huang R, et al. Coenzyme engineering of a hyperthermophilic 6-phosphogluconate dehydrogenase from NADP+ to NAD+ with its application to biobatteries. Sci Rep, 2016, 6: 36311. |
| [80] |
Kim EJ, Kim JE, Zhang YHPJ. Ultra-rapid rates of water splitting for biohydrogen gas production through in vitro artificial enzymatic pathways. Energ Environ Sci, 2018, 11(8): 2064-2072. DOI:10.1039/C8EE00774H |
| [81] |
Opgenorth PH, Korman TP, Iancu L, et al. A molecular rheostat maintains ATP levels to drive a synthetic biochemistry system. Nat Chem Biol, 2017, 13(9): 938-942. DOI:10.1038/nchembio.2418 |
| [82] |
Rollin JA, Tam TK, Zhang YHP. New biotechnology paradigm: cell-free biosystems for biomanufacturing. Green Chem, 2013, 15(7): 1708-1719. DOI:10.1039/c3gc40625c |
| [83] |
Ji DB, Wang L, Hou SH, et al. Creation of bioorthogonal redox systems depending on nicotinamide flucytosine dinucleotide. J Am Chem Soc, 2011, 133(51): 20857-20862. DOI:10.1021/ja2074032 |
| [84] |
Ji DB, Wang L, Liu WJ, et al. Synthesis of NAD analogs to develop bioorthogonal redox system. Sci China Chem, 2013, 56(3): 296-300. DOI:10.1007/s11426-012-4815-3 |
| [85] |
Liu YX, Feng YB, Wang L, et al. Structural insights into phosphite dehydrogenase variants favoring a non-natural redox cofactor. ACS Catal, 2019, 9(3): 1883-1887. DOI:10.1021/acscatal.8b04822 |
| [86] |
Campbell E, Wheeldon IR, Banta S. Broadening the cofactor specificity of a thermostable alcohol dehydrogenase using rational protein design introduces novel kinetic transient behavior. Biotechnol Bioeng, 2010, 107(5): 763-774. DOI:10.1002/bit.22869 |
| [87] |
Campbell E, Meredith M, Minteer SD, et al. Enzymatic biofuel cells utilizing a biomimetic cofactor. Chem Commun, 2012, 48(13): 1898-1900. DOI:10.1039/c2cc16156g |
| [88] |
Maurer SC, Kühnel K, Kaysser LA, et al. Catalytic hydroxylation in biphasic systems using CYP102A1 mutants. Adv Synth Catal, 2005, 347(7/8): 1090-1098. |
| [89] |
Ryan JD, Fish RH, Clark DS. Engineering cytochrome P450 enzymes for improved activity towards biomimetic 1, 4-NADH cofactors. Chem Bio Chem, 2008, 9(16): 2579-2582. DOI:10.1002/cbic.200800246 |
| [90] |
Lu XY, Liu YW, Yang YQ, et al. Constructing a synthetic pathway for acetyl-coenzyme A from one-carbon through enzyme design. Nat Commun, 2019, 10(1): 1378. DOI:10.1038/s41467-019-09095-z |
| [91] |
Chuaboon L, Wongnate T, Punthong P, et al. One-pot bioconversion of L-arabinose to L-ribulose in an enzymatic cascade. Angew Chem Int Ed, 2019, 58(8): 2428-2432. DOI:10.1002/anie.201814219 |
| [92] |
Su HH, Guo ZW, Wu XL, et al. Efficient bioconversion of sucrose to high-value-added glucaric acid by in vitro metabolic engineering. ChemSusChem, 2019, 12(10): 2278-2285. DOI:10.1002/cssc.201900185 |
| [93] |
Zhong C, Wei P, Zhang YHP. A kinetic model of one-pot rapid biotransformation of cellobiose from sucrose catalyzed by three thermophilic enzymes. Chem Eng Sci, 2017, 161: 159-166. |
| [94] |
Hoops S, Sahle S, Gauges R, et al. COPASI-a COmplex PAthway SImulator. Bioinformatics, 2006, 22(24): 3067-3074. DOI:10.1093/bioinformatics/btl485 |
| [95] |
Korman TP, Opgenorth PH, Bowie JU. A synthetic biochemistry platform for cell free production of monoterpenes from glucose. Nat Commun, 2017, 8: 15526. DOI:10.1038/ncomms15526 |
| [96] |
Tian CY, Yang JG, Zeng Y, et al. Biosynthesis of raffinose and stachyose from sucrose via an in vitro multienzyme system. Appl Environ Microbiol, 2019, 85(2): e02306-18. |
| [97] |
Meng DD, Wei XL, Zhang YHPJ, et al. Stoichiometric conversion of cellulosic biomass by in vitro synthetic enzymatic biosystems for biomanufacturing. ACS Catal, 2018, 8(10): 9550-9559. DOI:10.1021/acscatal.8b02473 |
| [98] |
Obert R, Dave BC. Enzymatic conversion of carbon dioxide to methanol: enhanced methanol production in silica sol-gel matrices. J Am Chem Soc, 1999, 121(51): 12192-12193. DOI:10.1021/ja991899r |
| [99] |
Wang XL, Li Z, Shi JF, et al. Bioinspired approach to multienzyme cascade system construction for efficient carbon dioxide reduction. ACS Catal, 2014, 4(3): 962-972. |
| [100] |
Ji XY, Su ZG, Wang P, et al. Tethering of nicotinamide adenine dinucleotide inside hollow nanofibers for high-yield synthesis of methanol from carbon dioxide catalyzed by coencapsulated multienzymes. ACS Nano, 2015, 9(4): 4600-4610. DOI:10.1021/acsnano.5b01278 |
 2019, Vol. 35
2019, Vol. 35