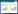中国科学院微生物研究所、中国微生物学会主办
文章信息
- 魏琪瑶, 徐晨晨, 王美艳, 叶海峰
- Wei Qiyao, Xu Chenchen, Wang Meiyan, Ye Haifeng
- 光遗传学工具的开发及其应用研究
- Development and application of optogenetic tools
- 生物工程学报, 2019, 35(12): 2238-2256
- Chinese Journal of Biotechnology, 2019, 35(12): 2238-2256
- 10.13345/j.cjb.190298
-
文章历史
- Received: July 4, 2019
- Accepted: November 29, 2019
- Published: December 5, 2019
细胞自身虽然会产生胞内信号动态变化,但环境的动态变化使细胞接受刺激会随着时间、空间变化作出响应,从而影响胞内信号的动态变化。观察信号通路的动态变化对于信号通路研究有极大的帮助。经典的信号通路研究常常通过基因的抑制/沉默或是过表达以观察信号通路的变化[1]。但这样的研究仅涉及单一条件下的调控,如不同基因表达调控直接导致的胞内信号网络改变的静态结果,而无法研究目标通路在时间或空间动态变化下的调控过程。此外,利用小分子药物或蛋白配体进行的调控由于扩散困难以及脱敏效应等原因而无法实现精确调控。相比之下,光控在时间、空间、强度各方面都具备极大优势。
光遗传学元件的引入推动了人们对于光响应蛋白的研究,它们一般来自植物、真菌、水母、珊瑚、细菌和藻类等[2-4]。光控研究最早在神经科学中被应用,被称为光遗传学。光遗传学是指结合光学与遗传学手段,精确控制特定神经元活动的技术。科学家们将光敏蛋白通过基因融合引入细胞,利用光控系统调控神经活动,在亚细胞水平定位蛋白活动,调控蛋白功能,促进或抑制基因表达,或者诱导蛋白降解(图 1)[5]。
然而,当下光遗传学的定义已经有所扩展,并不局限于神经科学领域,科学家们可以利用和改造多种类型的光敏蛋白控制细胞的活动。光敏蛋白响应特定波长的光,经过一系列构象改变、蛋白间可逆结合以及聚合反应,驱动特异性生理反应,包括(去)激活特异性蛋白活动、激活或抑制转录、亚细胞定位、局部活性氧生产和其他功能(表 1)。随着光遗传学研究的发展,科学家们在体外到体内、细胞组织到动物个体或群体等不同水平引入改造的光控开关进行研究,加深了对生物体微观机理以及相应生物功能的挖掘与理解,因此光遗传学元件的研究将推动着生物学一系列领域的共同发展。
| Opto system | Ta1/2 | Td1/2 | λon (nm) | λoff (nm) | Size (aa) | Chromophore | Mechanism | References |
| ChR2 | 0.2 ms | 15 ms | 470–480 | − | 737 | Retinal* | Ion channel | [4] |
| Bphp1 PpsR2 |
3–30 s | 15 min (dark) |
740–780 | 640–660 | BphP1:732 PpsR2:465 |
Biliverdin* | Dimerization | [8] |
| CRY2 CIBN |
10s (90%) | 12 min | 420–490 | − | CRY2:498 CIBN:170 |
FAD* | Dimerization | [9] |
| pMag nMag |
1.5 s | 6.8 s | < 500 | − | pMag:150 nMag:152 |
FMN* | Dimerization | [10] |
| AsLOV2 | Seconds | 30–50 s | < 500 | − | 143 | FAD* | Caging | [11] |
| PhyB PIF |
1.3 s | 4 s | 650 | 760 | PhyB:908 PIF:100 |
Phycocyanobilin | Dimerization | [12] |
| CRY2 (Oligo) | 30 s | 5.5 min | 420–490 | − | 498 | FAD* | Dimerization | [13] |
| UVR8 COPI |
Hours | Irreversible | 280–315 | − | UVR8:440 COPI:675 |
* | Dimerization | [14] |
| Abbreviations: aa, amino acid; FAD, flavin adenine dinucleotide; FMN, flavin mononucleotide; λ, wavelength; Ta1/2, half-life of association; Td1/2, half-life of dissociation; *endogenous. | ||||||||
近年来,光遗传学工具被广泛应用于人工改造信号通路,成为细胞信号转导中重要的调控元件。光控蛋白主要可分为两大类:视蛋白依赖光受体(Opsin-based photoreceptor)和光敏蛋白(Photosensitive protein)。视蛋白(Opsin)是一种光敏跨膜蛋白,广泛存在于各种生物中,根据其分布特点可分为Ⅰ型微生物类和Ⅱ型脊椎/非脊椎动物类[6]。光敏蛋白属于非视蛋白依赖光遗传学元件,其分子机制是通过吸收激发光的光子能量导致构象变化或者促进蛋白质之间的互作与重排[7]。
对光反应蛋白新功能的扩展将进一步拓宽其应用范围。虽然最佳光反应蛋白的设计取决于实际需求,但它们有一些共性特点:1)易于被遗传编码和表达;2)光响应活性具有较大的动态变化范围,且在黑暗条件下几乎没有或仅有较低的背景噪音;3)对细胞内源性蛋白功能和亚细胞定位造成的影响小;4)光激活波长具备特异性和快速反应性。根据不同作用机理,我们将光敏蛋白分为以下几类。
1.1 光控二聚化系统 1.1.1 光敏色素植物光敏色素是一类可响应红光调节植物生长发育的蛋白。在种子植物中有两类光敏色素——Ⅰ型和Ⅱ型。目前在拟南芥Arabidopsis thaliana中已发现5种属于Phy家族的蛋白(PhyA–PhyE)[15]。其中光敏色素A (PhyA)是唯一一种Ⅰ型光敏色素,其在光照条件下降解,在黑暗条件下积累[16]。区别于其他红光激活的Ⅱ型Phy蛋白,PhyA在716–720 nm远红光条件下被激活,并在与远红光受体伴侣蛋白FHY1 (Far-red elongated hypocotyl1)或FHL蛋白(FHY-LIKE)相互作用下转运入核[17];在红光条件下,核内的重组蛋白解聚,PhA被重新循环至胞浆[18] (图 1A)。
在Ⅱ型光敏色素中,红光是最有效的响应触发光,其工作机理已被深入研究且被广泛应用于合成生物学领域。以光敏色素B (PhyB)为例,其包含两个主要的结构域:N端光吸收结构域和C端效应结构域[19]。光敏色素二聚系统的局限在于Phy的发色团PCB不能在哺乳动物细胞内自然合成[20],需要外源引入。但与PhyA不同的是,PhyB通过结合转录因子PIF或自身核定位信号入核,不需要运输蛋白。在红光照射下PCB介导PhyB可逆构象变化从而结合光敏色素互作因子PIF (图 1A)。同时在远红光(760 nm)照射下可以抑制PhyB与PIF的结合[21]。
光敏色素不仅存在于植物中,近年来科学家们发现细菌中也存在类似的光敏色素。2016年,Vladislav V Verkhusha课题组Kaberniuk等[8]在沼泽红假单胞菌Rhodopseudomonas palustris中发现了细菌光敏色素BphP1可与天然配体PpsR2可逆结合(图 1B),建立了近红外光调控系统。与Phy/PIF系统相比,其优点是所需色素PCB在哺乳动物细胞内天然存在,无需额外引入。BphP1-PpsR2光控系统对740–780 nm近红外光敏感,650 nm红光可以促进BphP1与PpsR2蛋白分离。但只有在完全黑暗的条件下,两蛋白才完全分离[8]。该系统可用于调控基因表达、细胞信号转导、细胞形变与时空控制等[22-23]。但其实验数据表明,该BphP1-PpsR2系统调控基因表达效率较低,限制了其广泛应用。因此后期该课题组Redchuk等[23]科学家对PpsR2蛋白进行人工截短改造从而获得更小的PpsR2变体QPAS1,通过将BphP1-QPAS1系统与蓝光调控元件AsLOV2的LOV结构域结合,建立了蓝光与近红外光分别独立调控的胞内蛋白定位系统。
1.1.2 隐花色素隐花色素(Cryptochrome)最早在拟南芥中发现,主要参与光调节细胞伸长、开花的光周期等。隐花色素2 (CRY2)是一种蓝光响应蛋白,其天然结合配体为具有螺旋-环-螺旋结构的CIB蛋白。在光激活条件下,CRY2与CIB形成二聚物,并且在黑暗条件下二聚体解聚(图 1C)。同时Kennedy等[9]发现,CRY2蛋白中N端光裂合酶同源区域(PHR)与光响应结合CIB紧密相关,其在蓝光调控下可与缺失螺旋-环-螺旋结构的CIB1 (CIBN)或全长CIB1发生聚合或解聚作用。Taslimi等[24]发现一些CRY2的截短版本:CRY2 (515)与CRY2 (535),可以很大程度地降低黑暗条件下二聚作用产生的本底;L348F与W349R位点的突变分别可以减缓或加强CRY2-CIB1二聚物的解聚。此外在蓝光条件下高表达的CRY2蛋白自身可发生寡聚[24-25]。
1.1.3 紫外响应受体8UVR-8 (UV response locus 8)也是一种光敏蛋白,其作为植物UV-B响应信号通路的组成部分,帮助植物适应自然生长过程中的UVB (Ultraviolet B, 280–315 nm),并缓解UV-B产生的DNA损伤[26-27]。相较于其他光敏蛋白,UVR-8的优点在于,它通过色氨酸残基吸收UVB,激活不需要共因子[28],但其劣势在于不可逆性以及UV的较强光毒性和细胞损伤能力。在黑暗条件下,UVR-8以同型二聚体形式存在,而光形态建成调控因子(COP1)是一种E3泛素连接酶,通过结合降解UVB响应转录因子阻遏紫外响应作用的产生;在UVB存在条件下,UVR-8解聚为单体转运到核内,结合COP1的C端WD-40结构域并释放COP1转录结合因子,该过程是不可逆的[29-31] (图 1D)。
1.1.4 变构蛋白变构蛋白二聚化或解聚也可应用于光诱导蛋白互作,一般情况下依赖于LOV结构域。光-氧-电势结构域(LOV)最早是从植物光电感受器phot1中分离出的闭锁结构域(Caging domain),通过响应蓝光调控植物生长[32-33]。
黑暗条件下AsLOV2 (Arabidopsis sativa LOV2)或AtLOV2 (Arabidopsis thaliana LOV2)结构域与C末端螺旋Jα结合;蓝光激活条件下,由于半胱氨酸残基的高度保守,使LOV核心与黄素蛋白FMN之间的非共价作用转变为共价作用,由此发生光诱导构象变化,解除Jα螺旋结构对LOV核心结构域的抑制作用[34-35] (图 1E)。因此可以通过在LOV结构域N端或Jα螺旋的C端融合效应蛋白,调控蓝光释放原来闭锁在LOV结构域中的目的蛋白,从而将该系统应用于光控信号通路的改造[36]。此外,Strickland等[37]通过理性设计在AsLOV中引入4个突变位点,增强了Jα螺旋LOV结构域结合的稳定性。
虽然以上天然含有LOV结构域(光-氧-电结构域)的光受体蛋白本身就具有蓝光诱导异构的能力[26],但大部分情况下使用的是经过人工改造的具有多重异构结构域的变构蛋白。它可以感应光从而对下游通路产生影响。如TULIP系统利用LOV结构域响应蓝光发生构象变化,释放目标蛋白诱导二聚化的产生[38-39]。此外,LOV结构域相关的光遗传学工具开关的开启和关闭灵敏度取决于蛋白融合,对融合的方向和连接蛋白的长度较为敏感。
在真菌的光受体中,VVD是最小的含有LOV结构域的蛋白,其N端螺旋结构在光刺激的条件下发生空间构象变化[40],导致同源二聚化从而在空间上使互相作用的结构域靠近。但天然同源二聚化具有局限性且解聚半衰期长[10, 41],因此后续通过改造VVD蛋白,在其N末端螺旋处加上正电性氨基酸(pMag)与负电性氨基酸(nMag)利用蓝光激活LOV结构域,释放pMag与nMag残基,诱导正负电性的氨基酸相互吸引发生异源二聚化(图 1F);而该系统中带同种电荷氨基酸残基之间存在相互排斥,因此可以很大程度上消减自然情况下的同源二聚化作用[10, 42]。
此外,离子通道相关的光遗传学元件在机制上也属于变构蛋白,其蛋白种类主要属于Ⅰ型微生物类视蛋白家族,常应用于神经光遗传学领域调控神经元功能[4]。例如ChR2 (Channelrhodopsin-2)是一种光门控非特异性阳离子通道,可由蓝光介导使Na+、Ca2+、K+等阳离子泵入细胞内,Deisseroth课题组将其用来控制神经元活动[43-44] (图 1G)。此外,Kushibiki等通过在小鼠胰腺β-细胞中表达ChR2,建立了β-细胞分泌胰岛素的光依赖通路,并证明利用蓝光激活ChR2通道诱导胞内储存的Ca2+释放内流到小鼠胰腺β-细胞中,产生信号级联,可调节体内血糖水平[45]。
1.2 寡聚系统隐花色素CRY2PHR除了可与其结合蛋白CIBN在蓝光刺激下发生二聚作用以外,2013年,Bugaj等[25]发现CRY2自身具有寡聚作用(图 1H),并且在10 s内即在动物细胞中产生肉眼可见的蛋白质聚集,蛋白聚集量随着光照强度的增强与光照时间的增长而增多;在黑暗条件下,CRY2蛋白聚集随时间指数级减少,半衰期小于5.5 min。
该CRY2寡聚系统可应用于时空调控目的蛋白同源寡聚化,从而探究蛋白间相互作用以及细胞信号通路;并且该寡聚系统的稳定与否直接与CRY2蛋白浓度以及标记蛋白的种类和结构相关[46]。2017年,Park等[47]通过与CRY2融合表达不同的荧光蛋白发现融合蛋白或标记蛋白的四级结构对CRY2的寡聚效率存在显著影响,并且利用短肽标记构建了稳定高效的CRY2聚合系统(CRY2clust),该系统能够有效地调控活细胞在对光的响应过程中的精确胞内信号。但目前该系统CRY2蛋白寡聚机理仍未被揭示,并且无法有效应用于分子量较大的蛋白寡聚化调控,因此,该系统仍有很大的探索空间。
2 光控细胞信号通路在细胞的增殖、分化、衰老、凋亡等生理过程中,细胞信号通路起着主要调控作用且介导细胞执行一系列生物功能。细胞信号通路的相关研究是目前生物科学研究的主流,且功能性蛋白和信号网络间的互作关系的探究已获得一定成果与进展。然而,传统研究方法在空间与时间上缺乏灵活调控且应用面较为狭隘,因此导致细胞信号通路的探究仍具有很大局限性。
通过合成生物学手段人为构建改造的胞内信号通路,在原有信号通路中引入光遗传学元件来替代胞内响应蛋白和基因或作为额外的调控因子感受和响应外界刺激,可实现时空特异性的调控,也可通过光控蛋白调控多元信号通路探索信号网络间的联系。采用光遗传学改造胞内信号通路的应用已被广泛报道,其关键是选择合适的元件进行调控,以下举例阐述几条主要的胞内信号通路以及光遗传学工具在通路改造中的应用。
2.1 丝裂原活化蛋白激酶(MAPK)通路丝裂原活化蛋白激酶(Mitogen-activated protein kinase,MAPK)通路及其引起的一系列信号级联效应调控着细胞增殖、分化、凋亡等生理过程。MAPK的信号级联在胞内可被大量的细胞表面受体激活,如受体酪氨酸激酶(RTK)、细胞因子受体和G蛋白偶联受体(GPCRs);并且该信号级联主要由MAPK3K、MAP2K和MAPK相互激活、产生信号放大效应[48]。
MAPK信号通路相关的光遗传学改造最早以酵母为底盘细胞,成功实现了蓝光调控膜招募支架蛋白Ste5,从而激活下游一系列MAPK信号级联效应,在生物功能上表现为对酵母生长的抑制[38, 49] (图 2A)。在动物细胞中,Toettche等[50]建立了红光响应的Opto-SOS系统,通过引入光控系统PhyB-PIF (光敏色素互作因子)从而招募SOS至细胞膜,实现红光诱导激活MAPK信号级联反应(Ras/Raf/MEK/ERK) (图 2B)。
|
| 图 2 光控蛋白与细胞信号通路 Fig. 2 Photo-responsive proteins and relevant cell signaling pathways. (A–C) The MAPK pathway. (A)The TULIPs system fuses the ePDZ domain and the yeast scaffold protein Ste5 to optically control the MAPK pathway in yeast. In the dark, a peptide epitope is caged by docking of the Jα helix to the LOV core, while under the illumination of blue light, the Jα helix undocks and exposes the peptide epitope to recruit ePDZ-Ste5. The TULIPs system realizes the plasma membrane recruitment of Ste5 to activate a series of biological effects aroused by MAPK activation in budding yeast. (B) The Opto-SOS system is an engineered light-gate switch which can drive Ras activation. The membrane-localized Phy protein responds to red light driving heterodimerization with a cytoplasmic PIF-tagged SOScat construct. The membrane recruitment of SOScat leads to Ras activation and nuclear translocation of BFP-Erk2 to have impact on downstream of MAPK pathway. (C) Upon blue light stimulation, the membrane anchored CIBN interacts with CRY2PHR domain to recruit cytoplasmic CRY2PHR -Raf1 to the plasma membrane. Membrane recruitment of Raf1 should subsequently activate relevant MAPK cascades such as MEK and ERK. Without light activation, ERK is cinactivated because of the dissociation of CIBN-CRY2PHR which returns Raf1 to the cytoplasm. (D) Apoptosis caspase pathways. The system uses upconversion nanoparticles (UCNs) as a plasmid DNA carrier and a nanotransducer to convert external near-infrared (NIR) light to local blue light. Upon NIR illumination, the cytoplasmic CRY2-FADD can bind to membrane anchored fusion protein Fas-CIB because of the recruitment between protein CYR2 and CIB under local blue light activation. The interaction between Fas and FADD triggers Caspase cascades in cell leading to apoptosis. (E–F) PI3K-AKT signaling pathway. (E) When activated by red light, the membrane-fused fusion protein PhyB-mCherry-CAAX can recruit PIF-tagged protein iSH-PIF, the iSH domain of which is a constitutive binder of endogenous PI3K. The membrane recruitment of PI3K leads to upregulation of endogenous PI3K activating PI3K derivative AKT pathway. (F) Opto-Trk system. ED, extracellular domain; TKD, tyrosine kinase domain; CRYPHR, the photolyase homology region of CRY2c; and mCit, mCitrine. Blue light illumination drives the oligomerization of CRYPHR domain leading to the activation of the linked TrkB receptors which can further trigger PI3K-AKT signaling activation and regulate neurite outgrowth. (G) Rho family of GTPases. FKF1 contains a LOV domain that detects 450 nm red light. Illumination with red light induces fusion protein FKF1-Rac1 to bind to the membrane anchored nuclear protein GI resulting in the membrane recruitment of Rac1, small GTPase1. Subsequently, the covalent bond between FMN and FKF1 is hydrolyzed returning the LOV domain to its resting state followed by the dissociation of FKF1-GI complex. (H) STING signaling pathway. Upon light stimulation, endogenous GTP can be converts into c-di-GMP by diguanylate cyclase from NIR light-actived protein BphG1 or far-red light-responsive protein BphS. The upregulation of c-di-GMP level stimulates STING receptor leading to the dimerization of TBK1 which drives the phosphorylation and dimerization of free interferon-regulatory factor 3. Such dimerization and phosphorylation causes the translocation of IRF3 into nuclear to control the transcription of target gene. |
| |
此外,蓝光调控系统CRY2-CIBN也被应用于Raf/MEK/ERK信号通路的改造,该系统中CIBN定位于细胞膜,CRY2PHR与Raf1融合表达,通过蓝光激活调控蛋白CRY2PHR与CIBN二聚化招募Raf1激酶靶向细胞膜,从而激活下游激酶[51] (图 2C);Kim等则通过在FGFR (成纤维细胞生长因子受体)上引入可响应蓝光发生寡聚化的CRY2PHR蛋白,改造并建立了Opto-FGFR1系统。在蓝光激活条件下,FGFR二聚化并自磷酸化,从而对下游MAPK、PI3K、PLC等激酶产生影响,在Opto-FGFR1系统中表现为对FGFR受体的调控从而调控细胞的极化与迁移[52-53]。
2.2 细胞凋亡信号通路(Caspase cascade)程序性细胞死亡(Programming cell death,PCD),又称凋亡,是一种由基因编码的存在于所有细胞中的信号通路,在死亡信号介导下诱导细胞主动有序地生理性死亡。细胞凋亡是维持细胞内稳态的关键,凋亡通路的失调可能导致许多疾病,如癌症、自身免疫病和神经退行性疾病等,因此对于细胞凋亡通路的人为调控也是突破癌症等疾病治疗的关键所在[54]。
在PCD过程中,半胱氨酸-天冬氨酸蛋白水解酶(Caspase)是决定性的调控因子,根据不同的Caspase家族可分为执行者和效应者,对凋亡通路的级联起着重要作用。Mills等[55]通过引入LOV2结构域,构建了蓝光响应Opto-caspase-7系统,非激活状态下LOV2闭锁的空间结构抑制caspase-7活性,蓝光照射下诱导LOV2结构域构象变化释放caspase活性,并且系统可以在1 h内高效地引起细胞凋亡过程。Zheng等[56]通过将稀土上转换发光纳米颗粒(UCNPs)与蓝光调控系统CRY2-CIB相结合,建立了受红光间接调控的凋亡途径,实现调控凋亡通路下游一系列Caspase级联效应用于肿瘤治疗(图 2D)。但该系统中光转换纳米颗粒存在潜在的不稳定性与不可估测性,因此在临床肿瘤治疗中还存在诸多待解决问题。
2.3 磷脂酰肌醇3-激酶/蛋白激酶B通路(PI3K/AKT)PI3Ks激酶可以使磷脂酰肌醇磷酸化从而产生PIP3,PIP3则可激活下游相关通路AKT、PKC、Rac/actin来调节细胞生长、生存、迁移和细胞周期等过程[57-58]。同时控制着胞内多条重要的生存信号级联反应的AKT信号通路,其中蛋白激酶B (AKT/PKB) N端的PH结构域响应膜上PIP3水平的变化,随着PIP3水平的上升,PKB被激活并引发一系列细胞效应如细胞迁移极化、增殖和分化[59]。
利用光遗传学方法改造的PI3K/AKT通路目前已分别在红光系统与蓝光系统中实现。红光调控的PI3K/AKT通路通过利用光遗传学元件PhyB-PIF6,引入融合表达的PIF6因子与PI3K结合蛋白p85α的SH2结构域,从而在红光条件下膜招募SH2结构域,进而招募PI3K激酶,上调PIP3水平并激活下游AKT通路[60] (图 2E)。Chang等[61]利用蓝光响应蛋白CRY2的寡聚效应,将TrkB (原肌球蛋白受体激酶B)与CRY2PHR (CRY2光解酶同源区域)融合表达构建了Opto-trkB系统(图 2F),实现AKT/PI3K等酶表达水平的上调。此外,CRY2-CIB1系统也被用于改造PI3K/AKT信号通路,目前已建立了Opto-ish2、Opto-5-磷酸酶等系统,可分别用于调控网格蛋白介导的内吞作用以及细胞骨架的重组[62]。
2.4 Rho家族GTP酶相关通路Rho家族GTP酶是真核细胞中调控多条信号转导通路的分子开关,可以诱导细胞极化,调控微管动力学、膜运输通路、细胞器发育等,其中GTP酶最主要的生物功能是调控肌动蛋白细胞骨架的形成[63]。目前研究最为广泛的3种GTP酶分别为Rac1、Cdc42和RhoA,且它们之间的调控作用存在着非常紧密的联系——Cdc42可激活Rac1,Rac1与RhoA互相拮抗[64],3个酶对细胞骨架动力学调控具有重要影响。
光控Rho GTP酶系统的激活主要依赖于鸟苷酸交换因子(GEFs)的光响应膜招募过程,通过将光敏蛋白PhyB锚定于胞膜,融合表达PIF与多种GEFs (Rac1/Cdc42/Rho)从而构建Opto-GTPase,在红光调控下产生一定生物效应,如Tiam蛋白(Rac GEF)与交叉蛋白(Cdc42 GEF)的招募促进细胞产生板状与丝状伪足,Tim蛋白(Rho GEF)的膜招募可诱导细胞体积的收缩[7]。Yazawa等[65]利用拟南芥的FKF1-GI蛋白建立了蓝光激活二聚化系统(BLAD),可招募小G蛋白Rac1至细胞膜,同时诱导光照区域局部形成板状伪足(图 2G)。此外,LOV-PDZ以及LOV变构系统(LOV uncaging system)也被应用于Rho GTP酶的改造[11, 37-38]。
2.5 STING信号通路干扰素刺激因子(Stimulator of interferon genes, STING),也称为MITA或MPYS,由TMEM 173基因编码,是一种与内质网(ER)相关的信号分子,可识别细胞质内异常DNA或环状二核苷酸(CDNs),从而调控激活多种宿主防御基因的转录,其中包括Ⅰ型干扰素(IFNs)和促炎症细胞因子等[66-68]。早期研究表明,STING是人类天然免疫中的一种新分子,可响应入侵的DNA病毒、细菌以及转染的DNA,从而激活内源免疫相关基因的转录[69-70];进一步研究发现STING可作为环二核苷酸传感器(cGAMP,c-di-GMP,c-di-AMP),通过cGAMP合成酶(cGAS)介导合成cGAMP,从而可以检测胞浆DNA及胞内病原体释放的第二信使(c-di-AMP,c-di-GMP)[71]。STING被激活后在TKB1激酶(Tank-binding Kinase 1)介导下分别使干扰素调节因子3 (Interferon-regulatory Factor 3, IRF3)与核因子(Nuclear factor-κB, NF-κB)磷酸化,从而使这些转录因子入核激活下游内源基因转录[72-73]。
2014年,Folcher等[74]通过改造STING通路,利用脑电图脑-机接口BCI (EEG-based brain-computer interface)开发了光诱导连接的无线电植入装置来调控分泌型碱性磷酸酶SEAP (Secreted alkaline phosphatase)的表达。该人造STING通路包含的元件有红光响应的细菌BphG1突变体蛋白、细菌二鸟苷环化酶DGCL结构域(Diguanylate cyclase)以及STING受体蛋白,可用于检测细胞内c-di-GMP水平并调控干扰素IFN-β启动子激活,驱动特定转基因的转录(PIFN(AC+)-SEAP-pA) (图 2H)。
2017年,本课题组利用合成生物学技术将STING通路改造为光控信号通路,利用干扰素基因刺激因子(STING)作为细胞内环二核苷酸传感器的原理,采用细菌光敏蛋白BphS (c-di-GMP合酶)以及c-di-GMP的专一磷酸二酯酶YhjH调控信号通路[75-77],将IFN的响应元件((hIFN-RE)- (ISRE)3)作为GLP-1和Insulin的启动响应元件,建立了远红光响应的GLP-1和Insulin分泌系统用于糖尿病治疗[76] (图 2H)。但该系统的弊端在于对内源性STING通路的严格依赖,系统易与人体免疫的重要内源性过程(如环鸟苷的激增)产生交叉干扰,在应用上有潜在不可控性。因此,我们后续改进并提出了正交性更强、调控性能更好的改进系统,详见3.3。
3 光控细胞信号通路的应用生物体的发展高度依赖于空间和时间的信号级联与基因表达的调节,一个信号分子的定位可能引发细胞不对称分裂或向组织层的特异性迁移,特定时间的基因表达也可能决定细胞分化过程中的命运,异常的时空变化产生的信号配体或基因表达可能是癌症、遗传性相关疾病的基础。通过光遗传学工具能够精确了解到至少刺激多少特定种类的神经元才能启动目标生物体,并能确定神经元对特定神经递质永久脱敏的信号剂量或持续时间;同时光遗传学工具具备精确调控目标通路的空间、时间和强度的能力。因此,光遗传学工具在细胞信号通路中的引入能够在多种基础研究如神经生物学(调控神经兴奋的发生、突触可塑性研究)、基因修饰与调控、干细胞再生与分化、细胞信号通路和蛋白定位等研究中提供定制化的技术支持,并能够突破传统研究的限制,推动其他相关领域的技术发展,如光感蛋白的发现,显微技术、成像技术和微创技术的发展等。此外,目前已经解析了异常的细胞信号通路变化与疾病间联系,而如何实现精确调控细胞信号通路将基础研究的成果转化到医学领域,光控细胞信号通路可能会成为这一突破的关键所在。
3.1 光控在神经生物学研究中的应用光遗传学最早就被应用于调控神经兴奋,而实现对突触的调控是也光遗传工具目前应用最深入的研究领域[78]。2005年,Karl Deisseroth等在哺乳动物神经元中引入ChR2,实现了蓝光光照下1 ms内引发单个神经兴奋和一系列神经兴奋,在当时引起了巨大的轰动。由此衍生出的研究工作也被广泛报道,如全息光学刺激视网膜神经节细胞恢复视力[79]以及光遗传学工具在测量微秒级亚细胞电生理的发展等。2019年Claire N. Bedbrook[80]等结合机器学习建立计算机模型从而改造了大量ChR变体,其中变体ChRger1、ChRger3展现出优良的神经系统的光遗传学激活,尤其ChRger2无需光纤植入即可实现神经元的光遗传学激活,初步解决了哺乳动物颅内光遗传学外源电子元件植入带来的问题。
3.2 光控系统在基因表达调控中的应用光遗传是一种能够实现时空特异性地调控基因表达(转录、翻译和基因编辑)的强大工具。不同于传统的扩散性高、控制性低的化学诱导剂,特殊波长的光触发基因调控系统能够实现精确正交调控,且无明显脱靶效应。Shimizu-Sato等[81]通过在光敏色素上融合Gal4-DNA结合域(Phy-GBD),在Gal4-DNA激活域上融合PIF3 (PIF3-GAD),实现在红光照射下Phy-GBD结合PIF3-GAD融合蛋白,从而在Gal4启动子控制下刺激基因转录;远红光则能关闭转录。Ravi S Kane等[82]利用CRY2PHR-CIBN蛋白在蓝光下使翻译激活蛋白eIF4E定位至目标mRNA,从而激活其翻译过程。
然而上述方法都存在一个限制因素:需要向胞内基因外源引入复杂的启动子区域。近期,光控研究也被用来招募效应器到内源性基因组位点而应用于基因转录调节和基因组编辑。Polstein等[83]利用Gigantea和FKF1相互作用的光控系统,引入锌指结构DNA结合蛋白(Zinc finger protein,ZFP)以招募VP16转录激活子至目标DNA序列,并在蓝光下开启转录过程。Konermann等[84]设计了利用转录激活因子类似效应器DNA结合域和转录激活子VP64结合构建的CRY2-CIB1光控系统,实现修改组蛋白乙酰化,从而展现出其潜在的协调表观遗传状态和翻译后修饰的功能。
虽然调控基因表达的Cre/loxP、CRISPR/Cas9等系统能够高效调控基因表达,但想要对系统进行更加精确的调控,实现在时间、空间上特异性调控基因表达,光遗传学元件给了人们很多启发。
3.2.1 光控Cre/loxP系统调控基因表达Cre/loxP系统来源于F1噬菌体,能够介导位点特异的DNA重组。该系统含有两种组分:loxP位点(Locus of X-over P1)和Cre重组酶(Cyclization recombination)。loxP位点作为重组酶识别的位点,是一段长34 bp的DNA序列,含有两个13 bp的反向重复序列和一个8 bp的核心序列;Cre重组酶是一种由343个氨基酸组成的单体蛋白,生物活性较稳定,能在生物体不同的组织内、不同的生理条件下引发loxP位点的DNA重组。Cre/loxP系统不需要细胞或者生物体提供其他的辅助因子,重组酶的编码基因受响应启动子调控。
近期Won Do Heo课题组开发了一种高度光敏和高效的光活化Flp重组酶(PA-Flp)[85],可用于体内基因调控。该系统中重组酶PA-Flp可由病毒载体介导与Cre-loxP系统结合,形成Flp依赖性Cre表达系统,可同时激活Flp和Cre;并且实验证明了PA-Flp依赖性、Cre介导的内侧隔膜中的Cav3.1沉默增加了小鼠的物体探索行为。PA-Flp是非侵入性的、高效的、易于使用的光遗传模块,其高度光敏特性能通过无创发光二极管照明激活小鼠大脑深部区域,为神经科学研究提供了无副作用和可扩展的基因操作工具[86]。
3.2.2 光控CRISPR/Cas9系统调控基因编辑CRISPR/Cas9系统是细菌在长期演化过程中为了对抗入侵的病毒及外源DNA而形成的一种适应性免疫防御[87]。在利用CRISPR/Cas9进行基因编辑过程中,可以通过外源导入Cas9和sgRNA对靶基因进行剪切从而实现基因定点敲除、突变或敲入。
有关光响应的CRISPR/Cas9基因编辑方式已经有了很多报道。如单体无活性的Split Cas9系统可融合光开光蛋白pMag和nMag后在光照下使Cas9活性重组[88]。2018年Felix Bubeck等利用LOV2蓝光元件,引入了李斯特菌Listeria monocytogenes原噬菌体中的AcrIIA4蛋白作为效应蛋白改造了抗CRISPR变体(Anti-CRISPR)。在蓝光刺激下,该CASANOVA系统可以很容易地在Cas9抑制状态和活性状态之间进行精确地外部切换,可用于研究活细胞中Cas9 DNA靶向定位的动力学。
光控的Cas9系统改变了过去单向的、不可控的基因编辑,实现了时空特异性的、非侵入性的、可控的基因编辑模式,扩展了基因编辑工具箱。
3.2.3 光控CRISPR/dCas9系统调控基因表达dCas9是Cas9突变(D10A和H840A)后失去核酸酶活性的蛋白,但其仍具有结合基因组的能力,因而将dCas9与其他效应蛋白融合可用于内源基因修饰、调控以及DNA位点的标记等[89-90]。为了实现特定时间、特定细胞的基因调控表达,Cas9或dCas9系统被不断优化。近几年里很多化学药物调控dCas9系统不断被提出,但多数化学药物调控存在药物扩散难以控制、反应速度慢、反应效率低、毒性大、无法满足时空特异性和可逆调控的问题。而光在时间、空间、强度各方面都能很容易地实现调控,因而能够很好地解决化学药物调控存在的一些问题。一些光响应蛋白对特异性波长光触发的基因调控系统能够实现精确正交调控。
近期研究已经开发了光响应的CRISPR/dCas9基因编辑方式。2015年有研究报道[75],dCas9介导的蓝光响应系统被用于靶向内源基因组位点调控其转录。融合CIB1的dCas9在sgRNA引导下靶向内源基因组位点,融合转录激活子的CRY2在蓝光响应下与CIB1结合,从而调控目标基因转录。靶向内源基因修饰和转录表达的光响应系统在近几年已经有较多的研究报道[91-92]。
3.3 光控系统在干细胞研究中的应用光遗传学元件也可应用于干细胞的改造,通过内源性转录因子组成的通信网络控制细胞的可塑性、多能性,并且该网络可持续对内环境与信号作出反应从而重调细胞类型特异性基因表达[92-93]。传统干细胞的诱导需要一系列生长因子的激活,其过程低效、复杂且可重复性低,通过光遗传学手段在基因或信号水平控制分化关键基因可有效解决该问题。例如Sokolik等[94]利用蓝光响应VVD结构域的二聚化,上调神经分化因子Brn2的表达,高表达的Brn2可竞争性抑制Oct4、促进Sox2上调从而诱导神经分化基因的表达[94-95]。本课题组通过引入外源光敏蛋白(BphS)以及相关响应蛋白(BldD),构建了完全绝缘的远红光响应的人造基因回路调控转录激活子,进而构建了远红光调控的CRISPR-dCas9系统(Far-red light-activated CRISPR-dCas9 effector, FACE)[96]。该系统可高效诱导内源或外源基因表达,通过上调单个神经转录因子NEURON2有效促进多能干细胞向功能神经元的分化。此外,利用干细胞改造原理建立人工改造肿瘤干细胞样细胞(CSC),其中天然CSC细胞是诱导肿瘤复发的关键因素,通过光遗传系统调控Sox2、OCT4、KIf4和c-Myc的表达可以控制分化细胞转化为iCSC,可应用于筛选CSCs靶向药物,为进一步研究肿瘤药物抗性以及肿瘤再生提供技术支持[97-99]。
3.4 光控细胞信号通路与疾病治疗随着合成生物学技术的发展,基因治疗与细胞治疗成为了新一代药物研发的潜力股。通过结合光遗传学方法与合成生物学理念而建立的精准治疗的创新体系,能够改善传统药物或基因治疗的失准或脱靶效应,并且现已有多种体系实现了以光为调控因素,在体外与体内水平达到较好的时空特异性与细胞行为学调控[100-101]。此外,通过结合交叉领域生物电子药物技术与原理,从而系统性地将分子生物学、细胞生物学、生物工程、电子信息技术串联一体,建立智能化药物诊疗体系。目前,光控细胞信号通路在癌症、糖尿病以及神经性疾病(见3.1)等重大疾病中都有良好的进展。
在癌症治疗方面,近几年来免疫疗法如过继细胞疗法等技术迅速发展,在多种癌症治疗的临床应用中呈现出显著疗效,但传统细胞疗法存在组织特异弱、穿透性差等局限[102-104]。而引入光遗传学系统可以精确调控免疫细胞有效识别肿瘤特异性抗体从而消除肿瘤细胞;应用特定光谱的光遗传学工具,可以一定程度上提高组织穿透能力,有效解决了传统细胞疗法存在的问题。He等[105]建立了Opto-CRAC系统(光控钙离子释放-激活钙离子通道),通过引入响应蓝光发生构象变化的LOV2结构域来模拟基质互作分子(STIM)自然体内构象变化的过程,从而通过光诱导LOV2释放STIM与膜上CRAC蛋白ORAI1结合,产生Ca2+并且介导免疫细胞内一系列特征性钙依赖反应(图 3A)[106-107]。该系统除了可用于精确控制CAR-T细胞中的Ca2+/NFAT信号;引入Opto-CRAC系统改造的细胞与小鼠模型都呈现树突状细胞加速成熟且抗原递呈能力上调的特点,从而促进T细胞活化[105]。另一基于CAR-T (Chimeric antigen receptor T-cell immunotherapy)治疗的光控系统PA-CXCR4,利用趋化因子受体与光响应视紫红质之间的结构与功能关系,通过505 nm光激活诱导产生胞内趋化因子的信号,控制T细胞迁移与运输(图 3A)。Xu等通过体外实验证明经过PA-CXCR4改造的CD8+ T细胞在光照条件下,能显著减缓肿瘤细胞增殖[108]。此外2019年Molly E Allen等[109]引入逻辑门-“与”门,设计了TamPA-Cre系统。通过将蓝光响应蛋白(pMag, nMag)与他莫昔芬(Tamoxifen)依赖性核定位结构域整合Split-Cre重组酶,构建了药物小分子他莫昔芬与蓝光共同调控嵌合抗原受体的表达,实现CAR-T细胞的特异性调控,进一步改善了CAR-T治疗脱靶的问题。

|
| 图 3 光控细胞信号通路的改造应用 Fig. 3 Engineered optogenetic signaling pathway. (A) (left) Opto-CRAC system and cancer therapy. To simulate a conformational switch in STIM1 in cell, LOV2 domain is fused with STIM. Upon blue light illumination, LOV2 domain undocks and releases STIM which can bind to membrane-localized CRAC protein ORAI1to trigger Ca2+ influx and Ca2+-dependent responses in immune cells. With light activation, secretion of cytokines can be upregulated in opto-CRAC engineered DCs, TILs, and macrophages (Mac) to kill tumor cells. (right) PA-CXCR system. Photoactivatable rhodopsin (Rhod)-CXCR4 chimeric chemokine receptor engineered T cell can traffick and kill tumors with manipulation of light. (B) The FACE system controls stem cell differentiation. Based on endogenous STING signaling pathway, the FACE system utilizes bacteria diguanylate cyclase-BphS to convert free GTP into c-di-GMP which can bind to the domain of BldD leading to the dimerization and formation of the FRL-dependent transactivator FRTA (p65-VP64-NLS-BldD) when illuminated by far-red light. The FRTA translocates into nuclear and binds to the chimeric promoter PFRLx to initiate the expression of the FRL-inducible genome transactivator (FGTA4) by assembling MS2, p65, and HSF1 recruited by the MS2 box of the sgRNA-dCas9 complex. The FACE system optically controls the MS2-mediated recruitment of transcriptional activators to improve the activation efficiency of dCas9-targeted endogenous genes. |
| |
影响着全球约4.6亿的不死“癌症”——糖尿病,其引起的一系列并发症以及慢性疾病的无法根治性带来的痛苦不亚于肿瘤[110]。糖尿病治疗的传统手段是通过严格的饮食控制和终生注射胰岛素(Ⅰ型糖尿病)或GLP-1 (Ⅱ型糖尿病)来实现血糖稳态的控制。目前运用合成生物学思路建立的光控糖尿病治疗系统已实现。2011年,本课题组[111]构建了蓝光调控光遗传学装置来调控血糖内稳态,通过在细胞中异位表达蓝光响应G蛋白偶联受体黑视素(Melanopsin),在蓝光响应下可以介导一系列胞内信号级联磷酸化激活转录因子NFATs,因此该系统可以通过光控NFAT控制特异性GLP-1启动子来驱动GLP-1的转录与表达。华东理工大学杨弋课题组[96]构建了另一光控胰岛素分泌的系统,利用蓝光激活下LOV蛋白VVD结构域二聚化,通过将DNA结合结构域Gal4 (1–65)与VVD结构域、转录激活结构域VP16融合表达构建了GAVV。蓝光条件下GAVV二聚化从而入核结合启动子Gal启动胰岛素的转录与表达。近期Zhang等[112]将光敏腺苷酸环化酶(PAC)引入胰腺β细胞中,通过蓝光激活上调胞内cAMP水平,从而增强葡萄糖刺激胰岛素分泌过程(Glucose-stimulated insulin secretion, GSIS)来实现控制血糖稳态。
为了解决蓝光组织透性低、光毒性较大、体内应用局限性大等问题,本课题组[76]成功构建了一种远红光调控转基因表达的控制开关。利用红细菌中响应远红光的受体蛋白BphS作为感受器, BphS在远红光照射下可产生c-di-GMP分子。进一步设计合成了c-di-GMP调控的杂交型转录激活子p65-VP64-BldD及其相对应的启动子,最终获得了远红光调控基因表达的光控基因线路(图 3B)。最后通过利用合成生物学、光遗传学、电子软件工程等多学科交叉技术,巧妙地开发了一种集糖尿病诊断和治疗为一体的半自动化诊疗新系统(图 4)。首次实现通过智能手机APP超远程调控胰岛素表达就能实现远程控制血糖稳态的目的。这一研究将为糖尿病患者提供极大的便捷性,颠覆了传统口服和注射药物治疗糖尿病的方法。这种通过无线信号超远程控制药物精准释放的电子药物胶囊系统将是未来电子药物的新趋势。
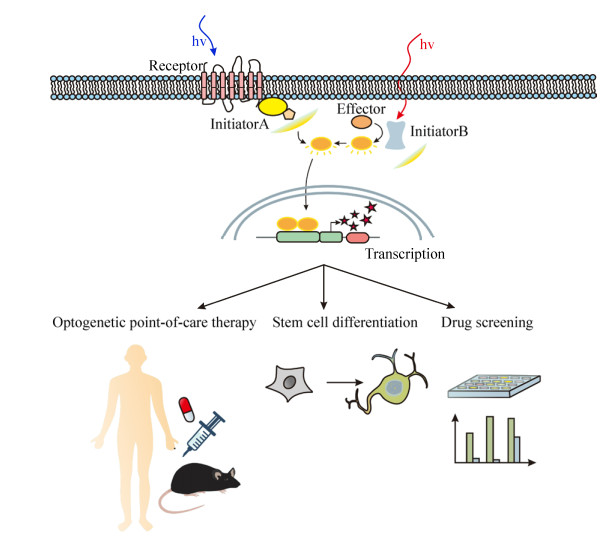
|
| 图 4 人工改造光控信号通路应用于精准化治疗、干细胞诱导分化、药物筛选的示意图 Fig. 4 Schematic diagram of the application of engineered optogenetic signaling pathway for point-to-care therapy, stem cell differentiation and drug screening. |
| |
光控细胞信号通路的研究对现代生物学具有深远意义。目前,光控改造细胞信号通路的应用已扩展到实现控制蛋白质与蛋白质的相互作用、观察信号网络动态、上调或下调基因表达或蛋白活性、亚细胞定位甚至走向转化医学与疾病治疗的道路。
时空特异性是光遗传学工具的最大优势,这一特性恰好弥补了传统研究方法中的缺点和劣势。但是,光控系统也不是完美无缺,目前主要面临着移植元件的组织损伤性、光毒性以及光控特异性与高效性等问题。尽管现有的手段已经在不断降低元件植入的创伤,但仍无法完全忽略创口给实验或是临床应用带来的影响。同时,应用在活体生物内时,需要考虑不同光遗传学工具对应不同光谱,其组织穿透能力的差异也是一个极大的影响因素。此外,在照射细胞一段时间后,光毒性仍是一个需要考虑的因素。而且光响应蛋白的大小、特性对光遗传学工具的开发也均有着很大的影响。
针对以上问题,为了实现更加安全精准的调控,光遗传学工具的开发目标应集中于研发设计更为精确、智能化的控制手段,例如结合显微技术、医学影像技术、软件工程、电子信息工程等交叉领域实现宏观元件对微观分子水平的高效调控。近年来结合交叉领域优化光控系统在一定程度上改善了光控的部分问题。一方面,光学元件、软件编程和电子信息技术的发展为可控光源提供了技术支持[113-117];动物手术和临床手术技术的不断优化使微创成为可能;另一方面,一些灵敏度高、响应强烈以及毒性低、组织穿透性强的光控系统也不断地被开发出来[118-119]。值得关注的是,近几年来研究者们在尝试整合多个光发生系统实现多方向、多方面地调控细胞信号网络。利用不同波长的光设计构建的多个光调控系统将能用来探索两个信号通路、一个通路中的两个节点或多个蛋白激活的组合、顺序或正交激活的下游效应等[120]。
近年来光遗传学领域在工具开发和扩展方面有了很大的发展,为细胞在不同空间和时间的动态变化的研究提供了新的研究手段,也为细胞信号通路的基础研究提供了强有力的技术支持,对未来在转化医学方面的研究也有着广阔的应用前景。
| [1] |
Fenno L, Yizhar O, Deisseroth K. The development and application of optogenetics. Ann Rev Neurosci, 2011, 34: 389-412. DOI:10.1146/annurev-neuro-061010-113817 |
| [2] |
Ellis-Davies GCR. Caged compounds: Photorelease technology for control of cellular chemistry and physiology. Nat Methods, 2007, 4(8): 619-628. DOI:10.1038/nmeth1072 |
| [3] |
Nerbonne JM. Caged compounds: Tools for illuminating neuronal responses and connections. Curr Opin Neurobiol, 1996, 6(3): 379-386. DOI:10.1016/S0959-4388(96)80123-1 |
| [4] |
Boyden ES, Zhang F, Bamberg E, et al. Millisecond-timescale, genetically targeted optical control of neural activity. Nat Neurosci, 2005, 8(9): 1263-1268. DOI:10.1038/nn1525 |
| [5] |
Hegemann P, Nagel G. From channelrhodopsins to optogenetics. EMBO Mol Med, 2013, 5(2): 173-176. DOI:10.1002/emmm.201202387 |
| [6] |
Deisseroth K. Optogenetics: 10 years of microbial opsins in neuroscience. Nat Neurosci, 2015, 18(9): 1213-1225. DOI:10.1038/nn.4091 |
| [7] |
Zhang K, Cui BX. Optogenetic control of intracellular signaling pathways. Trends Biotechnol, 2015, 33(2): 92-100. DOI:10.1016/j.tibtech.2014.11.007 |
| [8] |
Hegemann P, Nagel G. From channelrhodopsins to optogenetics. EMBO Mol Med, 2013, 5(2): 173-176. DOI:10.1002/emmm.201202387 |
| [9] |
Kennedy MJ, Hughes RM, Peteya LA, et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods, 2010, 7(12): 973-975. DOI:10.1038/nmeth.1524 |
| [10] |
Kawano F, Suzuki H, Furuya A, et al. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun, 2015, 6: 6256. DOI:10.1038/ncomms7256 |
| [11] |
Wu YI, Frey D, Lungu OI, et al. A genetically encoded photoactivatable Rac controls the motility of living cells. Nature, 2009, 461(7260): 104-108. DOI:10.1038/nature08241 |
| [12] |
Shao JW, Wang MY, Yu GL, et al. Synthetic far-red light-mediated CRISPR-dCas9 device for inducing functional neuronal differentiation. Proc Natl Acad Sci USA, 2018, 115(29): E6722-E6730. DOI:10.1073/pnas.1802448115 |
| [13] |
Taslimi A, Vrana JD, Chen D, et al. An optimized optogenetic clustering tool for probing protein interaction and function. Nat Commun, 2014, 5: 4925. DOI:10.1038/ncomms5925 |
| [14] |
Favory JJ, Stec A, Gruber H, et al. Interaction of COP1 and UVR8 regulates UV-B-induced photomorphogenesis and stress acclimation in Arabidopsis. EMBO J, 2009, 28(5): 591-601. DOI:10.1038/emboj.2009.4 |
| [15] |
Mathews S, Sharrock R. Phytochrome gene diversity. Plant Cell Environ, 1997, 20(6): 666-671. DOI:10.1046/j.1365-3040.1997.d01-117.x |
| [16] |
Li JG, Li G, Wang HY, et al. Phytochrome signaling mechanisms. Arabidopsis book, 2011, 9: e0148. DOI:10.1199/tab.0148 |
| [17] |
Sheerin DJ, Hiltbrunner A. Molecular mechanisms and ecological function of far‐red light signalling. Plant Cell Environ, 2017, 40(11): 2509-2529. DOI:10.1111/pce.12915 |
| [18] |
Hiltbrunner A, Tscheuschler A, Viczián A, et al. FHY1 and FHL act together to mediate nuclear accumulation of the phytochrome A photoreceptor. Plant Cell Physiol, 2006, 47(8): 1023-1034. DOI:10.1093/pcp/pcj087 |
| [19] |
Quail PH. Phytochrome photosensory signalling networks. Nat Rev Mol Cell Biol, 2002, 3(2): 85-93. |
| [20] |
Wiltbank LB, Kehoe DM. Diverse light responses of cyanobacteria mediated by phytochrome superfamily photoreceptors. Nat Rev Microbiol, 2018, 17(1): 37-50. |
| [21] |
Müller K, Engesser R, Timmer J, et al. Synthesis of phycocyanobilin in mammalian cells. Chem Commun, 2013, 49(79): 8970-8972. DOI:10.1039/c3cc45065a |
| [22] |
Kawano F, Shi F, Yazawa M. Optogenetics: Switching with red and blue. Nat Chem Biol, 2017, 13(6): 573-574. DOI:10.1038/nchembio.2387 |
| [23] |
Redchuk TA, Kaberniuk AA, Verkhusha VV. Near-infrared light-controlled systems for gene transcription regulation, protein targeting and spectral multiplexing. Nat Protocols, 2018, 13(5): 1121-1136. DOI:10.1038/nprot.2018.022 |
| [24] |
Taslimi A, Zoltowski B, Miranda JG, et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nat Chem Biol, 2016, 12(6): 425-430. DOI:10.1038/nchembio.2063 |
| [25] |
Bugaj LJ, Choksi AT, Mesuda CK, et al. Optogenetic protein clustering and signaling activation in mammalian cells. Nat Methods, 2013, 10(3): 249-252. DOI:10.1038/nmeth.2360 |
| [26] |
Repina NA, Rosenbloom A, Mukherjee A, et al. At light speed: advances in optogenetic systems for regulating cell signaling and behavior. Ann Rev Chem Biomol Eng, 2017, 8: 13-39. DOI:10.1146/annurev-chembioeng-060816-101254 |
| [27] |
Heijde M, Ulm R. UV-B photoreceptor-mediated signalling in plants. Trends Plant Sci, 2012, 17(4): 230-237. DOI:10.1016/j.tplants.2012.01.007 |
| [28] |
Crefcoeur RP, Yin RH, Ulm R, et al. Ultraviolet-B- mediated induction of protein-protein interactions in mammalian cells. Nat Commun, 2013, 4: 1779. DOI:10.1038/ncomms2800 |
| [29] |
Di W, Hu Q, Yan Z, et al. Structural basis of ultraviolet-B perception by UVR8. Nature, 2012, 484(7393): 214-219. DOI:10.1038/nature10931 |
| [30] |
Cloix C, Kaiserli E, Heilmann M, et al. C-terminal region of the UV-B photoreceptor UVR8 initiates signaling through interaction with the COP1 protein. Proc Natl Acad Sci USA, 2012, 109(40): 16366-16370. DOI:10.1073/pnas.1210898109 |
| [31] |
Müller K, Engesser R, Schulz S, et al. Multi-chromatic control of mammalian gene expression and signaling. Nucleic Acids Res, 2013, 41(12): e124. DOI:10.1093/nar/gkt340 |
| [32] |
Christie JM, Salomon M, Nozue K, et al. LOV (light, oxygen, or voltage) domains of the blue-light photoreceptor phototropin (nph1): Binding sites for the chromophore flavin mononucleotide. Proc Natl Acad Sci USA, 1999, 96(15): 8779-8783. DOI:10.1073/pnas.96.15.8779 |
| [33] |
Harper SM, Christie JM, Gardner KH. Disruption of the LOV−Jα helix interaction activates phototropin kinase activity. Biochemistry, 2004, 43(51): 16184-16192. DOI:10.1021/bi048092i |
| [34] |
Crosson S, Rajagopal S, Moffat K. The LOV domain family: photoresponsive signaling modules coupled to diverse output domains. Biochemistry, 2003, 42(1): 2-10. |
| [35] |
Pudasaini A, El-Arab KK, Zoltowski BD. LOV-based optogenetic devices: light-driven modules to impart photoregulated control of cellular signaling. Front Mol Biosci, 2015, 2: 18. |
| [36] |
Harper SM, Neil LC, Gardner KH. Structural basis of a phototropin light switch. Science, 2003, 301(5639): 1541-1544. DOI:10.1126/science.1086810 |
| [37] |
Strickland D, Yao XL, Gawlak G, et al. Rationally improving LOV domain-based photoswitches. Nat Methods, 2010, 7(8): 623-626. DOI:10.1038/nmeth.1473 |
| [38] |
Strickland D, Lin Y, Wagner E, et al. TULIPs: Tunable, light-controlled interacting protein tags for cell biology. Nat Methods, 2012, 9(4): 379-384. DOI:10.1038/nmeth.1904 |
| [39] |
Woolley GA. Designing chimeric LOV photoswitches. Chem Biol, 2012, 19(4): 441-442. DOI:10.1016/j.chembiol.2012.04.003 |
| [40] |
Zoltowski BD, Schwerdtfeger C, Widom J, et al. Conformational switching in the fungal light sensor vivid. Science, 2007, 316(5827): 1054-1057. DOI:10.1126/science.1137128 |
| [41] |
Lamb JS, Zoltowski BD, Pabit SA, et al. Time-resolved dimerization of a PAS-LOV protein measured with photocoupled small angle X-ray scattering. J Am Chem Soc, 2008, 130(37): 12226-12227. |
| [42] |
Furuya A, Kawano F, Nakajima T, et al. Assembly domain-based optogenetic system for the efficient control of cellular signaling. ACS Synth Biol, 2017, 6(6): 1086-1095. DOI:10.1021/acssynbio.7b00022 |
| [43] |
Kato HE, Zhang F, Yizhar O, et al. Crystal structure of the channelrhodopsin light-gated cation channel. Nature, 2012, 482(7385): 369-374. DOI:10.1038/nature10870 |
| [44] |
Nagel G, Szellas T, Huhn W, et al. Channelrhodopsin-2, a directly light-gated cation-selective membrane channel. Proc Natl Acad Sci USA, 2003, 100(24): 13940-13945. DOI:10.1073/pnas.1936192100 |
| [45] |
Kushibiki T, Okawa S, Hirasawa T, et al. Optogenetic control of insulin secretion by pancreatic β-cells in vitro and in vivo. Gene Therapy, 2015, 22(7): 553-559. DOI:10.1038/gt.2015.23 |
| [46] |
Lee S, Park H, Kyung T, et al. Reversible protein inactivation by optogenetic trapping in cells. Nat Methods, 2014, 11(6): 633-636. DOI:10.1038/nmeth.2940 |
| [47] |
Park H, Kim NY, Lee S, et al. Optogenetic protein clustering through fluorescent protein tagging and extension of CRY2. Nat Commun, 2017, 8(1): 30. |
| [48] |
Turjanski AG, Vaqué JP, Gutkind JS. MAP kinases and the control of nuclear events. Oncogene, 2007, 26(22): 3240-3253. DOI:10.1038/sj.onc.1210415 |
| [49] |
Pathak GP, Strickland D, Vrana JD, et al. Benchmarking of optical dimerizer systems. ACS Synth Biol, 2014, 3(11): 832-838. DOI:10.1021/sb500291r |
| [50] |
Toettcher JE, Weiner OD, Lim WA. Using optogenetics to interrogate the dynamic control of signal transmission by the Ras/Erk module. Cell, 2013, 155(6): 1422-1434. DOI:10.1016/j.cell.2013.11.004 |
| [51] |
Zhang K, Duan LT, Ong QX, et al. Light-mediated kinetic control reveals the temporal effect of the Raf/MEK/ERK pathway in PC12 cell neurite outgrowth. PLoS ONE, 2014, 9(3): e92917. DOI:10.1371/journal.pone.0092917 |
| [52] |
Mohammadi M, Dikic I, Sorokin A, et al. Identification of six novel autophosphorylation sites on fibroblast growth factor receptor 1 and elucidation of their importance in receptor activation and signal transduction. Mol Cell Biol, 1996, 16(3): 977-989. DOI:10.1128/MCB.16.3.977 |
| [53] |
Kim N, Kim JM, Lee M, et al. Spatiotemporal control of fibroblast growth factor receptor signals by blue light. Chem Biol, 2014, 21(7): 903-912. DOI:10.1016/j.chembiol.2014.05.013 |
| [54] |
Degterev A, Boyce M, Yuan JY. A decade of caspases. Oncogene, 2003, 22(53): 8543-8567. DOI:10.1038/sj.onc.1207107 |
| [55] |
Mills E, Chen X, Pham E, et al. Engineering a photoactivated caspase-7 for rapid induction of apoptosis. ACS Synth Biol, 2011, 1(3): 75-82. |
| [56] |
Zheng B, Wang HJ, Pan HZ, et al. Near-infrared light triggered upconversion optogenetic nanosystem for cancer therapy. ACS Nano, 2017, 11(12): 11898-11907. DOI:10.1021/acsnano.7b06395 |
| [57] |
Vanhaesebroeck B, Guillermet-Guibert J, Graupera M, et al. The emerging mechanisms of isoform-specific PI3K signalling. Nat Rev Mol Cell Biol, 2010, 11(5): 329-341. |
| [58] |
Cantley LC. The phosphoinositide 3-kinase pathway. Science, 2002, 296(5573): 1655-1657. DOI:10.1126/science.296.5573.1655 |
| [59] |
Leopold AV, Chernov KG, Verkhusha VV. Optogenetically controlled protein kinases for regulation of cellular signaling. J Chem Soc Rev, 2018, 47(7): 2454-2484. DOI:10.1039/C7CS00404D |
| [60] |
Toettcher JE, Gong D, Lim WA, et al. Light-based feedback for controlling intracellular signaling dynamics. Nat Methods, 2011, 8(10): 837-839. DOI:10.1038/nmeth.1700 |
| [61] |
Chang KY, Woo D, Jung H, et al. Light-inducible receptor tyrosine kinases that regulate neurotrophin signalling. Nat Commun, 2014, 5: 4057. DOI:10.1038/ncomms5057 |
| [62] |
Idevall-Hagren O, Dickson EJ, Hille B, et al. Optogenetic control of phosphoinositide metabolism. Proc Natl Acad Sci USA, 2012, 109(35): E2316-E2323. DOI:10.1073/pnas.1211305109 |
| [63] |
Etienne-Manneville S, Hall A. Rho GTPases in cell biology. Nature, 2002, 420(6916): 629-635. DOI:10.1038/nature01148 |
| [64] |
Machacek M, Hodgson L, Welch C, et al. Coordination of Rho GTPase activities during cell protrusion. Nature, 2009, 461(7260): 99-103. DOI:10.1038/nature08242 |
| [65] |
Yazawa M, Sadaghiani AM, Hsueh B, et al. Induction of protein-protein interactions in live cells using light. Nat Biotechnol, 2009, 27(10): 941-945. DOI:10.1038/nbt.1569 |
| [66] |
Ishikawa H, Barber GN. STING is an endoplasmic reticulum adaptor that facilitates innate immune signalling. Nature, 2008, 455(7213): 674-678. DOI:10.1038/nature07317 |
| [67] |
Ishikawa H, Ma Z, Barber GN. STING regulates intracellular DNA-mediated, type Ⅰ interferon-dependent innate immunity. Nature, 2009, 461(7265): 788-792. DOI:10.1038/nature08476 |
| [68] |
Burdette DL, Monroe KM, Sotelo-Troha K, et al. STING is a direct innate immune sensor of cyclic di-GMP. Nature, 2011, 478(7370): 515-518. DOI:10.1038/nature10429 |
| [69] |
Zhong B, Yang Y, Li S, et al. The adaptor protein MITA links virus-sensing receptors to IRF3 transcription factor activation. Immunity, 2008, 29(4): 538-550. DOI:10.1016/j.immuni.2008.09.003 |
| [70] |
Jin L, Waterman PM, Jonscher KR, et al. MPYS, a novel membrane tetraspanner, is associated with major histocompatibility complex class Ⅱ and mediates transduction of apoptotic signals. Mol Cell Biol, 2008, 28(16): 5014-5026. DOI:10.1128/MCB.00640-08 |
| [71] |
Sun LJ, Wu JX, Du FH, et al. Cyclic GMP-AMP synthase is a cytosolic DNA sensor that activates the type Ⅰ interferon pathway. Science, 2013, 339(6121): 786-791. DOI:10.1126/science.1232458 |
| [72] |
Barber GN. STING: infection, inflammation and cancer. Nat Rev Immunol, 2015, 15(12): 760-770. DOI:10.1038/nri3921 |
| [73] |
Konno H, Konno K, Barber GN. Cyclic dinucleotides trigger ULK1 (ATG1) phosphorylation of STING to prevent sustained innate immune signaling. Cell, 2013, 155(3): 688-698. DOI:10.1016/j.cell.2013.09.049 |
| [74] |
Folcher M, Oesterle S, Zwicky K, et al. Mind-controlled transgene expression by a wireless-powered optogenetic designer cell implant. Nat Commun, 2014, 5: 5392. DOI:10.1038/ncomms6392 |
| [75] |
Piatkevich KD, Subach FV, Verkhusha VV. Engineering of bacterial phytochromes for near-infrared imaging, sensing, and light-control in mammals. Chem Soc Rev, 2013, 42(8): 3441-3452. DOI:10.1039/c3cs35458j |
| [76] |
Shao JW, Xue S, Yu GL, et al. Smartphone-controlled optogenetically engineered cells enable semiautomatic glucose homeostasis in diabetic mice. Sci Trans Med, 2017, 9(387): eaal2298. DOI:10.1126/scitranslmed.aal2298 |
| [77] |
Tarutina M, Ryjenkov DA, Gomelsky M. An unorthodox bacteriophytochrome from Rhodobacter sphaeroides involved in turnover of the second messenger c-di-GMP. J Biol Chem, 2006, 281(46): 34751-34758. DOI:10.1074/jbc.M604819200 |
| [78] |
Stirman JN, MM, Husson SJ, et al. Real-time multimodal optical control of neurons and muscles in freely-behaving Caenorhabditis elegans. Nat Methods, 2011, 8(2): 153-158. DOI:10.1038/nmeth.1555 |
| [79] |
Reutsky-Gefen I, Golan L, Farah N, et al. Holographic optogenetic stimulation of patterned neuronal activity for vision restoration. Nat Commun, 2013, 4: 1509. DOI:10.1038/ncomms2500 |
| [80] |
Yang KK, Wu Z, Arnold FH. Machine-learning-guided directed evolution for protein engineering. Nat methods, 2019, 16(8): 687-694. DOI:10.1038/s41592-019-0496-6 |
| [81] |
Shimizu-Sato S, Huq E, Tepperman JM, et al. A light-switchable gene promoter system. Nat Biotechnol, 2002, 20(10): 1041-1044. DOI:10.1038/nbt734 |
| [82] |
Cao J, Arha M, Sudrik C, et al. Bidirectional regulation of mRNA translation in mammalian cells by using PUF domains. Angewand Chem Int Ed, 2014, 53(19): 4900-4904. DOI:10.1002/anie.201402095 |
| [83] |
Polstein LR, Gersbach CA. Light-inducible spatiotemporal control of gene activation by customizable zinc finger transcription factors. J Am Chem Soc, 2012, 134(40): 16480-16483. DOI:10.1021/ja3065667 |
| [84] |
Konermann S, Brigham MD, Trevino AE, et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature, 2013, 500(7463): 472-476. DOI:10.1038/nature12466 |
| [85] |
Jung H, Kim SW, Kim M, et al. Noninvasive optical activation of Flp recombinase for genetic manipulation in deep mouse brain regions. Nat Commun, 2019, 10: 314. DOI:10.1038/s41467-018-08282-8 |
| [86] |
Chuong AS, Miri ML, Busskamp V, et al. Noninvasive optical inhibition with a red-shifted microbial rhodopsin. Nat Neurosci, 2014, 17(8): 1123-1129. DOI:10.1038/nn.3752 |
| [87] |
Burstein D, Harrington LB, Strutt SC, et al. New CRISPR-Cas systems from uncultivated microbes. Nature, 2017, 542(7640): 237-241. DOI:10.1038/nature21059 |
| [88] |
Nihongaki Y, Kawano F, Nakajima T, et al. Photoactivatable CRISPR-Cas9 for optogenetic genome editing. Nat Biotechnol, 2015, 33(7): 755-760. DOI:10.1038/nbt.3245 |
| [89] |
Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096. DOI:10.1126/science.1258096 |
| [90] |
Ma DC, Peng SG, Xie Z. Integration and exchange of split dCas9 domains for transcriptional controls in mammalian cells. Nat Commun, 2016, 7: 13056. DOI:10.1038/ncomms13056 |
| [91] |
Ma GL, Liu JD, Ke YP, et al. Optogenetic control of voltage-gated calcium channels. Angewand Chem Int Ed, 2018, 57(24): 7019-7022. DOI:10.1002/anie.201713080 |
| [92] |
Nguyen NT, He L, Martinez-Moczygemba M, et al. Rewiring calcium signaling for precise transcriptional reprogramming. ACS Synth Biol, 2018, 7(3): 814-821. DOI:10.1021/acssynbio.7b00467 |
| [93] |
Graf T, Enver T. Forcing cells to change lineages. Nature, 2009, 462(7273): 587-594. DOI:10.1038/nature08533 |
| [94] |
Sokolik C, Liu YX, Bauer D, et al. Transcription factor competition allows embryonic stem cells to distinguish authentic signals from noise. Cell Syst, 2015, 1(2): 117-129. |
| [95] |
Wang X, Chen XJ, Yang Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nat Methods, 2012, 9(3): 266-269. DOI:10.1038/nmeth.1892 |
| [96] |
Shao JW, Wang MY, Yu GL, et al. Synthetic far-red light-mediated CRISPR-dCas9 device for inducing functional neuronal differentiation. Proc Natl Acad Sci USA, 2018, 115(29): E6722-E6730. DOI:10.1073/pnas.1802448115 |
| [97] |
Nishi M, Sakai Y, Akutsu H, et al. Induction of cells with cancer stem cell properties from nontumorigenic human mammary epithelial cells by defined reprogramming factors. Oncogene, 2014, 33(5): 643-652. DOI:10.1038/onc.2012.614 |
| [98] |
Friedmann-Morvinski D, Bushong EA, Ke E, et al. Dedifferentiation of neurons and astrocytes by oncogenes can induce gliomas in mice. Science, 2012, 338(6110): 1080-1084. DOI:10.1126/science.1226929 |
| [99] |
Kiełbus M, Czapiński J, Odrzywolski A, et al. Optogenetics in cancer drug discovery. Exp Opin Drug Discov, 2018, 13(5): 459-472. DOI:10.1080/17460441.2018.1437138 |
| [100] |
Pathak GP, Vrana JD, Tucker CL. Optogenetic control of cell function using engineered photoreceptors. Biol Cell, 2013, 105(2): 59-72. DOI:10.1111/boc.201200056 |
| [101] |
Ye HF, Fussenegger M. Optogenetic medicine: synthetic therapeutic solutions precision-guided by light. Cold Spring Harbor Perspect Med, 2018, 9(9): a034371. |
| [102] |
Kalos M, June CH. Adoptive T cell transfer for cancer immunotherapy in the era of synthetic biology. Immunity, 2013, 39(1): 49-60. DOI:10.1016/j.immuni.2013.07.002 |
| [103] |
Vivier E, Ugolini S, Blaise D, et al. Targeting natural killer cells and natural killer T cells in cancer. Nat Rev Immunol, 2012, 12(4): 239-252. DOI:10.1038/nri3174 |
| [104] |
Restifo NP, Dudley ME, Rosenberg SA. Adoptive immunotherapy for cancer: harnessing the T cell response. Nat Rev Immunol, 2012, 12(4): 269-281. DOI:10.1038/nri3191 |
| [105] |
He L, Zhang YW, Ma GL, et al. Near-infrared photoactivatable control of Ca2+ signaling and optogenetic immunomodulation. eLife, 2015, 4: e10024. DOI:10.7554/eLife.10024 |
| [106] |
Soboloff J, Rothberg BS, Madesh M, et al. STIM proteins: dynamic calcium signal transducers. Nat Rev Mol Cell Biol, 2012, 13(9): 549-565. DOI:10.1038/nrm3414 |
| [107] |
Tan P, He L, Han G, et al. Optogenetic immunomodulation: shedding light on antitumor immunity. Trends Biotechnol, 2017, 35(3): 215-226. DOI:10.1016/j.tibtech.2016.09.002 |
| [108] |
Xu Y, Hyun YM, Lim K, et al. Optogenetic control of chemokine receptor signal and T-cell migration. Proc Natl Acad Sci USA, 2014, 111(17): 6371-6376. DOI:10.1073/pnas.1319296111 |
| [109] |
Allen ME, Zhou W, Thangaraj J, et al. An AND-Gated Drug and Photoactivatable Cre-loxP System for Spatiotemporal Control in Cell-Based Therapeutics. ACS Synth Biol, 2019, 8(10): 2359-23711. DOI:10.1021/acssynbio.9b00175 |
| [110] |
IDF Diabetes Atlas GroupGroup IDA. Update of mortality attributable to diabetes for the IDF diabetes atlas: estimates for the year 2013. Diabetes Research Clinical Practice, 2015, 109(3): 461-465. DOI:10.1016/j.diabres.2015.05.037 |
| [111] |
Ye H, Baba MDE, Peng RW, et al. A synthetic optogenetic transcription device enhances blood-glucose homeostasis in mice. Science, 2011, 332(6037): 1565-1568. DOI:10.1126/science.1203535 |
| [112] |
Zhang F, Tzanakakis ES. Amelioration of diabetes in a murine model upon transplantation of pancreatic β-cells with optogenetic control of cyclic adenosine monophosphate. ACS Synth Biol, 2019, 8(10): 2248-2255. DOI:10.1021/acssynbio.9b00262 |
| [113] |
McCall JG, Kim TI, Shin G, et al. Fabrication and application of flexible, multimodal light-emitting devices for wireless optogenetics. Nat Protocols, 2013, 8(12): 2413-2428. DOI:10.1038/nprot.2013.158 |
| [114] |
Vaziri A, Emiliani V. Reshaping the optical dimension in optogenetics. Curr Opin Neurobiol, 2012, 22(1): 128-137. DOI:10.1016/j.conb.2011.11.011 |
| [115] |
Chow BY, Boyden ES. Optogenetics and translational medicine. Sci Transl Med, 2013, 5(177): 177ps5. |
| [116] |
Wu F, Stark E, Im M, et al. An implantable neural probe with monolithically integrated dielectric waveguide and recording electrodes for optogenetics applications. J Neural Eng, 2013, 10(5): 056012. DOI:10.1088/1741-2560/10/5/056012 |
| [117] |
Packer AM, Roska B, Häuser M. Targeting neurons and photons for optogenetics. Nat Neurosci, 2013, 16(7): 805-815. DOI:10.1038/nn.3427 |
| [118] |
Wojtovich AP, Foster TH. Optogenetic control of ROS production. Redox Biol, 2014, 2(1): 368-376. |
| [119] |
Gradinaru V, Zhang F, Ramakrishnan C, et al. Molecular and cellular approaches for diversifying and extending optogenetics. Cell, 2010, 141(1): 154-165. DOI:10.1016/j.cell.2010.02.037 |
| [120] |
Müller K, Engesser R, Timmer J, et al. Orthogonal optogenetic triple-gene control in mammalian cells. ACS Synth Biol, 2014, 3(11): 796-801. DOI:10.1021/sb500305v |
 2019, Vol. 35
2019, Vol. 35