中国科学院微生物研究所、中国微生物学会主办
文章信息
- 方欢, 钟碧溪, 魏磊, 张祥林, 张威, 汪小我
- Fang Huan, Zhong Bixi, Wei Lei, Zhang Xianglin, Zhang Wei, Wang Xiaowo
- 血浆游离DNA全基因组甲基化测序的实用稳定性评估
- Practical stability of whole-genome bisulfite sequencing using plasma cell-free DNA
- 生物工程学报, 2019, 35(12): 2284-2294
- Chinese Journal of Biotechnology, 2019, 35(12): 2284-2294
- 10.13345/j.cjb.190281
-
文章历史
- Received: June 26, 2019
- Accepted: August 30, 2019
细胞游离DNA (Cell-free DNA,cfDNA)广泛存在于人体的血液、唾液、尿液、粪便、脑脊液及其他体液中[1],是无创液体活检技术中的重要标记物之一[2-3]。血浆游离DNA由全身各组织细胞的基因组经过细胞凋亡、细胞坏死、主动分泌等断裂释放到血液中[4],携带了其来源细胞的基因组变异、DNA甲基化、核小体排布等多方面信息[5],可应用于孕妇的无创产前检测、器官移植受体的术后排异评估和癌症的溯源检测等领域[6-8],具有极高的研究价值和应用潜力。
血浆游离DNA的检测方式多种多样,按照检测位点数量来分,包括基于PCR的单位点检测[9-10]、基于杂交捕获的基因芯片和靶向测序[11-13]、基于高通量测序的全基因组检测[6, 14]等;按照检测的信息来分,包括检测突变的有无[15]、染色体拷贝数的多少[16]、DNA甲基化的程度[17]、病原体的感染[18]等。随着二代测序技术的发展,全基因组测序、多维度信息整合是液体活检未来的发展趋势[2]。血浆游离DNA的全基因组甲基化测序数据除了用以获取DNA甲基化程度外,还蕴含着部分点突变、拷贝数变异、片段化模式的信息,具有极大的应用潜力。然而,目前尚缺乏对血浆游离DNA全基因组甲基化数据中非甲基化信息的详细论证与研究。
目前最常见的血浆游离DNA的全基因组甲基化检测是基于全基因组重亚硫酸盐测序(Whole genome bisulfite sequencing,WGBS)。其基本原理为:在重亚硫酸盐的作用下,DNA序列中非甲基化的胞嘧啶C会转化为尿嘧啶U,经过PCR扩增后变成胸腺嘧啶T,而甲基化的C则不变,将测序片段与人的基因组比对后,即可定量C位点的甲基化状态。这一过程主要分为建库测序实验和数据分析两部分,其中,甲基化建库方式分为转化前建库(Pre-BS)和转化后建库(Post-BS)两种[19]。在重亚硫酸盐转化过程中DNA有可能被打断。Pre-BS建库在转化之前连接测序接头,部分DNA被打断丢失,但测序获得的序列均为真实的原始片段;Post-BS建库在转化之后连接测序接头,避免了DNA因断裂失去接头而损失,但人工引入了被打断的片段。因此,两种建库方式在检出甲基化和片段化模式中各有利弊,Pre-BS保留了片段长度信息,Post-BS所需DNA的起始量更低。目前,测序公司对于Pre-BS建库的送样要求为1 μg,对于Post-BS建库的送样要求则至少为20 ng。而在研究型实验室中,在使用更高成本的试剂的前提下,Pre-BS的起始量可低至10 ng,Post-BS的起始量则仅需0.5 ng。按照血浆游离DNA提取浓度为20 ng/mL计算,仅需抽取5 mL血液,获取2–3 mL血浆即可满足公司的Post-BS建库和实验室的Pre-BS建库需求。
本研究从血浆游离DNA全基因组甲基化测序的实用性和稳定性角度出发,探究血浆游离DNA的采集提取过程的稳定性、不同甲基化建库方式的异同点与质量控制评价、甲基化测序中的片段化模式信息,为血浆游离DNA全基因组甲基化测序应用于癌症检测、无创产前检测等液体活检领域提供实用性的基础参考。
1 材料与方法 1.1 血浆游离DNA测序数据的获取 1.1.1 血液采集及血浆分离为了比较不同人、不同采血时间、是否为冻存血样等因素对血浆游离DNA采集的影响,研究采集了两名志愿者(P1为男性,P2为女性)在两个时间点(第一次采血时间记为w0,6周后第二次采血时间记为w6)的血样。血液采集方式使用标准采血流程,使用10 mL EDTA抗凝管低温运输,并在采血后的2 h内分离血浆。血浆分离步骤为:将混有EDTA的血液置于冷冻高速离心机中,4 ℃、1 600×g离心10 min;在超净台中将上层血浆转移至离心管中,再次4 ℃、16 000×g离心10 min;将上清分装至1.5 mL EP管中,用于后续提取血浆游离DNA,或−80 ℃冷冻保存。
1.1.2 血浆游离DNA的提取不同于组织或细胞的基因组DNA,血浆游离DNA在血液中的含量极低,不能使用基因组DNA的试剂盒提取血浆游离DNA。通过文献调研与比较,我们选择了QIAamp Circulating Nucleic Acid试剂盒提取血浆游离DNA。同时,将相同的血浆样品通过干冰运输分别送至市面上主流的两家测序服务提供商(公司A、公司B),进行血浆游离DNA的提取服务,每家公司重复一次。通过Qubit荧光剂测量血浆游离DNA的浓度,并计算每毫升血浆中的得率;通过安捷伦2100生物分析仪测量血浆游离DNA的片段大小,合格的样品主峰应该在170 bp左右,且无大片段的基因组污染;通过重复试验间的浓度比较,确定提取的稳定性。
1.1.3 血浆游离DNA的建库和测序对于血浆游离DNA,市场上主流测序公司仅提供微量全基因组甲基化建库服务(Post-BS建库方式),我们在实验室进行了全基因组甲基化的微量Pre-BS建库,并将两种建库方式的结果进行比较。同时,为比较A和B两家公司提取血浆游离DNA的得率和稳定性,选择公司A对两名志愿者的3次血样进行提取建库和测序,公司B对一次血样进行提取建库和测序,每个血样设置一次重复。公司A与B一共构建完成8个甲基化文库。此外,为了探究不同甲基化建库方式对DNA甲基化、片段长度的影响,我们对同样的血浆样品进行了非甲基化的常规全基因组建库测序作为参照。本研究使用的测序平台包括Illumina的NovaSeq 6000和Hiseq X Ten,测序读长均为双端150 bp。本研究中关于血浆游离DNA样本的血液来源志愿者、采血时间、建库方式、测序平台、建库实验室等详细信息见表 1。
| Sample name | Blood source | Blood drawing time | Library preparation strategy | Sequencing strategy | Library preparation laboratory |
| SP1 | P1 | w0 | Post-BS | NovaSeq 6000, PE150 | Company A |
| SP2 | P1 | w0 | Post-BS | NovaSeq 6000, PE150 | Company A |
| SP3 | P2 | w6 | Post-BS | NovaSeq 6000, PE150 | Company A |
| SP4 | P2 | w6 | Post-BS | NovaSeq 6000, PE150 | Company A |
| SP5 | P1 | w6 | Post-BS | NovaSeq 6000, PE150 | Company A |
| SP6 | P1 | w6 | Post-BS | NovaSeq 6000, PE150 | Company A |
| SP7 | P1 | w6 | WGS | NovaSeq 6000, PE150 | Company A |
| SP8 | P1 | w6 | Post-BS | Hiseq X Ten, PE150 | Company B |
| SP9 | P1 | w6 | Post-BS | Hiseq X Ten, PE150 | Company B |
| SP10 | P1 | w6 | Pre-BS | NovaSeq 6000, PE150 | Our lab |
| SP11 | P1 | w6 | Pre-BS | NovaSeq 6000, PE150 | Our lab |
针对血浆游离DNA测序数据的特点,本研究开发了一套血浆游离DNA预处理与质量控制的流程,包括检验测序质量、去除接头序列、比对基因组、去除PCR重复、计算转化效率、片段长度分布、覆盖度和深度等。
1.2.1 cfDNA全基因组甲基化数据的预处理对于SP7之外的全基因组甲基化数据,进行如下预处理步骤:首先,使用FastQC软件对数据的测序质量、碱基分布、序列重复次数、接头污染情况等进行初步统计。由于基因组大部分C位点无甲基化修饰,测序为T碱基,因此在甲基化文库序列中,T的比例最高,C的比例最低,造成碱基不均衡现象。然后,由于血浆游离DNA的片段长度较短,当片段长度短于测序读长时,双端测序读段的3′端包含部分测序接头序列。我们通过Cutadapt软件截去接头序列。下一步,使用BS-Seeker2软件[20]将甲基化测序数据比对到人的hg19基因组上,匹配模式为局部比对。最后,使用Picard软件去除比对到同一位置的重复片段,计算全基因组每一CpG位点上比对的片段数目和甲基化片段数目。
1.2.2 cfDNA全基因组测序数据的预处理由于本研究涉及的血浆游离DNA全基因组甲基化测序受到多因素影响,而采集的样本有限,因此,设置cfDNA全基因组测序数据(Whole genome sequencing, WGS)作为基础对照,并搜集公共数据集中健康人的血浆游离DNA全基因组数据,弥补样本数量的不足,增强结论的可信度。本研究搜集的公共数据集包括GEO数据库中的GSE71378数据集(样本编号BH01,美国多个健康人的血浆游离DNA混合测序数据)[21]以及EGA数据库中的EGAS00001001024数据集(样本编号C309–314,中国香港健康人血浆游离DNA测序数据)[22]。与甲基化测序数据相比,血浆游离DNA的全基因组数据处理流程类似,使用esATAC包[23]去除接头序列和比对基因组,使用Picard去除PCR重复扩增的片段。
1.2.3 cfDNA文库的质量控制标准在测序数据的预处理过程中,分别统计测序片段数、成功比对片段数、去重后片段数,计算比对率、PCR重复率和测序有效率,其中PCR重复率越低越好,比对率和测序有效率越高越好。对有效片段统计其长度分布,观测主峰位置是否符合血浆游离DNA的长度特点。再基于片段的实际长度,用片段覆盖到的基因组区域除以基因组总长度计算覆盖度,使用片段总碱基数除以覆盖基因组范围得到平均测序深度。我们以上述参数为指标,比较不同建库方式、不同实验室建库获得的血浆游离DNA甲基化测序数据的质量。
PCR重复率与测序深度息息相关,对同一文库来说,测序较浅时,我们发现重复片段的概率较低,PCR重复率低;随着测序加深,PCR重复率逐渐升高。对原始测序数据进行降采样,以5 M测序片段为步长设计采样点,每个采样点重复两次,分别计算重复率,取两次重复的平均值观测PCR重复率与测序深度的关系。此外,自建库的SP10和SP11测序过程分为两步,首先测序5G原始数据,根据上述标准判断文库质量,然后根据文库复杂度进行加测,由此得到的两次独立上机的数据可用于探索同一文库在两次测序中的结果异同。后续分析比较使用两次测序合并的数据。
1.3 cfDNA甲基化数据的片段化模式评估借鉴文献中的短片段比例作为片段化模式特征[14],分别计算SP1–11和美国人血浆游离DNA混合测序数据BH01、中国香港人血浆游离DNA测序数据C309–314的全基因组片段化模式图谱。具体步骤如下:首先,将基因组划分为不重叠的5 M宽的窗口,计算每个窗口内长度为100–150 bp的短片段数量和长度为150–220 bp的长片段数量,短片段占比为短片段数量除以长短片段总数。为了避免性别对片段化模式特征的影响,将X和Y性染色体排除在外。为了使不同样本间可比,对短片段占比进行标准化,通过减去均值使片段化模式特征分布于0附近,再用于后续聚类分析。
为了定量刻画不同血浆游离DNA文库的片段化模式差异,以全基因组的短片段占比为特征向量,计算两两样本间的皮尔森相关系数,再采用非加权配对算术平均法进行聚类,得到血浆游离DNA甲基化数据的片段化模式关系。
2 结果与分析 2.1 血浆游离DNA的提取稳定性为了探究人群差异、采血时间、血浆冻存时间、起始血浆体积等因素对血浆游离DNA提取的影响,本研究采集了两名志愿者在两个时间点的血样,将血浆冻存不同时间后使用同一流程进行多次提取,血浆游离DNA的提取浓度见表 2。结果表明,两名志愿者的血浆游离DNA浓度存在显著差异;同一个人在不同时间采集的血浆游离DNA趋于稳定,采血时间对血浆游离DNA浓度的影响不及不同人的影响大;冻存时间与游离DNA浓度并不是正比关系,而是呈现游离DNA浓度随着冻存时间加长而先增大后减小的趋势。这可能是因为:在新鲜血浆中,细胞的基因组DNA较为完整,容易在提取过程中去除;当血浆冻存一段时间后,基因组DNA逐渐降解,混入游离DNA中一起提取,从而增加了血浆游离DNA的浓度;当血浆经过长期冷冻后,DNA降解消失现象增多,血浆游离DNA的浓度逐渐减少。通过使用安捷伦2100生物分析仪对冻存血浆中游离DNA的长度分布进行检测,证实了冻存血浆中大片段基因组污染增多的观点。实际上,DNA浓度不仅与血浆中片段的摩尔量有关,而且与片段长度直接相关。因此,当存在基因组污染时,使用质量浓度定量血浆游离DNA是不准确的,即使大片段的摩尔量很少,百倍的片段长度也会使测得的游离DNA浓度虚高。可能更精确定量血浆游离DNA的方案有:方案1是通过片段选择去除长片段后检测游离DNA的质量浓度,排除长片段的干扰;方案2是使用摩尔浓度替代质量浓度,通过qPCR定量血浆中游离DNA的摩尔数量,避免片段长度对定量准确性的影响。
| Blood source | Blood drawing time | Freezing time (d) | Input plasma volume (mL) | Plasma concentration (ng/mL) |
| P1 | w0 | 0 | 4 | 17.13 |
| P1 | w0 | 42 | 3 | 38.00 |
| P1 | w0 | 125 | 11 | 30.00 |
| P1 | w6 | 0 | 3 | 22.93 |
| P1 | w6 | 0 | 3 | 24.93 |
| P1 | w6 | 0 | 3 | 25.20 |
| P1 | w6 | 0 | 3 | 24.27 |
| P1 | w6 | 90 | 2 | 22.25 |
| P2 | w0 | 0 | 4 | 10.00 |
| P2 | w0 | 42 | 3 | 30.67 |
| P2 | w0 | 125 | 15 | 15.51 |
| P2 | w6 | 0 | 3 | 14.93 |
| P2 | w6 | 0 | 3 | 11.33 |
| P2 | w6 | 0 | 3 | 14.47 |
| P2 | w6 | 0 | 3 | 12.47 |
| P2 | w6 | 90 | 3 | 16.89 |
| The lines in bold corresponding to SP10 and SP11 in table 3. | ||||
为了比较不同实验室提取血浆游离DNA的浓度与稳定性差异,将两名志愿者的多个血浆样本分别送至公司A、B进行血浆游离DNA提取,最终提取浓度与本实验室的两样本提取结果汇总见表 3。从表中可以观察到不同实验室是影响血浆游离DNA提取浓度的关键因素,公司A提取的SP1–7浓度较低,公司B提取的SP8–9浓度较高,但重复实验间的一致性较差。此外,在公司A提取的样本中,来自P2个体的样本(SP3–4)的cfDNA浓度低于来自P1个体的样本(SP1–2、SP5–7),与前面本实验室提取的结果一致。最后,使用安捷伦2 100生物分析仪检测血浆游离DNA的片段长度分布,确认SP1–11无基因组污染,提取结果达到了建库要求,可以进行后续建库和测序。
| Sample name | Input plasma volume (mL) | Total cfDNA mass (ng) | Plasma concentration (ng/mL) |
| SP1 | 1 | 19.12 | 19.12 |
| SP2 | 1 | 19.12 | 19.12 |
| SP3 | 2 | 13.35 | 6.68 |
| SP4 | 2 | 11.44 | 5.72 |
| SP5 | 2 | 16.78 | 8.39 |
| SP6 | 2 | 17.55 | 8.78 |
| SP7 | 2 | 15.64 | 7.82 |
| SP8 | 1 | 65.00 | 65.00 |
| SP9 | 1 | 25.00 | 25.00 |
| SP10 | 3 | 72.80 | 24.27 |
| SP11 | 2 | 44.50 | 22.25 |
| SP1 and SP2 are different libraries from the same extracted DNA. | |||
本研究涉及Pre-BS和Post-BS两种甲基化建库方式。10个血浆游离DNA甲基化文库的Qubit浓度和2 100片段分布都符合上机测序要求,血浆游离DNA甲基化测序数据的预处理和质量控制结果见表 4。公司A建库的SP1–6的局部比对率不超过40%,远低于公司B建库的SP8–9。当使用全局匹配模式进行比对时,SP1–6的成功比对率只有25%左右。在排除了接头序列污染等流程不当的因素后,我们在未成功比对的片段中观测到了部分PCR异源双链核酸分子,确认造成比对率低的原因发生于公司A的甲基化建库过程中。由于比对率的不足,造成与SP8–9相似测序深度的SP1–4、SP6的总有效片段数量较少,基因组覆盖度偏低,平均有效深度仅(1–2)×;对于测序量是SP8或SP9三倍的SP5,其获得的有效片段数、平均深度与SP8、SP9持平。综合来看,公司B的血浆游离DNA甲基化建库的数据有效率比公司A更高。考虑Pre-BS与Post-BS两种建库方式的特点,可见SP10–11的PCR重复率略高于SP8–9,这可能是因为Pre-BS建库打断损失了部分血浆游离DNA,文库复杂度降低导致PCR重复片段增多。
| Sample name | Raw fragments (M) |
Local alignment rate (%) |
Duplication rate (%) |
Effective fragments (M) | Genome coverage (%) |
Sequencing depth (×) |
| SP1 | 84.85 | 38.67 | 28.84 | 23.35 | 49.18 | 2.33 |
| SP2 | 87.99 | 39.17 | 24.53 | 26.01 | 55.54 | 2.36 |
| SP3 | 72.30 | 34.30 | 25.91 | 18.37 | 46.07 | 1.96 |
| SP4 | 82.18 | 33.97 | 26.48 | 20.52 | 53.07 | 1.98 |
| SP5 | 230.78 | 38.46 | 40.90 | 52.46 | 70.01 | 3.70 |
| SP6 | 62.16 | 39.04 | 28.31 | 17.39 | 44.22 | 1.95 |
| SP8 | 77.81 | 77.10 | 17.05 | 49.77 | 85.62 | 3.63 |
| SP9 | 77.40 | 77.72 | 16.38 | 50.30 | 85.24 | 3.68 |
| SP10 | 218.31 | 74.55 | 42.92 | 91.27 | 83.26 | 6.52 |
| SP11 | 69.19 | 76.27 | 23.05 | 40.60 | 72.13 | 3.30 |
在表 4中,可见PCR重复率与测序深度高度相关,测序最深的SP5和SP10是重复率最高的两个样本。为了更精确地定量比较血浆游离DNA甲基化文库的复杂度差异,我们对有效片段数较多的SP5、SP8–11的原始甲基化测序数据作了降采样分析,结果如图 1所示。随着测序数据的采样下降,PCR重复率下降,符合正相关关系。对比同一降采样深度下不同文库的重复率,发现公司A的SP5文库重复率最高;公司B的SP8、SP9文库重复率最低;SP10、SP11两个Pre-BS文库理论上应该重复率较高,但实际上介于公司A和公司B的Post-BS文库之间。重复率的高低直接关系到测序成本和文库的极限测序量,是建库水平的一个直观反映。有趣的一点是,同一文库在两次独立测序中得到的重复率曲线不完全相同。具体来说,图 1中test_SP10与test_SP11的重复率高于SP10和SP11,当预期测序深度较低时,所得数据的重复率高于高深度数据的降采样重复率。这可能是因为:血浆游离DNA甲基化文库的复杂度有限,不同预期测序量下的上机文库量不同,造成文库的复杂度、重复率差异。

|
| 图 1 测序深度与文库重复率的关系 Fig. 1 Relationship between sequencing depth and library duplication rate. |
| |
除了成功比对率、重复率、覆盖度深度等质量控制指标之外,血浆游离DNA甲基化文库的评价标准还包括片段长度分布。根据成功比对的片段绘制血浆游离DNA甲基化文库的长度分布曲线,同时对比非甲基化建库的SP7和多个健康人混合数据BH01,所得结果如图 2所示。图 2中虚线为170 bp,BH01和SP7–11文库的主峰均在170 bp附近,而公司A建库的SP1–6主峰明显偏离170 bp。此外,更为严重的问题是公司A实验的Post-BS文库和SP7都存在长度分布截断的现象。事实上,实验中的片段选择步骤难以做到完全截断,不会是垂直的长度分布曲线。经过反复排查测序数据的预处理流程,采取局部比对基因组的策略,问题依然存在。考虑到片段长度截断发生在150 bp以内,有可能是因为测序公司在生成原始数据时,默认过滤了含有部分接头序列的片段。对于基因组DNA的常规建库来说,文库大小(200–400 bp)远高于测序的读长(150 bp),因此舍弃短片段不会影响测序数据的分析,但是对于血浆游离DNA来说,过滤短片段会对后续分析的影响较大。例如,癌症病人血浆游离DNA比健康人更短[24],使用长度为90–150 bp的血浆游离DNA可以更灵敏地区分癌症病人和健康人[25]。换句话说,血浆游离DNA的短片段中,癌症来源片段的比例更高,舍弃短片段将直接影响血浆游离DNA的应用。

|
| 图 2 血浆游离DNA文库的长度分布(A:WGS文库的长度分布;B:公司A构建的Post-BS文库的长度分布;C:公司B构建的Post-BS文库的长度分布;D:Pre-BS甲基化文库的长度分布) Fig. 2 Length distribution of plasma cfDNA library. (A) Length distribution of WGS library. (B) Length distribution of Post-BS library accomplished by company A. (C) Length distribution of Post-BS library accomplished by company B. (D) Length distribution of Pre-BS library. |
| |
对比Pre-BS和Post-BS建库的片段长度区别,可见SP10与SP11这两个Pre-BS文库的片段长度分布呈现10 bp左右的周期,与非甲基化建库(WGS)的公共数据BH01中的周期一致,而两家公司的Post-BS数据中,血浆游离DNA片段长度分布比较平滑。由于Pre-BS与Post-BS两种建库方式是分别在不同实验室完成的,所以不能排除实验室差异的因素造成片段长度分布的差异。但至少,这一现象证实了Pre-BS可以保留部分片段长度信息,而公司A、B的Post-BS建库对精细的片段长度信息损失较多。
2.3 血浆游离DNA甲基化数据的长度信息为了进一步阐述血浆游离DNA甲基化文库中的片段长度信息,研究参考了文献中的血浆游离DNA长度特征[14],使用全基因组5 M窗口内的短片段比例作为特征,绘制甲基化与常规文库的全基因组片段化模式图谱,所得结果见图 3。

|
| 图 3 血浆游离DNA全基因组数据中的片段化模式 Fig. 3 Fragmentation pattern in whole-genome plasma cfDNA data. |
| |
由于在考察血浆游离DNA的长度特征时,全基因组测序比全基因组甲基化测序更能反映真实情况,因此,本研究将全基因组甲基化数据中的片段长度特征与全基因组测序数据进行比较,以期评估血浆游离DNA甲基化数据中的片段长度信息。此外,为了反映片段长度在人群中的差异,还搜集了两个公共数据集中的健康人血浆游离DNA全基因组测序数据(美国多个健康人混合血浆测序数据BH01、中国香港健康人血浆测序数据C309–314)。从图 3可以看出,SP10–11中Pre-BS甲基化数据得到的片段化模式特征在全基因组的方差较大,信息量更大,其分布模式与WGS数据BH01、SP7相似,而与SP1–6与SP8–9中Post-BS数据存在显著差异。这一现象再次印证了Pre-BS保留片段长度信息的特点。
我们根据血浆游离DNA的全基因组片段化模式图谱,计算样本间的相关关系,聚类分析推断样本间相似性,聚类结果如图 4所示。SP10–11中Pre-BS数据与SP7、BH01相关性高;公司B建库的Post-BS数据和C309–314聚在一起;而公司A建库的Post-BS数据单独聚在一起。从聚类结果可以观察到血浆游离DNA的Pre-BS数据可以保留WGS的片段化模式特征;公司B建库Post-BS数据也捕获了部分片段化模式特征,对应到图 3的基因组图谱中,例如1号、10号染色体的图谱模式印证了Post-BS B与C309–314更相似。总体来说,不管是Pre-BS还是Post-BS甲基化建库,都含有血浆游离DNA的片段化模式信息,不同甲基化建库方式的数据差异与不同实验室的WGS数据差异相当,说明甲基化与非甲基化建库方式对片段化模式的影响甚至不及人群因素的影响大。但是,对于公司A产出的Post-BS数据,其片段化模式与WGS数据相关性差,这一点有可能与公司A的短片段截断有关。
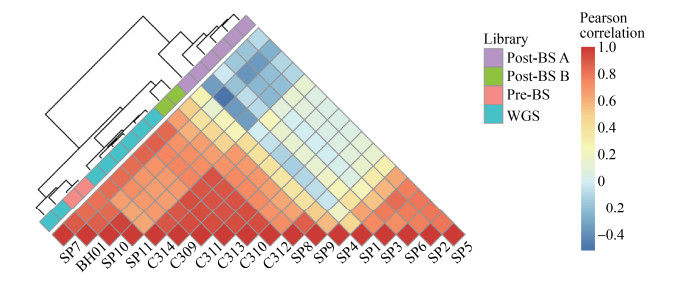
|
| 图 4 血浆游离DNA文库间的片段化模式相关性 Fig. 4 Correlations of fragmentation pattern among different cfDNA libraries. |
| |
血浆游离DNA在液体活检领域有着广泛的应用,在癌症检测领域,血浆游离DNA中部分位点的检测试剂盒已经得到国家食品药品监督管理总局的批准,应用于人群的癌症筛查中。随着新一代测序技术的不断发展,血浆游离DNA的全基因组甲基化测序在早期癌症的检测与肿瘤溯源方面具有良好的应用前景。
本研究通过对不同人、不同采血时间、不同冻存时间、不同建库方式、不同测序平台、不同公司等变量的分析,探究了血浆游离DNA的采集、提取、建库、测序、信息分析过程,综合评估实验的稳定性和甲基化数据的实用性。研究结果表明:针对血浆游离DNA的采集提取稳定性,两名健康异性志愿者的血浆游离DNA浓度在5–65 ng/mL范围内,两人的血浆游离DNA浓度有显著差异,而不同采血时间的浓度较为稳定;针对血浆游离DNA的建库测序稳定性,我们发现不同测序公司间的建库质量存在较大差异,并存在短序列读段的长度截断等干扰因素;针对Pre-BS与Post-BS甲基化建库的实用性,结果表明抽取5 mL血液即可满足Pre-BS与Post-BS的建库需求,同时Pre-BS保留了更精细的片段长度信息;针对血浆游离DNA甲基化数据中的片段化模式,我们发现Pre-BS甲基化测序数据中能提取到与WGS相似的片段化模式特征。这些实用性与稳定性的评估结果将为血浆游离DNA全基因组甲基化测序应用于液体活检领域提供有力的参考和支撑。
| [1] |
Chan AK, Chiu RW, Lo YM, et al. Cell-free nucleic acids in plasma, serum and urine: a new tool in molecular diagnosis. Ann Clin Biochem, 2003, 40(2): 122-130. DOI:10.1258/000456303763046030 |
| [2] |
Heitzer E, Haque IS, Roberts CES, et al. Current and future perspectives of liquid biopsies in genomics-driven oncology. Nat Rev Genet, 2019, 20(2): 71-88. DOI:10.1038/s41576-018-0071-5 |
| [3] |
Schwarzenbach H, Hoon DS, Pantel K. Cell-free nucleic acids as biomarkers in cancer patients. Nat Rev Cancer, 2011, 11(6): 426-437. DOI:10.1038/nrc3066 |
| [4] |
Lui YY, Chik KW, Chiu RW, et al. Predominant hematopoietic origin of cell-free DNA in plasma and serum after sex-mismatched bone marrow transplantation. Clin Chem, 2002, 48(3): 421-427. |
| [5] |
Wan JCM, Massie C, Garcia-Corbacho J, et al. Liquid biopsies come of age: towards implementation of circulating tumour DNA. Nat Rev Cancer, 2017, 17(4): 223-238. DOI:10.1038/nrc.2017.7 |
| [6] |
Sun K, Jiang PY, Chan KCA, et al. Plasma DNA tissue mapping by genome-wide methylation sequencing for noninvasive prenatal, cancer, and transplantation assessments. Proc Natl Acad Sci USA, 2015, 112(40): E5503-E5512. DOI:10.1073/pnas.1508736112 |
| [7] |
De Rubis G, Rajeev Krishnan S, Bebawy M. Liquid biopsies in cancer diagnosis, monitoring, and prognosis. Trends Pharmacol Sci, 2019, 40(3): 172-186. DOI:10.1016/j.tips.2019.01.006 |
| [8] |
Li WY, Li QJ, Kang SL, et al. Cancer Detector: ultrasensitive and non-invasive cancer detection at the resolution of individual reads using cell-free DNA methylation sequencing data. Nucleic Acids Res, 2018, 46(15): e89. DOI:10.1093/nar/gky423 |
| [9] |
Ooki A, Maleki Z, Tsay JJ, et al. A panel of novel detection and prognostic methylated DNA markers in primary non-small cell lung cancer and serum DNA. Clin Cancer Res, 2017, 23(22): 7141-7152. DOI:10.1158/1078-0432.CCR-17-1222 |
| [10] |
Cohen JD, Li L, Wang YX, et al. Detection and localization of surgically resectable cancers with a multi-analyte blood test. Science, 2018, 359(6378): 926-930. DOI:10.1126/science.aar3247 |
| [11] |
Liu L, Toung JM, Jassowicz AF, et al. Targeted methylation sequencing of plasma cell-free DNA for cancer detection and classification. Ann Oncol, 2018, 29(6): 1445-1453. DOI:10.1093/annonc/mdy119 |
| [12] |
Jamal-Hanjani M, Wilson GA, Horswell S, et al. Detection of ubiquitous and heterogeneous mutations in cell-free DNA from patients with early-stage non-small-cell lung cancer. Ann Oncol, 2016, 27(5): 862-867. DOI:10.1093/annonc/mdw037 |
| [13] |
Guo SC, Diep D, Plongthongkum N, et al. Identification of methylation haplotype blocks aids in deconvolution of heterogeneous tissue samples and tumor tissue-of-origin mapping from plasma DNA. Nat Genet, 2017, 49(4): 635-642. DOI:10.1038/ng.3805 |
| [14] |
Cristiano S, Leal A, Phallen J, et al. Genome-wide cell-free DNA fragmentation in patients with cancer. Nature, 2019, 570(7761): 385-389. DOI:10.1038/s41586-019-1272-6 |
| [15] |
Qu CF, Wang YT, Wang P, et al. Detection of early-stage hepatocellular carcinoma in asymptomatic HBsAg- seropositive individuals by liquid biopsy. Proc Natl Acad Sci USA, 2019, 116(13): 6308-6312. DOI:10.1073/pnas.1819799116 |
| [16] |
Chan KCA, Jiang PY, Chan CWM, et al. Noninvasive detection of cancer-associated genome-wide hypomethylation and copy number aberrations by plasma DNA bisulfite sequencing. Proc Natl Acad Sci USA, 2013, 110(47): 18761-18768. DOI:10.1073/pnas.1313995110 |
| [17] |
Feng H, Jin P, Wu H. Disease prediction by cell-free DNA methylation. Brief Bioinform, 2019, 20(2): 585-597. DOI:10.1093/bib/bby029 |
| [18] |
Lam WKJ, Jiang PY, Chan KCA, et al. Sequencing-based counting and size profiling of plasma Epstein-Barr virus DNA enhance population screening of nasopharyngeal carcinoma. Proc Natl Acad Sci USA, 2018, 115(22): E5115-E5124. DOI:10.1073/pnas.1804184115 |
| [19] |
Olova N, Krueger F, Andrews S, et al. Comparison of whole-genome bisulfite sequencing library preparation strategies identifies sources of biases affecting DNA methylation data. Genome Biol, 2018, 19(1): 33. DOI:10.1186/s13059-018-1408-2 |
| [20] |
Guo WL, Fiziev P, Yan WH, et al. BS-Seeker2: a versatile aligning pipeline for bisulfite sequencing data. Bmc Genomics, 2013, 14: 774. DOI:10.1186/1471-2164-14-774 |
| [21] |
Snyder MW, Kircher M, Hill AJ, et al. Cell-free DNA comprises an in vivo nucleosome footprint that informs its tissues-of-origin. Cell, 2016, 164(1/2): 57-68. |
| [22] |
Jiang PY, Chan CWM, Chan KCA, et al. Lengthening and shortening of plasma DNA in hepatocellular carcinoma patients. Proc Natl Acad Sci USA, 2015, 112(11): E1317-E1325. DOI:10.1073/pnas.1500076112 |
| [23] |
Wei Z, Zhang W, Fang H, et al. esATAC: an easy-to-use systematic pipeline for ATAC-seq data analysis. Bioinformatics, 2018, 34(15): 2664-2665. DOI:10.1093/bioinformatics/bty141 |
| [24] |
Underhill HR, Kitzman JO, Hellwig S, et al. Fragment length of circulating tumor DNA. PLoS Genet, 2016, 12(7): e1006162. DOI:10.1371/journal.pgen.1006162 |
| [25] |
Mouliere F, Chandrananda D, Piskorz AM, et al. Enhanced detection of circulating tumor DNA by fragment size analysis. Sci Transl Med, 2018, 10(466): eaat4921. DOI:10.1126/scitranslmed.aat4921 |
 2019, Vol. 35
2019, Vol. 35