中国科学院微生物研究所、中国微生物学会主办
文章信息
- 涂春田, 汪洋, 易力, 王瑜欣, 刘宝宝, 宫胜龙
- Tu Chuntian, Wang Yang, Yi Li, Wang Yuxin, Liu Baobao, Gong Shenglong
- 信号分子调控细菌生物被膜形成的分子机制
- Roles of signaling molecules in biofilm formation
- 生物工程学报, 2019, 35(4): 558-566
- Chinese Journal of Biotechnology, 2019, 35(4): 558-566
- 10.13345/j.cjb.180326
-
文章历史
- Received: August 10, 2018
- Accepted: January 28, 2019
2. 洛阳师范学院 生命科学院,河南 洛阳 471022
2. College of Life Sciences, Luoyang Normal University, Luoyang 471022, Henan, China
生物被膜(Bacterial biofilm,BF)是指黏附于机体黏膜或生物材料表面、由细菌分泌的胞外聚合物(EPS)组成的基质包裹菌体形成的被膜状生物群体[1]。EPS主要是由细菌自发产生,由多糖、蛋白质、脂质、细胞外DNA (eDNA)和生物表面活性剂组成。据报道,自然界中任何成熟的细菌均可形成BF,成熟BF可以是同种或不同种细菌共同形成的。细菌BF状态被认为是一种细菌适应不良环境而形成的保护模式。已经证明,与浮游细胞相比,生活在BF内的细菌抵抗抗生素和免疫系统的能力更强[2-3],这加强了人们对这种细菌生命形式的关注。研究发现,BF能增强细菌的耐受性有诸多原因,主要包括细菌在BF内缓慢生长和由各种生物聚合物组成的细胞外基质的存在。BF与其周围基质的形成和被膜内细菌增殖受到许多因素的影响,包括参与BF生命周期的不同阶段的多个复杂的调节系统。
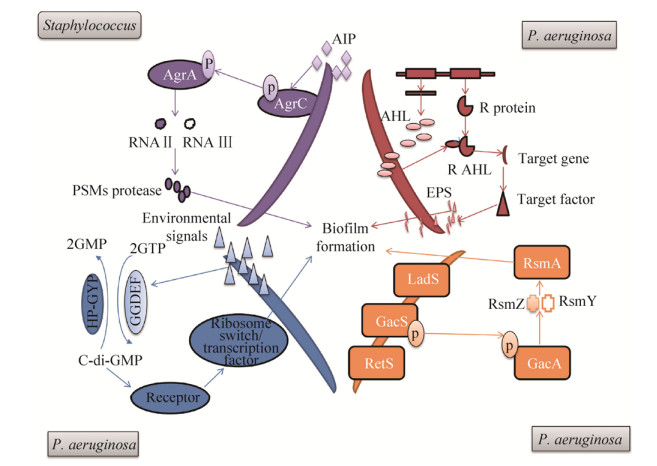
信号分子是细菌在代谢过程中自主产生的代谢产物,它不仅能在胞内调控细菌个体的生命活动,还能分泌到胞外调节细胞间的生命活动,BF的形成已被证实受多种信号分子的调控[4]。近年来,有关细菌BF的形成过程和结构成分的研究已有报道,但就信号分子与细菌BF形成的关系却报道较少,而揭示二者之间的关系会为解决病原菌的持续性感染提供一个全新的思路。本文概述了4个调控系统QS、c-di-GMP、双组分系统和sRNA,重点阐述了相关信号分子与BF形成的关系(图 1表示QS、c-di-GMP、双组分系统和sRNA对BF的调控机制),以期寻找出合适的能抑制BF相关信号分子传递的抑制剂。
20世纪70年代,Hasting等[6]通过对海洋鱼类共生细菌费氏弧菌Vibrio fisheri和哈维氏弧菌Vibrio harveyi的发光机制的研究,揭示了细菌与细菌间是存在信息交流的。这种细菌通过自身产生的信号分子进行交流和协调群体行为的过程称为密度感应。至今已经发现多种介导密度感应有关的信号分子,如N-酰化高丝氨酸内酯(N-Acyl homoserine lactones,AHL)、自诱导多肽(Autoinducing peptide,AIP)、自诱导分子Ⅱ (Autoinducer-2,AI-2)和扩散性信号分子(Diffusible signal factor,DSF)等,本文以AIP、AHL和AI-2这3种研究比较广泛的QS信号分子为例,阐述其调控BF形成的相关机制。
1.1 自诱导多肽(Autoinducing peptide,AIP)AIP是革兰阳性菌中的寡肽类自诱导信号分子,是参与细菌种内信息交流的QS信息分子。目前对于革兰阳性菌AIP信号分子的研究较多的是葡萄球菌,其调控基因是附属基因调节子(Accessory gene regulator,agr),能产生和感应的信号分子为自诱导物(AIP)。agr是由RNA Ⅱ和RNA Ⅲ两个转录单元组成,其启动子分别是P2和P3,RNA Ⅱ的转录框包括agrD、agrB、agrC和agrA 4个基因的agr操纵子,RNA Ⅲ的转录框是agr系统的效应分子,编码α-溶血素和δ-毒素等毒力因子。agrB和agrD产物参与AIP的产生;agrA和agrC基因编码的蛋白质形成一个双组分系统(Two component system,TCS),AgrC是双组分系统的感应激酶,能结合AIP,并磷酸化激活AgrA,激活的AgrA与P2和P3结合从而诱导RNA Ⅱ和RNA Ⅲ的转录。同时,活化的agr可进一步增强AIP的表达,对agr的自激活机制起到信号放大的作用。
大量研究表明,agr介导的群体密度感应系统对葡萄球菌BF的形成有着极其重要的影响,具有上调细胞与BF分离、下调BF初始黏附的作用。agr可上调酚溶解性调理素(Phenol-soluble modulins,PSMs)和蛋白酶的表达,从而促进后期细胞与BF的分离[7]。研究浮游态金黄色葡萄球菌和BF状态下金黄色葡萄球菌agr活性发现,浮游态的细菌表现出高水平的agr活性,而BF中的细胞主要抑制agr系统,这表明agr系统参与了BF的脱落[8]。agr还可以通过下调如自溶血素E (Autolysin E,AtIE)等与BF初期形成有关的粘附分子的形成从而调控BF,Dai等[9]通过对野生表皮葡萄球菌与agr突变株形成BF能力对比发现,agr突变株形成更厚的BF,而agr和AtIE双突变菌株BF形成功能严重受损,在agr突变株中,AtlE表达量增加,而在agr与AtlE双突变株中几乎没有AtlE表达。由此表明,agr突变株通过增加AtIE的产生量,从而加强细菌的初始黏附功能,使BF形成能力增强。
1.2 N-酰化高丝氨酸内酯(N-acyl homoserine lactones,AHL)由信号分子AHL介导的QS系统,是革兰阴性菌种内通讯语言,它在生物体内调节多种生理功能,如毒力因子的产生和BF的形成。革兰阴性菌中的QS系统被称为LuxI/LuxR型自诱导系统,其信号分子是AHL。在LuxI/LuxR调控网络中,LuxI型蛋白是负责产生AHL分子的酶,而LuxR型蛋白是负责接收AHL分子的受体。
AHL分子的碳链长度不同,短链分子(4–8个碳原子)可以通过自由扩散进出细胞,长链分子(10–12个碳原子)需要主动运输通过细胞膜。AHL通过细胞膜扩散到细菌周围并富集到特定密度后,AHL分子会重新进入细胞结合到LuxR受体蛋白上,激活LuxR蛋白并形成R-AHL复合体,随后R-AHL复合体与目的激活子相互作用诱导目的基因表达,如合成胞外多糖、毒力因子及藻酸盐等。铜绿假单胞菌野生株形成的BF高度结构化、较厚,但临床分离获得的不产生AHL信号分子的突变株则不能形成成熟的BF[10]。研究发现,铜绿假单胞菌PA01突变株(不能产生AHL)形成的BF扁平、均质,人工添加AHL信号分子后,又能形成与野生型菌株相同的有结构的、异质化的BF[11],这证实了AHL在铜绿假单胞菌BF的形成过程中具有重要作用。大多数革兰阴性菌如荧光假单胞菌、嗜水气单胞菌等都使用LuxI/LuxR型自诱导系统,沙门菌属、埃希菌属和克雷伯杆菌属在革兰阴性菌中是例外,因为它们的LuxI/LuxR型自体诱导系统不完善[12-13],这些属的细菌不具有LuxI家族或其他AHL合酶,然而,它们有LuxR家族蛋白质,可以感知某些AHL分子,从而被调控。
1.3 自诱导物Ⅱ(AI-2)由LuxS/AI-2介导的群体感应系统广泛存在于革兰阳性菌和革兰阴性菌中,其信号分子AI-2被认为是种间通用信号分子[14]。AI-2的合成需要luxS基因编码的LuxS蛋白酶的催化作用,细菌产生的AI-2分泌到细胞外环境,随着细胞的分裂增殖,胞外的AI-2的浓度也会不断增加,当AI-2浓度达到阈值后细菌通过感应周围AI-2信号分子的浓度而对其周围细菌进行“计数”,并会通过一系列的调控系统调控与QS相关的基因的表达,如BF的形成和毒力因子等基因的表达[14]。
本课题组一直致力于猪链球菌(Streptococcus suis,SS) LuxS/AI-2 QS系统的研究,利用同源重组原理构建了猪链球菌2型luxS基因缺失株、互补株和过表达株,研究发现缺失株不能产生AI-2信号分子[15],而luxS过表达菌株AI-2信号分子产生量也并未增加,缺失luxS基因对SS的BF的形成产生了显著的影响,过表达菌株形成生物被膜的能力有一定增强,并随培养时间增加而增加[16]。外源添加0–2 µmol/L的AI-2分子能够显著增加SS生物被膜的形成,而添加2–15 µmol/L的AI-2分子后生物被膜的形成能力受到抑制,外源添加AI-2分子能显著提高luxS缺失株的生物被膜形成能力[17]。研究发现SS随细菌密度的增加胞外AI-2信号分子的浓度也在不断增加,并在对数生长末期达到最大,稳定期和衰亡期迅速减少,这说明SS能将胞外AI-2运至胞内,细胞内存在AI-2的摄取机制和与之结合的受体蛋白。添加不同浓度AI-2分子可以显著提高毒力基因cps2、sly和mrp的表达,而ef、fbps和gapdh基因表达却有一定下调[17]。AI-2信号分子可能通过与受体蛋白结合来调控相关基因的表达而影响BF的形成。课题组解析了猪链球菌LuxS蛋白晶体的三维晶体结构,发现位于底物结位点附近80位和87位氨基酸发生了可能的进化突变,通过定点突变发现2个氨基酸的突变对其底物结合和酶的催化能力有较大影响,并影响猪链球菌AI-2的产生和BF的形成能力[18]。
2 环二鸟苷酸(c-di-GMP)小分子核苷酸c-di-GMP是细菌中普遍存在的第二信使,它在BF的形成和运动的切换之中作为胞内的信号分子而起着重要作用,近年来在生物界引起了广泛的关注。研究发现,它是通过改变病原体的生活方式,从而提高病原体的生存潜力。病原菌生活方式的转换受c-di-GMP在细胞内的浓度调控,低水平的c-di-GMP能促进细胞运动基因的表达,高水平c-di-GMP表现出促进细胞粘附因子的产生和胞外基质组分的增加,使细菌更易粘附在细胞或生物材料表面,有利于BF的形成[19]。c-di-GMP在细胞内的浓度受两种酶的调节:鸟苷酸环化酶(DGCs),含GGDEF结构域由两分子GTP合成c-di-GMP;磷酸二酯酶(PDEs),含一个EAL或HD-GYP结构域,可降解c-d-GMP,具有EAL结构域的磷酸二酯酶将c-di-GMP降解为线性的二核苷酸pGpG,HD-GYP结构域催化c-di-GMP降解为两分子的GMP分子。此外,还鉴定出了多种同时含有GGDEF和EAL结构域的蛋白质能同时调控c-di-GMP的合成和降解[20]。c-di-GMP调控BF形成中的重要作用是通过调控一些生物因子例如Psl、Pel、藻酸盐胞外多糖、表面粘附素CdrA和Ⅳ型菌毛的合成来实现的。许多研究表明,c-di-GMP在细胞内浓度的变化往往伴随着BF形成的变化[21]。
铜绿假单胞菌是研究BF的模型生物。研究表明,c-di-GMP能促进铜绿假单胞菌中的多种粘附素的产生。如Ⅳ型菌毛的合成依赖于具有c-di-GMP结合域的蛋白质PilZ和FimX的调控,此外,杯状菌毛的合成也受到c-di-GMP的调节[22]。CupA的表达依赖于具有GGDEF或c-di-GMP结构域的蛋白质WspR、MorA和PA1120[23]。PDE RocR被证明参与了CupB和CupC菌毛的合成,PDE PvrR参与了CupD菌毛的合成[24-25]。此外,CupA已被证明参与SDS诱导的自动聚集,这取决于由SiaD DGC介导的c-di-GMP在细胞内增长的水平。此外,表面蛋白CdrA已被证明能使绿脓杆菌细胞结合在Psl多糖上,c-di-GMP在转录水平正调控表面蛋白CdrA的产生。
研究发现,c-di-GMP还能促进psl多糖基因的转录[26]。当RsmA不被sRNA RsmY和RsmZ屏蔽时,发现psl的表达通过RsmA蛋白与psl mRNA的结合而在转录后水平被调节。Irie等报道Psl多糖能通过SiaD和SadC DGCs刺激c-di-GMP的合成[27],c-di-GMP浓度的升高会促进Psl多糖的合成和BF基质中其他组分产量的增加。胞外多糖可以通过特异性的正反馈调节通路促进BF的形成,这可能对浮游细菌吸附在现有的BF上起到一定的作用。藻酸盐是粘液型铜绿假单胞菌分泌的细胞外多糖和主要粘附因子,是组成BF的重要成分,c-di-GMP能够与作为藻酸盐合酶一部分的膜锚定蛋白Alg44的结构域PilZ相结合,从而正调控藻酸盐的产生。Pel多糖的合成在转录和翻译后水平都受到c-di-GMP的调节,对铜绿假单胞菌野生型菌株及wspF突变体(具有升高c-di-GMP水平的能力)进行的微阵列分析表明在wspF突变体中pel基因的转录水平增加。此外,还发现c-di-GMP与转录调节物FleQ结合,从而诱导pel基因的转录[28]。Pel多糖合成在翻译后水平通过c-di-GMP与PelD蛋白的结合而被激活[29]。
大量研究表明,c-di-GMP参与将急性感染转变为持续性感染[4, 30],与持续性感染相关的粘液表型在一定程度上受到c-di-GMP的调节,且高浓度的c-di-GMP可将细菌分泌系统从T3切换到T6,c-di-GMP的这些功能均对BF的形成具有调节作用。
3 双组分系统(Two component system,TCS)与sRNA双组分系统(TCS)是QS系统的核心部分,TCS参与调控细菌的多种重要生物学功能,包括与宿主的识别、细菌生长和营养代谢、毒力因子的表达、致病性及耐药性等[31]。TCS由两个部分组成:位于内膜中的组氨酸激酶(Histidine protein kinases,HPK)即“受体”和位于细胞质中的调节蛋白(Regulatory protein,RR)即“调控子”。HPK能感受胞外生物信号和环境条件的刺激并发生磷酸化,之后将磷酸基团转移给其耦联的RR保守的天冬氨酸残基上,磷酸化使RR被激活,激活后的RR可以结合在受调控基因的启动子上,进而调控基因的表达。
在铜绿假单胞菌中,目前已经鉴定出60多种双组分系统,其中Gac系统的研究是最深入的,它最初在丁香假单胞菌中被发现,由跨膜传感器激酶GacS (HPK)和其偶联调节物GacA (RR)组成,GasS具有一个自动磷酸化的保守组氨酸残基,被激活后的磷酸基团能转移到其偶联调节物GacA上,影响RsmZ和RsmY两个sRNA的转录[32]。这两个sRNA能结合在CsrA同源物RsmA上,从而影响RsmA蛋白的功能。RsmA是一个全局性的转录调控蛋白,可抑制QS、胞外产物和运动等不同靶基因的表达从而影响BF的形成[33]。最近在铜绿假单胞菌中又鉴定出了一个CsrA同系物RsmF,RsmF (也称为RsmN)是一种与mRNA结合的蛋白,与RsmA有多个共同的结合靶标,包括RsmY和RsmZ。然而与RsmA相比,RsmF与这些sRNA的结合亲和力明显较低。另一方面,RsmF不能结合pslA显示出了与RsmA的不同结合特性[34]。
SagS/BfiS是GacS调控网路的一个分支。SagS是细菌浮游态和生物被膜态的一个开关,并且是可以磷酸化的磷酸激酶。SagS通过不同调节途径调控RsmY和RsmZ,其对RsmY的调节依赖于一种组氨酸磷酸转移蛋白HptB[35],而其对RsmZ的调节是依赖于HptB与另一个磷酸激酶BfiS的相互作用。BfiS是生物被膜形成过程中细胞向不可逆附着过渡所必需的。BfiS、BfiR的同源RR可激活CafA (RNase G)的表达,CafA可以降低RsmZ的表达水平,而RsmZ的表达水平的降低是生物被膜成熟和维持所必需的[36]。当然,RsmY和RsmZ水平也受到其他调节因子的影响,例如Anr/NarL,它能在低氧条件下下调这两种sRNA,而β-内酰胺酶调节剂AmpR,可以上调RsmZ[37-38]。在细菌中这些sRNA的转录水平由多个交叉调节系统紧密调控,以调控细菌从浮游态生长模式到生物被膜生长模式的转变。
除传感器激酶GacS外,最近又发现了两种传感器激酶RetS (调节胞外多糖和Ⅲ型分泌系统)和LadS (降低细菌粘附能力)是通过GacA调节基因表达进而影响BF的形成。在探索RetS对GacS/A的影响中,其实是通过在RetS和GacS之间形成异二聚体,进而导致GacS的自磷酸化被阻断,表明RetS是GacS的拮抗剂[39]。LadS通过GacA激活RsmY和RsmZ而具有与RetS相反的功能[40-41],这表明LadS有助于BF的形成。最近研究报道了一种新的与RsmA结合的RsmY/RsmZ型sRNA,被称为RsmW,研究发现它不受GacA调控,与rsmY和rsmZ表达相反,并发现它的表达上调会促进BF的形成[42]。
Gac系统除了通过调节QS影响BF的形成外,通过体外研究发现它还通过调节胞外多糖Pel、Psl和Ⅳ型菌毛,参与细菌浮游态与BF生长模式之间的转换。Gac系统被认为通过RsmA来调节与type-Ⅲ和type-Ⅳ分泌系统(T3SS和T6SS)相关的基因的表达,在急性和持续性感染之间起转换作用[26]。体外研究表明,急性感染通常与T3SS产生高毒力因子有关,而与BF有关的持续性感染则表现出较弱的T6SS表达[43]。通过对突变体的研究,得出这一转变是由两个传感器RetS和LadS来促成的。retS和rsmA的突变体表现出相似的表型,胞外多糖产生增多,BF增多,T3SS的表达减少,以及Ⅳ型菌毛活力降低[44]。与此相反,ladS突变体表现出T3SS的表达增强BF形成量降低。近年来,尽管对Gac系统进行了深入研究,但是由传感器激酶GacS、LadS和RetS检测到的触发磷酸化反应的信号仍未鉴定出来,寻找出传感器激酶的激活剂是绝大多数双组分系统尚未解决的问题[45]。然而,寻找传感器激酶的激活剂有其重要意义,因为可以通过它们来控制传感器激酶的激活与否,从而调控细菌的行为,这对于细菌感染的治疗是非常有用的。
总之,Gac调控网络是一种复杂的多激酶网络,在调控细菌浮游态生长模式和生物被膜生长模式之间起主要作用。调控网络的复杂性和大量不同的传感器是整合大量信号分子并作出最适合细菌生存指令所必需的。
4 展望信号分子可以在细菌生长的特定环境中调节其生命活动,与BF相关的信号分子能使细菌对周围其他细菌的存在作出反应,进而调整自身的生命活动。在过去十年中,有关信号分子调控致病菌BF形成机制的研究已取得显著进步,但寻找信号分子识别的受体仍然是信号转导研究的瓶颈。而且生物被膜的形成是由QS、c-di-GMP和双组分系统等共同调节,有意思的是这3个系统彼此之间存在着一些相同功能,如上文所述高水平c-di-GMP和Gac系统的激活均能诱导生物被膜形成和持续性感染,这种特性增加了寻找这些信号分子受体蛋白的难度,也使得生物被膜形成网络更加复杂。因此,充分了解参与形成复杂聚合物的细胞间信号转导过程是一项艰巨的挑战。但是换言之,与QS、TCS和c-di-GMP相关的信号分子均可以作为细菌生物被膜的研究靶标。通过发掘QS、c-di-GMP和TCS对生物被膜形成的机制,将为寻找新的靶点作用药物奠定基础,通过干扰生物被膜形成的某个环节,将有望产生有效的抗感染治疗方法,为彻底治疗临床难治的持续性感染带来新希望。
| [1] | Flemming HC, Wingender J. The biofilm matrix. Nat Rev Microbiol, 2010, 8(9): 623–633. |
| [2] | Høiby N, Bjarnsholt T, Givskov M, et al. Antibiotic resistance of bacterial biofilms. Int J Antimicro Agents, 2010, 35(4): 322–332. DOI: 10.1016/j.ijantimicag.2009.12.011 |
| [3] | Costerton JW, Stewart PS, Greenberg EP. Bacterial biofilms: a common cause of persistent infections. Science, 1999, 284(5418): 1318–1322. DOI: 10.1126/science.284.5418.1318 |
| [4] | Jakobsen TH, Tolker-Nielsen T, Givskov M. Bacterial biofilm control by perturbation of bacterial signaling processes. Int J Mol Sci, 2017, 18(9): 1970. |
| [5] | Solano C, Echeverz M, Iñigo L. Biofilm dispersion and quorum sensing. Curr Opin Microbiol, 2014, 18(4): 96–104. |
| [6] | Nealson KH, Hastings JW. Bacterial bioluminescence: its control and ecological significance. Microbiol Rev, 1979, 43(4): 496–518. |
| [7] | Loughran AJ, Atwood DN, Anthony AC, et al. Impact of individual extracellular proteases on Staphylococcus aureus biofilm formation in diverse clinical isolates and their isogenic sarA mutants. Microbiologyopen, 2014, 3(6): 897–909. DOI: 10.1002/mbo3.2014.3.issue-6 |
| [8] | Yarwood JM, Bartels DJ, Volper EM, et al. Quorum sensing in Staphylococcus aureus biofilms. J Bacteriol, 2004, 186(6): 1838–1850. DOI: 10.1128/JB.186.6.1838-1850.2004 |
| [9] | Dai L, Yang L, Parsons C, et al. Staphylococcus epidermidis recovered from indwelling catheters exhibit enhanced biofilm dispersal and "self-renewal" through downregulation of agr. BMC Microbiol, 2012, 12(1): 102. DOI: 10.1186/1471-2180-12-102 |
| [10] | Schaber JA, Carty NL, Mcdonald NA, et al. Analysis of quorum sensing-deficient clinical isolates of Pseudomonas aeruginosa. J Med Microbiol, 2004, 53(9): 841–853. DOI: 10.1099/jmm.0.45617-0 |
| [11] | Sauer K, Cullen MC, Rickard AH, et al. Characterization of nutrient-induced dispersion in Pseudomonas aeruginosa PAO1 biofilm. J Bacteriol, 2004, 186(21): 7312–7326. |
| [12] | Whitehead NA, Barnard AML, Slater H, et al. Quorum-sensing in Gram-negative bacteria. FEMS Microbiol Rev, 2001, 25(4): 365–404. |
| [13] | Fuqua WC, Winans SC, Greenberg EP. Quorum sensing in bacteria: the LuxR-LuxI family of cell density-responsive transcriptional regulators. J Bacteriol, 1994, 176(2): 269–275. |
| [14] | Wang Y, Wang YX, Sun LY, et al. The LuxS/AI-2 system of Streptococcus suis. Appl Microbiol Biotechnol, 2018, 102(17): 7231–7238. DOI: 10.1007/s00253-018-9170-7 |
| [15] | Wang Y, Li Y, Zhang ZC, et al. Overexpression of luxS cannot increase autoinducer-2 production, only affect the growth and biofilm formation in Streptococcus suis. Sci World J, 2013, 2013: 924276. |
| [16] | Wang Y, Zhang W, Wu ZF, et al. Functional analysis of luxS in Streptococcus suis reveals a key role in biofilm formation and virulence. Vet Microbiol, 2011, 152(1/2): 151–160. |
| [17] | Wang Y, Yi L, Wu ZF, et al. Comparative proteomic analysis of Streptococcus suis biofilms and planktonic cells that identified biofilm infection-related immunogenic proteins. PLoS ONE, 2012, 7(4): e33371. DOI: 10.1371/journal.pone.0033371 |
| [18] | Wang Y, Yi L, Wang SH, et al. Crystal structure and identification of two key amino acids involved in AI-2 production and biofilm formation in Streptococcus suis LuxS. PLoS ONE, 2015, 10(10): e0138826. DOI: 10.1371/journal.pone.0138826 |
| [19] | Valentini M, Filloux A. Biofilms and cyclic di-GMP (c-di-GMP) signaling: lessons from Pseudomonas aeruginosa and other bacteria. J Biol Chem, 2016, 291(24): 12547–12555. |
| [20] | Galperin MY. Bacterial signal transduction network in a genomic perspective. Environ Microbiol, 2004, 6(6): 552–567. |
| [21] | Díaz M, Castro M, Copaja S, et al. Biofilm Formation by the acidophile bacterium Acidithiobacillus thiooxidans involves c-di-GMP pathway and Pel exopolysaccharide. Genes, 2018, 9(2): 113. DOI: 10.3390/genes9020113 |
| [22] | Francis VI, Stevenson EC, Porter SL. Two-component systems required for virulence in Pseudomonas aeruginosa. FEMS Microbiol Lett, 2017, 364(11): fnx104. |
| [23] | Meissner A, Wild V, Simm R, et al. 78 Pseudomonas aeruginosa cupA-encoded fimbriae expression is regulated by a GGDEF and EAL domain-dependent modulation of the intracellular level of cyclic diguanylate. Environ Microbiol, 2007, 9(10): 2475–2485. |
| [24] | Rao F, Yang Y, Qi Y, et al. Catalytic Mechanism of Cyclic Di-GMP-specific phosphodiesterase: a Study of the EAL domain-containing RocR from Pseudomonas aeruginosa. J Bacteriol, 2008, 190(10): 3622–3631. DOI: 10.1128/JB.00165-08 |
| [25] | Kulasekara HD, Ventre I, Kulasekara BR, et al. A novel two-component system controls the expression of Pseudomonas aeruginosa fimbrial cup genes. Mol Microbiol, 2005, 55(2): 368–380. |
| [26] | Irie Y, Starkey M, Edwards AN, et al. Pseudomonas aeruginosa biofilm matrix polysaccharide Psl is regulated transcriptionally by RpoS and post-transcriptionally by RsmA. Mol Microbiol, 2010, 78(1): 158–172. |
| [27] | Irie Y, Borlee BR, O'Connor JR, et al. Self-produced exopolysaccharide is a signal that stimulates biofilm formation in Pseudomonas aeruginosa. Proc Natl Acad Sci USA, 2012, 109(50): 20632–20636. DOI: 10.1073/pnas.1217993109 |
| [28] | Hickman JW, Harwood CS. Identification of FleQ from Pseudomonas aeruginosa as ac-di-GMP-responsive transcription factor. Mol Microbiol, 2008, 69(2): 376–389. |
| [29] | Lee VT, Matewish JM, Kessler JL, et al. A cyclic-di-GMP receptor required for bacterial exopolysaccharide production. Mol Microbiol, 2007, 65(6): 1474–1484. |
| [30] | Klockgether J, Tümmler B. Recent advances in understanding Pseudomonas aeruginosa as a pathogen. F1000 Res, 2017: 61261. |
| [31] | Guo XP, Sun YC. New insights into the non-orthodox two component rcs phosphorelay system. Front Microbiol, 2017, 8: 2014. DOI: 10.3389/fmicb.2017.02014 |
| [32] | Kay E, Humair B, Dénervaud V, et al. Two GacA-dependent small RNAs modulate the quorum-sensing response in Pseudomonas aeruginosa. J Bacteriol, 2006, 188(16): 6026–6033. DOI: 10.1128/JB.00409-06 |
| [33] | Lapouge K, Schubert M, Allain FH, et al. Gac/Rsm signal transduction pathway of gamma-proteobacteria: from RNA recognition to regulation of social behaviour. Mol Microbiol, 2008, 67(2): 241–253. |
| [34] | Marden JN, Diaz MR, Walton WG, et al. An unusual CsrA family member operates in series with RsmA to amplify posttranscriptional responses in Pseudomonas aeruginosa. Proc Natl Acad Sci USA, 2013, 110(37): 15055–15060. DOI: 10.1073/pnas.1307217110 |
| [35] | Bordi C, Marie-Cécile L, Ventre I, et al. Regulatory RNAs and the HptB/RetS signalling pathways fine-tune Pseudomonas aeruginosa pathogenesis. Mol Microbiol, 2010, 76(6): 1427–1443. DOI: 10.1111/j.1365-2958.2010.07146.x |
| [36] | Petrova OE, Sauer K. The novel two-component regulatory system BfiSR regulates biofilm development by controlling the small RNA rsmZ through CafA. J Bacteriol, 2010, 192(20): 5275–5288. DOI: 10.1128/JB.00387-10 |
| [37] | O'Callaghan J, Reen FJ, Adams C, et al. Low oxygen induces the type Ⅲ secretion system in Pseudomonas aeruginosa via modulation of the small RNAs rsmZ and rsmY. Microbiology, 2011, 157(12): 3417–3428. DOI: 10.1099/mic.0.052050-0 |
| [38] | Balasubramanian D, Kumari H, Mathee K. Pseudomonas aeruginosa AmpR: an acute-chronic switch regulator. Pathogens & Disease, 2015, 73(2): 1. |
| [39] | Goodman AL, Merighi M, Hyodo M, et al. Direct interaction between sensor kinase proteins mediates acute and chronic disease phenotypes in a bacterial pathogen. Genes Dev, 2009, 23(2): 249–259. |
| [40] | Chambonnier G, Roux L, Redelberger D, et al. The hybrid histidine kinase lads forms a multicomponent signal transduction system with the GacS/GacA two-component system in Pseudomonas aeruginosa. PLoS Genetics, 2016, 12(5): e1006032. DOI: 10.1371/journal.pgen.1006032 |
| [41] | Broder UN, Jaeger T, Jenal U. LadS is a calcium-responsive kinase that induces acute-to-chronic virulence switch in Pseudomonas aeruginosa. Nat Microbiol, 2016, 2: 16184. |
| [42] | Miller CL, Romero M, Karna SL, et al. RsmW, Pseudomonas aeruginosa small non-coding RsmA-binding RNA upregulated in biofilm versus planktonic growth conditions. BMC Microbiol, 2016, 16(1): 155. DOI: 10.1186/s12866-016-0771-y |
| [43] | Mougous JD, Cuff ME, Raunser S, et al. A virulence locus of Pseudomonas aeruginosa encodes a protein secretion apparatus. Science, 2006, 312(5779): 1526–1530. DOI: 10.1126/science.1128393 |
| [44] | Mikkelsen H, Mcmullan R, Filloux A. The Pseudomonas aeruginosa reference strain PA14 displays increased virulence due to a mutation in ladS. PLoS ONE, 2011, 6(12): e29113. DOI: 10.1371/journal.pone.0029113 |
| [45] | Jimenez PN, Koch G, Thompson JA, et al. The multiple signaling systems regulating virulence in Pseudomonas aeruginosa. Microbiol Mol Biol Rev, 2012, 76(1): 46–65. DOI: 10.1128/MMBR.05007-11 |
 2019, Vol. 35
2019, Vol. 35