中国科学院微生物研究所、中国微生物学会主办
文章信息
- 乔郅钠, 徐美娟, 龙梦飞, 杨套伟, 张显, 中西秀树, 饶志明
- Qiao Zhina, Xu Meijuan, Long Mengfei, Yang Taowei, Zhang Xian, Hideki Nakanishi, Rao Zhiming
- TCA循环关键节点对L-谷氨酸合成的影响
- Effect of key notes of TCA cycle on L-glutamate production
- 生物工程学报, 2020, 36(10): 2113-2125
- Chinese Journal of Biotechnology, 2020, 36(10): 2113-2125
- 10.13345/j.cjb.200038
-
文章历史
- Received: January 20, 2020
- Accepted: March 2, 2020
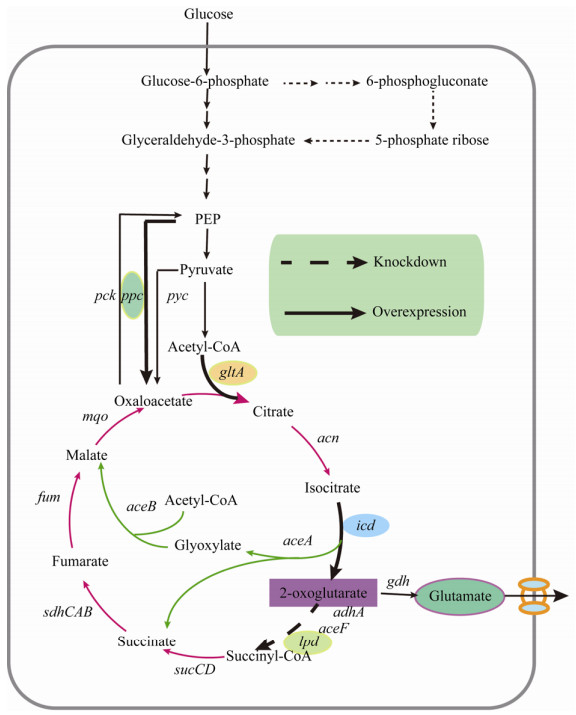
谷氨酸是一种重要的氨基酸,在食品、医药、饲料和化妆品等行业具有广泛应用,市场需求量大[1],且谷氨酸高值化产品(如聚谷氨酸[2]、γ-氨基丁酸[3]等)已成为目前研究热点,因此选育和开发一株谷氨酸高产菌株是非常有研究意义的,谷氨酸棒杆菌中谷氨酸的生物合成途径如图 1所示。实验室前期以谷氨酸生产菌株谷氨酸棒杆菌Corynebacterium glutamicum E01 (其谷氨酸产量可达40.8 g/L)为出发菌株,经逐级诱变(包括基因组改组技术和等离子诱变ARTP等)筛选,获得1株谷氨酸产量较高的菌株C. glutamicum G01,其产量高达93.53 g/L。对C. glutamicum E01和C. glutamicum G01进行全局RNA-seq和重测序分析,找出引起谷氨酸高产的关键基因,进一步对C. glutamicum G01菌株进行代谢改造以改善谷氨酸产量。
|
| 图 1 谷氨酸棒杆菌中谷氨酸生物合成途径 Fig. 1 Glutamate biosynthetic pathway in C. glutamicum. |
| |
在谷氨酸棒杆菌中,中心代谢途径对谷氨酸的生产非常重要。已有研究表明,谷氨酸高产菌株中α-酮戊二酸脱氢酶复合体ODHC (由3个亚基组成,其中E1由odhA基因编码,E2由aceF基因编码,E3由lpd基因编码)酶活水平较低[4],ODHC能够催化谷氨酸的前体物质α-酮戊二酸形成琥珀酰-CoA,因此,ODHC活性的降低能够改变代谢分布,使其集中流向谷氨酸合成方向,有利于谷氨酸的大量合成[5]。磷酸烯醇式丙酮酸羧化酶PEPC (编码基因ppc)在TCA循环到氨基酸合成的碳流中起关键作用,该酶被认为是谷氨酸棒杆菌氨基酸生产菌株的重要潜在靶标,近年来,已有很多研究者研究了其对谷氨酸生产的影响[6-8]。柠檬酸合酶CS (编码基因gltA)催化TCA循环的初始反应,控制进入TCA循环的速度,CS的过表达可以将更多的碳通量重定向到TCA循环中[9]。异柠檬酸脱氢酶ICD (编码基因icd)能够将异柠檬酸氧化去甲酸酯化为谷氨酸合成前体物质α-酮戊二酸[10],相关研究表明,ICD的过表达可能会使更多的碳流量流向α-酮戊二酸以积累更多的α-酮戊二酸[11]。
本研究通过对C. glutamicum E01和C. glutamicum G01进行转录组与重测序分析,挑选中心代谢途径中转录水平和基因水平上存在差异的基因进行研究,测试其对谷氨酸生产的扰动影响。结合核糖体结合位点优化(RBS优化)和CRISPR/dCas9敲弱系统构建了谷氨酸高产菌株,实现了谷氨酸产量和糖酸转化率的提高,为谷氨酸棒杆菌的代谢工程改造提供了理论基础。
1 材料与方法 1.1 菌株与质粒菌株C. glutamicum E01 (专利公开号为103215198B)、C. glutamicum G01 (中国典型培养物保藏中心,保藏号:M2013418)、大肠杆菌Escherichia coli BL21(DE3) (基因型:F– ompT gal dcm lon hsdSB(rB– mB–)λ(DE3 lacI lacUV5-T7 gene 1 ind1 sam7 nin5),表达宿主)和质粒pDXW-10 (E. coli-C. glutamicum穿梭表达质粒,Ptac启动子,Kmr)、pFSC-dCas9 (pXMJ19包含dCas9基因、SD序列和Ptac启动子,Cmr)为实验室保存,pFST质粒(pEC-XK99E温敏型质粒,Ptrc启动子,Kmr)为江南大学白仲虎教授馈赠。
1.2 培养基LB培养基(g/L):蛋白胨10,酵母粉5,NaCl 10,固体培养基添加琼脂15–20,根据需要加入相应浓度抗生素。
LBG培养基(g/L):LB培养基+葡萄糖5,固体培养基添加琼脂15–20,根据需要加入相应浓度抗生素;谷氨酸棒杆菌平板活化和诱导表达用。
种子培养基(g/L):葡萄糖25,K2HPO4 1.5,MgSO4 0.6,玉米浆30,FeSO4·7H2O 0.005,MnSO4·H2O 0.005,尿素2.5 (与其他组分分开灭菌),用200 g/L NaOH溶液调节pH至7.0,115 ℃灭菌20 min;谷氨酸棒杆菌上罐发酵养种子用。
发酵培养基(g/L):葡萄糖140,K2HPO4 1.0,MgSO4 0.6,玉米浆5.0,FeSO4·7H2O 0.005,MnSO4·H2O 0.005,尿素7.0 (与其他组分分开灭菌),用200 g/L NaOH调pH至7.0,115 ℃灭菌20 min;谷氨酸棒杆菌发酵用。
补料培养基:80%葡萄糖;谷氨酸棒杆菌发酵补料用。
1.3 引物本研究引物如表 1所示。
| Primer name | Primer sequence (5′–3′) | Restriction site |
| pDXW-10-ppc-F | TTCACACAGGAAACAGAATTC-Sequence of Synthetic RBS-ATGACTGATTTTTTACGCGATGACA Sequence of Synthetic RBS 1: AAAGGAGGTGCGC 2: AAAGGAGTTGCTT 3: AAAGGAGCGGTCC 4: AAAGGAGGGAAATC 5: AAAGGAGGTTGTC 6: AAAGGAGGCTACT |
EcoRⅠ |
| pDXW-10-ppc-R | CATCCGCCAAAACAGAAGCTTTTAGCCGGAGTTGCGCAGCGCA | Hind Ⅲ |
| pDXW-10-gltA-F | TTCACACAGGAAACAGAATTCAAAGGAGGGAAATCATGAGTGACAGCCAAGTCCGCAAAGGACTC | EcoRⅠ |
| pDXW-10-gltA-R | CATCCGCCAAAACAGAAGCTT TTAGGTTCTCTCCGAAATGGGC | Hind Ⅲ |
| pDXW-10-icd-F | TTCACACAGGAAACAGAATTCAAAGGAGGGAAATCATGGCTAAGATCATCTGGACCCGCACCGAC | EcoRⅠ |
| pDXW-10-icd-R | CATCCGCCAAAACAGAAGCTTTTACTTCTTCAGTGCGTCAACG | Hind Ⅲ |
| dCas9-F | AGGAAACAGACCATGGAATTCAAAGGAGGACAACTAATGGATAAAAAGTATTCCATTGGCCTGGCA | EcoRⅠ |
| dCas9-Ptrc-R1 | TGCACCGTGCAGTCGTTAGTCGCCACCCAG | |
| sgRNA-F | AGGAAACAGACCATGGAATTC-N20-GTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCC N20 Sequences: 1-TAGTACTCGGAGCCGGCCCC 2-CGGATGCACGACAATGACCC 3-ACACCTGCATTTCTGTGCCC 4-AAACACGTATCCTTGAATGC 5-ATGGAAAACCAACCGCACCC |
EcoRⅠ |
| sgRNA-R | TCCGCCAAAACAGCCAAGCTTAAAAAAGCACCGACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTT | Hind Ⅲ |
| dCas9-Ptrc-F1 | CTGGGTGGCGACTAACGACTGCACGGTGCA | |
| dCas9-Ptrc-R | TCCGCCAAAACAGCCAAGCTTAAAAAAGCACCGACCGACTCGG | Hind Ⅲ |
| 16S rRNA F | GCCCAGGTAAGGTTCTT | |
| 16S rRNA R | GGTGTAGCGGTGAAATGC | |
| lpd F | CGTTGGAGGCGTTCATTA | |
| lpd R | GGAGACTTCAGCATCTTCAT |
将出发菌株C. glutamicum E01和谷氨酸高产菌C. glutamicum G01进行5 L发酵罐发酵,取对数期(发酵18 h,胞外L-谷氨酸开始积累)的发酵样品,4 ℃、6 000 r/min离心10 min,液氮冻存10 min后,置于-80 ℃冰箱储存,送苏州金唯智有限公司进行总RNA-seq和重测序分析。
1.5 重组菌株的构建与表达 1.5.1 过表达菌株的构建与表达以C. glutamicum G01基因组为模板,以相应引物为模板PCR扩增获得各基因片段,回收后与经EcoRⅠ和Hind Ⅲ双酶切获得的pDXW-10线性化载体进行同源重组连接,各连接产物分别转化E. coli BL21(DE3)。筛选后,提取重组质粒进行PCR验证,送苏州金唯智有限公司进行测序分析。验证正确的各重组质粒分别电转C. glutamicum G01,涂布卡那霉素平板进行筛选,验证正确的重组菌株进行命名。
构建好的各重组谷氨酸棒杆菌及菌株C. glutamicum G01在LBG固体平板上划线活化后,挑取单菌落接入10 mL LBG液体小瓶,于30 ℃、180 r/min培养16–24 h后按1%接种量转接至50 mL LBG液体培养基,30 ℃、180 r/min培养4–5 h后,添加IPTG至终浓度为0.5 mmol/L,继续于30 ℃、180 r/min诱导表达8–12 h后,4 ℃下离心收集菌体细胞。收集的细胞分别用PBS缓冲液(0.1 mol/L,pH 7.4)洗涤2次后,重新悬浮于PBS缓冲液中,控制菌体浓度一致的情况下,用超声破碎仪破碎,破碎液在4 ℃下离心20 min,收集上清,进行SDS-PAGE分析,所得粗酶液用于后续的酶活测定。
1.5.2 敲弱菌株的构建以质粒pFSC-dCas9为模板,dCas9-F和dCas9-Ptrc-R1为引物进行PCR,扩增出含Ptac部分片段的dCas9基因片段。
以表 1所列引物(引物上设同源臂) PCR扩增获得含不同N20序列的sgRNA片段,胶回收后与经EcoRⅠ酶切获得的pFST线性化载体进行同源重组连接并将各连接产物分别转化E. coli BL21(DE3)。经筛选后,提取重组质粒进行PCR验证,将重组质粒分别命名为pFST-S1、pFST-S2、pFST-S3、pFST-S4和pFST-S5,即不同sgRNA载体构建完成[12-14]。
之后,以含Ptac部分片段的dCas9基因片段和pFST-S1 (pFST-S2、pFST-S3、pFST-S4、pFST-S5)重组质粒为模板进行融合PCR[15],获得含dCas9-Ptrc- sgRNA的融合片段,胶回收后的各融合片段与经EcoRⅠ和Hind Ⅲ双酶切获得的pFST线性化载体进行同源重组连接并化转至E. coli BL21(DE3)。经筛选后,提取重组质粒进行PCR验证,将重组质粒分别命名为pFST-dCas9-S1、pFST-dCas9-S2、pFST-dCas9-S3、pFST-dCas9-S4和pFST-dCas9-S5。
将质粒pFST-dCas9-S1、pFST-dCas9-S2、pFST-dCas9-S3、pFST-dCas9-S4和pFST-dCas9-S5分别电转C. glutamicum E01,筛选后验证正确的重组菌株分别命名为CG1、CG2、CG3、CG4和CG5。
1.6 相关酶的酶活测定磷酸烯醇式丙酮酸羧化酶酶活测定:采用苹果酸脱氢酶偶联法测定,酶活定义为在30 ℃下每分钟氧化1 μmol NADH所需的酶量为1 U[16]。
柠檬酸合酶酶活测定:通过5, 5′-二硫代双(2-硝基苯甲酸)测定在412 nm下的吸光值[9]。
异柠檬酸脱氢酶酶活测定:酶活力定义为30 ℃下每分钟催化底物NADP+产生1 μmol NADPH所需的酶量[17]。
LPD酶活的测定:于30 ℃下进行反应,每5 min读数1次,连续测15 min。酶活性单位定义为在上述反应条件下每分钟氧化1 μmol NADH所需的酶量为1 U[18-19]。
1.7 RT-qPCR收集培养至对数期的细胞,根据FastPure Cell/Tissue Total RNA Isolation Kit的使用说明书提取RNA。使用HiScript Q Setect RT SuperMix for qPCR试剂盒,将RNA (1 μg)转录成cDNA。使用ABI QuantStudio™ 3D通过实时PCR定量产物。反应混合物(20 μL)含有10 μL 2×ChamQ Universal SYBR qPCR Master Mix和各0.4 μL基因的上下游引物(表 1)。以RNase-free ddH2O作为阴性对照,验证Mix和引物有无污染。以基因组DNA作为阳性对照,验证提取的cDNA有无污染。
1.8 重组菌株发酵菌株经平板活化后,挑取单菌落分别接入含10 mL种子培养基的50 mL摇瓶中,30 ℃、180 r/min摇床培养24 h至OD600约为20.0作为一级种子液。10 mL一级种子液全部接入含200 mL种子培养基的1.0 L带挡板三角瓶中,30 ℃、180 r/min摇床培养18 h至OD600约为30.0作为二级种子液。二级种子全部接入装有2.0 L发酵培养基的5 L发酵罐中。发酵罐发酵温度30 ℃,搅拌转速600 r/min,通气量3 L/min,pH 7.0 (补加50%氨水自动控制)。发酵过程中当葡萄糖浓度降至约30 g/L时补加80%的葡萄糖溶液。
1.9 发酵过程中相关参数测定发酵液菌体浓度适当稀释后,在600 nm波长下检测其吸光度,OD600代表菌体浓度,葡萄糖和谷氨酸含量用生物传感分析仪SBA-40E进行测定[20-21]。
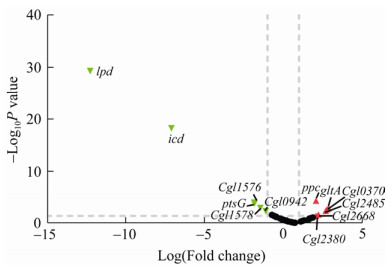
2 结果与分析 2.1 RNA-seq数据分析对RNA-seq的中心代谢途径相关基因进行集中分析,期望能够找到对谷氨酸产量有显著影响的基因。图 2中对葡萄糖吸收的3个编码基因ptsG、ptsI和ptsH,EMP途径的20个相关基因,戊糖磷酸途径的15个相关基因和TCA循环的15个相关基因进行转录水平差异分析,其中lpd和icd基因转录水平下调显著,gltA和ppc基因转录水平上调显著。
|
| 图 2 中心代谢路径基因表达差异图 Fig. 2 Difference map of gene expression in central metabolic pathway. |
| |
为了验证转录水平显著差异的基因lpd、icd、gltA和ppc在基因水平上是否发生变化,进行了重测序分析,结果显示,lpd、icd、gltA和ppc基因的氨基酸序列均发生突变,在基因水平上发生了变化(表 2)。
| Genes | C. glutamicum G01 |
| lpd | K67R, I108T, L122I, T125A, I134T, D154E, F162Y, E295D, D305P, T341A, S359F, E370Q |
| icd | I372V |
| gltA | A4T, S79C, S156T, A243E, A252D |
| ppc | Q14R, Y85H, G241D, E267G, H274R, K303E, A310E, K383T, A408S, A433T, A441E, Q542R, D549G, R634K, A704T, L773F, T829R, R840H |
以上分析所得差异基因主要位于TCA循环中草酰乙酸节点和α-酮戊二酸代谢节点处,可见,这两个节点对谷氨酸生产影响较为显著,后面将展开研究。
2.2 草酰乙酸节点重组谷氨酸棒杆菌的构建草酰乙酸OAA节点在谷氨酸合成途径中扮演着重要角色。因此,探索研究OAA节点处转录水平和基因水平均发生了变化的ppc和gltA基因在C. glutamicum G01中对生产谷氨酸的影响具有重要意义。
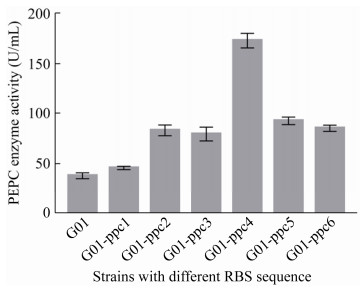
2.2.1 C. glutamicum G01/pDXW-10-ppc菌株的构建和酶活力测定首先,将参考文献[22]中效果较好的RBS序列用于ppc基因的优化表达,构建含不同RBS序列的PEPC过表达菌株(G01-ppc1、G01-ppc2、G01-ppc3、G01-ppc4、G01-ppc5和G01-ppc6,原始菌株用G01表示),测试PEPC酶活水平以确定最优RBS序列。
酶活测定结果显示,G01-ppc4菌株的PEPC酶活为(172.89±7.23) U/mL,较原始菌的(38.52±2.87) U/mL提高了3.5倍(图 3),RBS-4序列的表达效果较好。采用RBS-4序列构建的重组菌株命名为C. glutamicum G01/pDXW-10-ppc,RBS-4序列将用于后面其他基因的过表达。
|
| 图 3 PEPC过表达菌株的酶活水平 Fig. 3 Enzyme activity level of PEPC overexpression strains. |
| |
将RBS-4序列用于gltA基因的过表达,PCR鉴定结果如图 4A所示,条带大小为1 146 bp,与gltA基因大小相符,即菌株C. glutamicum G01/pDXW-10-gltA构建成功。在控制菌体浓度一致的条件下,C. glutamicum G01/pDXW-10-gltA菌株中CS的表达量均明显高于对照菌株,其大小约为42.6 kDa,即CS在C. glutamicum G01中成功表达(图 4B)。
|
| 图 4 C. glutamicum G01/pDXW-10-gltA菌株的PCR鉴定结果(A)和表达情况(B) Fig. 4 PCR results (A) and expression (B) of C. glutamicum G01/pDXW-10-gltA strain. (A) M: 10 000 bp nucleic acid marker; 1 and 2: PCR results of gltA gene. (B) M: protein marker; 1: crude enzyme solution of control strain; 2: crude enzyme solution of C. glutamicum G01/pDXW-10-gltA strain. |
| |
酶活测定结果显示,C. glutamicum G01/pDXW-10-gltA菌株CS酶活为(148.36±2.56) U/mL,较C. glutamicum G01的(18.87±1.68) U/mL提高了约6.9倍,即gltA基因在C. glutamicum G01中的过表达使得C. glutamicum G01/pDXW-10-gltA中的CS酶活提高。
2.3 α-酮戊二酸代谢节点重组谷氨酸棒杆菌的构建降低ODHC复合体活性是将α-酮戊二酸通量重定向至谷氨酸合成而不是琥珀酰CoA合成的一种必不可少的方法[4, 16]。敲除odhA虽然完全消除了ODHC活性,但是会阻断TCA循环,严重影响菌体生长[5]。有研究者通过改变RBS序列[23]或翻译起始密码子[24],或者使用odhA反义RNA[25]和odhA抑制蛋白OdhI[26-28]来降低ODHC活性。此外,理论上异柠檬酸脱氢酶ICD可能会改变α-酮戊二酸节点处的代谢流,ICD酶活力的提高有利于α-酮戊二酸的积累[11]。因此,接下来将会对异柠檬酸脱氢酶和ODHC的E3亚基展开研究,探索其对谷氨酸生产的影响。
2.3.1 CRISPR/dCas9系统用于C. glutamicum E01中lpd基因的弱化及弱化程度测试从转录组测序结果看,C. glutamicum E01菌株中LPD蛋白的表达量较高,因此,选择在该菌株中进行lpd基因弱化实验,筛选出对lpd基因弱化程度最高的sgRNA后再在C. glutamicum G01中实验,以获得谷氨酸产量的进一步提高。
酶活测定结果显示,LPD敲弱菌株CG1、CG2、CG3、CG4和CG5中LPD酶活分别较原始菌下降了3.1%、5.4%、21.1%、3.0%和6.0% (图 5A,原始菌株用CG0表示)。RT-qPCR结果显示(图 5B),各敲除菌株中lpd基因的转录水平均呈现一定程度的降低,CG3菌株中lpd基因转录水平最低,且相比于其他sgRNA,sgRNA-3对lpd基因的弱化程度更高。后续将会使用sgRNA-3来实现lpd基因在C. glutamicum G01中的弱化。
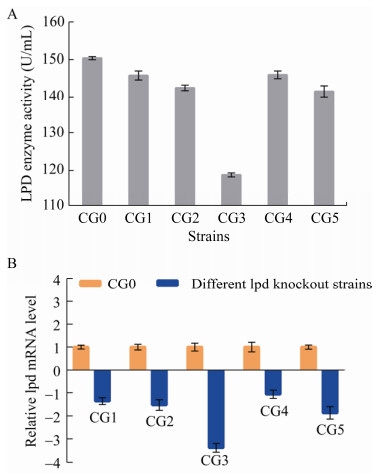
|
| 图 5 lpd敲弱菌株中LPD酶活(A) x与lpd基因转录水平(B) Fig. 5 LPD enzyme activity (A) and lpd gene transcription level (B) of lpd knockout strains. |
| |
将pFST-dCas9-S3敲弱质粒电转C. glutamicum G01,提取重组质粒用dCas9-F和sgRNA-R引物进行PCR验证,结果如图 6所示,泳道中条带大小为4 467 bp左右,与dCas9-Ptrc-sgRNA融合片段大小相符,即dCas9-Ptrc-sgRNA片段与pFST质粒连接成功,即lpd基因的敲弱菌株C. glutamicum DL01构建成功。
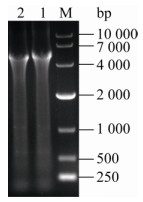
|
| 图 6 C. glutamicum DL01菌株的PCR鉴定结果 Fig. 6 PCR identification results of C. glutamicum DL01 strain. |
| |
将RBS-4序列用于icd基因在C. glutamicum G01中的过表达,PCR鉴定结果显示,泳道中条带大小约为2 217 bp,与基因icd大小相符,即C. glutamicum G01/pDXW-10-icd构建成功(图 7A)。在控制菌体浓度一致的条件下,C. glutamicum G01/pDXW-10-icd菌株中ICD的表达量均明显高于对照菌株,其大小约为80.1 kDa,即ICD在C. glutamicum G01中成功表达(图 7B)。
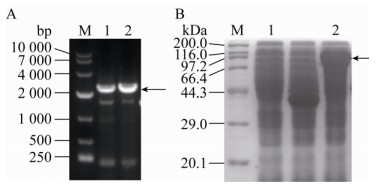
|
| 图 7 C. glutamicum G01/pDXW-10-icd菌株的PCR鉴定结果(A)和表达情况(B) Fig. 7 PCR results (A) and expression (B) of C. glutamicum G01/pDXW-10-icd strain. (A) M: 10 000 bp nucleic acid marker; 1 and 2: PCR results of icd gene. (B) M: protein marker; 1: crude enzyme solution of control strain; 2: crude enzyme solution of C. glutamicum G01/pDXW-10-icd strain. |
| |
酶活测定结果显示,C. glutamicum G01/pDXW-10-icd菌株中ICD酶活为(506.45± 4.81) mU/mL,较原始菌的(60.87±2.57) mU/mL提高了7.3倍,即icd基因在C. glutamicum G01中的过表达提高了C. glutamicum G01/pDXW-10-icd中ICD的酶活力。
2.4 发酵测试重组菌株产谷氨酸水平由图 8可知,发酵48 h后,菌株C. glutamicum G01谷氨酸产量达到(93.53±4.52) g/L,糖酸转化率为45.2%;C. glutamicum G01/pDXW-10-ppc和C. glutamicum G01/pDXW-10-gltA菌株生长速率和葡萄糖消耗速率均较原始菌有所提高,可能是PEPC酶活的提高增强了磷酸烯醇式丙酮酸对OAA的供应,加快了TCA循环,有利于菌体的生长,葡萄糖的消耗速率加快;而CS酶活的提高直接强化了碳流进入TCA循环,使得菌体生长和葡萄糖消耗速率提高;C. glutamicum G01/pDXW- 10-ppc菌株发酵48 h后谷氨酸产量提高至(108.31±1.72) g/L,较原始菌提高了15.8%,可能是突变位点和RBS优化使得PEPC酶活得到一定程度提高,有利于改善谷氨酸产量;C. glutamicum G01/pDXW-10-gltA菌株生长较快但谷氨酸产量并未得到较大程度的改善,谷氨酸产量仅达到(90.23±3.64) g/L,即仅提高CS活性并不能增强谷氨酸合成能力[29-30]。C. glutamicum DL01菌株菌体生长速率较原始菌慢,但最终谷氨酸产量高达(123.86±4.75) g/L,较原始菌提高了32.4%;糖酸转化率为56.1%,较原始菌提高了10.9%。可见,lpd基因的弱化表达减弱了α-酮戊二酸下游代谢通量,影响了菌体的生长,降低了耗糖速率,提高了糖酸转化率,同时使得更多的α-酮戊二酸用于谷氨酸合成,谷氨酸产量得到改善。C. glutamicum G01/pDXW-10-icd菌株生长速率和葡萄糖消耗速率与原始菌相近,但谷氨酸产量并无明显提高,且从转录组数据来分析,虽然icd基因在对照菌株中的表达量明显高于谷氨酸高产菌C. glutamicum G01,但是对照菌株谷氨酸产量却很低,这也进一步说明了ICD的过表达可能对谷氨酸合成影响不大;这与之前的研究是相符的,之前Shimizu等在谷氨酸棒杆菌中过表达了ICD,提高了ICD酶活力,但是对谷氨酸生产的α-KG节点处的代谢流分配没有很大影响,谷氨酸产量也并无明显提高[31-32]。
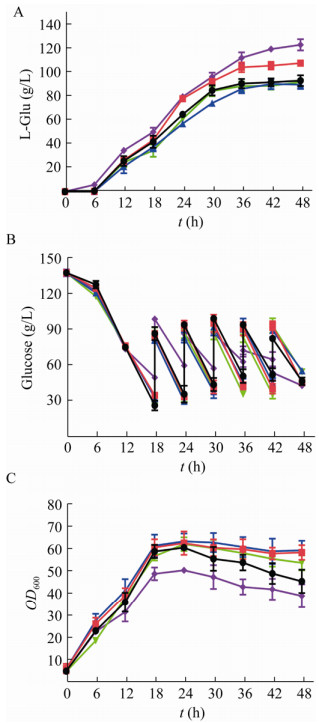
|
| 图 8 重组谷氨酸棒杆菌的补料分批发酵 Fig. 8 The fed-batch fermentation of recombinant strains. (A) L-glutamate concentration. (B) Sugar consumption rate. (C) Cell growth rate. C. glutamicum G01 (black lines), C. glutamicum G01/pDXW-10-ppc (red lines), C. glutamicum G01/pDXW-10-gltA (blue lines), C. glutamicum G01/pDXW-10-icd (green lines) and C. glutamicum DL01 (purple lines). |
| |
此外,为进一步确定gltA和icd基因对谷氨酸生产的影响,将gltA和icd基因在对照菌株C. glutamicum E01中也进行了过表达,重组菌株发酵数据表明,其谷氨酸产量与C. glutamicum E01菌株相近,没有明显提高(数据未显示),即gltA和icd基因的过表达对谷氨酸生产基本没有影响。
2.5 谷氨酸高产菌株的构建 2.5.1 ppc基因在C. glutamicum DL01中的过表达将构建好的pDXW-10-ppc重组质粒电转化至C. glutamicum DL01中,PCR鉴定结果如图 9A所示,泳道中条带大小为2 759 bp,C. glutamicum DL01/pDXW-10-ppc菌株构建成功;在控制菌体浓度一致的条件下,C. glutamicum DL01/pDXW- 10-ppc菌株中PEPC的表达量均明显高于对照菌株C. glutamicum DL01,其大小约为103.2 kDa,即PEPC在C. glutamicum DL01/pDXW-10-ppc中成功表达(图 9B)。
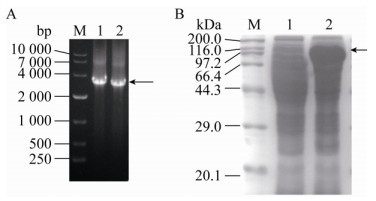
|
| 图 9 C. glutamicum DL01/pDXW-10-ppc菌株的构建结果(A)与表达情况(B) Fig. 9 Construction results (A) and expressions (B) of C. glutamicum DL01/pDXW-10-ppc strain. (A) M: 10 000 bp nucleic acid marker; 1 and 2: PCR results of ppc gene. (B) M: protein marker; 1: crude enzyme solution of C. glutamicum DL01 control strain; 2: crude enzyme solution of C. glutamicum DL01/pDXW-10-ppc strain. |
| |
测定C. glutamicum DL01/pDXW-10-ppc菌株中PEPC和LPD的酶活,结果表明该菌株中PEPC酶活提高至(162.64±5.18) U/mL,较原始菌的(38.52±2.87) U/mL提高了3.2倍,即ppc基因在C. glutamicum DL01中的过表达,提高了C. glutamicum DL01/pDXW-10-ppc中PEPC的酶活力。LPD酶活下降为58.7 U/mL,较原始菌的72.3 U/mL下降了18.8%,可能是CRISPR/dCas9系统成功实现了lpd基因在C. glutamicum DL01/pDXW-10-ppc中的弱化,因此,LPD酶活呈现一定程度的下降。
2.5.3 发酵罐发酵测试谷氨酸高产菌株谷氨酸水平将C. glutamicum DL01/pDXW-10-ppc菌株进行5 L罐发酵,在整个发酵过程中该菌株的生长速率比原始菌略有降低,可能是lpd基因的弱化使得α-酮戊二酸下游代谢流减弱引起的。C. glutamicum DL01/pDXW-10-ppc菌株发酵48 h后,其谷氨酸产量为(136.09±5.53) g/L,较原始菌提高了45.5%,正是PEPC酶活的提高,使得谷氨酸产量在C. glutamicum DL01菌株的基础上再提高13.1%;此外,该菌株的糖酸转化率为58.9%,较原始菌提高了13.7%。本文构建的整合菌株,谷氨酸产量由93.53 g/L提高至(136.09±5.53) g/L,且糖酸转化率提高了13.7% (图 10),这一策略将为谷氨酸生产菌株的代谢改造提供理论指导,以进一步获得高产量、高糖酸转化率的可工业化谷氨酸生产菌株,同时将会用作高值氨基酸细胞工厂实现谷氨酸高值化产品的高产出。
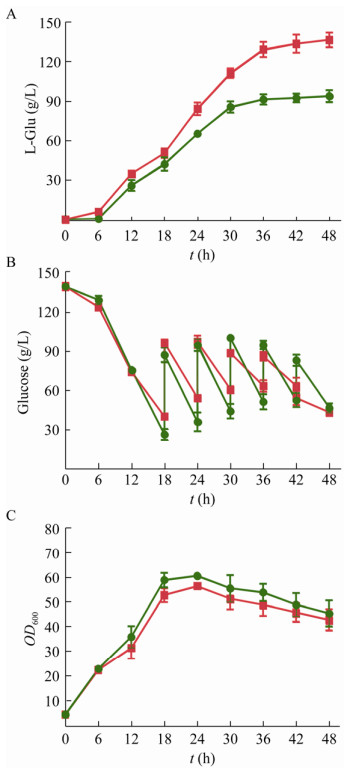
|
| 图 10 重组菌株C. glutamicum DL01/pDXW-10-ppc菌株的补料分批发酵 Fig. 10 The fed-batch fermentation of recombinant strain C. glutamicum DL01/pDXW-10-ppc. (A) L-glutamate concentration. (B) Sugar consumption rate. (C) Cell growth rate. C. glutamicum G01 (green lines), C. glutamicum DL01/pDXW-10-ppc (red lines). |
| |
实验室前期通过逐级诱变筛选得到了一株谷氨酸高产菌株C. glutamicum G01,将其与谷氨酸生产菌株C. glutamicum E01进行全局RNA-seq分析和重测序分析,挑选出中心代谢路径转录水平和基因水平上存在差异的基因进行研究,以进一步改善谷氨酸的产量。由于OAA节点和α-酮戊二酸节点在谷氨酸合成途径中扮演重要角色,因此,探究了增强OAA供应的磷酸烯醇式丙酮酸羧化酶、OAA节点处的柠檬酸合酶、α-酮戊二酸节点处的异柠檬酸脱氢酶和ODHC复合体E3亚基对谷氨酸生产的扰动影响。基于以上研究构建的整合菌株,谷氨酸产量提高了45.5%,糖酸转化率提高了13.7%,在一定程度上降低了工业生产成本,为谷氨酸高产菌株的基因工程改造提供了理论依据,为谷氨酸高值化产品的生产奠定了基础。
鲍杰等通过RBS序列弱化odhA表达,使得谷氨酸产量提高16.8%;同时研究了中心代谢路径中pyc、ppc、gltA和icd基因对谷氨酸生产的影响,结果显示谷氨酸产量均未得到改善[11]。目前,很多研究者通过诱变手段虽然获得了谷氨酸高产菌株,但并未进一步去阐述高产机理,如白长胜等通过对获得的谷氨酸高产菌株进行发酵培养基优化,谷氨酸产量由120.3 g/L提高至135.6 g/L,培养基优化水平上谷氨酸产量未得到明显提高[33];陈宁等通过控制谷氨酸温度敏感突变株发酵过程中的温度转换,完成了从谷氨酸非积累型细胞向谷氨酸积累型细胞的转换,实现了谷氨酸的高产,其产量高达181 g/L,为目前已报道最高产量[34]。本文期望能够通过转录组与重测序分析结果,挖掘关键基因以阐述谷氨酸高产菌株C. glutamicum G01高产谷氨酸的机理。但本研究仅针对中心代谢途径差异基因对谷氨酸生产的影响,还不够全面;谷氨酸的高产出是多因子协同作用的,本文未对能量代谢、谷氨酸分泌等相关差异基因入手探索对谷氨酸生产的影响。下一步将会继续挖掘其他功能基因对谷氨酸生产的影响,进一步解析谷氨酸高产机理。
| [1] |
Yang Y, Zhang MM, Gao Y, et al. Research progress on key molecular mechanism of high yield glutamic acid fermentation by Corynebacterium glutamicum. Sci Technol Food Ind, 2019, 40(5): 311-315, 321 (in Chinese). 杨阳, 张苗苗, 高越, 等. 谷氨酸棒状杆菌高效发酵谷氨酸的关键分子机理研究进展. 食品工业科技, 2019, 40(5): 311-315, 321. |
| [2] |
Xu GQ, Zha J, Cheng H, et al. Engineering Corynebacterium glutamicum for the de novo biosynthesis of tailored poly-γ-glutamic acid. Metabol Eng, 2019, 56: 39-49. |
| [3] |
Wang NN, Ni YL, Shi F. Deletion of odhA or pyc improves production of γ-aminobutyric acid and its precursor L-glutamate in recombinant Corynebacterium glutamicum. Biotechnol Lett, 2015, 37(7): 1473-1481. |
| [4] |
Kawahara Y, Takahashi-Fuke K, Shimizu E, et al. Relationship between the glutamate production and the activity of 2-Oxoglutarate dehydrogenase in Brevibacterium lactofermentum. Biosci Biotechnol Biochem, 1997, 61(7): 1109-1112. |
| [5] |
Asakura Y, Kimura E, Usuda Y, et al. Altered metabolic flux due to deletion of odhA causes L-glutamate overproduction in Corynebacterium glutamicum. Appl Environ Microbiol, 2007, 73(4): 1308-1319. |
| [6] |
Wada M, Sawada K, Ogura K, et al. Effects of phosphoenolpyruvate carboxylase desensitization on glutamic acid production in Corynebacterium glutamicum ATCC 13032. J Biosci Bioeng, 2016, 121(2): 172-177. DOI:10.1016/j.jbiosc.2015.06.008 |
| [7] |
Yao WJ, Deng XZ, Zhong H, et al. Double deletion of dtsR1 and pyc induce efficient L-glutamate overproduction in Corynebacterium glutamicum. J Ind Microbiol Biotechnol, 2009, 36(7): 911-921. |
| [8] |
Sato H, Orishimo K, Shirai T, et al. Distinct roles of two anaplerotic pathways in glutamate production induced by biotin limitation in Corynebacterium glutamicum. J Biosci Bioeng, 2008, 106(1): 51-58. DOI:10.1263/jbb.106.51 |
| [9] |
Buch AD, Archana G, Kumar GN. Enhanced citric acid biosynthesis in Pseudomonas fluorescens ATCC 13525 by overexpression of the Escherichia coli citrate synthase gene. Microbiology, 2009, 155(8): 2620-2629. |
| [10] |
Man ZW, Xu MJ, Rao ZM, et al. Systems pathway engineering of Corynebacterium crenatum for improved L-arginine production. Sci Rep, 2016, 6: 28629. DOI:10.1038/srep28629 |
| [11] |
Wen JB, Bao J. Engineering Corynebacterium glutamicum triggers glutamic acid accumulation in biotin-rich corn stover hydrolysate. Biotechnol Biofuels, 2019, 12: 86. DOI:10.1186/s13068-019-1428-5 |
| [12] |
Peng F, Wang XY, Sun Y, et al. Efficient gene editing in Corynebacterium glutamicum using the CRISPR/Cas9 system. Microb Cell Factor, 2017, 16: 201. DOI:10.1186/s12934-017-0814-6 |
| [13] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. |
| [14] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [15] |
Ho SN, Hunt HD, Horton RM, et al. Site-directed mutagenesis by overlap extension using the polymerase chain reaction. Gene, 1989, 77(1): 51-59. |
| [16] |
Hasegawa T, Hashimoto KI, Kawasaki H, et al. Changes in enzyme activities at the pyruvate node in glutamate-overproducing Corynebacterium glutamicum. J Biosci Bioeng, 2008, 105(1): 12-19. DOI:10.1263/jbb.105.12 |
| [17] |
Chen RD, Yang H. A highly specific monomeric isocitrate dehydrogenase from Corynebacterium glutamicum. Arch Biochem Biophys, 2000, 383(2): 238-245. |
| [18] |
Wu AM, Jiang ZQ, Wei Y, et al. Purification of an extracellular dihydrolipoamide dehydrogenase from Bacillus subtilis. J China Agric Univ, 2008, 13(1): 16-19 (in Chinese). 武爱民, 江正强, 韦赟, 等. 枯草芽孢杆菌胞外二氢硫辛酰胺脱氢酶的纯化. 中国农业大学学报, 2008, 13(1): 16-19. |
| [19] |
Guest JR, Creaghan IT. Further studies with lipoamide dehydrogenase mutants of Escherichia coli k12. J Gen Microbiol, 1974, 81(1): 237-245. |
| [20] |
Zhang HL, Li YY, Wang XY. Metabolic engineering of L-valine synthesis and secretory pathways in Corynebacterium glutamicum for higher production. Chin J Biotech, 2018, 34(10): 1606-1619 (in Chinese). 张海灵, 李颜颜, 王小元. 代谢工程改造谷氨酸棒状杆菌合成及分泌途径生产L-缬氨酸. 生物工程学报, 2018, 34(10): 1606-1619. |
| [21] |
Xue N, Li ZX, Zhan JJ, et al. Effect of aceA and gogat knockout combined gltA overexpression in GKGD on α-ketoglutarate production. Food Fermentat Ind, 2017, 43(8): 1-7 (in Chinese). 薛宁, 李智祥, 战俊杰, 等. 敲除aceA和gogat及过表达gltA对谷氨酸棒状杆菌GKGD合成α-甜酮戊二酸的影响. 食品与发酵工业, 2017, 43(8): 1-7. |
| [22] |
Zhang B, Zhou N, Liu YM, et al. Ribosome binding site libraries and pathway modules for shikimic acid synthesis with Corynebacterium glutamicum. Microb Cell Factor, 2015, 14: 71. DOI:10.1186/s12934-015-0254-0 |
| [23] |
Zhang B, Yu M, Zhou Y, et al. Systematic pathway engineering of Corynebacterium glutamicum S9114 for l-ornithine production. Microb Cell Factor, 2017, 16: 158. DOI:10.1186/s12934-017-0776-8 |
| [24] |
Nguyen AQD, Schneider J, Reddy GK, et al. Fermentative production of the diamine putrescine: system metabolic engineering of Corynebacterium Glutamicum. Metabolites, 2015, 5(2): 211-231. |
| [25] |
Kim J, Hirasawa T, Sato Y, et al. Effect of odhA overexpression and odhA antisense RNA expression on Tween-40-triggered glutamate production by Corynebacterium glutamicum. Appl Microbiol Biotechnol, 2009, 81(6): 1097-1106. |
| [26] |
Kim J, Hirasawa T, Saito M, et al. Investigation of phosphorylation status of OdhI protein during penicillin- and Tween 40-triggered glutamate overproduction by Corynebacterium glutamicum. Appl Microbiol Biotechnol, 2011, 91(1): 143-151. |
| [27] |
Komine-Abe A, Nagano-Shoji M, Kubo S, et al. Effect of lysine succinylation on the regulation of 2-oxoglutarate dehydrogenase inhibitor, OdhI, involved in glutamate production in Corynebacterium glutamicum. Biosci Biotechnol Biochem, 2017, 81(11): 2130-2138. |
| [28] |
Schultz C, Niebisch A, Gebel L, et al. Glutamate production by Corynebacterium glutamicum: dependence on the oxoglutarate dehydrogenase inhibitor protein OdhI and protein kinase PknG. Appl Microbiol Biotechnol, 76(3): 691-700.
|
| [29] |
Wiegand G, Remington SJ. Citrate synthase: structure, control, and mechanism. Ann Rev Biophys Biophys Chem, 1986, 15(1): 97-117. |
| [30] |
Eikmanns BJ, Thum-Schmitz N, Eggeling L, et al. Nucleotide sequence, expression and transcriptional analysis of the Corynebacterium glutamicum gltA gene encoding citrate synthase. Microbiology, 1994, 140(8): 1817-1828. |
| [31] |
Shimizu H, Tanaka H, Nakato A, et al. Effects of the changes in enzyme activities on metabolic flux redistribution around the 2-oxoglutarate branch in glutamate production by Corynebacterium glutamicum. Bioproc Biosyst Eng, 2003, 25(5): 291-298. |
| [32] |
Eikmanns BJ, Rittmann D, Sahm H. Cloning, sequence analysis, expression, and inactivation of the Corynebacterium glutamicum icd gene encoding isocitrate dehydrogenase and biochemical characterization of the enzyme. J Bacteriol, 1995, 177(3): 774-782. |
| [33] |
Bai CS, Han J, Wang G, et al. Optimization of fermentation medium for L-glutamic production by suboptimai dose. China Condim, 2016, 41(8): 87-90 (in Chinese). 白长胜, 韩隽, 王刚, 等. 亚适量法L-谷氨酸发酵培养基的优化. 中国调味品, 2016, 41(8): 87-90. |
| [34] |
Chen N, Xu QY, Xie XX, et al. Method for improving fermentation acid yield of glutamic acid temperature-sensitive mutant: CN, 101705263A. 2010-05-12 (in Chinese). 陈宁, 徐庆阳, 谢希贤, 等.一种提高谷氨酸温度敏感突变株发酵产酸率的方法: 中国, 101705263A. 2010-05-12. |
 2020, Vol. 36
2020, Vol. 36




