中国科学院微生物研究所、中国微生物学会主办
文章信息
- 胡若琳, 袁超, 牛义, 汤青林, 魏大勇, 王志敏
- Hu Ruolin, Yuan Chao, Niu Yi, Tang Qinglin, Wei Dayong, Wang Zhimin
- 植物MYB转录因子在花药发育中的调控作用
- Regulation of plant MYB transcription factors in anther development
- 生物工程学报, 2020, 36(11): 2277-2286
- Chinese Journal of Biotechnology, 2020, 36(11): 2277-2286
- 10.13345/j.cjb.200125
-
文章历史
- Received: March 16, 2020
- Accepted: May 18, 2020
- Published: August 21, 2020
2. 重庆市蔬菜学重点实验室,重庆 400715
2. Chongqing Key Laboratory of Olericulture, Chongqing 400715, China
在植物的生长发育过程中,细胞内基因的表达具有时间特异性和空间特异性,导致各细胞间出现分化、产生差异,造成这种现象的主要原因之一是转录因子(Transcription factor,TF)在转录水平上的调节作用[1]。花作为开花植物的生殖器官,通过传粉、受精作用产生下一代。花主要由花萼、花瓣、雄蕊和雌蕊4部分组成,其中,花药作为雄蕊最重要的组成部分,其发育过程复杂且精细,与转录因子的调控息息相关。MYB转录因子家族是植物中最大的转录因子家族之一,参与植物多种生命活动的调控[2],特别是对花药发育的调控。本文就近年来关于MYB转录因子调控植物花药发育的相关研究进行回顾和总结,从而为深入研究转录因子在植物花药发育中的分子调控机制提供一些参考。
1 MYB转录因子的发现科学家们最初在禽急性成髓细胞白血病病毒中发现MYB基因,1982年Klempnauer等又克隆了禽髓母细胞瘤病毒的原癌基因v- myb和其细胞前体c-myb,分析发现c-myb转化为v-myb可能是由于DNA重排和随后使用剪接RNA作为中间产物所致[3]。此后,在动物、植物和真菌中陆续发现大量MYB基因的存在,并且与动物和酵母相比,植物中的MYB转录因子在结构和功能上的保守性更高[4]。Paz-Ares等在野生型玉米中发现的ZmMYBC1基因是第一个植物MYB类基因,该基因参与了色素的合成[5]。随着分子生物技术的发展,在模式植物拟南芥Arabidopsis thaliana中已鉴定出200多个MYB基因[6],同时,在水稻Oryza sativa[4]、葡萄Vitis vinifera[8]、棉花Gossypium spp[9]和谷子[7]等植物中也发现存在大量MYB基因,并且大多数MYB基因的功能已逐渐被挖掘鉴定。许多研究表明,MYB类转录因子参与调控植物细胞分化、器官形成、叶片形态建成、初生和次生代谢、生物和非生物胁迫反应等,在植物的生长发育过程中发挥着重要的作用[10-12]。
2 MYB转录因子结构与分类植物中结构上含有MYB结构域的转录因子被称为MYB类转录因子。MYB结构域的N端是高度保守的DNA结合结构域,该结构域的基本单位是1–3个不完全重复R结构(R1、R2和R3),R结构大约由52个氨基酸残基组成。每个重复形成3个α螺旋,第2和第3螺旋构成一个规则间隔的螺旋-旋转-螺旋(Helix-turn-helix,HTH)结构基序,其中HTH由3个保守色氨酸残基(W)组成并形成一个疏水中心,每个色氨酸残基间隔18–19个氨基酸[13-14]。根据每个MYB蛋白中含有的R结构个数和重复种类的不同,MYB蛋白可分为4R-MYB、2R-MYB (R2R3-MYB)、3R-MYB (R1R2R3-MYB)和1R-MYB (MYB-related)这4类相关蛋白[15]。其中,最小类的4R-MYB蛋白中的每个基因包含4个R1/R2重复序列,且只在极少数植物基因组中进行编码[16],目前仅在模式植物拟南芥[17]、葡萄[8]、杨树[18]中对4R-MYB蛋白进行了相关研究,但是对这些蛋白在植物中的作用仍知之甚少。在大多数真核生物基因组中发现存在3个连续重复序列的3R-MYB (R1R2R3- MYB),可能是从R2R3-MYB基因获得R1重复序列进化而来。研究发现,植物中的蛋白成员3R-MYB与动物、真菌中的3R-MYB蛋白高度同源,它们调节细胞周期和细胞的分化,与植物的抗逆性有着紧密的联系[19]。MYB家族的1R-MYB包含1个或部分MYB重复[20-21],调控植物生物钟和端粒DNA结合蛋白[22],有些1R-MYB蛋白还具有维持染色体结构完整性和调节基因转录的作用[23]。在这4类MYB蛋白中,发现2R-MYB (R2R3-MYB)在植物中数目最多,推测它可能是从3R-MYB (R1R2R3-MYB)基因中丢失R1后进化而来,也有研究者认为它可能是从获得R1重复的1R-MYB基因进化而来的[24-25]。在R2R3-MYB蛋白的结构中存在高度保守的MYB结构域,它们的C端具有高度的可变性,负责决定蛋白质之间的相互作用[8],参与细胞发育与形态建成、调节代谢、植物应激反应等方面。
3 MYB转录因子对花药发育的调控植物花药发育过程主要分为两个阶段,第一阶段由最初的雄蕊原基开始,经组织特化、减数分裂后,形成完整的花粉囊壁,从外到内依次为表皮层、纤维层、中层、绒毡层,且药室中心聚集着小孢子的四分体结构;第二阶段花药继续膨大,花丝伸长,小孢子由四分体结构发育成花粉粒,花粉囊开裂释放花粉,完成授粉受精过程,最后花药组织逐渐衰老退化[26]。
Sanders等对模式植物拟南芥花药的形态发育进行了研究[27]。根据花药组织细胞切片观察,将拟南芥花药发育过程分为14个时期,其中在1–5期,由雄蕊原基经细胞分裂先形成表皮层和孢原细胞等各层组织,孢原细胞再进一步分裂形成内外两层细胞,内层为造孢细胞,外层逐渐发育形成药室内壁、中间层、绒毡层。在第10期,绒毡层开始降解,为花粉发育和花粉外壁形成提供营养。13–14期裂口细胞破裂,花药仅含2个药室,成熟花粉由此释放,花药逐渐衰老并脱落。
3.1 调控绒毡层发育绒毡层是维管植物孢子囊最内侧的细胞层,与小孢子的释放和花粉壁的形成有关。在花药发育过程中,绒毡层为花粉发育提供养分,并合成和分泌与花粉形成直接有关的酶——胼胝质酶。因此,若绒毡层发育异常,则会造成花粉败育,最终导致不育。研究表明,拟南芥AtMYB33和AtMYB65基因在花药发育中起功能冗余作用,调节绒毡层的发育,atmyb33和atmyb65双突变体绒毡层细胞在减数分裂前产生过多的空泡化、肿胀和肥大,导致花粉败育[28]。拟南芥AtMYB80转录因子参与绒毡层和花粉的发育,AtMYB80的表达受到抑制,导致完全雄性不育且绒毡层程序性细胞死亡(Programmed cell death,PCD)提前[29]。棉花上发现GhMYB80基因在绒毡层降解开始前表达停止,并且棉花MYB80蛋白能完全恢复atmyb80突变体的育性,此外,还发现GhMYB80和甘蓝型油菜BnMYB80与一个类似EAR阻遏基序(LDLNLELRISPP)融合后可诱导拟南芥雄性不育[30]。在花药发育过程中,多种TFs参与绒毡层的发育,包括DYSFUNCTIONAL TAPETUM1 (DYT1)[31]、DEFECTIVE INTAPETAL DEVELOPMENT AND FUNCTION1 (TDF1)[32]、ABORTED MICROSPORES (AMS)[33-34]和MALE STERILITY1 (MS1)[35]等。其中,DYT1和AMS编码bHLH转录因子,TDF1和MS188(也称AtMYB103/MYB80)编码R2R3 MYB型转录因子,MS1编码一个PHD(Plant homeodomain)转录因子。研究表明遗传途径DYT1-TDF1-AMS- MS188-MS1严格控制绒毡层发育[32]。DYT1直接调控TDF1的表达从而影响绒毡层的发育和花粉壁的形成[36]。而在tdf1突变体中,绒毡层细胞严重空泡化,并伴有一个额外的分裂,这些细胞不能过渡到分泌的绒毡层,从而阻碍了四分体小孢子的释放[32]。研究还发现,水稻中的OsTDF1与拟南芥中的AtTDF1有着相似的作用,即突变体中间层的降解受到阻碍[37]。2019年,Zhang等为了确定乌菜ogu-CMS花粉的败育阶段,找到与绒毡层发育相关的重要转录因子,通过形态学比较和石蜡切片观察发现乌菜ogu-CMS雄蕊中的花药败育,减数分裂后绒毡层出现异常空泡,导致小孢子败育;BrTDF1在花序组织中的表达量比其他组织中高;与可育系相比,BrTDF1在不育系芽的各个发育阶段均表现出较低的表达。此外,对TDF1的直接靶基因A6和EXPB5的检测表明,它们在ogu不育系中的表达低于其保持系;BrTDF1在拟南芥tdf1突变体转基因株系中的表达可恢复其育性,因此认为乌菜绒毡层的发育需要MYB转录因子BrTDF1[38]。关于绒毡层的遗传途径虽已被研究报道,但途径中各类型转录因子的靶基因以及各靶基因间是否会相互调控进而影响绒毡层发育仍需深入研究。
3.2 调控花药开裂花药作为雄蕊的重要组成部分,是花粉发育的场所。成熟花粉的及时释放依赖于植物花药的开裂,以完成授粉受精过程。花药药室内壁的次生加厚、花药壁组织的脱水和酶解等过程都会影响花药的开裂,其中药室内壁的次生加厚为花药开裂提供一种机械力量,对花药开裂至关重要[39]。NAC结构域家族中的NST1和NST2可促进花药的次生增厚,即花药内皮层的选择性扩张。研究发现,AtMYB26蛋白存在于花药内,受生长素应答因子ARF8.4直接调控,MYB26位于NST1、NST2的上游,直接诱导NST1和NST2的表达使次生增厚在花药内形成,从而确保了次生增厚位置的特异性,推测AtMYB26蛋白是促进花药开裂的关键[40-41]。对RUS4基因沉默拟南芥突变体(RUS4-amiRNA)株系的花药不同发育阶段进行细胞学观察,通过qRT-PCR对一些与花药药室内壁次生加厚相关的基因进行分析,发现RUS4基因可能通过间接激活转录因子MYB103和MYB85的表达影响药室内壁次生壁的合成过程[42]。在花药开裂的过程中,MYB转录因子不仅仅只调控药室内壁的次生加厚,它如何通过其他代谢途径或与激素互作共同调控花药开裂过程也是一个重要的研究方向。
3.3 调控花粉发育植物雄性配子体,即花粉粒,产生于被子植物的花药内。在花粉形成过程中,小孢子母细胞的分化、绒毡层与胼胝质的正常发育、花粉壁的形成、孢粉素的合成与沉积以及同化物的分配等各个步骤都会受到MYB转录因子的调节。因此,MYB转录因子超量、异常表达或基因沉默,都会使花药的正常发育受到影响,形成无生殖能力的花粉,导致花粉败育。
近年来,花粉壁发育的转录调控已被广泛研究,特别是在拟南芥和水稻的雄性不育突变体的研究中[43]。目前已鉴定出一系列转录因子,包括DYT1、AMS、MS1以及R2R3-MYB型转录因子MYB35[44]和MYB80/MS188[32]等,这些基因的敲除突变体在绒毡层或花粉壁发育中表现出畸变。Zhu等[32]通过突变植株的RNA原位杂交,建立了拟南芥花粉壁发育调控的遗传途径,即bHLH蛋白DYTI位于MYB35的上游,其次是AMS、MS188,最后是MS1。在水稻中已部分证实了相似的遗传途径[43]。拟南芥MYB99蛋白作为矮牵牛Petunia hybrida ODORANT1 (ODO1)的一种密切同源蛋白,存在于拟南芥绒毡层细胞中,其表达受MS1转录因子调控,影响花粉壁的形成[45]。除影响花粉壁的形成外,研究还发现,GhMYB24转录本在花药中表达量较高,且主要在花粉中表达,它在拟南芥中的过表达会导致花药不开裂和花粉粒减少,造成雄性不育。另外,经酵母双杂交试验表明GhMYB24与细胞内GhJAZ1/2相互作用影响茉莉酸(Jasmonic acid,JA)途径进而影响雄蕊发育[46]。拟南芥的AtMYB103受NST1、NST2的直接调控,通过调控花药次生壁的合成,调节绒毡层的发育,影响花粉发育,其异常表达还导致了雄性不育[47]。拟南芥AtMYB21突变体或AtMYB24超表达株系表现出较短的花药丝,延迟或无花药开裂,以及较少的活花粉粒[48]。从油菜中分离得到的编码R2R3-MYB转录激活因子的油菜Brassica campestris Male Fertility 28 (BcMF28)在拟南芥雄蕊中特异表达,它的超量表达引起了雄蕊发育缺陷,包括花丝缩短、花药不开裂和花粉败育,最终导致雄性不育[49]。MYB转录因子在花粉发育中主要通过其下游靶基因进行调控,但多数靶基因还未完全确定,且与靶基因间的具体调控关系也需进一步探索。
3.4 调控糖类物质参与花药发育作为光自养生物,植物光合作用产生的碳水化合物通过韧皮部转运到植物的各个部分,为其生长发育提供碳和能量[50]。蔗糖是碳水化合物的主要形式,不仅为植物的生长发育提供能量和碳骨架,而且还是调节植物生长发育过程的信号分子[51]。蔗糖对拟南芥花粉萌发[52]和烟草Nicotiana tabacum花粉管生长至关重要[53]。一种R2R3- MYB转录因子CSA (Carbon starved anther)通过上调MST8、SUT3、UGP2等糖分配基因的表达,可促进蔗糖向花药的转运,从而保证花粉正常发育[54]。油菜素类固醇(Brassinosteroids,BRs)在调控水稻花粉粒和种子形成中起关键作用,OsBZR1可直接促进CSA的表达,使与糖分裂、吸收和淀粉合成的相关基因进行表达,促进水稻花粉的形成[55]。糖醇作为许多植物光合作用和碳水化合物运输的最终产物,会影响植物的雄蕊发育。山梨醇是苹果雄蕊发育和花粉管生长的必需物质,Dong等发现MdMYB39L通过调控其下游靶基因MdHT、MdPMEI、MdPLIM和MdLRX,参与己糖的吸收、细胞壁的形成和修饰,以及小孢子的发生,影响雄蕊和花粉管发育,表明MdMYB39L在山梨醇调节苹果雄蕊发育和花粉管生长中发挥了重要作用[56]。然而,关于糖类物质的研究多集中于植物生长发育的其他方面,在花器官分配机制和有关MYB转录因子的调控网络机制研究上还较少。
3.5 参与激素途径调控花药发育 3.5.1 茉莉酸途径茉莉酸(JA)是一种脂肪酸衍生物,在植物生长发育中起重要作用。作为雄蕊和花粉发育成熟的一个关键信号,拟南芥中的F-box蛋白CORONATINE INSENSITIVE1 (COI1)受JA信号诱导,通过SCFCOI1-26S蛋白酶体途径降解JAZIM结构域(JAZ)蛋白,激活MYB转录因子(如MYB21和MYB24)来调节雄蕊的发育,最终激活花药相关基因的表达[57]。R2R3-MYB转录因子MYB21和MYB24与Ⅲe bHLH因子(MYC2、MYC3、MYC4和MYC5)形成MYB-MYC配合物,并与JAZs相互作用介导雄蕊发育的后期[58]。MYB21和MYB24通过它们的N端R2R3结构域与大多数JAZs相互作用,并且MYB24的N端和C端介导MYB21和MYB24的二聚体相互作用;适当水平的MYB24过量表达可以挽救JA缺陷突变体Opr3的雄性不育,但过高水平的MYB24却无法挽救,MYB24 N-末端的过量表达,又会导致野生型拟南芥雄性不育[59]。在番茄中SIMYB21对JA生物合成有正调控作用,而对生长素和赤霉素有负调控作用,JA会促进番茄中SlMYB21的表达,对番茄的开花、花粉成熟等有一定的影响[60]。在之前的研究中,尽管JA在诱导AtMYB21/24或SlMYB21基因表达中的作用在拟南芥和番茄中很常见,但这两种植物的MYB基因功能却存在差异,因此,在进一步发掘研究MYB基因功能时,不同植物间差异需要具体分析。此外,转录因子MYB108与MYB24也一起协调JA途径参与花药开裂过程[61]。Sun等鉴定了一种JA诱导的R2R3-MYB转录因子CaMYB108,参与辣椒素(Capsaicinoids,Caps)生物合成和雄蕊发育的调控,沉默CaMYB108后植株出现花药延迟开裂、花粉活力降低的现象[62]。
3.5.2 生长素途径除JA外,生长素在花药开裂中也起着重要作用[63],生长素应答因子ARF17对花药发育和花粉形成具有重要意义。在绒毡层中,ARF17的表达受到microRNA160的严格控制,ARF17过表达导致绒毡层发育缺陷并产生雄性不育型缺陷[64]。拟南芥arf17突变体表现花药不开裂,MYB108与NST1在arf17中表达下调,通过染色质免疫沉淀和电泳迁移率转移实验表明ARF17直接与MYB108启动子结合并调节其表达,ARF17启动子成功驱动MYB108的表达可修复arf17的花药开裂缺陷,说明ARF17直接调节MYB108在花药开裂中的表达[65]。
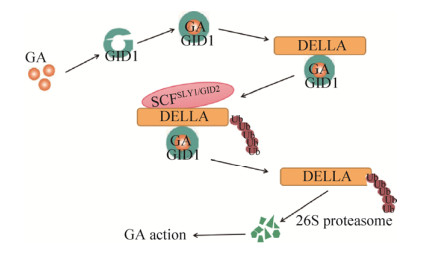
3.5.3 赤霉素途径赤霉素(GA)通过GA信号途径影响植物的开花诱导和花药发育等过程,特别在花粉的形成和花粉管伸长过程中起重要作用。在GA信号途径中,GA首先结合GA INSENSITIVE DWARF1 (GID1)受体,促进DELLA蛋白与GA-GID1复合体的结合,形成GA-GID1-DELLA三聚体,减弱DELLA蛋白的抑制作用并促进DELLA同泛素连接酶复合体(SCFSLY1/GID2)结合,随后使DELLA蛋白通过26S蛋白酶快速降解,从而释放DELLA蛋白的抑制作用,促进GA发生作用[66] (图 1)。GA可通过JA调控MYB21、MYB24、MYB57的表达,从而调节雄蕊晚期发育。在水稻GA信号转导中,GA4直接与GID1结合形成GA4-GID1复合体,该复合体与DELLA蛋白SLENDER RICE1 (SLR1)相互作用并通过蛋白酶使其降解,而SLR1可通过负调控GA应答基因如GAMYB的表达控制GA信号转导,GAMYB的下游基因CYP703A3和KAR参与花粉形成,可见激活GA信号可能导致花粉败育[67]。在拟南芥和水稻等植物中,GA可以通过GAMYB转录因子调节雄蕊和花药的发育[68]。黄瓜的同源基因CsGAMYB1蛋白定位于细胞核,主要在雄性花蕾中表达;其异位表达可部分挽救拟南芥myb33-myb65双突变体的雄蕊发育和育性表型。然而,野生型拟南芥CsGAMYB1的过表达却导致了雄性不育,且黄瓜CsGAMYB1基因敲除可以导致雌雄花比例的降低,调节黄瓜的性别表达[69]。这一研究结果提高了对黄瓜性别决定的认识,为花诱导分子机制和高产栽培提供了依据,对研究其他两性植物的性别表达具有重要参考价值。目前,JA、IAA以及GA等生长物质在花药开裂、花粉发育等过程中的调控机制为揭示植物花药发育机理奠定了一定的基础,但关于各激素调控花药发育的大量基因以及各激素间的相互作用机制都还有待进一步研究。
植物的花药发育过程精准、复杂,是花药发育相关基因选择性表达以及多种因素共同调节的结果。MYB转录因子家族庞大且功能多样,在所有真核生物中都有表达,并参与植物生长发育、代谢过程、各种生物和非生物胁迫的反应以及激素合成和信号转导等方面。随着分子生物学和实验技术的发展,MYB转录因子如何调控植物花药发育的机制被逐渐发掘。在花药发育中,MYB转录因子可与DYT1、TDF1、AMS、MS1转录因子相互作用,共同调节绒毡层发育,调控花粉壁的形成,进而影响花药发育;MYB26与NST1、NST2相互作用,使花药内形成次生增厚,成为花药开裂的关键;此外,MYB转录因子还可通过茉莉酸、生长素、赤霉素等激素途径影响花药发育。这些过程都受到众多基因的调控,彼此间既相对独立又紧密联系。针对目前已经鉴定的影响花药发育的基因以及对这些基因功能的不断研究,可以得出MYB转录因子在植物花药发育中的调控作用途径,如图 2所示。
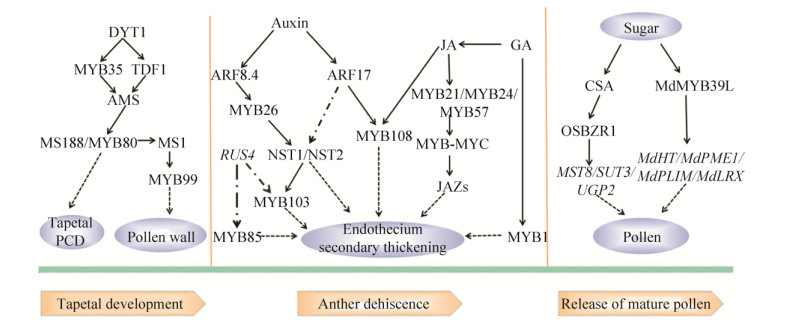
|
图 2 MYB转录因子在花药发育中的调控作用示意图(“      |
| |
虽然目前已通过突变体的表型分析、基因定位、表达时期以及生理指标测定等对MYB转录因子在植物花药发育中的作用已开展了一些研究,但植物花药发育过程中仍有很多调控机理尚未揭示,如MYB转录因子在不同属种植物中的作用机制是否一致,在花药的不同结构、不同的发育时期的调控作用是否相同。在MYB转录因子功能的开发应用上,是否可以将其在植物育性上的调控作用应用到育种工作中,以降低育种工作的难度,加快育种进程。这些问题都还有待进一步大量的研究和探索。基于转录组学和基因编辑技术的发展,今后的研究方向应该从分子或细胞水平上揭示MYB转录因子对花药发育各个时期不同部位的调控机理,这不仅为植物花药发育调控网络的研究提供更多的参考,也可为MYB转录因子在参与植物其他生命活动中的研究以及应用奠定基础。
| [1] |
Veenstra GJC, Wolffe AP. Gene-selective developmental roles of general transcription factors. Trends Biochem Sci, 2001, 26(11): 665-671. DOI:10.1016/S0968-0004(01)01970-3 |
| [2] |
Li CN, Ng CKY, Fan LM. MYB transcription factors, active players in abiotic stress signaling. Environ Exp Bot, 2015, 114: 80-91. DOI:10.1016/j.envexpbot.2014.06.014 |
| [3] |
Klempnauer KH, Gonda TJ, Bishop JM. Nucleotide sequence of the retroviral leukemia gene v-myb and its cellular progenitor c-myb: the architecture of a transduced oncogene. Cell, 1982, 31(2): 453-463. DOI:10.1016/0092-8674(82)90138-6 |
| [4] |
Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in Arabidopsis. Trends Plant Sci, 2010, 15(10): 573-579. DOI:10.1016/j.tplants.2010.06.005 |
| [5] |
Paz-Ares J, Ghosal D, Wienand U, et al. The regulatory c1 locus of Zea mays encodes a protein with homology to myb proto-oncogene products and with structural similarities to transcriptional activators. EMBO J, 1987, 6(12): 3553-3558. DOI:10.1002/j.1460-2075.1987.tb02684.x |
| [6] |
Bai H, Song ZJ, Wang YF, et al. Identification and expression analysis of MYB transcription factors related to rust resistance in Foxtail Millet. Sci Agric Sin, 2019, 52(22): 4016-4027 (in Chinese). 白辉, 宋振君, 王永芳, 等. 谷子抗锈病反应相关MYB转录因子的鉴定与表达. 中国农业科学, 2019, 52(22): 4016-4027. |
| [7] |
Muthamilarasan M, Khandelwal R, Yadav CB, et al. Identification and molecular characterization of MYB transcription factor superfamily in C4 model plant foxtail millet (Setaria italica L.). PLoS ONE, 2014, 9(10): e109920. DOI:10.1371/journal.pone.0109920 |
| [8] |
Matus JT, Aquea F, Arce-Johnson P. Analysis of the grape MYB R2R3 subfamily reveals expanded wine quality-related clades and conserved gene structure organization across Vitis and Arabidopsis genomes. BMC Plant Biol, 2008, 8: 83. DOI:10.1186/1471-2229-8-83 |
| [9] |
Suo JF, Liang XE, Pu L, et al. Identification of GhMYB109 encoding a R2R3 MYB transcription factor that expressed specifically in fiber initials and elongating fibers of cotton (Gossypium hirsutum L.). Biochim Biophys Acta -Gene Struct Express, 2003, 1630(1): 25-34. DOI:10.1016/j.bbaexp.2003.08.009 |
| [10] |
Guan SY, Jiao P, Jiang ZZ, et al. Research progress of MYB transcription factors in plant abiotic stress. J Jilin Agric Univ, 2019, 41(3): 253-260 (in Chinese). 关淑艳, 焦鹏, 蒋振忠, 等. MYB转录因子在植物非生物胁迫中的研究进展. 吉林农业大学学报, 2019, 41(3): 253-260. |
| [11] |
Wang HQ, Guo XR, Yang XB, et al. Screening of the proteins interacting with SmPAP1 of R2R3-MYB transcription factor from Salvia miltiorrhiza Bunge by yeast two-hybrid system. Genom Appl Biol, 2016, 35(10): 2819-2826 (in Chinese). 王怀琴, 郭晓荣, 杨新兵, 等. 利用酵母双杂交筛选与丹参R2R3-MYB类转录因子SmPAP1互作的蛋白. 基因组学与应用生物学, 2016, 35(10): 2819-2826. |
| [12] |
Kocábek T, Mishra AK, Matoušek J, et al. The R2R3 transcription factor HlMYB8 and its role in flavonoid biosynthesis in hop (Humulus lupulus L.). Plant Sci, 2018, 269: 32-46. DOI:10.1016/j.plantsci.2018.01.004 |
| [13] |
Ogata K, Kanei-Ishi C, Sasaki M, et al. The cavity in the hydrophobic core of Myb DNA-binding domain is reserved for DNA recognition and trans-activation. Nat Struct Biol, 1996, 3(2): 178-187. DOI:10.1038/nsb0296-178 |
| [14] |
Ambawat S, Sharma P, Yadav NR, et al. MYB transcription factor genes as regulators for plant responses: an overview. Physiol Mol Biol Plants, 2013, 19(3): 307-321. DOI:10.1007/s12298-013-0179-1 |
| [15] |
Katiyar A, Smita S, Lenka SK, et al. Genome-wide classification and expression analysis of MYB transcription factor families in rice and Arabidopsis. BMC Genomic, 2012, 13: 544-563. DOI:10.1186/1471-2164-13-544 |
| [16] |
Chen YH, Yang XY, He K, et al. The MYB transcription factor superfamily of Arabidopsis: expression analysis and phylogenetic comparison with the rice MYB family. Plant Mol Biol, 2006, 60(1): 107-124. DOI:10.1007/s11103-005-2910-y |
| [17] |
Du H, Wang YB, Xie Y, et al. Genome-wide identification and evolutionary and expression analyses of MYB-related genes in land plants. DNA Res, 2013, 20(5): 437-448. DOI:10.1093/dnares/dst021 |
| [18] |
Wilkins O, Nahal H, Foong J, et al. Expansion and diversification of the Populus R2R3-MYB family of transcription factors. Plant Physiol, 2009, 149(2): 981-993. DOI:10.1104/pp.108.132795 |
| [19] |
Haga N, Kato K, Murase M, et al. R1R2R3-Myb proteins positively regulate cytokinesis through activation of KNOLLE transcription in Arabidopsis thaliana. Development, 2007, 134(6): 1101-1110. DOI:10.1242/dev.02801 |
| [20] |
Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in Arabidopsis thaliana. Curr Opin Plant Biol, 2001, 4(5): 447-456. DOI:10.1016/S1369-5266(00)00199-0 |
| [21] |
Deng J, Li M, Huang LY, et al. Genome-wide analysis of the R2R3 MYB subfamily genes in lotus (Nelumbo nucifera). Plant Mol Biol Rep, 2016, 34(5): 1016-1026. DOI:10.1007/s11105-016-0981-3 |
| [22] |
Schaffer R, Landgraf J, Accerbi M, et al. Microarray analysis of diurnal and circadian- regulated genes in Arabidopsis. Plant Cell, 2001, 13(1): 113-123. |
| [23] |
Yu EY, Kim SE, Kim JH, et al. Sequence-specific DNA recognition by the Myb-like domain of plant telomeric protein RTBP1. J Biol Chem, 2000, 275(31): 24208-24214. DOI:10.1074/jbc.M003250200 |
| [24] |
Rosinski JA, Atchley WR. Molecular evolution of the Myb family of transcription factors: evidence for polyphyletic origin. J Mol Evol, 1998, 46(1): 74-83. DOI:10.1007/PL00006285 |
| [25] |
Jiang CZ, Gu JY, Chopra S, et al. Ordered origin of the typical two- and three-repeat Myb genes. Gene, 2004, 326: 13-22. DOI:10.1016/j.gene.2003.09.049 |
| [26] |
Li DD. Gene regulatory network for tapetum and middle layer in Arabidopsis thaliana[D]. Shanghai: Shanghai Normal University, 2017 (in Chinese). 李丹丹.拟南芥绒毡层和中层的基因调控网络[D].上海: 上海师范大学, 2017. |
| [27] |
Sanders PM, Bui AQ, Weterings K, et al. Anther developmental defects in Arabidopsis thaliana male-sterile mutants. Sexual Plant Reproduct, 1999, 11(6): 297-322. DOI:10.1007/s004970050158 |
| [28] |
Millar AA, Gubler F. The Arabidopsis GAMYB-like genes, MYB33 and MYB65, are microRNA-regulated genes that redundantly facilitate anther development. Plant Cell, 2005, 17(3): 705-721. DOI:10.1105/tpc.104.027920 |
| [29] |
Phan HA, Iacuone S, Li SF, et al. The MYB80 transcription factor is required for pollen development and the regulation of tapetal programmed cell death in Arabidopsis thaliana. Plant Cell, 2011, 23(6): 2209-2224. DOI:10.1105/tpc.110.082651 |
| [30] |
Xu Y, Iacuone S, Li SF, et al. MYB80 homologues in Arabidopsis, cotton and Brassica: regulation and functional conservation in tapetal and pollen development. BMC Plant Biol, 2014, 14: 278. DOI:10.1186/s12870-014-0278-3 |
| [31] |
Zhang W, Sun YJ, Timofejeva L, et al. Regulation of Arabidopsis tapetum development and function by DYSFUNCTIONAL TAPETUM1 (DYT1) encoding a putative bHLH transcription factor. Development, 2006, 133(16): 3085-3095. DOI:10.1242/dev.02463 |
| [32] |
Zhu J, Lou Y, Xu XF, et al. A genetic pathway for tapetum development and function in Arabidopsis. J Integr Plant Biol, 2011, 53(11): 892-900. DOI:10.1111/j.1744-7909.2011.01078.x |
| [33] |
Sorensen AM, KröberS, Unte US, et al. The Arabidopsis ABORTED MICROSPORES (AMS) gene encodes a MYC class transcription factor. Plant J, 2003, 33(2): 413-423. DOI:10.1046/j.1365-313X.2003.01644.x |
| [34] |
Xu J, Yang CJ, Yuan Z, et al. The ABORTED MICROSPORES regulatory network is required for postmeiotic male reproductive development in Arabidopsis thaliana. Plant Cell, 2010, 22(1): 91-107. DOI:10.1105/tpc.109.071803 |
| [35] |
Ito T, Nagata N, Yoshiba Y, et al. Arabidopsis MALE STERILITY1 encodes a PHD-type transcription factor and regulates pollen and tapetum development. Plant Cell, 2007, 19(11): 3549-3562. DOI:10.1105/tpc.107.054536 |
| [36] |
Gu JN, Zhu J, Yu Y, et al. DYT1 directly regulates the expression of TDF1 for tapetum development and pollen wall formation in Arabidopsis. Plant J, 2014, 80(6): 1005-1013. DOI:10.1111/tpj.12694 |
| [37] |
Cai CF, Zhu J, Lou Y, et al. The functional analysis of OsTDF1 reveals a conserved genetic pathway for tapetal development between rice and Arabidopsis. Sci Bull, 2015, 60(12): 1073-1082. DOI:10.1007/s11434-015-0810-3 |
| [38] |
Zhang SY, Wang J, Chen GH, et al. Functional analysis of a MYB transcription factor BrTDF1 in the tapetum development of Wucai (Brassica rapa ssp.). Sci Hortic, 2019, 257: 108728. DOI:10.1016/j.scienta.2019.108728 |
| [39] |
Zhao SQ, Dong JJ. Genetic regulation of anther and pollen development in Arabidopsis thaliana. J Shanxi Univ: Nat Sci Ed, 2015, 38(1): 177-184 (in Chinese). 赵淑清, 董晶晶. 拟南芥花药和花粉发育的分子调控机制. 山西大学学报:自然科学版, 2015, 38(1): 177-184. |
| [40] |
Yang CY, Song J, Ferguson AC, et al. Transcription factor MYB26 is key to spatial specificity in anther secondary thickening formation. Plant Physiol, 2017, 175(1): 333-350. DOI:10.1104/pp.17.00719 |
| [41] |
Ghelli R, Brunetti P, Napoli N, et al. A newly identified flower-specific splice variant of AUXIN RESPONSE FACTOR8 regulates stamen elongation and endothecium lignification in Arabidopsis. Plant Cell, 2018, 30(3): 620-637. DOI:10.1105/tpc.17.00840 |
| [42] |
Li WC, Zhang Y, Zhao SQ. Silencing of Arabidopsis RUS4 impairs anther endothecium secondary cell wall thickening. Chin J Cell Biol, 2019, 41(4): 619-626 (in Chinese). 李文超, 张艺, 赵淑青. 拟南芥RUS4基因沉默对花药药室内壁次生加厚的影响. 中国细胞生物学学报, 2019, 41(4): 619-626. |
| [43] |
Shi JX, Cui MH, Yang L, et al. Genetic and biochemical mechanisms of pollen wall development. Trends Plant Sci, 2015, 20(11): 741-753. DOI:10.1016/j.tplants.2015.07.010 |
| [44] |
Zhu J, Chen H, Li H, et al. Defective in tapetal development and function 1 is essential for anther development and tapetal function for microspore maturation in Arabidopsis. Plant J, 2008, 55(2): 266-277. DOI:10.1111/j.1365-313X.2008.03500.x |
| [45] |
Battat M, Eitan A, Rogachev I, et al. A MYB triad controls primary and phenylpropanoid metabolites for pollen coat patterning. Plant Physiol, 2019, 180(1): 87-108. |
| [46] |
Li Y, Jiang J, Du ML, et al. A cotton gene encoding MYB-like transcription factor is specifically expressed in pollen and is involved in regulation of late anther/pollen development. Plant Cell Physiol, 2013, 54(6): 893-906. DOI:10.1093/pcp/pct038 |
| [47] |
Zhang ZB, Zhu J, Gao JF, et al. Transcription factor AtMYB103 is required for anther development by regulating tapetum development, callose dissolution and exine formation in Arabidopsis. Plant J, 2007, 52(3): 528-538. DOI:10.1111/j.1365-313X.2007.03254.x |
| [48] |
Mandaokar A, Thines B, Shin B, et al. Transcriptional regulators of stamen development in Arabidopsis identified by transcriptional profiling. Plant J, 2006, 46(6): 984-1008. DOI:10.1111/j.1365-313X.2006.02756.x |
| [49] |
Shen XP, Hu ZW, Xiang X, et al. Overexpression of a stamen-specific R2R3-MYB gene BcMF28 causes aberrant stamen development in transgenic Arabidopsis. Biochem Biophys Res Commun, 2019, 518(4): 726-731. |
| [50] |
Lucas WJ, Groover A, Lichtenberger R, et al. The plant vascular system: evolution, development and functions. J Integr Plant Biol, 2013, 55(4): 294-388. DOI:10.1111/jipb.12041 |
| [51] |
Braun DM, Wang L, Ruan YL. Understanding and manipulating sucrose phloem loading, unloading, metabolism, and signalling to enhance crop yield and food security. J Exp Bot, 2014, 65(7): 1713-1735. DOI:10.1093/jxb/ert416 |
| [52] |
Sivitz AB, Reinders A, Ward JM. Arabidopsis sucrose transporter AtSUC1 is important for pollen germination and sucrose-induced anthocyanin accumulation. Plant Physiol, 2008, 147(1): 92-100. DOI:10.1104/pp.108.118992 |
| [53] |
Goetz M, Guivar'ch A, Hirsche J, et al. Metaboliccontrol of tobacco pollination by sugars and invertases. Plant Physiol, 2017, 173(2): 984-997. DOI:10.1104/pp.16.01601 |
| [54] |
Zhang H, Liang WQ, Yang XJ, et al. Carbon starved anther encodes a MYB domain protein that regulates sugar partitioning required for rice pollen development. Plant Cell, 2010, 22(3): 672-689. |
| [55] |
Zhu XL, Liang WQ, Cui X, et al. Brassinosteroids promote development of rice pollen grains and seeds by triggering expression of CSA, a MYB domain protein. Plant J, 2015, 82(4): 570-581. DOI:10.1111/tpj.12820 |
| [56] |
Meng D, He MY, Bai Y, et al. Decreased sorbitol synthesis leads to abnormal stamen development and reduced pollen tube growth via an MYB transcription factor, MdMYB39L, in apple (Malus domestica). New Phytol, 2018, 217(2): 641-656. DOI:10.1111/nph.14824 |
| [57] |
Song SS, Qi TC, Huang H, et al. The jasmonate-ZIM domain proteins interact with the R2R3-MYB transcription factors MYB21 and MYB24 to affect jasmonate-regulated stamen development in Arabidopsis. Plant Cell, 2011, 23(3): 1000-1013. DOI:10.1105/tpc.111.083089 |
| [58] |
Qi TC, Huang H, Song SS, et al. Regulation of jasmonate mediated stamen development and seed production by a bHLH-MYB complex in Arabidopsis. Plant Cell, 2015, 27(6): 1620-1633. DOI:10.1105/tpc.15.00116 |
| [59] |
Huang H, Gao H, Liu B, et al. Arabidopsis MYB24 regulates jasmonate-mediated stamen development. Front Plant Sci, 2017, 8: 1525. DOI:10.3389/fpls.2017.01525 |
| [60] |
Niwa T, Suzuki T, Takebayashi Y, et al. Jasmonic acid facilitates flower opening and floral organ development through the upregulated expression of SlMYB21 transcription factor in tomato. Biosci Biotechnol Biochem, 2018, 82(2): 292-303. DOI:10.1080/09168451.2017.1422107 |
| [61] |
Mandaokar A, Browse J. MYB108 acts together with MYB24 to regulate jasmonate-mediated stamen maturation in Arabidopsis. Plant Physiol, 2009, 149(2): 851-862. DOI:10.1104/pp.108.132597 |
| [62] |
Sun BM, Zhu ZS, Chen CJ, et al. Jasmonate- inducible R2R3-MYB transcription factor regulates capsaicinoid biosynthesis and stamen development in Capsicum. J Agric Food Chem, 2019, 67(39): 10891-10903. DOI:10.1021/acs.jafc.9b04978 |
| [63] |
Cardarelli M, Costantino P. An auxin switch for male fertility. Nat Plants, 2018, 4(7): 408-409. DOI:10.1038/s41477-018-0200-3 |
| [64] |
Wang B, Xue JS, Yu YH, et al. Fine regulation of ARF17 for anther development and pollen formation. BMC Plant Biol, 2017, 17: 243. DOI:10.1186/s12870-017-1185-1 |
| [65] |
Xu XF, Wang B, Feng YF, et al. AUXIN RESPONSE FACTOR17 directly regulates MYB108 for anther dehiscence. Plant Physiol, 2019, 181(2): 644-655. |
| [66] |
Li QX, Zhang L, Wang Y, et al. The research progress of gibberellin on the regulation of flowering and floral organ development in plant. Chin J Cell Biol, 2019, 41(4): 746-758 (in Chinese). 李巧峡, 张丽, 王玉, 等. 赤霉素调控植物开花及花器官发育的研究进展. 中国细胞生物学学报, 2019, 41(4): 746-758. |
| [67] |
Kwon CT, Kim SH, Kim D, et al. The rice floral repressor Early flowering1 affects spikelet fertility by modulating gibberellin signaling. Rice (N Y), 2015, 8: 23. DOI:10.1186/s12284-015-0058-1 |
| [68] |
Liu ZH, Bao WJ, Liang WQ, et al. Identification of gamyb-4 and analysis of the regulatory role of GAMYB in rice anther development. J Integr Plant Biol, 2010, 52(7): 670-678. DOI:10.1111/j.1744-7909.2010.00959.x |
| [69] |
Zhang Y, Zhang XL, Liu B, et al. A GAMYB homologue CsGAMYB1 regulates sex expression of cucumber via an ethylene-independent pathway. J Exp Bot, 2014, 65(12): 3201-3213. DOI:10.1093/jxb/eru176 |
 2020, Vol. 36
2020, Vol. 36