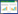中国科学院微生物研究所、中国微生物学会主办
文章信息
- 段云峰, 律娜, 蔡峰, 朱宝利
- Duan Yunfeng, Lü Na, Cai Feng, Zhu Baoli
- 不同保存液和保存期限下肠道微生物组的变化
- Variations of gut microbiome composition under different preservation solutions and periods
- 生物工程学报, 2020, 36(12): 2525-2540
- Chinese Journal of Biotechnology, 2020, 36(12): 2525-2540
- 10.13345/j.cjb.200475
-
文章历史
- Received: July 31, 2020
- Accepted: November 4, 2020
2. 中国科学院大学,北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
肠道微生物对维持人体健康是必不可少,保持肠道微生物的平衡对促进健康和维持体内稳态至关重要。肠道菌群失调造成菌群组成或功能异常,可以导致多种疾病发生。肠道菌群的改变与多种急性和慢性疾病的发病机制有关,如糖尿病[1]、肥胖[2]、代谢综合征[3]、炎症性肠病[4]、癌症[5]、哮喘[6]、自闭症[7]、阿尔茨海默病[8]、帕金森病[9]、焦虑和抑郁[10]等。通过检测肠道菌群组成和功能,对疾病的早期检测和治疗具有重要意义。
正常的肠道菌群主要包括厚壁菌门和拟杆菌门,在不同个体之间高度可变,并且不同研究之间经常出现数据不一致。在研究过程中,有多种因素可能导致数据变化和不一致,尤其是样本存储和运输条件及元基因组提取过程,并且存储条件引起的变异大于存储时间和测序方法的变异[11]。此外,对肠道菌群的研究需要大规模人群,需要采集不同健康状态下个体的粪便样本,提取元基因组DNA后进行高通量基因组测序,比较肠道菌群构成。影响微生物DNA稳定性的因素可能会导致肠道微生物组成发生重大变化,从而影响研究结果。因此,对粪便样品处理和储存方法研究对于微生物组研究至关重要。
粪便是研究肠道菌群构成最常用样本,样本最佳保存方式是−80 ℃冷冻[12]。粪便样本的收集,尤其是非住院患者,通常采取居家取样的方式,但是几乎所有家庭都不具备快速低温冷冻条件,在保存和运输过程中就会导致菌群比例变化[13-14]。即使将这些样品包装在冰袋中或采取其他保护措施,长时间的运输也可能使样品解冻,导致菌群组成发生改变[15]。有研究对室温下粪便样品的稳定性进行了分析,结果显示24 h内粪便样品成分还算稳定,在室温下保存3 d后,不同细菌门水平的相对比例也没有明显变化[16-17]。短期内的常温保存还可以接受,但是在进行标准化和大规模人群采样时,由于采集样本数量多、地域分布广、现场采样条件多样、工作量大、运输条件差等因素限制,对新鲜样本的采集、保存、运输等存在诸多不便,3 d时间很难满足实际需求。
将粪便样本保存在专用溶液中,可以保持长达7 d的稳定性,这就为常温运输和保存提供了便利。在粪便样本采集条件限制下,使用保存液是一种较为理想的解决方案。针对此类问题,本研究选取了5种市售微生物样本采集常温保存液,检测其对新鲜粪便样本的影响,在室温下把粪便放置于不同保存液和不放保存液条件下,放置不同时间后通过16S rRNA基因高通量测序方法,分析其中的微生物组成,比较不同保存液相比新鲜样本对肠道菌群组成的影响,目的是确定适合大规模、多地域研究的粪便保存方法。
1 材料与方法 1.1 样品采集和DNA提取从健康人群中招募5位志愿者,每人提供一份新鲜粪便样品。S1为25岁男性,S2为28岁男性,S3为24岁男性,S4为26岁女性,S5为30岁女性。
采样时使用一次性无菌粪便取样袋(MEDISHARK,GF18B-SL80),用无菌取样勺取粪便中段内部,在粪便多位点挖取样本,每人取15 g左右粪便后加入10 mL PBS缓冲液中,涡旋后充分混匀。每份样本再分别取2.5 mL混匀的样本于5 mL不同品牌的商品化常温液体保存液中(A:Hcode,深圳泓睿康;B:SGENE,深圳赛因生物;C:MEDISHARK (GL18V1-2),北京麦迪时刻;D:PSP® Spin Stool DNA Kit,德国Stratec;E:OMNIgene•GUT,加拿大DNA Genotek)并充分混匀。混合均匀后将上述混有保存液的样本,分别取1.3 mL保存到2.0 mL冻存管中(各5管),上述样品室温保存,取一份不加保存液的样本作为新鲜对照,其余样本分别于第1、3、7、15、30天提取基因组DNA,剩余样本转存于−80 ℃冰箱保存,因保存液E数量不足,仅采集了第3、7、30天的样本,所有样本收集结束后统一提取DNA。
粪便总基因DNA提取,所有样品使用改进的Bead-beater法,通过采用特定粒径的研磨物(玻璃或氧化锆颗粒,粒径1 mm)与破碎仪(QIAGEN涡旋振荡仪和适配器(Cat.No 13000-v1-24的组合) 3 200 r/min研磨10 min后再配合使用QIAampPowerFecal DNA Kit试剂盒(QIAGEN,德国)提取粪便样品总基因组DNA。操作步骤按照试剂盒说明书进行,获得的DNA溶液保存于−20 ℃冰箱中备用。
1.2 PCR扩增和扩增子测序以提取的样品DNA为模板进行PCR扩增,采用二次扩增法,引物使用细菌16S rRNA基因V3–V4通用引物343F (341F:5′-CCTACGGGNBG CASCAG-3′;805R:5′-GACTACNVGGGTATCTA ATCC-3′),第二次扩增用来引入不同index序列。第一次扩增,设定反应条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,25个循环;72 ℃延伸5 min。第二次扩增反应条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,8个循环;72 ℃延伸5 min。
扩增所用引物委托生工生物工程(上海)股份有限公司合成。主要试剂、设备及厂家:KAPA HiFi GC缓冲液(5×)、KAPA dNTP Mix、KAPA HiFiHotStart DNA Polymerase (KAPA Biosystems, 美国);无水乙醇、Na2EDTA·2H2O (北京化工厂,中国);Tris-base (NoVoN,中国)、硼酸(沪试,中国)、琼脂糖(Life Technologies,美国);6×DNA上样缓冲液、100 bp Ladder marker、λ DNA/Hind Ⅲ marker (TIANGEN,德国);Midori Green Advance DNA Stain (Midori Green,德国);AMPure XP (Beackman,德国)。最后采用Illumina公司的Hiseq 2500进行高通量基因测序(PE250)。
1.3 测序结果统计分析测序后将序列分配到其所属样品中,并去掉引物序列,获得有效序列文件。对有效文件进行处理,除去测序质量低的序列。分析数据只保留高质量(Q值≥25)碱基比例大于等于90%的reads。采用QIIME软件中的usearch8.0软件进行嵌合体序列的检测及过滤[18],按照97%的序列相似性进行OTUs (Operational taxonomic units)聚类,采用Silva (https://www.arb-silva.de/)数据库获得样品的分类信息[19]。对每个样品的OTUs进行物种分类学(Taxonomy)注释。分类至门、纲、目、科、属、种各分类水平各自含有的微生物类群数。
Alpha多样性指数和系统发育分析使用Mothur (Version 1.39.5)[20]完成多样性指数的分析,计算样品的Shannon指数,并根据所得到多样性指数数据绘制箱线图。
Beta多样性分析采用R语言工具分析,主坐标分析(Principal coordinate analysis,PCoA)和NMDS (Non-metric multidimensional scaling)非度量多维尺度分析采用vegan软件包进行分析和作图。热图、层次聚类(Hierarchical clustering)分析采用Bray-Curtis Index算法,ward方法绘制聚类树。
差异显著性用Kruskal-Wallis秩和检验比较,结果以平均值±标准差
经过对原始序列的测序质量和序列长度进行质量控制后,共获得120个粪便样品,测序后获得5 529 120条有效序列,每个样本最少序列数46 076,共获得594个OTU。具体每个样本的OUT数量见图 1。从图中可以看出,各样本的OUT数量比较接近,差异不明显。
|
| 图 1 各样本在不同分类级别的OUT数量分布 Fig. 1 OUT number distribution of each sample at different taxonomy levels. |
| |
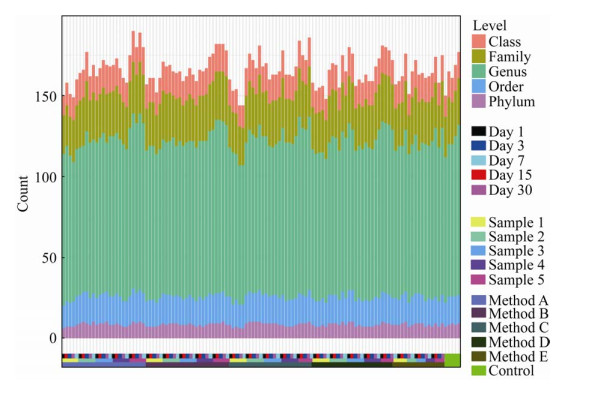
Alpha多样性分析显示,保存液中的粪便样品在室温下保存不同时间后16S rRNA V3–V4区的Shannon多样性指数存在一定差异(图 2)。测试的5种保存液,都存在随时间延长细菌α多样性指数下降趋势。在各时间点,除保存液E之外,其他保存液显示的α多样性波动不明显。与对照(S0)相比,除保存液D外,其他保存液的菌群多样性大幅降低,其中,保存液E降低幅度最大。
|
| 图 2 不同保存液在不同时间点Shannon多样性指数分布 Fig. 2 Shannon diversity index distribution of different preservation solutions at different time points. T1–T5 represent the storage time of 1, 3, 7, 15, and 30 days respectively, and S0 represents the control. A–D represents 5 kinds of preservation solutions. The prism in the figure represents the median. |
| |
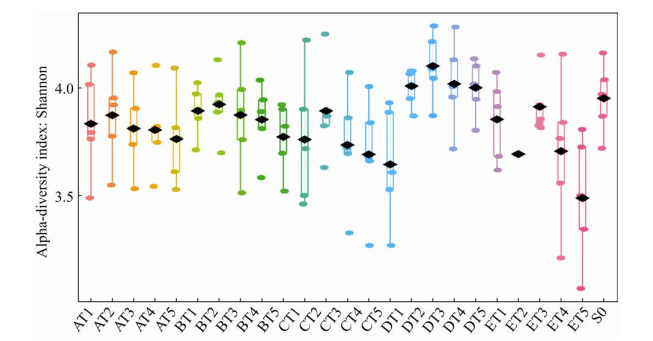
由于每个人的肠道菌群构成不同,在α多样性指数方面也会表现出差异。对每个人的多样性指数分析形成了每个样本的不同保存液在不同时间点Shannon多样性指数分布图(图 3)。结果发现,不同个体的肠道菌群多样性存在差异,其中样本2和5的多样性更高,样本3的多样性最少。保存液D在不同样本中,与对照相比表现出升高多样性的趋势。在样本2中,保存液B和C表现较为理想,在样本5中,保存液B和D表现较为理想。
|
| 图 3 每个样本在不同保存液在不同时间点Shannon多样性指数分布 Fig. 3 The Shannon diversity index distribution of each sample in different preservation solutions at different time points. A–E represent different preservation solutions; S represents control; the sample number is represented by numbers 1-5, and numbers 1, 3, 7, 15 and 30 represent the number of days of storage. |
| |
通过层次聚类(Hierarchical clustering)分析比较不同保存液在不同时间点各样品OTU的群落结构相似度,利用树形图描述和对比了120个样品间的相似性和差异性(图 4),从图中可以看出,来自同一人的大部分样本可以聚在一起,说明个体差异性大于不同保存液引起的差异。然而,在样本3和样本2中存在互换,这两个人的样本采用不同保存液后存在交叉,并且这种交叉只发生在第30天的样本,涉及的保存液有A、B、C和D。此外,不同保存液之间有一定的差异性,其中,在样本2中,保存液B、C和D保存的样本能够聚类在一起,其中保存液D在各样本中都有聚类。
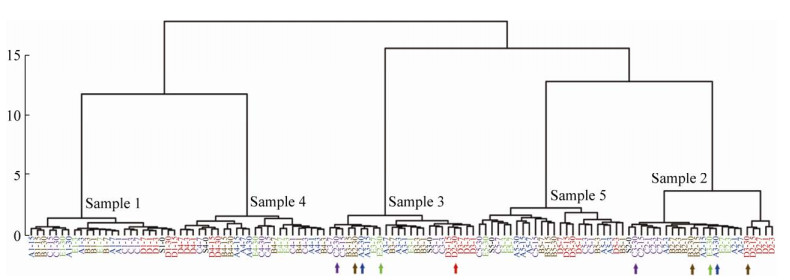
|
| 图 4 不同样本群落结构相似度树形图 Fig. 4 Tree diagram of similarity of community structure in different samples. The tree diagram was constructed by Bray-Curtis Index and ward method. |
| |
根据beta多样性距离矩阵进行的分析显示,不同保存方法,在不同时间点的分布与样本来源联系最密切,有5个人的不同样本分别聚在一起(图 5)。在不同保存方法中,保存液D和E与各样本的距离较远,特别是样本2和样本3,经过保存液D保存的样本在多个时间点上都明显偏离了其对照样本。
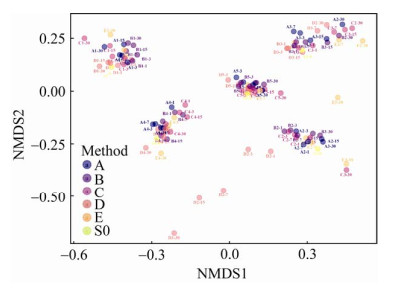
|
| 图 5 Beta多样性距离矩阵NMDS分析结果 Fig. 5 NMDS analysis result of beta diversity distance matrix. |
| |
具体到每一个人的样本,与对照相比,保存液A、C、D更接近对照(图 6)。比如在样本4中,与对照最接近的是保存液C第7天的样本、保存液A第7天的样本和保存液D第7天的样本。
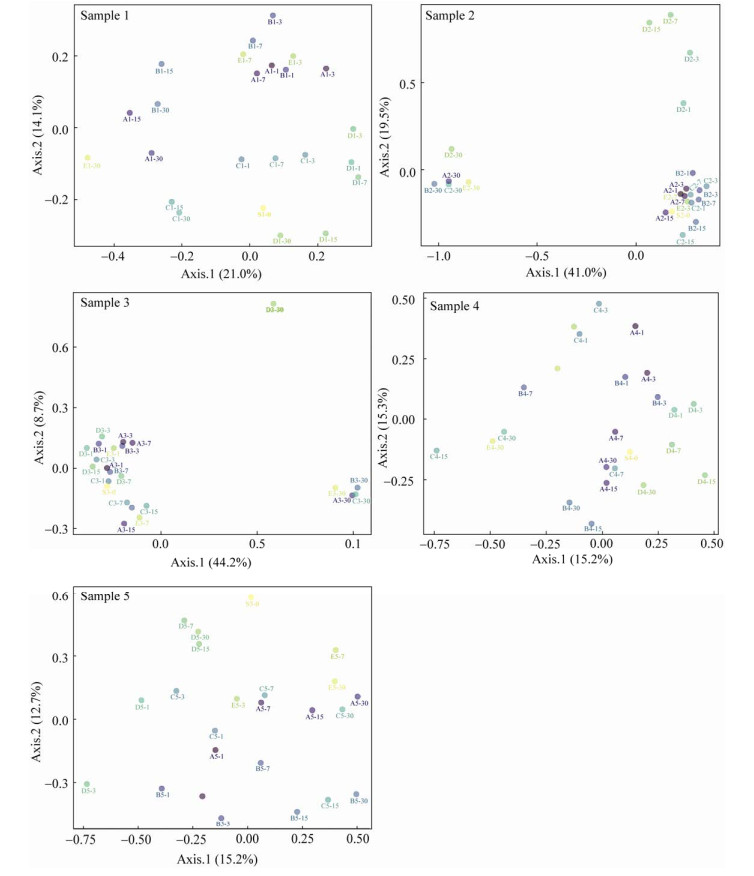
|
| 图 6 Beta多样性PCoA分析结果 Fig. 6 PCoA analysis results of beta diversity. |
| |
为了分析不同保存液在不同时间对粪便样品微生物群落构成的影响,通过对高通量测序结果进行分析,物种注释之后的比对分析结果显示,在门水平,与对照(S0)相比,保存液D使放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)增加,而其他几种保存液都使厚壁菌门减少,而拟杆菌门(Bacteroidetes)比例相对增加(图 7A)。
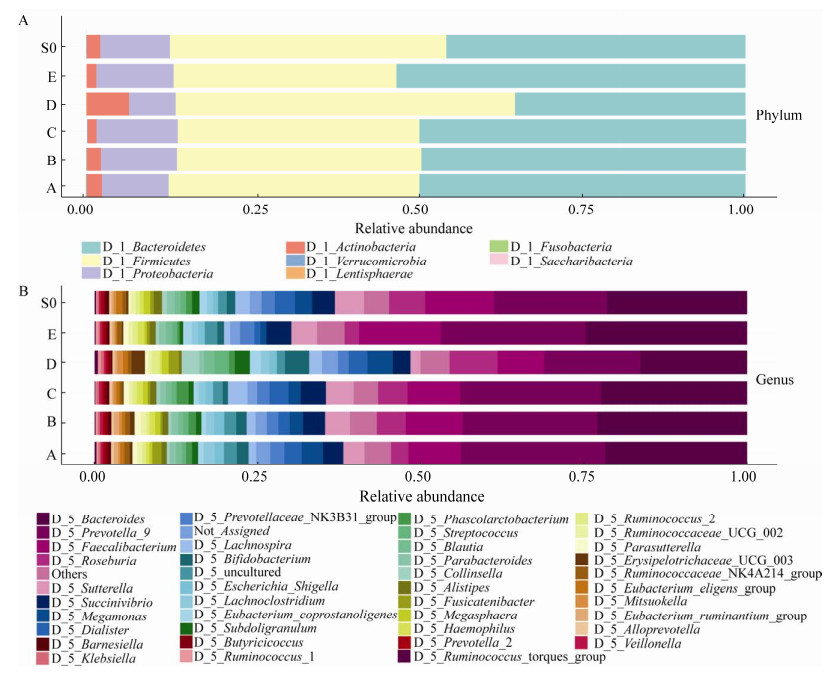
|
| 图 7 各样品中微生物在门和属分类水平的分布 Fig. 7 The distribution of microbiota in the phylum and genus classification levels in each sample. (A) Distribution of bacteria at the phylum level. (B) Distribution of bacteria at the genus level. |
| |
在属的分类水平,保存液D与对照组的差异较大,A、B、C三种保存液与对照组在菌群构成上更接近(图 7B)。
在属水平,通过热图分析和层次聚类(Hierarchical clustering)分析可以看出,来源不同的5个人,肠道菌群的构成本身存在差异(图 8)。从聚类结果上看,在样本1、2、5中,保存液C与对照最接近。在样本4中最接近的是保存液B和C,在样本3中最接近的是保存液E和C。保存液D在不同个体中都是聚类在一起,与对照样本S距离较远。
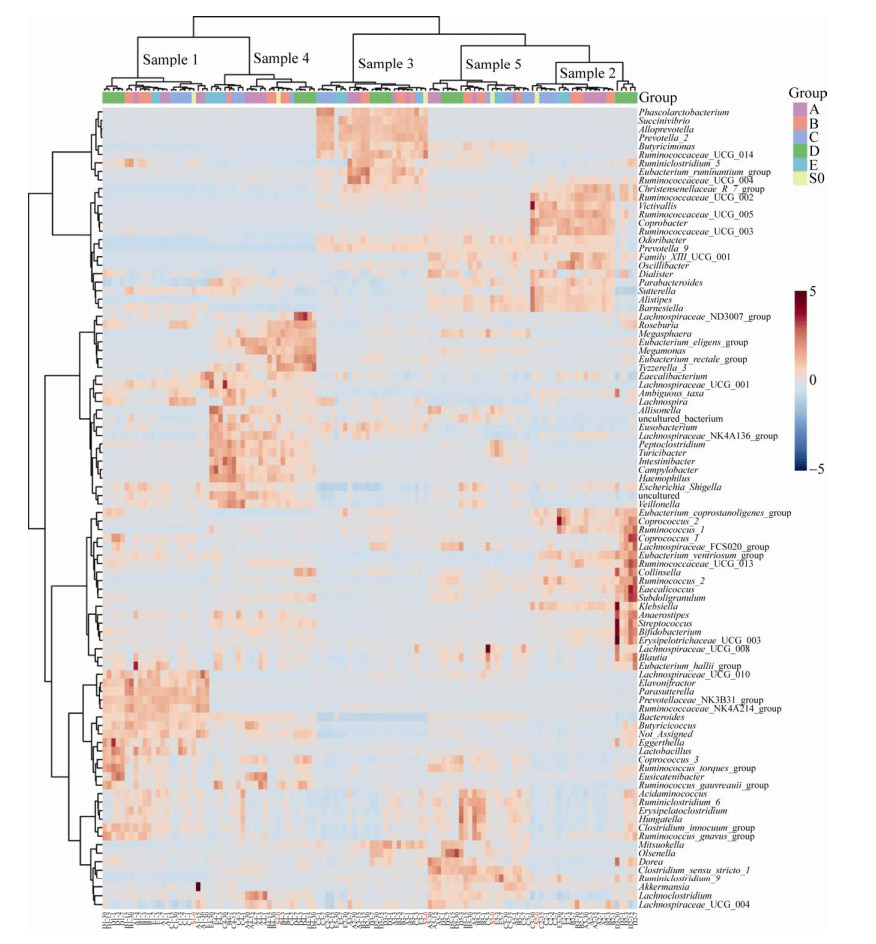
|
| 图 8 不同样本的肠道菌群在属水平的细菌构成热图 Fig. 8 Heat map of gut microbiome composition of different samples at the genus level. |
| |
在门水平上,通过对常见的6个门的细菌进行对比,结果显示,使用不同保存液后,保存液D会明显导致拟杆菌门(Bacteroidetes)和变形菌门(Proteobacteria)降低,而导致厚壁菌门(Firmicutes)和放线菌门(Actinobacteria)增加,而其他几种保存液引起的这几种菌的变化跟对照组相差不大(图 9)。
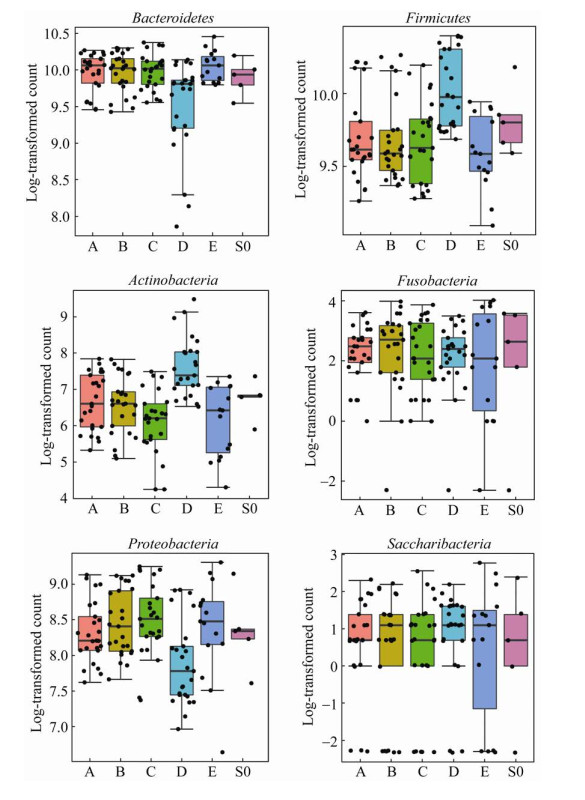
|
| 图 9 门水平不同细菌在不同保存液中的丰度变化 Fig. 9 Changes in the abundance of different bacteria at different phylum levels in different preservation solutions. |
| |
在属水平,通过对常见的6种属水平的细菌进行对比,结果显示,革兰氏阳性杆菌,也是常见的有益菌,双歧杆菌(Bifidobacterium)和乳酸杆菌(Lactobacillus)受保存液影响会出现变动。对双歧杆菌来说,相比对照组,保存液A、B与之相当,保存液C、E相对降低,而保存液D则相对升高其丰度。对乳酸杆菌来说,除保存液E相对降低丰度之外,保存液A、B、C、D与对照都相当。
除革兰氏阳性杆菌外,对革兰氏阳性球菌的分析结果表明,各类保存液对链球菌(Streptococcus)的影响,除保存液D差异较大之外,保存液C与对照组最接近;而各类保存液对瘤胃球菌科UCG 003 (Ruminococcaceae UCG 003)的影响则不同,保存液D比对照组降低,其他各类保存液与对照组差异不大。
对于革兰氏阴性菌,大肠-志贺氏杆菌(Escherichia-Shigella)的表现则是不同保存液都比对照组增加了丰度,而克雷伯氏菌(Klebsiella)的表现则是保存液A、B会增加其丰度,保存液C、D和E与对照组相当。
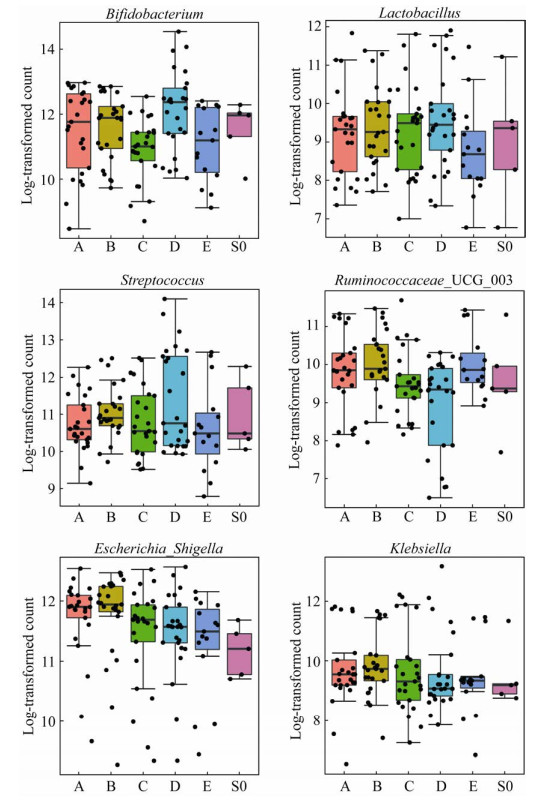
|
| 图 10 属水平不同细菌在不同保存液中的丰度变化 Fig. 10 Changes in the abundance of bacteria at different genus levels in different preservation solutions. |
| |
根据不同保存液在不同时间点肠道菌群构成相关性分析显示,在第1、7、15和30天时间点5种保存液中,保存液A和B在第1和7天时相关性强;在第7、15天时,保存液A还和E的相关性强;与对照组相关性最强的是保存液C,在4个时间点,两者都表现出较强的相关性(图 11)。
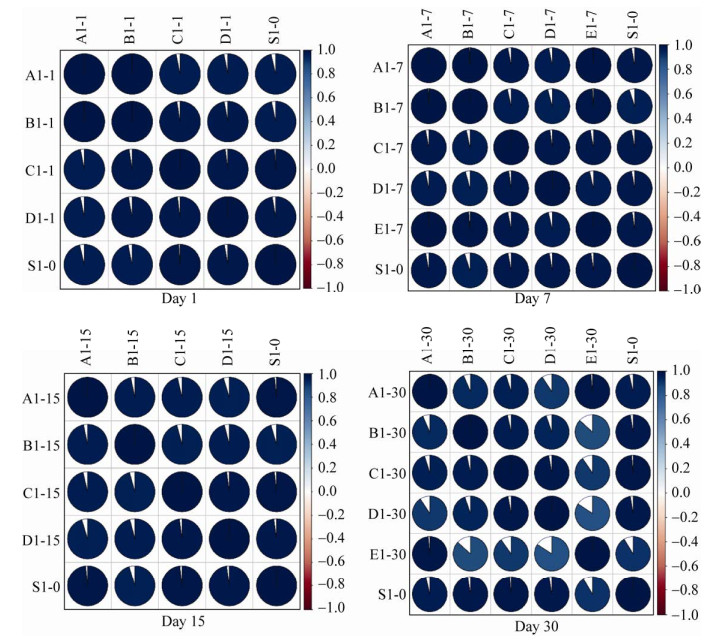
|
| 图 11 不同保存条件下的肠道菌群在不同保存液中的相关性图 Fig. 11 The gut microbiome correlation under different storage conditions in different storage solutions. |
| |
随着室温放置时间的延长,各类保存液中的菌群结构相似性呈现明显变低的趋势,在第30天时尤为明显。以样本1为例,菌群结构相关性的趋势是随样品在室温下放置时间延长,与对照样品菌群结构的相似度逐渐降低(图 12)。与对照样本相关性最强的分别是保存液C在第1、3、15、30天的样本。
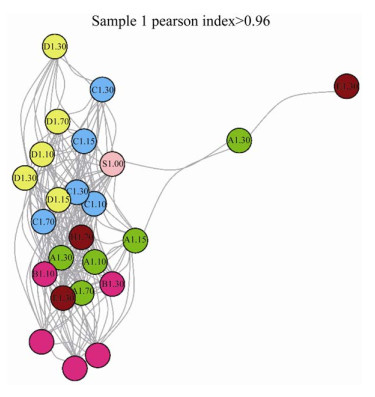
|
| 图 12 样本1在不同保存条件下的肠道菌群在不同保存液中的相关性网路图 Fig. 12 The correlation network diagram of the gut microbiome of sample 1 in different storage solutions under different storage conditions. |
| |
高通量基因测序技术是常用的检测多种复杂微生物群落,如土壤、空气、水体、发酵物、人体及动植物胃肠道系统等的菌群组成及遗传多样性的常用技术。该方法具有无需培养、简单、快捷、可重复性、高通量的特征,可以检测和分析大量样品中的微生物群,因此,在比较不同生态环境中的微生物群落的组成差异和动态监测方面具有显著优势。
肠道微生物相关研究已经发现了大量跟人类健康息息相关的微生物。通过分析微生物的构成可以反映人体的健康状态。由于个体之间的肠道菌群构成较为多样,同时,样本采集之后很难做到及时送检,这就给微生物的活动提供了条件,容易导致菌群组成发生改变。通过抑制微生物的繁殖,保护基因组DNA不被降解的技术可以解决样本采集和保存问题。为此,市面上已经出现了多种粪便样本保存液,可以在常温下保存样本,方便运输和后续检测。
为了建立方便可靠的粪便保存方法,以确保肠道微生物组研究的准确性,需要比较不同保存液在不同时间点对样本中菌群结构的影响。本文采用16S rRNA扩增子高通量测序技术对市售的5种保存液进行对比,评测这些保存液在最长30 d的时间里对粪便样本中的微生物组成,菌群多样性以及具有代表性的细菌丰度的影响。
结果显示,不同保存液在30 d内确实会对菌群组成产生影响,有些保存液引起的偏差较大。一项研究系统地评估了6种粪便样品储存条件,包含3种非商业性储存方案:−80 ℃、−80 ℃含70%乙醇、4 ℃含70%乙醇和3种商业保存液。结果发现,不同的保存液对样本产生的变化存在差异[11]。所以,非常有必要评估不同保存液的保存效果。
如果能及时提取DNA,即使短期室温存储,对微生物组和代谢组谱的影响极小,短期粪便样品存储可以在52 h之内处理[21]。在不能及时提取DNA的条件下,将样本冷冻在−80 ℃条件下,可以在长达6个月的时间里确保粪便微生物群落没有显著变化[22],甚至婴儿粪便样品的在−80 ℃下放置2年,微生物群落也几乎没有显著变化,除了细菌OTU数量有所减少,某些特定OTU的丰度发生了变化[23]。
对样本中微生物多样性的分析显示,随时间延长,各类保存液都存在α多样性指数下降的趋势。这一趋势与−80 ℃条件下保存一致,可能随时间延长细菌DNA降解所致[22]。从层次聚类结果图中可以看出,经过大多数保存液保存后,只在第30天时样本之间在聚类上存在交叉,说明30 d已经会引起肠道菌群组成的明显改变。以往的研究已经证实,在室温下保存2周后,样品中观察到了微生物群落的显著变化[16, 22]。本研究结果将时间延长到30 d后,也观察到了类似现象,因此,两周内的保存时间对肠道菌群的组成影响是可以接受的。
在保持物种多样性方面,保存液D在不同样本中,与对照相比表现出升高多样性的趋势。这可能是由于保存液中的成分不能快速杀死其中的微生物,导致部分微生物出现了增殖,低丰度微生物的增加可能导致了整体物种多样性指数的增加。在保持物种构成方面,保存液D会使放线菌门和厚壁菌门增加,并且在属水平,也表现出与对照组菌群构成差异较大。这些差异与不良的保存条件导致的菌群组成改变一致。有研究发现不加保护剂,常温下肠杆菌科,特别是肠杆菌属的OTU的相对丰度会随着时间延长而增加,表明这类细菌并没有死亡,而是继续繁殖。
与保存液D不同,保存液A、B、C的菌群构成与对照组在门和属水平上都更接近。此外,在不同样本中,热图分析显示,保存液C与对照最接近。在样本3中,最接近的是保存液E和保存液C。而保存液D在不同的个体中都是聚类在一起,与对照样本S距离较远。这就说明保存液D在保持菌群组成稳定方面表现不佳。
具体到某类微生物,对革兰氏阳性杆菌双歧杆菌和乳酸杆菌来说,相比对照组,保存液A、B、C与对照组丰度相当,而保存液E会相对降低,保存液D则相对升高。有研究也观察到了类似现象,不良保存条件会导致乳酸杆菌丰度增加。对革兰氏阳性球菌的分析显示,保存液D会降低该类菌的丰度,保存液C与对照组最接近。对于革兰氏阴性菌,不同保存液都会升高该类菌的比例,而对于克雷伯氏菌则是保存液A、B会导致该菌增加,保存液C、D和E与对照组相当。
从菌群整体构成来看,在第1天时,保存液C就与对照组表现出最强的相关性。此外,在4个时间点,与对照组相关性最强的也是保存液C,在第30天时,仍是保存液C表现最强。在不同时间点,相关性较低的是保存液B和E。
按时间来看,保存液A和C在15 d内都表现较好,可以满足样本运输要求。15 d内甚至一周时间内快递可以运输样本至全国。在中国,快递服务时间一般在3 d左右,保存液对肠道菌群的稳定性能够维持3 d以上理论上都是可以用的。只要样品在收集之日迅速采用合适的保存液并用最快的邮寄方式寄出,并注意避免周末,就可以很方便地将样品送至实验室,并不会明显降低检测质量。但是,在快递服务不可靠的环境中,应该优先考虑使用保护作用更持久的保存液进行样品递送,以确保样本质量。
在本研究中,不同个体的肠道菌群结构差异明显,在α和β多样性方面,都可以看出所选取的5个人的菌群组成有所不同。饮食、身体状况和生活方式等差异会导致菌群结构改变,导致个体菌群组成的差异[24]。但是,通过热图分析可以看出同一个体的优势菌群在不同保存时间内,其相对比例较稳定,占优势的菌群基本没有变化,说明同一个体的肠道菌群即使用不同保存液,在不同时间也具有一定稳定性。
在室温下保藏后,不同保存液对样本的菌群结构变化影响不同,变化的剧烈程度也不同。粪便样品在保存过程中某些细菌种类的损失很可能是由于代谢和氧气条件的改变而造成的[21]。有氧条件,渗透压以及乳酸和短链脂肪酸比例都进一步提高,对粪便中的某些细菌产生致命影响,而一些耐受性较好的菌会部分增殖。保存液可以使粪便样品在保存30 d后与对照样品菌群结构相似度基本在90%以上。可见粪便样本保存液确实能起到稳定样品菌群结构的作用。
采用保存液可以对粪便样品中的肠道菌群进行不超过30 d的常温保存,在室温条件下15 d内可以有效保持肠道菌群结构及多样性,维持主要菌群的相对比例,这样可以降低肠道菌群保存条件,减低采集、运输和和保存成本,可以实现居家采样、大范围采样、跨地域样品运输等特殊条件下对粪便菌群的有效保存。
| [1] |
Graessler J, Qin Y, Zhong H, et al. Metagenomic sequencing of the human gut microbiome before and after bariatric surgery in obese patients with type 2 diabetes: correlation with inflammatory and metabolic parameters. Pharmacogenomics J, 2013, 13(6): 514-522. |
| [2] |
Turnbaugh J, Ley RE, Mahowald MA, et al. An obesity-associated gut microbiome with increased capacity for energy harvest. Nature, 2006, 444(7122): 1027-1031. |
| [3] |
Perry RJ, Peng L, Barry NA, et al. Acetate mediates a microbiome-brain-β-cell axis to promote metabolic syndrome. Nature, 2016, 534(7606): 213-217. |
| [4] |
Dickson I. Gut microbiota: Diagnosing IBD with the gut microbiome. Nat Rev Gastroenterol Hepatol, 2017, 14(4): 195. |
| [5] |
Nakatsu G, Li XC, Zhou HK, et al. Gut mucosal microbiome across stages of colorectal carcinogenesis. Nat Commun, 2015, 6: 8727. |
| [6] |
Thorburn AN, McKenzie CI, Shen S, et al. Evidence that asthma is a developmental origin disease influenced by maternal diet and bacterial metabolites. Nat Commun, 2015, 6: 7320. |
| [7] |
Kang DW, Ilhan ZE, Isern NG, et al. Differences in fecal microbial metabolites and microbiota of children with autism spectrum disorders. Anaerobe, 2018, 49: 121-131. |
| [8] |
Vogt NM, Kerby RL, Dill-Mcfarland KA, et al. Gut microbiome alterations in Alzheimer's disease. Sci Rep, 2017, 7: 13537. |
| [9] |
Hill-Burns EM, Debelius JW, Morton JT, et al. Parkinson's disease and Parkinson's disease medications have distinct signatures of the gut microbiome. Mov Disord, 2017, 32(5): 739-749. |
| [10] |
Chen Z, Li J, Gui SW, et al. Comparative metaproteomics analysis shows altered fecal microbiota signatures in patients with major depressive disorder. Neuroreport, 2018, 29(5): 417-425. |
| [11] |
Ma JL, Sheng LL, Hong Y, et al. Variations of gut microbiome profile under different storage conditions and preservation periods: a multi-dimensional evaluation. Front Microbiol, 2020, 11: 972. |
| [12] |
Shaw AG, Sim K, Powell E, et al. Latitude in sample handling and storage for infant faecal microbiota studies: the elephant in the room? Microbiome, 2016, 4: 40. Microbiome, 2016, 4: 40. |
| [13] |
Voigt AY, Costea PI, Kultima JR, et al. Temporal and technical variability of human gut metagenomes. Genome Biol, 2015, 16: 73. |
| [14] |
Lauber CL, Zhou N, Gordon JI, et al. Effect of storage conditions on the assessment of bacterial community structure in soil and human-associated samples. FEMS Microbiol Lett, 2010, 307(1): 80-86. |
| [15] |
Gorzelak MA, Gill SK, Tasnim N, et al. Methods for improving human gut microbiome data by reducing variability through sample processing and storage of stool. PLoS ONE, 2015, 10(8): e0134802. |
| [16] |
Cardona S, Eck A, Cassellas M, et al. Storage conditions of intestinal microbiota matter in metagenomic analysis. BMC Microbiol, 2012, 12: 158. |
| [17] |
Dominianni C, Wu J, Hayes RB, et al. Comparison of methods for fecal microbiome biospecimen collection. BMC Microbiol, 2014, 14: 103. |
| [18] |
Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. |
| [19] |
Pruesse E, Quast C, Knittel K, et al. SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB. Nucleic Acids Res, 2007, 35(21): 7188-7196. |
| [20] |
Schloss D, Westcott SL, Ryabin T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities. Appl Environ Microbiol, 2009, 75(23): 7537-7541. |
| [21] |
Liang YL, Dong TY, Chen MJ, et al. Systematic analysis of impact of sampling regions and storage methods on fecal gut microbiome and metabolome profiles. mSphere, 2020, 5(1): e00763-19. |
| [22] |
Carroll IM, Ringel-Kulka T, Siddle JP, et al. Characterization of the fecal microbiota using high-throughput sequencing reveals a stable microbial community during storage. PLoS ONE, 2012, 7(10): e46953. |
| [23] |
Bai GY, Gajer P, Nandy M, et al. Comparison of storage conditions for human vaginal microbiome studies. PLoS ONE, 2012, 7(5): e36934. |
| [24] |
de Paepe K, Kerckhof FM, Verspreet J, et al. Inter-individual differences determine the outcome of wheat bran colonization by the human gut microbiome. Environ Microbiol, 2017, 19(8): 3251-3267. |
 2020, Vol. 36
2020, Vol. 36