中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈天达, 张婷婷, 杨娅楠, 赵柏闻, 吴崇明
- Chen Tianda, Zhang Tingting, Yang Yanan, Zhao Bowen, Wu Chongming
- DNA提取过程中混入的实验室空气微生物DNA定量
- Quantification of microbial DNA in laboratory environment during DNA extraction
- 生物工程学报, 2020, 36(12): 2541-2547
- Chinese Journal of Biotechnology, 2020, 36(12): 2541-2547
- 10.13345/j.cjb.200526
-
文章历史
- Received: August 19, 2020
- Accepted: December 14, 2020
2. 中国医学科学院药用植物研究所,北京 100193
2. Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100193, China
人体共生微生物与我们的健康密切相关。人体皮肤、母乳等微生物群在人类健康中发挥关键的作用。但由于采样方法的限制,来自皮肤和母乳等样本中的微生物DNA总量很低,一般只有100 pg左右。由于实验室环境如空气中存在一定量的环境微生物,如果在DNA提取过程未加注意,会不可避免地混入到实验样品中,对实验样品,特别是痕量DNA样品的检测结果造成影响[1]。然而迄今为止,环境DNA混杂对于样品检测结果的影响尚未见报道。
对空气混杂进来的DNA进行相对精确的定量是比较困难的。如此微量的DNA已经低于NanoDrop以及Qubit的检测下限[2]。常规的食品级无菌检测,主要通过培养活菌进行检测。药品如疫苗中的DNA残留检测,分别为杂交法及qPCR法[3-4],但两种方法都是已知DNA残留的宿主来源前提下进行DNA定量。而实验室空气微生物组成未知,因此上述两种方法均无法使用。还有一些研究使用大肠杆菌16S rRNA进行定量[5],但是这种方法仅对单纯大肠杆菌DNA残留是准确的。不同微生物的基因组大小不同,16S rRNA基因拷贝数有差异[6],仅用大肠杆菌16S rRNA定量方法可能会产生比较大的误差。因此,本课题利用空气净化器收集实验室中的空气微生物并以其16S rRNA的qPCR标准曲线进行DNA定量。
1 材料与方法 1.1 实验室空气微生物样本的采集及标准曲线绘制为了绘制实验室环境微生物DNA量的标准曲线,我们通过空气净化器在洁净的实验室中采集空气微生物DNA达30 d。此操作过程参考文献[7],并进行微调。
1.1.1 实验室空气微生物样本的采集(1) 全新小米空气净化器,内外用75%乙醇擦拭干净,并更换全新滤膜。净化器置于实验室中,调至净化模式,时长30 d;(2)关闭净化器,用75%乙醇擦拭过的剪刀和镊子将净化器里面的滤纸剪出一小部分;(3)滤纸的白色面朝里,绿色面朝外,撕开滤纸,使其中的灰色物质暴露出来。(4)将滤纸剪开,分装在8个50 mL离心管中,向其中分别加入50 mL PBS溶液,使滤纸全部被溶液浸渍。(5)用VORTEX-5振荡2 min。(6)用0.2 µm的水系滤膜减压过滤。然后将滤膜放置在通风橱中干燥,约10 min。
1.1.2 DNA提取DNA提取试剂盒采用QIAGEN DNeasy PowerSoil Kit。具体操作如下:(1)将滤膜用剪刀剪碎,收集到装有石榴石的2 mL离心管中,分别加入60 μL C1溶液,在65 ℃金属浴放置15 min。(2)转移至漩涡振荡器上振荡10 min,随后10 000×g离心30 s,将其中的液体全部转移至新管中。(3)加入250 μL C2溶液,振荡5 s,冰浴5 min。随后10 000×g离心60 s,转移上清。(4)加入200 μL C3溶液,振荡5 s (奶白色),冰浴5 min。10 000×g离心60 s,转移上清。(5)取750 μL上清于新管中,加入1 200 μL C4溶液,振荡5 s。(6)取675 μL加入到带有硅胶柱的管子中,10 000×g离心1 min,弃掉下面管子中的液体。重复此步骤,直至取完。(7)加500 μL C5溶液,10 000×g离心30 s。弃掉下面管子中的液体。再10 000×g离心30 s。(8)将柱子转移至新管中,加入50 μL C6,10 000×g离心30 s。
1.1.3 DNA定量使用QubitTM dsDNA HS Assay Kit (Invitrogen),按使用说明书操作。
1.2 实验室开放环境下纯水样品DNA的提取 1.2.1 DNA提取为了检验常规样品DNA提取过程中,实验室环境DNA的混杂对样品检测的影响,我们使用无菌水作为空白样品,使用重鼎生物的微量DNA提取试剂盒(XJ-Micro)在开放的实验室环境下进行常规的DNA提取操作。具体流程如下:(1)取200 μL无菌水作为空白样本。(2)向管中加入Proteinase K 15 μL,混匀。56 ℃孵育30 min。(3)向管中加入600 μL溶液GH及600 μL无水乙醇,充分混匀。12 000×g离心2 min。(4)将上清液转入套有收集管的微量离心柱中。8 000×g离心2 min。取出离心柱后弃去收集管中废液,将离心柱放回收集管中。(每次加700 μL,共6次)。(5)向离心柱中加入200 μL溶液W1,8 000×g离心1 min。(6)向离心柱中加入200 μL溶液W2,8 000×g离心1 min。弃废液,将离心柱放回。(7)重复步骤(6)一次。12 000×g离心2 min。(8)将离心柱取出后放入新的1.5 mL离心管中。向柱中央加入5 μL无菌去离子水,室温放置3 min,12 000×g离心1 min。再向柱中央加入5 μL无菌去离子水,室温放置2 min,12 000×g离心1 min,DNA溶液被收集在离心管中。
1.2.2 DNA定量我们使用细菌通用16S rRNA引物,通过Realtime PCR方法对空白样品中的细菌DNA含量进行定量。所用引物序列如下:515F正向引物为5ʹ-GTGYCAGCMGCCGCG GTAA-3ʹ;806R反向引物为5ʹ-GGACTACNVGGGTWT CTAAT-3ʹ。
Realtime PCR使用KAPA SYBR® FAST qPCR Master Mix (2×) Kit,按照试剂盒的使用说明书,在Bio-Rad CFX96仪器上运行。
1.3 皮肤样品DNA的提取 1.3.1 皮肤样本的采集将灭菌拭子在无菌生理盐水中浸湿,在实验目标区域擦拭约30 s (在不产生不适感的前提下尽量用力);取样后,将棉拭子头伸入无菌取样管(含有无菌水)中,折断拭子头与杆部的连接,弃掉拭子杆。在VORTEX-5上振荡1 min后,移除拭子头,并高速离心10 min,去掉上清。
1.3.2 DNA提取分别在生物安全柜和实验室开放环境下使用DNeasy PowerSoil pro试剂盒(QIAGEN)对皮肤DNA进行提取。
1.3.3 鸟枪法元基因组测序使用KAPA HyperPlus试剂盒(KAPA Biosystems)对DNA进行文库构建。构建的文库的插入大小约为350 bp,然后进行高通量测序。在Illumina NovaSeq 6000平台(Illumina)上进行测序,每个样本的测序深度为3 Gb。原始的下机数据,使用KneadData进行质量控制和宿主基因的去除(v0.7.2,宿主基因为H. sapiens,ucschg19,默认参数)。质控后的clean reads,使用PathoScope 2.0 (默认参数),与NCBI的nt数据库进行比对,得到物种注释结果。
2 结果与分析 2.1 实验室环境微生物DNA的掺杂量为了检测在实验室开放环境下提取DNA导致的环境细菌DNA掺杂量,我们首先用空气净化器采集实验室空气微生物样本,并用细菌16S rRNA引物建立了基于qPCR的DNA含量标准曲线(图 1)。

|
| 图 1 实验室环境微生物DNA标准曲线 Fig. 1 Standard curve for laboratory environmental microbial DNA |
| |
随后我们在实验室开放环境下进行纯水样本的DNA提取,并根据标准曲线计算提取过程中掺杂的环境微生物DNA量。实验结果显示,在实验室开放环境中提取的纯水样品中,空气微生物DNA的残留量为28.9 pg。
2.2 开放环境DNA提取掺杂的微生物种类进一步在实验室开放环境下对纯水进行DNA提取和元基因组分析,以确定掺杂的主要环境微生物种类。结果显示,有22种环境细菌掺杂到了无菌的纯水样品中,其中丰度最高的是痤疮杆菌Cutibacterium acnes (77.060%)、大肠杆菌Escherichia coli (8.073%)、表皮葡萄球菌Staphylococcus epidermidis (4.329%)、金黄色葡萄球菌Staphylococcus aureus (2.324%)和头状葡萄球菌Staphylococcus capitis (1.597%) (表 1)。这些掺杂菌都是空气和皮肤上常见的细菌。
| Species | Relative abundance(%) |
| Cutibacterium acnes | 77.060 |
| Escherichia coli | 8.073 |
| Staphylococcusepidermidis | 4.329 |
| Staphylococcus aureus | 2.324 |
| Staphylococcus capitis | 1.597 |
| Burkholderia contaminans | 0.927 |
| Unclassified Gordonia | 0.514 |
| Acinetobacter johnsonii | 0.412 |
| Olsenella uli | 0.354 |
| Klebsiella pneumoniae | 0.336 |
| Pseudopropionibacteriumpropionicum | 0.287 |
| Kocuria rhizophila | 0.270 |
| UnclassifiedStreptococcus | 0.267 |
| Enterobacter cloacae | 0.229 |
| Brevundimonas sp. DS20 | 0.193 |
| Corynebacteriumglutamicum | 0.167 |
| Corynebacterium jeikeium | 0.142 |
| Bifidobacterium pseudocatenulatum | 0.130 |
| Kocuria palustris | 0.129 |
| Altererythrobacteratlanticus | 0.119 |
| Unclassified Curtobacterium | 0.090 |
| Micrococcus luteus | 0.086 |
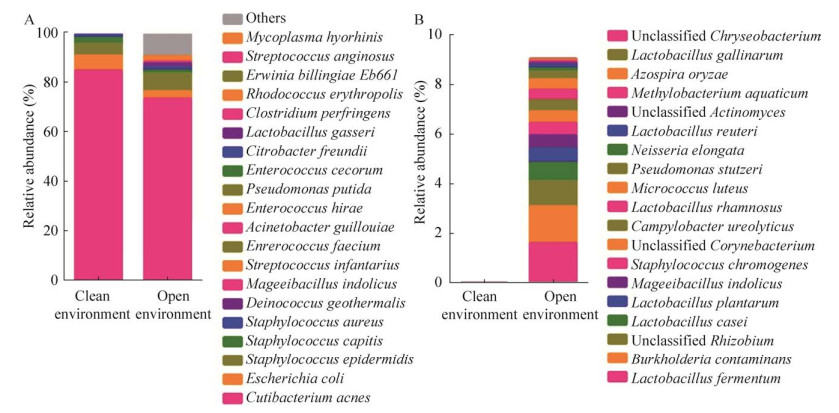
我们进一步分别在生物安全柜和实验室开放环境下对同一份皮肤样本进行DNA提取,考察环境微生物掺杂对所得分析结果的影响。与洁净环境(生物安全柜)下提取的皮肤样品相比,在开放环境下提取的皮肤样品中出现了83种新的菌种,其中7种掺杂细菌发酵乳杆菌Lactobacillus fermentum、洋葱伯克氏菌Burkholderia contaminans、未分类的根瘤菌unclassified Rhizobium、干酪乳杆菌族Lactobacillus caseigroup、植物乳杆菌Lactobacillus plantarum、产色葡萄球菌Staphylococcus chromogenes和未分类的棒状杆菌unclassified Corynebacterium的相对丰度超过了0.5%,而Lactobacillus fermentum和Burkholderia contaminans的掺杂量达到了1.5%左右(图 2)。由于环境细菌的掺杂,导致皮肤样本中原本的优势菌种,如Cutibacterium acnes和Escherichia coli的相对丰度分别降低了11.35%和3.11%,而Staphylococcus epidermidis的相对丰度升高了2.45% (图 2)。
|
| 图 2 开放环境DNA提取对皮肤样本微生物组检测结果的影响 Fig. 2 Impact of DNA extraction under open environment on the microbial analysis results of skin sample. (A) The microbial composition obtained under the two conditions. (B) The incorporated bacteria by DNA extraction under open environment. |
| |
空气中漂浮的灰尘微粒上附着有大量的环境微生物,在DNA提取过程会不可避免地混入到所得的样品DNA中。虽然对于微生物DNA含量异常丰富的样品,例如粪便样品,环境微生物掺入所造成的影响可以忽略不计,但是对于微生物DNA含量本就非常微小的样品,如皮肤样品和母乳样品等,在DNA提取过程中如果不注意防护,那么混入的环境微生物DNA有可能大大影响检测结果的准确性。然而,这些掺入的环境微生物DNA会对这些微量样品的元基因组检测结果造成多大的影响,目前尚没有明确的结论。
我们的结果显示,DNA提取过程中实验室环境微生物的掺入有可能对微量样品的元基因组检测结果造成严重的影响。首先,在开放环境下提取DNA可能导致大约30 pg的DNA掺杂。对于粪便、唾液等样本(样本DNA提取量可达1 μg),空气微生物的比例小于万分之一,因此对于这类样本中万分之一以上丰度的微生物不会产生很大影响。但在某些样本类型,如皮肤样本等,样本DNA总量可能小于1 ng,母乳样本中的微生物DNA总量可能更低。而且这类样本中,人源DNA比例可高达90%以上,微生物DNA总量约为100 pg以下[8]。实验空气微生物在DNA提取过程中的残留量可达到其总量的30%以上,对这类样本的元基因组检测可能会产生较大影响。
我们利用纯水样品进一步分析了在实验室开放环境下进行DNA提取所掺杂的主要环境微生物种类。虽然所在实验室在肉眼检测下非常干净整洁,但是在DNA提取过程中依然有大量微生物掺杂进来。我们在纯水样品中检测到了22种环境细菌,其中含量最丰富的是空气和皮肤上常见的细菌。由于皮肤菌群主要来自于同空气等接触得到的细菌,并且在人活动过程中皮肤上的微生物也会扩散到环境空气中,因此,我们的实验结果非常容易理解。但是这也可能为皮肤样本的菌群分析结果造成更加隐蔽的影响,导致所得元基因组数据不可靠。
为了深入了解DNA提取过程中环境微生物DNA的掺杂对皮肤菌群分析结果的影响,我们使用了同一个皮肤样品并分别在无菌的生物安全柜和表面上看似洁净的实验台上进行DNA提取。元基因组测序结果显示,由于环境细菌的掺杂,导致皮肤样本中优势菌种的相对丰度发生了明显的改变,最多降低了11.35%,从而导致所得细菌组成结果的准确性大大降低。更重要的,在开放环境下提取的皮肤样品中掺杂了83种环境细菌,其中7种掺杂细菌的丰度超过了0.5%。而该皮肤样品中真实含有的细菌种类只有34种。换言之,在开放环境下进行DNA提取会使所检测到的细菌种类增加49种,提高将近1.5倍(144%)。这个结果会大大增加皮肤微生物组数据的多样性指数,从而得到完全错误的整体皮肤菌群分析结果。
因此,当对极低量样本进行DNA提取时,为了进一步提高其准确性,需要注意空气微生物的影响。可以提高实验室的洁净度级别,或尽量在超净台中处理样本[9]。同时,也需要注意DNA提取及建库过程中使用的试剂和耗材的DNA残留问题[10]。
| [1] |
Cheema AS, Stinson LF, Lai CT, et al. DNA extraction method influences human milk bacterial profiles. J Appl Microbiol, 2020. DOI:10.1111/jam.14780 |
| [2] |
Yang P, Zhang Y, Li W, et al. Development of a method for determination of residual exogenous DNA in recombinant human enterovirus 71 virus-like particle vaccine. Chin J Biol, 2020, 33(3): 308-311, 315 (in Chinese). 杨萍, 张艳, 李微, 等. 重组人肠道病毒71型病毒样颗粒疫苗外源DNA残留量检测方法的建立. 中国生物制品学杂志, 2020, 33(3): 308-311, 315. |
| [3] |
Cafiero C, Re A, Stigliano E, et al. Optimization of DNA extraction from dental remains. Electrophoresis, 2019, 40(14): 1820-1823. |
| [4] |
Ponti G, Maccaferri M, Manfredini M, et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clin Chim Acta, 2018, 479: 14-19. |
| [5] |
Peng Y, Yang XM. Comparison of testing methods for residual DNA in biotech drugs. Int J Biol, 2016, 39(2): 73-76 (in Chinese). 彭燕, 杨晓明. 生物技术药物中残留DNA检测方法的比较. 国际生物制品学杂志, 2016, 39(2): 73-76. |
| [6] |
Lang-Yona N, Mazar Y, Pardo M, et al. Air-sampled filter analysis for endotoxins and DNA content. J Vis Exp, 2016(109): 53444. |
| [7] |
Kobayashi A, Sano D, Taniuchi A, et al. Use of a genetically-engineered Escherichia coli strain as a sample process control for quantification of the host-specific bacterial genetic markers. Appl Microbiol Biotechnol, 2013, 97(20): 9165-9173. |
| [8] |
Bjerre RD, Hugerth LW, Boulund F, et al. Effects of sampling strategy and DNA extraction on human skin microbiome investigations. Sci Rep, 2019, 9: 17287. |
| [9] |
Hung JC, Anderson MM. Mayo Clinic approaches to meet United States Pharmacopeia <797>requirements for facility design and environmental controls of nuclear pharmacy. J Nucl Med, 2009, 50(1): 156-164. |
| [10] |
Lee C, Lee S, Shin SG, et al. Real-time PCR determination of rRNA gene copy number: absolute and relative quantification assays with Escherichia coli. Appl Microbiol Biotechnol, 2008, 78(2): 371-376. |
 2020, Vol. 36
2020, Vol. 36