中国科学院微生物研究所、中国微生物学会主办
文章信息
- 韩娜, 彭贤惠, 张婷婷, 强裕俊, 李秀文, 张雯
- Han Na, Peng Xianhui, Zhang Tingting, Qiang Yujun, Li Xiuwen, Zhang Wen
- 一种新的定量16S rRNA基因扩增子测序方法
- A new quantitative 16S rRNA amplicon sequencing method
- 生物工程学报, 2020, 36(12): 2548-2555
- Chinese Journal of Biotechnology, 2020, 36(12): 2548-2555
- 10.13345/j.cjb.200353
-
文章历史
- Received: June 16, 2020
- Accepted: September 27, 2020
- Published: October 30, 2020
2. 感染性疾病诊治协同创新中心,浙江 杭州 310003
2. Collaborative Innovation Center for Diagnosis and Treatment of Infectious Disease, Hangzhou 310003, Zhejiang, China
16S rRNA编码基因是细菌染色体上编码16S rRNA相对应的DNA序列,其种类少、含量大,且存在于所有的细菌基因组中,是细菌系统分类研究中最有用和最常用的分子钟。16S rRNA基因序列包括9个可变区和10个保守区,保守区序列反映了物种间的亲缘关系,而可变区序列则能体现物种间的差异。通过在16S rRNA的两个保守区域设计通用引物,可对样本中所有细菌的相应区域进行扩增,且由于扩增片段大小适中,可利用测序技术对上述基因序列某个或者某几个高变区(如16S rRNA基因的V1–V2、V3–V4、V4或V5等)进行测序,测序数据通过与16S rRNA基因参考数据库(如RDP、SILVA、GreenGene等)[1-3]比对可获得序列变异和相对丰度,进而获得样本物种分类、物种丰度、种群结构、系统进化、群落比较等诸多信息。该技术被称为16S rRNA基因扩增子测序技术,近年来被广泛用于肠道微生物菌群结构和组成的测定[4-6],同时也可用于临床样本的未知病原菌筛查[7]。
目前16S扩增子测序技术主要是基于二代测序技术,受限于二代测序的读长(50–600 bp),目前最多只能测16S rRNA基因9个可变区中的2个,较常采用的为V3–V4区,对微生物群落中大部分物种的分辨率仅能到属水平的相对丰度,只有部分菌可以到种水平。且实验过程中不同因素对结果皆具有一定影响,如样本浓度、PCR循环数、扩增引物等[8-9]。由于不同研究课题采用的测序目标区段和测序平台不完全一致,由实验因素引入的与实际情况的偏差也各不相同,因此不同项目的16S扩增子测序结果之间的可比性较差,已有的多个国际、国内公开的16S扩增子测序项目的数据也不能直接应用到自己的研究课题和检测项目中。
同时由于16S rRNA基因扩增子测序手段通过某一分类单元的序列数占总序列数的比值获得相对丰度数据,而相对丰度信息不能很好地反映样本中物种绝对丰度情况,忽略了微生物总量的影响,微生物群的潜在病理学、生理学和生态学意义可能会被相对丰度所掩盖。且仅有相对丰度数据,无法实现同一样本不同检测方法(如传统的培养法、定量PCR法和数字PCR法等)获得的结果直接相比较,同时也削弱了不同样本间的可比性。
目前细菌微生物绝对定量的方法主要有3种:1)运用荧光定量PCR的方法计算细菌绝对量。需要特定物种设计特定的引物,对引物特异性要求比较高,且引物优化难度较大[10]。2)通过加入已知拷贝数的外源菌估算特定样本中微生物的绝对量。外源菌内标的方法对外源菌的要求比较高,必须为所检测样本中不存在的菌,并且要有特异性的探针[11-12]。3)通过流式细胞计数法计算微生物细菌绝对量。该方法操作复杂,且操作过程中或其他因素引入的死亡细胞没有计入[13-15]。
鉴于以上问题,开发一个受实验因素影响较低的、能直接反映复杂样本微生物群落结构中不同物种实际含量的16S扩增子检测方法对于菌群多样性研究和临床未知病原检测都非常必要。本研究采用随机标签和内参法相结合的方法,开发了一套定量16S rRNA基因扩增子测序方法,能够获得样本中各物种的绝对丰度数据,较为有效地解决了以上问题。
1 方法 1.1 内参设计人工设计两种含有插入序列的质粒作为测序内参(Marker)。Marker序列模拟16S V3/V4区,其结构为341F扩增引物+随机序列(300 bp)+805R引物。所插入的随机序列已通过Blast方法验证,与已知自然物种序列无匹配。根据合成的质粒浓度和分子量可换算出质粒拷贝数。每样本建库时添加1 μL总量50 000拷贝(浓度为50 000 copies/μL)的Marker1和1 μL总量10 000拷贝(10 000 copies/μL)的Marker2作为内参。
采用RT-PCR方法对两种Marker进行定量,上游引物为5′-CCTACGGGNGGCWGCAG-3′,下游引物为5′-GACTACHVGGGTATCTAATCC-3′,探针为5′-GTGCCAGCAGCCGCGGTAA-3′ (5′-FAM标记)。重复7次实验,含有50 000拷贝的Marker1的样本CT值平均为25.91,含有10 000拷贝的Marker2的样本CT值平均为27.03。
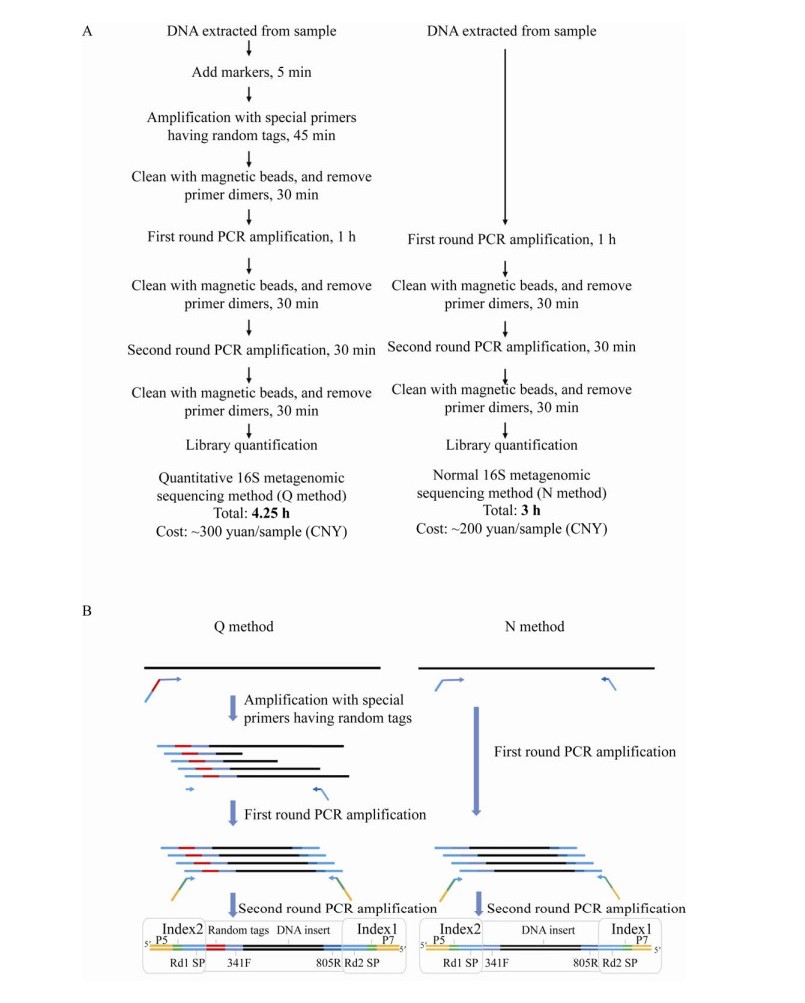
1.2 定量16S rRNA基因扩增子测序方法(Q方法)实验流程采用设计5′端含有18 bp随机序列标签的扩增引物,对16S V3–V4区进行序列片段的扩增(上游引物为5′-CCTACGGGNGGCWGCAG-3′,下游引物为5′-GACTACHVGGGTATCTAATCC-3′),实验流程如图 1A所示,扩增后的产物结构如图 1B所示。
|
| 图 1 实验流程图(A)和文库结构图(B) Fig. 1 Flow chart of the experiment (A) and library structure (B) |
| |
采用问卷调查的方式对志愿者进行个人情况调查。为避免粪便被马桶水污染,符合条件者以发放一次性便盆的方式采集样本。最终纳入7名符合标准的志愿者,以一个月为间隔采集粪便样本,共收集40份粪便样本。收集的粪便样本,按QIAamp DNA Stool Mini Kit试剂盒说明操作提取样本DNA。核酸提取后采用Qubit® dsDNA HS分析试剂盒定量。
以提取的粪便样本核酸为模板,分别按照常规的16S两步建库法(16S V3/V4,N法)和定量16S (Q法)进行文库构建(图 1)。两种方法中都在建库时添加1 μL总量50 000拷贝(浓度为50 000 copies/μL)的Marker1和1 μL总量10 000拷贝(10 000 copies/μL)的Marker2作为内参。构建好的文库使用Qubit® dsDNA HS分析试剂盒测定文库浓度,并采用Agilent 2100或1%琼脂糖凝胶电泳检测文库片段大小。对于含有小片段多的文库,采用割胶纯化方法处理或重新建库。合格的文库采用Illumina Miseq高通量测序平台完成样本的16S rDNA V3–V4区段测序,测序模式为PE 300。每个样本拼接后获得的高质量序列数目不低于3万条reads,碱基数据质量值Q30不低于80%。未符合要求的样本未纳入后续分析。
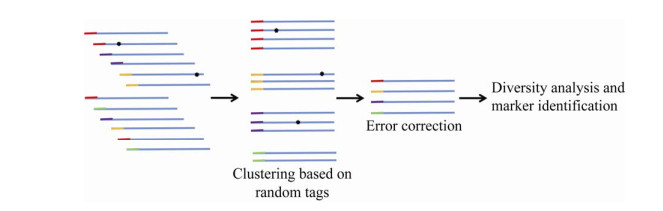
1.4 数据分析下机序列经PEAR软件将双端序列低质量过滤、拼接成高质量的tags。对于N法测序数据,基于Parallel-meta3[16]计算OTU数、Genus相对含量和各样本的多样性指数,进行PCoA分析。对于Q法测序数据,基于本课题组自主开发的Q16Spipeline分析流程,首先对具有相同标签的16S序列聚类,基于聚类结果进行测序数据的自我矫正,并进行计数和Marker统计等功能,然后基于实验中实际添加的Marker数,换算出样本中检测出的微生物种属的实际含量。利用Alpha diversity wilcoxon test方法比较不同方法处理的两组样本的Shannon指数、Simpson指数和Chao1指数。利用SPSS 16.0软件进行统计学计算(图 2)。
|
| 图 2 数据分析流程图 Fig. 2 Flow chart of data analysis. |
| |
定量16S建库法相比较于常规16S建库方法增加了一轮随机引物扩增和清洗步骤,因此实验成本(约300元/样本vs约200元/样本)和时间(4.25 h vs 3 h)都有提高(图 1)。
2.2 多样性比较取15份粪便样本DNA,平分为2份,分别按照Q法和N法建库,然后利用Miseq测序平台PE300模式测序。
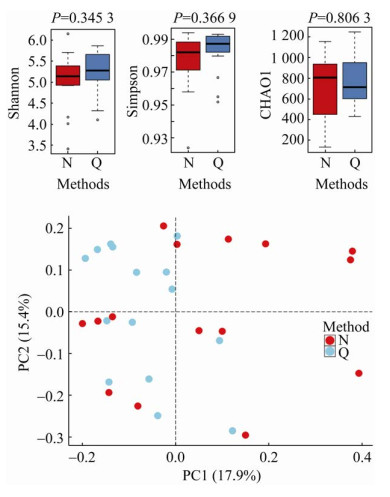
对测序数据进行筛选、拼接和校正后,比较两组的Shannon指数、Simpson指数和Chao1指数,无显著差异(t-test,P > 0.05),如图 3所示。PCoA分析也未发现两组具有显著性差异(图 3)。
|
| 图 3 Alpha指数和PCoA分析 Fig. 3 Alpha diversity and PCoA analysis. |
| |
计算样本间Unweighted Distance也显示,相同样本不同方法检测的距离显著小于不同样本间(t-test,P < 0.05) (图 4)。
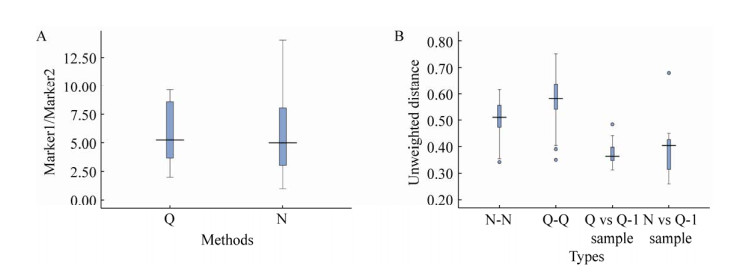
|
| 图 4 Q方法和N方法的比较和稳定性评测(A:Q方法和N方法样本中Marker1/Marker2的比值. B:不同样本间的Unweighted distance箱式图,从左至右分别为采用N方法的不同样本间距离(N-N)、采用Q方法的不同样本间距离(Q-Q)、相同样本采用相同方法(Q方法)的距离(Q vs Q-1 sample)、相同样本采用不同方法间的距离(N vs Q-1 sample)) Fig. 4 Comparison and stability assessment of method Q and method N. (A) The ratio of Marker1/Marker2 using method Q and method N, respectively. (B) Boxplots for unweighted unifrac distance intra- and inter samples. From left to right were distances distribution from different samples using method N (N-N), distance distribution from different samples using method Q (Q-Q), distances distribution from same samples using method Q (Q vs Q-1 sample), distances distribution from same samples using different methods (N vs Q-1 sample). |
| |
以上结果皆显示Q方法和N方法在多样性检测方面具有较高的一致性,未发现具有显著差异。
2.3 基于Q方法的序列纠错基于随机标签,可对扩增和测序阶段引入的测序错误进行纠错,提高测序数据的准确度。纠错策略如下:第一步,将具有相同标签的读序归到一组;第二步,同组数据比对,找出不一致的位点;第三步,基于打分的方法判断不一致的位点的碱基情况;第四步,生成纠错后的序列。
采用Q方法中的随机标签对40份粪便样本测序,平均每1.95条序列拥有一个相同的标签。将相同的标签聚类后同组比对,序列不一致时,首先排除低质量测序引入的碱基不一致,逐个对碱基位点进行计数打分,分高的碱基作为正确的碱基,生成纠错后的一致性序列,发现所得测序序列中有平均15.6%序列存在纠错位点。
2.4 Marker计数采用Q方法和N方法建库的样本中均人为添加了两种Marker序列,数量分别为Marker1 (50 000拷贝)和Marker2 (10 000拷贝)。对所有样本分别进行Q方法和N方法测序后,统计测序数据中Marker1和Marker2的数目,Marker1在Q方法和N方法中的平均丰度为0.2%和0.03%。Marker2在Q方法和N方法中的平均丰度为0.03%和0.01%。计算各样本中Marker1/Marker2的比值,绘制箱式图,如图 4A所示。相较于实际添加比率5,Q方法得到两种Marker丰度比为2–9.67,略优于方法N的丰度比范围(1–14) (图 4A)。
2.5 重复测序评估Q方法稳定性选取16份粪便样本DNA,一分为二,均采用Q方法建库测序,分别上机。利用Q16Spipeline分析流程对测序获得序列开展自矫正和计数计算,并进行多样性指数计算。结果显示两次重复间的Distance (图 4B,Q vs Q-1sample)低于样本间Distance (图 4B,Q和N,t-test)。
2.6 基于Q16流程的定量分析为配套Q方法产生的16S测序数据分析,本研究开发了一套Q16Spipeline分析流程,实现对测序数据的自我矫正、计数和Marker统计等功能,并基于添加的Marker实际含量,换算样本中各检测的微生物种属的实际含量。分析流程已部署到公开的微生物分析云平台(https://analysis.mypathogen.org/),作为公共的分析工具,可供科研工作者使用。以样本17ZYH03为例,样本中检出高于50 000拷贝数的属有15个,其中Bacteroides含量最高,换算所得检测样本中Bacteroides含量有275万拷贝。换算公式为:
| $ {C_{\rm{n}}} = \frac{{{C_{\rm{m}}}}}{{{D_{\rm{m}}}}} \times {D_{\rm{n}}} $ |
其中,Cm为所添加的Marker实际数量,Dm为Marker的检出序列数量,Dn为该菌属的检出序列数量,Cn为该菌属在样本中的实际数量。
3 讨论人体由复杂多样的微生物群落定殖。人体微生态相关的微生物组已经被16S rRNA基因、ITS扩增子测序等方法广泛研究,但这种经典的分析方法仅关注了微生物类群的相对丰度,未能揭示微生物总量和个体微生物的绝对定量,加大了临床判断感染性疾病病原体的难度,阻碍了该项技术在临床感染性疾病以及突发和新发疫情中的应用。
本研究建立了一种新的16S rRNA扩增子文库构建方法(Q方法),该方法相较于常规的16S文库构建方法,具有可定量检测复杂环境微生物组的优点。采用Q方法建库,我们可以基于读序上的随机标签剔除建库过程中对不同序列的扩增偏好性,同时实验中设计了两种内参,可用于将测序结果中OTU数换算成实际加入的拷贝数。该方法有效地提高了肠道菌群结构检测的精准性,降低了实验操作对结果的影响。同时由于将常规的16S测序结果中的相对丰度百分比结果转化成为拷贝数结果,基于Q方法建库的16S rRNA扩增子测序结果与数字PCR、定量PCR等分子生物学方法结果更具有横向可比性,有利于未来技术的进一步研发和改进。
本研究加入的内参是人工合成的spike-in质粒,侧翼是细菌16S基因V3–V4区段的保守区序列,中间是与待扩增片段等长、且与目前已测序物种均无相似性匹配的随机序列。这与最近Guo等构建的侧翼是真菌ITS和细菌16S rRNA基因保守区串联、中间插入宿主特异基因区段的spike-in质粒是一致的[17]。这种设计能够排除微生物组群内基因的干扰,可通过qPCR对内参基因进行绝对定量,标准化内参拷贝数。本研究设计的内参质粒具有普适性,可用于人体、动物、环境等微生物群落的定量检测研究中。
PCR扩增和测序阶段均会引入碱基错配,进而导致错误的碱基识别,形成背景噪音。已有研究探索了多种方法降低和矫正这一过程引入的错误。PCR扩增过程中引入的错配可通过使用尿嘧啶- DNA糖基化酶(Uracil-DNA glycocasylase,UDG)和甲酰嘧啶-DNA-糖基化酶(Formamidopyrimidine- DNA glycosylase,Fpg)修复损伤的DNA模板,使用高保真酶扩增有效降低碱基错配率[18]。测序过程中引入的碱基识别错误可以通过以下几种方式降低和矫正:(1)滚环测序,将目标区段采用滚环复制的方式串联成多个拷贝的分子序列,根据目标区段多次测序提高覆盖度来自我纠错[18]。(2)读序配对矫正,去除左右测序序列不一致的读序[19-20]。(3)分子标签方式,在最原始的DNA模板分子上加上一段随机序列,通过分子标签归类纠错,生成一致性序列[19, 21]。本研究中采用的是分子标签的方式,发现所得测序序列中有平均15.6%序列存在变异位点,通过分子标签分组纠错方法可排除文库构建和测序阶段引入的测序错误,还原了样本中起始DNA模板的真实序列,提高了后续物种识别的精确度。本研究目前是基于16S V3–V4区进行的,该区域是16S宏基因组测序最常采用的扩增区域之一。除此之外,还有16S V1–V3区、V4–V5区等不同扩增区域用于16S宏基因组研究。原则上,只需在本研究基础上替换引物中扩增序列区域即可实现在以上区域中的应用。同时,该建库方法理论上也可应用于真菌ITS扩增。本课题组也在开展相应实验验证基于以上扩增区域进行微生物群落定量检测的可行性。
相较于常规16S建库方法,Q方法拥有可定量且准确度较高的优点,但是其建库成本和操作时间相较于常规16S测序较高,因此未来实际应用过程仍有待进一步的优化和改进。在后续分析方面,目前尚未考虑到不同菌种16S的拷贝数的差异对定量结果的影响,后续仍需进一步优化。在后续的工作中,课题组也计划结合数字PCR和RT-PCR的方法对Q方法的结果进一步评估和优化。
| [1] |
Maidak BL, Cole JR, Lilburn TG, et al. The RDP-Ⅱ (Ribosomal Database Project). Nucleic Acids Res, 2001, 29(1): 173-174. |
| [2] |
Pruesse E, Quast C, Knittel K, et al. SILVA: a comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB. Nucleic Acids Res, 2007, 35(21): 7188-7196. |
| [3] |
DeSantis TZ, Hugenholtz P, Larsen N, et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Appl Environ Microbiol, 2006, 72(7): 5069-5072. |
| [4] |
Integrative HMPRNC. The Integrative Human Microbiome Project: dynamic analysis of microbiome-host omics profiles during periods of human health and disease. Cell Host Microbe, 2014, 16(3): 276-289. |
| [5] |
Lloyd-Price J, Mahurkar A, Rahnavard G, et al. Strains, functions and dynamics in the expanded Human Microbiome Project. Nature, 2017, 550(7674): 61-66. |
| [6] |
Zhang W, Li J, Lu S, et al. Gut microbiota community characteristics and disease-related microorganism pattern in a population of healthy Chinese people. Sci Rep, 2019, 9(1): 1594. |
| [7] |
Miao J, Han N, Qiang Y, et al. 16SPIP: a comprehensive analysis pipeline for rapid pathogen detection in clinical samples based on 16S metagenomic sequencing. BMC Bioinformatics, 2017, 18(Suppl 16): 255-259. |
| [8] |
Muinck EJ de, Trosvik P, Gilfillan GD, et al. A novel ultra high-throughput 16S rRNA gene amplicon sequencing library preparation method for the Illumina HiSeq platform. Microbiome, 2017, 5(1): 68. |
| [9] |
Claesson MJ, Wang Q, O'Sullivan O, et al. Comparison of two next-generation sequencing technologies for resolving highly complex microbiota composition using tandem variable 16S rRNA gene regions. Nucleic Acids Res, 2010, 38(22): e200. |
| [10] |
Philippot L, Bru D, Saby NP, et al. Spatial patterns of bacterial taxa in nature reflect ecological traits of deep branches of the 16S rRNA bacterial tree. Environ Microbiol, 2009, 11(12): 3096-3104. |
| [11] |
Tkacz A, Hortala M, Poole PS. Absolute quantitation of microbiota abundance in environmental samples. Microbiome, 2018, 6(1): 110. |
| [12] |
Stammler F, Glasner J, Hiergeist A, et al. Adjusting microbiome profiles for differences in microbial load by spike-in bacteria. Microbiome, 2016, 4(1): 28. |
| [13] |
Vandeputte D, Kathagen G, D'Hoe K, et al. Quantitative microbiome profiling links gut community variation to microbial load. Nature, 2017, 551(7681): 507-511. |
| [14] |
Props R, Kerckhof FM, Rubbens P, et al. Absolute quantification of microbial taxon abundances. ISME J, 2017, 11(2): 584-587. |
| [15] |
Vieira-Silva S, Sabino J, Valles-Colomer M, et al. Quantitative microbiome profiling disentangles inflammation- and bile duct obstruction-associated microbiota alterations across PSC/IBD diagnoses. Nat Microbiol, 2019, 4(11): 1826-1831. |
| [16] |
Jing G, Sun Z, Wang H, et al. Parallel-META 3: Comprehensive taxonomical and functional analysis platform for efficient comparison of microbial communities. Sci Rep, 2017, 7: 40371. |
| [17] |
Guo X, Zhang X, Qin Y, et al. Host-associated quantitative abundance profiling reveals the microbial load variation of root microbiome. Plant Communications, 2020, 1(1): 100003. |
| [18] |
Lou DI, Hussmann JA, McBee RM, et al. High-throughput DNA sequencing errors are reduced by orders of magnitude using circle sequencing. Proc Natl Acad Sci USA, 2013, 110(49): 19872-19877. |
| [19] |
Zhang TH, Wu NC, Sun R. A benchmark study on error-correction by read-pairing and tag-clustering in amplicon-based deep sequencing. BMC Genomics, 2016, 17: 108. |
| [20] |
Pan L, Shah AN, Phelps IG, et al. Rapid identification and recovery of ENU-induced mutations with next-generation sequencing and Paired-End Low-Error analysis. BMC Genomics, 2015, 16: 83. |
| [21] |
Jabara CB, Jones CD, Roach J, et al. Accurate sampling and deep sequencing of the HIV-1 protease gene using a Primer ID. Proc Natl Acad Sci USA, 2011, 108(50): 20166-20171. |
 2020, Vol. 36
2020, Vol. 36