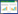中国科学院微生物研究所、中国微生物学会主办
文章信息
- 杨雪, 吴边, 柳陈坚, 董勇宏, 曾学琴, 李晓然
- Yang Xue, Wu Bian, Liu Chenjian, Dong Yonghong, Zeng Xueqin, Li Xiaoran
- 基于不同通用引物比较分析肠道微生物群落变化
- Comparison of intestinal microbial community succession based on different universal primer sets
- 生物工程学报, 2020, 36(12): 2556-2565
- Chinese Journal of Biotechnology, 2020, 36(12): 2556-2565
- 10.13345/j.cjb.200330
-
文章历史
- Received: June 7, 2020
- Accepted: November 16, 2020
- Published: November 25, 2020
2. 云南省第一人民医院 普通外科,云南 昆明 650032;
3. 昆明理工大学附属医院 普通外科,云南 昆明 650032
2. Department of General Surgery, the First People's Hospital of Yunnan Province, Kunming 650032, Yunnan, China;
3. Department of General Surgery, the Affiliated Hospital of Kunming University of Science and Technology, Kunming 650032, Yunnan, China
肠道微生物是生活在消化道中复杂多样的微生物群体,与身体其他部位相比,肠道微生物不论是在种类还是数量上都是最多的[1]。肠道菌群可以和多种免疫因素共同作用,协调肠道内平衡[2]。研究表明,肠道菌群与多种疾病发生的关联性已经被确认,肠道菌群可以调控免疫系统,与细菌相关的结直肠癌中,从原发灶到转移灶都有细菌的共存,小鼠粪便抑制了炎症菌群及个别菌中的免疫调节特性,菌群成为新的肿瘤预后性生物标志物和治疗靶点[3];肠道菌群与免疫性疾病的关联性已经被确认,肠道菌群的多样性降低使菌群群落功能丧失、免疫失衡和菌群定植位置异常,最终导致疾病易感性增加[4-5];对比2型糖尿病和健康个体的肠道微生物发现,2型糖尿病患者的肠道噬菌体显著增多[6]。
肠道中繁多的微生物大多数种类不能进行体外培养,随着分子生物学技术的发展,运用高通量测序研究人类肠道微生物群落,已经是非常普遍的研究方法,然而,目前大多研究仅关注肠道微生物中的细菌,多使用V3-V4区的细菌16S rRNA基因引物进行扩增和分析。古菌在人类肠道微生物中的丰度较低[7],一般为产甲烷古菌[8],是严格厌氧菌,能够将无机或有机物厌氧发酵转化成甲烷和CO2。研究表明,产甲烷古菌多与不健康的胃肠道有关,主要是慢性便秘和便秘为主的肠易激综合征[9],甚至与结直肠癌相关[10],与肥胖等代谢性疾病也有相关性[11]。因此,对于肠道微生物的研究,关注其中的古菌变化也具有重要的意义。
细菌和古菌都具有16S rRNA基因,且具有较高的同源性和保守性,在以往的研究中,有研究者设计了一些针对细菌和古菌的通用引物,但是并未见报道将这类引物直接用于肠道微生物分析。本研究通过分析文献报道过的通用引物,选择了一对可以同时扩增细菌以及产甲烷古菌,且长度适合二代高通量测序的引物。本研究比较了这一对引物(AB引物)[12-13]与常用的只能扩增细菌的引物(B引物)在分析肠道微生物群落结构变化的能力,通过几个常用rRNA基因序列数据库的结果,全面评估AB引物在分析肠道微生物群落变化的可行性和优势。
1 材料与方法 1.1 样品采集和DNA提取从健康人群中选取样品,入选标准为:(1)身体质量指数(BMI,Body mass index)在正常范围内(18.5–23.9);(2)半年内没有服用过抗生素;(3)取样前1个月内没有服用益生菌制品;(4)没有严重基础疾病,取样期间未患病。志愿者在实验过程中连续服用1周混合乳酸菌发酵乳(乳酸乳球菌、干酪乳杆菌和植物乳杆菌)[14],采集服用前后粪便样品。所有样品置于无菌保藏管后,于−20 ℃冰箱保存。使用QIAamp Fast DNA Stool Mini Kit试剂盒(QIAGEN,德国)提取DNA。操作步骤按照试剂盒说明书进行,获得DNA溶液,并保存在−20 ℃冰箱中备用。
1.2 PCR扩增和测序以提取的样品DNA为模板进行PCR扩增,由于单一使用古菌和细菌引物成本较高且难以定量分析,所以引物分别使用细菌16S rDNA基因通用引物(B引物) 343F (5′-TAC GGR AGG CAG CAG-3′)和798R (5′-AGG GTA TCT AAT CCT-3′)与细菌和古菌通用引物(AB引物) 515F (5′-GTG CCA GCM GCC GCG GTA A-3′)和909R (5′-TTT CAG YCT TGC GRC CGT AC-3′),在引物的5′端加入Illumina所需要的接头,30个循环之后将PCR产物稀释10倍后作为模板,按照之前的条件再做5个循环,以此减少扩增过程中非特异性扩增异源双链DNA[15]。PCR扩增使用Premix Ex TaqTM (TaKaRa,大连),在50 μL的反应体系中,加入10 ng模板,引物4 μmol/L,按照试剂盒说明书加入其他试剂。PCR退火温度为55 ℃,4 ℃保存PCR产物。PCR产物使用UltraClean PCR Clean-up kit (MOBIO,美国)进行纯化,所有纯化后的PCR产物按照等量混匀,使用Illumina Miseq测序(Illumina,美国)。
1.3 测序结果分类鉴定测序后将序列分配到其所属样品中,并去掉引物序列,获得有效序列文件。然后对有效文件进行处理,除去测序质量不好和长度小于500 bp的序列。对样品序列使用Mothur等[16]的classify.seqs命令,分别参照Silva (https://www.arb-silva.de/)、Greengenes (http://greengenes.secondgenome.com/)和RDP (http://rdp.cme.msu.edu/)数据库获得样品的分类信息。
1.4 多样性指数和系统发育分析使用Mothur Version 1.39.5来完成多样性指数的分析,以97%的序列相似度来划分可操作分类单元(Operational taxonomic units,OTU),计算样品的Chao1[17]、ACE[18-19]、Shannon[20]和Simpson[21]指数,并根据所得到rarefaction文件绘制rarefaction曲线。确定用于构建系统发育树的OTU数目,然后从每个OTU中选取一条代表性序列,将其与NCBI的GenBank中的序列进行Blast比对,然后将结果中具有确定分类信息的序列用Mega v5.0进行比对,将比对结果用Neighbour-joining方法构建系统发育树。
1.5 样品间的聚类分析使用Mothur Version 1.39.5对所有样品序列进行群落相似性分析,按照非加权组平均法(Unweighted pair-group method with arithmetic means,UPGMA)构建样品间的系统发育树,利用树枝结构描述和比较同一样品不同引物间的相似性和差异关系;利用韦恩(Venn)图显示同一样品不同引物间共有和特有的OTU,直观地表现出样品中OTU的重叠情况。
1.6 国际基因库接受号该论文所涉及到的基因序列已提交到NCBI中的SRA数据库,国际基因库接受号:PRJNA464415。
2 结果与分析 2.1 基于不同引物分析肠道微生物群落结构的差异本研究共选取4个志愿者入组实验。经过对原始序列进行质量和长度的筛选后,8个粪便样品分别用两种不同引物进行扩增后共获得37万余条有效序列,样品中OTU数目、Chao1和ACE分别按照序列相似度为100%和97%进行分析(表 1),发现除了4号样品外,其他样品的第2次取样微生物丰度均大于第一次取样的微生物丰度,说明益生菌的摄入确实会改善肠道微生物群落。
| Sample name* | Sequence number | OTU | Chao index | ACE index | Coverage |
| H_11_B | 23 250 | 1 194 | 2 868 | 5 168 | 97.48 |
| H_12_B | 29 516 | 1 647 | 3 882 | 6 669 | 97.11 |
| H_21_B | 11 878 | 1 275 | 3 624 | 7 472 | 96.56 |
| H_22_B | 19 615 | 1 397 | 3 169 | 4 616 | 96.87 |
| H_31_B | 23 690 | 959 | 2 412 | 3 532 | 98.10 |
| H_32_B | 34 115 | 1 154 | 2 819 | 4 370 | 98.28 |
| H_41_B | 18 595 | 1 001 | 2 483 | 3 792 | 97.59 |
| H_42_B | 25 139 | 906 | 2 462 | 4 156 | 98.17 |
| H_11_AB | 14 757 | 881 | 1 852 | 2 854 | 97.81 |
| H_12_AB | 32 380 | 1 661 | 3 284 | 4 424 | 97.78 |
| H_21_AB | 12 885 | 1 333 | 3 158 | 5 467 | 96.04 |
| H_22_AB | 23 897 | 1 472 | 3 099 | 4 483 | 97.38 |
| H_31_AB | 23 948 | 674 | 1 781 | 2 971 | 98.67 |
| H_32_AB | 37 056 | 551 | 911 | 1 110 | 99.47 |
| H_41_AB | 25 596 | 692 | 1 377 | 1 980 | 98.90 |
| H_42_AB | 15 520 | 456 | 840 | 1 112 | 99.07 |
| *Sample naming rules: H: healthy volunteers; 1–4: sample No. 1–4; 1 and 2: before and after human interference; B: amplification sequencing results of B primers that can only amplify bacteria; AB: amplification sequencing results of AB primers that can amplify both bacteria and archaea. |
|||||
将本研究中B引物和AB引物扩增的序列与Silva数据库进行微生物群落结构分析,结果(图 1)显示,在门的水平上AB引物中含有B引物中没有的广古菌门Euryarchaeota。其他门也有一定的差异,这是引物不同所引起的差异,其中拟杆菌门Bacteroidetes和厚壁菌门Firmicutes在两种引物所得序列中所占比例均较高,而且可以看出AB引物所得序列与B引物所得序列除古菌外差异较小。在属的分类水平上,瘤胃球菌属Ruminococcus、拟杆菌属Bacteroides和粪杆菌属Faecalibacterium为主要优势菌属,而且从属的水平可以看出AB引物中Ruminococcus和萨特氏菌属Sutterella的丰度明显高于B引物,在B引物中丰度最高的是Bacteroides,而其他菌属丰度都相对较低。由此可以看出AB引物对样品中的微生物群落能进行更全面的分析。
|
| 图 1 样品中微生物参照Silva数据库在不同分类水平的分布 Fig. 1 Distribution of major phyla and genera in the different samples. (A) The distribution of AB primer set amplification at phylum level. (B) The distribution of B primer set amplification at phylum level. (C) The distribution of AB primer set amplification at genus level. (D) The distribution of B primer set amplification at genus level. |
| |
从样品中的古菌分布情况(图 2)可以看出,除4号样品和3号样品取样后的样品没有古菌外,其他样品中都有微量的古菌,1号样品的第2次取样中的古菌丰度为0.083%,其中89.2%是甲烷短杆菌属Methanobrevibacter。1号样品第1次取样和2号样品第2次取样中,检测到的主要古菌是热杆菌属Methanothermobacter。

|
| 图 2 样品中古菌的分布情况 Fig. 2 Distribution of Archaea in the samples. |
| |
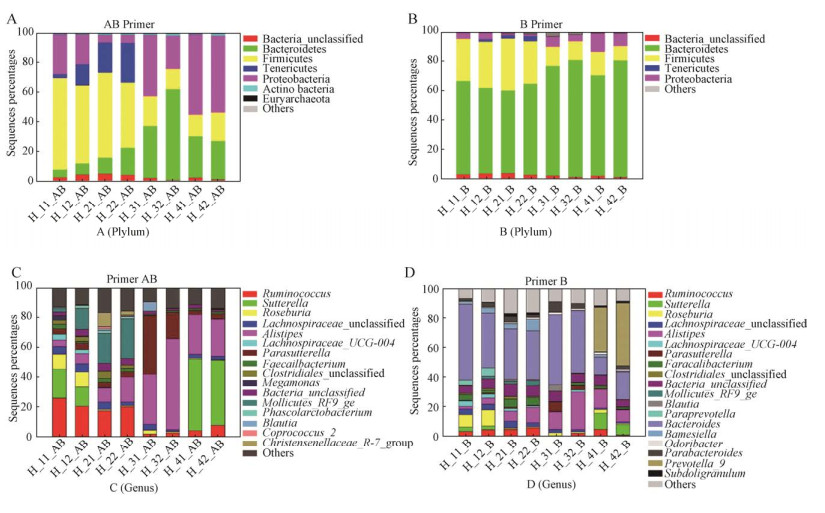
通过分析服用益生菌前后样品共享的OTU数目可以看出,虽然短时间的益生菌干扰并不会对健康人群的肠道微生物产生较大的影响,但是肠道微生物仍然出现了一定的变化(图 3),当然,在没有严格限定饮食的前提下,也不排除这些变化是由于饮食变化引起的。
|
| 图 3 不同样品的共有OTU数目 Fig. 3 Shared OTU numbers in different samples. (A) Volunteers 1 and 2. (B) Volunteers 3 and 4. |
| |
通过RDP的Probe Match进行in silico 分析,B引物和AB引物都能够与大部分的细菌16S rRNA基因序列配对,且B引物只能扩增出微量的产甲烷古菌,AB引物能够扩增超过80%的产甲烷古菌(表 2)。
| Primer | Bacteria | Euryarchaeota |
| 343F | 78.8% | < 0.01% |
| 798R | 65.1% | < 0.01% |
| 515F | 83.0% | 82.6% |
| 909R | 58.5% | 84.5% |
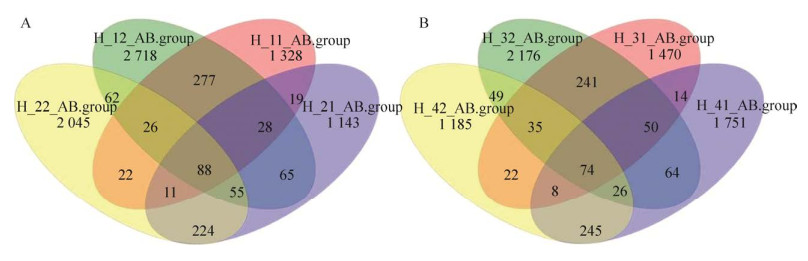
常用的rRNA序列数据库主要有Silva、Greengenes和RDP,为了全面分析AB引物的覆盖度,将AB引物的测序结果分别以3个数据库为参考序列进行了分析比较(图 4)。在门的水平,RDP数据库中Firmicutes与Bacteroidetes的比例相对较高,相应的变形菌门Proteobacteria比例相对较低,Sliva和Greengenes之间基本没有区别。在属的水平上,在RDP数据库中另枝菌属Alistipes比例属于相对较高的,Silva和Greengenes在属的水平上并没有明显区别,这一结果差异主要是由于数据库中的序列组成造成的,在以后的分析中,可以优先选择Silva和Greengenes作为参考数据库。
|
| 图 4 AB引物扩增序列分别以Silva、RDP和Greengenes为数据库在门和属分类水平的分布 Fig. 4 Reference Silva, RDP and Greengene database distribution of major phylum and genus in the different samples. (A) The Silva database consists of species at phylum level. (B) The Silva database consists of species at genus level. (C) The RDP database consists of species at phylum level. (D) The RDP database consists of species at genus level. (E) The Greengenes database consists of species at phylum level. (F) The Greengenes database consists of species at genus level. |
| |
对同一样品的不同引物获得OTU进行聚类分析(图 5),结果显示虽然每个样品不同引物的相同OTU只占总OTU的一小部分,但是在H_11样品中,AB引物和B引物的相同OTU个数为328,只占总OTU的14.1%,而序列相同占总序列数的82%,同样的,H_12、H_21、H_22、H_31、H_32、H_41和H_42中两种引物的相同OTU分别为516、362、405、236、268、277和214,占总OTU的比例基本为15%左右,占总序列数的比例均高于80%,且样品H_32的序列相似比例达到90.9%,因此说明两种引物都能够扩增出样品的大多数丰度较高的微生物,在需要扩增古菌的条件下,是可以单独选用AB引物的。
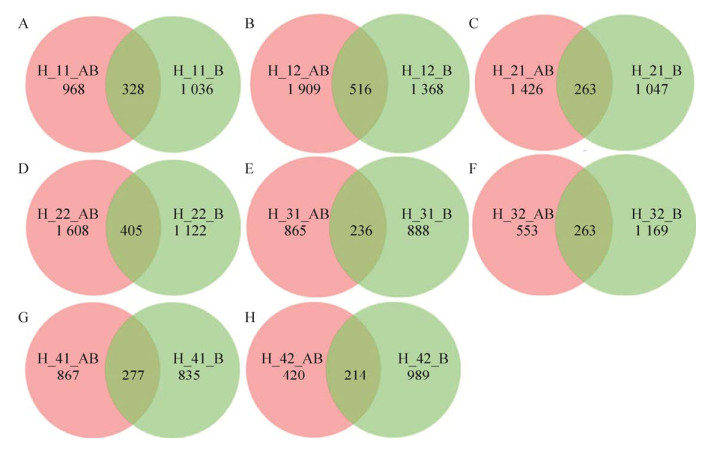
|
| 图 5 同一样品不同引物的OTU聚类 Fig. 5 OTU clustering of different primers in the same samples. (A) Volunteer 1 before probiotics intake. (B) Voluntee r 1 after probiotics intake. (C) Volunteer 2 before probiotics intake. (D) Volunteer 2 after probiotics intake. (E) Volunteer 3 before probiotics intake. (F) Volunteer 3 after probiotics intake. (G) Volunteer 4 before probiotics intake. (H) Volunteer 4 after probiotics intake. |
| |
本文证实了一对可以同时扩增细菌和古菌的16S rRNA基因通用引物,通过志愿者的肠道微生物群落结构变化展示了这对引物非常适宜用于人体肠道微生物分析,既可以展示细菌群落的前后变化,又能够同时展示肠道中产甲烷古菌的情况,已有研究表明肠道中古菌基本为产甲烷古菌[24],该引物可以通过一次扩增和高通量测序同时分析肠道样品中的细菌和古菌群落结构变化,既经济又快速,也可以对细菌和古菌的比例作大致比较。
通过这对引物扩增和测序得到的细菌群落与以往研究差异不大。谢尚奎等[25]的研究曾发现便秘大鼠和正常大鼠的粪便中总细菌和Bacteroidetes及Firmicutes等肠道优势菌群没有明显变化,这与上述结果类似。在属的分类水平上,Ruminococcus、Bacteroides和Faecalibacterium为主要优势菌属,Bacteroides是哺乳动物胃肠道微生物主要组成之一,可以利用单糖,但其能量主要来源于宿主和植物多糖[26];Faecalibacterium是健康成年人肠道最常见的细菌之一,能够改善人的免疫系统[27];Faecalibacterium已知菌种中丰度最高的是普拉梭菌Faecalibacterium prausnitzii[28],它是人肠道重要的共生菌,研究表明肠道中Faecalibacterium prausnitzii比例与肥胖哮喘、抑郁症等相关[29-30]。另外,在1号样品中,Sutterella的比例达到10% (图 1C),研究发现其与儿童自闭症有密切的联系,自闭症患者肠道中产碱菌属水平比正常儿童高,主要是由于Sutterella水平较高[31]。
产甲烷古菌是古菌中非常重要的一种,也是动物肠道中的主要类群。通过Probe Match比对显示AB引物能够扩增超过80%的产甲烷古菌,在本研究检测到的古菌中,Methanobrevibacter是严格厌氧菌,大多数生长在大型动物肠道中,会产生大量的温室气体,在人体肠道中也有发现,是与人肥胖相关的肠道微生物[32],Methanothermobacter可以利用CO2和氢气产生甲烷[33]。
随着微生物分子生态学和微生物分类学的发展,对人体内微生物的研究也在不断深入,通过高通量测序技术和微生物分类学的方法对人体内微生物进行分析,能够更加全面地了解人体内的微生物群落结构,合理的引物选择有利于发现肠道中的稀有菌群,帮助人们认识肠道微生物的丰富性和多样性,为人类肠道菌群研究提供基础。
| [1] |
Quigley EMM. Gut bacteria in health and disease. Gastroenterol Hepatol (N Y), 2013, 9(9): 560-569. |
| [2] |
Zhou L, Sonnenberg GF. Essential immunologic orchestrators of intestinal homeostasis. Sci Immunol, 2018, 3(20): eaao1605. |
| [3] |
Kroemer G, Zitvogel L. Cancer immunotherapy in 2017: The breakthrough of the microbiota. Nat Rev Immunol, 2018, 18(2): 87-88. |
| [4] |
Round JL, Palm NW. Causal effects of the microbiota on immune-mediated diseases. Sci Immunol, 2018, 3(20): eaao1603. |
| [5] |
Bach JF. The hygiene hypothesis in autoimmunity: the role of pathogens and commensals. Nat Rev Immunol, 2017, 18(2): 105-120. |
| [6] |
Ma YF, You XY, Mai GQ, et al. A human gut phage catalog correlates the gut phageome with type 2 diabetes. Microbiome, 2018, 6: 24. |
| [7] |
Manichanh C, Reeder J, Gibert P, et al. Reshaping the gut microbiome with bacterial transplantation and antibiotic intake. Genome Res, 2010, 20(10): 1411-1419. |
| [8] |
Schulz K, Hunger S, Brown GG, et al. Methanogenic food web in the gut contents of methane-emitting earthworm Eudrilus eugeniae from Brazil. ISME J, 2015, 9(8): 1778-1792. |
| [9] |
Sahakian AB, Jee SR, Pimentel M. Methane and the gastrointestinal tract. Digest Dis Sci, 2010, 55(8): 2135-2143. |
| [10] |
Jang SI, Jahng JH, Lim HC, et al. M2049 Relationship between intestinal gas and the development of right colonic diverticula. Gastroenterology, 2010, 138: S-466. |
| [11] |
Mathur R, Amichai M, Chua KS, et al. Methane and hydrogen positivity on breath test is associated with greater body mass index and body fat. J Clin Endocrinol Metabo, 2013, 98(4): E698-E702. |
| [12] |
Reysenbach AL, Pace NR. Archaea: A Laboratory Manual—Thermophiles. New York: Cold Spring Harbour Laboratory Press, 1995, 16: 101-107. |
| [13] |
Brunk CF, Eis N. Quantitative measure of small subunit rRNA gene sequences of the Kingdom Korarchaeota. Appl Environ Microbiol, 1998, 64(12): 5064-5066. |
| [14] |
Liu CJ, Tang XD, Yu J, et al. Gut microbiota alterations from different Lactobacillus probiotic-fermented yoghurt treatments in slow-transit constipation. J Funct Foods, 2017, 38: 110-118. |
| [15] |
Thompson JR, Marcelino LA, Polz MF. Heteroduplexes in mixed-template amplifications: formation, consequence and elimination by 'reconditioning PCR'. Nucleic Acids Res, 2002, 30(9): 2083-2088. |
| [16] |
Ercolini D, Filippis F De, Storia A La, et al. "Remake" by high-throughput sequencing of the microbiota involved in the production of water buffalo mozzarella cheese. Appl Environ Microbiol, 2012, 78(22): 8142-8145. |
| [17] |
Nie ZQ, Zheng Y, Wang M, et al. Exploring microbial succession and diversity during solid-state fermentation of Tianjin duliu mature vinegar. Bioresour Technol, 2013, 148: 325-333. |
| [18] |
Ninane V, Mukandayambaje R, Berben G. Identification of lactic acid bacteria within the consortium of a kefir grain by sequencing 16S rDNA variable regions. J Aoac Int, 2007, 90(4): 1111-1117. |
| [19] |
Kim YS, Kim MC, Kwon SW, et al. Analyses of bacterial communities in meju, a korean traditional fermented soybean bricks, by cultivation-based and pyrosequencing methods. J Microbiol, 2011, 49(3): 340-348. |
| [20] |
Fernández E, Alegría A, Delgado S, et al. Comparative phenotypic and molecular genetic profiling of wild Lactococcus lactis subsp. Appl Environ Microbiol, 2011, 77(15): 5324-5335. |
| [21] |
Quintela-Baluja M, Böhme K, Fernández-No IC, et al. Characterization of different food-isolated Enterococcus strains by MALDI-TOF mass fingerprinting. Electrophoresis, 2013, 34(15): 2240-2250. |
| [22] |
Stackebrandt E, Ebers J. Taxonomic parameters revisited: the tarnished "gold standard". Microbiol Today, 2006, 33: 152-155. |
| [23] |
Cole JR, Wang Q, Cardenas E, et al. The Ribosomal Database Project: improved alignments and new tools for rRNA analysis. Nucleic Acids Res, 2009, 37(S1): D141-D145. |
| [24] |
Moissl-Eichinger C, Pausan M, Taffner J, et al. Archaea are interactive components of complex microbiomes. Trends Microbiol, 2018, 26(1): 70-85. |
| [25] |
Xie SK, Ren DL, Xian ZY, et al. Changes of intestinal flora and effects of corresponding intervention in colon slow transit constipation rats. Guangdong Med J, 2016, 37(3): 325-327 (in Chinese). 谢尚奎, 任东林, 鲜振宇, 等. 结肠慢传输型便秘大鼠肠道菌群的变化及益生菌的干预效果. 广东医学, 2016, 37(3): 325-327. |
| [26] |
Martens EC, Chiang HC, Gordon JI. Mucosal glycan foraging enhances fitness and transmission of a saccharolytic human gut bacterial symbiont. Cell Host Microbe, 2008, 4(5): 447-457. |
| [27] |
Miquel S, Martín R, Rossi O, et al. Faecalibacterium prausnitzii and human intestinal health. Curr Opin Microbiol, 2013, 16(3): 255-261. |
| [28] |
Duncan SH, Hold GL, Harmsen HJ, et al. Growth requirements and fermentation products of Fusobacterium prausnitzii, and a proposal to reclassify it as Faecalibacterium prausnitzii gen. Int J Syst Evol Microbiol, 2002, 52(Pt 6): 2141-2146. |
| [29] |
Newton RJ, McLellan SL, Dila DK, et al. Sewage reflects the microbiomes of human populations. mBio, 2015, 6(2): e02574-14. |
| [30] |
Jiang HY, Ling ZX, Zhang YH, et al. Altered fecal microbiota composition in patients with major depressive disorder. Brain Behav Immun, 2015, 48(4): 186-194. |
| [31] |
Williams BL, Hornig M, Parekh T, et al. Application of novel PCR-based methods for detection, quantitation, and phylogenetic characterization of Sutterella species in intestinal biopsy samples from children with autism and gastrointestinal disturbances. mBio, 2012, 3(1): e00261-11. |
| [32] |
Dighe AS, Jangid K, González JM, et al. Comparison of 16S rRNA gene sequences of genus Methanobrevibacter. BMC Microbiol, 2004, 4: 20. |
| [33] |
Dworkin M, Falkow S, Rosenberg E, et al. The Prokaryotes. 3rd ed. New York: Springer-Verlag, 2006.
|
 2020, Vol. 36
2020, Vol. 36