中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘东来, 张春涛, 王佑春, 许四宏
- Liu Donglai, Zhang Chuntao, Wang Youchun, Xu Sihong
- 病原宏基因组高通量测序技术质量控制与评价的挑战和思考
- Challenges and considerations on quality control and evaluation of pathogen metagenomic next-generation sequencing
- 生物工程学报, 2020, 36(12): 2598-2609
- Chinese Journal of Biotechnology, 2020, 36(12): 2598-2609
- 10.13345/j.cjb.200377
-
文章历史
- Received: June 24, 2020
- Accepted: September 22, 2020
感染性疾病作为全球十大死亡原因之一,时刻威胁着患者的生命健康,其根本原因在于现有的病原鉴定技术无法满足临床需求[1-5]。传统分子诊断技术通常只能检测非常有限的数个靶标,一定程度上限制了其临床应用[2, 5-8]。目前,60%–70%的中枢神经系统感染患者和40%–50%的脓毒症、重症肺炎患者的治疗缺乏病原学依据[9-11]。此外,近年来WHO和国家卫生健康委员会多次强调控制抗生素滥用的重要性,对临床病原诊断提出了更高的要求[12]。病原宏基因组高通量测序技术(Metagenomic next-generation sequencing,mNGS)通过对待测样本总核酸进行测序,理论上能“无偏倚”地检出全部潜在病原体,包括病毒、细菌、真菌和寄生虫等[13-14],尤其适用不明病因感染的临床诊断[15-19]。正是得益于mNGS技术,才能在新型冠状病毒疫情初期迅速发现并鉴定出这一新发病原体[20-21]。然而,由于该技术复杂程度高,更新迭代速度快,且临床疑难感染的诊断极具风险,因此对其转化应用的监管,即按照三类传染病体外诊断试剂进行质量评价和审评审批,面临巨大的技术和政策挑战[22]。
为了促进mNGS技术的普及和规范发展,国内外多家行业机构和政府监管部门均制定并发布了相关学术共识和技术指南[22-28]。近两年,已有多家国内外公司和科研机构公开发表该技术的分析性能验证和临床研究[16-18, 28-30]。例如,美国Karius公司于2019年发表了病原体游离核酸检测的相关研究结果[16];同年,美国加州大学旧金山分校发表了脑脊液中病原体核酸检测的相关研究结果[18, 28]。在我国,已有多家公司研发了适用于多种临床适应症的mNGS检测方法,同时该技术也被写入相关临床诊疗指南[25, 27]。然而,目前国内外仍没有任何基于mNGS技术的体外诊断试剂获得监管机构批准。
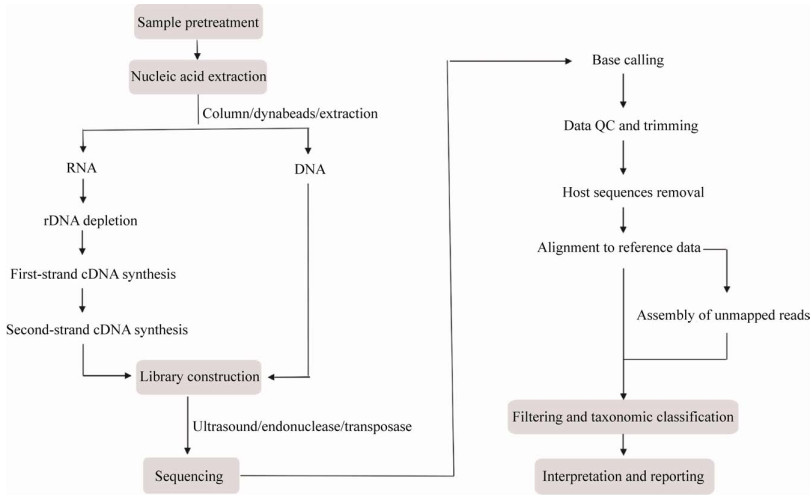
本文通过对mNGS技术原理、检测流程及关键环节(图 1)进行综述,着重阐述关于该技术质量控制与评价方法的思考,以期尽可能完整、科学和清晰地展现其技术特点,特别是其独特的、不同于传统单重或多重病原核酸检测试剂的质量控制与评价的策略和方法。本文所述的mNGS技术原理不包括靶向扩增或16S rRNA基因高通量测序。
|
| 图 1 mNGS技术流程及关键质量控制节点 Fig. 1 Metagenomic next-generation sequencing technical workflow and key quality control points. |
| |
mNGS技术以高通量测序为工具,将临床感染患者样本中的全部核酸片段化后进行测序,再应用生物信息学分析(以下简称“生信”)软件将测序结果与相关病原体数据库中的参考序列进行比对、分析和鉴定[2],如图 2所示。
与传统的病原体核酸诊断技术最大的不同在于,mNGS技术不需要提前针对一种或多种病原体的基因组进行特异性核酸扩增,因此,该技术原理上能够检测样本中的全部病原体[31]。然而,该技术的实际检测范围可能受多个技术因素影响,这些因素包括但不限于[13]:测序深度、不同病原体核酸的提取效率、随机引物逆转录的效率及偏倚,以及人源核酸去除不当导致的病原体丢失等;生信流程的准确性和特异性,以及数据库中病原体信息的全面性及其数据质量。
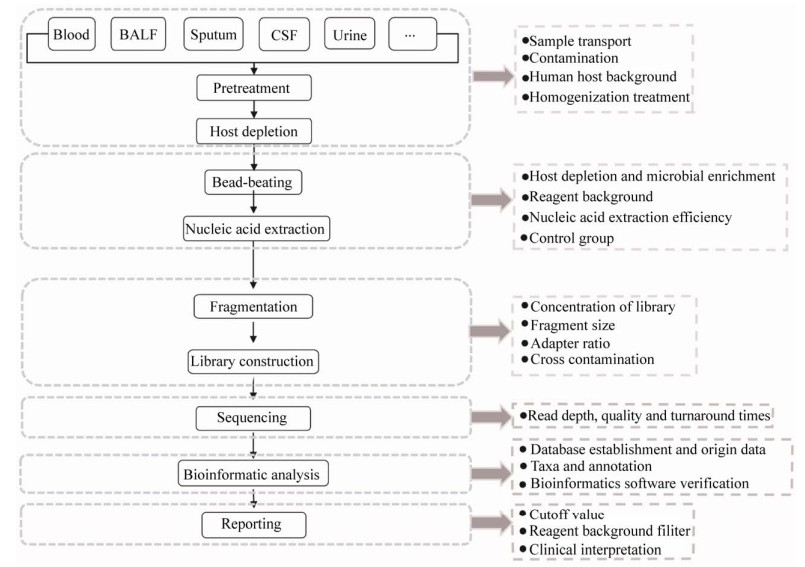
2 检测流程与挑战mNGS技术的检测周期一般约为24 h,分为湿实验(Wet lab或wet bench)和干实验(Dry lab或dry bench),流程大致分为6个步骤:样本前处理、核酸提取、文库构建、上机测序、生信分析和报告解读。针对多样化的临床样本,核酸提取前需进行不同的前处理,比如液化、破壁、去宿主等以提高病原体检出率[22];核酸提取和文库构建是对核酸进行纯化分离后,在片段化的核酸两端加上已知序列信息的接头以便于测序。单样本的文库构建完成后通常需要经历文库扩增,混合(Polling)后进行上机测序。测序完成后,下机数据进入生信流程,完成数据质量控制及微生物比对注释等步骤。最后,经报告解读完成报告的审核及发放。一般地,测序平台的测序长度为75–200 bp,少数仪器可达300–400 bp;测序数据量至少为10兆(Million,M)条核酸片段(Reads),如湿实验包含人源核酸去除,数据量低于10 M reads也可能满足设计要求,但需充分验证[32-33]。
2.1 湿实验在上述湿实验流程中,高通量测序技术应用于临床病原感染诊断面临着四大技术挑战[34-35]:
第一,如何克服样本中宿主核酸和样本病理特性的影响。mNGS技术是对样本的总核酸进行检测,所有的人体样本,包括组织、血液、脑脊液、痰液、肺泡灌洗液、关节腔积液以及粪便等,均可作为待测样本。临床样本中微生物的含量和基因组相对于宿主细胞来说占比低,样本测序数据中宿主基因组信息占比常高达90%–99%,影响检测的灵敏度。再者,感染患者标本多为病理性样本,各个样本的核酸总量、基因组大小、核酸质量以及人源核酸占比存在显著多样性,因而对mNGS技术的兼容性提出了更高的要求。比如,应用特异性探针杂交捕获或特异性酶降解等方法去除人核糖体或线粒体核酸,能提高病原体核酸的相对占比,有利于提高检测性能;但是,去除人源核酸的同时也降低了样本核酸总量,对建库技术的兼容性提出了更高的要求[36]。
第二,如何真正实现对病原微生物无偏倚地检测。由于细菌、真菌、病毒和寄生虫各种微生物特性不同,样本前处理和核酸提取的方法需要兼容不同微生物的特征。例如,增加玻璃珠研磨样本有利于提高难破壁细菌和真菌的核酸提取效率,但可能损失病毒核酸;对样本进行离心或过滤,有利于某一种类的病原体富集,但可能损失其他种类的病原体[34, 37]。核酸提取的方法,如DNA和RNA分别或者同时提取,可能影响检测灵敏度;核酸提取流程也需要适配不同的样本类型,如不同的血浆游离核酸提取试剂对病原体游离核酸的提取效率不同;应用机器自动化提取核酸有利于缩短实验时间、提高操作便易性,但可能降低核酸提取效率等等。样本前处理和核酸提取在方法优化时会进行“样本处理到结果报告”的全流程评价,当流程“固定”后,通常选择特定的核酸浓度和体积参数用以保证检测的整体稳定性。此外,在核酸提取前通常应加入内参或内部质控品对检测流程进行质量控制。
第三,如何针对不同种类的临床样本,包括血液、痰液、肺泡灌洗液、脓液、脑脊液、新鲜组织和组织切片等,选择合适的前处理程序,如痰液液化、组织研磨和切片脱蜡[35]。充分了解并根据不同样本的核酸特点设计样本前处理的具体方法,对于保证检测的可靠性尤为重要。例如,脑脊液样本核酸总量有时可能低于测序文库制备所需的最低核酸起始量,需要考虑对样本中的核酸进行富集,使用核酸回收率高的纯化试剂盒;血浆样本中人源和病原体核酸均以短片段游离形式存在,需要考虑使用可回收段片化核酸的纯化试剂盒;甲醛固定石蜡包埋处理会使组织样本中核酸发生断裂或降解,需要对原始样本进行脱蜡和DNA修复后再进行提取;肠道样本中病原体核酸占比远大于其他类型样本,在样本前处理时需要特别注意防护对其他样本的污染。
第四,如何克服检测背景的干扰。目前mNGS技术的检测流程中所用到的试剂,如核酸提取试剂中包含常见环境微生物,这些微生物统称“试剂背景菌”。在生信流程和报告解读过程中,应过滤试剂背景菌及这些背景微生物带来的潜在的分析干扰[38-39]。当然,尽可能的减少样本和环境来源的背景菌的存在和干扰也正是mNGS技术面临的挑战。
2.2 干实验生信流程是mNGS技术不可或缺的一部分。经过湿实验获得的原始测序数据,需要经过生信流程分析处理才能最终得到检测结果。mNGS技术的生信流程与其他NGS技术的设计截然不同,肿瘤或遗传缺陷相关基因检测的生信流程专门为检测人类生殖或体细胞基因突变设计,而mNGS技术的生信流程则在分析的最初阶段就将人类基因组数据全部过滤掉,然后从剩下的数据中挖掘病原体基因组信息[40-41]。这也意味着独特的技术性挑战,即其分析对象,不再是总长度约3 Gb的人类基因组,而是复杂度高一两个数量级的复杂微生物基因组。
mNGS技术的生信流程通常包括以下步骤[40]:原始测序数据预处理,如低质量及低复杂性数据过滤、接头和标签序列裁剪、标签分类等;人源数据过滤;经处理的数据与病原体数据库中参考序列进行比对分析;比对分析、统计及注释等;根据预设的结果判断阈值或阈条件,明确并清晰地完成结果报告和解释。其中,病原体数据库中参考序列的数量、种类及数据质量,决定了mNGS技术的理论检测范围,同时也是影响检测结果准确性的重要因素[42-43]。因此,应根据其技术特点和临床需要建立与之匹配的病原体数据库。一种方法是以开源的大型复杂型数据库,如美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)的GenBank基因数据库为基础,对其中序列信息进行人为调整,如过滤人源基因、质粒基因、区分病原体全基因组或部分基因组序列等。另一种方法是从头构建,汇总大量高质量病原体序列信息组成数据库。例如美国食品药品监督管理局(U.S. Food and Drug Administration,FDA)与多部门联合建立的适用于传染病NGS诊断试剂的开源数据库,即FDA参考级数据库(FDA database for reference-grade microbial sequences,FDA-ARGOS)[44]。对于纳入病原体数据库的序列,应建立明确的筛选标准并尽可能注明其基因分类特征,如属、种和亚种等,型和亚型等,以及标准株、流行株和分离株等。此外,对于某些罕见的、基因组不完整或难以区分基因型的病原体序列应予以特别注明。由于无论采用哪种构建策略均无法避免病原体数据库的偏倚性,因此对其进行充分验证并尽可能完整地了解其对检测结果的影响,对保证检测结果的可靠性尤为重要。特别需要注意的是,数据库的建设不是一次性的,而是需要不断补充和完善的,但每次“升级”均应重新进行验证。
参考基因组数据库是生信流程的基石,在搭建时要格外注重对参考基因组数据库的质量控制。基因组参考数据库主要包括宿主(人类)参考基因组数据库和微生物参考基因组数据库等。宿主(人类)参考基因组数据库主要用于人源宿主序列的过滤,由于人类参考基因组与部分微生物存在一些同源序列,以及部分公共数据库来源的部分微生物参考基因组可能污染了人类基因组序列,如果人源序列过滤不完全则可能导致一些病原体的假阳性检出。如人类参考基因组Hg38来源于欧美白种人,因此人源参考基因组也需要考虑人种基因组的差异,面向中国人群检测的生信流程应该需要包含黄种人的特异序列[45-46]。此外,对于RNA检测流程,由于人的基因存在内含子,信使RNA (Messenger RNA,mRNA)经过剪接后生成,因此人的参考基因组也需要考虑纳入mRNA参考序列。
微生物参考基因组数据用于比对和鉴定微生物序列,数据库的质量直接影响检测结果。在进行微生物基因组数据库构建时需要格外注意质量控制,需要细致检查每一个物种参考基因组的完整性(完成图、精细图、草图等)、质量(碱基质量值、覆盖率、深度等)以及可能存在的污染片段。由于质粒是可以在不同的物种间穿梭的,所以一个物种的参考基因组里面是否需要包含质粒序列,一定需要事先评估该物种的质粒序列是否有足够的物种特异性,否则不能轻易将质粒序列包含于物种的参考基因组。数据库还应该进行周期性的更新和维护,以尽可能地完善数据库的质量,但每次更新后需要重新进行性能评估,因为基因组的改变可能会导致检测结果的差异。
微生物鉴定和注释的算法是生信流程另一个极其重要的支柱,鉴定和注释的准确性也直接影响了最后的检测结果。比对软件或算法如何选择、比对结果如何注释都是需要质量控制的。目前已经有多款针对宏基因组测序物种鉴定的软件,但不同的软件性能表现不一[47]。分析时间的长短、计算资源的需求、鉴定结果的准确性等方面需要进行综合评估,选择最优的方案。如果采用了非开源的第三方流程,自主优化的空间会大大降低,在使用前应该充分评估检测能力的全面性,目前的生信流程在相近物种和病毒分型方面都存在一定的缺陷。在物种注释方面,如何划定比对阈值,以及对于一条序列多物种比对时,如何鉴定出真正的病原体是一个难点。在病毒分型方面,因为病毒进化速率较快,特别是RNA病毒不同株之间的差异都可能比较大,准确分型是对生信流程的考验。根据临床需求,病毒的分型有时不能只分到“种”的层级(几乎所有的第三方流程只能分到种的层级),还应考虑“型”甚至“亚型”的层级(如肠道病毒物种下面的71型和11型导致的临床症状有差异;此外分型在病毒的溯源和院内感染的发现和控制也有重要价值);甚至需要考虑到“株”的层级,比如“野生株”和“疫苗株”,在海关对发热患者检疫的时候可以区分是感染或者是刚打疫苗的缘故。但这些详细的层级在NCBI的taxonomy上是没有足够的分类信息的,需要人工进行整理加工。深层次的分型信息要求更多、更详细的基因组纳入到数据库中,可以避免因为“型/亚型/株”之间的差异过大导致的漏检,从而保证了生信流程分析的可靠性。
mNGS技术的生信流程还应该具备背景菌、定植菌、致病菌等不同类型的微生物的训练和注释能力,不同类型微生物的注释有助于更准确、更高效地对检测结果进行解读。但这些不同类型的病原体会受到湿实验条件的改变、不同样本类型等因素的影响,因此在湿实验环节如果有条件的改变,特别是试剂更换等,一定需要重新进行背景的训练[38-39]。不同的样本类型也需要分别进行背景菌和定植菌的训练,因为定植菌的致病性是相对的,例如口腔有大量特异的定植菌,一般不会引起疾病,但如果脑脊液或胸腹水等其他样本类型检测出这些病原体,就可能是严重的致病菌。此外,在数据库更新时也需要做背景菌的训练,因为数据库的改变可能会影响到微生物检出结果,背景菌的检出可能也会随着改变。
结果判断阈值或阈条件的设置和结果报告与解释的格式均没有统一标准或明确定。mNGS技术的检测结果报告格式一般分为正式报告和附录:正式报告中的病原体均为满足阳性检测结果判断阈值或阈条件的;附录中的病原体则是既不能确定检出,也不能明确排除。导致附录中的病原体的原因比较复杂,可能有:阈值或阈条件设置不合理;生信流程难以区分基因型相似的病原体;对试剂内源性、环境污染及定植或机会致病病原体的验证不充分等等[34, 38]。尽管目前mNGS技术的生信流程的质量控制没有标准方法,但为了保证检测的稳定性和准确性,当生信流程发生改变时,如调整分析流程顺序、升级软件算法、增加病原体数据库容或修改结果报告格式等,应制定相应验证方案和评价标准进行严格的版本控制,详细地记录生信流程的版本演化历史以及各版本之间的差异[24]。
3 质量控制与评价的思考任何体外诊断试剂在临床应用前均应对其性能指标进行严格的质量评价。上文详述了mNGS技术的湿实验和干实验两部分内容,且两者均可直接影响基于该技术的三类体外诊断试剂的可靠性和准确性,因此应进行优化和充分验证。目前已了解到的mNGS技术的转化应用多为定性检测,参考其他传染病体外诊断试剂的监管模式,其质量控制与评价的指标包括检测范围、准确性、特异性、最低检出限、精密度、干扰和稳定性等[24],且可以通过相应的标准品或参考品进行性能评价。目前,相关监管机构暂未发布病原宏基因组高通量测序技术适用的国家参考品,以下仅提出对其质量评价的思考和建议。
3.1 参考品配置对于mNGS技术的检测范围,理论上不会超过其生信流程中所使用的数据库中病原体的种类和数量,实际上还取决于湿实验和干实验各环节的优化程度,如核酸提取效率、RNA的逆转录效率、结果判断阈值或阈条件的合理性等因素。然而,严格验证理论检测范围的所有病原体不具有可操性,目前国际公认的方法是选择具有“代表性”的病原体用于验证,其“代表性”特征包括:不同病原体种类,如细菌、真菌、寄生虫、DNA病毒及RNA病毒;不同基因组长度、核酸GC含量;不同核酸提取难易程度与核酸稳定程度等[3]。当检测范围改变时,一般是病原体数据库发生调整,应重新进行验证。如果湿实验未发生改变,可以仅重新验证干实验,使用基于上述病原体测序数据,或FDA-ARGOS参考数据库中选择的完整干净的病原体核酸数据制备的数字参考品进行验证[44]。在检测范围验证的基础上,还应进一步根据具体临床诊断需求、临床意义及检测局限性等因素进一步缩小范围、验证并明确mNGS试剂的报告范围,严格区分试剂内源性、环境污染以及定植或机会致病病原体。在选择病原体验证报告范围时,其“代表性”还应满足具有明确临床诊断价值,以及基因相似的种或亚种、型或亚型等。
病原mNGS参考品是指感染性临床样本的模拟样品,其组成包括样本基质、宿主和目标病原微生物[48]。针对技术的预期用途,不同的临床样本,其中所含的基质,以及宿主和微生物的存在形式和种类不同,因此参考品的设置不同。例如,应用mNGS技术对血浆细胞游离DNA (Cell free DNA,cfDNA)进行检测,从而实现对血流感染相关病原体的检测,在进行质量控制和性能评价时,设置的参考品是以健康人捐献的血浆作为样本基质,以健康人群血浆中的cfDNA长度和含量的分布模拟了3个梯度的宿主基因组,以片段化的病原微生物基因组模拟病原在血浆中的状态,并选择基因组大小、GC含量、病原体种类和临床意义有代表性的物种作为模拟的病原体,同时考虑基因组相似性可能带来的假阳性[3, 48]。血液和尿液样本较容易收集,使用的参考品首选患者的临床样本,对于脑脊液、肺泡灌洗液、深部痰以及组织等可以使用分离培养的病原体和健康人的样本混合制备而成的模拟参考品,或者也可以选择分离培养的病原体和与临床样本特征相似的基质混合制备而成的模拟参考品,例如脑脊液参考品可选择人工脑脊液作为样本基质[49-50]。选择用于参考品的病原体时应满足上文的“代表性”,病原体浓度应与真实临床样本中的浓度相似。
对干实验流程性能的独立验证可以使用虚拟参考品进行,即使用数字模拟或真实的高通量测序数据,单独对病原宏基因组学流程的干实验部分进行测试[47, 51-52]。虚拟参考品可以通过模拟程序[51],灵活地使用调整的错误模型参数和质量概要文件以进行更全面的测试,但同时,虚拟参考品应与真实产生的数据相似,模拟数据集应包含与现实比例接近的宿主基因组、常见的病原微生物和定植菌群以及环境菌群的基因组,对于相似性较高的物种,应增加验证和分析工作。
3.2 性能指标的制定对于mNGS技术,具有非靶向性检测所有种类的病原体的技术特点和潜力,但对于其转化的应用于临床诊断的三类体外诊断试剂,仍需要按照现行的规定进行监管,需要有明确的预期用途和临床意义,且经过严格的分析性能验证、临床性能验证等审评审批。因此,申请注册审批的产品必须有明确的检测范围,定义可报告的生物谱,被确定为背景污染物或临床无关紧要的生物应加以描述,最大程度地保证临床检测的可靠性和可读性。
在一定的检测范围内,在分析性能和临床性能验证过程,使用已知微生物种类的标准参考品和其他已上市的或默认的金标准方法进行对比,对准确性进行验证,在最低检测限以上的阴阳性符合率应该达到一定的要求。例如,根据免疫层析、RT-PCR、PCR、细菌真菌培养以及16S rRNA和ITS测序等方法对mNGS检测脑脊液的结果进行验证,确定方法学准确性[18]。进行准确性验证时,首选来自感染患者的样本,这些样本包含待检测的微生物,但如果很难获得代表性样本,则可以阴性临床样本掺入微生物或者纯化后的核酸作为模拟样本。由于感染性标本病理性、多样性的特征,模拟样本和真实临床样本可能仍然存在差别。如果检测试剂对样本有特殊处理步骤,如去宿主或病原微生物富集等,需考虑其适用性[32, 36]。
最低检出限(Limit of detection,LOD)是指某个病原体在95%置信度下的最低可检出的浓度或滴度(或核酸浓度),通常可以通过重复检测一定浓度梯度的系列稀释样本进行验证。例如,实验室性能验证表明mNGS检测脑脊液样本中巨细胞病毒(CMV)、人免疫缺陷病毒(HIV)、无乳链球菌、肺炎克雷伯菌、黑曲霉、新型隐球菌和弓形虫的LOD分别是14 copies/mL、313 copies/mL、10 CFU/mL、8 CFU/mL、0.2 CFU/mL和81 reads/mL[18]。即当脑脊液样本中含有上述微生物的浓度为以上浓度时,mNGS有95%的概率可以检测到该微生物,也可以理解为对含检测限浓度样本独立进行20次实验,应该是少于等于1次未能检测到目标微生物。对于单个病原体,mNGS技术对病原体的检测性能取决于病原体的核酸总量在整个样本中的占比,即病原体与宿主核酸的比例。简单来讲,可以将临床样本中的人源核酸比作草垛、病原体核酸比作针。基于核酸扩增的分子诊断技术先特异性地屏蔽了草垛,仅需考虑针的数量即可;而mNGS技术则是“草垛寻针”,既要考虑草垛的数量,也要考虑针的数量,即草垛和针的比例。此外,微生物的多样性,一方面体现在基因组跨度较大,真菌的基因组可达几十Mb,病毒的基因组小至几kb,另一方面体现在微生物的细胞结构差异导致潜在的核酸提取效率差异,这些都可能对最低检测限产生影响。因此,对于不同的微生物,其最低检测限均需要经过独立的实验验证。
对于不同的临床样本,需要确定临床样本中宿主含量的分布,从而确定不同宿主含量条件下的最低检出限。由于实际检测过程中,无法对宿主含量进行定量,因此应考虑以不同宿主含量和95%置信度的最低可检测滴度为最低检测限,从两个维度,即人源核酸不变、病原体核酸梯度改变或反之,分别进行验证及评价。此外,还应充分评价干实验对最低检出限的影响。例如,当病原体的滴度较低时,仅能测到病原体基因组的部分核酸序列,进行序列比对时可能会错误地比对到与之基因组相似的病原体参考序列上,降低试剂最低检出限,同时增加假阳性和假阴性。
精密度通常是指在规定条件下相互独立的测试结果之间的一致程度,用偏差、标准偏差和变异系数(Coefficient of variation,CV)表示,可以通过对同一个样本多次重复检测进行验证及评价。例如,对于可定量方法学,通过批内和批间结果确定mNGS对血液中病原微生物的游离核酸分子定量结果的批内CV为16.7%–18.9%,批间CV为17.9%–22.2%[3]。对于定性方法学,制备微生物含量接近(高于) LOD的样本,称为精密度参考品。其批内和批间检测结果的阳性率应符合一定的要求[18],并可通过批内和批间微生物检测结果,如微生物reads数/M数据量(Reads per million,RPM),大概确定方法学的精密度。mNGS技术的湿实验周期长,操作步骤多,流程复杂,实验过程中的破壁、文库构建和上机测序等步骤均可能产生结果偏差,也是与其他方法学的差别之处;干实验流程相对固定,变量较少,因此不能通过重复对一个文库多次上机来评估变异系数,其精密度指标的制定也可能与其他定性方法不同。监管部门在制定精密度指标之前应该组织大规模的试剂质量评价联合研究,对mNGS技术特征,以及根据具体预期用途设计开发的试剂的性能进行综合评估,随着实验流程的优化和方法学的日趋成熟,精密度的指标也需要不断作出修改。此外,由于计算变异系数用到的质量控制参数可能并不会体现在常规的结果报告中,因此可能需要对生信流程进行特殊设计并进行验证及版本控制。
干扰的验证及评价方法与传统分子诊断技术既有相似之处又有不同之处。相似的是,mNGS技术可以根据样本类型的特点将已知的核酸提取或酶反应抑制物质,一般包括血红素、高浓度蛋白质和胆红素等,添加到病原体参考品中,验证是否可能产生假阴性结果。不同的是,能够干扰检测结果的因素还包括人源核酸,试剂内源性、环境污染及定植或机会致病病原体,数据库中病原体基因组信息广泛性、完整性及准确性等,上述因素对检测结果的干扰均应充分验证并建立质量评价标准。例如,内源性病原体的源头,可能是湿实验流程中使用的试剂或耗材携带的[39]。此外,高同源性物种之间的干扰应引起关注,物种间的基因组可能相似,但致病能力和临床意义可能完全不同。例如,脑膜炎奈瑟菌与延长奈瑟菌等奈瑟菌属物种的临床意义完全不同,脑膜炎奈瑟菌是引起流行性脑脊髓膜炎的病原体,高发于青少年和青壮年。因此在研发过程中需要明确内源性病原体以及对检测结果的干扰,并建立有效的原材料质量评价方法和标准。
4 总结对于不明病因感染的临床诊断,mNGS技术具有无法替代的优势,因此其应用转化的速度远超人们想象。现阶段,制约其发展的主要瓶颈,逐渐由技术本身的局限和挑战转变为相关质量控制与评价方法以及质量标准等的缺失。这些缺失不同程度地阻碍了mNGS技术转化为可受监管的三类体外诊断试剂,也限制了行业的发展。相比基于靶向扩增的传统分子诊断技术,mNGS技术具有完全不同的技术原理和更为复杂的检测流程,因此需要更严谨的质量控制方案来保证稳定的质量,以及更科学的质量评价方案来保证技术性能可以满足临床需求。然而,目前国内外仍然缺少系统的、详细的质量控制与评价的指南或质量标准。本文阐述了关于mNGS技术质量控制与评价方面的思考,总结了mNGS全技术流程中的关键质量控制节点,以期为行业从业者和监管部门提供详实准确的参考信息。
| [1] |
WHO. The top 10 causes of death, 2018, https://www.who.int/en/news-room/fact-sheets/detail/the-top-10-causes-of-death.
|
| [2] |
Chiu CY, Miller SA. Clinical metagenomics. Nat Rev Genet, 2019, 20(6): 341-355. |
| [3] |
Bharucha T, Oeser C, Balloux F, et al. STROBE-metagenomics: a STROBE extension statement to guide the reporting of metagenomics studies. Lancet Infect Dis, 2020. DOI:10.1016/S1473-3099(20)30199-7 |
| [4] |
Fenollar F, Raoult D. Molecular diagnosis of bloodstream infections caused by non-cultivable bacteria. Int J Antimicrob Agents, 2007, 30(S1): 7-15. |
| [5] |
Mancini N, Carletti S, Ghidoli N, et al. The era of molecular and other non-culture-based methods in diagnosis of sepsis. Clin Microbiol Rev, 2010, 23(1): 235-251. |
| [6] |
Ramanan P, Bryson AL, Binnicker MJ, et al. Syndromic panel-based testing in clinical microbiology. Clin Microbiol Rev, 2018, 31(1): e00024-17. |
| [7] |
Schreckenberger PC, McAdam AJ. Pointcounterpoint: Large multiplex PCR panels should be first-line tests for detection of respiratory and intestinal pathogens. J Clin Microbiol, 2015, 53(10): 3110-3115. |
| [8] |
Kothari A, Morgan M, Haake DA. Emerging technologies for rapid identification of bloodstream pathogens. Clin Infect Dis, 2014, 59(2): 272-278. |
| [9] |
Grumaz S, Grumaz C, Vainshtein Y, et al. Enhanced performance of next-generation sequencing diagnostics compared with standard of care microbiological diagnostics in patients suffering from septic shock. Crit Care Med, 2019, 47(5): e394-e402. |
| [10] |
Glaser CA, Gilliam S, Schnurr D, et al. In search of encephalitis etiologies: diagnostic challenges in the California Encephalitis Project, 1998-2000. Clin Infect Dis, 2003, 36(6): 731-742. |
| [11] |
Glaser CA, Honarmand S, Anderson LJ, et al. Beyond viruses: clinical profiles and etiologies associated with encephalitis. Clin Infect Dis, 2006, 43(12): 1565-1577. |
| [12] |
中华人民共和国国家卫生健康委员会.抗菌药物临床应用指导原则, 2015, http://www.gov.cn/xinwen/2015-08/27/content_2920799.htm.
|
| [13] |
Gu W, Miller S, Chiu CY. Clinical metagenomic next-generation sequencing for pathogen detection. Ann Rev Pathol Mech Dis, 2019, 14: 319-338. |
| [14] |
Chiu CY. Viral pathogen discovery. Curr Opin Microbiol, 2013, 16(4): 468-478. |
| [15] |
Li LH, Chen LD, Xiao B, et al. Application of metagenomic sequencing in detecting the pathogens of infectious diseases. Infect Dis Inf, 2018, 31(1): 15-18 (in Chinese). 李林海, 陈丽丹, 肖斌, 等. 宏基因组测序在感染性疾病病原体检测中的应用. 传染病信息, 2018, 31(1): 15-18. |
| [16] |
Blauwkamp TA, Thair S, Rosen MJ, et al. Analytical and clinical validation of a microbial cell-free DNA sequencing test for infectious disease. Nat Microbiol, 2019, 4(4): 663-674. |
| [17] |
Hong DK, Blauwkamp TA, Kertesz M, et al. Liquid biopsy for infectious diseases: sequencing of cell-free plasma to detect pathogen DNA in patients with invasive fungal disease. Diagn Microbiol Infect Dis, 2018, 92(3): 210-213. |
| [18] |
Miller S, Naccache SN, Samayoa E, et al. Laboratory validation of a clinical metagenomic sequencing assay for pathogen detection in cerebrospinal fluid. Genome Res, 2019, 29(5): 831-842. |
| [19] |
Miao Q, Ma YY, Wang QQ, et al. Microbiological diagnostic performance of metagenomic next-generation sequencing when applied to clinical practice. Clin Infect Dis, 2018, 67(S2): S231-S240. |
| [20] |
Ren LL, Wang YM, Wu ZQ, et al. Identification of a novel coronavirus causing severe pneumonia in human: a descriptive study. Chin Med J (Engl), 2020, 133(9): 1015-1024. |
| [21] |
Zhu N, Zhang DY, Wang WL, et al. A novel coronavirus from patients with pneumonia in China, 2019. N Engl J Med, 2020, 382(8): 727-733. |
| [22] |
Gargis AS, Kalman L, Lubin IM. Assuring the quality of next-generation sequencing in clinical microbiology and public health laboratories. J Clin Microbiol, 2016, 54(12): 2857-2865. |
| [23] |
Food and Drug Administration. Infectious disease next generation sequencing based diagnostic devices: microbial identification and detection of antimicrobial resistance and virulence markers; draft guidance for industry and Food and Drug Administration staff, 2016. https://www.fda.gov/regulatory-information/search-fda-guidance-documents/infectious-disease-next-generation-sequencing-based-diagnostic-devices-microbial-identification-and.
|
| [24] |
Schlaberg R, Chiu CY, Miller S, et al. Validation of metagenomic next-generation sequencing tests for universal pathogen detection. Arch Pathol Lab Med, 2017, 141(6): 776-786. |
| [25] |
宏基因组分析和诊断技术在急危重症感染应用专家共识组. 宏基因组分析和诊断技术在急危重症感染应用的专家共识. 中华急诊医学杂志, 2019, 28(2): 151-155. |
| [26] |
中国食品药品检定研究院.第二代测序技术检测试剂质量评价通用技术指导原则, 2016, https://www.nifdc.org.cn/nifdc/xshjl/8495.html.
|
| [27] |
中华医学会呼吸病学分会感染学组. 中国成人医院获得性肺炎与呼吸机相关性肺炎诊断和治疗指南(2018年版). 中华结核和呼吸杂志, 2018, 41(4): 255-280. |
| [28] |
Wilson MR, Sample HA, Zorn KC, et al. Clinical metagenomic sequencing for diagnosis of meningitis and encephalitis. N Engl J Med, 2019, 380(24): 2327-2340. |
| [29] |
Langelier C, Kalantar KL, Moazed F, et al. Integrating host response and unbiased microbe detection for lower respiratory tract infection diagnosis in critically ill adults. Proc Natl Acad Sci USA, 2018, 115(52): E12353-E12362. |
| [30] |
Sichtig H, Minogue T, Yan Y, et al. FDA-ARGOS is a database with public quality-controlled reference genomes for diagnostic use and regulatory science. Nat Commun, 2019, 10(1): 3313. |
| [31] |
Doan T, Wilson MR, Crawford ED. Erratum to: Illuminating uveitis: metagenomic deep sequencing identifies common and rare pathogens. Genome Med, 2016, 8: 123. |
| [32] |
Nelson MT, Pope CE, Marsh RL, et al. Human and extracellular DNA depletion for metagenomic analysis of complex clinical infection samples yields optimized viable microbiome profiles. Cell Rep, 2019, 26(8): 2227-2240.e2225. |
| [33] |
Simner PJ, Miller HB, Breitwieser FP, et al. Development and optimization of metagenomic next-generation sequencing methods for cerebrospinal fluid diagnostics. J Clin Microbiol, 2018, 56(9): e00472-18. |
| [34] |
Martin TC, Visconti A, Spector TD, et al. Conducting metagenomic studies in microbiology and clinical research. Appl Microbiol Biotechnol, 2018, 102(20): 8629-8646. |
| [35] |
Bachmann NL, Rockett RJ, Timms VJ, et al. Advances in clinical sample preparation for identification and characterization of bacterial pathogens using metagenomics. Front Public Health, 2018, 6: 363. |
| [36] |
Gu W, Crawford ED, O'Donovan BD, et al. Depletion of abundant sequences by hybridization (DASH): using Cas9 to remove unwanted high-abundance species in sequencing libraries and molecular counting applications. Genome Biol, 2016, 17: 41. |
| [37] |
Stevens KA, Jaykus LA. Bacterial separation and concentration from complex sample matrices: a review. Crit Rev Microbiol, 2004, 30(1): 7-24. |
| [38] |
Goig GA, Blanco S, Garcia-Basteiro AL, et al. Contaminant DNA in bacterial sequencing experiments is a major source of false genetic variability. BMC Biol, 2020, 18: 24. |
| [39] |
Salter SJ, Cox MJ, Turek EM, et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biol, 2014, 12: 87. |
| [40] |
Gargis AS, Kalman L, Bick DP, et al. Good laboratory practice for clinical next-generation sequencing informatics pipelines. Nat Biotechnol, 2015, 33(7): 689-693. |
| [41] |
Olson ND, Lund SP, Colman RE, et al. Best practices for evaluating single nucleotide variant calling methods for microbial genomics. Front Genet, 2015, 6: 235. |
| [42] |
Brinkmann A, Andrusch A, Belka A, et al. Proficiency testing of virus diagnostics based on bioinformatics analysis of simulated in silico high-throughput sequencing data sets. J Clin Microbiol, 2019, 57(8): e00466-19. |
| [43] |
Naccache SN, Federman S, Veeraraghavan N, et al. A cloud-compatible bioinformatics pipeline for ultrarapid pathogen identification from next-generation sequencing of clinical samples. Genome Res, 2014, 24(7): 1180-1192. |
| [44] |
Sichtig H, Minogue T, Yan Y, et al. FDA-ARGOS is a database with public quality-controlled reference genomes for diagnostic use and regulatory science. Nat Commun, 2019, 10: 3313. |
| [45] |
Shepelev VA, Uralsky LI, Alexandrov AA, et al. Annotation of suprachromosomal families reveals uncommon types of alpha satellite organization in pericentromeric regions of hg38 human genome assembly. Genom Data, 2015, 5: 139-146. |
| [46] |
Pan BH, Kusko R, Xiao WM, et al. Similarities and differences between variants called with human reference genome HG19 or HG38. BMC Bioinformatics, 2019, 20(S2): 101. |
| [47] |
Ye SH, Siddle KJ, Park DJ, et al. Benchmarking metagenomics tools for taxonomic classification. Cell, 2019, 178(4): 779-794. |
| [48] |
Hardwick SA, Deveson IW, Mercer TR. Reference standards for next-generation sequencing. Nat Rev Genet, 2017, 18(8): 473-484. |
| [49] |
Seehusen DA, Reeves MM, Fomin DA. Cerebrospinal fluid analysis. Am Fam Physician, 2003, 68(6): 1103-1108. |
| [50] |
Cold Spring Harbor Laboratory Press. Artificial cerebrospinal fluid (ACSF) (10×). Cold Spring Harb Protoc, 2017, DOI: 10.1101/pdb.rec094342.
|
| [51] |
Huang WC, Li LP, Myers JR, et al. ART: a next-generation sequencing read simulator. Bioinformatics, 2012, 28(4): 593-594. |
| [52] |
Roy S, Coldren C, Karunamurthy A, et al. Standards and guidelines for validating next-generation sequencing bioinformatics pipelines: a joint recommendation of the association for molecular pathology and the college of american pathologists. J Mol Diagn, 2018, 20(1): 4-27. |
 2020, Vol. 36
2020, Vol. 36