中国科学院微生物研究所、中国微生物学会主办
文章信息
- 胡宗福, 常杰, 佟庆, 于建华, 李树国, 牛化欣
- Hu Zongfu, Chang Jie, Tong Qing, Yu Jianhua, Li Shuguo, Niu Huaxin
- 高通量测序方法分析两种草食性淡水螺肠道菌群多样性
- High-throughput sequencing analysis of intestinal flora diversity of two freshwater snails (Radix auricularia and Planorbella trivolvis)
- 生物工程学报, 2020, 36(12): 2622-2634
- Chinese Journal of Biotechnology, 2020, 36(12): 2622-2634
- 10.13345/j.cjb.200322
-
文章历史
- Received: June 4, 2020
- Accepted: November 13, 2020
- Published: December 15, 2020
2. 东北农业大学 动物医学院,黑龙江 哈尔滨 150038
2. College of Veterinary Medicine, Northeast Agricultural University, Harbin 150038, Heilongjiang, China
淡水螺是水生生态环境中常见的生物类群,在水生态系统中具有重要的地位和作用。在缺少鱼类等大型消费者的小水体中,淡水螺类是主要的初级消费者,对初级生产力的生物量和能量的转化具有重要的作用[1-2]。淡水螺还是很多寄生虫的中间宿主,在某些重要人畜寄生虫传染病的防治中有重要地位[3-4]。
动物的肠道菌群不但在动物的能量代谢和抵抗病原体方面起着重要的作用,还在生长、发育、免疫、繁殖等多个方面发挥重要的功能[5-7]。当前,脊椎动物肠道菌群的研究已经取得了广泛的进展[8-9],但软体动物肠道微生物的研究相对滞后。对软体动物腹足纲的肠道微生物研究主要集中于贝类肠道微生物区系的研究和功能性肠道微生物的研究。淡水螺肠道菌群区系研究方面仅见一篇报道,Horn等[10]采用16S rRNA基因高通量测序方法,调查了来自非洲和北美的3种淡水扁卷螺(Planorbidae,腹足纲Pulmonata)的肠道菌群,观察到的细菌分布在18个门中,证明了在3种淡水螺中存在高度多样化的肠道细菌群落,并表明需要进一步的研究来确定肠道细菌在螺生理和免疫学中的作用。陆生蜗牛是软体动物腹足类肠道微生物研究相对较多的种类。Nicolai等[11]使用PCR- DGGE指纹分析技术揭示了蜗牛Helix pomatia肠道微生物在不同生境群体间的差异和季节性的变化,还发现有些菌群属于不随季节变化而影响的常驻菌群。贝类功能性肠道微生物方面的研究目前主要集中于纤维素分解菌的挖掘。Cardoso等[12]对非洲大蜗牛Achatina fulica肠道微生物进行了宏基因组研究,发现了很多与木质纤维素分解相关的基因,在2 700个GH (Glycoside hydrolase)编码基因中约有36%的基因序列与纤维素酶和半纤维素酶编码基因序列相似,这有助于通过分解木质纤维素产生乙醇燃料来缓解目前紧张的全球能源问题。Joynson等[13]采用宏基因组学的方法研究了一种黑蛞蝓Arion ater的肠道菌群的多样性及功能,揭示了肠道菌群中大量存在的木质纤维素酶的基因。
三旋卷丽螺源自北美,食谱范围广和适应力强的特性使其能够随观赏鱼贸易携带进入我国[14]。耳萝卜螺则广泛分布于各种淡水水体中,从湖泊水库到小型水体。耳萝卜螺以水草、藻类和落叶为食[1]。淡水肺螺类肠道微生物群落的研究尚处于起步阶段,为补充这种研究匮乏,本实验以三旋卷丽螺和耳萝卜螺两种淡水螺为研究对象,采用16S rRNA基因Miseq高通量测序技术,对其肠道微生物群落进行研究,分析了两种草食性淡水螺的核心菌群,为研究软体动物淡水腹足类肠道微生物生态学和功能提供数据支撑。
1 材料与方法 1.1 样本采集与处理实验材料包括两种淡水螺,耳萝卜螺(Radix auricularia,RA)采自内蒙古通辽市一小池塘内。池塘水深约0.5 m,水温约23 ℃,池塘内主要食物种类为刚毛藻、落叶及底栖微藻。所采螺的平均壳高约(9.7±0.3) mm,平均壳长约(20±0.5) mm。三旋丽卷螺(Planorbella trivolvis,PL)来自实验室养殖,螺壳直径为(10.53±0.45) mm,缸内水温约26 ℃,螺的食物来源为干苜蓿及缸内孽生的藻类。
实验螺采回实验室后,5 h内取样完毕。先用自来水洗净螺壳上的杂质,然后用70%酒精擦拭螺体表面。在超净台内进行无菌操作,破坏螺壳,解剖、分离和取出肠道。将肠道连同内容物一起装入无菌1.5 mL离心管,−80 ℃保存,用于细菌DNA的提取。鉴于螺个体微小,为满足取样要求,将5个螺的肠道及其内容物装于一个离心管内成为一个重复样本,每种螺各有4个重复样本。
1.2 肠道菌群多样性的测定取螺肠道样品约0.5 g,按照使用MP-bio土壤DNA提取试剂盒说明提取细菌基因组,将提取的DNA进行1.5%琼脂糖凝胶电泳,并用NanoDrop对DNA的浓度和OD260/OD280值进行测定。
采用16S rRNA基因V3–V4区引物(338F:5′-ACTCCTACGGGAGGCAGCA-3′,806R:5′-GG ACTACHVGGGTWTCTAAT-3′)进行PCR扩增,引物两端带有12 bp的条形码,用以识别不同样品的序列。PCR扩增使用TransStart FastPfu (全式金,北京,中国)试剂盒。反应体系:4.0 μL 5×FastPfu缓冲液,2 μL dNTPs (各2.5 mmol/L),0.4 μL FastPfu DNA聚合酶,10 ng DNA模板,0.8 μL正向引物338F (5 mmol/L),0.8 μL反向引物806R (5 mmol/L),然后用ddH2O补足20 μL。程序如下:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,循环数为26个循环,最后72 ℃延伸10 min。扩增产物使用1.5%琼脂糖凝胶进行电泳检测,使用Thermo Scientific公司的GeneJET胶回收试剂盒回收产物,使用Illumina测序专用的NEB Next® Ultra DNA Library Prep Kit试剂盒建立基因克隆文库。构建好的文库送交上海美吉生物工程有限责任公司通过Illumina Miseq PE300平台进行高通量测序。
1.3 数据统计使用Usearch (version 7.0)软件(http://drive5.com/uparse/),将所得序列根据97%相似度关系进行操作分类单元(Operational taxonomic units,OTU)聚类分析,产生OTU表,并与Silva数据库(http://www.arb-silva.de)比对进行分类学分析,所用平台为QIIME平台(http://qiime.org/scripts/assign_taxonomy.html),统计后得到各样本的群落组成。然后对群落进行基于OTU水平的α多样性分析,所用软件为Mothur (version v.1.30.1,http://www.mothur.org/wiki/Schloss_SOP#Alpha_diversity),分析指标包括Sobs指数、Shannon指数、Chao指数、Simpson指数、Ace指数以及覆盖度(Coverage)。肠道菌群组成分析及热图使用R语言工具作图,主成分分析(Principal component analysis,PCA)采用R语言的vegan软件包进行分析和作图。肠道菌群的基因功能预测使用PICRUSt (Phylogenetic Investigation of Communities by Reconstruction of Unobserved States)软件进行分析。系统发育进化树使用IQ-TREE进行分析制作。差异显著性用Kruskal-Wallis秩和检验比较,结果以平均值±标准差表示,P < 0.05表示差异显著。
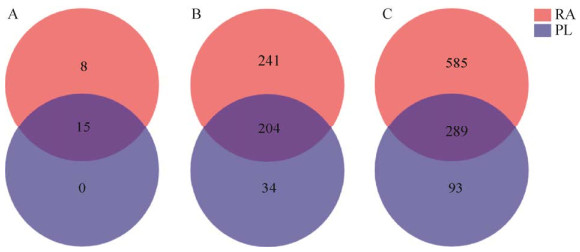
2 结果与分析 2.1 基于16S rRNA基因测序的肠道菌群多样性及组成分析测序得到的双端序列经过质量控制,包括去除质量值低于20的尾部碱基及含氮的碱基,根据双端序列间的重叠关系进行双端序列的拼接,通过末端条形码识别得到每个样本的序列,8个样品最终得到有效序列数367 979条,平均序列长度为425.48 bp。其中三旋卷丽螺(PL) 4个样共获得有效序列225 592条,平均每个样品56 398条,耳萝卜螺(RA) 4个样共获得有效序列142 837条,平均每个样品为35 597条。基于97%序列同源性划分共得到1 117个OTU,其中PL组有505个OTU,分属297个属、173科、105目、36纲、19门;RA组有949个OTU,分属457个属、218科、129目、42纲、24门。在后续分析中,将每个样本序列数抽平至25 733条,过滤掉含Chloroplast的OTU,最后获得967个OTU,其中PL组有382个OTU,分属238个属、137科、86目、28纲、15门;RA组有874个OTU,445个属、209科、126目、41纲、23门。维恩图显示,虽然两种螺种类和采集水域不同,但其共有OTU达289个,分别占PL及RA的75.65%及33.07%。RA特有OTU (585个)远高于PL特有OTU数(93个)。在属水平上,共有菌属有204个,分别占PL及RA的85.71%及45.84%,RA特有菌属(241个)远高于PL特有菌属(34个)。此外,在门水平上,共有菌门15个,占PL总门数的100%,占RA总门数的64.22% (图 1)。
|
| 图 1 基于门水平(A)、属水平(B)及OTU水平(C)的Venn分析 Fig. 1 Venn analysis of gut bacterial community based on phylum (A), genus (B) and OTU (C) level. |
| |
由表 1可见,两组样本的α多样性指数显著差异(P < 0.05),Sobs指数RA组显著大于PL组(P < 0.05),表明耳萝卜螺样本中所观察到的物种数目少于三旋卷丽螺;Shannon指数RA组显著高于PL组(P < 0.05),Simpson指数RA显著低于PL组(P < 0.05),表明耳萝卜螺肠道菌群多样性比三旋卷丽螺高;Chao指数RA组高于PL组(P < 0.05),Ace指数RA组高于PL组(P < 0.05),表明耳萝卜螺肠道菌群物种丰富度较三旋卷丽螺组高。两组螺菌群样本的稀释曲线显示均已进入平台期,表明测序深度可靠,能够真实反映样本中大多数菌群组成(图 2)。
| Samples | Shannon | Simpson | Ace | Chao | Sequences |
| PL | 3.41±0.21b | 0.09±0.03a | 324.38±10.47b | 329.06±14.05b | 56 318.50±12 717.32a |
| RA | 5.00±0.26a | 0.02±0.01b | 774.08±30.88a | 780.70±41.74a | 33 777.33±1 977.36b |
| Notes: PL stands for the gut microflora of Planorbella trivolvis; RA stands for the gut microflora of Radix auricularia. The letter “a” and “b” represent the significant different between two groups (P < 0.05). | |||||

|
| 图 2 稀释曲线和香农指数曲线 Fig. 2 Sobs and Shannon indexes of snail gut microbiota. (A) Rarefaction curves of snail gut microbiota. (B) Shannon curves of snail gut microbiota. PL represents the samples of Planorbella trivolvis group, RA represents the samples of Radix auricularia group. |
| |
在门水平上,PL和RA肠道菌群含有的菌门分别为13和23个,其中相对丰度大于1%的门分别有7个和10个(表 2)。变形菌门(Proteobacteria)在两组中都是丰度最大的菌门,在PL和RA中的相对丰度分别为54.88%和33.63%。此外,PL组相对丰度较高的有拟杆菌门(Bacteroidetes,28.49%)、放线菌门(Actinobacteria,7.65%)、绿弯菌门(Chloroflexi,2.55%)、疣微菌门(Verrucomicrobia,2.76%)和衣原体门(Chlamydiae,1.22%)。在RA肠道菌群中,除变形菌门外,蓝细菌门(Cyanobateria,15.33%)、放线菌门(Actinobacteria,12.99%)和绿弯菌门(Chloroflexi,13.95%)也具有一定优势。此外,RA肠道菌群中丰度大于1%的还有厚壁菌门(Firmicutes,7.09%)、拟杆菌门(Bacteroidetes,2.27%)、软壁菌门(Tenericutes,6.10%)、一种未鉴定的菌门(Unclassified,4.80%)、疣微菌门(Verrucomicrobia,1.49%)和梭杆菌门(Fusobacteria,1.23%) (表 2)。
| Phylum | Groups | |||
| PL | RA | PL-Sd | RA-Sd | |
| Proteobacteria | 54.88 | 33.63 | 4.10 | 4.32 |
| Bacteroidetes | 28.49 | 2.27 | 7.12 | 1.18 |
| Actinobacteria | 7.65 | 12.99 | 1.76 | 2.85 |
| Cyanobacteria | 1.35 | 15.33 | 0.56 | 5.75 |
| Chloroflexi | 2.55 | 13.95 | 0.56 | 5.09 |
| Firmicutes | 0.58 | 7.09 | 0.52 | 3.28 |
| Tenericutes | 0 | 6.101 | 0 | 5.19 |
| Unclassified | 0 | 4.80 | 0 | 3.14 |
| Verrucomicrobia | 2.76 | 1.49 | 1.59 | 0.97 |
| Fusobacteria | 0.18 | 1.23 | 0.11 | 0.34 |
| Chlamydiae | 1.22 | 0.010 | 0.56 | 0.12 |
| Planctomycetes | 0.25 | 0.20 | 0.14 | 0.10 |
| Acidobacteria | 0.06 | 0.35 | 0.02 | 0.15 |
| Deinococcus-Thermus | 0 | 0.19 | 0 | 0.27 |
在属水平上,RA组中丰度高于1%的菌属有17个,有9个不能分类到属水平,可在高一级的水平得到分类(科、目或纲),丰度低于1%的属的总丰度达52.18% (图 3)。各属间丰度依次下降,其中丰度相对较高的菌属有绿屈挠菌科未知属(Unclassified_f_Chloroflexaceae,6.92%)、支原体科未知属(Norank_f_Mycoplasmataceae,5.42%)、厚皮藻属(Pleurocapsa,4.27%)、硫网菌属(Thiodictyon,3.70%)和暖绳菌科未知属(Norank_f_Caldilineaceae,4.08%)。其他还有席藻科(Phormidiaceae,2.04%)、Nodosilinea (1.58%)、类诺卡氏菌属(Nocardioides,1.49%)、鞘丝藻属(Leptolyngbya,1.44%)、纤毛菌属(Leptotrichia,1.23%)、红杆菌科未知属(Unclassified_f_ Rhodobacteraceae,1.64%)和根瘤菌科未知属(Norank_f_Rhizobiales_Incertae_Sedis,1.48%)等(图 3)。

|
| 图 3 基于属水平的淡水螺肠道菌群组成 Fig. 3 The composition of gut bacterial community in Radix auricularia and Planorbella trivolvis. Notes: all the genera with abundance below 0.03 was showed as others; norank or unclassified represent the bacteria can not identified in genus level; f_ represents family; o_ represents order; R_1–R_4 stand for the intestinal microflora of Radix auricularia (RA); P_1–P_4 stand for the intestinal microflora of Planorbella trivolvis (PL). |
| |
而PL组中丰度高于1%的菌属有14个,有6个不能分类到属水平,可在高一级的水平得到鉴定(科、目或纲)。其他丰度低于1%的菌属占总丰度的14.25% (图 3)。以Cloacibacterium的丰度最高(达24.33%)。其他丰度大于1%的菌属由高到低依次为根瘤菌科未知属(Norank_f_Rhizobiales_ Incertae_Sedis,7.33%)、OM60NOR5_ clade (6.25%)、红杆菌科未知属(Unclassified_f_Rhodobacteraceae,5.76%)、假单胞菌属(Pseudomonas,4.45%)、红杆菌属(Rhodobacter,3.86%)、Candidatus_Alysiosphaera (3.39%)、肠杆菌科未知属(Unclassified_ Enterobacteriaceae,2.28%)、固氮螺菌属(Azospirillum,3.01%)、分枝杆菌属(Mycobacterium,2.15%)、气单胞菌属(Aeromonas,1.97%)及Luteolibacter (1.85%)等(图 3)。
2.2 共有核心菌群分析该螺所有的肠道样本都享有的菌群,任何一个样品不含有的菌属都需要剔除。经过分析,获得了93个共有核心菌群,其中丰度大于0.5%的共有核心菌群有27个属(表 3)。这些菌属主要分布在变形菌门(Proteobacteria)内,个别也分布在绿弯菌门(Chloroflexi)、放线菌门(Actinobacteria)、疣微菌门(Verrucomicrobia)及梭杆菌门(Fusobacteria)。
| Phylum | Family | Genus | PL-mean | PL-SD | RA-mean | RA-SD |
| Proteobacteria | Rhizobiales_Incertae_Sedis | Norank | 7.33 | 0.49 | 1.48 | 0.45 |
| Proteobacteria | Halieaceae | OM60NOR5_clade | 6.25 | 1.68 | 0.08 | 0.07 |
| Proteobacteria | Rhodobacteraceae | Unclassified | 5.76 | 1.05 | 1.64 | 0.99 |
| Proteobacteria | Pseudomonadaceae | Pseudomonas | 4.45 | 0.40 | 0.97 | 1.06 |
| Proteobacteria | Rhodobacteraceae | Rhodobacter | 3.86 | 0.44 | 0.96 | 0.31 |
| Proteobacteria | Geminicoccaceae | Candidatus_Alysiosphaera | 3.39 | 0.75 | 0.13 | 0.06 |
| Chloroflexi | JG30-KF-CM45 | Norank | 2.54 | 0.55 | 1.36 | 0.65 |
| Proteobacteria | Enterobacteriaceae | Unclassified | 2.28 | 0.84 | 0.94 | 0.81 |
| Actinobacteria | Mycobacteriaceae | Mycobacterium | 2.15 | 0.48 | 0.39 | 0.08 |
| Proteobacteria | Aeromonadaceae | Aeromonas | 1.97 | 0.71 | 0.78 | 0.66 |
| Verrucomicrobia | Rubritaleaceae | Luteolibacter | 1.85 | 1.29 | 0.54 | 0.31 |
| Proteobacteria | Sphingomonadaceae | Polymorphobacter | 1.62 | 0.36 | 0.14 | 0.05 |
| Proteobacteria | Reyranellaceae | Reyranella | 1.34 | 0.37 | 0.10 | 0.11 |
| Proteobacteria | Acetobacteraceae | Roseomonas | 1.13 | 0.09 | 0.41 | 0.15 |
| Bacteroidetes | Chitinophagaceae | Dinghuibacter | 1.09 | 0.36 | 0.035 | 0.06 |
| Bacteroidetes | Flavobacteriaceae | Flavobacterium | 1.08 | 0.32 | 0.40 | 0.49 |
| Bacteroidetes | Bacteroidaceae | Bacteroides | 0.88 | 1.65 | 0.12 | 0.15 |
| Actinobacteria | Microbacteriaceae | Aurantimicrobium | 0.68 | 0.16 | 0.74 | 0.20 |
| Proteobacteria | Burkholderiaceae | Polynucleobacter | 0.62 | 0.09 | 0.12 | 0.11 |
| Proteobacteria | Burkholderiaceae | Norank | 0.52 | 0.18 | 0.23 | 0.12 |
| Proteobacteria | Hyphomicrobiaceae | Hyphomicrobium | 0.47 | 0.17 | 0.56 | 0.09 |
| Fusobacteria | Leptotrichiaceae | Leptotrichia | 0.18 | 0.11 | 1.23 | 0.34 |
| Proteobacteria | Burkholderiaceae | Hydrogenophaga | 0.14 | 0.05 | 0.56 | 0.27 |
| Proteobacteria | Rhodobacteraceae | Paracoccus | 0.03 | 0.01 | 0.78 | 0.38 |
| Proteobacteria | Acetobacteraceae | Unclassified | 0.01 | 0.01 | 0.51 | 0.21 |
| Chloroflexi | Caldilineaceae | Norank | 0.01 | 0.01 | 4.08 | 1.43 |
| Actinobacteria | Microbacteriaceae | Microbacterium | 0.01 | 0.01 | 0.55 | 0.40 |
PCoA分析用来表示不同样品间的菌群多样性,如图 4所示,在非加权丰度因素(Unweighted unifrac)的条件下分析(图 4A),两种螺各自的样本都能显著分为一个群体(R2=0.735,P=0.027),PC1坐标轴的影响因素占总成分的73.59%。在加权丰度因素的条件下(Weighted unifrac) (图 4 B),两种螺的样本分群同样显著(R2=0.843,P=0.027),PC1坐标轴的影响因素占总成分的84.65%。两种螺样本在两种分析水平上(非加权丰度因素和加权丰度因素)均显著分群(P < 0.05),表示两种螺的微生物群落的结构和丰度都有自己明显的特征。

|
| 图 4 基于OTU水平的PCoA分析 Fig. 4 PCoA analysis of gut bacterial community based on OTU level. (A) Analysed by unweighted unifrac calculate mode. (B) Analysed by weighted unifrac calculate mode. R2 and P were the correlation coefficient and significance of Adonis of two snail gut microflora. |
| |
为反映两种淡水螺的肠道菌群的关系,构建了含物种聚类关系树、样品聚类关系树的热图分析(图 5),图中样品的颜色代表了菌群的相对丰度,图左侧即为基于属水平的菌群聚类关系树,图中顶端代表样品的聚类关系。物种聚类关系树显示,所有的菌群按其在样品中的丰度分为两个大的聚类分支,其中PL肠道菌群丰度较高的菌群聚类为一个大的聚类分支,而RA的肠道菌群可聚类为一个大的聚类分支。样品聚类关系分析显示,来自RA的4个样本彼此临近,同样来自PL的4个样本彼此临近,说明2种螺在组内各样品之间肠道菌群相似度较大。

|
| 图 5 样品中肠道菌群热图分析 Fig. 5 Heatmap analysis of intestinal flora. Norank or unclassified represent the bacteria can not identified in genus level; f_ represents family; o_ represents order; R_1–R_4 stand for the intestinal microflora of Radix auricularia (RA); P_1–P_4 stand for the intestinal microflora of Planorbella trivolvis (PL). |
| |
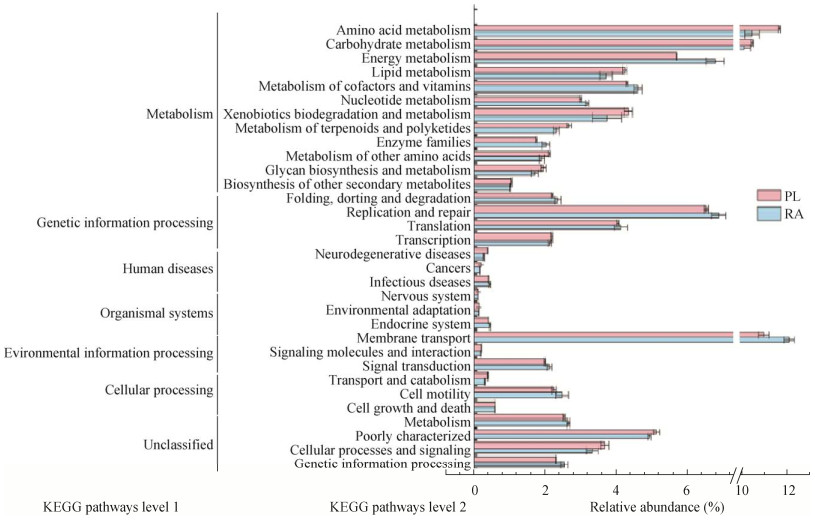
通过对三旋卷丽螺(PL)和耳萝卜螺(RA)肠道菌群KEGG pathway (level-2)进行丰度组成分析,结果显示,与物种组成相比,两组样本的KEGG功能组成较为相似(图 6)。丰度相对较大的代谢通路如氨基酸代谢(RA,10.44%;PL,11.65%)、碳水化合物代谢(RA,10.10%;PL,10.45%)及膜转运(RA,12.10%;PL,10.95%);丰度居中的如能量代谢(RA,6.78%;PL,5.70%)、遗传信息的复制与修复(RA,6.87%;PL,6.53%)、脂类代谢(RA,3.71%;PL,4.24%)、辅酶因子和维生素代谢(RA,4.60%;PL,4.30%)及外源物质降解与代谢(RA,3.73%;PL,4.33%) (图 6)。
|
| 图 6 基于PICRUSt的肠道菌群基因功能预测 Fig. 6 Genomic functional prediction of snail gut bacterial community by PICRUSt. |
| |
通常认为无脊椎动物肠道与微生物群落联系有限[15],但相关研究表明无脊椎动物也具有复杂的肠道微生物群落,曾有研究提出无脊椎动物可能与更复杂的微生物群落共同进化[16]。本文基于16S rRNA基因高通量测序分析显示,两种淡水螺具有很高的肠道菌群多样性,OTU数达到了967个,Shannon指数在两种螺都大于3.4,Chao指数都大于350。本研究中螺肠道菌群多样性要高于Horn等[10]对3种不同地区淡水螺肠道菌群的研究。从菌群多样性指数来看,耳萝卜螺显著高于三旋卷丽螺,这可能与耳萝卜螺采自野生群体有关,野外水体具有更多样性的食物和更复杂的环境,而三旋卷丽螺为室内养殖,饵料与环境较为单一,由于这种环境和饵料的差异,会对多样性结果造成影响[11]。
由结果可见,尽管在门水平上,变形菌门是优势菌门,但是在较低的分类水平上,优势菌群之间丰度差距不大,例如在耳萝卜螺中,最大的菌属绿屈挠菌科未知属(Unclassified_F_ Chloroflexaceae)丰度仅为6.92%,丰度大于4%的菌属有4个,而丰度大于1%的菌属达到了17个,三旋卷丽螺也存在相同的趋势。因此,对菌群的关注不应局限于丰度较大的菌属。
本研究显示,变形菌门是两种螺共同的优势菌门,在三旋卷丽螺中占比达54.88%,在耳萝卜螺占比33.63%。在其他腹足类肠道菌群中,变形菌门也占优势地位,如在黑蛞蝓肠道菌群中丰度达到了88.15%,在非洲大蜗牛A. fulica、淡水螺Potamopyrgus antipodarum、淡水扁卷螺Biomphalaria pfeifferi也是优势菌群[17-18]。变形菌门还在其他食物组成富含木质纤维素的草食性昆虫肠道菌群中占优势地位[19-20]。对Biomphalaria pfeifferi (非洲)、Bulinus africanus (非洲)和Helisoma duryi (北美)等3种扁卷螺肠道菌群进行的16S rRNA扩增子测序研究显示,细菌多样性分布在18个门中,其中来自变形菌门γ亚群、拟杆菌门和酸杆菌门的序列数最多。
Cloacibacterium是PL的特有物种,在PL样本中呈优势存在,而不分布在RA中。Cloacibacterium属于黄杆菌科(Flavobacteriaceae),是一种具有黄色色素的兼性厌氧、革兰氏阳性杆菌,可从淡水底泥中分离得到[21],富含各种脂肪酸,该菌还在人类帕金森病患者血液及腺瘤患者肠道内富集 [22-23],对人类可能有致病作用。此外,红杆菌科、假单胞菌属、红杆菌属、固氮螺菌属等也在PL组中显著存在。在新西兰的一种淡水螺Potamopyrgus antipodarum肠道菌群内也发现了红杆菌科假单胞菌属的存在[18]。红杆菌属属于变形菌门,广泛分布在海水和淡水中,是一种可进行光合作用的具有新能源利用价值的菌种,红杆菌属在螺肠道内的出现说明了其通过水环境向动物肠道迁徙的路线。Vesbach等[18]认为红杆菌已经定植于淡水螺Potamopyrgus antipodarum肠道内与宿主形成共生体,另外在海绵Halichondria panicea和水蚤Daphnia体内也发现了红杆菌的共生现象[24-25]。
在耳萝卜螺肠道中显著富集的物种有绿弯菌科未知属、支原体科未知属、暖绳菌科未知属、厚皮藻属、网硫菌属、席藻科未知属等。绿弯菌科未知属和暖绳菌科未知属都属绿弯菌门,是一种能够进行光合作用的革兰氏染色阴性的自养菌[26-27],这种菌在耳萝卜螺的出现说明其作为水体环境菌群进入螺肠道。支原体是动物和环境中广泛存在的一类原生动物,有些种类可引起动物疾病,因此耳萝卜螺肠道菌群内支原体的作用需要进一步关注和研究。厚皮藻属是一种淡水藻类,网硫菌属是一种能进行光合作用的革兰氏阴性菌,属丙型变形菌门,都作为水体微生物进入螺肠道,属于螺肠道暂居菌群。席藻科属于蓝藻门,是一类淡水中常见的自养藻类。可见,在耳萝卜螺中显著富集的菌群很大一部是来自其水生环境。
研究显示有289个OTU、204个菌属及15个门为两种螺肠道菌群所共有,进一步的核心菌群分析显示有93个菌属为两种淡水螺共有的核心菌群,其中有27个菌属丰度大于0.5%,这些菌群一定程度上反映了作为草食性淡水螺肠道菌群的共同特征,分类学显示以变形菌门为主,其他还有拟杆菌门及放线菌门[11, 13]。较为代表性的有气单胞菌属、假单胞菌属、Luteolibacter、黄杆菌属、拟杆菌属、细杆菌属及Aurantimicrobium。在非洲大蜗牛及黑蛞蝓肠道中也发现了丰度较大的气单胞菌属、假单胞菌属、黄杆菌属及肠杆菌属[13, 28]。
本研究通过菌群基因组的功能预测分析发现,两种螺肠道菌群功能丰度的组成基本相似,丰度较大的均以氨基酸代谢、碳水化合物分解代谢、膜转运及能量代谢为主。这与以往对于非洲大蜗牛及欧洲黑蛞蝓肠道微生物功能组成的研究结果保持了一致,提示肺螺类软体动物在肠道菌群功能组成方面存在着相似性[12-13]。而基于欧洲黑蛞蝓及非洲大蜗牛的肠道微生物宏基因组分析发现,螺类之间的肠道菌群功能组成的差异要小于其与奶牛、大蜗牛、白蚁及长角亚洲甲虫等物种肠道菌群功能组成的差异[13]。此外,两种螺肠道菌群在外源物质降解与代谢及萜类和聚酮化合物的代谢两种代谢通路的丰度也较大,提示两种螺肠道菌群在分解外源性有毒有害化合物等方面有一定的作用,这使其具有较高的生理适应性[12]。由于三旋卷丽螺的外来物种属性,这种适应性提高了其适应新环境的能力[14]。
综上所述,两种螺肠道菌群存在高度的多样性,二者之间既有物种分布和丰度的差异,又有多种共有核心菌群。本文对淡水螺肠道菌群的研究有助于发掘微生物资源库,深化对淡水软体动物肠道菌群对认识,对研究肠道菌群在淡水软体动物生理学、免疫学及生态学中的作用具有重要意义。
| [1] |
Dewitt TJ, Sih A, Hucko JA. Trait compensation and cospecialization in a freshwater snail: size, shape and antipredator behaviour. Anim Behav, 1999, 58(2): 397-407. |
| [2] |
Eckblad J. Biomass and energy transfer by a specialized predator of aquatic snails. Freshwater Biol, 2010, 6(1): 19-21. |
| [3] |
Soldánová M, Selbach C, Sures B, et al. Larval trematode communities in Radix auricularia and Lymnaea stagnalis in a reservoir system of the Ruhr river. Parasite Vector, 2010, 3: 56. |
| [4] |
Bargues MD, Vigo M, Horak P, et al. European Lymnaeidae (Mollusca: Gastropoda), intermediate hosts of trematodiases, based on nuclear ribosomal DNA ITS-2 sequences. Infect Genet Evol, 2001, 1(2): 85-107. |
| [5] |
Sommer F, Bäckhed F. The gut microbiota-masters of host development and physiology. Nat Rev Microbiol, 2013, 11(4): 227-238. |
| [6] |
Maslowski KM, Mackay CR. Diet, gut microbiota and immune responses. Nat Immunol, 2011, 12(1): 5-9. |
| [7] |
Morrison DJ, Preston T. Formation of short chain fatty acids by the gut microbiota and their impact on human metabolism. Gut Microbes, 2016, 7(3): 189-200. |
| [8] |
Bäckhed F, Ley RE, Sonnenburg JL, et al. Host-bacterial mutualism in the human intestine. Science, 2005, 307(5717): 1915-1920. |
| [9] |
Fan PX, Bian BL, Teng L, et al. Host genetic effects upon the early gut microbiota in a bovine model with graduated spectrum of genetic variation. ISME J, 2020, 14(1): 302-317. |
| [10] |
Van Horn DJ, Garcia JR, Loker ES, et al. Complex intestinal bacterial communities in three species of Planorbid snails. J Mollus Stud, 2012, 78(1): 74-80. |
| [11] |
Nicolai A, Rouland-lefèvre C, Ansart A, et al. Inter-population differences and seasonal dynamic of the bacterial gut community in the endangered land snail Helix pomatia (gastropoda: helIcIdae). Malacologia, 2015, 59(1): 177-190. |
| [12] |
Cardoso AM, Cavalcante JJV, Cantão ME, et al. Metagenomic analysis of the microbiota from the crop of an invasive snail reveals a rich reservoir of novel genes. PLoS ONE, 2012, 7(11): e48505. |
| [13] |
Joynson R, Pritchard L, Osemwekha E, et al. Metagenomic analysis of the gut microbiome of the common black slug Arion ater in search of novel lignocellulose degrading enzymes. Front Microbiol, 2017, 8: 2181. |
| [14] |
Li XH, Gao ST, Gu WB, et al. Species identification of freshwater snail Planorbella trivolvis and analysis of its potential distribution. Chin J Schistosom Control, 2015, 27(3): 268-272 (in Chinese). 李晓恒, 高世同, 顾文彪, 等. 淡水螺类三旋丽卷螺的物种鉴定及其潜在适生区分析. 中国血吸虫病防治杂志, 2015, 27(3): 268-272. |
| [15] |
Mcfall-Ngai M. Adaptive immunity: care for the community. Nature, 2007, 445(7124): 153. |
| [16] |
Loker ES, Adema CM, Zhang SM, et al. Invertebrate immune systems-not homogeneous, not simple, not well understood. Immunol Rev, 2004, 198(1): 10-24. |
| [17] |
Cardoso AM, Cavalcante JJV, Vieira RP, et al. Gut bacterial communities in the giant land snail Achatina fulica and their modification by sugarcane-based diet. PLoS ONE, 2012, 7(3): e33440. |
| [18] |
Takacs-Vesbach C, King K, Van Horn D, et al. Distinct bacterial microbiomes in sexual and asexual Potamopyrgus antipodarum, a New Zealand freshwater snail. PLoS ONE, 2016, 11(8): e0161050. |
| [19] |
Dillon RJ, Dillon VM. The gut bacteria of insects: nonpathogenic interactions. Annu Rev Entomol, 2004, 49: 71-92. |
| [20] |
Russell JA, Moreau CS, Goldman-Huertas B, et al. Bacterial gut symbionts are tightly linked with the evolution of herbivory in ants. Proc Natl Acad Sci USA, 2009, 106(50): 21236-21241. |
| [21] |
Allen TD, Lawson PA, Collins MD, et al. Cloacibacterium normanense gen. nov., sp. nov., a novel bacterium in the family Flavobacteriaceae isolated from municipal waste water. Int J Syst Evol Microbiol, 2006, 56(6): 1311-1316. |
| [22] |
Qian YW, Yang XD, Xu SQ, et al. Detection of microbial 16S rRNA gene in the blood of patients with Parkinson's disease. Front Aging Neurosci, 2018, 10: 156. |
| [23] |
Sanapareddy N, Legge RM, Jovov B, et al. Increased rectal microbial richness is associated with the presence of colorectal adenomas in humans. ISME J, 2012, 6(10): 1858-1868. |
| [24] |
Althoff K, Schütt C, Steffen R, et al. Evidence for a symbiosis between bacteria of the genus Rhodobacter and the marine sponge Halichondria panicea: harbor also for putatively toxic bacteria?. Mar Biol, 1998, 130(3): 529-536. |
| [25] |
Qi WH, Nong G, Preston JF, et al. Comparative metagenomics of Daphnia symbionts. BMC Genomics, 2009, 10: 172. |
| [26] |
Overmann J. The family Chlorobiaceae. In: Dworkin M, Falkow S, Rosenberg E, et al, Eds. The Prokaryotes. New York: Springer, 2006: 359–378.
|
| [27] |
Martins MD, Rigonato J, Taboga S R, et al. Proposal of Ancylothrix gen. nov., a new genus of Phormidiaceae (Cyanobacteria, Oscillatoriales) based on a polyphasic approach. Int J Syst Evol Microbiol, 2016, 66(6): 2396-2405. |
| [28] |
Pinheiro GL, Correa RF, Cunha RS, et al. Isolation of aerobic cultivable cellulolytic bacteria from different regions of the gastrointestinal tract of giant land snail Achatina fulica. Front Microbiol, 2015, 6: 860. |
 2020, Vol. 36
2020, Vol. 36