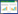中国科学院微生物研究所、中国微生物学会主办
文章信息
- 徐铮, 李娜, 陈盈利, 张雯瑞, 朱婉莹
- Xu Zheng, Li Na, Chen Yingli, Zhang Wenrui, Zhu Wanying
- 人乳寡糖2’-FL和3-FL的生物制备研究进展
- Recent advances in the bio-production of human milk oligosaccharides 2'-FL and 3-FL
- 生物工程学报, 2020, 36(12): 2767-2778
- Chinese Journal of Biotechnology, 2020, 36(12): 2767-2778
- 10.13345/j.cjb.200201
-
文章历史
- Received: April 12, 2020
- Accepted: June 23, 2020
- Published: July 7, 2020
母乳具有独一无二的活性功能,对新生儿健康至关重要。研究表明,母乳喂养的新生儿患肠胃炎、急性中耳炎以及其他免疫类疾病的概率较非母乳喂养显著降低,甚至在未来的智商发育上都占据优势,产生肥胖和糖尿病的概率也会下降[1]。母乳中含量最高的成分是乳糖和脂类,此外含有大量岩藻糖基化且无法被消化的寡糖(即人乳寡糖HMO),而牛奶、羊奶中的寡糖含量却非常少,因此母乳是最丰富的天然HMO源。母乳中至今已发现200种以上的HMO,初乳中总含量为20–25 g/L,成熟母乳中的含量为10–15 g/L[2-3]。HMO大多包含3–6个糖基,以乳糖或唾液酸结尾,可分为3类,包括被岩藻糖基修饰的中性HMO (35%–50%)、包含唾液酸及其硫酸盐的酸性HMO (12%–14%)以及非岩藻糖基化的中性HMO (42%–55%)。2’-岩藻基乳糖(2’-FL)的含量最高,达到HMO总量的近30%,母乳中浓度为0.5–2.0 g/L[4-6]。2’-FL的合成研究对HMO的商业化应用至关重要,尤其是寻找到高效率、低成本、绿色环保的合成路线。学者们首先尝试了有机合成法,但难以突破若干瓶颈问题,例如反应立体选择性差、产率低、使用毒性试剂等,不利于在食品工业中推广[2]。近年来,2’-FL和3-FL的生物制备研究发展迅速,并体现出优于化学合成路线的诸多特性,在国外已达到产业化水平[3]。本文针对这两种HMO的最新研究成果尤其是生物法合成路线予以综述,并对未来发展趋势进行深入讨论。
1 2’-FL和3-FL的功效研究HMO是母乳中的主要活性成分,对婴幼儿健康起到显著促进作用。HMO可以促进婴儿双歧杆菌Bifidobacterium infantis以及其他双歧杆菌类益生菌的生长,促进B. infantis分泌短链脂肪酸,构建健康的肠道微环境来抑制有害菌生长,进而减少腹泻发生。HMO在结构上与肠道表皮细胞表面的多糖受体结构相似,可以作为诱饵诱导病毒、病原菌、或细菌毒素与之结合,从而降低这些有害物质定殖或入侵人体的风险。研究表明,奶粉中添加2’-FL和乳糖-N-新四糖(LNnT)喂养的婴儿其体内的肠道菌群种类与母乳喂养的婴儿一致;持续喂养3个月,观察到粪便中的微生物双歧杆菌数量上升而病原菌种类减少[7-8]。肠杆菌科细菌被认为与早产儿的坏死性小肠结肠炎相关,研究表明肠杆菌科菌株可以在含有低聚半乳糖的环境下繁殖,但却无法在含有2’-FL、6’-唾液酸乳糖(6’-SL)或LNnT的环境下存活,表明2’-FL对肠道有害菌具有显著抑制作用[9];Weichert等[10]证明2’-FL和3-FL能够抑制绿脓杆菌和肠道致病型大肠杆菌在肠道和呼吸系统的定殖。2019年德国巴斯夫公司针对婴幼儿营养品市场推出了首款2’-FL产品PREBILAC,并与Glycosyn LLC公司合作将其专利技术应用于婴儿营养以外的肠道健康领域,包括膳食补充剂、功能性营养和医疗食品等。
|
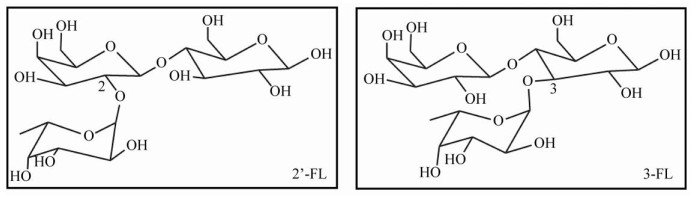
| 图 1 2’-FL和3-FL的结构示意图 Fig. 1 The chemical structures of 2'-FL and 3-FL. |
| |
2’-FL的生物合成路径如图 2所示。2’-FL的生物合成最早通过酶催化来实现,Albermann等[12]在2000年通过外源表达和纯化大肠杆菌Escherichia coli K12来源的Gmd酶(GDP-D-甘露糖-4, 6-脱水酶)和WcaG酶(GDP-4-酮-6-脱氧-D-甘露糖-3, 5-差向异构酶-4-还原酶),在体外将GDP-D-甘露糖转化为GDP-L-岩藻糖,即2’-FL合成最关键的前体。同时他们发现Gmd的活性能够被GDP-L-岩藻糖显著抑制,因此Gmd是一个较关键的限速步骤酶。岩藻糖基转移酶(Fucosyltransferases,FTs)可以催化GDP-L-岩藻糖的岩藻糖基转糖苷到乳糖上去,FTs大多来源于真核生物,但真核类FTs具有一个膜结合结构域,活性受翻译后修饰影响,因此这类基因在缺乏后修饰能力的原核宿主中难以获得活性表达。直到1997年Ge等[13]发现了第一个原核来源的FTs,来源于幽门螺杆菌Helicobactor pylori。Albermann等[14]进一步表达了幽门螺杆菌来源的FucT2酶,并与Gmd和WcaG联用,以GDP-D-甘露糖为出发底物,乳糖为最终底物,在体外成功合成了2’-FL,其中GDP-D-甘露糖到GDP-L-岩藻糖的转化率为78%,以上研究表明3种关键酶能够通过联用有效合成2’-FL。
纯酶催化仅限于实验室小规模研究,产业化应用还需要将关键酶基因构建到底盘微生物中来完成。已知的合成途径包括Salvage和De novo两种(图 2),其中Salvage途径需要外源添加L-岩藻糖作为合成前体;De novo途径则直接利用D-葡萄糖或甘油为碳源来合成关键前体GDP-L-岩藻糖,进而生产出人乳寡糖。2006年,法国国家科研中心的Drouillard等[15]首次在大肠杆菌中构建出2’-FL合成途径,表达了幽门螺杆菌FutC酶基因,最高产量达到14 g/L,其中胞外含量11 g/L。这是首次采用De novo途径,该途径成为研究的焦点,Lee等[16]在E. coli BL21 star(DE3)菌株中表达了幽门螺杆菌来源的FucT2酶以及ManB、ManC、Gmd、WcaG酶,但该菌株分解乳糖的能力较强,因此难以大量积累2’-FL。更换无法水解乳糖的E. coli JM109(DE3)后,乳糖仅被用于合成2’-FL,产量上升到1.2 g/L,但仅为理论产量的20%。此外,作者发现FucT2表达后可溶性很差,多为包涵体,因此解决该酶的可溶性问题将提高2’-FL的合成能力。例如,Chin等[17]在其N端融合了3个天冬氨酸(D3标签),使得酶可溶性提升,酶活力提高了3.4倍,这种添加负电荷氨基酸标签的方式早先也被用于提高脂肪酶在大肠杆菌中的可溶性表达,获得了较好的效果,因而具备一定的普适性。
由于E. coli JM109(DE3)菌株存在一些产业化应用的缺陷,包括生长缓慢、易产乙酸、易形成生物膜等,Chin等[18]尝试使用BL21 star(DE3)菌株,他们使用λ-Red重组技术改造了乳糖操纵子,使得BL21 star(DE3)水解乳糖的能力下降了97%,解决了乳糖易被宿主分解的问题。尽管最终产量并不如JM109(DE3),但每克乳糖对应的2’-FL产率提高了3.6倍。最终通过分批发酵实现了2’-FL产量6.4 g/L,最高细胞干重71.1 g/L,产率0.118 g/(L·h)。Huang等[11]对不同的大肠杆菌宿主进行了比较,结果表明对2’-FL而言JM109(DE3)产量最高,对3-FL而言JM109(DE3)和NovaBlue(DE3)产量最高,而这两种宿主都不能利用乳糖,因此避免了乳糖分解。另一方面,加强乳糖的转运能力对提高2’-FL产量有较大帮助,已有报道通过表达乳酸克鲁维酵母来源的乳糖转运蛋白基因lac12来实现该目标。
补救途径(Salvage pathway)也获得了较多的关注,Baumgärtner等[18]利用λ-Red重组技术整合2’-FL表达元件到JM109的基因组,表达元件包括gmd、wcaG、manB、manC、futC (双拷贝)和fkp基因,每个基因单独配置tac启动子,整个体系没有使用质粒,因此无需使用抗生素。重组菌以甘油为碳源,岩藻糖和乳糖为底物,合成2’-FL获得了20.28 g/L的产量,产率0.57 g/(L·h)。由于大肠杆菌本身具有L-岩藻糖的利用途径,因而造成胞内L-岩藻糖的损耗;Chin等[19]在E. coli BL21 star(DE3)菌株中敲除了fucI和fucK两个L-岩藻糖利用途径的关键酶基因,敲除了β-半乳糖苷酶基因lacZ,同时用质粒表达了fkp和fucT2两个基因,成功以甘油为碳源将2’-FL产量提高到23.1 g/L,产率达到0.389 g/(L·h),2’-FL和L-岩藻糖的摩尔转化比为每摩尔L-岩藻糖转化出0.36 mol 2’-FL。岩藻糖在大肠杆菌胞内有代谢途径,该途径会显著影响菌体的生长速率,因此敲除该途径的3个关键基因fucI (表达L-岩藻糖异构酶)、fucK (表达L-墨角藻糖激酶)、fucA (表达L-墨角藻糖-1-磷酸醛缩酶)将重组菌生长速率提高了14%–78%。进一步地,在此基础上Jung等[20]敲除了araA和rhaA两个基因(分别表达L-阿拉伯糖异构酶和鼠李糖异构酶),使得重组菌能够产出47 g/L的2’-FL,是文献报道的最高产量;产率也达到最高的0.6 g/(L·h)。然而作者发现L-岩藻糖到2’-FL的摩尔转化率为0.52,表明仍然有接近一半的L-岩藻糖被其他代谢途径消耗,造成了浪费。
Salvage途径具有技术局限性,原因是L-岩藻糖价格昂贵且难以获得,产业化难度较大[3]。相比较而言,De novo途径不引入L-岩藻糖而以葡萄糖或甘油为碳源合成2’-FL前体GDP-L-岩藻糖。显然,De novo途径的增产需要足够的胞内GDP-L-岩藻糖。为了实现过量积累,多个课题组优化了GDP-L-岩藻糖合成途径。Byun等[21]优化大肠杆菌de novo途径产GDP-L-岩藻糖的产量为55.2 mg/L,共表达了gmd、wcaG、zwf三个基因;其中zwf基因属于磷酸戊糖途径,编码负责NADPH再生的葡萄糖-6-磷酸脱氢酶(G6PDH),从而为WcaG催化的反应供应更多的NADPH。Lee等[22]通过在大肠杆菌中表达gmd、wcaG、manB、manC获得了170.3 mg/L的GDP-L-岩藻糖含量,是仅表达gmd和wcaG对照组的4.4倍,表明manB和manC的过表达较为重要。但继续表达manA并没有进一步增强GDP-L-岩藻糖的产量,表明该基因无需过表达。大肠杆菌的最高GDP-L-岩藻糖积累量为235.2 mg/L,是通过表达gmd、wcaG、manB、manC、g6pdh、icd、maeB七个基因实现的,其中G6PDH、Icd、MaeB均负责产生还原力NADPH,十分有利于GDP-L-岩藻糖的积累[23]。Koizumi等[24]利用大肠杆菌和产氨短杆菌Corynebacterium ammoniagenes来混菌发酵,产氨短杆菌具有很强的转化GMP为GTP的能力,GTP被大肠杆菌中的ManC酶催化产生GDP-D-甘露糖,在经过Gmd和WcaG的作用形成GDP-L-岩藻糖,并通过FucT催化与N-乙酰-D-乳糖胺(LacNAc)反应生成终产物Lewis X (也称CD15),这步反应释放的GDP再次通过产氨短杆菌转化为GTP;该混菌体系获得了惊人的18.4 g/L的反应中间体GDP-L-岩藻糖,最终收获了21 g/L的Lewis X,这为2’-FL和3-FL的大量合成提供了一种新思路,但混菌发酵较为复杂,研究难度大,因此尚未见更多的应用报道。3-FL的合成研究较2’-FL少,Jung等[25]通过表达幽门螺杆菌FutA,实现了3-FL的从头生物合成(de novo),其表达宿主基于大肠杆菌BL21 star(DE3) ∆L-YA菌株,作者发现以甘油为碳源的3-FL产量要远高于葡萄糖为碳源,并且敲除了可拉酸合成途径的关键基因wcaJ后菌株∆LW-YA中3-FL产量达到了11.5 g/L,摩尔得率0.39 mol/mol,产率0.22 g/(L·h)。Choi等[26]发现BL21(DE3)乙酸产生量更低,因此更适合生产3-FL。结合上述报道,2’-FL和3-FL的生物制备工艺总结为表 1,对中间体GDP-L-岩藻糖的增强合成工艺总结为表 2。
| Microbial hosts and pathway selection |
Expressed genes |
Deleted genes |
Titer (g/L) |
Productivity g/(L·h) (fermentation time) |
Molar yield (mol product/ mol substrate) |
References |
| E. coli BL21(DE3) (salvage) |
fkp, gsk, mfutA | lacZ∆m15*, fucIK | 3-FL, 4.6 (extracellular) |
0.076 (60 h) |
0.33 (mol 3-FL/mol lactose) | [26] |
| E. coli JM109 (salvage) | fkp, futC, gmd, wcaG, manB, manC | 2’-FL, 20.28 (50% for extracellular) |
0.57 (35.5 h) |
NR | [18] | |
| E. coli BL21star(DE3) (salvage) |
fkp, fucT2 | fucI, fucK, lacZ | 2’-FL, 23.1 (extracellular) |
0.389 (> 70 h) |
0.36 (mol 2’-FL/mol fucose) | [19] |
| E. coli BL21star(DE3) (salvage) |
fkp, fucT2 | araA, rhaA, fucI, fucK, lacZ | 2’-FL, 47 (extracellular) |
0.60 (80 h) |
0.52 (mol 2’-FL/mol fucose) | [20] |
| E. coli JM109(DE3) (de novo) |
manB, manC, gmd, wcaG, fucT2 | 2’-FL, 1.23 (extracellular) |
0.013 (96 h) |
0.09 (g 2’-FL/g lactose) |
[16] | |
| E. coli BL21star(DE3) (de novo) |
manB, manC, gmd, wcaG, fucT2 (fus aspartic acid tag) | lacZ∆m15* | 2’-FL, 6.4 (extracellular) |
0.118 (78 h) |
0.225 (g 2’-FL/g lactose) |
[17] |
| E. coli BL21(DE3) (de novo) |
lacY, rcsA, zwf, icd, gnd, maeB, pntAB, udhA, gapC, gapN | lacZ, ion, wcaJ | 2’-FL, 9.12 3-FL, 12.43 (50% for extracellular) |
NR | NR | [11] |
| E. coli (de novo) | futC, rcsA | 2’-FL, 14 (intracellular 3, extracellular 11) | 0.31 (45 h) |
NR | [15] | |
| E. coli ΔL M15 (de novo) |
manB, manC, gmd, wcaG, wcfB | lacZ | 2’-FL, 15.4 (extracellular) |
0.53 (46 h) |
0.6 (mol 2’-FL/ mol lactose) | [28] |
| E. coli BL21star(DE3) (de novo) |
gmd, wcaG, manB, manC | lacZ∆m15* | 2’-FL, 0.49 (flask, 87.7% for extracellular) |
0.007 (72 h) |
NR | [40] |
| E. coli | NR | NR | 3-FL, 0.26 | NR | NR | [32] |
| E. coli BL21star(DE3) (de novo) |
futC, gmd, wcaG, manB, manC | lacZ, wcaj | 3-FL, 11.5 (extracellular) |
0.22 (65 h) |
0.39 (mol 3-FL/mol lactose) | [25] |
| Saccharomyces cerevisiae (salvage) | lac12, fkp, fucT2 | 2’-FL, 0.5 (25% for extracellular) |
0.004 (120 h) |
0.63 (mol 2’-FL/mol fucose) | [27] | |
| Saccharomyces cerevisiae (de novo) | lac12, gmd, wcaG, fucT2 | 2’-FL, 0.56 (75% for extracellular) |
0.0058 (96 h) |
0.229 (mol 2’-FL/mol lactose) | [34] | |
| Saccharomyces cerevisiae (de novo)
Yarrowia lipolytica (de novo) |
NR | NR | 2’-FL 15 24 |
0.22 (68 h) 0.44 (55 h) |
NR | [35] |
| B. subtilis (salvage) | lac12, fkp, fucT, glcP, several genes for GTP synthesis | yesZ | 2’-FL, 5.01 | 0.1 (48 h) |
0.85 (mol 2’-FL/mol fucose) | [36] |
| *: lacZ was knocked-out partially. | ||||||
| Microbial hosts | Expressed genes | GDP-L-fucose concentration | Productivity | References |
| E. coli (de novo) | gmd, wcaG, zwf | 55.2 mg/L | 2.5 mg/(L·h) | [21] |
| C. glutamicum (de novo) | gmd, wcaG, manB, manC | 86.2 mg/L | NR | [37] |
| E. coli (salvage) | fkp, gpt, gmk, ndk | 122.0 mg/L | NR | [41] |
| E. coli (de novo) | gmd, wcaG, manB, manC | 170.3 mg/L | 6.7 mg/(L·h) | [22] |
| E. coli (de novo) | gmd, wcaG, manB, manC, G6PDH (zwf), icd, maeB | 235.2 mg/L | NR | [23] |
| E. coli and Corynebacterium ammoniagenes (mixed fermentation) | gmd, wcaG, manB, manC | 18.4 g/L | NR | [24] |
| NR: not reported. | ||||
2’-FL和3-FL的合成关键酶较多,主要包括Gmd、WcaG、1, 2-FT、1, 3-FT等。近年来,针对以上关键酶的酶源挖掘与分子改造研究逐步深入。多篇文献针对关键酶酶源进行了挖掘与筛选分析,其中研究最为详细的是1, 2-FT酶(α1, 2-岩藻糖基转移酶)。南开大学的Huang等[11]对α1, 2-岩藻糖基转移酶基因futC、futL、futF、wbsJ、wbwK、wbgL、wbiQ、futG、futN、wcfB、wcfW进行了比较,结果表明来源于幽门螺杆菌的futC效果最好。此外他们还对futB、futA、futD、futE、futH、futJ、futK、futM等1, 3-FT基因(α1, 3-岩藻糖基转移酶)进行了比较研究,结果表明表达FutA酶获得的3-FL产量最高。Albermann等和Yu等[14, 27]发现FucT2酶的底物特异性不强,会造成副产物积累,例如在重组酵母以及大肠杆菌合成2’-FL过程中测到了乳糖二岩藻糖四糖(Difucosyllactose),这是由于FucT2酶以第一次合成出的2’-FL为底物,二次转糖苷形成了四糖产物。与FucT2酶相比较,WcfB酶的底物特异性要强得多,利用WcfB酶催化的产物中没有发现副产物,且2’-FL的产量提高了[28];这表明有必要对α1, 2-岩藻糖基转移酶的底物特异性进行研究,提高合成效率。关键酶的改造研究对于提高酶的可溶性以及产物合成能力至关重要,已报道文献主要针对3-FL合成酶1, 3-FT。2006年Lin等和Ma等[29-30]分别研究了幽门螺杆菌来源1, 3-FT酶的可溶性问题。研究表明,改善关键酶的可溶性能够有效提升3-FL产量,Choi等[31]也发现幽门螺杆菌1, 3-FT酶可溶性可以通过密码子优化和截短C末端来获得大幅提升,截短体∆52的蛋白可溶性上升到0.15–0.20 g/L,3-FL初筛产量提高了18倍。进一步利用HotSpot Wizard在线预测来定点突变获得了突变体A128N,活性又提高3.4倍,最终构建的∆52四位点突变体A128N/H129E/Y132I/S46F是野生型酶催化效率的近20倍(对底物乳糖的kcat/Km值达到4.97 L/(mmol·min)),这表明截短体是增强这类酶催化活性的一种途径。Yu等[32]对幽门螺杆菌来源的3种1, 3-FTs (ft1、ft2、pylT)进行了研究,这类酶是细胞膜结合的二聚体蛋白,C末端都具有重复型七肽结构(2–10个)来保持二聚体结构,还具有一个细胞膜铆钉区域。当增加重复七肽数量以及去除膜结合区域后二聚体结构更加稳定且活性上升,使得3-FL产量能够提升10–20倍。除截短改造外,基于高通量筛选的定向进化方法也是提高关键酶产物合成能力的有效手段。上海交通大学研究团队对3-FL合成酶FutA进行了定向进化研究[33],利用荧光激活细胞分离仪FACS技术对3-FL进行标记,以易错PCR产生的大肠杆菌单克隆的胞内3-FL含量作为筛选指标;经过3轮筛选获得了突变体M32 (7点突变:S45F/ D127N/R128E/H131I/Y199N/E340D/V368A),该突变体的3-FL合成能力提升了14倍(kcat/Km值由2.53 L/(mmol·s)提升到15.59 L/(mmol·s)),其主要原因是突变体的Km值大幅下降,仅为野生型酶的9.2% (9.2 mmol/L)。晶体结构解析和分子动力学MD模拟分析表明S45F突变在活性中心形成了一个钳形结构,增强了对半乳糖基团的亲和力,而α螺旋α5的灵活性获得提升,可能增强了其岩藻糖糖基化能力。综上所述,2’-FL和3-FL合成关键酶在新酶源挖掘与比较、酶分子改造等方面已取得了成果,为提升人乳寡糖产量奠定基础。
4 利用食品安全型底盘微生物合成2’-FL由于大肠杆菌会分泌内毒素(热源)且极易感染噬菌体,因此开发食品安全型微生物作为细胞工厂来生产2’-FL具有重要意义。相比较而言,酵母已被人类使用几千年非常安全,不易被噬菌体感染,是食药品行业非常有竞争力的宿主微生物,常用的酵母底盘包括酿酒酵母、毕赤酵母、解脂耶氏酵母等。伊利诺伊大学香槟分校的Liu等[34]首先在酿酒酵母中构建了2’-FL的de novo合成途径,成功合成出了2’-FL,产量0.51 g/L,作者还通过表达一个α-岩藻糖苷酶基因将2’-FL水解为L-岩藻糖,从而获得了0.41 g/L的L-岩藻糖。尽管产量很低,但还是对于酵母底盘的应用作了探索性贡献。Yu等[27]在酿酒酵母中表达乳糖透过酶基因lac12、fkp、fucT2,发酵120 h获得了0.5 g/L的2’-FL,但75%的2’-FL被发现位于胞内,仅有25%分泌到了胞外,而在大肠杆菌也只有不到50%的2’-FL能够分泌到胞外。2019年,杜邦公司的Hollands等[35]分别开发了酿酒酵母和解脂耶氏酵母两个底盘来尝试de novo途径,其中酿酒酵母底盘做到了15 g/L产量,产率0.22 g/(L·h),发酵时间约70 h;解脂耶氏酵母更是达到了24 g/L,产率0.44 g/(L·h),发酵时间不到60 h,该研究是食品级宿主生产2’-FL的较大突破,为产业化应用提供了可能。当然,酵母底盘也存在一些缺点,例如发酵延滞期长、总周期长等。枯草芽孢杆菌是安全性较好的食品级微生物宿主,江南大学研究团队尝试利用重组枯草芽孢杆菌生产2’-FL,采用了补救途径。通过提高底物L-岩藻糖和乳糖的转运(过表达glcP和lac12基因)、去除乳糖水解途径(敲除yesZ基因)、引入补救途径关键基因fkp和fucT、增强GTP的再生(调节GTP合成的数个关键基因)这几种方法,将2’-FL在补料发酵中的产量提升到5.01 g/L (合成36 h),是摇瓶实验的4.84倍。L-岩藻糖的摩尔转化率达到最高的0.85,由于理论摩尔转化率为1.0,因此作者认为枯草芽孢杆菌中可能存在未知的L-岩藻糖消耗途径[36]。Chin等[37]在谷氨酸棒杆菌中表达了gmd、wcaG、manB、manC四个基因来构建de novo途径,最终获得了86.2 mg/L的GDP-L-岩藻糖;这是首次在大肠杆菌以外的菌株实现关键前体GDP-L-岩藻糖的积累。综上所述,多个课题组开展了食品级微生物宿主生产2’-FL或3-FL的研究。结果表明酵母、枯草芽孢杆菌、谷氨酸棒杆菌均具备了开发为工业生产菌株的潜力,其中酵母底盘的效果最优。
5 对2’-FL运出蛋白的研究由于胞内合成的2’-FL有时难以有效地被输送到胞外,导致部分残留在胞内(大部分文献中残留率达50%),需要破胞才能获得所有2’-FL,这对下游产物分离和提取造成了很大困难,这也是目前的一大技术瓶颈。Hollands等[35]对2’-FL的转运蛋白进行了系统研究,他们依据3个基本原则,包括:(1)挑选一切有可能转运2’-FL的转运蛋白,最终锁定大肠杆菌SetA为参考对象,使用隐马尔可夫模型(HMM)检索到数个候选蛋白。(2)挑选真菌来源能够转运二糖、三糖、四糖的转运蛋白,最终锁定粗糙脉孢菌Neurospora crassa来源的CDT1和CDT2转运蛋白。(3)不依赖ATP水解、能够在同向和反向协同过程中保持质子或金属离子动态平衡的转运蛋白,这让他们锁定植物源转运蛋白SWEET家族。根据以上原则筛选了27种不同的转运蛋白,结果表明CDT2和大肠杆菌SetA对2’-FL的转运效果最好,SWEET家族蛋白和CDT1则没有效果,但SetA会降低菌体生长速率,因此最终选择CDT2作为酵母底盘的2’-FL转运蛋白,通过荧光显微镜可以观察到CDT2在细胞膜上的聚集,酿酒酵母合成2’-FL的胞外/胞内含量比例提高到约2.5倍(对照组不到1.5倍),解脂耶氏酵母的该比例由不到1倍提升到1.5倍以上,为2’-FL的产物释放提供了有力借鉴;然而,对大肠杆菌的人乳寡糖转出蛋白研究还未见。
6 2’-FL的专利保护情况截止目前,2’-FL和3-FL的发明专利情况如表 3所示。中国发明专利获得授权的较少,国际专利申请人包括德国巴斯夫公司、丹麦Glycom公司、美国杜邦公司、美国雅培制药公司、美国Glycosyn公司、德国Jennewein Biotechnologie公司等知名HMO供应商,专利内容包括2’-FL和3-FL的发酵合成、关键酶改造、产品分离纯化、产品结晶、产品应用等多个方面。其中多数专利由欧洲企业申报,表明欧洲已成为人乳寡糖产品的研发与生产中心。
| Patent title | Patent number | Patent applicant | Application time |
| 促进有益微生物群生长的益生菌和人乳寡糖的合益素组合(授权中国发明专利) | CN201180068703.2 | Abbott Laboratories | 2011/12 |
| Increasing activity of 2’-fucosyllactose transporters endogenous to microbial cells | US201815959573 | DuPont Nutrition & Biosciences | 2018/04 |
| Hydrated and anhydrous polymorphs of 2’-fucosyllactose and their production methods | US201916393866 | BASF SE | 2019/04 |
| Use of purified 2’-fucosyllactose, 3-fucosyllactose and lactodifucotetraose as prebiotics | US201916403095 | Glycosyn LLC Trustees Boston College | 2019/05 |
| Process for purification of neutral human milk oligosaccharide using simulated moving bed chromatography | US201916440100 | Jennewein Biotechnologie GmbH | 2019/06 |
| Separation of 2’-O-fucosyllactose from fermentation broth | WO2015DK50158 | Glycom AS | 2015/06 |
| Crystalline 3-O-fucosyllactose | US201314442017 | Glycom AS | 2013/09 |
| 2’-Fucosyllactose producing mutant microorganisms and method for producing 2’-fucosyllactose using same | WO2016KR03013 | Seoul National University R & db Foundation | 2016/03 |
| Method for the production of fucosyllactose in bacterial cells | US201314100825 | Jennewein Biotechnologie GmbH | 2013/12 |
| Polymorphs of 2’-O-fucosyllactose and producing thereof | PCT/DK2011/050192 | Glycom AS | 2011/06 |
| Crystalline 3-O-fucosyllactose | PCT/DK2013/050300 | Glycom A/S | 2013/09 |
| *: Searched in European Patent Office (EPO) database. | |||
近期丹麦Glycom公司的Agoston等[38]也报道了化学合成2’-FL的工艺,并获得了公斤级的高纯度产品,该路线是将乳糖经过2步反应获得中间体3、岩藻糖经过4步反应获得中间体8,3和8再经过4步反应得到2’-FL,所有的反应步骤均在接近常温的条件下完成,能耗相对经济。尽管总得率不高,但化学工艺在生产稳定性上往往占有优势,因此化学法的进步也对人乳寡糖的生物法研发提出了更高的要求,但生物制备技术仍有瓶颈问题待进一步突破:(1)大幅提升关键酶的活力和可溶性,尤其是2’-FL和3-FL合成关键酶,都存在外源表达可溶性差、酶活性低的问题,探索其与分子伴侣的适配性来提升表达效果,开发新酶源替代现有酶极为重要,而这其中必须借助高通量的筛选技术并对基因资源库进行充分探索。(2)将关键酶基因整合到宿主染色体,避免基于抗生素的质粒体系带来的生产不稳定性以及食品安全问题,但这其中伴随着因染色体基因拷贝数降低而产量下降的可能,需要更多的实验来佐证效果。(3)增强底盘宿主对2’-FL和3-FL的胞外分泌能力,筛选更好的产物运输蛋白,免去破胞步骤,减小下游产品分离提取的难度,这是目前最应该解决的瓶颈问题。(4)进一步提升底盘微生物胞内合成GDP-L-岩藻糖的能力,改造其合成途径的关键酶,例如解决Gmd酶的产物抑制作用,大幅提升限速酶的合成力。(5)开发高效的2’-FL和3-FL下游分离提取技术,降低分离成本并提高产物提取收率。
8 展望国外如韩国、欧盟等对人乳寡糖的研究已非常深入,国内对2’-FL和3-FL的研究起步较晚,但近期关注度很高。由于对人乳寡糖的重视程度与日俱增,2016年国家食品安全风险评估中心(CFSA)已对2’-FL作为食品添加剂新品种公开征求意见,因此2’-FL的法规出台有望提上日程。尽管欧洲对大肠杆菌生产2’-FL持开放态度[39],开发食品安全级别的宿主(如具有GRAS认证或国标GB2760认证)仍然很有必要,尤其是开发酵母、谷氨酸棒杆菌、枯草芽孢杆菌等食品行业非常成熟的底盘微生物。此外,对于转基因菌种的使用在法规上较为模糊,有待国家相关部门和企业的产业标准出台。从技术角度来看,对合成途径中的新酶挖掘与改造至关重要,需要生物信息学、基因组学、高通量筛选等技术的介入,例如近年来兴起的蛋白质从头设计法(De novo design)和基于微流控的荧光激活细胞分选技术(FACS)可分别从理性设计和定向进化两种角度深入研究。在知识产权方面,应尽快建立专利壁垒,形成我国自有知识产权的HMO生产技术。由于中国人普遍存在乳糖不耐受症,故乳糖是不受欢迎的乳业废弃物,未经处理排放还会造成严重水体污染,因此开发HMO将极大提升乳糖的利用附加值,具有重要的经济价值与乳品产业升级意义。
| [1] |
Zhai YF, Yu X, Xiang QS, et al. Research progress of human milk oligosaccharides synthesis in vitro. Sci Technol Food Ind, 2018, 39(5): 348-352 (in Chinese). 翟娅菲, 禹晓, 相启森, 等. 人乳寡糖体外合成研究进展. 食品工业科技, 2018, 39(5): 348-352. |
| [2] |
Han NS, Kim TJ, Park YC, et al. Biotechnological production of human milk oligosaccharides. Biotechnol Adv, 2012, 30(6): 1268-1278. DOI:10.1016/j.biotechadv.2011.11.003 |
| [3] |
Bych K, Mikš MH, Johanson T, et al. Production of HMOs using microbial hosts—from cell engineering to large scale production. Curr Opin Biotech, 2019, 56: 130-137. |
| [4] |
Grollman AP, Hall CW, Ginsburg V. Biosynthesis of fucosyllactose and other oligosaccharides found in milk. J Biol Chem, 1965, 240(3): 975-981. |
| [5] |
Sprenger GA, Baumgärtner F, Albermann C. Production of human milk oligosaccharides by enzymatic and whole-cell microbial biotransformations. J Biotechnol, 2017, 258: 79-91. DOI:10.1016/j.jbiotec.2017.07.030 |
| [6] |
Anderson A, Donald ASR. Improved method for the isolation of 2'-fucosyllactose from human milk. J Chromatogr A, 1981, 211(1): 170-174. |
| [7] |
Vandenplas Y, Berger B, Carnielli VP, et al. Human milk oligosaccharides: 2'-fucosyllactose (2'-FL) and lacto-N-neotetraose (LNnT) in infant formula. Nutrients, 2018, 10(9): 1161. DOI:10.3390/nu10091161 |
| [8] |
Reverri EJ, Devitt AA, Kajzer JA, et al. Review of the clinical experiences of feeding infants formula containing the human milk oligosaccharide 2'-fucosyllactose. Nutrients, 2018, 10(10): 1346. DOI:10.3390/nu10101346 |
| [9] |
Facinelli B, Marini E, Magi G, et al. Breast milk oligosaccharides: effects of 2'-fucosyllactose and 6'-sialyllactose on the adhesion of Escherichia coli and Salmonella fyris to Caco-2 cells. J Matern-Fetal Neo Med, 2019, 32(17): 2950-2952. DOI:10.1080/14767058.2018.1450864 |
| [10] |
Weichert S, Jennewein S, Hüfner E, et al. Bioengineered 2'-fucosyllactose and 3-fucosyllactose inhibit the adhesion of Pseudomon as aeruginosa and enteric pathogens to human intestinal and respiratory cell lines. Nutr Res, 2013, 33(10): 831-838. DOI:10.1016/j.nutres.2013.07.009 |
| [11] |
Huang D, Yang KX, Liu J, et al. Metabolic engineering of Escherichia coli for the production of 2'-fucosyllactose and 3-fucosyllactose through modular pathway enhancement. Metab Eng, 2017, 41: 23-38. DOI:10.1016/j.ymben.2017.03.001 |
| [12] |
Albermann C, Distler J, Piepersberg W. Preparative synthesis of GDP-β-l-fucose by recombinant enzymes from enterobacterial sources. Glycobiology, 2000, 10(9): 875-881. DOI:10.1093/glycob/10.9.875 |
| [13] |
Ge ZM, Chan NW, Palcic MM, et al. Cloning and heterologous expression of an α1, 3-fucosyltransferase gene from the gastric pathogen Helicobacter pylori. J Biol Chem, 1997, 272(34): 21357-21363. DOI:10.1074/jbc.272.34.21357 |
| [14] |
Albermann C, Piepersberg W, Wehmeier UF. Synthesis of the milk oligosaccharide 2'-fucosyllactose using recombinant bacterial enzymes. Carbohyd Res, 2001, 334(2): 97-103. DOI:10.1016/S0008-6215(01)00177-X |
| [15] |
Drouillard S, Driguez H, Samain E. Large-scale synthesis of H-antigen oligosaccharides by expressing Helicobacter pylori α1, 2-fucosyltransferase in metabolically engineered Escherichia coli cells. Angew Chem Int Edit, 2006, 45(11): 1778-1780. DOI:10.1002/anie.200503427 |
| [16] |
Lee WH, Pathanibul P, Quarterman J, et al. Whole cell biosynthesis of a functional oligosaccharide, 2'-fucosyllactose, using engineered Escherichia coli. Microb Cell Fact, 2012, 11: 48. DOI:10.1186/1475-2859-11-48 |
| [17] |
Chin YW, Kim JY, Lee WH, et al. Enhanced production of 2'-fucosyllactose in engineered Escherichia coli BL21star (DE3) by modulation of lactose metabolism and fucosyltransferase. J Biotechnol, 2015, 210: 107-115. DOI:10.1016/j.jbiotec.2015.06.431 |
| [18] |
Baumgärtner F, Seitz L, Sprenger GA, et al. Construction of Escherichia coli strains with chromosomally integrated expression cassettes for the synthesis of 2'-fucosyllactose. Microb Cell Fact, 2013, 12: 40. DOI:10.1186/1475-2859-12-40 |
| [19] |
Chin YW, Seo N, Kim JH, et al. Metabolic engineering of Escherichia coli to produce 2'-fucosyllactose via salvage pathway of guanosine 5'-diphosphate (GDP)-L-fucose. Biotechnol Bioeng, 2016, 113(11): 2443-2452. DOI:10.1002/bit.26015 |
| [20] |
Jung SM, Chin YW, Lee YG, et al. Enhanced production of 2'-fucosyllactose from fucose by elimination of rhamnose isomerase and arabinose isomerase in engineered Escherichia coli. Biotechnol Bioeng, 2019, 116(9): 2412-2417. DOI:10.1002/bit.27019 |
| [21] |
Byun SG, Kim MD, Lee WH, et al. Production of GDP-L-fucose, L-fucose donor for fucosyloligosaccharide synthesis, in recombinant Escherichia coli. Appl Microbiol Biotechnol, 2007, 74(4): 768-775. |
| [22] |
Lee WH, Han NS, Park YC, et al. Modulation of guanosine 5'-diphosphate-d-mannose metabolism in recombinant Escherichia coli for production of guanosine 5'-diphosphate-l-fucose. Bioresour Technol, 2009, 100(24): 6143-6148. DOI:10.1016/j.biortech.2009.07.035 |
| [23] |
Lee WH, Chin YW, Han NS, et al. Enhanced production of GDP-L-fucose by overexpression of NADPH regenerator in recombinant Escherichia coli. Appl Microbiol Biot, 2011, 91(4): 967-976. DOI:10.1007/s00253-011-3271-x |
| [24] |
Koizumi S, Endo T, Tabata K, et al. Large-scale production of GDP-fucose and Lewis X by bacterial coupling. J Ind Microbiol Biot, 2000, 25(4): 213-217. DOI:10.1038/sj.jim.7000055 |
| [25] |
Jung SM, Park YC, Seo JH. Production of 3-fucosyllactose in engineered Escherichia coli with α-1, 3-fucosyltransferase from Helicobacter pylori. Biotechnol J, 2019, 14(6): 1800498. DOI:10.1002/biot.201800498 |
| [26] |
Choi YH, Park BS, Seo JH, et al. Biosynthesis of the human milk oligosaccharide 3-fucosyllactose in metabolically engineered Escherichia coli via the salvage pathway through increasing GTP synthesis and β-galactosidase modification. Biotechnol Bioeng, 2019, 116(12): 3324-3332. DOI:10.1002/bit.27160 |
| [27] |
Yu S, Liu JJ, Yun EJ, et al. Production of a human milk oligosaccharide 2'-fucosyllactose by metabolically engineered Saccharomyces cerevisiae. Microb Cell Fact, 2018, 17: 101. DOI:10.1186/s12934-018-0947-2 |
| [28] |
Chin YW, Kim JY, Kim JH, et al. Improved production of 2'-fucosyllactose in engineered Escherichia coli by expressing putative α-1, 2-fucosyltransferase, WcfB from Bacteroides fragilis. J Biotechnol, 2017, 257: 192-198. DOI:10.1016/j.jbiotec.2016.11.033 |
| [29] |
Lin SW, Yuan TM, Li JR, et al. Carboxyl terminus of Helicobacter pylori α1, 3-fucosyltransferase determines the structure and stability. Biochemistry, 2006, 45(26): 8108-8116. DOI:10.1021/bi0601297 |
| [30] |
Ma B, Audette GF, Lin SJ, et al. Purification, kinetic characterization, and mapping of the minimal catalytic domain and the key polar groups of Helicobacter pylori α-(1, 3/1, 4)-fucosyltransferases. J Biol Chem, 2006, 281(10): 6385-6394. DOI:10.1074/jbc.M511320200 |
| [31] |
Choi YH, Kim JH, Park BS, et al. Solubilization and iterative saturation mutagenesis of α-1, 3-fucosyltransferase from Helicobacter pylori to enhance its catalytic efficiency. Biotechnol Bioeng, 2016, 113(8): 1666-1675. DOI:10.1002/bit.25944 |
| [32] |
Yu J, Shin J, Park M, et al. Engineering of α-1, 3-fucosyltransferases for production of 3-fucosyllactose in Escherichia coli. Metab Eng, 2018, 48: 269-278. DOI:10.1016/j.ymben.2018.05.021 |
| [33] |
Tan YM, Zhang Y, Han YB, et al. Directed evolution of an α-1, 3-fucosyltransferase using a single-cell ultrahigh-throughput screening method. Sci Adv, 2019, 5(10): eaaw8451. DOI:10.1126/sciadv.aaw8451 |
| [34] |
Liu JJ, Kwak S, Pathanibul P, et al. Biosynthesis of a functional human milk oligosaccharide, 2'-fucosyllactose, and L-fucose using engineered Saccharomyces cerevisiae. ACS Synth Biol, 2018, 7(11): 2529-2536. DOI:10.1021/acssynbio.8b00134 |
| [35] |
Hollands K, Baron CM, Gibson KJ, et al. Engineering two species of yeast as cell factories for 2'-fucosyllactose. Metab Eng, 2012, 52: 232-242. |
| [36] |
Deng JY, Gu LY, Chen TC, et al. Engineering the substrate transport and cofactor regeneration systems for enhancing 2'-fucosyllactose synthesis in Bacillus subtilis. ACS Synth Biol, 2019, 8(10): 2418-2427. DOI:10.1021/acssynbio.9b00314 |
| [37] |
Chin YW, Park JB, Park YC, et al. Metabolic engineering of Corynebacterium glutamicum to produce GDP-L-fucose from glucose and mannose. Bioproc Biosyst Eng, 2013, 36(6): 749-756. DOI:10.1007/s00449-013-0900-z |
| [38] |
Agoston K, Hederos MJ, Bajza I, et al. Kilogram scale chemical synthesis of 2'-fucosyllactose. Carbohyd Res, 2019, 476: 71-77. DOI:10.1016/j.carres.2019.03.006 |
| [39] |
EU approved lactose-N-neotetrasaccharide as a new food ingredient. Chin J Food Hyg, 2016, 28(2): 262 (in Chinese). 欧盟批准乳糖-N-新四糖等作为新食品成分使用.中国食品卫生杂志, 2016, 28(2): 262. http://www.chinabeverage.org/standard/show/detail.php?id=7818. |
| [40] |
Seydametova E, Yu J, Shin J, et al. Search for bacterial α-1, 2-fucosyltransferases for whole-cell biosynthesis of 2'-fucosyllactose in recombinant Escherichia coli. Microbiol Res, 2019, 222: 35-42. DOI:10.1016/j.micres.2019.02.009 |
| [41] |
Zhai YF, Han DL, Pan Y, et al. Enhancing GDP-fucose production in recombinant Escherichia coli by metabolic pathway engineering. Enzyme Microb Technol, 2015, 69: 38-45. DOI:10.1016/j.enzmictec.2014.12.001 |
 2020, Vol. 36
2020, Vol. 36