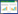中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王弋
- Wang Yi
- 槲皮素与DNA的相互作用
- Interaction between quercetin and DNA
- 生物工程学报, 2020, 36(12): 2877-2891
- Chinese Journal of Biotechnology, 2020, 36(12): 2877-2891
- 10.13345/j.cjb.200127
-
文章历史
- Received: March 18, 2020
- Accepted: August 19, 2020
DNA是生命体最重要的4种生物大分子之一,是遗传信息的载体和基因的物质基础,它在生命体的组成发展与存活中占有举足轻重的地位。有机小分子,尤其是药物分子与DNA之间的相互作用,在分子生物学高度发展的今天,已是疾病治疗与药物设计的重要研究方向,因为有机小分子在体内结合DNA后可能影响相应基因的表达。本文的研究也是基于这样的研究热点,设计出对有机小分子的分析流程或者分析模型,用来检测它们与DNA之间的相互作用,完成有机小分子在分子水平与DNA的相互作用模式分析。本文选用槲皮素和鲱鱼精DNA作为研究对象,发现二者间存在相互作用并证明了二者体外相互作用的结合模式,也同时证明了本文所采用的光谱学结合分子对接分析的研究策略,对于研究有机小分子与DNA之间的体外作用方式是行之有效的。
既往的研究结果认为:当有机小分子与DNA发生嵌插作用时,其二者结合反应后的紫外可见吸收光谱对各自的紫外-可见吸收光谱的最大吸收峰发生红移或蓝移[1-6],且存在增色效应或者减色效应[5-10]。当药物分子与DNA发生静电作用时,不会改变紫外-可见吸收光谱的最大吸收峰相应波长,只改变吸收强度[9, 11]。因而紫外-可见吸收光谱法是可以独立作为DNA与有机小分子相互作用模式判断的凭据。
此外,利用Turro等以及Liu等使用荧光光谱法的研究,说明了有机小分子与DNA之间相互作用前后,荧光强度的变化可以作为DNA与有机小分子相互作用存在的证据[12-13]。
电化学分析方法通过研究有机小分子存在与否对体系伏安特性的影响,来判断DNA与有机小分子之间的相互作用[14-20],获得结合位点与结合常数之类的信息,通过信息的进一步处理得出相互作用性质转变过程及相互作用模式[21-26];生物学方法,尤其是凝胶电泳法所获得的药物与DNA结合的动力学数据可以辅助判断DNA的结合位点[26-28],Fiel等[29]认为嵌插作用会导致有机小分子与DNA结合的复合物电泳迁移率明显降低,而二者之间若只是静电作用则对凝胶电泳的迁移率影响不大。
1 材料与方法超纯水(Ultrapure water,UP水),电阻率是18.25兆欧,测量时室温为16 ℃,pH 6.91 (取3次测量的平均值);磷酸二氢钠-磷酸氢二钠缓冲液(0.19 mol/L磷酸二氢钠+0.16 mol/L磷酸氢二钠)的pH为6.97 (取3次测量的平均值),该缓冲液使用UP水配置,磷酸二氢钠与磷酸氢二钠为分析纯;槲皮素,编号4951-10,鲱鱼精DNA,编号D3159-10G购自Sigma公司。
有关实验材料的说明:槲皮素与鲱鱼精DNA在使用前没有进行进一步纯化。使用的鲱鱼精DNA的260/280 nm紫外吸光度的比率是1.89,表明该鲱鱼精DNA样品基本不存在蛋白质污染[30]。鲱鱼精DNA样品的储备液使用UP水配置,终浓度6 g/L。槲皮素样品的储备液使用无水乙醇配置,终浓度1.99×10-2 mol/L,所用无水乙醇为分析纯。每次实验使用的鲱鱼精DNA储备液、槲皮素储备液以及其他试剂都是新鲜配置的。
主要使用的器材设备如下:日立F-7000荧光分光光度仪;Varian CARY-100 Bio紫外-可见分光光度计;石英比色皿;微量移液器(100-1 000 µL),微量移液器(2-20 µL),微量移液器(0.1-1.0 µL);微量注射器;1 mL枪头,20 µL枪头;1 µL枪头;擦镜纸。
1.1 鲱鱼精DNA储备液的配置鲱鱼精DNA的储备液是使用6 mg鲱鱼精DNA用UP水定容至1 mL配制而成的,终浓度为6 g/L。配置时首先将1.5 mL EP管用UP水洗净再用干净的称量纸包裹,再在65 ℃烘箱中烘干。称量鲱鱼精DNA样品时首先将准备好的1.5 mL EP管置于10万分之一的电子天平上清零,然后用准备好的蓝色枪头沾取6 mg鲱鱼精DNA样品并将其置于1.5 mL EP管内。使用0-1 000 µL微量移液器移取UP水至准备好的鲱鱼精DNA样品中定容至1 mL,混匀。得到终浓度为6 g/L鲱鱼精DNA储备液。
1.2 槲皮素储备液的配置终浓度1.99×10-2 mol/L的槲皮素储备液的配置方法与上述鲱鱼精DNA储备液的配置方法相同。但是考虑到槲皮素在UP水中溶解度非常低,所以本文选取了分析纯的无水乙醇溶解槲皮素,来进行槲皮素的储备液配置。已经有文献表明虽然在有机小分子-DNA复合物的溶液中,终浓度高于70%的乙醇会影响DNA的构象,使其从B型构象转变为A型[31],但是终浓度低于25%的乙醇并不会影响DNA的构象[32]。本文在接下来的实验中使用的槲皮素储备液所引入的乙醇量非常少,因此不影响DNA构象。
1.3 分子对接实验方法本试验采用Discovery Studio 4.1进行分子对接研究。对接的对象选择了B型DNA的十二聚物,序列为d (CAGTACGCTACG),槲皮素的结构挑选自Drug-Bank。使用的分析流程是cdocker流程,该流程会通过考核槲皮素与B型DNA十二聚体上相应对接区域的几何形状互补特征进行配对,考察能量互补特征进行匹配,最后输出不同构象条件的槲皮素与鲱鱼精DNA结合前后其吉布斯自由能的状态,从而挑选能量条件最优的个体。
本实验主要目的是验证槲皮素与鲱鱼精DNA之间是否存在相互作用以及这种相互作用的模式是哪一种,对接的B型DNA作为刚性受体与不同的构象状态的槲皮素小分子进行分子对接,最终结果显示出槲皮素与该B型DNA十二聚体是否能够产生相互作用,以及这种相互作用的类型,经计算得出槲皮素与DNA对接前后吉布斯自由能的变化量,以供与上述其他光谱学实验相互印证。
2 结果与分析荧光光谱分析,通过比较鲱鱼精DNA与槲皮素混合前后的发射光变化,认为鲱鱼精DNA与槲皮素存在体外相互作用。共振散射荧光光谱实验分别控制槲皮素或者鲱鱼精DNA的终浓度不变:控制槲皮素浓度不变的实验中,槲皮素终浓度1×10-4 mol/L,鲱鱼精DNA终浓度6×10-3 g/L、1.2×10-2 g/L、1.8×10-2 g/L、2.4×10-2 g/L和3×10-2 g/L;在控制鲱鱼精DNA终浓度不变的实验中,鲱鱼精DNA终浓度4×10-3 g/L,槲皮素终浓度6.6×10-6 mol/L、1.32×10-5 mol/L、1.98×10-5 mol/L、2.64×10-5 mol/L和3.3×10-5 mol/L;紫外可见分光光度法实验采用的是固定鲱鱼精DNA终浓度4×10-2 g/L,逐次滴加入槲皮素使其终浓度依次达到1.98×10-5 mol/L、3.97×10-5 mol/L、5.95×10-5 mol/L、7.94×10-5 mol/L和9.92×10-5 mol/L的试验方法,获得相对应浓度的槲皮素的紫外-可见吸收光谱;荧光偏振实验中,固定槲皮素终浓度为9.92×10-5 mol/L (对应储备液体积15 µL),鲱鱼精DNA终浓度分别为0 g/L (对应储备液体积0 µL)、6×10-3 g/L (对应储备液体积为3 µL)、1.2×10-2 g/L (对应储备液体积为6 µL)、1.8×10-2 g/L (对应储备液体积为9 µL)、3×10-2 g/L (对应储备液体积为15 µL),研究固定槲皮素终浓度,随着鲱鱼精DNA的浓度增加,总体系偏振度的变化;最后通过分子对接实验佐证光谱学结论——槲皮素与DNA之间的体外相互作用属于沟槽嵌合。
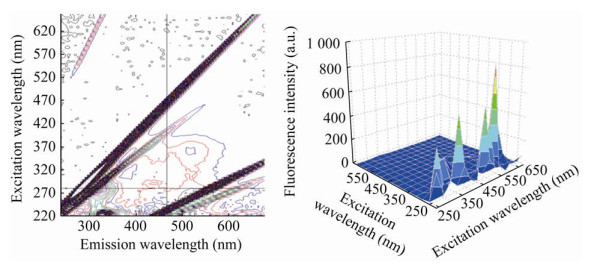
2.1 荧光光谱分析通过三维荧光光谱的扫描结果预实验并结合文献观点,本文选取了激发光波长280 nm,发射光波长为467 nm。三维荧光光谱扫描的参数设定为:激发光的范围220-660 nm,发射光的范围是240-680 nm,狭缝宽度均为5 nm,光电倍增管的电压为820 V。获得的三维荧光光谱中,UP水、加入槲皮素储备液的UP水(槲皮素终浓度6.61×10-5 mol/L)以及加入鲱鱼精DNA的UP水(鲱鱼精DNA终浓度3×10-2 g/L)在280 nm激发光条件下,在467 nm发射光处没有明显的荧光特征峰,但是在鲱鱼精DNA溶液中加入槲皮素(鲱鱼精DNA终浓度3×10-2 g/L,槲皮素终浓度7.94×10-5 mol/L)充分混合并静置10 min之后的混合溶液,在280 nm的激发光条件下产生了在467 nm发射光波长处的较为明显的荧光特征峰。这与文献结论相一致。所以,本文选择激发光波长为280 nm,观察固定槲皮素浓度,改变体系中的鲱鱼精DNA终浓度后的467 nm处的荧光特征峰的变化,来初步确定槲皮素与鲱鱼精DNA体外相互作用的存在。三维荧光光谱预实验结果如图 1-4所示。
|
| 图 1 6.61×10-5 mol/L槲皮素的三维荧光光谱结果 Fig. 1 Three-dimensional fluorescence spectrum of 6.61×10-5 mol/L quercetin at the emitted wavelength of 467 nm and the fluorescence intensity of 40.39 a.u. at the excitation wavelength of 280 nm. |
| |

|
| 图 2 水的三维荧光光谱结果 Fig. 2 Under the condition of 280 nm excitation light wavelength, the result of three-dimensional fluorescence spectrum was obtained at 467 nm emitting light wavelength and the fluorescence intensity was 11.71 a.u.. |
| |
|
| 图 3 3×10-2 g/L鲱鱼精DNA的三维荧光光谱结果 Fig. 3 Three-dimensional fluorescence spectrum of 3×10-2 g/L herring sperm DNA at the excitation wavelength of 280 nm and the emitted wavelength of 467 nm, with an fluorescence intensity of 252.6 a.u.. |
| |

|
| 图 4 3×10-2 g/L DNA+7.94×10-5 mol/L槲皮素的三维荧光光谱结果 Fig. 4 3×10-2 g/L DNA+7.94×10-5 mol/L quercetin at the excitation wavelength of 280 nm, the three-dimensional fluorescence spectrum at the emitted wavelength of 467 nm, with an fluorescence intensity of 437.8 a.u.. |
| |
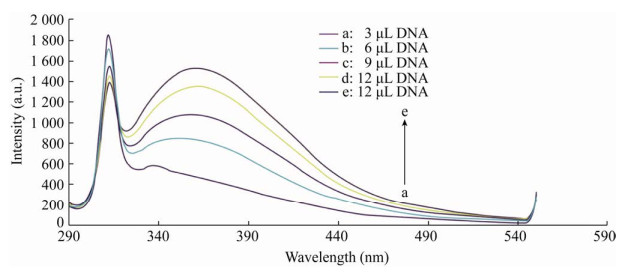
荧光光谱法是以槲皮素能够光致发光为前提,设定280 nm激发光,能够获得其310 nm处的特征发射波长,而其在467 nm处左右发射光相对微弱(图 5)。

|
| 图 5 槲皮素与不同浓度DNA结合后的荧光光谱 Fig. 5 The fluorescence spectrum of quercetin combined with different concentrations of DNA. The final concentration of quercetin used was 1×10-4 mol/L. The final DNA concentrations of herring sperm used were 0 g/L, 6×10-3 g/L, 1.2×10-2 g/L, 1.8×10-2 g/L, 2.4×10-2 g/L and 3×10-2 g/L, respectively. |
| |
在已经加有槲皮素(终浓度1×10-4 mol/L)的总体系中加入不同终浓度的鲱鱼精DNA (鲱鱼精DNA终浓度分别为6×10-3 g/L、1.2×10-2 g/L、1.8×10-2 g/L、2.4×10-2 g/L和3×10-2 g/L,形成混合体系分别经过10 min的反应时间以使得槲皮素与DNA充分结合)后,混合体系的荧光光谱曲线中出现了467 nm附近处的特征荧光峰,并且这一特征荧光强度会随着混合体系中鲱鱼精DNA终浓度的增加而增大,而在仅有槲皮素的体系中这一特征荧光峰比较微弱,如表 1所示。将相对应终浓度的纯的鲱鱼精DNA (鲱鱼精DNA终浓度分别为6×10-3 g/L、1.2×10-2 g/L、1.8×10-2 g/L、2.4×10-2 g/L和3×10-2 g/L)用UP水定容至3 mL的体系,荧光光谱结果表明,鲱鱼精DNA在310 nm和350 nm附近处存在特征荧光峰,而在467 nm处的荧光强度较低(图 6)。所以,综合以上结论,推测槲皮素与DNA存在相互作用,并且生成了在实验参数条件为280 nm激发光、稳压820 V、两个狭缝宽度均为5 nm时具有467 nm附近特征荧光峰的新的复合物。
| The mixed solution of quercetin and herring sperm DNA (mol/L) | Wavelength (nm) | Intensity (a.u.) |
| 9.92×10-5 mol/L quercetin | 467 | 91.02 |
| 9.92×10-5 mol/L quercetin+6.0×10-3 g/L DNA | 467 | 105.9 |
| 9.92×10-5 mol/L quercetin+2.0×10-2 g/L DNA | 467 | 126.9 |
| 9.92×10-5 mol/L quercetin+1.8×10-2 g/L DNA | 467 | 156.9 |
| 9.92×10-5 mol/L quercetin+2.4×10-2 g/L DNA | 467 | 194.0 |
| 9.92×10-5 mol/L quercetin+3.0×10-2 g/L DNA | 467 | 226.7 |
|
| 图 6 不同浓度鲱鱼精DNA的荧光光谱 Fig. 6 Fluorescence spectra of different concentrations of herring sperm DNA. The final concentrations of herring sperm DNA were 0 g/L, 6×10-3 g/L, 1.2×10-2 g/L, 1.8×10-2 g/L, 2.4×10-2 g/L and 3×10-2 g/L, respectively. |
| |
通过比较纯的槲皮素与纯的鲱鱼精DNA在自身的特征荧光光谱以及二者混合反应充分之后的特征荧光光谱后发现,鲱鱼精DNA在与槲皮素混合反应充分之后其在310 nm与350 nm处的特征吸收峰发生了明显的减色效应,如表 2和表 3所示。考虑到槲皮素DNA扁平的小分子共轭结构比较适合于嵌插进DNA碱基或者结合在鲱鱼精DNA的沟槽之中,对这一减色效应的原因推测有二。
| Herring sperm DNA (mol/L) | Wavelength (nm) | Intensity (a.u.) |
| 6.0×10-3 g/L DNA | 350 | 435.8 |
| 1.2×10-2 g/L DNA | 350 | 610.7 |
| 1.8×10-2 g/L DNA | 350 | 760.3 |
| 2.4×10-2 g/L DNA | 350 | 865.0 |
| 3.0×10-2 g/L DNA | 350 | 960.6 |
| The mixed solution of quercetin and herring sperm DNA (mol/L) | Wavelength (nm) | Intensity (a.u.) |
| 9.92×10-5 mol/L quercetin+6.0×10-3 g/L DNA | 350 | 190.6 |
| 9.92×10-5 mol/L quercetin+1.2×10-2 g/L DNA | 350 | 246.3 |
| 9.92×10-5 mol/L quercetin+1.8×10-2 g/L DNA | 350 | 310.9 |
| 9.92×10-5 mol/L quercetin+2.4×10-2 g/L DNA | 350 | 368.6 |
| 9.92×10-5 mol/L quercetin+3.0×10-2 g/L DNA | 350 | 400.1 |
其一,槲皮素通过静电相互作用、共价相互作用、嵌插作用或者沟槽相互作用(这几种作用方式互不排斥可以共存)与鲱鱼精DNA结合使得新产生的鲱鱼精DNA-槲皮素复合物共轭程度增大,并通过鲱鱼精DNA的大量的共轭体系吸收较高能量的光量子,通过共振体系的传递利用新生成的复合物的光致发光效应产生了新的波长更长能量更低的发射光,同时,这一新生成的复合物的共振体系将原本纯槲皮素DNA产生的荧光光量子(310 nm与350 nm附近)也吸收了。这一结论依赖于槲皮素与鲱鱼精DNA较低的结合常数。
其二,槲皮素与鲱鱼精DNA形成复合物的方式依赖于纯鲱鱼精DNA上的能够产生310 nm附近与350 nm附近特征荧光峰的共振环结构,并且通过槲皮素与鲱鱼精DNA这种结合作用,产生了共振程度更大的复合物体系,这使得原先在纯鲱鱼精DNA荧光图谱中310 nm和350 nm处的荧光峰发生强烈的减色效应,而在原先纯鲱鱼精DNA并不存在荧光峰的467 nm处产生了比310 nm和350 nm处波长更长能量更低的荧光峰,这种推测不需要槲皮素与鲱鱼精DNA存在很低的结合常数。需要补充说明的是,槲皮素储备液虽然使用无水乙醇作为溶剂进行配置,但荧光光谱检测时由于其含量微乎其微,并且无水乙醇自身的荧光光谱结构在本实验内各种缓冲体系中作为本底已经被比较并消除,所以并不影响反应混合物各自的物理化学特征,也不会影响鲱鱼精DNA的B型双螺旋结构或者最终的分析结果。槲皮素与不同终浓度鲱鱼精DNA混合反应后的荧光光谱在310 nm处对纯槲皮素自身荧光光谱在310 nm处特征峰的增色效应,很可能是由于大量加入鲱鱼精DNA引起的,并且这一对槲皮素自身荧光光谱在310 nm处的增色效应,相对于纯鲱鱼精荧光光谱310 nm处特征峰是减色效应。
基于以上的实验结论,可以进行进一步实验论证,来辨明槲皮素与DNA之间的相互作用模式究竟属于哪一种。并探讨这种被动观测到的槲皮素与鲱鱼精DNA之间的低选择性结合的光谱现象是否会存在于其他光谱学方法中。另外,仅仅根据荧光光谱的实验结论,不能够排除槲皮素与鲱鱼精DNA的结合导致DNA双螺旋构象改变的情况。这种构象的改变可能导致鲱鱼精DNA原本在280 nm激发光条件下,310 nm和350 nm处的荧光特征峰产生减色效应,而新生成的槲皮素-鲱鱼精DNA复合物作为一个新产物的整体,其变化了的三级结构将对其自身的荧光特性产生重要的影响。如果这种三级结构改变较为明显,DNA构象变化很大,槲皮素与鲱鱼精DNA之间的结合可以是静电相互作用、嵌插作用、沟槽结合、共价相互作用,或者它们之中的几种相互作用方式并存。尤其是,本实验的目的之一就是研究槲皮素对抗癌、抗炎、抗菌、抗病毒、降糖降压、免疫调节及保护心血管的作用机理,造成这种药物作用结果的可能原因之一,是槲皮素与DNA的结合导致DNA构象的转变,三级结构发生重要变化,影响DNA作为转录模板的功能。最值得注意的是,按照这种推测,槲皮素与鲱鱼精DNA之间的相互作用所产生的槲皮素-鲱鱼精DNA复合物,可以在不产生明显的共轭程度增强的前提下,仍旧产生荧光波长为467 nm附近的特征峰。
因此,认为荧光光谱法的实验结果比较明确地显示了槲皮素与鲱鱼精DNA存在体外相互作用,并且在结合后对鲱鱼精DNA在350 nm处的特征峰产生了明显的减色效应,在467 nm附近处荧光特征峰对槲皮素本身产生了明显的增色效应。同时,也对槲皮素与鲱鱼精DNA之间的相互作用模式验证提出了新的要求,因为基于本次荧光光谱法的实验结果,并不能够明确表示出槲皮素与鲱鱼精DNA之间存在哪一种相互作用,只能够对这类相互作用模式提出推测。
2.2 共振散射荧光光谱分析本实验检测鲱鱼精DNA在加入不同浓度的槲皮素之后其共振散射荧光光谱的变化,旨在佐证荧光光谱的实验结果:槲皮素与鲱鱼精DNA的确存在相互作用,而且这种相互作用很可能是可以增加体系的共轭程度的。所以,使用共振散射荧光光谱法检测,在固定鲱鱼精DNA终浓度4×10-3 g/L (检测总体系为3 mL不足部分用UP水补足),改变槲皮素终浓度(分别为6.6×10-6 mol/L、1.32×10-5 mol/L、1.98×10-5 mol/L、2.64×10-5 mol/L和3.3×10-5 mol/L)。获得的共振散射荧光光谱,如图 7所示。
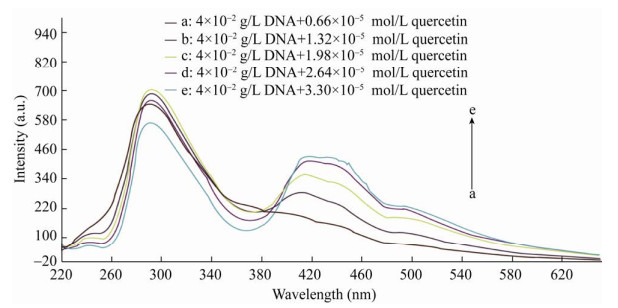
|
| 图 7 使用共振散射荧光光谱法进行分析 Fig. 7 Resonance scattering fluorescence spectroscopy was used for the analysis. The final DNA concentration of herring sperm was 4×10-3 g/L. The final concentrations of quercetin were 6.6×10-6 mol/L, 1.32×10-5 mol/L, 1.98×10-5 mol/L, 2.64×10-5 mol/L and 3.3×10-5 mol/L, respectively. |
| |
随着槲皮素终浓度的增加,总体系的共振散射荧光光谱在439 nm处的特征峰强度逐渐增大,如图 8所示。

|
| 图 8 共振散射荧光光谱(439 nm处) Fig. 8 Interaction between quercetin and DNA by resonance scattering fluorescence spectroscopy. The final DNA concentration of herring sperm used in this experiment was 4×10-3 g/L. The final concentrations of quercetin were 6.6×10-6 mol/L, 1.32×10-5 mol/L, 1.98×10-5 mol/L, 2.64×10-5 mol/L and 3.3×10-5 mol/L, respectively. The ordinate in the picture is the light intensity and the abscissa is the number of samples from low concentration to high concentration. |
| |
这首先说明了槲皮素与鲱鱼精DNA之间存在明显的相互作用,考虑槲皮素的加入使得鲱鱼精DNA的粒子体积逐渐增大,这种粒子体积的增大可能是由于鲱鱼精DNA的大沟与槲皮素相结合所导致的,这种结合键合力类型可能是疏水相互作用、氢键或者静电相互作用。其次,这种相互作用所导致的直接结果是槲皮素-鲱鱼精DNA复合物的形成,也就是在固定鲱鱼精DNA终浓度4×10-3 g/L (检测总体系为3 mL不足部分用UP水补足),改变槲皮素终浓度(分别为6.6×10-6 mol/L、1.32×10-5 mol/L、1.98×10-5 mol/L、2.64×10-5 mol/L和3.3×10-5 mol/L)的情况下,溶液当中粒子体积不断增大,粒子的生色团电耦合程度增加,电子离域程度增大,混合溶液的共振散射光谱中439 nm处特征峰强度因而不断增大。
2.3 紫外分光光度法分析基于荧光光谱法与共振散射荧光光谱的实验结论,可以认为槲皮素与鲱鱼精DNA之间是存在相互作用的。所以在对紫外-可见分光光度法的实验结论的分析中倾向于寻找槲皮素与鲱鱼精DNA之间相互作用的模式。实验采用的是固定鲱鱼精DNA终浓度为4×10-2 g/L,逐次滴加入槲皮素使其终浓度依次达到1.98×10-5 mol/L、3.97×10-5 mol/L、5.95×10-5 mol/L、7.94×10-5 mol/L和9.92×10-5 mol/L,获得的紫外吸收光谱,如图 9所示。
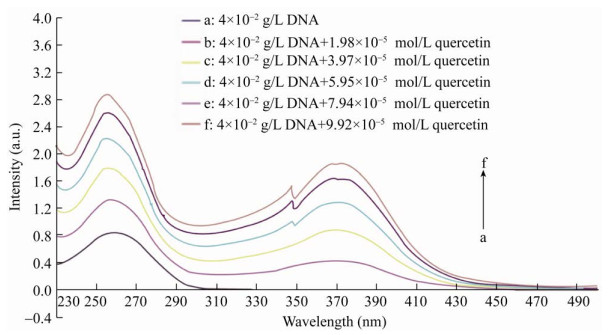
|
| 图 9 紫外-可见分光光度法分析槲皮素与鲱鱼精DNA的相互作用 Fig. 9 The interaction between quercetin and herring sperm DNA was analyzed by ultraviolet spectrophotometry. In the experiment, the final concentration of herring sperm DNA was fixed at 4×10-2 g/L and quercetin was added dropwise to make the final concentration reach 1.98×10-5 mol/L, 3.97×10-5 mol/L, 5.95×10-5 mol/L, 7.94×10-5 mol/L and 9.92×10-5 mol/L, respectively. |
| |
理论上DNA自身在260 nm附近处具有比较强烈的光吸收,经实验测定终浓度为4×10-2 g/L的鲱鱼精DNA的紫外-可见吸收光谱在259 nm处达到最大光吸收0.84 ABS。终浓度4×10-2 g/L的鲱鱼精DNA在加入了不同终浓度的槲皮素之后,其260 nm附近处的紫外光吸收明显增强,而鲱鱼精DNA在该处吸光度的增强值并不等于槲皮素在该处自身的紫外光吸收,说明槲皮素与鲱鱼精DNA的确存在相互作用,并且这种相互作用产生了一种新的槲皮素-鲱鱼精DNA复合物,这种复合物在260 nm附近处有比相应浓度的槲皮素和鲱鱼精DNA都更加强烈的紫外光吸收。通常认为有机小分子的加入如果引起了DNA吸光度发生强烈的增色或者减色效应,那么这种有机小分子与DNA之间可能是存在静电相互作用的。通过对鲱鱼精DNA结合不同终浓度槲皮素之后的紫外吸收光谱与鲱鱼精DNA自身的紫外吸收光谱在260 nm附近的最大吸收波长的比对,发现在固定终浓度的鲱鱼精DNA中加入不同终浓度的槲皮素会使得最大吸收波长发生不同程度的蓝移;而这一新产生的复合物的紫外光谱在该处的最大吸收波长对于相应浓度的槲皮素则发生了不同程度的红移。如表 4所示。
| The mixed solution of quercetin and herring sperm DNA (mol/L) | Maximum absorption wavelength (nm) | Relative DNA displacement after binding (blue shift) (nm) | Relative quercetin displacement after binding (red shift) (nm) |
| 4×10-2 g/L DNA | 259 | 0 | / |
| 5.95×10-5 mol/L quercetin | 254 | / | 0 |
| 4×10-2 g/L DNA+1.98×10-5 mol/L quercetin | 257 | 2 | 3 |
| 4×10-2 g/L DNA+3.97×10-5 mol/L quercetin | 256 | 3 | 2 |
| 4×10-2 g/L DNA+5.95×10-5 mol/L quercetin | 256 | 3 | 2 |
| 4×10-2 g/L DNA+7.94×10-5 mol/L quercetin | 255 | 4 | 1 |
| 4×10-2 g/L DNA+9.92×10-5 mol/L quercetin | 255 | 4 | 1 |
对这一结果的分析,主要考虑槲皮素与鲱鱼精DNA之间的相互作用可能具有比较低的选择性,这使得在混合溶液体系中存在各种分别具有微弱结构差异(结合方式与结合强度)的槲皮素-鲱鱼精DNA复合物,也就使得该复合物的紫外-可见光谱结果受到不同程度跨环效应的影响。又由于跨环效应要求两个或数个发色基团虽然不共轭,但由于空间的排列,使它们的电子云仍能相互影响,这就要求槲皮素的相对刚性的环结构与鲱鱼精DNA的共轭碱基具有一定的相对取向以及相当近的距离,因此要求槲皮素与鲱鱼精DNA之间存在一定程度的沟槽相互作用。槲皮素与鲱鱼精DNA之间的这种沟槽结合又进一步把鲱鱼精DNA的沟槽“撑开”,使得鲱鱼精DNA在加入了不同浓度的槲皮素并充分反应之后,在260 nm附近处的紫外光吸收显著增加。基于上述的结果分析,推测槲皮素与鲱鱼精DNA之间可能存在沟槽结合或静电相互作用。但还不能够排除的一种可能就是槲皮素与DNA的结合导致DNA构象的转变,三级结构发生重要变化,影响DNA作为转录模板的功能,最值得注意的是,按照这种推测,槲皮素与鲱鱼精DNA之间的相互作用所产生的槲皮素-鲱鱼精DNA复合物,可以在不产生明显的静电相互作用与特征性的沟槽结合的前提下,以较为复杂与低选择性的相互作用模式,生成新的槲皮素-鲱鱼精DNA复合物,同样有可能获得上述紫外-可见分光光度法的实验结果。因此,对于槲皮素与鲱鱼精DNA之间的相互作用模式需要借助其他实验方法进行辅助推断。假若按照前一种情况来推断,即假设槲皮素与鲱鱼精DNA之间的相互作用可能是明显的沟槽结合或静电相互作用(考虑到槲皮素较为刚性的小分子结构,这一推论更为可信),根据Knakis的方法[33]可以对槲皮素(Quercetin,QUE)与鲱鱼精DNA之间的相互作用进行如下运算:
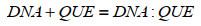
|
(1) |
其中DNA代表与槲皮素结合的一个寡聚脱氧核糖核酸双链片段,QUE代表一个单位的槲皮素。
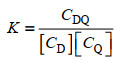
|
(2) |
其中CDQ,CD,CQ,分别是槲皮素与鲱鱼精DNA复合物的浓度,鲱鱼精DNA的浓度以及槲皮素的浓度。K是(1)中所描述反应的平衡常数。
Beer lambert法则对消光系数的要求是假定消光系数本身是依据配基的结合会减少吸光度的原理。

|
(3) |
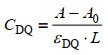
|
(4) |
和

|
(5) |
在该式中的CD0是纯鲱鱼精DNA的浓度,A0和A是纯鲱鱼精DNA与在当前槲皮素浓度之下的混合液的260 nm处的吸光度。εD和εDQ分别是鲱鱼精DNA和槲皮素-鲱鱼精DNA复合物的摩尔消光系数。l是比色皿的光程(1 cm)。
将上述CD和CDQ带入(2)式得
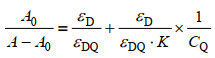
|
(6) |
令1/(A-A0)对1/CC做双倒数图像,如图 10所示。
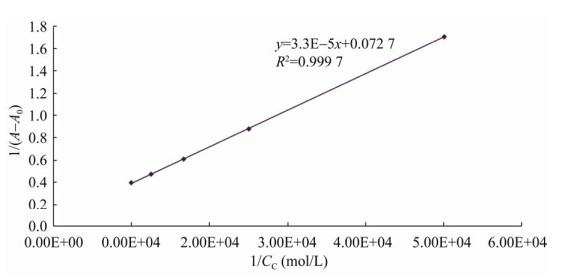
|
| 图 10 采用的鲱鱼精DNA终浓度固定为4×10-2 g/L Fig. 10 The final DNA concentration of herring sperm was fixed at 4×10-2 g/L. The final concentrations of quercetin were 1.98×10-5 mol/L, 3.97×10-5 mol/L, 5.95×10-5 mol/L, 7.94×10-5 mol/L and 9.92×10-5 mol/L, respectively. |
| |
于是可以得到线性结果,并且能够通过计算截距与斜率的比值求出结合常数(K=2 203)。
将K值代入公式:
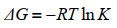
|
(7) |
其中,∆G是反应前后的吉布斯自由能变化量,R是气体常数1.98 cal/(mol·K),T是测量时温度289 K。
经计算得到槲皮素与鲱鱼精DNA结合后所得的槲皮素-鲱鱼精DNA复合物对应反应物的吉布斯自由能变化量约为:∆G=-4.4 kcal/mol。
本实验以紫外-可见吸收光谱的光吸收程度的变化,在固定鲱鱼精DNA终浓度不变的情况下,对不同的槲皮素终浓度做线性回归,如图 11和图 12所示。

|
| 图 11 槲皮素254 nm线性回归分析 Fig. 11 Linear regression analysis of characteristic peak of quercetin at 254 nm. The final concentration of herring sperm DNA was fixed at 4×10-2 g/L. The final concentrations of quercetin were 1.98×10-5 mol/L, 3.97×10-5 mol/L, 5.95×10-5 mol/L, 7.94×10-5 mol/L and 9.92×10-5 mol/L, respectively. |
| |
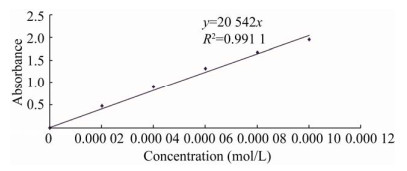
|
| 图 12 槲皮素368 nm线性回归分析 Fig. 12 Linear regression analysis of characteristic peak of quercetin at 368 nm. The final concentration of herring sperm DNA was fixed at 4×10-2 g/L. The final concentrations of quercetin were 1.98×10-5 mol/L, 3.97×10-5 mol/L, 5.95×10-5 mol/L, 7.94×10-5 mol/L and 9.92×10-5 mol/L, respectively. |
| |
基于前述实验结论,发现槲皮素与鲱鱼精DNA之间是存在相互作用的,这种相互作用可能是沟槽结合或者静电相互作用,结合的选择性不高,结合力较弱。荧光偏振实验可以有力地佐证槲皮素与鲱鱼精DNA之间的相互作用属于结合力较弱、选择性相对有限的沟槽结合或者静电相互作用,如图 13所示。

|
| 图 13 荧光偏振分析 Fig. 13 Fluorescence polarization analysis. The final concentration of quercetin was 9.92×10-5 mol/L and the final concentrations of herring sperm DNA was 0 g/L, 6×10-3 g/L, 1.2×10-2 g/L, 1.8×10-2 g/L and 3.0×10-2 g/L, respectively. |
| |
因为在水溶液中,溶液的粘度相对较小,小分子的旋转运动不太受到限制,这时小分子的运动相对比较快,荧光偏振值相对较小。随着槲皮素-鲱鱼精DNA混合总体系中鲱鱼精DNA终浓度的增加,总体系的荧光偏振强度并没有明显的上升趋势。已经知道当有机小分子与DNA之间的作用模式为嵌插作用或者较强的静电相互作用时,随着DNA的终浓度增加,混合总体系的荧光偏振值会有较为明显的上升趋势,而当槲皮素与鲱鱼精DNA之间的结合模式为沟槽结合或者其他的较弱的相互作用,例如与磷酸基团的较弱的相互作用时,随着鲱鱼精DNA终浓度的增加,荧光偏振度的上升趋势不明显[34]。所以判断槲皮素与鲱鱼精DNA之间的相互作用是沟槽作用模式或者较弱的静电相互作用模式。该分析选择的激发光波长是280 nm,设置波速为1 200 nm/min。
2.5 分子对接实验结果分析为了明确槲皮素与鲱鱼精DNA之间的相互作用究竟是哪一种,本文采用分子对接实验来进行佐证。分子对接实验的对象是槲皮素小分子与B型DNA十二聚体。实验结果表明,槲皮素与鲱鱼精DNA之间的相互作用主要依靠氢键和疏水相互作用来维系,没有静电相互作用模式的存在。主要包括槲皮素与鲱鱼精DNA沟槽中碱基的相互作用,即槲皮素的共轭环结构与脱氧核糖核酸十二聚体的B链上的DG5、DC6和DG7的碱基共轭环结构之间的疏水相互作用;同时还存在槲皮素与鲱鱼精DNA之间的氢键如槲皮素上的H20与DNA十二聚体中的DG5的N7之间的氢键,槲皮素上的H31与DG7的N7之间的氢键之间的相互作用。槲皮素与鲱鱼精DNA之间发生相互作用的吉布斯自由能变化量是-5.710 7 kcal/moL,与紫外-可见分光光度法的计算结果大致相似,因此,认为分子对接实验结果佐证了上述其他实验的结论,认为槲皮素与鲱鱼精DNA之间的相互作用模式是沟槽结合(图 14)。

|
| 图 14 分子对接实验结果显示槲皮素与鲱鱼精DNA之间的相互作用模式是沟槽结合 Fig. 14 Molecular docking experiments revealed that the pattern of interaction between quercetin and herring sperm DNA was groove binding. |
| |
根据荧光光谱法和共振散射荧光光谱法的实验结果,认为槲皮素与鲱鱼精DNA之间的确存在相互作用;通过紫外-可见分光光度法和荧光偏振实验,可以判断槲皮素与鲱鱼精DNA之间的相互作用模式不属于嵌插作用,而是沟槽嵌合或者静电相互作用;通过分子对接实验,成功佐证槲皮素与鲱鱼精DNA之间的相互作用模式是沟槽结合;因此,建立在上述结论之上,荧光光谱法、共振散射荧光光谱法、紫外-可见分光光度法、荧光偏振实验以及分子对接实验,这一系列实验方法组合,将可以作为研究有机小分子与DNA之间的体外相互作用的普适而简便的模式化实验流程。
本实验从实验结果来看,证明了槲皮素与鲱鱼精DNA之间在体外的确存在相互作用,并且这一相互作用模式是沟槽结合。与此同时也证明了本实验选用的实验方法组合切实可行,将可以用来作为研究有机小分子与DNA之间的相互作用的模式化流程,辅助对有机小分子药理的研究以及包括中药作用机理的说明和新药的筛选。但通过本实验挑选的研究方法并不能够说明槲皮素与鲱鱼精DNA之间在人体内的实际过程就一定是沟槽结合,只能够猜测槲皮素可以通过对鲱鱼精DNA的沟槽进行结合,影响以鲱鱼精DNA为代表的DNA或者特定的DNA序列作为转录模板的功能,从而达到抑制特定基因片段表达的效果,影响细胞的功能从而产生抗癌、抗炎、抗菌、抗病毒、降糖降压、免疫调节及保护心血管的作用。
致谢 感谢上海大学宋红生、陈旭老师,中国海洋大学包振民老师和北京大学夏传信老师对本文的指导与帮助。
| [1] |
Yang Z, Zhang D, Long H, et al. Electrochemical behavior of gallic acid interaction with DNA and detection of damage to DNA. J Electroanal Chem, 2008, 624(1/2): 91-96. |
| [2] |
Wang RY, Ji MN, Wang R, et al. Stopped-flow kinetic fluorimetric studies of the interaction of Ru (Ⅱ) complex with DNA and its analytical application. Spectrochim Acta A Mol Biomol Spectrosc, 2008, 71(3): 1042-1048. DOI:10.1016/j.saa.2008.02.043 |
| [3] |
Cheng ZH, Lin XQ, Shao XG, et al. Application and advance of chemometrics in electroanalytical chemistry. J Anal Sci, 2002, 18(1): 74-79. |
| [4] |
Li XM, Ye SL, Zhang SS, et al. Study on the action mechanism with DNA and their biological function of complexes prepared from glycoside by electroanalytical chemistry. Chem Res Appl, 2003, 15(6): 741-743. |
| [5] |
Lan W, Wang R, Lu XQ, et al. New developments of chemometrics in electroanalytical chemistry. Chemistry, 2007, 70(5): 338-342. |
| [6] |
Wang M, Sun C, Wang L, et al. Electrochemical detection of DNA immobilized on gold colloid particles modified self-assembled monolayer electrode with silver nanoparticle label. J Pharm Biomed Anal, 2003, 33(5): 1117-1125. DOI:10.1016/S0731-7085(03)00411-4 |
| [7] |
Liu J, Li J, Dong S. Interaction of brilliant cresyl blue and methylene green with DNA studied by spectrophotometric and voltammetric methods. Electroanalysis, 1996, 8(8/9): 803-807. |
| [8] |
Jiang X, Shang L, Wang Z, et al. Spectrometric and voltammetric investigation of interaction of neutral red with calf thymus DNA: pH effect. Biophys Chem, 2005, 118(1): 42-50. |
| [9] |
Liu Q, Li J, Tao W, et al. Comparative study on the interaction of DNA with three different kinds of surfactants and the formation of multilayer films. Bioelectrochemistry, 2007, 70(2): 301-307. DOI:10.1016/j.bioelechem.2006.03.041 |
| [10] |
He JB, Wang Y, Deng N, et al. Study of the adsorption and oxidation of antioxidant rutin by cyclic voltammetry-voltabsorptometry. Bioelectrochemistry, 2007, 71(2): 157-163. DOI:10.1016/j.bioelechem.2007.03.003 |
| [11] |
Zhang KJ, Liu WY. Electrochemical and spectroscopic study on the interaction between G-quadruplex DNA and (R)-/(S)-2-(5-fluorouracil- 1-acetyl) amido-1, 5-dimethyl glutarate. Int J Electrochem Sc, 2011, 6: 4006-4015. |
| [12] |
Turro C, Bossmann SH, Jenkins Y, et al. Proton transfer quenching of the MLCT excited state of Ru (phen) 2dppz2+ in homogeneous solution and bound to DNA. J ACS, 1995, 117(35): 9026-9032. |
| [13] |
Liu N, Li GJ, Liu SF, et al. Electrochemical DNA biosensor for the detection of interaction between di[azino-di (5, 6-azafluorene)-κ2-NN'] dichlormanganous and DNA. Sens Actuators B Chem, 2008, 133(2): 582-587. |
| [14] |
Carter MT, Bard AJ. Voltammetric studies of the interaction of tris (1, 10-phenanthroline) cobalt (Ⅲ) with DNA. J ACS, 1987, 109(24): 7528-7530. |
| [15] |
Wang S, Peng T, Yang CF. Electrochemical determination of interaction parameters for DNA and mitoxantrone in an irreversible redox process. Biophys Chem, 2003, 104(1): 239-248. |
| [16] |
Ferancová A, Adamovski M, Gründler P, et al. Interaction of tin (Ⅱ) and arsenic (Ⅲ) with DNA at the nanostructure film modified electrodes. Bioelectrochemistry, 2007, 71(1): 33-37. |
| [17] |
Heli H, Bathaie SZ, Mousavi MF. An electrochemical study of neutral red-DNA interaction. Electrochimica acta, 2005, 51(6): 1108-1116. |
| [18] |
Li X, Chen Y, Huang X. Electrochemical behavior of neomycin at DNA-modified gold electrodes. J Inorg Biochem, 2007, 101(6): 918-924. |
| [19] |
Tabassum S, Parveen S, Arjmand F. New modulated metallic macrocycles: electrochemistry and their interaction with calf thymus DNA. Acta Biomater, 2005, 1(6): 677-689. |
| [20] |
Tian X, Li F, Zhu L, et al. Study on the electrochemical behavior of anticancer herbal drug rutin and its interaction with DNA. J Electroanal Chem, 2008, 621(1): 1-6. |
| [21] |
Niu SY, Zhang SS, SHI X, et al. Electrochemical study on the interaction betwwen neutral red and DNA. Chem J ChineseU, 2011, 21(1): 24-27. |
| [22] |
Li H, Mei W, Xu Z, et al. Electrochemistry of a novel monoruthenated porphyrin and its interaction with DNA. J Electroanal Chem, 2007, 600(2): 243-250. |
| [23] |
Wang L, Lin L, Ye B. Electrochemical studies of the interaction of the anticancer herbal drug emodin with DNA. J Pharm Biomed Anal, 2006, 42(5): 625-629. |
| [24] |
Liu JF, Li YQ, Guo MD. Present status of the researches on electrochemical DNA biosensors. Phys T Chem Anal B Chem Anal, 2007, 43(8) (in Chinese). 刘俊芳, 李彦青, 郭满栋. 电化学DNA生物传感器的研究现状. 理化检验-化学分册, 2007, 43(8): b701-704. |
| [25] |
Farhadi K, Yamchi RH, Sabzi R. Electrochemical study of interaction between clozapine and DNA and its analytical application. Anal Lett, 2007, 40(9): 1750-1762. |
| [26] |
Wang F, Xu Y, Zhao J, et al. Electrochemical oxidation of morin and interaction with DNA. Bioelectrochemistry, 2007, 70(2): 356-362. |
| [27] |
Park HI, Sanchez D, Cho SK, et al. Bacterial communities on electron-beam Pt-deposited electrodes in a mediator-less microbial fuel cell. Environ Sci Technol, 2008, 42(16): 6243-6249. |
| [28] |
Spiess PC, Morin D, Jewell WT, et al. Measurement of protein sulfhydryls in response to cellular oxidative stress using gel electrophoresis and multiplexed fluorescent imaging analysis. Chem. Res. Toxicol, 2008, 21(5): 1074-1085. |
| [29] |
Fiel RJ, Munson BR. Binding of meso-tetra (4-N-methylpyridyl) porphine to DNA. Nucleic Acids Res, 1980, 8(12): 2835-2842. |
| [30] |
Potaman VN, Bannikov YA, Shlyachtenko LS. Sedimentation of DNA in ethanol-water solutions within the interval of B→ A transition. Nucleic Acids Res, 1980, 8(3): 635-642. |
| [31] |
Charak S, Shandilya M, Tyagi G, et al. Spectroscopic and molecular docking studies on chlorambucil interaction with DNA. Int J Biol Macromol, 2012, 51(4): 406-411. |
| [32] |
Tataurov AV, You Y, Owczarzy R. Predicting ultraviolet spectrum of single stranded and double stranded deoxyribonucleic acids. Biophys Chem, 2008, 133(1/3): 66-70. |
| [33] |
Kanakis CD, Tarantilis PA, Pappas C, et al. An overview of structural features of DNA and RNA complexes with saffron compounds: Models and antioxidant activity. J Photochem Photobiol B, 2009, 95(3): 204-212. |
 2020, Vol. 36
2020, Vol. 36