中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘星, 陈奇
- Liu Xing, Chen Qi
- 鲨源单域抗体的研究进展
- Progress in shark single-domain antibody
- 生物工程学报, 2020, 36(6): 1069-1082
- Chinese Journal of Biotechnology, 2020, 36(6): 1069-1082
- 10.13345/j.cjb.190460
-
文章历史
- Received: October 13, 2019
- Accepted: December 6, 2019
- Published: March 11, 2020
自Kohler和Milstein[1]发明杂交瘤技术以来,理化性状均一、生物活性单一的单克隆抗体被广泛应用于免疫检测、医学诊断以及疾病治疗等领域。其中,首个抗体药物抗CD3抗体(OKT3)于1986年通过美国FDA的审查,随后,由于靶向性好、副作用小等优点,抗体药物逐渐成为生物制药行业发展最快的种类之一。到2018年底,据不完全统计已有69种抗体药物通过FDA批准上市[2-3]。据Grilo及Mantalaris统计,2017年抗体药物的市场销售额为980亿美元,预计到2022年销售额将达到1 370亿至2 000亿美元,领跑所有的生物类药物[3]。
回顾整个抗体应用(药物及免疫试剂)的发展历程,可以发现传统抗体的固有属性一直是制约其发展的关键因素。首先传统抗体尺寸大,难以穿越肿瘤组织以及人体屏障,限制了抗体药物的应用范围。其次,传统抗体结构复杂、稳定性差,导致其作为免疫试剂时易失效,从而限制了相关抗体试剂的应用领域。为了克服上述问题,通过基因工程技术对传统抗体进行了多种形式的小型化改造,先后开发出了抗体可变区片段(Antigen-binding fragment,Fab)、单链抗体(Single-chain variable fragment,ScFv)等小分子抗体,然而这些小分子抗体往往表现出较差的稳定性以及亲和力[4-5]。
1993年天然缺失轻链的重链抗体(Heavy- chain antibodies,HcAbs)首先在骆驼科动物中被发现[6]。随后,1995年,在护士鲨Ginglymostoma cirratum体内发现了具有类似重链抗体结构的免疫球蛋白,被称为IgNAR[7]。HcAbs及IgNAR的发现为抗体小型化研究提供了新的发展方向,并引发了对单域抗体(VHH及VNAR)的研究热潮。单域抗体所固有的分子量小、亲和力高、稳定性强、溶解度好、组织穿透性强以及可识别隐藏抗原表位等优点使其在免疫检测[8]、医学成像[9]、体外诊断[10-11]以及疾病治疗[12-13]等领域中受到广泛的关注。由于骆驼科动物的饲养较鲨鱼更为容易等原因,早期对单域抗体的研究主要集中于骆驼科动物。目前国内外已有大量文献综述了VHH的结构及功能特性及其在各领域的应用。近年来针对鲨源单域抗体的研究日益增多,对其结构及功能的认识不断加深,应用也日渐广泛。但是,目前国内未见关于鲨源单域抗体的综述报道。因此,文中详细综述了鲨源单域抗体的结构及功能特性、制备及人源化改造技术、亲和力成熟策略以及应用领域,并系统性分析了鲨源单域抗体的优缺点,最后对其发展前景进行了展望。
1 IgNAR的发现20世纪60年代首次证明,鲨鱼能够对抗原刺激产生保护性免疫应答[14],表明其存在适应性免疫系统。首先在鲨鱼体内发现的免疫球蛋白为IgM,其具有2种不同沉降系数(19S及7S)的类型。其中,7S型IgM的亲和力经免疫后会提高,并且存在记忆效应[15-16]。第2种被发现的免疫球蛋白为IgW,其与高等脊椎动物IgD同源性较高。分泌型IgW存在2种类型,分别包括3个和7个结构域[17]。
1995年,Andrew等在护士鲨体内发现了不同于IgM和IgW的新型免疫球蛋白IgNAR,其最初被发现与T细胞抗原受体(T cell receptor,TCR)同源性较高,从而被命名为新抗原受体(New antigen receptor,NAR)[7],但是后续研究发现该分子与免疫球蛋白存在诸多类似的结构及功能特性,因此被重新命名为IgNAR[18],IgNAR与传统抗体不同,它是由2条重链组成的同源二聚体,缺乏轻链(图 1)。与经典的B细胞受体一样,IgNAR以分泌型和膜结合型的形式存在,由被称为VNAR的可变区和不同数量的恒定区组成。其中,分泌型IgNAR具有5个恒定区[19]。IgNAR在适应性免疫中的作用由Dooley等首次证实,其以鸡卵清溶菌酶(HEL)为抗原免疫护士鲨,经过长达3年的监控,发现在多次间隔免疫之后,护士鲨产生了快速且显著增强的IgNAR免疫应答反应[20]。

|
| 图 1 不同类型抗体的结构示意图 Fig. 1 Schematic diagram of different types of antibodies |
| |
软骨鱼纲(Chondrichthyes)是与哺乳动物在进化上距离最远的具有真正适应性免疫系统的脊椎动物,由全头亚纲(Holocephali)和板鳃亚纲(Elasmobranchii)组成[21]。其中板鳃亚纲可分为鲨总目(Selachimorpha)和鳐总目(Batoidea)[22]。
2006年Criscitiello等在护士鲨等体内发现NAR-TCR的存在[23]。最初认为,NAR-TCR是IgNAR基因簇和TCR位点基因重组的产物。但是,2007年Venkatesh等对全头亚纲鱼类象鼻鲨Callorhinchus milii的全基因组数据分析,只发现了NAR-TCR的存在,而没有找到IgNAR存在的证据[24-25],表明NAR-TCR的出现早于IgNAR,因此推测IgNAR的可变区可能源于NAR-TCR。同时由于IgNAR中最后4个恒定区与IgW的恒定区同源性较高,因此有学者认为在2.2亿年前软骨鱼纲分化为全头亚纲和板鳃亚纲前后,NAR-TCR与IgW发生基因重组,并丢失CH1,导致在板鳃亚纲鱼类(鲨、鳐)中出现了IgNAR[23, 26-27],因此基于IgNAR所开发的VNAR包括鳐源及鲨源2种。至于VNAR的起源,到目前为止依然没有定论,因为它与任何已知的抗原受体都没有很高的相似性[28]。
据不完全统计目前鲨总目存在超过450个不同的种[29],鳐总目存在约560个不同的种[22],由此可知板鳃亚纲鱼类种类远多于骆驼科动物,为VNAR的开发提供了丰富的材料,由于当前针对VNAR的研究集中于鲨总目,为叙述方便,本文所述VNAR特指鲨源单域抗体。
3 IgNAR的多样性来源与在其他脊椎动物中发现的转座子(VnDnJnC)基因系统不同,板鳃亚纲鱼类的免疫球蛋白(IgM、IgW和IgNAR)基因以基因簇(VDJC)n形式存在(图 2)。每个基因簇由1个V基因片段(Variable gene segment)、多个D基因片段(Diversity gene segment)、1个J基因片段(Joining gene segment)和恒定区外显子组成[30-31]。IgNAR也由基因簇中的基因编码,其每个基因簇中含有3个D基因片段,因此通常需要4次基因重排,才能产生1个完整的VNAR基因序列[30]。由于IgNAR不含轻链,缺少重链和轻链的组合多样性,同时IgNAR基因簇数量较少,因此IgNAR最主要的多样性来源于长且高度突变的CDR3区[32-33]。同时,CDR1、HV2和HV4区的突变也能增加IgNAR的多样性[34]。此外对VNAR的结构分析表明,非典型半胱氨酸残基的数量及位置的不同会导致VNAR抗原结合区的构象存在巨大的差异[35-37]。因此,VNAR中的二硫键不仅可以增加结构的稳定性,还能产生各种各样的构象,从而进一步扩展IgNAR的多样性。
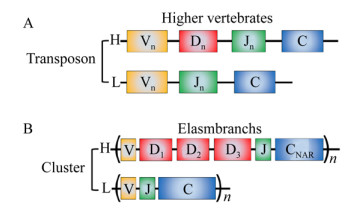
|
| 图 2 高等脊椎动物和板鳃亚纲鱼类免疫球蛋白基因的转座子及基因簇排列方式比较 Fig. 2 Comparison of transposons and cluster arrangement of immunoglobulin genes in higher vertebrates and elasmbranchs |
| |
根据CDR区和FR区中非典型半胱氨酸残基及保守色氨酸残基的位置和数量可以将VNAR划分为不同的亚型[38]。到目前为止,已经定义了3种主要的VNAR,分别称为类型Ⅰ、Ⅱ和Ⅲ[39-40]。此外有些VNAR只包含2个高度保守的典型半胱氨酸残基,Liu等[41]和Streltsov等[28]将这些VNAR称为Ⅱb型。同时将在CDR1区含有保守色氨酸残基以及只含有典型半胱氨酸残基的VNAR称为Ⅲb型[38]。所有当前命名的VNAR类型见图 3。此外,2019年Feng等[42]通过对约119万条来源于护士鲨的VNAR序列分析发现,护士鲨中存在约5%其他类型的VNAR,其不含或只含有1个半胱氨酸残基。
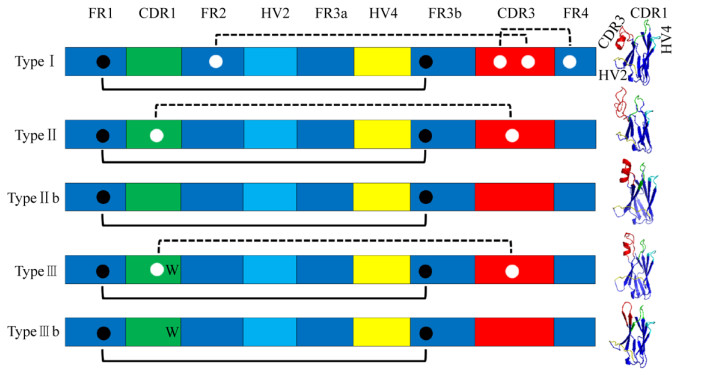
|
| 图 3 不同类型的VNAR示意图(左侧为VNAR序列的特征图,其中黑点为典型半胱氨酸残基,白点为非典型氨基酸残基,黑色实线和虚线为二硫键.右侧为不同类型VNAR的三维结构模型,其是利用Pymol软件对已报道的VNAR序列[41, 44]进行同源建模而获得) Fig. 3 Schematic diagram of different types of VNAR. The characteristic diagrams of VNAR are on the left. The canonical cysteine residues (black dots), non-Canonical cysteine residues (white dots), disulphide bonds (solid and dashed lines), and conserved tryptophan (W) are shown in their relative positions. The three-dimensional structural models of different types of VNAR are on the right, which are obtained by homology modeling of reported VNAR sequences[41, 44] using Pymol software |
| |
迄今为止,Ⅰ型VNAR只在护士鲨中被发现,可能为该物种所特有[40]。该亚型VNAR分别在FR2和FR4区中存在2个非典型半胱氨酸残基,同时在CDR3区中也存在2个以上的半胱氨酸残基。晶体结构分析发现,FR2和FR4中存在的半胱氨酸残基会与CDR3中的2个半胱氨酸残基分别形成二硫键,从而形成1个紧密结构[37] (图 3)。需要注意的是,Ⅰ型VNAR由于二硫键的束缚导致CDR3区形成的指凸结构的长度较短,难以识别蛋白裂隙深处的抗原表位[36]。
Ⅱ型VNAR与Ⅰ型的不同之处是CDR1区和CDR3区中的2个非典型半胱氨酸会形成二硫键,使上述区域靠近,同时CDR3区可形成1个较长的指凸结构,从而易于与蛋白裂隙等隐藏抗原表位结合。迄今为止,在所有研究的鲨鱼中都发现了Ⅱ型VNAR[40]。
Ⅲ型VNAR与Ⅱ型类似,在CDR1和CDR3区分别存在1个非典型半胱氨酸,此外该亚型在CDR1区半胱氨酸残基附近存在1个保守的色氨酸残基。需要注意的是Ⅲ型VNAR的多样性较低。据推测,Ⅲ型VNAR是保护新生鲨鱼抵御常见病原体的早期广谱抗体,通常只在幼鲨(< 1岁)中表达[32]。除Ⅲ型VNAR外,所有类型的VNAR都能产生高亲和力[43]。
5 VNAR的主要特性 5.1 结构特性VNAR与哺乳动物传统VH及驼源动物VHH在结构上最大的不同是,其缺乏包含有CDR2区的C′和C″的β链,同时在C-D和D-E的β链之间分别存在额外的高变区(HV2和HV4) (图 4)。2条β链的缺失,使得VNAR的分子量约为12 kDa,比VHH小约20%,是已知脊椎动物中尺寸最小的抗原结合域[37, 43]。
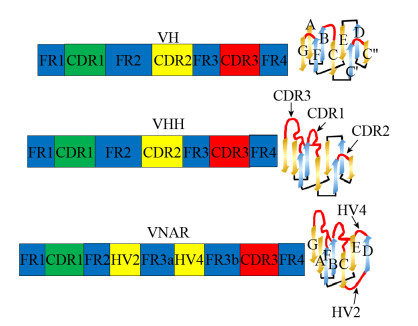
|
| 图 4 VH、VHH和VNAR结构示意图 Fig. 4 Structural schematic diagram of VH, VHH and VNAR |
| |
基于有限的VNAR晶体结构可知[36-37, 44-45],HV2区通常与CDR1、CDR3以及HV4区距离较远(图 5),有可能作为独立抗原结合区而存在。Zielonka等[46]以上皮细胞粘附分子EpCAM特异性VNAR为模板,通过随机化HV2区(包含9个氨基酸残基)构建了酵母展示文库,经淘选获得特异性针对细胞表面分子CD3ε以及人Fcγ的VNAR,新分离获得的VNAR依然保留了识别EpCAM的高亲和力。该研究表明同一VNAR的不同区域可以作为独立的抗原识别区识别不同的抗原。更为重要的是,该研究为双特异性抗体的开发提供了新思路。
针对一系列抗酶抗体的研究发现,传统抗体与单域抗体可能优先识别不同的抗原表位[37, 47-48]。这主要是因为传统抗体的VH-VL界面通常是平面或者凹面,无法识别蛋白裂隙等隐藏抗原表位,而单域抗体的CDR3区通常较长,可以形成指凸结构,从而能够深入到酶活性中心等蛋白裂隙。目前有限的VNAR与酶免疫复合物的晶体结构研究证实了上述观点,VNAR的CDR3区可以深入酶活性中心[37] (图 5)。
不仅酶活性中心,VNAR也能识别其他折叠蛋白的裂隙。恶性疟原虫顶端膜抗原1 (AMA1)是一种重要的蛋白抗原,存在于目前所有已验证的疟原虫中,其含有1个进化上保守的蛋白裂隙。Nuttall等利用半合成库,淘选得到AMA1特异性的VNAR,命名为12Y-2[49]。随后作者对12Y-2进行一系列突变,并对2株突变体与AMA1的晶体结构进行研究,结果显示,VNAR的CDR3区能够深入到AMA1的疏水裂隙中[44]。因此有学者认为识别裂隙等隐藏抗原表位可能是单域抗体的基本特征[47]。
5.2.2 高水溶性和稳定性IgNAR可在含有350 mmol/L尿素和1 000 mOsm/kg渗透压盐离子的鲨鱼血液中保持生物活性,主要是因为IgNAR具有极强的稳定性[50-51]。通常认为IgNAR的稳定性可以归因于以下2个因素:1)高水溶性,与传统抗体VH-VL界面相比,VNAR带有大量的带电和亲水氨基酸残基,如Tyr37、Glu46、Lys82、Gln84、Arg101和Lys104,其中Glu46、Lys82和Lys104氨基酸残基的保守性最强,其通过相互之间以及与水分子之间的氢键形成1个带电的内陷结构。此外Tyr37作为免疫球蛋白超家族的保守氨基酸残基与Gln84、Arg101参与形成CDR3区与FR区的氢键网络,最终使得VNAR形成亲水性界面,提高溶解性并保护保守的免疫球蛋白疏水性骨架[28];2) VNAR的特殊结构,Buchner等发现与哺乳动物抗体相比,VNAR包含1个额外的盐桥和延伸的疏水核。将这些增强稳定性的结构转移到人抗体的可变区之后,可显著提高其稳定性[52]。此外VNAR内部的二硫键也有利于提高其稳定性[28, 37, 51]。
5.2.3 较强的组织穿透性由于眼部特殊的构造,抗体等大分子药物通常只能通过玻璃体注射等方式给药,往往存在感染以及视网膜脱落等副作用。因此,急需开发组织穿透性强的药物,以便改变给药途径。葡萄膜炎是眼科常见的致盲性疾病,其成因复杂,多属于自身免疫性疾病。诱导共刺激因子配体(ICOSL)与共刺激因子(ICOS)相互作用,在T细胞活化过程中起着重要作用。通过阻断这种相互作用可以抑制T细胞的完全活化,从而改善疾病症状。Kovaleva等[53]利用ICOSL免疫的护士鲨建立免疫库,并淘选得到特异性识别ICOSL的VNAR。作者利用角膜划伤的小鼠模型研究了VNAR穿越组织的能力,通过滴加的方式给小鼠模型用药,药物包括VNAR、VNAR-Fc以及mAb,给药20 min之后,只有VNAR可以穿透角膜,在小鼠眼睛的前房被发现。该结果显示了VNAR具有较强的组织穿透性。
6 VNAR的制备技术 6.1 免疫库制备免疫库首先需要对鲨鱼等动物进行免疫,目前用于产生高亲和力IgNAR的免疫方案已有报道[54-55]。然而,与哺乳动物相比,鲨鱼的免疫周期通常较长[27, 38]。Dooley等以鸡蛋溶菌酶(HEL)为抗原免疫护士鲨,建立了首个VNAR噬菌体展示免疫文库,并从中淘选获得多株VNARs,其中性能最好的被命名为5A7,其亲和力为20 nmol/L[20]。需要注意的是,并不是所有测试的鲨鱼种类都能产生适应性IgNAR免疫应答。Crouch等[56]以人血清白蛋白(HSA)以及HEL作为抗原免疫斑点猫鲨Scyliorhinus canicula,经过长达37周的免疫程序,没有发现机体产生抗原特异性IgNAR的证据。
6.2 天然库由于天然库缺乏免疫过程中刺激机体所发生的体细胞突变,导致使用该策略淘选得到的VNARs的亲和力通常较低。不过使用数量较多的动物,通过增加文库库容以及多样性的方式,有助于淘选获得高亲和力的VNARs。Feng等[42]基于EASeL技术用6条护士鲨构建噬菌体展示天然库,其库容可达1.2×1010,作者从该天然库中淘选获得特异性识别癌症治疗相关抗原(GPC3、HER2和PD1)、病毒刺突蛋白(SARS和MERS)以及假单胞菌外毒素(PE38)的VNARs,其中1个识别PE38的VNAR具有较高的亲和力(10.1 nmol/L)。该研究表明利用库容大、多样性丰富的天然库也可淘选得到高亲和力的VNAR。
6.3 半合成库随着对VNAR结构及功能的认知,发现体内VNAR的多样性主要来源于长且高度突变的CDR3区。因此以VNAR作为框架,通过CDR3区的随机化构建半合成VNAR文库,可以获得类似甚至优于天然库的效果。Nuttall等以斑纹须鲨Orectolobus maculates VNAR为框架,通过随机化CDR3区(包含16或者18个氨基酸残基)构建了第1个VNAR噬菌体展示半合成文库,并利用其淘选得到针对AMA1的VNAR,其亲和力为200 nmol/L[57]。
7 VNAR的亲和力成熟策略 7.1 随机突变Nuttal等[49]利用易错PCR在针对AMA1的VNAR (12Y-2)的CDR1和CDR3区进行随机突变并建立突变库,随后通过严格淘选,使得VNAR突变株的亲和力提高了10倍。此外该研究团队还利用来源于Qβ噬菌体的低保真RNA聚合酶以及核糖体展示文库技术构建突变库,通过严格淘选,使得VNAR突变株的亲和力提高了23倍[58]。
7.2 定点突变Fennell等[59]以HEL特异性结合的VNAR为模型,利用核糖体展示技术以及易错PCR对VNAR序列中各氨基酸的功能进行了系统性研究。通过反复突变以及淘选,获得320个含有突变氨基酸并保留抗体活性的序列,经测序及分析,作者确定了保持结构及功能完整性的关键位点、与亲和力密切相关的位点以及允许高度突变的位点。其中3个位点氨基酸的突变(A1D、S61R和G62R)可以提高亲和力,最终结合这3种突变,使VNAR突变体的亲和常数达到460 pmol/L,比野生型5A7提高了20倍。该工作通过对VNAR序列中各氨基酸功能的系统性研究,为VNAR的定点突变提供了参考。
7.3 分步式突变Zielonka等[60]首先以条纹斑竹鲨Chiloscyllium plagiosum VNAR为框架,通过随机合成CDR3区(包含12个氨基酸残基)构建了酵母展示文库,并以3种肿瘤标志物上皮细胞粘附分子(EpCAM)、肝配蛋白A受体2 (EphA2)以及人丝氨酸蛋白酶(HTRA1)为靶分子淘选VNAR,经过3轮淘选后,收集VNARs并以其为模板随机合成CDR1区(包含5个氨基酸残基),并进行再次淘选。最后通过对淘选得到的VNARs序列分析,发现针对EpCAM的3株VNARs拥有完全相同的CDR3区且与最初淘选得到的VNAR序列相同,通过亲和力分析发现,CDR1区突变的3株VNARs的平均亲和常数为11.2 nmol/L,比原始VNAR的亲和力提高了65倍。针对HTRA1的VNAR也存在类似的现象。以上结果证明,通过对CDR区的分步式突变可以获得高亲和力的VNAR,这种CDR区的分步式体外亲和力成熟策略与护士鲨免疫系统中IgNAR亲和力成熟的过程类似。
8 VNAR的人源化改造通过序列分析,发现VNAR与人源抗体可变区之间氨基酸的同源性约为25%–30%[61],然而基于现有VNAR的晶体数据可知,VNAR的结构与人源抗体可变区的结构具有较高的相似度,因此可以通过框架移植实现对VNAR的人源化改造。目前首个被人源化改造的VNAR是由Müller等从人血清白蛋白(HSA)免疫的白斑角鲨Squalus acanthias噬菌体展示文库中分离获得,其最初被命名为E06。E06自身并不能用于疾病治疗,药物分子通过与E06进行融合可以增加在血清中的半衰期,从而提高药物的疗效[62]。为了降低E06的免疫原性,利用与VNAR相似度最高的人类免疫球蛋白可变区框架DPK9对E06进行人源化改造。结果发现,人源化E06的亲和力及特异性得到了较好的保留。随后该团队以人源化huE06v1.10为模板利用易错PCR进行随机突变并构建突变库,经过淘选获得一系列突变株。作者利用树突细胞-T细胞系统(DC-T)测试突变株的免疫原性,发现BA11等突变株几乎没有免疫原性。最终基于亲和力、免疫原性以及半衰期等数据,认为突变株BA11最具临床应用价值,并将其重命名为NDureTM[63-64]。
值得注意的是,除了上述传统人源化改造技术外,Nuttall等基于VNAR晶体结构类似于免疫球蛋白超家族I-set类群的现象,提出以人源I-set类群分子(如人类神经细胞粘附分子)为模板进行VNAR的人源化改造,从而降低VNAR免疫原性的设想[65]。
9 VNAR的应用VNAR所具备的分子量小、亲和力高、稳定性强、溶解度好、组织穿透性强以及可识别隐藏抗原表位等特性,使得其在应用时具有产品质量稳定、生产成本低、易储存以及货架期长等优点,在药物开发、体外诊断以及免疫检测等领域受到广泛的关注(表 1)。
| Name | Target | Species | Library type | Affinity | Application fields | References |
| T43 | TNFα | Heterodontus francisci | Immune library | NR | Drug | [66] |
| V13 | VEGF165 | Heterodontus francisci | Immune library | NR | Drug | [69] |
| VNAR1 | ICOSL | Ginglymostoma cirratum | Immune library | 1–11 nmol/L | Drug | [53] |
| 5A7 | HEL | Ginglymostoma cirratum | Immune library | 20 nmol/L | Drug | [48] |
| E06 | HSA | Squalus acanthias | Immune library | 200 pmol/L | Drug | [62] |
| D01 | Aurora-A | Orectolobus maculatus | Naive library | 2 μmol/L | Drug | [67] |
| 12A-9 | Kgp protease | Orectolobus maculatus | Semi-synthetic library | 130 nmol/L | Drug | [70] |
| H6 | HBeAg | Orectolobus maculatus | Semi-synthetic library | 53 nmol/L | Drug | [68] |
| 12F-11 | Tom70 | Orectolobus maculatus | Semi-synthetic library | 2 nmol/L | Drug | [71] |
| A05 | BAFF | Ginglymostoma cirratum | Semi-synthetic library | NR | Drug | [72] |
| 653 | VHVS | Triakis scyllium | Semi-synthetic library | NR | Drug | [73] |
| H8 | PfHRP2 | Orectolobus maculatus | Immune library | NR | Diagnostic | [74] |
| 12Y-1 | AMA-1 | Orectolobus maculatus | Semi-synthetic library | 200 nmol/L | Diagnostic | [57] |
| NR | EpCAM | Chiloscyllium plagiosum | Semi-synthetic library | 0.02–1 μmol/L | Diagnostic | [60] |
| DSTL096 | Ebola virus | Ginglymostoma cirratum | Immune library | NR | Immunoassay | [75] |
| SP1 | Cholera toxin | Squalus acanthias | Naive library | NR | Immunoassay | [41] |
| PE38-B6 | PE38 | Ginglymostoma cirratum | Naive library | 10.1 nmol/L | Immunoassay | [42] |
| NR | SEB | Squalus acanthias | Semi-synthetic library | 0.1–0.4 μmol/L | Immunoassay | [76] |
| TNFα: tumor necrosis factor α; VEGF165: vascular endothelial growth factor 165; ICOSL: inducible co stimulator ligand; HEL: hen egg lysozyme; HAS: human serum albumin; HBeAg: hepatitis B e Antigen; Tom70: human mitochondrial translocase receptor; BAFF: B-cell activating factor; VHVS: viral hemorrhagic septicemia virus; PfHRP2: histidine-rich protein 2; AMA-1: malarial apical membrane protein 1; EpCAM: epithelial cell adhesion molecule; PE38: pseudomonas exotoxin 38; SEB: staphylococcal enterotoxin B; NR: not reported. | ||||||
人肿瘤坏死因子TNFα (rhTNFα)是由免疫细胞分泌的一种小分子蛋白。细菌内毒素等刺激机体时会产生过量的rhTNFα,从而引发中毒性休克、肾上腺损害等严重疾病。因此,rhTNFα抑制剂的开发备受研究者的关注。Camacho-Villegas等[66]利用佛氏虎鲨Heterodontus francisci制备噬菌体免疫文库,经过4轮淘选分离获得针对rhTNFα的VNAR,并命名为T43。通过细胞实验证实T43具有中和rhTNFα的能力。同时利用脂多糖诱导的内毒素休克小鼠模型证实T43能显著降低小鼠的死亡率。T43所表现出的良好的治疗效果以及组织渗透性等,使其有潜力成为免疫治疗药物。
9.2 VNAR作为酶抑制剂的应用抗体作为酶抑制剂时,可以通过改变酶活性位点构象或者直接结合酶活性位点等方式实现对酶活性的抑制[47]。Aurora-A激酶是一类进化上高度保守的苏氨酸激酶,参与细胞有丝分裂过程,当其过量表达时,会导致出现非整数倍染色体,使得细胞增殖异常,最终诱发癌症。因此,Aurora-A激酶抑制剂的开发受到广泛关注。Burgess等[67]以斑纹须鲨VNAR为框架制备半合成库,获得特异性识别Aurora-A激酶的VNAR,命名为D01。晶体结构显示D01通过破坏Aurora-A激酶的Lys-Glu盐桥使得酶构象改变,从而失去活性。
Dooley等[37]利用鸡卵清溶菌酶(HEL)免疫的护士鲨构建噬菌体免疫文库,并淘选得到多株高亲和力的VNARs,包括5A7等[48]。随后Stanfield等通过5A7与HEL共结晶的方式,系统性研究了VNARs的晶体结构以及5A7与HEL的结合,发现5A7中只有CDR1和CDR3区会与HEL接触,且CDR3区的Arg100与Tyr101会深入到HEL的酶活性中心,从而抑制HEL的酶活性。
9.3 VNAR作为细胞内抗体的应用乙型肝炎病毒前体蛋白在内质网中被加工成分泌型乙型肝炎病毒抗原(HBeAg),后者可引起慢性感染。Walsh等[68]以斑纹须鲨VNAR为框架制备半合成库,并淘选到1株特异性结合乙型肝炎病毒前体蛋白的VNAR,命名为H6。为了评估H6作为细胞内抗体结合乙型肝炎病毒前体蛋白以减少HBeAg分泌的效果,H6基因被插入到pCMV/ER表达载体,并转入pTRE细胞中,通过蛋白印迹分析发现H6能够显著降低前体蛋白及HBeAg的含量,表明VNAR能在真核细胞内的还原性环境下功能性表达并发挥效应,显示了VNAR作为细胞内抗体的潜力。
9.4 VNAR在疾病诊断中的应用早期诊断对于疾病的及时发现及治疗至关重要,尤其是对于肿瘤等严重疾病。目前针对多种肿瘤标志物的VNAR被成功开发。Zielonka等[60]以条纹斑竹鲨VNAR为框架通过分步式随机合成CDR3区和CDR1区,构建了酵母展示文库,分别淘选得到识别3种肿瘤标志物(EpCAM、EphA2以及HTRA1)的VNAR,其亲和力都为纳摩尔级,为后续建立免疫学检测方法奠定了基础。
9.5 VNAR在致病微生物检测中的应用Goodchild等[75]利用埃博拉病毒免疫的小鼠和护士鲨制备噬菌体免疫文库,通过固相淘选法得到2株特异性识别病毒核蛋白(Viral nucleoprotein,NP)的VNAR,以及2株特异性识别埃博拉病毒基质蛋白(Viral matrix protein,VP40)的ScFv。其中NP蛋白与病毒RNA形成复合物并被病毒包膜包裹,通常难以产生针对NP的传统抗体。上述淘选结果证明VNAR倾向于识别传统抗体难以识别的抗原表位。作者发现VNAR与mAb及ScFv相比,对热变性具有更强的抵抗力。VNAR在80 ℃条件下处理3 h依然能保留约50%的生物活性,而mAb及ScFv则分别在70 ℃以及50 ℃下处理3 h后完全丧失生物活性。VNAR优越的热稳定性使其具有成为现场免疫检测试剂的潜力。
9.6 VNAR在食品安全危害因子检测中的应用Liu等[76]以白斑角鲨VNAR为框架通过随机合成不同长度的CDR3区构建了噬菌体合成文库,利用固相淘选法,获得特异性识别葡萄球菌肠毒素B (SEB)、蓖麻毒素A (BoNT/A)和肉毒杆菌毒素(Ricin)的VNAR。作者发现VNARs针对SEB、BoNT/A以及Ricin的最低检测限分别是10 ng/mL、40 ng/mL以及1 μg/mL,展示了VNAR应用于食品安全危害因子检测的潜力。
10 VNAR作为免疫试剂和治疗药物的优势和劣势VNAR是目前在脊椎动物中发现的尺寸最小的抗原结合域,以下因素使其作为免疫试剂或药物时具有优势:1)源于适应性免疫系统:IgNAR是板鳃亚纲鱼类适应性免疫系统的重要组成部分。漫长的进化过程以及严酷的生理环境使得VNAR形成独特的结构,具备极强的稳定性以及重折叠能力。同时通过抗原免疫利用IgNAR的体内亲和成熟可获得高亲和力的VNAR。2)尺寸小以及组织穿透性强:由于缺乏C′和C″的β链,导致VNAR的分子量仅为12 kDa。小尺寸使得VNAR作为药物时,具有较强的组织穿透性[53, 69]。3)识别隐藏抗原表位的倾向:与驼源VHH类似,VNAR的CDR3区能形成指凸结构,识别传统抗体无法识别的隐藏抗原表位。4) VNAR结构多样性大:同为单域抗体的驼源VHH仅存在类似于Ⅱ型VNAR的结构,即具有连接CDR1和CDR3的二硫键,而VNAR到目前为止至少存在5种不同的结构类型。因此,多样化的VNAR结构为其识别不同类型的靶分子奠定了基础。5)与人类进化距离远:由于鲨总目鱼类与人类在进化上距离较远,可用于开发针对保守的哺乳动物蛋白等靶点的抗体药物。当前离临床应用最近的NDureTM,其靶分子即为保守的哺乳动物蛋白HAS。6)易于大规模生产:VNAR结构简单,由单基因表达,可以通过各种表达系统进行大规模生产,有利于降低成本。7)商业竞争少:当前对于VNAR的研究较少,没有形成严密的专利保护。
当然,VNAR作为免疫试剂或治疗药物时也存在诸多不可回避的问题:1)鲨鱼饲养困难:普通实验室饲养及免疫鲨鱼存在较大困难。2)免疫周期长:鲨鱼免疫周期较长(4–6个月),而骆驼科动物则只需要3个月左右。较长的免疫周期会增加饲养成本以及抗体开发周期。3)免疫原性大:由于鲨总目鱼类与人类进化关系较远,因此在开发基于VNAR的药物时,VNAR可能存在较大的免疫原性,需要进行人源化改造。
11 结论与展望VNAR是迄今为止在脊椎动物中发现的尺寸最小的抗原结合域。由于制备困难等原因,目前国内外对其研究较少,特别是国内,鲜有团队开展相关工作。近年来,随着驼源VHH在各个领域日渐广泛的应用,尤其是首个VHH药物(Cablivi®)先后被EMA和FDA批准上市之后,同属单域抗体的VNAR也日益成为抗体领域的研究热点。与VHH相比,虽然针对多种靶分子的VNAR被淘选得到,但大多数只是研究了VNAR的特异性、亲和力和稳定性等性能,其作为免疫试剂或者药物的应用性研究则较少。然而VNAR较传统抗体所拥有的尺寸小、稳定性强、亲和力高、识别隐藏抗原表位以及易于大规模生产等优点,注定其在免疫检测和治疗领域具有广泛的应用潜力。更为重要的是,目前用于VNAR开发的鲨鱼种类不到10种,且未见鳐源VNAR的报道,而现存板鳃亚纲鱼类超过千余种(鲨总目约450种,鳐总目约560种),能够为VNAR的研究提供极为丰富的材料。有理由相信,随着研究的深入能够发现VNAR更多的结构及功能特性,在拓展VNAR来源的同时有望进一步丰富抗体小型化的理论基础。
| [1] |
Köhler G, Milstein C. Continuous cultures of fused cells secreting antibody of predefined specificity. Nature, 1975, 256(5517): 495-497. DOI:10.1038/256495a0 |
| [2] |
de la Torre BG, Albericio F. The pharmaceutical industry in 2018. An analysis of FDA drug approvals from the perspective of molecules. Molecules, 2019, 24(4): 809. DOI:10.3390/molecules24040809 |
| [3] |
Grilo AL, Mantalaris A. The increasingly human and profitable monoclonal antibody market. Trends Biotechnol, 2019, 37(1): 9-16. DOI:10.1016/j.tibtech.2018.05.014 |
| [4] |
Nelson AL. Antibody fragments: hope and hype. Mabs, 2010, 2(1): 77-83. DOI:10.4161/mabs.2.1.10786 |
| [5] |
Muyldermans S. Nanobodies: Natural single-domain antibodies. Annu Rev Bio, 2013, 82(1): 775-797. DOI:10.1146/annurev-biochem-063011-092449 |
| [6] |
Hamers-Casterman C, Atarhouch T, Muyldermans S, et al. Naturally occurring antibodies devoid of light chains. Nature, 1993, 363(6428): 446-448. DOI:10.1038/363446a0 |
| [7] |
Greenberg AS, Avila D, Hughes M, et al. A new antigen receptor gene family that undergoes rearrangement and extensive somatic diversification in sharks. Nature, 1995, 374(6518): 168-173. DOI:10.1038/374168a0 |
| [8] |
He T, Zhu J, Nie Y, et al. Nanobody technology for mycotoxin detection: Current status and prospects. Toxins, 2018, 10(5): 180. DOI:10.3390/toxins10050180 |
| [9] |
Chakravarty R, Goel S, Cai WB. Nanobody: the "Magic Bullet" for molecular imaging?. Theranostics, 2014, 4(4): 386-398. DOI:10.7150/thno.8006 |
| [10] |
Huang L, Muyldermans S, Saerens D. Nanobodies®: proficient tools in diagnostics. Exp Rev Mol Diagnost, 2010, 10(6): 777-785. DOI:10.1586/erm.10.62 |
| [11] |
Kong QM, Yao YB, Chen R, et al. Progress in nanobody and its application in diagnosis. Chin J Biotech, 2014, 30(9): 1351 (in Chinese). 孔庆明, 姚亚波, 陈睿, 等. 纳米抗体及其在诊断检测中的研究进展. 生物工程学报, 2014, 30(9): 1351. |
| [12] |
Siontorou CG. Nanobodies as novel agents for disease diagnosis and therapy. Int J Nanomedicine, 2013, 8: 4215-4227. |
| [13] |
Wu Y, Hao XJ, Li M. Application of nanobody in cancer treatment. Chin J Biotech, 2017, 33(7): 1085-1090 (in Chinese). 吴越, 郝秀静, 李敏. 纳米抗体在癌症治疗中的应用. 生物工程学报, 2017, 33(7): 1085-1090. |
| [14] |
Papermaster BW, Condie RM, Finstad J, et al. Evolution of the immune response: I. The phylogenetic development of adaptive immunologic responsiveness in vertebrates. J Exp Med, 1964, 119(1): 105-130. DOI:10.1084/jem.119.1.105 |
| [15] |
Shankey TV, Clem LW. Phylogeny of immunoglobulin structure and function-Ⅷ: Intermolecular heterogeneity of shark 19S IgM antibodies to pneumococcal polysaccharide. Mol Immunol, 1980, 17(3): 365-375. DOI:10.1016/0161-5890(80)90057-7 |
| [16] |
Morrow WJW, Harris JE, Davies D, et al. Isolation and partial characterization of dogfish (Scyliorhinus canicula) antibody. J Mar Biol Associat United Kingdom, 1983, 63(2): 409-418. DOI:10.1017/S0025315400070764 |
| [17] |
Rumfelt LL, Lohr RL, Dooley H, et al. Diversity and repertoire of IgW and IgM VH families in the newborn nurse shark. BMC Immunol, 2004, 5: 8. DOI:10.1186/1471-2172-5-8 |
| [18] |
Roux KH, Greenberg AS, Greene L, et al. Structural analysis of the nurse shark (new) antigen receptor (NAR): Molecular convergence of NAR and unusual mammalian immunoglobulins. Proc Natl Acad Sci USA, 1998, 95(20): 11804-11809. DOI:10.1073/pnas.95.20.11804 |
| [19] |
Flajnik MF, Kasahara M. Origin and evolution of the adaptive immune system: genetic events and selective pressures. Nat Rev Genet, 2010, 11(1): 47-59. |
| [20] |
Dooley H, Flajnik MF. Shark immunity bites back: affinity maturation and memory response in the nurse shark, Ginglymostoma cirratum. Eur J Immunol, 2005, 35(3): 936-945. DOI:10.1002/eji.200425760 |
| [21] |
Inoue JG, Miya M, Lam K, et al. Evolutionary origin and phylogeny of the modern holocephalans (Chondrichthyes: Chimaeriformes): a mitogenomic perspective. Mol Biol Evolut, 2010, 27(11): 2576-2586. DOI:10.1093/molbev/msq147 |
| [22] |
Nelson JS, Grande TC, Wilson MV. Fishes of the World. 5th ed. Hoboken: John Wiley & Sons, 2016.
|
| [23] |
Criscitiello MF, Saltis M, Flajnik MF. An evolutionarily mobile antigen receptor variable region gene: doubly rearranging NAR-TcR genes in sharks. Proc Natl Acad Sci USA, 2006, 103(13): 5036-5041. DOI:10.1073/pnas.0507074103 |
| [24] |
Venkatesh B, Kirkness EF, Loh YH, et al. Survey sequencing and comparative analysis of the elephant shark (Callorhinchus milii) genome. PLoS Biol, 2007, 5(4): e101. DOI:10.1371/journal.pbio.0050101 |
| [25] |
Venkatesh B, Lee AP, Ravi V, et al. Elephant shark genome provides unique insights into gnathostome evolution. Nature, 2014, 505(7482): 174-179. DOI:10.1038/nature12826 |
| [26] |
Pettinello R, Dooley H. The immunoglobulins of cold-blooded vertebrates. Biomolecules, 2014, 4(4): 1045-1069. DOI:10.3390/biom4041045 |
| [27] |
Flajnik MF. A cold-blooded view of adaptive immunity. Nat Rev Immunol, 2018, 18(7): 438-453. DOI:10.1038/s41577-018-0003-9 |
| [28] |
Streltsov VA, Varghese JN, Carmichael JA, et al. Structural evidence for evolution of shark Ig new antigen receptor variable domain antibodies from a cell-surface receptor. Proc Natl Acad Sci USA, 2004, 101(34): 12444-12449. DOI:10.1073/pnas.0403509101 |
| [29] |
Criscitiello MF. What the shark immune system can and cannot provide for the expanding design landscape of immunotherapy. Expert Opin Drug Dis, 2014, 9(7): 725-739. DOI:10.1517/17460441.2014.920818 |
| [30] |
Hsu E, Pulham N, Rumfelt LL, et al. The plasticity of immunoglobulin gene systems in evolution. Immunol Rev, 2006, 210(1): 8-26. |
| [31] |
Flajnik MF. Comparative analyses of immunoglobulin genes: surprises and portents. Nat Rev Immunol, 2002, 2(9): 688-698. DOI:10.1038/nri889 |
| [32] |
Diaz M, Stanfield RL, Greenberg AS, et al. Structural analysis, selection, and ontogeny of the shark new antigen receptor (IgNAR): identification of a new locus preferentially expressed in early development. Immunogenetics, 2002, 54(7): 501-512. DOI:10.1007/s00251-002-0479-z |
| [33] |
Diaz M, Velez J, Singh M, et al. Mutational pattern of the nurse shark antigen receptor gene (NAR) is similar to that of mammalian Ig genes and to spontaneous mutations in evolution: the translesion synthesis model of somatic hypermutation. Int Immunol, 1999, 11(5): 825-833. DOI:10.1093/intimm/11.5.825 |
| [34] |
Diaz M, Greenberg AS, Flajnik MF. Somatic hypermutation of the new antigen receptor gene (NAR) in the nurse shark does not generate the repertoire: Possible role in antigen-driven reactions in the absence of germinal centers. Proc Natl Acad Sci USA, 1998, 95(24): 14343-14348. DOI:10.1073/pnas.95.24.14343 |
| [35] |
Stanfield RL, Dooley H, Flajnik MF, et al. Crystal structure of a shark single-domain antibody V region in complex with lysozyme. Science, 2004, 305(5691): 1770-1773. DOI:10.1126/science.1101148 |
| [36] |
Stanfield RL, Dooley H, Verdino P, et al. Maturation of shark single-domain (IgNAR) antibodies: evidence for induced-fit binding. J Mol Biol, 2007, 367(2): 358-372. DOI:10.1016/j.jmb.2006.12.045 |
| [37] |
Streltsov VA, Carmichael JA, Nuttall SD. Structure of a shark IgNAR antibody variable domain and modeling of an early-developmental isotype. Prot Sci, 2005, 14(11): 2901-2909. DOI:10.1110/ps.051709505 |
| [38] |
Kovaleva M, Ferguson L, Steven J, et al. Shark variable new antigen receptor biologics-a novel technology platform for therapeutic drug development. Exp Opin Biol Ther, 2014, 14(10): 1527-1539. DOI:10.1517/14712598.2014.937701 |
| [39] |
Ubah OC, Barelle CJ, Buschhaus MJ, et al. Phage display derived IgNAR V region binding domains for therapeutic development. Curr Pharm Des, 2016, 22(43): 6519-6526. |
| [40] |
Matz H, Dooley H. Shark IgNAR-derived binding domains as potential diagnostic and therapeutic agents. Dev Comp Immunol, 2019, 90: 100-107. |
| [41] |
Liu JL, Anderson GP, Delehanty JB, et al. Selection of cholera toxin specific IgNAR single-domain antibodies from a naïve shark library. Mol Immunol, 2007, 44(7): 1775-1783. DOI:10.1016/j.molimm.2006.07.299 |
| [42] |
Feng MQ, Bian HJ, Wu XL, et al. Construction and next-generation sequencing analysis of a large phage-displayed VNAR single-domain antibody library from six naive nurse sharks. Antibody Therapeut, 2019, 2(1): 1-11. DOI:10.1093/abt/tby011 |
| [43] |
Zielonka S, Empting M, Grzeschik J, et al. Structural insights and biomedical potential of IgNAR scaffolds from sharks. mAbs, 2015, 7(1): 15-25. DOI:10.4161/19420862.2015.989032 |
| [44] |
Henderson KA, Streltsov VA, Coley AM, et al. Structure of an IgNAR-AMA1 complex: targeting a conserved hydrophobic cleft broadens malarial strain recognition. Structure, 2007, 15(11): 1452-1466. DOI:10.1016/j.str.2007.09.011 |
| [45] |
Streltsov VA, Varghese JN, Masters CL, et al. Crystal structure of the amyloid-β p3 fragment provides a model for oligomer formation in alzheimer's disease. J Neurosci, 2011, 31(4): 1419-1426. DOI:10.1523/JNEUROSCI.4259-10.2011 |
| [46] |
Zielonka S, Empting M, Konning D, et al. The shark strikes twice: hypervariable loop 2 of Shark IgNAR antibody variable domains and its potential to function as an autonomous paratope. Mar Biotechnol, 2015, 17(4): 386-392. DOI:10.1007/s10126-015-9642-z |
| [47] |
Lauwereys M, Ghahroudi MA, Desmyter A, et al. Potent enzyme inhibitors derived from dromedary heavy-chain antibodies. EMBO J, 1998, 17(13): 3512-3520. |
| [48] |
Dooley H, Flajnik MF, Porter AJ. Selection and characterization of naturally occurring single-domain (IgNAR) antibody fragments from immunized sharks by phage display. Mol Immunol, 2004, 40(1): 25-33. |
| [49] |
Nuttall SD, Humberstone KS, Krishnan UV, et al. Selection and affinity maturation of IgNAR variable domains targeting Plasmodium falciparum AMA1. Proteins, 2004, 55(1): 187-197. |
| [50] |
Liu JL, Zabetakis D, Brown JC, et al. Thermal stability and refolding capability of shark derived single domain antibodies. Mol Immunol, 2014, 59(2): 194-199. |
| [51] |
Griffiths K, Dolezal O, Parisi K, et al. Shark Variable New Antigen Receptor (VNAR) single domain antibody fragments: stability and diagnostic applications. Antibodies, 2013, 2(1): 66-81. |
| [52] |
Feige MJ, Grawert MA, Marcinowski M, et al. The structural analysis of shark IgNAR antibodies reveals evolutionary principles of immunoglobulins. Proc Natl Acad Sci USA, 2014, 111(22): 8155-8160. DOI:10.1073/pnas.1321502111 |
| [53] |
Kovaleva M, Johnson K, Steven J, et al. Therapeutic potential of shark anti-ICOSL VNAR domains is exemplified in a murine model of autoimmune non-infectious uveitis. Front Immunol, 2017, 8: 1211. DOI:10.3389/fimmu.2017.01211 |
| [54] |
Müller MR, O'Dwyer R, Kovaleva M, et al. Generation and isolation of target-specific single-domain antibodies from shark immune repertoires//Chames P, Ed. Antibody Engineering. Totowa, NJ: Humana Press, 2012, 907: 177-194.
|
| [55] |
Flajnik MF, Dooley H. The generation and selection of single-domain, V region libraries from nurse sharks//Aitken R, Ed. Antibody Phage Display. New York: Humana Press, 2009: 71-82.
|
| [56] |
Crouch K, Smith LE, Williams R, et al. Humoral immune response of the small-spotted catshark, Scyliorhinus canicula. Fish Shellf Immunol, 2013, 34(5): 1158-1169. DOI:10.1016/j.fsi.2013.01.025 |
| [57] |
Nuttall SD, Krishnan UV, Hattarki M, et al. Isolation of the new antigen receptor from wobbegong sharks, and use as a scaffold for the display of protein loop libraries. Mol Immunol, 2001, 38(4): 313-326. DOI:10.1016/S0161-5890(01)00057-8 |
| [58] |
Kopsidas G, Roberts AS, Coia G, et al. In vitro improvement of a shark IgNAR antibody by Qβ replicase mutation and ribosome display mimics in vivo affinity maturation. Immunol Lett, 2006, 107(2): 163-168. |
| [59] |
Fennell BJ, Darmanin-Sheehan A, Hufton SE, et al. Dissection of the IgNAR V domain: molecular scanning and orthologue database mining define novel IgNAR hallmarks and affinity maturation mechanisms. J Mol Biol, 2010, 400(2): 155-170. DOI:10.1016/j.jmb.2010.04.061 |
| [60] |
Zielonka S, Weber N, Becker S, et al. Shark Attack: High affinity binding proteins derived from shark vNAR domains by stepwise in vitro affinity maturation. J Biotechnol, 2014, 191: 236-245. DOI:10.1016/j.jbiotec.2014.04.023 |
| [61] |
Dooley H, Flajnik MF. Antibody repertoire development in cartilaginous fish. Dev Comparat Immunol, 2006, 30(1/2): 43-56. |
| [62] |
Müller MR, Saunders K, Grace C, et al. Improving the pharmacokinetic properties of biologics by fusion to an anti-HSA shark VNAR domain. mAbs, 2012, 4(6): 673-685. DOI:10.4161/mabs.22242 |
| [63] |
Kovalenko OV, Olland A, Piche-Nicholas N, et al. Atypical antigen recognition mode of a shark immunoglobulin new antigen receptor (IgNAR) variable domain characterized by humanization and structural analysis. J Bio Chem, 2013, 288(24): 17408-17419. |
| [64] |
Steven J, Müller MR, Carvalho MF, et al. In vitro maturation of a humanized shark vnar domain to improve its biophysical properties to facilitate clinical development. Front Immunol, 2017, 8: 1361. DOI:10.3389/fimmu.2017.01361 |
| [65] |
Nuttall SD. Overview and discovery of IgNARs and generation of VNARs//Saerens D, Muyldermans S, Eds. Single Domain Antibodies. Totowa, NJ: Humana Press, 2012, 911: 27-36.
|
| [66] |
Camacho-Villegas T, Mata-Gonzalez T, Paniagua-Solis J, et al. Human TNF cytokine neutralization with a vNAR from Heterodontus francisci shark: a potential therapeutic use. mAbs, 2013, 5(1): 80-85. DOI:10.4161/mabs.22593 |
| [67] |
Burgess SG, Oleksy A, Cavazza T, et al. Allosteric inhibition of Aurora-A kinase by a synthetic vNAR domain. Open Biol, 2016, 6(7): 160089. DOI:10.1098/rsob.160089 |
| [68] |
Walsh R, Nuttall S, Revill P, et al. Targeting the hepatitis B virus precore antigen with a novel IgNAR single variable domain intrabody. Virology, 2011, 411(1): 132-141. DOI:10.1016/j.virol.2010.12.034 |
| [69] |
Camacho-Villegas TA, Mata-Gonzalez MT, Garcia-Ubbelohd W, et al. Intraocular penetration of a vNAR: in vivo and in vitro VEGF165 neutralization. Mar Drugs, 2018, 16(4): 113. DOI:10.3390/md16040113 |
| [70] |
Nuttall SD, Krishnan UV, Doughty L, et al. A naturally occurring NAR variable domain binds the Kgp protease from Porphyromonas gingivalis. FEBS Lett, 2002, 516(1/3): 80-86. |
| [71] |
Nuttall SD, Krishnan UV, Doughty L, et al. Isolation and characterization of an IgNAR variable domain specific for the human mitochondrial translocase receptor Tom70. Eur J Biochem, 2003, 270(17): 3543-3554. DOI:10.1046/j.1432-1033.2003.03737.x |
| [72] |
Häsler J, Flajnik MF, Williams G, et al. VNAR single-domain antibodies specific for BAFF inhibit B cell development by molecular mimicry. Mol Immunol, 2016, 75: 28-37. DOI:10.1016/j.molimm.2016.05.009 |
| [73] |
Ohtani M, Hikima JI, Jung TS, et al. Variable domain antibodies specific for viral hemorrhagic septicemia virus (VHSV) selected from a randomized IgNAR phage display library. Fish Shellf Immunol, 2013, 34(2): 724-728. |
| [74] |
Leow CH, Fischer K, Leow CY, et al. Isolation and characterization of malaria PfHRP2 specific VNAR antibody fragments from immunized shark phage display library. Malaria J, 2018, 17: 383. DOI:10.1186/s12936-018-2531-y |
| [75] |
Goodchild SA, Dooley H, Schoepp RJ, et al. Isolation and characterisation of Ebolavirus-specific recombinant antibody fragments from murine and shark immune libraries. Mol Immunol, 2011, 48(15/16): 2027-2037. |
| [76] |
Liu JL, Anderson GP, Goldman ER. Isolation of anti-toxin single domain antibodies from a semi-synthetic spiny dogfish shark display library. BMC Biotechnol, 2007, 7: 78. DOI:10.1186/1472-6750-7-78 |
 2020, Vol. 36
2020, Vol. 36





