中国科学院微生物研究所、中国微生物学会主办
文章信息
- 许航, 朱思橙, 张文辉, 季荣
- Xu Hang, Zhu Sicheng, Zhang Wenhui, Ji Rong
- 好氧活性污泥体系中磺胺类抗生素生物降解的研究进展
- Advances in biodegradation of sulfonamides antibiotics in aerobic activated sludge system
- 生物工程学报, 2021, 37(10): 3459-3474
- Chinese Journal of Biotechnology, 2021, 37(10): 3459-3474
- 10.13345/j.cjb.210428
-
文章历史
- Received: June 5, 2021
- Accepted: August 17, 2021
2. 南京大学泉州环保产业研究院,福建 泉州 362000
2. Quanzhou Institute for Environmental Protection Industry, Nanjing University, Quanzhou 362000, Fujian, China
磺胺类抗生素(Sulfonamides,SAs) 是一类被广泛应用的抗菌药物,具有广谱抗菌、性质稳定、使用便捷、产量大、价格低、品种多等特点[1]。该类化合物是以对位氨基苯磺酰胺为基本结构的衍生物(图 1),是对氨基苯甲酸的结构类似物,因此可通过竞争二氢蝶酸合成酶(Dihydropteroate synthase,DHPS) 的活性位点来抑制细菌中叶酸的生物合成进而发挥抗菌作用[2]。由于SAs在人类及畜禽肠道的可降解性较差,相当一部分该类药物会伴随人类及畜禽粪便和尿液的排放进入水生生态系统[3-5]。污水处理厂作为生活及农业废水收集并进一步处理的重要场所,成为SAs重要的汇。住房与城乡建设部数据显示,2019年我国城市污水排放量为5 546 474万m3,污水处理厂达到2 471座,处理能力达到17 863.17万m3/d。然而现有污水处理工艺并不能完全去除SAs[6-7],其主要原因是现有工艺更关注氮、磷及总有机碳(Total organic carbon,TOC) 的去除而忽视SAs等有机微污染物的存在。我国是抗生素使用大国,每年抗生素的使用量可达到90 000 t以上,长期的抗生素暴露会产生环境风险。有研究表明,SAs在水环境中浓度为ng/L水平[8-10],该浓度水平的SAs长期暴露会改变水环境中的微生物群落结构和功能[11],如活性污泥中磺胺甲噁唑(SMX) 浓度提高会增加其中微生物的耐药性并加速抗性基因(如sul 1、sul 2) 的传播[12-13],且显著抑制水体中氨氮的去除[11]。
目前,已有研究通过物理、化学和生物方法尝试在污水处理中的各个环节加速对SAs的降解转化,以实现对该类药物的高效去除。其中,吸附法作为最常用的物理方法,被用于去除水中微量有机污染物。但由于吸附剂的能力有限,故该方法未被广泛应用[14]。此外,高级氧化技术,包括Fenton法、紫外光照射、臭氧氧化、γ射线分解、超声波分解和电化学氧化等是最常用的化学方法,该类方法通过一系列的自由基链式反应进攻抗生素分子,使抗生素发生断键、开环和羟基化反应,从而实现SAs的有效转化。然而,应用该类化学方法时,SAs的矿化极低,这表明转化过程的中间产物很难被彻底降解[14-17]。与物理和化学方法相比,生物方法具有成本低、效率高和环境友好等特点。污水处理厂最常使用好氧活性污泥法,即利用悬浮生长的微生物絮体对有机污水进行好氧处理,该方法已被证实可以实现SAs的有效去除[18]。目前已有好氧活性污泥对SAs生物降解的相关研究,本文将围绕这些典型的SAs,对其在好氧活性污泥体系中的生物降解机制、生物降解途径及影响生物降解的环境因素的相关研究进展加以总结,以期为今后SAs生物降解的研究提供参考。
1 磺胺类抗生素的生物降解机制目前已被广泛应用的SAs种类繁多,主要包括SMX和磺胺嘧啶(SDZ) 等较常使用的典型SAs (表 1),但却可以被同种降解菌进行转化,这是因为这类药物降解时所发生的酶催化反应模式相似[19-20]。污水处理厂采用的好氧活性污泥体系对SAs的生物降解机理主要包括两种,即被作为唯一碳源或氮源直接利用和共代谢作用。
| Names | Chemical structures | Molecular weight (g/mol) | pKa | Water solubility (mg/L) |
| Sulfacetamide (SA) | 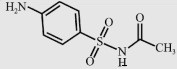 |
214.2 | 5.22 | 100 |
| Sulfadimethoxine (SDM) | 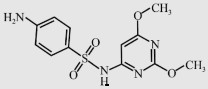 |
310.3 | 5.94 | 46.3 |
| Sulfadiazine (SDZ) |  |
250.3 | 1.6/6.5 | 77 |
| Sulfamonomethoxine (SMM) |
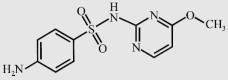 |
280.3 | 5.94 | 4 030 |
| Sulfamerazine (SMR) | 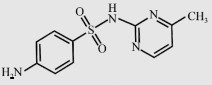 |
264.3 | 2.29 | 202 |
| Sulfamethizole (SMT) |  |
270.3 | 5.45 | 529 |
| Sulfamethoxazole (SMX) | 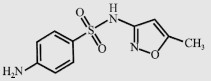 |
253.3 | 1.6/6.4 | 610 |
| Sulfadimidine (SM2)/Sulfamethazine (SMZ) | 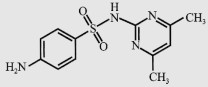 |
278.3 | 2.8/7.6 | 1 500 |
| Sulfapyridine (SPY) | 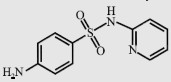 |
249.3 | 8.48 | < 1 000 |
| Sulfaquinoxaline (SQX) |
 |
300.3 | 2.3/6.0 | 47.9 |
| Sulfamethoxypyridazine (STP) |  |
280.3 | 6.7 | 579.5 |
| Sulfathiazole (STZ) | 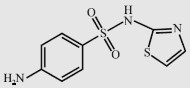 |
255.3 | 7.2 | 500 |
当体系中污染物浓度较高时,硝化活性污泥中的某些异养菌可以利用SAs作为唯一碳源或氮源生长。目前已有一些研究从驯化的活性污泥中分离出以SAs为碳源或氮源的菌株[21-27],如无色杆菌属Achromobacter、不动杆菌属Acinetobacter、微杆菌属Microbacteriu、假单胞菌属Pseudomonas和红球菌属Rhodococcus,在以SAs为唯一碳源时会将其矿化(表 2)。其中,从好氧活性污泥中驯化分离得到的Achromobacter、Acinetobacter和Microbacterium可以利用SAs作为底物进行初级代谢,被证实可以实现SAs的矿化[25, 28-30]。Achromobacter可以利用SAs作为唯一碳源生长。在30 ℃、pH 7.0时,以SMX为底物培养Achromobacter sp. BR3 16 d后,SMX的矿化率可达到24%–44%。Achromobacter sp. S-3暴露于磺胺二甲嘧啶(SMZ) 5–25 d后,对SMZ的去除率可达到45%–80%[28, 30]。反硝化无色杆菌Achromobacter denitrificans除了可以在25 d内实现SMX 70%的矿化以外,还可以利用SDZ、SMZ、磺胺吡啶(SPY) 和磺胺甲氧基哒嗪(STP)为唯一碳源和氮源在56 h内实现95%以上的降解[25],这表明Achromobacter对不同种类的SAs可能存在相同的利用方式。Acinetobacter在暴露于磺胺二甲氧嗪(SDM)、磺胺甲噻二唑(SMT)、SMX和SMZ的反应器中为主要的优势种,说明隶属于该属的菌株可能具有降解这类物质的潜力。另外,已经有研究通过分离得到Acinetobacter纯培养,结果表明Acinetobacter能够在25 h内实现对SDZ、SMT和SMX的完全降解,矿化率分别为17.5%、20.5%和98.8%,这一结果证明了Acinetobacter是SAs的降解菌;结合代谢产物鉴定的结果,作者认为该类物质杂环中的甲基可诱导Acinetobacter分泌甲基化酶进而催化降解反应[1, 15, 30-32]。还有研究从处理抗生素污染废水的膜生物反应器和驯化的活性污泥中分离出微杆菌属的菌株(Microbacterium sp. BR1、Microbacterium sp. SMXB24和SMX348),在以SMX为唯一碳源时,能够实现对SMX的快速转化和矿化,Microbacterium sp. BR1在25 d内对SMX的矿化率可达到40%[23, 28, 33]。除了对SMX的降解效果明显外,Microbacterium也能够降解其他类型的SAs。Ricken等[33]的研究结果表明Microbacterium在3.0–24.5 h内可以实现对SDZ、SMZ、SDM和SMT的完全降解。
| Strains or consortium contributing to sulfonamides biodegradation | Removal rate | Mechanism | References |
| Sulfamethoxazole | |||
| Acinetobacter sp. | 100%
(Initial concentration: 5–240 mg/L Exposure time: 7 h–10 d) |
/ | [44] |
| Achromobacter sp. JL9 | 0.575 mg/(L·h) | Cometabolism | [24] |
| Acidobacteria GP7 Arthrobacter SMK-1 |
/ | / | [35] |
| Microbacterium sp. | 1.25–1.70 mg/(L·d) | / | [23]
|
| Rhodococcus equi | 15%–29%
(Initial concentration: 6 mg/L Exposure time: 120 h) |
Aromatic amine N-acetyltransferase |
[21]
|
| Pseudomonas aeruginosa | < 5.6%
(Initial concentration: 6 mg/L Exposure time: 120 h) |
Aromatic amine N-acetyltransferase and DHPS | [21] |
| Paracoccus meganema | / | / | [22] |
| Pseudomonas sp. | 2.5 mg/(L·d) | Carbon source | [23] |
| Microbacterium sp. BR1 and Rhodococcus sp. BR2 | Mineralization rate 50%
(Initial concentration: 0.33 MBq/mmol Exposure time: 400 h) |
Carbon source | [28]
|
| Achromobacter denitrificans PR1 and Leucobacter sp. GP | / | Flavin monooxygenase | [27] |
| Conventional activated sludge (CAS) | 24%–100%
(Initial concentration: 100–200 mg/L Exposure time: 0–10 d) |
/ | [1, 19, 45-49] |
| CAS | 100%
(Initial concentration: 10 mg/L Exposure time: 14 d) |
Co-metabolism/
Carbon source |
[50]
|
| Acetyl-sulfamethoxazole | |||
| CAS | 34%– > 94%
(Initial concentration: 8 μg/L Exposure time: 144 h) |
Peptidase | [51]
|
| Sulfamethoxazole | |||
| Achromobacter denitrificans PR1 | 100%
(Initial concentration: 0.1 mmol/L Exposure time: 56 h) |
Carbon/Nitrogen source | [25]
|
| Paenarthrobacter ureafaciens | 80%
(Initial concentration: 100 mg/L Exposure time: 60 d) |
/ | [52]
|
| CAS | 20%– > 90%
(Initial concentration: 8 μg/L Exposure time: 600 h) |
/ | [1, 19, 53-54]
|
| Nitrifying activated sludge (NAS) | 45%
(Initial concentration: 500 μg/L Exposure time: 50 h) |
Co-metabolism | [55]
|
| Sulfadiazine | |||
| Arthrobacter sp.
(Arthrobacter sp. D2) and Pimelobacter sp. LG209 |
100%
(Initial concentration: 50–200 mg/L Exposure time: 25–140 h) |
Carbon/Nitrogen source | [26, 30]
|
| CAS | 73%
(Initial concentration: (95.63±5.82) ng/L) |
Co-metabolism | [43]
|
| NAS | 35%–97.4%
(Initial concentration: 8–50 μg/L Exposure time: 46–144 h) |
Co-metabolism | [55-56]
|
| Sulfapyridine | |||
| Achromobacter denitrificans PR1 | 100%
(Initial concentration: 0.1 mmol/L Exposure time: 56 h) |
Carbon/Nitrogen source | [25]
|
| Sulfamethoxypyridazine | |||
| Achromobacter denitrificans PR1 | 98%±2%
(Initial concentration: 0.1 mmol/L Exposure time: 56 h) |
Carbon/Nitrogen source | [25]
|
| Sulfathiazole | |||
| Achromobacter denitrificans PR1 | 47%±13%
(Initial concentration: 0.1 mmol/L Exposure time: 56 h) |
Carbon/Nitrogen source | [25]
|
| CAS | 30%–97%
(Initial concentration: 20 μg/L Exposure time: 20 d) |
/ | [1, 19, 47] |
| NAS | 85%
(Initial concentration: 500 μg/L Exposure time: 50 h) |
Co-metabolism | [55]
|
| Sulfacetamide | |||
| Achromobacter denitrificans PR1 | 10%±1%
(Initial concentration: 0.1 mmol/L Exposure time: 56 h) |
Carbon/Nitrogen source | [25]
|
实际上,这些微生物主要通过分泌某些酶进而催化磺胺类物质的降解。从活性污泥中分离的铜绿假单胞菌Pseudomonas aeruginosa和马红球菌Rhodococcus equi可以利用SMX为唯一碳源生长,17 d的矿化率可达40%。有研究表明P. aeruginosa和R. equi产生的芳胺N-乙酰转移酶(N-acetyltransferases,NATs) 对芳香胺类物质有底物特异性,可以将SMX作为底物反应。此外,SMX与P. aeruginosa释放的DHPS上活性位点结合,该过程对该酶的活性产生竞争性抑制。除了可以合成DHPS以外,一些微生物携带有与磺胺降解相关的基因,该类基因可指导合成与磺胺降解直接相关的酶进而催化反应。有研究报道Microbacterium sp. BR1中的某一基因簇与SMX的降解有关。该基因簇包括一组编码两种单加氧酶(Monooxygenases,Sad A and Sad B) 以及黄素单核苷酸还原酶(Flavin mononucleotide reductase,Sad C) 的基因,其中Sad A和Sad C攻击磺胺分子导致4-氨基苯酚的释放,4-氨基苯酚在Sad B和Sad C的催化作用下可产生1, 2, 4-三羟基苯[34]。Reis等从活性污泥中分离得到含无色杆菌Achromobacter和亮杆菌Leucobacter的复合体,该复合体可在50 min内实现SMX的快速转化。作者结合宏基因组学、qPCR和RT-qRCR等技术手段分析发现该复合体中的Leucobacter含有磺胺单加氧酶基因(sad A) 的同源物,该基因指导合成的酶可能催化SMX苯环上的羟基化反应[23, 27-28]。
除了利用传统的分离培养方法并通过矿化率判断微生物是否可以直接利用磺胺类物质作为碳源和氮源以外,近年来有相关研究利用稳定同位素探针(Stable isotope probing,SIP) 技术,更为直接地反映了真实环境中原位微生物对SAs的降解作用。Song等[35]通过在活性污泥中接入12C-SMX和13C-SMX,提取污泥样品DNA并通过氯化铯密度梯度离心法区分12C和13C标记的DNA进而找出可同化SMX的微生物。研究结合SIP技术、高通量测序技术和构建分子生态网络发现活性污泥体系中的γ变形菌门γ-proteobacteria、浮霉菌门Planctomycetaceae、酸杆菌门Acidobacteria和俭菌总门Parcubacteria的相对丰度与SMX降解相关。然而通过传统培养方法从该污泥中分离获得的Arthrobacter SMK-1并不属于这4个菌门。因此,SIP技术和其他分子生物学技术的联用可以揭示无法通过传统培养方法得到的潜在SMX降解菌。
1.2 共代谢作用微生物的共代谢作用(Co-metabolism) 是指微生物以某一初级底物作为生长的能量来源,同时该底物可诱导产生某种具有广谱性的酶,这类酶可以在催化初级底物的同时催化其他物质的转化。在污水处理系统中,一些有机微污染物(Organic micropollutants,OMPs) 浓度较低,无法提供微生物生长所需的全部能源,故其更易通过共代谢的方式被生物转化[35]。目前,SAs已被证实可通过共代谢作用被好养活性污泥中的自养或异养微生物转化。诱导共代谢作用的初级底物主要包括醋酸盐、琥珀酸盐、葡萄糖、氨氮等参与重要生命过程或较易被微生物利用的物质[36-38]。这些初级底物的存在,一方面可以通过增加微生物的生物量来提高对SAs的降解,另一方面可以使微生物在利用初级底物的同时释放某些酶进而催化抗生素的转化。
近年来,已有一些研究从驯化的活性污泥中分离出可通过共代谢作用降解SAs的菌株[21-27],如无色杆菌属Achromobacter[24, 27, 29, 39]和红球菌属Rhodococcus[29, 40] (表 2)。当以醋酸盐或琥珀酸盐与SMX作为底物培养Achromobacter sp. JL9和Achromobacter denitrificans PR1时,SMX的降解效率显著升高[24-25, 39]。当以葡萄糖和SMX/SMZ为底物时,SMX/SMZ的降解效率同样显著提高。此外,马红球菌Rhodococcus equi可以转化SMX为乙酰化的SMX。这种乙酰-SMX在仅以SMX为唯一碳源的处理组中并未被检出,这表明葡萄糖的存在有利于R. equi的生长从而介导某些酶(如对芳香胺有特异性的芳胺N-乙酰转移酶、酰胺酶、水解苯胺的氨基甲酸酯酶或水解N-乙酰化化合物的N-乙酰-苯乙胺水解酶) 的产生,进而催化产生N-乙酰化SMX和N-羟基乙酰化SMX等转化产物[28, 40]。除以上已经验证可通过共代谢作用转化SAs的微生物以外,还有一些异养微生物被认为具有共代谢该类药物的潜力。SMX连续暴露8 d后,活性污泥反应器中的甲基杆菌属Meganema和副球菌属Paracoccus微生物丰度增加。这两类微生物在利用乙酸钠的同时可能通过共代谢作用实现对SMX的转化[22]。
除了异养微生物以外,一些自养微生物也可以实现SAs的共代谢。在污水处理过程中,发挥共代谢作用的是一类与氮循环有关的微生物[41],该类微生物参与的硝化过程可以通过共代谢作用实现SAs的生物降解[42]。Zheng等[43]发现随着硝化活性污泥中亚硝化单胞菌Nitrosomonas sp. 相对丰度的增加,体系中抗生素的浓度逐渐降低,这表明硝化细菌可能在硝化过程中通过酶促反应参与了某些抗生素的共代谢。实际上,发挥这种共代谢作用的主要是氨氧化细菌(Ammonia oxidizing bacteria,AOB),主要是通过氨氧化作用来实现SAs的生物转化[53, 55, 57-58]。
除了AOB的相对丰度,氨氧化速率(Ammonia oxiding rate,AOR) 也会影响SAs的去除,Kassotaki等[57]发现在硝化活性污泥中,氨氧化速率与SMX的去除率呈正相关。催化氨氧化反应的酶是位于AOB细胞膜上的氨单加氧酶(Ammonia monooxygenase,AMO),这类酶可以介导SMX的生物转化,而加入AMO酶抑制剂烯丙基硫脲(Allyl thiourea,ATU) 后SMX几乎不发生降解转化。该结果表明AMO酶在催化氨氮到羟胺这一步反应的同时介导了SMX的生物转化。事实上,AMO酶是通过氨单加氧酶基因(amo ABC) 指导合成的,SAs的暴露会影响amo基因的表达。有研究表明,SAs的暴露增加了amo A基因的表达,进而促进了抗生素的降解转化[59]。除了SMX以外,其他的SAs也可被AOB转化。在硝化活性污泥体系中,SDZ只在氨氧化活性组中有较高的降解效率,在无氨氮或加入ATU的氨氧化活性抑制组中降解效果差[56, 60]。两株氨氧化菌(Nitrosomomas nitrosa Nm90和Nitrospira inopinata) 分别与SDZ、SMZ、磺胺间甲氧嘧啶(SMM) 和磺胺噻唑(STZ) 共培养时,在氨氧化活性组中这些物质均可被转化;在氨氧化活性抑制组中,4种物质的浓度变化不大,该结果进一步证明了AOB对SAs转化主要通过AMO酶的共代谢作用实现[61]。
2 好氧活性污泥中磺胺类抗生素降解的环境影响因素好氧活性污泥体系中环境因子(如温度、pH值、底物浓度、培养时间等) 是影响这类酶催化的生物降解效率的重要因素,因此可以通过调整体系中的环境因子来提高降解菌群的活性,从而直接或间接加速SAs的生物降解速率。
2.1 温度温度主要通过影响SAs的溶解度和微生物的活性来影响降解效率。一方面,温度升高使得SAs的溶解度增大[17],这会直接影响SAs的环境行为;另一方面,SAs的降解菌有其最适生长温度,在一定范围内相关降解酶类的活性和降解效率才能保持最佳[44]。这实际上是因为温度会影响微生物某些关键基因的表达。例如在4 ℃、15 ℃、25 ℃和37 ℃条件下分别培养了含有氨氧化菌的沉积物样品,结果表明37 ℃时氨氧化菌的amo A基因丰度显著高于其他温度处理组[62],而由amo A基因表达合成的AMO酶被认为是参与SAs生物转化的重要催化剂。因此,通过控制合适的环境温度,可以强化某些关键基因的表达并提高关键酶的活性以促进生物降解反应。
2.2 pH物质形态受pH的影响进而影响生物降解。以SMX为例,当环境pH低于或高于其pKa时,SMX以离子形式存在。SAs形态不同,其环境行为也不同[63]。离子态的差异会影响其在污泥上的吸附和生物转化反应的发生,进而影响其在活性污泥体系中的降解。此外,环境pH还可以通过影响微生物的群落组成,进一步影响SAs的降解[64]。不同类型的磺胺降解菌,其最适生长pH范围也存在差异。以氨氧化菌为例,当环境中pH呈碱性时,氨氧化菌的活性更高,有利于其通过共代谢作用转化SAs[56, 61]。因此,pH主要通过影响SAs的存在形态和微生物生长这两方面进而影响降解效率。
2.3 抗生素的初始浓度当体系中的SAs浓度过低时,则无法为微生物的生长提供足量的能源,这时微生物将无法将其作为初级底物进行代谢,故降解效率较低。当底物浓度升高时,微生物的降解效率则增加。但当SAs浓度高于某一水平时,微生物的活性又会受到抑制从而降低药物的去除效率[45, 65]。
2.4 其他碳源其他碳源可通过竞争性和非竞争性抑制SAs降解酶的活性,以进一步影响这类药物的生物降解。有研究表明AMO酶可以通过共代谢作用,在催化氨氮的同时实现SAs的转化。AMO酶上的活性位点主要分为氨氮活性位点和非氨氮活性位点。一方面,其他碳源(如乙炔和乙烯) 可通过与氨氮竞争结合位点影响SAs的共代谢;另一方面,其他碳源通过与非氨氮活性位点结合影响酶活性,从而影响抗生素的生物转化效率[41, 66-68]。
此外,有研究表明,加入葡萄糖或其他易被微生物利用的碳源会提高SAs的去除效率,这是由于这些易利用的碳源促进了降解菌的生长。例如,在加入其他容易被利用的碳源后可以显著提高微杆菌属的菌株Microbacterium sp. BR1对SMX的降解效率。由于Microbacterium利用这些碳源生长后生物量增加,待易降解物质用完后其再继续利用SMX作为底物生长。因此,在污水处理过程中,为提高SAs的降解效率,可以考虑投加适量的其他碳源以促进降解菌的生长。
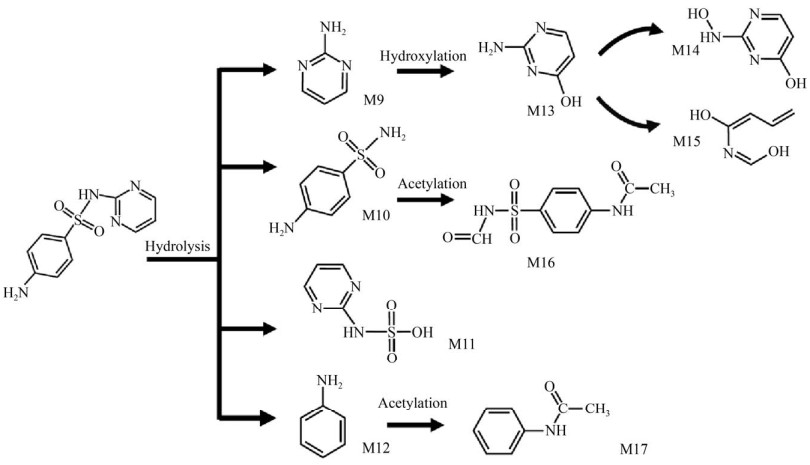
3 好氧活性污泥体系中磺胺类抗生素的生物降解途径SAs主要作为异养微生物的碳源和氮源被利用,或者在参与氨氧化菌的共代谢时被转化[69]。SAs的生物降解可能主要取决于磺酰基侧链部分的结构[25],主要的降解反应发生在苯胺或胺化杂芳侧基的羟基化、酰基化和磺胺基的水解裂解反应以及硝代反应[70],图 2为SMX在好氧活性污泥体系中的主要代谢途径。
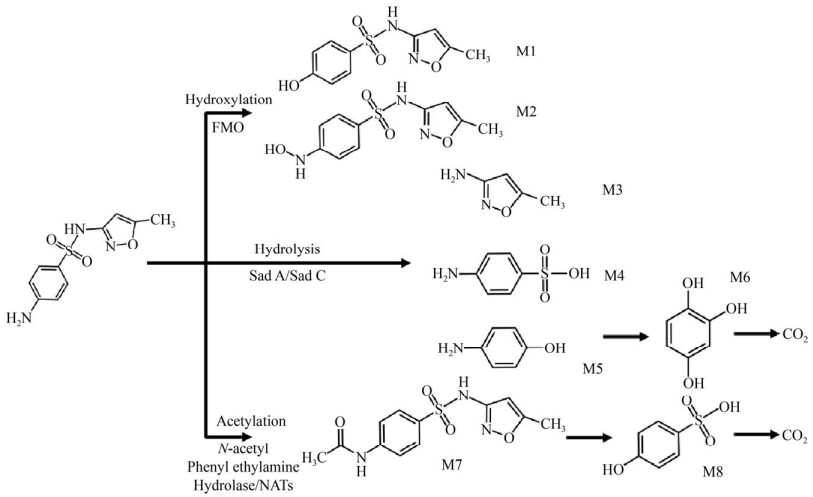
|
| 图 2 磺胺甲噁唑在好氧活性污泥中的降解途径[34, 40, 44, 50, 57, 61, 65, 71-72] Fig. 2 The typical biodegradation pathway of sulfamethoxazole in aerobic activated sludge[34, 40, 44, 50, 57, 61, 65, 71-72]. FMO: Flavin-containing monooxygenase; Sad A and Sad B: monooxygenases; Sad C: Flavin mononucleotide reductase; NATs: N-acetyltransferases. |
| |
当体系中有SAs存在时,便会激发降解菌中与羟基化反应相关酶的过量表达。有研究以SMX为底物培养Acinetobacter sp.、Rhodococcus rhodochrou和Microbacterium sp.时,SMX主要的代谢产物为羟基化反应生成的4-羟基甲噁唑(M1) 和N-羟基磺胺甲噁唑(M2),即芳香环上的氨基或氨基上的氢被羟基所取代。此外还发现了SMX经水解裂解反应产生的3-氨基-5-甲基异噁唑(M3)、4-氨基苯磺酸(M4) 和4-氨基苯酚(M5) 等代谢产物,这可能是由微生物体内的两种单加氧酶基因(sad A、sad C) 表达的酶促作用产生的;反应生产的4-氨基苯酚经Sad B和Sad C酶进一步转化为1, 2, 4-三羟基苯(M6) 而后矿化[34, 40, 44, 50, 71]。有研究表明从活性污泥中分离得到的Achromobacter denitrificans PR1对SMX的代谢和转化不稳定,但当其与菌株Leucobacter sp. GP共培养时,该菌群中含有sad A的同源基因,该基因过量表达可指导合成黄素单加氧酶(Flavin-containing monooxygenase,FMO) 进而催化SMX苯环上的羟基化取代反应[27]。除了羟基化反应和磺胺基的水解裂解反应以外,该类药物还可能发生乙酰化反应。Wang等的研究表明Acinetobacter sp.可通过体内的NATs或N-乙酰基苯乙胺水解酶等催化乙酰化反应转化SMX并产生N-乙酰磺胺甲噁唑(M7),M7进一步分解产生4-羟基苯磺酸(M8) 而后矿化[44, 57, 61, 65, 72]。
除SMX以外,SDZ也可以发生水解、羟基化和酰基化等反应。节杆菌Arthrobacter被报道是SDZ的降解菌,Reis等和Deng等[27, 30]从活性污泥中分离得到了菌株Arthrobacter sp. AK-YN10和Microbacterium sp. C448,并从它们的纯培养体系中检测到了SDZ代谢产物。根据SDZ代谢产物推测出了SDZ的生物转化途径(图 3):SDZ的磺胺键断裂产生2-氨基嘧啶(M9)、对氨基苯磺酰胺(M10)、嘧啶-2-氨基磺酸(M11) 和苯胺(M12)。然后,嘧啶环上发生羟基化反应产生2-氨基-4-羟基嘧啶(M13),该产物进一步发生羟基化和嘧啶环的水解反应;对氨基苯磺酰胺(M10) 和苯胺(M12) 进一步发生酰基化反应。但是目前的研究无法确定与SDZ生物转化相关的关键酶或基因,后续研究需要进一步关注该类基因或酶的定位[24, 28]。
此外,由于AAS中存在较多的AOB,AOB体内的amo基因指导合成AMO酶可直接和间接介导羟基化和硝代反应,产生羟基化和硝基化代谢产物。除了AMO酶,氨氧化菌如N. gargensis、N. nitrosa Nm90和N. inopinata基因组中的脱氨基酶基因可介导产生SAs的脱氨基反应[61]。图 4为AOB对SAs生物降解可能的代谢途径。
随着社会对环境保护和污染环境修复的日渐关注,关于SAs的生物转化过程及机理的研究已获得相当大的进展,例如降解菌的分离筛选、相关降解基因和酶的发现以及生物转化途径的阐明等,这些进展为人类生活环境中抗生素污染的去除提供了理论基础和应用依据。但是,由于目前研究的技术手段还存在一定的局限性,使得好氧活性污泥体系中的微生物协同降解机理和各类降解菌对SAs降解的贡献无法被准确评估,导致现有的体系无法最大化发挥降解菌的降解潜能,因此后续研究中可将以上问题作为研究重点和主要方向。
4.1 缺乏对原位环境中微生物组协同降解机理的研究目前的研究主要是通过传统纯培养的方法获得SAs的降解菌株,这为阐明降解菌对SAs的生物转化途径提供了有效手段。该方法通常通过向AAS中投加高浓度的抗生素并经历长期的驯化过程得到可降解SAs的富集液。然而,富集液中的微生物群落在驯化过程中发生了演替,这与真实环境条件中的原位微生物群落存在较大差异,故该方法无法反映原位真实环境条件,不能找到实际参与SAs降解和转化的微生物组;同时,该方法也无法体现出环境中不同种类的微生物在该类药物降解过程中的协同作用。因此,今后的研究可以考虑将标记技术(如SIP技术) 和组学技术(如高通量测序等) 应用到真实环境下微生物组的降解机制研究中,尽可能在还原真实环境条件的情况下,通过潜在降解菌生态网络的构建并结合同位素示踪溯源,寻找SAs的降解菌并阐明原位条件下SAs降解菌群的协同降解机理。
4.2 不同降解途径对磺胺类抗生素降解的贡献不明确氨氧化菌通过AMO酶介导的共代谢作用是实现SAs转化的重要途径。目前的研究中普遍使用ATU作为氨氧化菌的抑制剂,通过与氨氧化活性处理组的对比来研究这种共代谢作用在降解过程中的贡献。然而有研究表明,ATU在抑制氨氧化菌的AMO酶的同时可以抑制某些异养菌中酶的活性,这将直接导致体系中氨氧化菌的作用被高估。后续的研究可以尝试探索新型氨氧化菌活性抑制剂,以更准确地区分氨氧化菌和异养菌在SAs生物降解中的贡献。
此外,氨氧化菌的AMO酶在介导SAs的生物转化时产生的羟胺等物质可以进一步被转化成NO或HNO等,这些不同形态的含氮产物可以继续介导SAs的非生物转化。然而,目前的研究并没有对AMO酶介导的生物和非生物转化作出区分,这也会导致氨氧化菌在SAs生物转化中发挥的作用被高估。因此,今后的研究需要关注真实环境条件下不同形态的氮介导的非生物转化,从而区分生物和非生物转化途径的贡献。
4.3 无法最大化发挥体系中降解菌的降解潜能影响降解效率的环境因素主要包括pH和温度等,实际上这些环境因素通过改变与降解相关基因的表达进而影响某些酶的产生最终影响生物降解反应。污水处理厂无法完全满足降解菌的最适生长条件,故其无法发挥降解菌的最大潜能,例如,氨氧化菌在SAs的降解转化中发挥了重要作用,但是氨氧化菌对环境pH的要求较高,通常需要在偏碱性的条件下才能保持较高的氨氧化活性。若活性污泥体系无法满足氨氧化菌对pH的需求,也就无法充分发挥它的潜能。
此外,温度通过影响某些基因的表达和酶的活性影响物质的生物降解。一般来说,夏季中污水处理过程对SAs的去除效率要高于冬季,这主要由于酶催化反应受到了温度影响所导致的,故污水处理厂应在冬季采取相应的升温和保温措施,满足活性污泥对温度的需求。除了pH和温度外,污水处理过程中可考虑适量投加一些易被利用的碳源,充分利用异养菌的共代谢作用最终实现SAs的有效去除。除了投加额外碳源外,也可从改变活性污泥微生物组的角度入手,通过人为投加高效微生物菌剂充分发挥微生物组的协同作用进而提高去除效率。
4.4 新型磺胺类抗生素应引起关注在目前的研究中,SMX、SDZ等典型SAs在好氧活性污泥体系中的生物降解已经被密切关注。但是随着SAs种类的增多和广泛使用,除了典型SAs以外的其他药物逐渐被释放到水环境中,这会对人类健康及环境造成不利影响。目前,对于这类SAs的研究较少,故该类药物在污水处理过程中的生物降解机理(如环境归趋、原位降解菌的鉴定筛选、微生物组协同作用机制) 同样需要加以关注。
| [1] |
Brackett CC, Singh H, Block JH. Likelihood and mechanisms of cross-allergenicity between sulfonamide antibiotics and other drugs containing a sulfonamide functional group. Pharmacotherapy, 2012, 24(7): 856-870.
|
| [2] |
Masters PA, O'Bryan TA, Zurlo J, et al. Trimethoprim-sulfamethoxazole revisited. Arch Intern Med, 2003, 163(4): 402. DOI:10.1001/archinte.163.4.402
|
| [3] |
Xu ML, Li HL, Li SW, et al. The presence of tetracyclines and sulfonamides in swine feeds and feces: dependence on the antibiotic type and swine growth stages. Environ Sci Pollut Res, 2020, 27(34): 43093-43102. DOI:10.1007/s11356-020-10266-5
|
| [4] |
Tappe W, Herbst M, Hofmann D, et al. Degradation of sulfadiazine by Microbacterium lacus strain SDZm4, isolated from lysimeters previously manured with slurry from sulfadiazine-medicated pigs. Appl Environ Microbiol, 2013, 79(8): 2572-2577. DOI:10.1128/AEM.03636-12
|
| [5] |
Shanableh A, Bhattacharjee S, Alani S, et al. Assessment of sulfamethoxazole removal by nanoscale zerovalent iron. Sci Total Environ, 2021, 761: 143307. DOI:10.1016/j.scitotenv.2020.143307
|
| [6] |
Wu MH, Que CJ, Xu G, et al. Occurrence, fate and interrelation of selected antibiotics in sewage treatment plants and their receiving surface water. Ecotoxicol Environ Saf, 2016, 132: 132-139. DOI:10.1016/j.ecoenv.2016.06.006
|
| [7] |
Torresi E, Escolà Casas M, Polesel F, et al. Impact of external carbon dose on the removal of micropollutants using methanol and ethanol in post-denitrifying moving bed biofilm reactors. Water Res, 2017, 108: 95-105. DOI:10.1016/j.watres.2016.10.068
|
| [8] |
Chow LKM, Ghaly TM, Gillings MR. A survey of sub-inhibitory concentrations of antibiotics in the environment. J Environ Sci, 2021, 99: 21-27. DOI:10.1016/j.jes.2020.05.030
|
| [9] |
Lyu J, Yang L, Zhang L, et al. Antibiotics in soil and water in China-a systematic review and source analysis. Environ Pollut, 2020, 266(pt 1): 115147.
|
| [10] |
Zhang QQ, Ying GG, Pan CG, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance. Environ Sci Technol, 2015, 49(11): 6772-6782. DOI:10.1021/acs.est.5b00729
|
| [11] |
Guo X, Pang W, Dou C, et al. Sulfamethoxazole and COD increase abundance of sulfonamide resistance genes and change bacterial community structures within sequencing batch reactors. Chemosphere, 2017, 175: 21-27. DOI:10.1016/j.chemosphere.2017.01.134
|
| [12] |
Pan M, Chu LM. Occurrence of antibiotics and antibiotic resistance genes in soils from wastewater irrigation areas in the Pearl River Delta region, Southern China. Sci Total Environ, 2018, 624: 145-152. DOI:10.1016/j.scitotenv.2017.12.008
|
| [13] |
Proia L, Anzil A, Subirats J, et al. Antibiotic resistance along an urban river impacted by treated wastewaters. Sci Total Environ, 2018, 628/629: 453-466. DOI:10.1016/j.scitotenv.2018.02.083
|
| [14] |
Wang J, Wang S. Removal of pharmaceuticals and personal care products (PPCPs) from wastewater: a review. J Environ Manage, 2016, 182: 620-640. DOI:10.1016/j.jenvman.2016.07.049
|
| [15] |
Liu Y, Fan Q, Wang J. Zn-Fe-CNTs catalytic in situ generation of H2O2 for Fenton-like degradation of sulfamethoxazole. J Hazard Mater, 2018, 342: 166-176. DOI:10.1016/j.jhazmat.2017.08.016
|
| [16] |
Sui Q, Huang J, Lu SG, et al. Removal of pharmaceutical and personal care products by sequential ultraviolet and ozonation process in a full-scale wastewater treatment plant. Front Environ Sci Eng, 2014, 8(1): 62-68. DOI:10.1007/s11783-013-0518-z
|
| [17] |
Wang J, Wang S. Microbial degradation of sulfamethoxazole in the environment. Appl Microbiol Biotechnol, 2018, 102(8): 3573-3582. DOI:10.1007/s00253-018-8845-4
|
| [18] |
Wang S, Wang J. Degradation of emerging contaminants by acclimated activated sludge. Environ Technol, 2018, 39(15): 1985-1993. DOI:10.1080/09593330.2017.1345989
|
| [19] |
Pérez S, Eichhorn P, Aga DS. Evaluating the biodegradability of sulfamethazine, sulfamethoxazole, sulfathiazole, and trimethoprim at different stages of sewage treatment. Environ Toxicol Chem, 2005, 24(6): 1361-1367. DOI:10.1897/04-211R.1
|
| [20] |
Ingerslev F, Halling-Sorensen B. Biodegradability properties of sulfonamides in activated sludge. Environmental Toxicology and Chemistry, 2000, 19: 2467-2473. DOI:10.1002/etc.5620191011
|
| [21] |
Larcher S, Yargeau V. Biodegradation of sulfamethoxazole by individual and mixed bacteria. Appl Microbiol Biotechnol, 2011, 91(1): 211-218. DOI:10.1007/s00253-011-3257-8
|
| [22] |
Kor-Bicakci G, Pala-Ozkok I, Rehman A, et al. Chronic impact of sulfamethoxazole on acetate utilization kinetics and population dynamics of fast growing microbial culture. Bioresour Technol, 2014, 166: 219-228. DOI:10.1016/j.biortech.2014.05.042
|
| [23] |
Herzog B, Lemmer H, Horn H, et al. Characterization of pure cultures isolated from sulfamethoxazole-acclimated activated sludge with respect to taxonomic identification and sulfamethoxazole biodegradation potential. BMC Microbiol, 2013, 13: 276. DOI:10.1186/1471-2180-13-276
|
| [24] |
Liang DH, Hu Y. Simultaneous sulfamethoxazole biodegradation and nitrogen conversion by Achromobacter sp. JL9 using with different carbon and nitrogen sources. Bioresour Technol, 2019, 293(122061).
|
| [25] |
Reis PJ, Reis AC, Ricken B, et al. Biodegradation of sulfamethoxazole and other sulfonamides by Achromobacter denitrificans PR1. J Hazard Mater, 2014, 280: 741-749. DOI:10.1016/j.jhazmat.2014.08.039
|
| [26] |
Deng Y, Mao YP, Li B, et al. Aerobic degradation of sulfadiazine by Arthrobacter spp.: kinetics, pathways, and genomic characterization. Environ Sci Technol, 2016, 50(17): 9566-9575. DOI:10.1021/acs.est.6b02231
|
| [27] |
Reis AC, Reis AC, Čvančarová M, et al. Biodegradation of sulfamethoxazole by a bacterial consortium of Achromobacter denitrificans PR1 and Leucobacter sp. GP. Appl Microbiol Biotechnol, 2018, 102(23): 10299-10314. DOI:10.1007/s00253-018-9411-9
|
| [28] |
Bouju H, Ricken B, Beffa T, et al. Isolation of bacterial strains capable of sulfamethoxazole mineralization from an acclimated membrane bioreactor. Appl Environ Microbiol, 2012, 78(1): 277-279. DOI:10.1128/AEM.05888-11
|
| [29] |
Huang MH, Tian SX, Chen DH, et al. Removal of sulfamethazine antibiotics by aerobic sludge and an isolated Achromobacter sp. S-3. J Environ Sci, 2012, 24(9): 1594-1599. DOI:10.1016/S1001-0742(11)60973-X
|
| [30] |
Deng Y, Wang Y, Mao Y, et al. Partnership of Arthrobacter and Pimelobacter in aerobic degradation of sulfadiazine revealed by metagenomics analysis and isolation. Environ Sci Technol, 2018, 52(5): 2963-2972. DOI:10.1021/acs.est.7b05913
|
| [31] |
Wang S, Hu Y, Wang J. Biodegradation of typical pharmaceutical compounds by a novel strain Acinetobacter sp.. J Environ Manage, 2018, 217(240): 240-246.
|
| [32] |
Li N, Liu Q, Zhou GQ, et al. Contaminant removal and microorganism response of activated sludge in sulfamethazine wastewater treatment. Int Biodeterior Biodegrad, 2019, 143: 104705. DOI:10.1016/j.ibiod.2019.05.022
|
| [33] |
Ricken B, Corvini PF, Cichocka D, et al. Ipso-hydroxylation and subsequent fragmentation: a novel microbial strategy to eliminate sulfonamide antibiotics. Appl Environ Microbiol, 2013, 79(18): 5550-5558. DOI:10.1128/AEM.00911-13
|
| [34] |
Ricken B, Kolvenbach BA, Bergesch C, et al. MNH2-dependent monooxygenases initiate catabolism of sulfonamides in Microbacterium sp. strain BR1 subsisting on sulfonamide antibiotics. Sci Rep, 2017, 7(1): 15783. DOI:10.1038/s41598-017-16132-8
|
| [35] |
Song MK, Luo CL, Jiang LF, et al. The presence of in situ sulphamethoxazole degraders and their interactions with other microbes in activated sludge as revealed by DNA stable isotope probing and molecular ecological network analysis. Environ Int, 2019, 124: 121-129. DOI:10.1016/j.envint.2018.12.039
|
| [36] |
Alvarino T, Lema J, Omil F, et al. Trends in organic micropollutants removal in secondary treatment of sewage. Rev Environ Sci Biotechnol, 2018, 17(3): 447-469. DOI:10.1007/s11157-018-9472-3
|
| [37] |
Gonzalez-Gil L, Fernandez-Fontaina E, Singh RR, et al. Feeding composition and sludge retention time both affect co-metabolic biotransformation of pharmaceutical compounds in activated sludge systems. J Environ Chem Eng, 2021, 9(2): 105123. DOI:10.1016/j.jece.2021.105123
|
| [38] |
Song HL, Lu YX, Yang XL, et al. Degradation of sulfamethoxazole in low-C/N ratio wastewater by a novel membrane bioelectrochemical reactor. Bioresour Technol, 2020, 305: 123029. DOI:10.1016/j.biortech.2020.123029
|
| [39] |
Nguyen PY, Silva AF, Reis AC, et al. Bioaugmentation of membrane bioreactor with Achromobacter denitrificans strain PR1 for enhanced sulfamethoxazole removal in wastewater. Sci Total Environ, 2019, 648: 44-55. DOI:10.1016/j.scitotenv.2018.08.100
|
| [40] |
Gauthier H, Yargeau V, Cooper DG. Biodegradation of pharmaceuticals by Rhodococcus rhodochrous and Aspergillus niger by co-metabolism. Sci Total Environ, 2010, 408(7): 1701-1706. DOI:10.1016/j.scitotenv.2009.12.012
|
| [41] |
Su QX, Schittich AR, Jensen MM, et al. Role of ammonia oxidation in organic micropollutant transformation during wastewater treatment: insights from molecular, cellular, and community level observations. Environ Sci Technol, 2021, 55(4): 2173-2188. DOI:10.1021/acs.est.0c06466
|
| [42] |
Yuan XJ, Qiang ZM, Ben WW, et al. Distribution, mass load and environmental impact of multiple-class pharmaceuticals in conventional and upgraded municipal wastewater treatment plants in East China. Environ Sci Process Impacts, 2015, 17(3): 596-605. DOI:10.1039/C4EM00596A
|
| [43] |
Zheng WL, Wen XH, Zhang B, et al. Selective effect and elimination of antibiotics in membrane bioreactor of urban wastewater treatment plant. Sci Total Environ, 2019, 646: 1293-1303. DOI:10.1016/j.scitotenv.2018.07.400
|
| [44] |
Wang SZ, Wang JL. Biodegradation and metabolic pathway of sulfamethoxazole by a novel strain Acinetobacter sp.. Appl Microbiol Biotechnol, 2018, 102(1): 425-432. DOI:10.1007/s00253-017-8562-4
|
| [45] |
Wei Z, Li W, Zhao D, et al. Electrophilicity index as a critical indicator for the biodegradation of the pharmaceuticals in aerobic activated sludge processes. Water Res, 2019, 160: 10-17. DOI:10.1016/j.watres.2019.05.057
|
| [46] |
Min XB, Li W, Wei ZS, et al. Sorption and biodegradation of pharmaceuticals in aerobic activated sludge system: a combined experimental and theoretical mechanistic study. Chem Eng J, 2018, 342: 211-219. DOI:10.1016/j.cej.2018.01.012
|
| [47] |
Yang SF, Lin CF, Lin AY, et al. Sorption and biodegradation of sulfonamide antibiotics by activated sludge: experimental assessment using batch data obtained under aerobic conditions. Water Res, 2011, 45(11): 3389-3397. DOI:10.1016/j.watres.2011.03.052
|
| [48] |
Tobajas M, Verdugo V, Polo AM, et al. Assessment of toxicity and biodegradability on activated sludge of priority and emerging pollutants. Environ Technol, 2016, 37(6): 713-721. DOI:10.1080/09593330.2015.1079264
|
| [49] |
Drillia P, Dokianakis SN, Fountoulakis MS, et al. On the occasional biodegradation of pharmaceuticals in the activated sludge process: the example of the antibiotic sulfamethoxazole. J Hazard Mater, 2005, 122(3): 259-265. DOI:10.1016/j.jhazmat.2005.03.009
|
| [50] |
Müller E, Schüssler W, Horn H, et al. Aerobic biodegradation of the sulfonamide antibiotic sulfamethoxazole by activated sludge applied as co-substrate and sole carbon and nitrogen source. Chemosphere, 2013, 92(8): 969-978. DOI:10.1016/j.chemosphere.2013.02.070
|
| [51] |
Krah D, Ghattas AK, Wick A, et al. Micropollutant degradation via extracted native enzymes from activated sludge. Water Res, 2016, 95: 348-360. DOI:10.1016/j.watres.2016.03.037
|
| [52] |
Yu L, Wang Y, Su X, et al. Biodiversity, isolation and genome analysis of sulfamethazine-degrading bacteria using high-throughput analysis. Bioprocess Biosyst Eng, 2020, 43(8): 1521-1531. DOI:10.1007/s00449-020-02345-1
|
| [53] |
Peng J, Wang X, Yin F, et al. Characterizing the removal routes of seven pharmaceuticals in the activated sludge process. Sci Total Environ, 2019, 650(pt 2): 2437-2445.
|
| [54] |
Yang N, Wan JF, Zhao SJ, et al. Removal of concentrated sulfamethazine by acclimatized aerobic sludge and possible metabolic products. PeerJ, 2015, 3: e1359. DOI:10.7717/peerj.1359
|
| [55] |
Men Y, Achermann S, Helbling DE, et al. Relative contribution of ammonia oxidizing bacteria and other members of nitrifying activated sludge communities to micropollutant biotransformation. Water Res, 2017, 109: 217-226. DOI:10.1016/j.watres.2016.11.048
|
| [56] |
Wang B, Ni BJ, Yuan Z, et al. Insight into the nitrification kinetics and microbial response of an enriched nitrifying sludge in the biodegradation of sulfadiazine. Environ Pollut, 2019, 255(pt 1): 113160.
|
| [57] |
Kassotaki E, Buttiglieri G, Ferrando-Climent L, et al. Enhanced sulfamethoxazole degradation through ammonia oxidizing bacteria co-metabolism and fate of transformation products. Water Res, 2016, 94: 111-119. DOI:10.1016/j.watres.2016.02.022
|
| [58] |
Wang BZ, Li HY, Liu T, et al. Enhanced removal of cephalexin and sulfadiazine in nitrifying membrane-aerated biofilm reactors. Chemosphere, 2021, 263: 128224. DOI:10.1016/j.chemosphere.2020.128224
|
| [59] |
Katipoglu-Yazan T, Ubay-Cokgor E, Orhon D. Chronic impact of sulfamethoxazole: how does process kinetics relate to metabolic activity and composition of enriched nitrifying microbial culture?. J Chem Technol Biotechnol, 2018, 93: 1722-1732. DOI:10.1002/jctb.5545
|
| [60] |
Wang BZ, Ni BJ, Yuan ZG, et al. Unravelling kinetic and microbial responses of enriched nitrifying sludge under long-term exposure of cephalexin and sulfadiazine. Water Res, 2020, 173: 115592. DOI:10.1016/j.watres.2020.115592
|
| [61] |
Zhou LJ, Han P, Yu Y, et al. Cometabolic biotransformation and microbial-mediated abiotic transformation of sulfonamides by three ammonia oxidizers. Water Res, 2019, 159: 444-453. DOI:10.1016/j.watres.2019.05.031
|
| [62] |
Wu Y, Ke X, Hernández M, et al. Autotrophic growth of bacterial and archaeal ammonia oxidizers in freshwater sediment microcosms incubated at different temperatures. Appl Environ Microbiol, 2013, 79(9): 3076-3084. DOI:10.1128/AEM.00061-13
|
| [63] |
Qi CD, Liu XT, Lin CY, et al. Degradation of sulfamethoxazole by microwave-activated persulfate: kinetics, mechanism and acute toxicity. Chem Eng J, 2014, 249: 6-14. DOI:10.1016/j.cej.2014.03.086
|
| [64] |
Maspolim Y, Zhou Y, Guo C, et al. The effect of pH on solubilization of organic matter and microbial community structures in sludge fermentation. Bioresour Technol, 2015, 190: 289-298. DOI:10.1016/j.biortech.2015.04.087
|
| [65] |
Jiang BC, Li A, Cui D, et al. Biodegradation and metabolic pathway of sulfamethoxazole by Pseudomonas psychrophila HA-4, a newly isolated cold-adapted sulfamethoxazole-degrading bacterium. Appl Microbiol Biotechnol, 2014, 98(10): 4671-4681. DOI:10.1007/s00253-013-5488-3
|
| [66] |
Keener WK, Arp DJ. Kinetic studies of ammonia monooxygenase inhibition in Nitrosomonas europaea by hydrocarbons and halogenated hydrocarbons in an optimized whole-cell assay. Appl Environ Microbiol, 1993, 59(8): 2501-2510. DOI:10.1128/aem.59.8.2501-2510.1993
|
| [67] |
Wright CL, Schatteman A, Crombie AT, et al. Inhibition of ammonia monooxygenase from ammonia-oxidizing Archaea by linear and aromatic alkynes. Appl Environ Microbiol, 2020, 86(9): e02388-19.
|
| [68] |
Alvarez-Cohen L, Speitel GE. Kinetics of aerobic cometabolism of chlorinated solvents. Biodegradation, 2001, 12(2): 105-126. DOI:10.1023/A:1012075322466
|
| [69] |
Çeçen F, Gül G. Biodegradation of five pharmaceuticals: estimation by predictive models and comparison with activated sludge data. Int J Environ Sci Technol, 2021, 18(2): 327-340. DOI:10.1007/s13762-020-02820-y
|
| [70] |
Chen J, Xie S. Overview of sulfonamide biodegradation and the relevant pathways and microorganisms. Sci Total Environ, 2018, 640/641: 1465-1477. DOI:10.1016/j.scitotenv.2018.06.016
|
| [71] |
Fischer K, Majewsky M. Cometabolic degradation of organic wastewater micropollutants by activated sludge and sludge-inherent microorganisms. Appl Microbiol Biotechnol, 2014, 98(15): 6583-6597. DOI:10.1007/s00253-014-5826-0
|
| [72] |
Caranto JD, Lancaster KM. Nitric oxide is an obligate bacterial nitrification intermediate produced by hydroxylamine oxidoreductase. Proc Natl Acad Sci USA, 2017, 114(31): 8217-8222. DOI:10.1073/pnas.1704504114
|
 2021, Vol. 37
2021, Vol. 37