中国科学院微生物研究所、中国微生物学会主办
文章信息
- 史可, 郭晨蕾, 马晓丹, 梁斌, 王爱杰
- Shi Ke, Guo Chenlei, Ma Xiaodan, Liang Bin, Wang Aijie
- 一株氯霉素降解细菌的分离鉴定与代谢特性研究
- Isolation, identification and characterization of a chloramphenicol-degrading bacterium
- 生物工程学报, 2021, 37(10): 3653-3662
- Chinese Journal of Biotechnology, 2021, 37(10): 3653-3662
- 10.13345/j.cjb.210434
-
文章历史
- Received: June 7, 2021
- Accepted: August 30, 2021
- Published: September 2, 2021
2. 中国科学院生态环境研究中心,中国科学院环境生物技术重点实验室,北京 100085
2. Key Laboratory of Environmental Biotechnology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China
据报道全世界228个国家和地区2010年兽用抗生素的使用量约为63 151 t,预计2030年用量将增加67%,尤其是我国、美国、印度、巴西和德国等国用量较大[1]。未有效利用的抗生素主要汇入污水处理厂,而污水处理厂通常旨在去除常规污染物,难以有效地去除抗生素类难降解污染物和耐药细菌[2-4]。令人担忧的是,我国污水处理厂每年排放约53 800 t抗生素进入环境生态系统,污染极其严重[5]。氯霉素(Chloramphenicol,CAP) 自1949年以来被全世界广泛应用于人类疾病治疗和畜禽养殖产业发展,属于广谱抗生素[6-9]。尽管其对人类具有明显的致再生障碍性贫血和潜在的致癌性、遗传毒性[10-11],而且已被许多国家(包括美国、中国、澳大利亚等) 禁止使用[12],但是目前在不同国家和地区,特别是在中国,市政、医院、畜禽和水产养殖废水以及地表水中都普遍检测出CAP残留(ng–µg/L水平)[13]。此外,城市污泥、畜禽粪便、土壤和河流沉积物中的CAP残留浓度可能高达mg/kg水平,这将促进环境微生物的耐药性进化与传播[14],对人类与环境健康产生潜在威胁。因此,在传统的污(废) 水生物处理过程中如何深度消减抗生素残留和阻控抗生素抗性基因传播风险至关重要。
生物降解法具有绿色、运行成本低、环境友好等优势[15]。CAP乙酰化是导致其失活的重要细菌耐药机制[16],硝基还原是厌氧微生物脱毒转化CAP的主要代谢过程[17],然而生成的芳香胺物质在厌氧环境中难以进一步降解。一些好氧菌株如克雷伯氏菌Klebsiella sp.和埃希氏菌Escherichia sp.已经被证明能够通过水解CAP侧链酰胺键将其转化为对硝基苯基丝氨醇[18](p-nitrophenylserinol,1-对硝基苯-2-氨基-1, 3-丙二醇),但其仍无法对形成的产物进一步降解。最近报道了一株从污水处理厂活性污泥中分离获得的鞘脂菌Sphingobium sp. CAP‑1,首先将CAP氧化脱毒为对硝基苯甲醛(p-nitrobenzaldehyde,p-NBZ) 或氧化CAP产物(CAP的侧链C3位羟基氧合产物,O-CAP),接着两者均被依次降解为对硝基苯甲酸(p-nitrobenzoic acid,p-NBA) 和原儿茶酸(Protocatechuic acid,PCA),然后进入三羧酸循环实现CAP的完全分解代谢[19]。此外,从活性污泥中分离获得的CAP降解菌鞘氨醇单胞菌Sphingomonas sp. CL5.1,在CAP降解过程中除了检测到乙酰化CAP产物,也检测到菌株CAP-1研究中发现的O-CAP和p-NBA[20]。综上所述,亟待发掘新的CAP高效降解菌株资源,并解析其降解特性与代谢路径,丰富细菌代谢抗生素的基础理论。
基于CAP在环境中的普遍存在性和生态风险控制的必要性,本研究从长期受抗生素毒性选择的活性污泥中分离培养了一株CAP高效降解细菌,研究了该菌株的降解特性,解析了其降解途径。这些研究结果将为CAP在实际环境中(如污水处理厂活性污泥体系和畜禽/水产养殖/制药园区环境等) 由微生物介导的归趋与转化过程提供新见解,为CAP污染典型环境(尤其是制药和畜禽养殖废水) 的微生物强化处理提供高效降解菌株资源。
1 材料与方法 1.1 实验材料 1.1.1 实验药品与试剂实验所用CAP (纯度99%)、对硝基苯甲酸p-NBA (纯度99%)、对硝基苯甲醛p-NBZ (纯度97%) 和原儿茶酸PCA (纯度97%) 均购自上海阿拉丁生化科技股份有限公司。其余使用的药品均为分析纯及以上。
1.1.2 实验所用培养基Luria-Bertani (LB) 培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠10,调节pH为7.0。
无机盐培养基(MSM) (g/L):K2HPO4 1.50,KH2PO4 0.50,Mg2SO4·7H2O 0.20,NaCl 1.00,调节pH为7.0。
固体培养基:分别在以上培养基的基础上加入2% (W/V) 琼脂。
以上培养基使用前均在121 ℃下高温灭菌30 min后冷却备用。
1.2 实验方法 1.2.1 CAP降解菌的富集与分离以某制药厂二沉池污泥作为分离CAP降解菌的接种源,取5 g污泥接入含100 mL MSM的250 mL锥形瓶中(CAP浓度为50 mg/L)。将锥形瓶放置于恒温振荡培养箱,在30 ℃、150 r/min条件下进行富集培养,每隔7 d从富集液中以5% (V/V) 的接种量转接至新的含CAP无机盐培养基中。连续转接3次后,通过超高效液相色谱测试CAP的浓度,确定富集液是否具有CAP降解效果。将有降解活性的富集液进行梯度稀释,采用平板划线法纯化培养。将分离得到的CAP降解菌株与已灭菌的50%甘油按1︰1混合,−80 ℃保存。
1.2.2 CAP降解菌形态特征鉴定将CAP降解菌接种于含50 mg/L CAP的无机盐固体培养基上,30 ℃恒温培养5 d后观察菌落形态。挑取平板上的单菌于含有4 mL LB液体培养基的试管中,置于30 ℃、150 r/min的恒温振荡培养箱中培养至对数期,将分散后的菌体用磷钨酸染色再通过透射扫描电子显微镜(HT7650,HITACHI,Japan) 观察菌体显微结构。
1.2.3 16S rRNA基因序列测定及系统发育分析使用DNA提取试剂盒(DP302,Tiangen,Beijing) 提取CAP降解菌基因组总DNA,采用细菌通用引物27F/1492R进行扩增,PCR产物经电泳检验与TA克隆后送至生工生物工程(上海) 股份有限公司测序。将测序结果在NCBI和EzBioCloud数据库中进行比对,通过MEGA7.0构建系统发育树,该降解菌株16S rRNA序列的GenBank登录号为MZ314676。
1.2.4 CAP降解菌的代谢特性研究降解菌在LB液体培养基中活化至对数生长期,离心弃上清,用灭菌后的MSM洗涤3次,之后用等体积MSM重悬菌体,4 ℃保存备用。分别以葡萄糖和硝酸铵作为外加碳源和氮源,在MSM的基础上配置4种不同条件的培养基:CAP分别作为唯一碳氮源(记为C–N–)、唯一碳源(外加硝酸铵,记为C–N+)、唯一氮源(外加葡萄糖,记为C+N–) 和非唯一碳氮源(同时添加葡萄糖和硝酸铵,记为C+N+),探究降解菌株在不同营养条件下对CAP的降解效能。此外分别以50 mg/L p-NBZ、p-NBA、PCA为唯一碳源添加到MSM,探究降解菌株CAP-2的代谢途径。以上实验每组设置3个重复。接入一定体积的降解菌悬液使其终浓度为OD600=0.003,30 ℃、150 r/min恒温振荡培养,每隔一定时间取样1 mL经0.22 μm滤膜过滤后−20 ℃保存待测。
CAP及其中间产物浓度通过超高效液相色谱检测(Agilent 1290A,Agilent Co.,USA),色谱柱为C18柱(1.7 μm,2.1 mm×100 mm,186002854 ACQUITY BEH,Waters Co.,USA),进样量为5 μL,流动相为甲醇和0.1%的甲酸水(V︰V=25︰75),流速为0.2 mL/min,检测波长为275 nm,柱温为30 ℃,根据样品出峰时间鉴定为不同物质,使用峰面积定量物质浓度。总有机碳(TOC) 浓度通过TOC分析仪测定(N5-665-N,Jena,Germany)。
2 结果与分析 2.1 CAP降解富集液性能与微生物群落组成分析以受抗生素毒性选择的活性污泥作为初始接种源,CAP作为唯一碳氮源,经过20 d的驯化富集后,获得了一瓶CAP降解富集液,并通过连续传代培养提高了富集液对CAP的降解效能和矿化程度。对第5代富集液(5th) 的降解性能进行测试(图 1A),初始CAP浓度为50 mg/L,24 h后CAP降解率为36.29%±2.72%,且48 h内完全降解,TOC去除率为29.71%±1.55%。对于第35代富集液(35th),24 h后CAP降解率为69.62%±6.18%,48 h后TOC去除率达到76.57%±1.08%。

|
| 图 1 富集液降解CAP效率(A) 与微生物群落组成分析(B) Fig. 1 The analysis of CAP biodegradation and mineralization efficiencies using enrichment (A) and the corresponding microbial community composition (B). |
| |
将第5代和第35代CAP降解富集液进行基于16S rRNA基因的高通量测序(引物为338F/806R),解析其中微生物群落组成(图 1B)。第5代富集液中主要菌属包括丛毛单胞菌属Comamonas (相对丰度54.18%)、红球菌属Rhodococcus (相对丰度6.8%)、贪铜菌属Cupriavidus (相对丰度5.01%)。已有报道表明,丛毛单胞菌属对多种芳烃如对硝基苯甲酸和硝基苯等有矿化能力[21-24];贪铜菌属可代谢多种(氯代) 芳烃,如硝基酚、氯酚和卤代苯甲酸盐等[25];红球菌属可降解持久性有机污染物,如多氯联苯等;此外由于4, 5-双加氧酶的作用,Rhodopseudomonas sp.可以在好氧条件下,以3, 4-二羟基苯甲酸和对羟基苯甲酸为碳源进行生长[26]。第35代富集液中相对丰度最高的菌属为红球菌属(相对丰度71.41%),其次是Cupriavidus、Castellaniella和Hyphomicobium,相对丰度分别为9.54%、4.24%和1.60%。经过长期驯化富集,CAP降解菌群中红球菌属的相对丰度明显增加,表明该菌属在CAP降解过程中发挥关键作用。
2.2 CAP降解细菌的分离与鉴定对已获得的CAP降解富集液通过梯度稀释与平板划线的方法,分离得到一株CAP降解细菌,菌命名为CAP-2。其菌落呈浅褐色,不透明,形态呈规整圆形,表面干涩,中部微微凸起(图 2A)。经透射电镜观察,该菌株无芽孢、鞭毛及胞外聚合物,呈细杆状(图 2B)。

|
| 图 2 氯霉素降解细菌形态(A) 和透射电镜图(B) Fig. 2 Morphology observation (A) and transmission electron micrograph (B) of strain CAP-2. |
| |
对该菌株提取基因组DNA,经16S rRNA基因测序,在NCBI和EzBioCloud数据库中进行在线比对分析,发现该菌株的16S rRNA基因序列与模式菌株Rhodococcus erythropolis NBRC 15567T(BCRM01000055)、Rhodococcus qingshengii JCM 15477T (LRRJ01000016) 和Rhodococcus baikonurensis GTC1041T (AB071951) 的相似性分别达到了99.93%、99.31%和99.04%,并结合菌株形态特征,将降解菌株CAP-2鉴定为Rhodococcus sp.,应用MEGA 7.0软件将该菌株的16S rRNA基因序列与其他高度同源的模式红球菌属菌株构建系统发育树,如图 3所示。

|
| 图 3 菌株CAP-2基于16S rRNA基因序列同源性的系统发育树分析 Fig. 3 Phylogenetic analysis of strain CAP-2 and related Rhodococcus type strains by neighbor joining approach. Bootstrap values obtained with 1 000 resamplings are indicated as percentages at all branches. The scale bars represent 0.005 substitutions per nucleotide position. |
| |
通过设置不同的营养条件,探究菌株CAP-2对CAP的降解效能(图 4)。当CAP作为唯一碳氮源时(图 4A),24 h后CAP降解率为50.77%±2.27%,42 h后为99.61%±0.03%,代谢产物p-NBA在30 h后达到最大生成浓度(10.90±0.08) mg/L,120 h后其降解至(4.48±0.07) mg/L。外加碳源加快了CAP的降解速率(图 4B),24 h后降解率达到100%,同时中间产物p-NBA在42 h后降解至(1.25±0.008) mg/L。
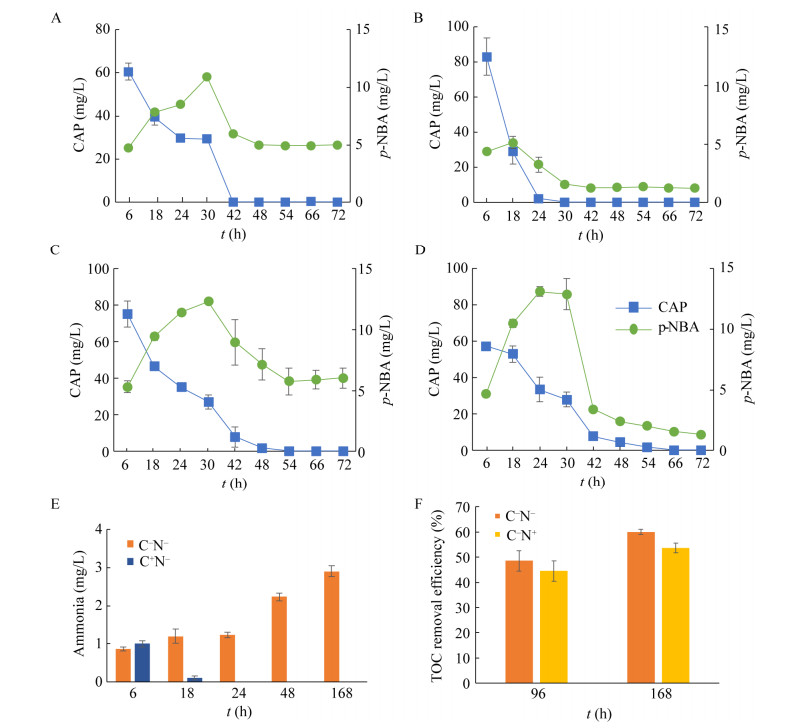
|
| 图 4 不同营养条件下菌株CAP-2降解CAP的特征(A:CAP作为唯一碳氮源;B:CAP作为唯一氮源;C:CAP作为唯一碳源;D:非唯一碳氮源;E:不同营养条件下CAP降解过程中氨氮释放;F:TOC去除特征) Fig. 4 The CAP degradation characteristics of strain CAP-2 under C–N– (A), C+N– (B), C–N+ (C) and C+N+ (D) four nutrient conditions. The characteristics of NH4+-N release (E) and TOC removal efficiencies during CAP degradation under different nutrient conditions (F). C–: free of carbon source. N–: free of nitrogen source. C+: with carbon source. N+: with nitrogen source. |
| |
外加氮源对CAP降解速率影响较小(图 4C),24 h后降解率为53.11%±2.75%,42 h后降解率达到89.41%±7.16%,p-NBA在30 h后达到最大累积浓度,120 h后降解至(5.75±0.67) mg/L;同时外加碳氮源,CAP降解速率没有增加(图 4D),24 h后降解率为41.34%±11.56%,42 h后为86.29%±0.11%,p-NBA在30 h后生成浓度达到最大值,120 h后降解至(1.15±0.01) mg/L。
CAP作为唯一碳源时,NH4+释放浓度随着CAP的降解逐渐升高(图 4E),168 h后达到(2.90±0.15) mg/L,表明菌株CAP-2能够以CAP作为氮源生长;外加碳源时,NH4+在6 h后的生成量为(1.00±0.08) mg/L,之后在较短时间(24 h) 内被消耗,表明碳源引入提高了菌株CAP-2对CAP的降解效率。通过测试TOC含量评估菌株CAP-2对CAP的矿化程度(图 4F),在CAP作为唯一碳氮源时,96 h后的TOC去除率为48.55%±4.43%,168 h后为60.02%±1.22%;外加氮源时,TOC在168 h后的去除率为53.70%±2.44%,表明外加氮源对CAP矿化程度影响较小。
综上所述,外加碳源(葡萄糖) 提高了菌株CAP-2对CAP与代谢产物p-NBA的降解效率,外加氮源(硝酸铵) 对CAP降解效能影响较小。
2.3.2 菌株CAP-2降解CAP途径解析菌株CAP-2在不同营养条件下降解CAP均伴随有p-NBA生成,并且该产物能够被进一步降解。根据目前已有文献报道,CAP好氧生物降解主要代谢产物包括p-NBZ、p-NBA和PCA[19],分别以上述中间产物作为底物,对菌株CAP-2降解CAP的途径进行初步解析。
菌株CAP-2降解p-NBZ速率较快(图 5A),24 h内90%以上p-NBZ被转化为p-NBA,且96 h后将产生的p-NBA降解至(2.48±0.45) mg/L,TOC去除率达到58.65%±0.87%;菌株CAP-2降解p-NBA的速率较慢(图 5B),24 h后其降解率为55.81%±4.77%,48 h后降解率明显升高,达到87.95%±1.18%,96 h后p-NBA降解率为93.09%±0.80%,120 h后TOC去除率为25.88%±1.70%,表明其降解过程可能存在未检测到的代谢产物;PCA通常是p-NBA好氧生物降解过程中的代谢产物,也是芳香烃开环矿化的重要中间产物[14],由于p-NBA转化为PCA后能够迅速被降解,故本研究尚未检测到PCA的积累。为了证明菌株CAP-2能够降解PCA,进一步地以PCA为底物研究了菌株CAP-2对其降解效能(图 5C),24 h后PCA降解率为96.48%±0.82%,TOC去除率达到63.64%±0.58%。以上结果表明菌株CAP-2首先可能将CAP的侧链氧化转化为p-NBZ,其再被进一步氧化为p-NBA,之后通过已报道的p-NBA部分硝基还原过程生成对羟基氨基苯甲酸,经其裂解酶作用进一步转化为PCA,进而通过双加氧酶初始介导的一套酶系最终进入三羧酸循环而开环矿化[19] (图 6)。
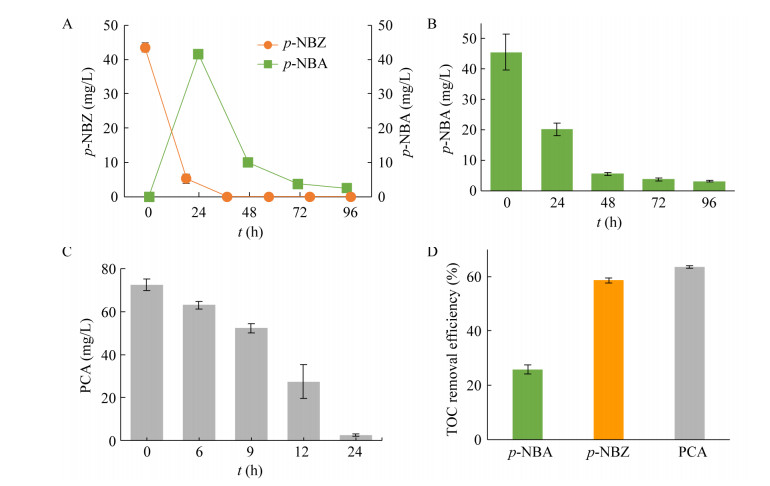
|
| 图 5 菌株CAP-2对CAP降解中间产物的降解特征(A:以p-NBZ为底物;B:以p-NBA为底物;C:以PCA为底物;D:菌株CAP-2对不同中间产物的矿化率) Fig. 5 The degradation characteristics of strain CAP-2 using CAP degradation intermediates (as the substrate p-NBZ (A), p-NBA (B), PCA (C) and the mineralization efficiency of strain CAP-2 on different intermediates (D)). |
| |

|
| 图 6 Rhodococcus sp. CAP-2降解氯霉素途径推测 Fig. 6 Putative CAP catabolic pathway in Rhodococcus sp. CAP-2. |
| |
本研究以受CAP污染的活性污泥为接种源首先富集获得一个由红球菌Rhodococcus主导(相对丰度为71.41%) 的CAP高效降解菌群,经过连续传代培养,24 h后CAP降解率为69.62%±6.18%,48 h后TOC去除率达到76.57%±1.08%,进一步从该富集液中分离培养了一株CAP高效降解菌CAP-2,通过16S rRNA基因鉴定为红球菌属,其能够以CAP作为唯一碳氮源实现60%以上的矿化效率,外加碳源(葡萄糖) 能够提升其对CAP及其关键降解产物p-NBA的降解效率。菌株CAP-2对CAP代谢中间产物具有降解能力,提出其降解途径为:首先CAP侧链被氧化断裂转化为p-NBZ,再被氧化为p-NBA,之后进一步转化为PCA进而开环矿化。与CAP侧链酰胺键水解细菌Klebsiella sp.、Escherichia sp.、Streptomyces venezuelae 13s和Streptomyces lividuns RM3相比,菌株CAP-2能将CAP降解矿化且没有芳香烃类产物的积累[18, 27-28];菌株CAP-2的TOC去除率与已报道的CAP降解菌Sphingomonas sp. CL5.1相比高出3%,且在CAP降解过程中并不涉及Sphingomonas sp. CL5.1中乙酰化和脱水的降解过程[20, 29]。此外,菌株CAP-2降解CAP的过程中也不生成氧化CAP的产物O-CAP (Sphingobium sp. CAP-1和Sphingomonas sp. CL5.1初始氧化降解CAP的关键产物)[9-20]。因此,推测菌株CAP-2初始氧化降解CAP的途径区别于上述报道,而是一个由CAP侧链氧化断裂生成p-NBZ和p-NBA主导的新型降解途径。
菌株CAP-2的获得丰富了CAP降解的生物资源,一方面能够以其作为模式菌株研究CAP在实际环境中由微生物介导的归趋转化,明晰其完整的降解途径与分子机理,挖掘并克隆表征其初始氧化降解的关键基因;另一方面将其作为降解菌株,研究在不同环境条件下的代谢能力并结合多孔生物炭材料和3D打印等新兴技术制备固定化菌剂应用于CAP污染场地的生物强化修复。这对于消除环境中CAP残留导致的微生物耐药性问题、保障人类与环境健康安全具有重要意义。
| [1] |
Van Boeckel TP, Brower C, Gilbert M, et al. Global trends in antimicrobial use in food animals. Proc Natl Acad Sci USA, 2015, 112(18): 5649-5654. DOI:10.1073/pnas.1503141112
|
| [2] |
Guo J, Li J, Chen H, et al. Metagenomic analysis reveals wastewater treatment plants as hotspots of antibiotic resistance genes and mobile genetic elements. Water Res, 2017, 123: 468-478. DOI:10.1016/j.watres.2017.07.002
|
| [3] |
Karkman A, Do TT, Walsh F, et al. Antibiotic- resistance genes in waste water. Trends Microbiol, 2018, 26(3): 220-228. DOI:10.1016/j.tim.2017.09.005
|
| [4] |
Michael I, Rizzo L, McArdell CS, et al. Urban wastewater treatment plants as hotspots for the release of antibiotics in the environment: a review. Water Res, 2013, 47(3): 957-995. DOI:10.1016/j.watres.2012.11.027
|
| [5] |
Zhang QQ, Ying GG, Pan CG, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance. Environ Sci Technol, 2015, 49(11): 6772-6782. DOI:10.1021/acs.est.5b00729
|
| [6] |
Balbi HJ. Chloramphenicol: a review. Pediatr Rev, 2004, 25(8): 284-288. DOI:10.1542/pir.25-8-284
|
| [7] |
Schwarz S, Kehrenberg C, Doublet B, et al. Molecular basis of bacterial resistance to chloramphenicol and florfenicol. FEMS Microbiol Rev, 2004, 28(5): 519-542. DOI:10.1016/j.femsre.2004.04.001
|
| [8] |
Scarzella M, Migazzo C, Cappio M. Typhoid fever treated with aureomycin and chloramphenicol. South Med J, 1949, 42(11): 983-985. DOI:10.1097/00007611-194911000-00013
|
| [9] |
Liang B, Cheng HY, Kong DY, et al. Accelerated reduction of chlorinated nitroaromatic antibiotic chloramphenicol by biocathode. Environ Sci Technol, 2013, 47(10): 5353-5361. DOI:10.1021/es400933h
|
| [10] |
吴昕. 临床应用氯霉素的不良反应. 临床合理用药杂志, 2008, 1(1): 36-37. Wu X. Adverse reactions of clinical application of chloramphenicol. Chin J Clinical Rational Drug Use, 2008, 1(1): 36-37 (in Chinese). DOI:10.3969/j.issn.1674-3296.2008.01.025 |
| [11] |
Gross BJ, Branchflower RV, Burke TR, et al. Bone marrow toxicity in vitro of chloramphenicol and its metabolites. Toxicol Appl Pharmacol, 1982, 64(3): 557-565. DOI:10.1016/0041-008X(82)90253-8
|
| [12] |
Klein EY, van Boeckel TP, Martinez EM, et al. Global increase and geographic convergence in antibiotic consumption between 2000 and 2015. Proc Natl Acad Sci USA, 2018, 115(15): E3463-E3470. DOI:10.1073/pnas.1717295115
|
| [13] |
Xu JY, Sangthong R, McNeil E, et al. Antibiotic use in chicken farms in northwestern China. Antimicrob Resist Infect Control, 2020, 9(1): 1-9. DOI:10.1186/s13756-019-0662-8
|
| [14] |
Basu S, Pal Chowdhury P, Deb S, et al. Degradation pathways of 2- and 4-nitrobenzoates in Cupriavidus sp. strain ST-14 and construction of a recombinant strain, ST-14: 3NBA, capable of degrading 3-nitrobenzoate. Appl Environ Microbiol, 2016, 82(14): 4253-4263. DOI:10.1128/AEM.00739-16
|
| [15] |
Yun H, Liang B, Qiu JG, et al. Functional characterization of a novel amidase involved in biotransformation of triclocarban and its dehalogenated congeners in Ochrobactrum sp. TCC-2. Environ Sci Technol, 2017, 51(1): 291-300. DOI:10.1021/acs.est.6b04885
|
| [16] |
Reis AC, Reis AC, Kolvenbach BA, et al. Biodegradation of antibiotics: the new resistance determinants-part Ⅱ. N Biotechnol, 2020, 54: 13-27. DOI:10.1016/j.nbt.2019.08.003
|
| [17] |
Smith AL, Erwin AL, Kline T, et al. Chloramphenicol is a substrate for a novel nitroreductase pathway in Haemophilus influenzae. Antimicrob Agents Chemother, 2007, 51(8): 2820-2829. DOI:10.1128/AAC.00087-07
|
| [18] |
Xin Z, Fengwei T, Gang W, et al. Isolation, identification and characterization of human intestinal bacteria with the ability to utilize chloramphenicol as the sole source of carbon and energy. FEMS Microbiol Ecol, 2012, 82(3): 703-712. DOI:10.1111/j.1574-6941.2012.01440.x
|
| [19] |
Ma XD, Liang B, Qi MY, et al. Novel pathway for chloramphenicol catabolism in the activated sludge bacterial isolate Sphingobium sp. CAP-1. Environ Sci Technol, 2020, 54(12): 7591-7600. DOI:10.1021/acs.est.9b07324
|
| [20] |
Zhang J, Gan W, Zhao R, et al. Chloramphenicol biodegradation by enriched bacterial consortia and isolated strain Sphingomonas sp. CL5.1: the reconstruction of a novel biodegradation pathway. Water Res, 2020, 187: 116397. DOI:10.1016/j.watres.2020.116397
|
| [21] |
Groenewegen PEJ, Bont JAM. Degradation of 4-nitrobenzoate via 4-hydroxylaminobenzoate and 3, 4-dihydroxybenzoate in Comamonas acidovorans NBA-10. Arch Microbiol, 1992, 158(5): 381-386. DOI:10.1007/BF00245369
|
| [22] |
Nishino SF, Spain JC. Degradation of nitrobenzene by a Pseudomonas pseudoalcaligenes. Appl Environ Microbiol, 1993, 59(8): 2520-2525. DOI:10.1128/aem.59.8.2520-2525.1993
|
| [23] |
Nishino SF, Spain JC. Oxidative pathway for the biodegradation of nitrobenzene by Comamonas sp. strain JS765. Appl Environ Microbiol, 1995, 61(6): 2308-2313. DOI:10.1128/aem.61.6.2308-2313.1995
|
| [24] |
Yabannavar AV, Zylstra GJ. Cloning and characterization of the genes for p-nitrobenzoate degradation from Pseudomonas pickettii YH105. Appl Environ Microbiol, 1995, 61(12): 4284-4290. DOI:10.1128/aem.61.12.4284-4290.1995
|
| [25] |
Kurosumi A, Kaneko E, Nakamura Y. Degradation of reactive dyes by ozonation and oxalic acid-assimilating bacteria isolated from soil. Biodegradation, 2008, 19(4): 489-494. DOI:10.1007/s10532-007-9153-3
|
| [26] |
Dutton PL, Evans WC. The metabolism of aromatic compounds by Rhodopseudomonas palustris. A new, reductive, method of aromatic ring metabolism. Biochem J, 1969, 113(3): 525-536. DOI:10.1042/bj1130525
|
| [27] |
Malik VS, Vining LC. Metabolism of chloramphenicol by the producing organism. Some properties of chloramphenicol hydrolase. Can J Microbiol, 1971, 17(10): 1287-1290. DOI:10.1139/m71-207
|
| [28] |
Mosher RH, Ranade NP, Schrempf H, et al. Chloramphenicol resistance in Streptomyces: cloning and characterization of a chloramphenicol hydrolase gene from Streptomyces venezuelae. J Gen Microbiol, 1990, 136(2): 293-301. DOI:10.1099/00221287-136-2-293
|
| [29] |
Zhang J, Zhao R, Cao L, et al. High-efficiency biodegradation of chloramphenicol by enriched bacterial consortia: kinetics study and bacterial community characterization. J Hazard Mater, 2020, 384: 121344. DOI:10.1016/j.jhazmat.2019.121344
|
 2021, Vol. 37
2021, Vol. 37




