中国科学院微生物研究所、中国微生物学会主办
文章信息
- 夏彬彬, 王军
- Xia Binbin, Wang Jun
- 基于深度学习的蛋白质建模与设计
- Protein modeling and design based on deep learning
- 生物工程学报, 2021, 37(11): 3863-3879
- Chinese Journal of Biotechnology, 2021, 37(11): 3863-3879
- 10.13345/j.cjb.210393
-
文章历史
- Received: May 27, 2021
- Accepted: July 15, 2021
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
蛋白质作为在机体中发挥主要作用的大分子,其在催化、转运、贮存、细胞结构、免疫等诸多方面发挥至关重要的作用[1]。蛋白质与DNA、RNA、其他蛋白质以及小分子物质的相互作用是其发挥多种生物学功能的物理基础。例如RNA聚合酶复合体与tRNA和DNA的相互作用、组蛋白与基因组DNA之间的相互作用、抗体与抗原表位的相互作用等。
随着蛋白质结构解析方法的发展,目前积累的已解析蛋白质结构数据越来越多。截止到2021年初,最大的蛋白质结构数据库(Protein data bank,PDB)[2]所积累的结构数据量已超过17万个。结构解析的本质是对于现有生物学现象和物质的描述性研究。深度学习是一种以统计学为手段,利用海量数据构建模型表征现实世界的方式。人们在获得了大量描述性信息之后,如何有效利用海量的结构数据信息并且应用到蛋白质设计中是一个至关重要的问题。蛋白质结构数据的处理和高效利用主要体现在两个方面:蛋白质结构预测和蛋白质设计,而设计又在很大程度上依赖于结构预测。
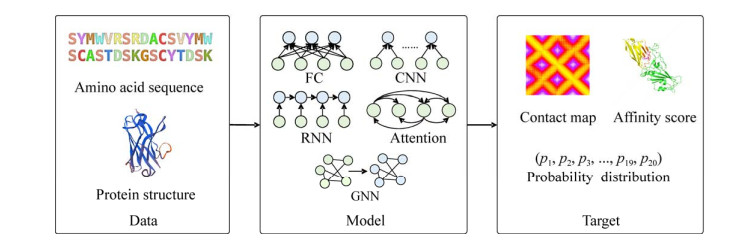
蛋白质结构预测(图 1A) 是指通过计算机算法根据氨基酸序列预测其空间结构,“至少对于标准生理环境中的小的球蛋白来说,它的天然结构仅由其氨基酸序列所决定”[3],这一表述于1973年被提出,称为Anfinsen法则。尽管如此,开发一种根据氨基酸序列就能预测蛋白质天然结构的方法仍然是一个巨大的挑战。这主要是由于我们当前还无法针对如此复杂的大分子,给出其折叠相关因素全面精确的物理描述,因此也推动研究者利用各种生物信息学方法开发预测算法。基于“相似的氨基酸序列可能拥有相似三维结构”而产生的同源建模方法将序列与已有结构的蛋白进行比对,利用已知的结构信息完成预测[4]。然而这种方法对同源性较低的蛋白质的预测效果不佳,这也是长久以来亟待突破的方向。随着深度学习的引入,这一问题得到了有效解决,先后有RaptorX-Contact[5]、RGN[6]、trRosetta[7]、AlphaFold[8]等一系列融入深度学习的新方法被提出。2020年由DeepMind研发的AlphaFold2人工智能系统在国际蛋白质结构预测竞赛CASP (The critical assessment of protein structure prediction) 上达到惊人的准确率,多数模型预测结构与实验测得的蛋白质真实结构高度一致,因而受到领域内高度关注。Nature杂志的新闻更是以“It will change everything”作为标题[9],指出该方法在解决蛋白结构问题上“迈出了一大步”,这是基于人工神经网络的深度学习算法在生物学领域重大问题上的一次跨越,也为包括蛋白质设计在内的相关领域奠定了坚实基础。
|
| 图 1 蛋白质结构预测与蛋白质设计的主要流程示意图 Fig. 1 Schematic representation of process for protein structure prediction (A) and protein design (B). |
| |
在结构预测任务成功的背后,微生物宏基因组数据发挥了至关重要的作用。蛋白质结构预测的首要步骤是基于大量氨基酸序列数据构建待预测序列的位置特异性矩阵(Position-specific scoring matrix,PSSM),该矩阵包含了序列保守性相关信息、氨基酸的权重与基序信息,因此氨基酸序列库的全面性与泛化水平就对结构预测的准确性有着直接影响。在蛋白质家族数据库Pfam中有接近15 000个蛋白质家族,其中近三分之一的蛋白质家族中至少存在一种已通过实验确定其结构的蛋白质;还有三分之一的家族,可根据计算与建模获得相对可靠的结构信息;而对于另外的5 211个蛋白家族,目前没有任何结构信息。研究人员通过整合宏基因组数据对蛋白质结构进行预测,发现有614个蛋白家族具有目前未知的结构,其中140个包含全新的蛋白质折叠模式[10]。由此可见,微生物宏基因组是重要的蛋白家族的数据来源,并且能增强蛋白结构预测的能力。
蛋白质设计的目标是基于序列和结构数据设计出与预期功能相符的蛋白质,这种设计可以是具有该功能的蛋白质序列,或者更进一步设计相应结构(图 1B)。目前该领域尚没有统一的框架和方法[11-12],其中较为知名的是RosettaDesign[13]以及基于它的一系列工具。对于理想的目标结构(能量上有利的规则构型,如ββ、βα、αβ及其衍生的复合构型) 此类工具可以获得很高成功率[14],但一般性的目标结构设计成功率仍然非常低[15]。因而也衍生出包括ABACUS2[16]、EvoDesign[17-18]等在内的一系列改进方法。通过蛋白质结构预测给予的先验知识,我们已知预定义的蛋白质局部二级结构在确定蛋白质整体构象、折叠动力学中的远距离接触(包括静电相互作用和疏水性堆积)具有关键作用,同时在给定构型下确定氨基酸种类至关重要。目前主流的设计方法主要基于采样和结构比对[13]。以全从头设计蛋白质为例,由于针对具体问题只有少数几种蛋白质骨架可以满足设计要求,首要问题是如何高效确定一种或多种蛋白骨架以满足核心残基的堆积和氢键的生成以获得稳定的蛋白,因此设计需要从大量的初始构象开始,这些初始的骨架构象可以来源于小肽片段的组装[19]或者基于代数方程以确定几何形状[20-21]。然而,这种骨架设计往往只在一些特定的典型构象中才具有较高的准确性,更具有普遍意义的构象有效性还需商榷[15]。因此实际操作中往往会先找出一个或多个已知结构的模板蛋白作为设计骨架(Scaffold)[22-23]。骨架确定后需要对关键残基进行精细化调节。对于非靶向的蛋白质设计,主要基于对蛋白稳定性进行评估;而针对特定靶点的设计则需要关注蛋白相互作用界面的热点(Hotspot) 残基,它被认为是维持相互作用的关键位点,因而需要进行精细化的分子对接以评估热点残基接触的稳定性,从而找出效果最佳的设计。但这样的设计方法仍存在一些问题,一方面,影响蛋白质设计成功率的因素很多,符合条件的骨架往往不易找到,结构预测的准确性、分子对接的准确性等都会影响到最终效果;另一方面,基于全柔性对接的方法在大量候选设计的筛选过程中也会消耗大量的计算资源,对于较大蛋白质的建模则代价更加高昂。
整体而言,蛋白质结构预测是蛋白质设计的基础。一方面,基于RosettaDesign的设计方法存在大量的突变与二次建模获得新结构的过程,可以说结构的预测是设计的必要条件;另一方面,基于深度学习方法的蛋白质结构预测也为蛋白质设计提供了先验知识,针对蛋白质结构与序列数据的建模方法在设计中必不可少,而现有的结构预测神经网络对此有大量的借鉴意义。但我们也要看到,蛋白质设计仍然面临精度不足,成功率不高和计算成本高昂的问题,尤其是在特定问题的定向设计中更是如此。
1 基于深度学习的蛋白质数据建模基于深度学习[24]的蛋白质设计作为一个新的应用领域,目的是希望克服现有蛋白质设计的缺陷,从解决计算成本问题、克服方法局限性的角度给予蛋白质设计新的可能性。其核心步骤是建立深度学习模型,将上游的海量数据与下游的建模目标结合,即蛋白质数据建模(也是深度学习建模) 的3个要素——数据、模型、目标。
1.1 数据对于一个蛋白质而言,所能存储的基本类型无外乎其氨基酸序列与三维结构信息两种(图 2-Data),除此之外还有基于它们生成的能够表征蛋白质的一系列衍生特征,例如氨基酸疏水性、二级结构和残基深度等。这些特征集合构成了蛋白质建模的数据基础。
|
| 图 2 蛋白质数据建模的三要素:数据、模型、目标 Fig. 2 The three essential elements of protein data modeling: data, model and target. |
| |
深度学习的输入形式为张量,在建模之前需要对原始数据进行转化,统一为张量形式,这样一个张量,就称为原始数据所对应的表示(Representation)[25-28]。当原始数据为疏水性、残基深度等的连续型变量时,可将数值与张量中的维度一一对应,从而完成转化;对于蛋白质结构数据,往往基于残基-残基或原子-原子之间的距离构建距离矩阵,从而完成张量化。氨基酸序列数据的张量化则需要针对每个氨基酸逐个获取其对应的表示,其表示方式分为局部表示(Local representation) 和分布式表示(Distributed representation) 两类(表 1)。
| Amino-acid residues | Local representation | Distributed representation | |
| Histidine | [1, 0, 0, ..., 0]T | [1.00, 0.23, 0.10, ..., 0.08]T | |
| Glycine | [0, 1, 0, ..., 0]T | [0.70, 0.62, 0.06, ..., 0.18]T | |
| Proline | [0, 0, 1, ..., 0]T | [0.52, 0.87, 1.00, ..., 0.78]T | |
| …… | …… | …… | |
| Serine | [0, 0, 0, ..., 1]T | [0.74, 0.28, 0.45, ..., 0.86]T | |
局部表示又称为独热编码(One-hot encoding),对于20种氨基酸,所有可能的氨基酸字符就可构成一个词表v,词表大小v=20,我们可以用一个|v|维的独热向量来表示每一种氨基酸残基,在第i种残基对应的向量中,第i维的值为1,其他都为0。但这样的表示每个残基对应的向量距离都相等,而实际上性质相似的氨基酸在蛋白质中发挥的作用往往也是相似的,即距离应当更近,故此这样的表示不能涵盖序列中的所有信息,就需要通过神经网络将局部表示空间 
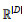
深度学习模型作为连接数据的纽带,它是否能够高效地从数据中抽取关键特征并且完成建模目标是评价其优劣的最基本标准。在高效抽取关键特征方面,需要对不同的蛋白质数据形式使用有针对性的网络类型。当前常用的网络类型主要分为:全连接网络、卷积神经网络、循环神经网络、注意力机制网络、图神经网络。
全连接网络(Fully connected,FC) 的核心操作是矩阵乘法,通过把一个特征空间线性变换到另一个特征空间,完成数据维度的转变(图 2-Model-FC),这一过程是明显的可并行化操作。实践中往往会将数据的特征空间(从神经网络得到或是通过特征工程构建) 映射到样本标签对应的空间。因此,FC往往不会单独使用,而是作为其他类型网络的最末几层,通过逐层减小特征空间的维度,发挥分类器的作用。
卷积神经网络(Convolutional neural network,CNN) 的建立是基于当前神经元的特征信息与它周边邻域特征相关的假设,所以CNN往往用于抽取数据的局部特征信息(图 2-Model-CNN)。对于氨基酸序列数据中的字符、接触图中的数值,如果将其顺序打乱,数据中蕴藏的信息也随即被破坏,即信息本身就在字符、数值的排列之中。因此,为了进行高效地预测,数据中的局部依赖性不能被忽视。在传统方法中,序列信息可以通过构建PSSM以及计算k-mers等方法获得其局部依赖性,但前者需要大量的序列比对,后者则丢失了序列本身的信息,只能作为序列信息的扩展。
卷积层是一种特殊形式的全连接层,相当于将多个低维的全连接层(称为卷积核或过滤器,convolution kernel or filter) 使用在序列或接触图中的每一个位置,类似于使用多个位置特异性矩阵(PSSM) 扫描序列。卷积层不仅能高效抽取数据的局部依赖性,还可以显著减少模型的参数量。每个卷积核局部扫描获得单个的标量作为下一层的输出维度之一,每个标量是对序列中某一局部相关性的量化。与全连接网络中的非线性激活函数类似,在一个卷积层之后通常会紧接着进行池化(Pooling) 操作。池化是将卷积得到的局部相关性信息通过最大化(Max pooling)、平均化(Average pooling) 等方式进一步抽提合并,获得显著减少的低维表示,所以池化过程实际上是一种下采样方法。最后,卷积层的输出可用作完全连接的神经网络的输入,以执行最终的预测任务。
卷积神经网络在蛋白质结构预测中获得广泛的应用。较为知名的RaptorX-Contact[5]、trRosetta[7]、AlphaFold[8]均为基于CNN的方法,它们以序列信息(具体来说为PSSM) 作为输入,预测其残基接触图,从而完成结构预测。实际操作中,CNN往往需要残差结构[30],该结构在深层网络的不同层之间引入短接(Shortcut connection),解决深度网络难以训练的问题,进而提升模型捕捉特征的能力。
循环神经网络(Recurrent neural network,RNN) 与CNN不同,它不再是对数据中的局部依赖进行建模,而是抽取数据中存在的长程依赖关系。RNN建立在这样一个基本假设上:当前神经元的特征信息与它之后的神经元特征相关(图 2-Model-RNN)。RNN可以用于表征具有顺序的结构化数据,即氨基酸序列的表征。与全连接网络和CNN只能接收固定长度的数据不同,RNN将相同的操作作用于每个序列数据元素,存储了之前元素特征的状态参数在网络中循环更新,因此RNN所能接收的数据形式更加广泛,对于序列数据,特别是长短不一的序列数据处理具有优势。从理论上讲,如果不存在内存容量的限制,RNN能够在无限长的序列中传递和抽取信息。但CNN结合诸如膨胀卷积在内各种技巧后能够达到与RNN相当甚至更好的性能。此外,由于RNN必须按照序列顺序逐个运算,因此难以并行化,相比CNN运算其速度慢得多。
递归几何神经网络(Recurrent geometric network,RGN)[6]是通过类比图像识别网络构建的基于序列信息的结构预测网络。它通过纯深度学习的方式进行结构预测,不依赖于结构模板等先验信息。其输入的是氨基酸序列信息,作者将氨基酸序列编码为41维的向量,其中包括蕴含20维氨基酸种类信息的One-Hot向量、20维PSSM位置向量和1维的位置编码,通过神经网络预期每个氨基酸对应的3个扭转角(φ、ψ、ω)。由于氨基酸和其上下文的氨基酸之间存在关联,故此使用双向长短期记忆(Long short-term memory,LSTM) 网络[31]最为合适,后者是RNN的一个变种,可以在一定程度上缓解梯度消失问题。
注意力机制网络[32-33] (Attention neural network) 最早由图像数据建模而引入,随后在自然语言处理中发展壮大。Attention机制模拟了人类的视觉,其核心是“从关注全部到关注重点”——人眼在观察图像时并不会首先看清图像中的每个细节,而是将注意力集中在接触图的焦点位置(图 2-Model- Attention)。与RNN类似,Attention机制也可以学习到序列信息中的长程依赖。对于单一序列建模所用的是基于自注意力机制(Self-attention) 的网络,它将每个序列元素表示为查询向量(Query,简写作Q)、键向量(Key,简写作K)、值向量(Value,简写作V),通过QKV的一系列运算,对于某一个Q向量(对应于序列的某个元素) 可以得到它与序列中其他所有元素的相关性大小,对每个Q向量重复这样的运算,就获得了整个序列的长程相关性表示。每个元素的运算都是独立且相同的,从而解决了RNN无法并行化的问题。与CNN多个卷积核并行运算相似,在注意力网络中也有相应的多头注意力机制(Multi-head attention),即对每个序列元素引入多套QKV向量,从不同角度对序列相关性进行建模。然而,这种模型结构也会引入另外的问题。RNN按照序列顺序逐个计算,天然包含了序列的顺序信息,但Attention机制并没有对此进行考虑,即对于相同字符的序列,打乱顺序后其Attention层的输出完全相同,这对字符种类有限的生物序列不利。因此需要在序列输入之前向其中加入顺序信息,把位置信息与序列元素信息相加,此过程称为位置编码(Positional encoding)。
在序列数据建模方面,基于Attention的机制往往被用于构建大型数据的预训练网络,通过大量以Attention机制为核心的网络模块叠加使用,在海量序列信息中抽取泛化特征,以供下游任务迁移学习使用[34-35]。一些研究也表明,预训练获得的氨基酸序列表示网络的特定层权重包含结构生物学中有意义的信息,即通过单纯对氨基酸序列的预训练可以获得其结构相关的特征[35-36]。
图神经网络(Graph neural network,GNN) 用于对非结构化数据中的依赖关系进行建模。蛋白质结构数据中的残基相互作用、原子三维结构关系等均为典型的非结构化的数据[37]。图神经网络的应用首先需要将现有的生物学网络建模成为图(Graph),以残基级表示为例,图中的每个节点(Node) 对应于氨基酸残基,而边(Edge) 对应于氨基酸间相互作用关系。GNN使用包括基于CNN和Attention机制的方法,将图中各个节点和边的特征进行聚合(Aggregator) 并获得一个新的图表示信息,在聚合结果中每个节点中包含邻居节点的特征信息,并且可以在下一个网络层再次聚合(图 2-Model-GNN)。与前面所述的所有网络相同,聚合过程也同样包含非线性函数的使用。可以训练GCN的任务包括节点分类[38]、无监督节点嵌入(旨在发现节点信息的低维表示)、边缘分类和图分类[39]。当前已知AlphaFold2在结构预测网络中将蛋白质定义为一个残基作为节点、残基关联作为边的空间图[40] (Spatial graph),并且使用注意力机制确定哪些残基之间的关联更加重要。
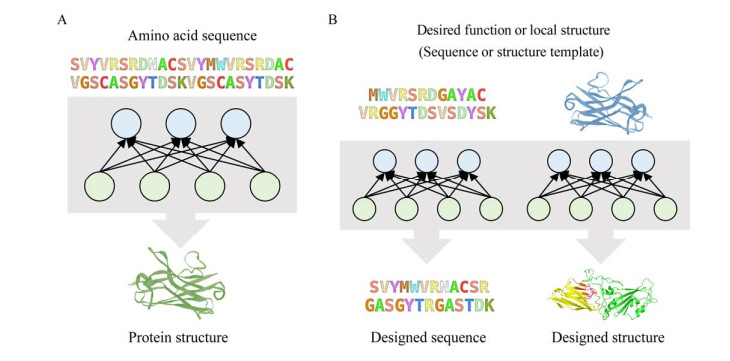
1.3 模型架构另一方面,描述某一对象的生物学数据是多模态的,并且针对研究对象所需解决的生物学问题又千差万别,神经网络作为一种结构灵活多变的建模形式,也需要顺应这种多样性引入不同的模型架构。为了适应模态多样、目标多变的数据建模的需求,神经网络可以大致划分为4种架构:单模态单任务架构(Monomodal single-task)、单模态多任务架构(Monomodal multitask)、多模态单任务架构(Multimodal single-task),以及迁移学习(Transfer learning)[41]。
对于基本的氨基酸序列分类或图分类问题,可将序列或结构作为输入,得到单一预测结果,因此使用单模态单任务架构即可(图 3A)。但对于一些复杂任务则需要给予模型更多的约束,这可以通过引入更多的预测目标来体现。例如,研究人员通过在自编码器的瓶颈层引入分类子网络,迫使瓶颈层在还原输入数据外还要保证本征向量与分类任务密切相关,从而将只能完成非监督任务的自编码器转换为一个有监督的分类器[42]。生物学数据建模也是如此,基于具体任务将网络的最后几层划分成两个子网络(图 3B),如果两子网络的预测任务存在较大关联,则新增预测任务可能会对原有预测任务产生有利影响,增加其预测准确率,从而达到比单任务架构更高的模型效率。在多任务架构中,总损失函数是每个任务的损失之和,当各个任务的损失差异很大时,可使用加权总和来平衡损失差距。
在复杂的生物学相互作用当中,即便设计出具有针对性的复杂网络架构,单一模态的数据所能提供的信息仍然是有限的。集成多个模态的最简单方式是在数据预处理阶段进行整合,类似于上文所述的建立数据表格,此方法也称为早期集成(Early integration)。然而,该方式仅能处理相似类型的数据,例如将分类型变量和数值型变量进行整合,但无法处理图像与序列数据这样差异巨大的数据整合。因此首先需要通过多个神经网络获得每个模态的对应低维表示,再将低维表示进行整合,这类神经网络称之为多模态网络。在该架构中,各个模态首先通过专用层进行处理,专用层的输出被合并后获得多个模态的集成数据,之后使用多个共享层进一步针对建模任务训练(图 3C),这类整合方式也称为中间集成(Intermediate integration)。中间集成的优势在于每个模态可使用与之对应的最适网络类型进行处理,因此可以更高效地提取更多有效特征。多模态的集成模型已在蛋白质结构数据的深度学习建模中得到应用,例如,利用分别表征几何与化学性质的蛋白质表面特征,使用不同网络分别对不同特征进行信息抽取,从而获得蛋白质相互作用指纹,还可以应用到下游的结合口袋预测、蛋白结合位点预测、蛋白质-蛋白质相互作用(Protein-protein interaction,PPI) 的筛选等任务中[43]。
迁移学习是解决数据稀缺的一种方式。虽然历史累计的生物学数据是海量的,但具体到某个细分问题,仍面临数据不足的问题,导致没有足够的数据从头训练一个模型。此时可使用经过类似任务训练的另一个模型中的大多数参数来初始化模型(图 3D),这种模型架构称之为迁移学习[44]。通过迁移学习,原有数据的先验知识被整合到当前的建模任务中,进一步的训练称为微调(Fine-tuning),在此过程中原有模型的参数可以进一步更新,也可保持不变。前者可以看作是在源模型所提取的特征之上构建一个独立的新模型。与使用随机初始化的参数从头开始训练的模型相比,迁移学习的训练过程可以更快地收敛,且需要的数据量更少。在生物图像分析中,研究人员成功地使用了来自ImageNet竞赛[45]的预训练模型对皮肤病变进行分类[46];在蛋白质序列数据上迁移学习的效用已被证明蕴含三维结构数据并可应用于蛋白质相互作用的预测[35, 47]。但由于缺乏针对多种模型性能的广泛评估,对于某一具体任务使用哪种模型可以获得更好的性能在当前的研究背景下仍缺少相应的指导信息。
需要指出的是,由于面临复杂多样的数据以及建模问题,多种网络架构往往会联合使用,特别是在基于无监督数据的预训练当中,例如:针对自然语言处理设计的预训练模型——基于变换器的双向编码器表示技术(Bidirectional encoder representations from transformers,BERT) 就是一个多任务架构模型[48],该模型通过自监督预训练的方式,构建针对自然语言数据的两种自监督任务,即掩码语言模型(Masked language model,MLM)和下文预测(Next sentence prediction)。该方式能够高效学习语料中的特征[35],获得序列的低维表示(Representation)。而通过无监督预训练获得低维表示的过程称为表示学习(Representation learning)[26]。学习到的表示想要应用于下游任务中,必须在当前的网络后添加全连接层,构建分类器,当BERT跟随下游任务继续训练从而更新其参数时,就进入微调阶段,这都是典型的迁移学习使用场景。类似于自然语言,当前已有不少对氨基酸序列进行预训练获得其表示的报道[35, 49-54],在蛋白质设计中基于序列信息的建模均可以通过相应表示作为输入,进而对设计目标进行预测。例如D-SCRIPT[47]以双氨基酸序列作为输入预测其亲和性,其氨基酸序列就直接使用了基于ProtTrans[54]预训练得到的表示。
1.4 建模目标从生物学角度而言,蛋白质设计的目的在于针对特定靶蛋白,设计出能够与之具有高亲和性或发挥其他特定生物学功能的蛋白质,或是给定蛋白质基本骨架,设计出满足骨架的蛋白质序列(图 2-Target)。但在方法学角度上,其建模目标又因为数据类型、模型架构的不同而存在差别。在从头蛋白质设计中,常需要构建多模态单任务架构的双输入神经网络以预测蛋白质结合能力,从而将候选结构筛选出来。例如,研究人员通过构建基于卷积的残差网络,以蛋白质-蛋白质的残基接触图作为预测目标[47];亦有其他报道更进一步试图通过接触图蕴含的信息,找出蛋白质互作的热点残基区域[55]。基于约束满足的设计方法试图找出符合特定三维结构的氨基酸序列,因此往往以一个20维的向量作为输出以预测不同种类氨基酸的概率。蛋白质结构生成则以蛋白质接触图作为预测目标,这也与结构预测相类似。
2 蛋白质设计方法 2.1 从数据生成:空间搜索与采样从数据中生成是一种典型的数据驱动方法,利用神经网络强大的信息抽取能力,捕捉数据特征的概率分布,并以此进行采样产生大量人造数据(图 4A)。生成式神经网络表征蛋白质序列、结构的概率分布,再依照分布信息产生新的蛋白质序列、结构,故此建模中的关键问题就在于如何针对不同的数据形式进行高效的信息编码与抽取,从而捕捉到数据中潜在的分布信息。该类方法在数据丰富的小分子生成中已经广泛应用,例如研究人员使用将变分自编码器与强化学习(Reinforcement learning) 相结合的深度神经网络模型,快速开发对治疗纤维化等疾病的靶标受体酪氨酸激酶(Discoidin domain receptor 1,DDR1)的新型抑制剂[56]。该研究利用包括广谱激酶抑制剂和DDR1特异抑制剂的生理生化性质在内的一系列数据,构建能够准确抽取DDR1抑制剂概率分布的深度生成模型,从概率分布中进行采样,自动生成大量潜在的合乎要求的化合物结构。在利用LSTM的数据生成方法中,有报道从大量短肽数据分布中进行采样,从而完成肽段设计[57]。
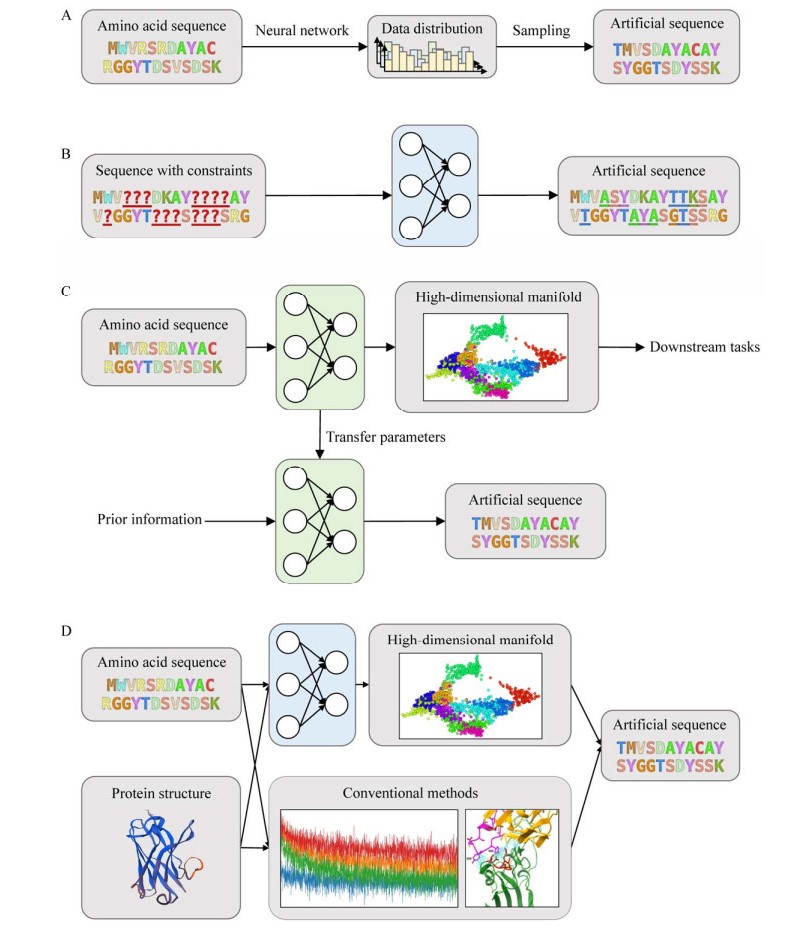
|
| 图 4 基于深度学习的蛋白质设计方法概述(A:利用空间搜索与采样方法从数据生成设计稿;B:通过求解约束满足问题完成改造与定向进化;C:基于迁移学习的设计方法;D:深度学习与传统方法结合的设计) Fig. 4 Protein design schemes based on deep learning. (A) Spatial search and sampling-based method by generation from data. (B) Determinate evolution by solving constraints problem. (C) Protein design based on transfer learning. (D) Combination of deep learning with conventional methods. |
| |
当前常用的深度生成模型包括:生成对抗网络、变分自编码器和长短期记忆网络3类。
2.1.1 生成对抗网络生成对抗网络(Generative adversarial network,GAN)[58-60]通过让两个神经网络相互博弈的方式进行学习,生成网络从潜在空间(Latent space) 中随机取样作为输入,其输出结果需要尽量模仿训练集中的真实样本。判别网络的输入是真实样本或生成网络的输出,其目的是将生成网络的输出从真实样本中尽可能分辨出来;而生成网络则要尽可能地欺骗判别网络。两个网络相互对抗、不断调整参数,最终目的是使判别网络无法判断生成网络的输出结果是否真实。整体收敛时认为生成器的结果足以以假乱真,也就编码了数据的分布信息。GAN可以对序列和结构信息进行生成,通过将结构信息表示为接触图的形式,当前已有方法基于GAN生成全新的接触图[61],之后通过Rosetta对其进行折叠获得全新设计的蛋白质结构,同时融合了基于残差网络的GAN也被用于生成与先验分布一致的氨基酸序列[62]。但对抗训练是一把双刃剑,其具有融合先验约束的训练优势,但具有训练困难的缺陷[63],限制了该方法的广泛应用。
2.1.2 变分自编码器变分自编码器(Variational autoencoder,VAE)[64]是具有附加分布假设的自动编码器,能够生成新的随机样本。该模型包含能够编码数据特征均值与方差信息的编码器,以及可以从编码信息中进行采样的生成器,通过生成结果与原数据的对比学习,构建出满足数据分布的一系列隐变量,并将其编码在中间的瓶颈层中。这样的方法常常被应用到小分子药物的生成与设计中[28, 65],蛋白质生成往往基于序列数据[66]。
2.1.3 长短期记忆网络长短期记忆网络[31]作为RNN的变体之一,其优势在于对序列信息的长程依赖编码上,因此常作为序列生成的方法。事实上对序列数据训练生成对抗网络时,生成器也往往选用长短期记忆网络,但与上述两种方法相比较,其单独作为生成方法时表征能力较为有限,现有方法也主要基于对短肽进行生成[57]。事实上,更多的基于LSTM的蛋白质设计是基于大规模预训练模型的应用[67],而非直接的蛋白质生成。
尤其在蛋白质结构数据的表征与序列生成中,上述方法均表征能力不足,无法针对包含序列排列、空间相对位置等一系列复杂信息在内的结构特征进行编码。因此,研究人员又提出基于图网络的蛋白质空间结构编码方案,从结构信息出发,产生符合要求的蛋白质序列信息[68]。
2.2 改造与定向进化:约束满足对于蛋白质设计而言,大量的实践仍然是基于对已有结构的改造,即对蛋白质靶点的成药性[69]、抗体的亲和性[70]、酶的催化效率[71]等进一步改进。这类方法都依赖于将先验信息作为一种约束,在该约束下进行新蛋白质的改进(图 4B)。在一些报道中,研究人员将这种方式比喻为数独游戏[72],依照数独游戏规则(约束信息),进行数字填写(新蛋白质的生成)。在该类方法中,作为约束信息的数据形式往往是蛋白质骨架,骨架强调蛋白质的三维结构信息与二级结构。在此约束下需要对大量可能的蛋白质排列组合进行采样,找出在该骨架下可以稳定存在的排布方式。正因如此,稳定性是该类设计中首先需要考量的因素,其优势也在于对单残基突变的敏感性,对于任一位点的突变往往可以较好地反映到模型的输出(表征稳定性) 中。但由于大量的蛋白质设计并不存在完善的蛋白质骨架先验信息,因此该方法往往作为设计中的一个环节出现,通过筛选剔除大量不合理设计,提高从头设计的效率与准确性。
2.3 基于迁移学习的设计方法迁移学习是神经网络的一种训练方法,通过将与当前任务相关联的神经网络迁移到当前任务中,利用其它领域的先验信息改善现有任务的性能表现(图 4C)。在一些基于序列信息的亲和测试模型中,研究任务需要以蛋白质序列作为神经网络的输入[47]。为了提高模型效率,往往需要引入氨基酸序列的预训练模型[35-36, 49-54],从中构建出蕴含大量先验信息的表示,之后再针对具体的下游任务对模型进行精修[67]。也有研究人员直接反用蛋白质结构预测网络,通过将trRosetta[7]的模型参数逆用,可依据蛋白质接触图产生大量符合条件的氨基酸序列[73-74],被称为trDesign。随后,研究人员又进一步开发出可生成大量不同结构类型但包含相同模体(Motif) 的网络架构[75],提示该方法对于功能蛋白的设计或许具有重要意义。
2.4 与传统方法的结合但我们仍然要清醒地认识到,当前对于蛋白质的设计仍需要分子对接、分子动力学模拟等方法的结合,以进一步提高准确性(图 4D)。研究人员通过分子动力学模拟获得蛋白质结构的时空数据,构建三维卷积神经网络对蛋白与小分子结合位点进行预测,验证蛋白的可成药性,并再次使用动力学模拟进行验证[69]。虽然传统方法准确性相对较高,但计算规模较大、算力成本高昂是制约其发展的因素,将深度学习的高效性与分子对接的准确性有机结合的方法报道不多,如何结合两者提出新的方法或许是未来的发展方向之一。
3 设计方案的评价 3.1 数据分布与模型解释性深度学习作为蛋白质设计领域的新方法,如何对现有的设计依据进一步解释,对生成数据的分布进行检验,是推动领域发展的关键问题。
对于氨基酸序列的生成而言,其多样性、偏好性是其评价指标。研究人员发现仅从氨基酸序列出发对序列信息进行表征,其深度神经网络的参数信息也同样能够包含二级结构[36]或者三级结构的残基接触信息[35],从而证明了使用深度学习对蛋白质序列表征的准确性和可靠性。
对于蛋白质结构的生成而言,其稳定性、合理性是衡量有效性的标准,而使用基于能量函数[76-77]的Rosetta打分系统对蛋白质结构进行评估是评价其优劣的常用方式。通过能量函数对兰纳-琼斯势(Lennard-Jones potential,L-J potential)、静电势、氢键相互作用、二硫键键能、溶剂化能等一系列与蛋白质结构密切相关的指标,从而对大量蛋白质设计稿进行筛选,剔除不合理结构,或者依照打分结果对设计稿排序,节约下游任务的计算量。打分系统可使用score_jd2命令对任意结构进行调用,并且对各个打分项的权重进行自定义。另有基于TM-align对生成的结构信息分布进行表征,从而识别模型的偏好性与采样过程,追溯不同设计结构的能量景观变化[73]。
3.2 对接与模拟深度学习方法相较于分子对接与动力学模拟具有计算效率上无可比拟的优势,但准确性还有待考量,因此与现有传统方法的准确性比较就是新方法创新性的考量因素之一。事实上,目前蛋白质设计方法主要基于分子对接与动力学模拟,当前较为成功的蛋白质设计仅针对特定的少数案例,并且主要是一些蛋白质超二级结构或由它们组合而成的复合体(对称蛋白质)。在一些设计场景中,可以使用分子对接对蛋白质结合界面进行分析[72],或使用分子动力学模拟验证蛋白质的稳定性[69],从而验证设计的有效性。
3.3 实验验证无论如何,实验仍然是任何蛋白质设计的金标准。当前基于深度学习的蛋白质设计,特别是一些创新性方法往往缺乏验证。一方面是由于方法准确性不足,距离落地验证仍然存在差距;另一方面也缺乏高效的验证手段。研究人员报道了一种基于酵母表面展示技术的微型蛋白(Mini-protein) 高通量筛选方法[78],该方法观察测试了15 000多种基于Rosetta方法新设计的在自然中不存在的微型蛋白是否形成折叠结构,对设计有效性与准确率进行验证,从而形成了“设计-验证-获得新数据-先验信息再设计”的迭代过程。但我们也看到,这样大规模的验证仍然只能基于对蛋白稳定性这类较为宽泛的指标进行测试,一些较大规模的功能性蛋白质设计也选择了绿色荧光蛋白[36, 67]这种功能单一、检测手段成熟的研究对象。总之,复杂功能的验证(例如:亲和性、可成药性等) 仍然会是一个低通量且具有挑战性的过程,这也对蛋白质设计方法的精准度(Precision) 提出了更高的要求。
4 总结与展望蛋白质设计是具有广阔应用前景的研究领域,其对于新药研发、药物递送、靶向治疗、材料科学均具有重要意义。特别是在微生物感染的治疗方面,通过高效方法设计的蛋白质药物可以靶向特定菌种,或有望克服抗生素滥用带来的耐药问题,有针对性地发挥治疗作用[79]。
当前基于深度学习方法对蛋白质数据从不同层面进行表征的方法已经趋于完善,尤其在蛋白质结构预测领域迁移的经验对于蛋白表征具有重要借鉴意义。通过将蛋白质的数据形式进行扩展,使用不同类型、不同架构的模型,针对蛋白质数据进行表征,完成预期目标的预测,亦能够获得较高的准确率。
即便如此,通过深度学习方法进行蛋白质设计仍然是一个新兴领域。当前研究大多局限于蛋白质设计的某一具体环节,仍缺乏系统性的创新研究。trDesign或许是未来的发展方向之一,但其准确率与先验信息的加入方法仍然会是一个长期困扰研究人员的问题。
综上所述,基于深度学习的蛋白质设计是一个意义深远的研究领域,但其研究目前仍处于初级阶段,我们已经看到与之相关技术领域的发展与成熟,如何将这些领域知识迁移、针对蛋白质设计的具体场景进行再创新,是未来要解决的关键问题。
| [1] |
利亚斯, 苏晓东. 结构生物学. 北京: 科学出版社, 2013. Liljas A, Su XD. Textbook of structural biology. Beijing: Science Press, 2013 (in Chinese). |
| [2] |
Berman H, Henrick K, Nakamura H. Announcing the worldwide protein data bank. Nat Struct Biol, 2003, 10(12): 980. DOI:10.1038/nsb1203-980
|
| [3] |
Anfinsen CB. Principles that govern the folding of protein chains. Science, 1973, 181(4096): 223-230. DOI:10.1126/science.181.4096.223
|
| [4] |
Waterhouse A, Bertoni M, Bienert S, et al. SWISS-MODEL: homology modelling of protein structures and complexes. Nucleic Acids Res, 2018, 46(w1): W296-W303. DOI:10.1093/nar/gky427
|
| [5] |
Wang S, Sun S, Li Z, et al. Accurate de novo prediction of protein contact map by ultra-deep learning model. PLoS Comput Biol, 2017, 13(1): e1005324. DOI:10.1371/journal.pcbi.1005324
|
| [6] |
AlQuraishi M. End-to-end differentiable learning of protein structure. Cell Syst, 2019, 8(4): 292-301. e3. DOI:10.1016/j.cels.2019.03.006
|
| [7] |
Yang J, Anishchenko I, Park H, et al. Improved protein structure prediction using predicted interresidue orientations. Proc Natl Acad Sci USA, 2020, 117(3): 1496-1503. DOI:10.1073/pnas.1914677117
|
| [8] |
Senior AW, Evans R, Jumper J, et al. Improved protein structure prediction using potentials from deep learning. Nature, 2020, 577(7792): 706-710. DOI:10.1038/s41586-019-1923-7
|
| [9] |
Callaway E. 'It will change everything': DeepMind's AI makes gigantic leap in solving protein structures. Nature, 2020, 588(7837): 203-204. DOI:10.1038/d41586-020-03348-4
|
| [10] |
Ovchinnikov S, Park H, Varghese N, et al. Protein structure determination using metagenome sequence data. Science, 2017, 355(6322): 294-298. DOI:10.1126/science.aah4043
|
| [11] |
曲戈, 朱彤, 蒋迎迎, 等. 蛋白质工程: 从定向进化到计算设计. 生物工程学报, 2019, 35(10): 1843-1856. Qu G, Zhu T, Jiang YY, et al. Protein engineering: from directed evolution to computational design. Chin J Biotech, 2019, 35(10): 1843-1856 (in Chinese). |
| [12] |
蒋迎迎, 曲戈, 孙周通. 机器学习助力酶定向进化. 生物学杂志, 2020, 37(4): 1. Jiang YY, Qu G, Sun ZT. Machine learning-assisted enzyme directed evolution. Journal of Biology, 2020, 37(4): 1 (in Chinese). |
| [13] |
Leaver-Fay A, Tyka M, Lewis SM, et al. ROSETTA3: an object-oriented software suite for the simulation and design of macromolecules. Methods Enzymol, 2011, 487: 545-574.
|
| [14] |
Koga N, Koga N, Tatsumi-Koga R, et al. Principles for designing ideal protein structures. Nature, 2012, 491(7423): 222-227. DOI:10.1038/nature11600
|
| [15] |
Li Z, Yang Y, Zhan J, et al. Energy functions in de novo protein design: current challenges and future prospects. Annu Rev Biophys, 2013, 42: 315-335. DOI:10.1146/annurev-biophys-083012-130315
|
| [16] |
Xiong P, Wang M, Zhou X, et al. Protein design with a comprehensive statistical energy function and boosted by experimental selection for foldability. Nat Commun, 2014, 5: 5330. DOI:10.1038/ncomms6330
|
| [17] |
Pearce R, Huang X, Setiawan D, et al. EvoDesign: designing protein-protein binding interactions using evolutionary interface profiles in conjunction with an optimized physical energy function. J Mol Biol, 2019, 431(13): 2467-2476. DOI:10.1016/j.jmb.2019.02.028
|
| [18] |
Huang X, Pearce R, Zhang Y. De novo design of protein peptides to block association of the SARS-CoV-2 spike protein with human ACE2. Aging, 2020, 12(12): 11263-11276. DOI:10.18632/aging.103416
|
| [19] |
Kuhlman B, Dantas G, Ireton GC, et al. Design of a novel globular protein fold with atomic-level accuracy. Science, 2003, 302(5649): 1364-1368. DOI:10.1126/science.1089427
|
| [20] |
Harbury PB, Plecs JJ, Tidor B, et al. High-resolution protein design with backbone freedom. Science, 1998, 282(5393): 1462-1467. DOI:10.1126/science.282.5393.1462
|
| [21] |
Thomson AR, Wood CW, Burton AJ, et al. Computational design of water-soluble α-helical barrels. Science, 2014, 346(6208): 485-488. DOI:10.1126/science.1257452
|
| [22] |
Fleishman SJ, Whitehead TA, Ekiert DC, et al. Computational design of proteins targeting the conserved stem region of influenza hemagglutinin. Science, 2011, 332(6031): 816-821. DOI:10.1126/science.1202617
|
| [23] |
Cao Y, Geddes TA, Yang JYH, et al. Ensemble deep learning in bioinformatics. Nat Mach Intell, 2020, 2(9): 500-508. DOI:10.1038/s42256-020-0217-y
|
| [24] |
LeCun Y, Bengio Y, Hinton G. Deep learning. Nature, 2015, 521(7553): 436-444. DOI:10.1038/nature14539
|
| [25] |
Baxter J. Learning internal representations. Proceedings of the eighth annual conference on computational learning theory — COLT '95. July 5-8, 1995. Santa Cruz, California, USA. New York: ACM Press, 1995: 311-320.
|
| [26] |
Bengio Y, Courville A, Vincent P. Representation learning: a review and new perspectives. IEEE Trans Pattern Anal Mach Intell, 2013, 35(8): 1798-1828. DOI:10.1109/TPAMI.2013.50
|
| [27] |
Meiler J, Müller M, Zeidler A, et al. Generation and evaluation of dimension-reduced amino acid parameter representations by artificial neural networks. Mol Model Annu, 2001, 7(9): 360-369. DOI:10.1007/s008940100038
|
| [28] |
Tavakoli M, Baldi P. Continuous representation of molecules using graph variational autoencoder. arXiv preprint arXiv: 2004.08152, 2020.
|
| [29] |
Xu JB, McPartlon M, Li J. Improved protein structure prediction by deep learning irrespective of co-evolution information. Nat Mach Intell, 2021, 1-9.
|
| [30] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. 2016 IEEE Conf Comput Vis Pattern Recognit CVPR, 2016, 770-778.
|
| [31] |
Hochreiter S, Schmidhuber J. Long Short-term memory. Neural Computation, 1997, 9(8): 1735-1780. DOI:10.1162/neco.1997.9.8.1735
|
| [32] |
Bahdanau D, Cho K, Bengio Y. Neural machine translation by jointly learning to align and translate. arXiv preprint arXiv: 1409.0473, 2014.
|
| [33] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Adv Neural Inf Process Syst, 2017, 30.
|
| [34] |
Rives A, Meier J, Sercu T, et al. Biological structure and function emerge from scaling unsupervised learning to 250 million protein sequences. Proc Natl Acad Sci USA, 2021, 118(15): e2016239118. DOI:10.1073/pnas.2016239118
|
| [35] |
Vig J, Madani A, Varshney L R, et al. BERTology meets biology: interpreting attention in protein language models. International Conference on Learning Representations, 2020.
|
| [36] |
Alley EC, Khimulya G, Biswas S, et al. Unified rational protein engineering with sequence-based deep representation learning. Nat Methods, 2019, 16(12): 1315-1322. DOI:10.1038/s41592-019-0598-1
|
| [37] |
Mitra K, Carvunis AR, Ramesh SK, et al. Integrative approaches for finding modular structure in biological networks. Nat Rev Genet, 2013, 14(10): 719-732. DOI:10.1038/nrg3552
|
| [38] |
Chen J, Ma TF, Xiao C. FastGCN: fast learning with graph convolutional networks via importance sampling. arXiv preprint arXiv: 1801.10247, 2018.
|
| [39] |
Battaglia PW, Hamrick JB, Bapst V, et al. Relational inductive biases, deep learning, and graph networks. arXiv preprint arXiv: 1806.01261, 2018.
|
| [40] |
Gilmer J, Schoenholz SS, Riley PF, et al. Neural message passing for quantum chemistry. Proceedings of the 34th International Conference on Machine Learning, PMLR, 2017, 70: 1263-1272.
|
| [41] |
Eraslan G, Avsec Ž, Gagneur J, et al. Deep learning: new computational modelling techniques for genomics. Nat Rev. Genet, 2019, 20(7): 389-403.
|
| [42] |
Hartono P. Mixing autoencoder with classifier: conceptual data visualization. IEEE Access, 2020, 8: 105301-105310. DOI:10.1109/ACCESS.2020.2999155
|
| [43] |
Gainza P, Sverrisson F, Monti F, et al. Deciphering interaction fingerprints from protein molecular surfaces using geometric deep learning. Nat Methods, 2020, 17(2): 184-192. DOI:10.1038/s41592-019-0666-6
|
| [44] |
Yosinski J, Clune J, Bengio Y, et al. How transferable are features in deep neural networks? Proceedings of the 27th International Conference on Neural Information Processing Systems - Volume 2, Cambridge, MA, USA: MIT Press, 2014: 3320-3328.
|
| [45] |
Russakovsky O, Deng J, Su H, et al. ImageNet large scale visual recognition challenge. Int J Comput Vis, 2015, 115(3): 211-252. DOI:10.1007/s11263-015-0816-y
|
| [46] |
Esteva A, Kuprel B, Novoa RA, et al. Dermatologist-level classification of skin cancer with deep neural networks. Nature, 2017, 542(7639): 115-118. DOI:10.1038/nature21056
|
| [47] |
Sledzieski S, Singh R, Cowen L, et al. Sequence-based prediction of protein-protein interactions: a structure-aware interpretable deep learning model. bioRxiv Preprint, 2021: 2021.01.22.427866.
|
| [48] |
Devlin J, Chang M-W, Lee K, et al. BERT: pre-training of deep bidirectional transformers for language understanding. arXiv preprint arXiv: 1810.04805, 2018.
|
| [49] |
Rao R, Bhattacharya N, Thomas N, et al. Evaluating protein transfer learning with TAPE. Adv Neural Inf Process Syst, 2019, 32: 9689-9701.
|
| [50] |
Strodthoff N, Wagner P, Wenzel M, et al. UDSMProt: universal deep sequence models for protein classification. Bioinformatics, 2020, 36(8): 2401-2409. DOI:10.1093/bioinformatics/btaa003
|
| [51] |
Min S, Park S, Kim S, et al. Pre-training of deep bidirectional protein sequence representations with structural information. arXiv preprint arXiv: 1912.05625, 2019.
|
| [52] |
Luo JW, Cai Y, Wu JL, et al. Self-supervised representation learning of protein tertiary structures (PtsRep): protein engineering as a case study. bioRxiv preprint, 2020: 2020.12.22.423916.
|
| [53] |
Brandes N, Ofer D, Peleg Y, et al. ProteinBERT: a universal deep-learning model of protein sequence and function. bioRxiv Preprint, 2021: 2021.05.24.445464.
|
| [54] |
Elnaggar A, Heinzinger M, Dallago C, et al. ProtTrans: towards cracking the language of life's code through self-supervised deep learning and high performance computing. IEEE Transactions on Pattern Analysis and Machine Intelligence, IEEE Computer Society, 2021(1): 1-1.
|
| [55] |
Liu Y, Zhu YH, Song XN, et al. Why can deep convolutional neural networks improve protein fold recognition? A visual explanation by interpretation. Brief Bioinform, 2021, bbab001.
|
| [56] |
Zhavoronkov A, Ivanenkov YA, Aliper A, et al. Deep learning enables rapid identification of potent DDR1 kinase inhibitors. Nat Biotechnol, 2019, 37(9): 1038-1040. DOI:10.1038/s41587-019-0224-x
|
| [57] |
Müller AT, Hiss JA, Schneider G. Recurrent neural network model for constructive peptide design. J Chem Inf Model, 2018, 58(2): 472-479. DOI:10.1021/acs.jcim.7b00414
|
| [58] |
Goodfellow I, Pouget-Abadie J, Mirza M, et al. Generative adversarial networks. Commun ACM, 2020, 63(11): 139-144. DOI:10.1145/3422622
|
| [59] |
Goodfellow I. NIPS 2016 tutorial: generative adversarial networks. arXiv preprint arXiv: 1701.00160, 2016.
|
| [60] |
Yu LT, Zhang WN, Wang J, et al. SeqGAN: sequence generative adversarial nets with policy gradient. arXiv preprint arXiv: 1609.05473, 2016.
|
| [61] |
Anand N, Huang P. Generative modeling for protein structures. Adv New Inf Proc Syst, 2018, 31.
|
| [62] |
Repecka D, Jauniskis V, Karpus L, et al. Expanding functional protein sequence spaces using generative adversarial networks. Nat Mach Intell, 2021, 3(4): 324-333. DOI:10.1038/s42256-021-00310-5
|
| [63] |
Arjovsky M, Bottou L. Towards principled methods for training generative adversarial networks. arXiv preprint arXiv arXiv: 1701.04862, 2017.
|
| [64] |
Kingma DP, Welling M. Auto-encoding variational bayes. arXiv preprint arXiv: 1312.6114, 2014.
|
| [65] |
Hong SH, Ryu S, Lim J, et al. Molecular generative model based on an adversarially regularized autoencoder. J Chem Inf Model, 2020, 60(1): 29-36. DOI:10.1021/acs.jcim.9b00694
|
| [66] |
Hawkins-Hooker A, Depardieu F, Baur S, et al. Generating functional protein variants with variational autoencoders. PLoS Comput Biol, 2021, 17(2): e1008736. DOI:10.1371/journal.pcbi.1008736
|
| [67] |
Biswas S, Khimulya G, Alley EC, et al. Low-N protein engineering with data-efficient deep learning. Nat Methods, 2021, 18(4): 389-396. DOI:10.1038/s41592-021-01100-y
|
| [68] |
Ingraham J, Garg V, Barzilay R, et al. Generative models for graph-based protein design. Adv Neur Inf Proc Syst, 2019, 32.
|
| [69] |
Kozlovskii I, Popov P. Spatiotemporal identification of druggable binding sites using deep learning. Commun Biol, 2020, 3(1): 618. DOI:10.1038/s42003-020-01350-0
|
| [70] |
Nimrod G, Fischman S, Austin M, et al. Computational design of epitope-specific functional antibodies. Cell Rep, 2018, 25(8): 2121-2131. e5. DOI:10.1016/j.celrep.2018.10.081
|
| [71] |
Kiss G, Çelebi-Ölçüm N, Moretti R, et al. Computational enzyme design. Angew Chem Int Ed, 2013, 52(22): 5700-5725. DOI:10.1002/anie.201204077
|
| [72] |
Strokach A, Becerra D, Corbi-Verge C, et al. Fast and flexible protein design using deep graph neural networks. Cell Syst, 2020, 11(4): 402-411. e4. DOI:10.1016/j.cels.2020.08.016
|
| [73] |
Anishchenko I, Chidyausiku TM, Ovchinnikov S, et al. De novo protein design by deep network hallucination. bioRxiv Preprint, 2020, 2020.
|
| [74] |
Norn C, Wicky BIM, Juergens D, et al. Protein sequence design by conformational landscape optimization. Proc Natl Acad Sci USA, 2021, 118(11): e2017228118. DOI:10.1073/pnas.2017228118
|
| [75] |
Tischer D, Lisanza S, Wang J, et al. Design of proteins presenting discontinuous functional sites using deep learning. bioRxiv Preprint, 2020: 2020.11.29.402743.
|
| [76] |
Park H, Bradley P, Greisen P, et al. Simultaneous optimization of biomolecular energy functions on features from small molecules and macromolecules. J Chem Theory Comput, 2016, 12(12): 6201-6212. DOI:10.1021/acs.jctc.6b00819
|
| [77] |
Alford RF, Leaver-Fay A, Jeliazkov JR, et al. The Rosetta all-atom energy function for macromolecular modeling and design. J Chem Theory Comput, 2017, 13(6): 3031-3048. DOI:10.1021/acs.jctc.7b00125
|
| [78] |
Rocklin GJ, Chidyausiku TM, Goreshnik I, et al. Global analysis of protein folding using massively parallel design, synthesis, and testing. Science, 2017, 357(6347): 168-175. DOI:10.1126/science.aan0693
|
| [79] |
Fjell CD, Hiss JA, Hancock REW, et al. Designing antimicrobial peptides: form follows function. Nat Rev Drug Discov, 2012, 11(1): 37-51. DOI:10.1038/nrd3591
|
 2021, Vol. 37
2021, Vol. 37