中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘涛, 田亚晨, 刘程, 方水琴, 吴有雪, 吴美娇, 李斌, 杨昊, 刘箐
- Liu Tao, Tian Yachen, Liu Cheng, Fang Shuiqin, Wu Youxue, Wu Meijiao, Li Bin, Yang Hao, Liu Qing
- 基于CRISPR/Cas体系的生物传感器在病原体核酸检测方面的应用
- Application of CRISPR/Cas-based biosensors for detecting nucleic acid of pathogens
- 生物工程学报, 2021, 37(11): 3890-3904
- Chinese Journal of Biotechnology, 2021, 37(11): 3890-3904
- 10.13345/j.cjb.200679
-
文章历史
- Received: October 22, 2020
- Accepted: January 25, 2021
病原体引起的传染性疾病是威胁人类健康的主要因素,据世界卫生组织统计,每年由传染病引起的死亡占全球死亡人数的1/4。近期暴发的新型冠状病毒SARS-CoV-2,截至2021年11月5日,全球已累计确诊2.49亿新型冠状病毒阳性病例,在面对突发病原体疫情时,快速准确地检测识别病原体对有效地管理和治疗传染性疾病至关重要[1-3]。目前对病原体的检测方法主要分为两类:第一类是传统病原体检测方法,包括分离培养[4]和免疫学检测方法[5]。酶联免疫吸附试验(Enzyme linked immunosorbent assay,ELISA) 作为应用最广泛的方法之一,是利用抗原和抗体的特异性识别,通过颜色变化鉴定病原微生物,但制备相应有效的抗体周期较长,并且灵敏度较低[6-7];第二种是基于分子生物学的检测[8],包括核酸扩增[9]、基因芯片[10]和基因测序[11]等方法,主要是基于病原体基因组信息。聚合酶链式反应(Polymerase chain reaction,PCR) 作为核酸检测的“金标准”,利用特异性引物和DNA聚合酶对靶标序列进行扩增,整个过程经过变性-退火-延伸3个反应步骤组成,扩增产物通过凝胶电泳判断产物大小[12]。在此基础上发展起来的实时荧光定量PCR (Quantitative real-time PCR,qRT-PCR) 因其能够精准可靠地检测病原体核酸,已被广泛地应用于各种临床样本的检测。然而,这些方法大多需要复杂的实验仪器和熟练的操作人员,且检测周转时间较长,使其无法在第一时间获得检测结果,这大大限制了其应用范围[13]。因此迫切地需要一种精确、灵敏、稳定且操作简便的新型检测分析方法,能够应用于现场诊断(Point-of-care testing,POCT)。
成簇规律间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)/成簇规律间隔短回文重复序列相关蛋白(CRISPR-associated protein,Cas) 作为继锌指核酸内切酶(Zinc finger endonuclease,ZFN)[14]和类转录激活因子效应物(Transcription activator-like effector nucleases,TALEN)[15]之后的一种新型基因编辑技术,能够更加简单、经济和高效地应用在基因编辑相关领域[16-18]。除此之外,CRISPR/Cas还可以应用在核酸检测方面[19-21]。得益于CRISPR/Cas体系对靶标核酸序列的精准识别,基于该体系的新型生物传感器在病原体核酸检测方面显示出了优良的性能,受到了人们的广泛关注。本文就基于CRISPR/Cas体系的生物传感器在病原体核酸检测中的最新研究进展进行综述,以期能够为病原体核酸检测方法的开发提供参考和帮助。
1 CRISPR系统的简介CRISPR/Cas作为古菌和绝大多数细菌的获得性免疫系统,在生物体内发挥抵御外源DNA、质粒和噬菌体入侵的功能[22-25]。CRISPR/Cas抵御外源核酸主要分为3个阶段[26]:首先,当外源基因入侵细菌后,细菌体内会启动自身免疫机制,将外源核酸整合到细菌自身的CRISPR回文重复序列中;然后在RNA聚合酶的作用下CRISPR序列开始转录,被剪切为成熟的CRISPR RNA (crRNA),形成向导RNA (Single-guide RNA,sgRNA),crRNA发挥向导功能;最后Cas蛋白通过识别外源核酸中的前间隔序列邻近基序(Protospacer adjacent motif,PAM),sgRNA引导Cas效应蛋白与靶标序列相互作用,引发对外源核酸序列的定向切割,从而消除外源基因。
基于CRISPR/Cas核酸检测体系的应用起源于Cas13a效应蛋白的“附带切割”能力的发现[27]。研究人员在进行Cas13a体外切割活性实验时发现,当靶标RNA添加到CRISPR/Cas13a体系中后,crRNA/Cas13a复合物不仅可以切割靶RNA,还可以非特异性地切割其他非靶标RNA[28]。利用这种切割活性成功检测到了amol/L浓度的靶标DNA/RNA,证明了CRISPR/Cas技术在核酸检测应用的可行性。随着研究的不断深入,越来越多的Cas蛋白被发现能够用作核酸识别和切割的工具,通过与核酸扩增相结合,基于CRISPR/Cas的核酸检测体系可实现单碱基分辨率并区分病原体的不同亚型[20]。
2 基于CRISPR/Cas体系的新型生物传感器生物传感器是一种能够将感应生物反应并将生物反应经过信号转导和信号放大元件转化为可处理和可定量的理化信号的生物传感装置,其主要构造包括生物敏感的识别元件(酶、抗体、抗原、微生物、细胞、组织、核酸等),适当的理化转化器及信号放大装置[29-30]。目前基于CRISPR/Cas体系的新型生物传感器是以核酸或者探针对病原体核酸序列的特异性识别为基础,将生物信号放大并转化为容易检测的理化信号,通过处理这些理化信号可实现定性或定量检测病原体的目的。按照传感器器件检测的原理可分为比色传感器、荧光传感器和电化学传感器等(表 1)。本文着重介绍基于CRISPR/Cas系统的几种常见检测传感器。
| Type | System name | Cas effector | Amplification | Sensitivity | Single-base resolution | Quantification | Time | Target | References |
| Fluorescence biosensors | CAS-EXPAR | Cas9 | EXPAR | Sub-amol/L | Yes | No | ≤1 h | DNA/RNA | [41] |
| dCas9/sgRNA-SG Ⅰ based DNA-FISH | dCas9 | No amplification | 10 CFU/mL | No | No | < 30 min | DNA | [42] | |
| PC reporter | dCas9 | PCR | Single copy/reaction | No | No | 10 min after PCR | DNA | [52] | |
| CRISDA | Cas9n | KF-madiated amplification | amol/L | Yes | Yes | 2–3 h | DNA | [53] | |
| Cas9nAR | Cas9n | KF-madiated amplification | 2 copies/reaction | Yes | Yes | < 1 h | DNA | [54] | |
| SHERLOCK | Cas13a | RPA | amol/L | Yes | No | 2–5 h | DNA/RNA | [35] | |
| SHERLOCKv2 | Cas13a/Cas13b | RPA | zmol/L | Yes | Yes | < 3 h | DNA/RNA | [55] | |
| DETECTR | Cas12a | RPA | amol/L | No | No | ~70 min | DNA | [39] | |
| HOLMES | Cas12a | PCR | amol/L | Yes | No | ~1 h | DNA/RNA | [36] | |
| HOLMESv2 | Cas12b | LAMP/RT-LAMP | amol/L | Yes | Yes | ~1 h | DNA/RNA | [56] | |
| Colorimetric biosensors | NASBACC | Cas9 | NASBA | fmol/L | Yes | No | 2–6 h | RNA | [44] |
| CASLFA | Cas9 | RPA/PCR | 150–200 copies/reaction | Yes | No | 3–4 h | DNA | [45] | |
| CRISPR/Cas12a-LFD | Cas12a | RAA | 20 copies/reaction | No | No | ~1 h | DNA | [46] | |
| SARS-CoV-2 DETECTR | Cas12a | RT-LAMP | 10 copies/reaction | No | No | ~30 min | RNA | [1] | |
| Electrochemical biosensors | CRISPR-Chip | dCas9 | No amplification | fmol/L | No | Yes | 45 min | DNA | [48] |
| E-DNA | Cas9/Cas12a | No amplification | fmol/L | Yes | Yes | < 1 h | DNA | [57] | |
| E-CRISPR | Cas12a | No amplification | pmol/L | Yes | Yes | ~1 h | DNA | [49-50] |
一些物质被紫外光照射后处于激发态,原子间会发生原子能级的跃迁,从而放射出多种可见光,使物质呈现荧光[31-32]。基于荧光产生的原理,荧光生物传感器(Fluorescent biosensors) 能够实现对病原体精准快速的检测[33]。加入特异性靶标后,整个体系会对这种刺激产生响应,并将产生的信号以荧光的形式输出,通过识别荧光信号实现对靶标的检测。作为最早应用于CRISPR/Cas体系的生物传感手段,也是目前应用最广泛的一种方法。目前基于CRISPR/Cas体系的荧光生物传感器主要是通过荧光切割探针和荧光染料这两种方式实现荧光检测。
2.1.1 基于荧光切割探针的荧光生物传感器基于荧光切割探针的这一类生物传感器主要利用Ⅴ型的Cas12和Cas14及Ⅵ型的Cas13的“附带切割”活性[28, 34]。在这一类生物传感器中使用荧光猝灭报道子(Fluorophore quencher-labeled reporter,FQ Reporter),利用单链DNA (Single- stranded DNA,ssDNA) 或单链RNA (Single- stranded RNA,ssRNA) 连接荧光基团和淬灭基团形成ssDNA-FQ或ssRNA-FQ[35-37]。在正常情况下体系无荧光,一旦体系中出现具有DNase或者RNase的活性物质时,报道子就会被切割,随即荧光基团和淬灭基团分离,释放荧光信号(图 1)。

|
| 图 1 基于荧光切割探针的荧光生物传感器 Fig. 1 Fluorescent biosensors based on fluorescent cutting probe. Schematic diagram of CRISPR/Cas12a-NER (A), SHERLOCK (B), HOLMES (C) and DETECTR (D). |
| |
这种荧光报道子的作用原理类似于qRT-PCR中使用的TaqMan荧光探针,不同的地方在于qRT-PCR中每扩增一条链,产生一个荧光分子,而基于Cas效应蛋白的“附带切割”活性,一旦被激活后切割大量荧光报道子,因而能够产生更多的荧光分子。基于此,Wang等[2]开发了CRISPR/ Cas12a-NER (CRISPR/Cas12a-based assay with a naked eye readout) 实现了对新型冠状病毒的检测,并对比了传统的qRT-PCR检测方法(图 1A)。与qRT-PCR相比[1-3, 38],CRISPR/Cas12a-NER使用RT-RAA (Reverse transcript recombinase-acid amplification) 对靶标进行扩增,简化了实验操作的复杂程度,缩短了检测所需时间,虽然检测灵敏度与qRT-PCR存在一定的差距,但已经能够达到检测要求(表 2)。作为一种新型的检测方法,CRISPR/Cas12a-NER能够在不需要精密仪器的前提下实现对新型冠状病毒的检测,有望弥补传统qRT-PCR无法在基层地区普及的现状。
| Detection method | CDC SARS-CoV-2 qRT-PCR[1] | CRISPR/Cas12a-NER[2] |
| Target | E gene | E gene |
| Sample control | RNase P | RNase P |
| Limit of detection | 3.2 copies/μL | 10 copies/μL |
| Assay reaction time | 120 min | 45 min |
| Assay components | UDG digestion (25 ℃, 2 min), reverse transcription (50 ℃, 15 min), denature (95 ℃, 2 min) amplification, (95 ℃, 3 s; 55 ℃ 30 s; 45 cycles) | RT-RAA (39 ℃, 30 min), CRISPR/Cas12a reaction and fluorescence readout (37 ℃, 15 min) |
| Bulky instrumentation required | Yes | No |
除了可实现新型冠状病毒的检测,还能够将其运用在其他病毒的检测。Gootenbery等[35]开发了一种基于LwCas13a的检测平台SHERLOCK (Specific high-sensitivity enzymatic reporter unlocking)。在SHERLOCK体系中,利用重组酶聚合酶扩增(Recombinase polymerase amplification,RPA) 或者逆转录重组酶聚合酶扩增(Reverse transcriptase-RPA,RT-RPA) 对靶标进行恒温扩增,将扩增后的产物添加到CRISPR/Cas13a体系后,crRNA会特异性识别靶标核酸序列并激发Cas13a的“附带切割”活性,随即切割体系中的ssRNA-FQ产生可被检测的荧光信号(图 1B),将病原体核酸序列转化为可测量的荧光值。相较于未采用扩增的CRISPR/Cas13a检测,使用RPA扩增极大地提高了检测的灵敏度,检测灵敏度可达amol/L并实现单碱基分辨率。利用SHERLOCK技术已成功实现了Zika病毒、Dengue病毒和致病菌的检测。此外经过验证,检测试剂经冻干可长期保存和携带,扩大了SHERLOCK的使用范围。
同样具有“附带切割”活性的Ⅴ型Cas12a也能够应用在基于CRISPR/Cas体系的荧光生物传感器中[39]。当Cas12a/crRNA结合靶标dsDNA后,CRISPR体系会显示出对任意ssDNA非特异性的切割活性[40]。Li等[36]首次报道了Cas12a的“反式切割”活性并将其运用在病原体核酸检测领域,利用LbCas12a开发了HOLMES (An one-hour low-cost multipurpose highly efficient system),该系统将CRISPR/Cas12a体系与PCR扩增技术结合,扩增后的产物添加到CRISPR/Cas12a体系中,crRNA会特异性识别dsDNA扩增子,随后激发Cas12a的非特异性切割功能,随即切割体系中的ssDNA-FQ,产生可被检测的荧光信号(图 1C)。整个检测体系可在1 h内完成对靶标DNA的检测,经过验证HOLMES可以检测amol/L的DNA病毒和RNA病毒并准确区分病毒的基因型和人的单核苷酸多态性(Single nucleotide polymorphism,SNP)。与此同时,Doudna团队[39]将CRISPR/Cas12a体系与RPA恒温扩增技术进行结合,开发出了一种能够实现amol/L灵敏度的核酸检测技术DETECTR (DNA endonuclease targeted CRISPR trans reporter)。利用这个检测体系可以在70 min内对临床样本中的人乳头瘤病毒(Human papillomavirus,HPV) 进行检测,并准确判断感染的HPV的类型(HPV16和HPV18) (图 1D)。相较于HOLMES,DETECTR使用了RPA恒温扩增替代了传统的PCR扩增,这不仅减少了对精密仪器的需求,降低了检测成本,且简化了扩增的操作步骤,提升了扩增效率。
2.1.2 基于荧光染料的荧光生物传感器基于荧光染料的生物传感器主要是利用CRISPR/Cas体系对靶标进行识别,使用荧光染料对扩增子进行标记,借助实时荧光PCR等工具实现对靶标的检测(图 2)。
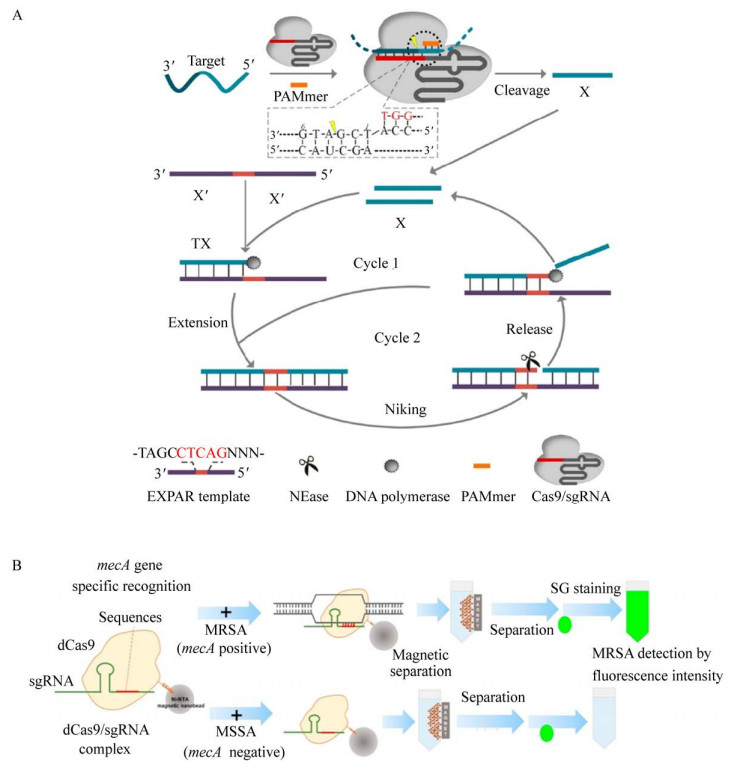
Huang等[41]利用指数式扩增反应(Exponential amplification reaction,EXPAR),将CRISPR/Cas9的切割效应与EXPAR高效扩增效应结合起来,开发了一种高效的核酸检测体系Cas-EXPAR。当体系中存在含有PAM序列的寡核苷酸(PAM-presenting oligonucleotide,PAMmer) 时,靶标ssDNA或RNA在Cas9/sgRNA的作用下在特定的位点被切割,切割后得到短片段充当恒温扩增EXPAR反应的引物,反应一段时间后生成大量DNA扩增子,采用实时荧光监测方法检测,将扩增产生的扩增子通过荧光信号的形式输出。在EXPAR强有力的扩增能力下,可将Cas-EXPAR的检测灵敏度提升至0.82 amol/L,并且能够区分切割位点的单碱基不匹配。此外,Cas-EXPAR可实现DNA甲基化和单核细胞增生李斯特氏菌mRNA的检测(图 2A)。相较于传统的核酸扩增反应,该体系并不需要添加外源引物,避免了传统扩增反应中的非特异性扩增,保证了扩增的效率,但由于检测步骤过多,所需试剂过于复杂,阻碍了Cas-EXPAR的进一步应用。
同样借助SYBR GREENⅠ荧光染料,Guk等[42]开发了基于dCas9/sgRNA-SGⅠDNA-FISH的检测方法。利用这一体系实现了对耐甲氧西林金黄色葡萄球菌(Methicillin-resistant Staphylococcus aureus,MRSA) 的检测。在这个体系中,设计识别mecA耐药基因的sgRNA序列,在添加靶标后,dCas9/sgRNA复合物能够特异性识别mecA基因并形成三元复合物,随后SYBR GREENⅠ与体系中的靶标DNA结合实现可视化检测。整个检测体系能够直接使用细胞裂解液进行检测,消除了传统核酸检测方法中DNA提取、分离和纯化的步骤,大大简化了实验步骤和检测时间,使得整个检测可在30 min内完成,并且检测最低浓度可达10 CFU/mL (图 2B)。该体系中最大的优势在于不需要添加额外的辅助扩增手段,通过dCas9/sgRNA实现对靶标核酸的富集,从而达到检测目的。Cas-EXPAR与dCas9/sgRNA-SGⅠbased DNA-FISH的最大区别在于两者使用的Cas效应蛋白的差异,前者利用了Cas9/sgRNA对靶标核酸的结合和切割功能,而dCas9/sgRNA复合物的作用类似于抗体,能够高特异性靶向结合靶标DNA,但不会对靶标进行切割。
2.2 基于CRISPR/Cas体系的比色生物传感器基于CRISRP/Cas体系的比色传感(Colorimetric biosensors) 是利用基于溶液对光的选择吸收产生的可视化颜色或者依靠光谱学仪器进行检测的一类生物传感器[43]。基于CRISPR/Cas体系的生物传感器利用光学特异性可实现快速高效的病原体检测(图 3)。相对于荧光生物传感器需要借助额外的工具来读取荧光值,基于比色分析方法的生物传感器能够凭借肉眼实现可视化检测。
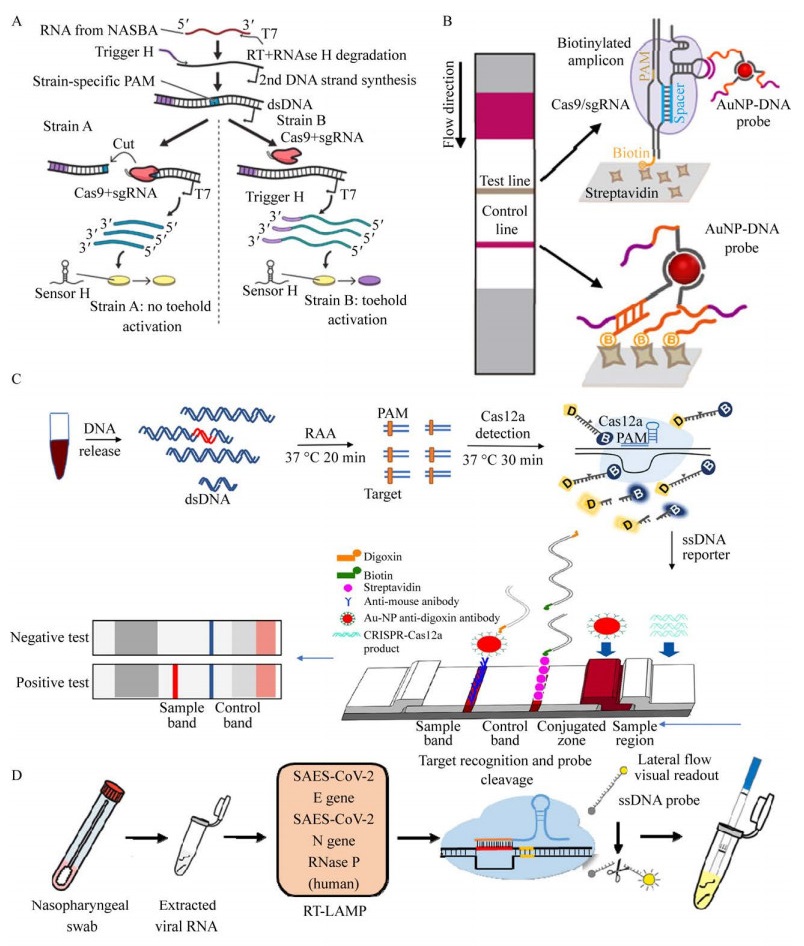
Keith等[44]首次将Cas9应用于检测病原体的比色传感器上,将核酸依赖扩增技术(Nuclear acid sequence-based amplification,NASBA) 与CRISPR/Cas9的靶标特异性识别整合在一起,建立了一种检测Zika病毒的纸基质的比色传感器NASBACC (NASBA-CRISPR cleavage)。利用NASBA对靶标RNA进行高效扩增,将扩增的中间产物dsDNA作为CRISRP/Cas9切割的底物,当dsDNA中含有切割所需的PAM序列时,dsDNA被剪切,扩增就无法继续进行;当dsDNA中不含切割所需的PAM序列时,在T7启动子的作用下,dsDNA被转录为ssRNA,随后将含有激发序列的toehold传感器添加到扩增产物中,含有靶标序列的扩增子可以打开其特殊发夹结构,通过生化反应将基因组信息转化为肉眼可见的颜色变化。经过验证该比色传感器可以检测到超低浓度的Zika病毒且能够区分不同亚型的病原体(图 3A)。作为首个使用基于Cas9的比色生物传感器,NASBASS能够在3 h内通过颜色变化读取实验结果,检出限可达fmol/L水平,并且利用了Cas9对靶标的特异性识别成功区分了美国Zika病毒与非洲Zika病毒。
虽然NASBASS展现了较高的灵敏度和特异性,但Cas9只能对靶标序列进行切割,一旦扩增后的靶标浓度过低就有可能导致颜色过浅,从而无法读取实验结果。为了解决这个问题,Wang等[45]将Cas9介导的检测技术整合至侧流层析试纸条(Lateral flow detection,LFD) 上,建立了一种新型的比色生物传感CASLFA (CRISPR/Cas9- mediated lateral flow nucleic acid assay)。在这个体系中,利用生物素标记的引物对靶标DNA进行扩增,将扩增产物添加到Cas9反应体系中后,Cas9/sgRNA会特异性识别靶标DNA并形成三元复合物。随后将反应底物滴加至侧流层析试纸条上,在毛细管力的作用下,液体不断地向前流动。当复合物流经结合垫时,AuNP-DNA结合探针会与sgRNA的环状区域结合,形成四元复合物,随后被固定在检测线的链霉亲和素捕获,过剩的AuNP-DNA会继续向前流动并被质控线上的捕获探针所捕获,由于AuNP的积累,会在检测线与质控线上产生颜色带,DNA靶标的存在或者不存在可以通过显色指示来判断(图 3B)。利用单核细胞增生李斯特氏菌、转基因35S启动子和非洲猪瘟病毒(African swine fever virus,ASFV) 对CASLFA检测方法的检测能力进行评估,结果表明检测灵敏度可达到150–200 copies/reaction,并能够从其他多种食源性致病菌中区分单核细胞增生李斯特氏菌,显示出较低的交叉反应。
相比于复杂的基于CRISPR/Cas9的比色传感器,基于CRISPR/Cas12a的比色传感在提升检测灵敏度的同时不仅减少了反应所需的复杂试剂,也降低了检测所需时间。Wang等[46]为了检测ASFV开发了一种基于CRISPR/Cas12a和LFD的一种快速、灵敏并且不需要检测仪器的新型比色生物传感CRISPR/Cas12a-LFD。使用地高辛和生物素标记的ssDNA报道子,当体系中含有ASFV靶标序列时,Cas12a的“附带切割”活性被激活并非特异性酶切ssDNA报道子,随后将切割产物滴加至试纸条上,从而在检测线产生颜色信号。整个检测可在1 h内完成,通过结合重组酶核酸扩增技术(Recombinase-acid amplification,RAA) 可将检测极限提高到2 copies/reaction的病毒基因。对149个临床样本进行检测,检测结果与RT-PCR结果一致,表明CRISPR/Cas12a-LFD具有高度准确性(图 3C)。同样利用CRISPR/Cas12a和LFD,James等[1]开发了SARS-CoV-2 DETECTR (DNA endonuclease targeted CRISPR trans reporter),通过结合环介导等温扩增(Loop-mediated isothermal amplification,LAMP) 同时进行逆转录和等温扩增,可在45 min内从呼吸道擦拭子RNA提取物中检测出10 copies/reaction的病毒基因(图 3D)。对比NASBACC和CASLFA,CRISPR/ Cas12a-LFD和SARS-CoV-2 DETECTR减少了所需的复杂试剂,同时也简化了实验操作,提升了检测效率。
2.3 基于CRISPR/Cas体系的电化学生物传感器电化学生物传感器(Electrochemical biosensors) 是利用生物体成分或者生物体本身作为感受元件,电极作为转换元件,通过电势或者电流作为信号输出的一类生物传感器[30, 47]。近年来,基于CRISPR/Cas体系的电化学生物传感器由于其高灵敏度、检测耗时短、仪器的便携性受到广泛关注(图 4)。这类传感器主要是将病原体的基因组信息转化为电信号,通过建立电信号与靶标浓度的关系,可实现靶标的定量检测。
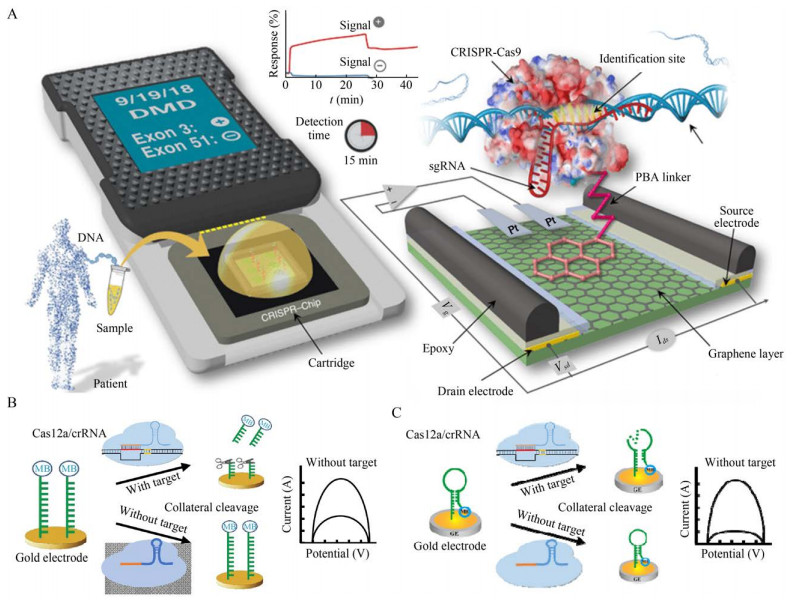
Reza等[48]将CRISPR/dCas9特异性靶标识别和石墨烯场效应晶体管(Graphene-based field-effect transistor,gFET) 的高灵敏度检测结合起来,建立了一种可在短时间内检测出特定基因突变的新型手持电化学生物传感器CRISPR-Chip。在这项研究中,将dCas9连接到由石墨烯制成的晶体管上,当dCas9/sgRNA复合物在其靶向的DNA上找到位点时,它会与其结合并触发石墨烯的电导率发生变化,从而改变了晶体管的电特性。作为一种便携式的电化学生物传感器,在不需要扩增的前提下能够在15 min内完成检测并且灵敏度可达1.7 fmol/L,且通过设计不同的sgRNA可实现对不同靶标的检测(图 4A)。与传统的病原体核酸检测不同的地方在于,它不需要对靶标片段进行复制扩增,可直接使用DNA提取物在野外工作环境下进行基因检测,这大大扩展了其检测的适用范围。
除了利用dCas9的靶向结合功能建立的电化学生物传感器,还可以利用Cas效应蛋白的“附带切割”活性建立超灵敏的电化学传感器。Dai等[49]建立了一种基于Cas12a的“附带切割”生物传感器E-CRISPR,利用这种新型的电化学生物传感器可实现超低浓度病原体的检测。在这个体系中,利用特异性修饰的ssDNA作为电化学报道子,这种报道子的3′端固定亚甲基蓝(Methylene blue,MB) 作为还原剂标记,5′端固定了硫醇键以便将报道子固定在金电极上。当体系内存在靶标时,Cas12a的非特异性ssDNA切割活性就会被激活,切割MB-ssDNA报道子使得MB从金电极表面分离,导致MB信号转换降低;在没有靶标存在的情况下,Cas12a的反式切割活性被抑制,保留了金电极表面的MB-ssDNA。可通过电信号判断靶标DNA的存在。经验证,E-CRISPR在不对靶标进行扩增的情况下,可检测到50 pmol/L浓度的HPV16。E-CRISPR除了适配Ⅴ型的CRISPR体系,和可用于Ⅲ和Ⅵ型的CRISPR体系(图 4B)。
不久后,Zhang等[50]基于E-CRISPR的检测原理,使用一种发夹状的DNA报道子代替了原有体系中的线性ssDNA报道子(图 4C)。这种发夹状的DNA电化学报道子不仅可以使得MB更接近电极,降低电子转化效率,还因其具有一个较低的表面覆盖率和疏松的形态结构,提高了Cas12a的切割效率,大大提升了检测的灵敏度。通过优化体系条件后,该体系可以在60 min内检测到30 pmol/L的靶标DNA。
此类基于电化学的生物传感器不需要额外的辅助扩增方法,通过添加靶标后分析体系中电信号的改变,就可以对靶标进行定量检测,减少了反应所需要的复杂试剂,降低了成本。
2.4 基于CRISPR/Cas体系的其他类型的生物传感器除了基于荧光、比色和电化学的生物传感器,还能将CRISPR/Cas体系应用在基于微流控芯片的生物传感器(图 5)。
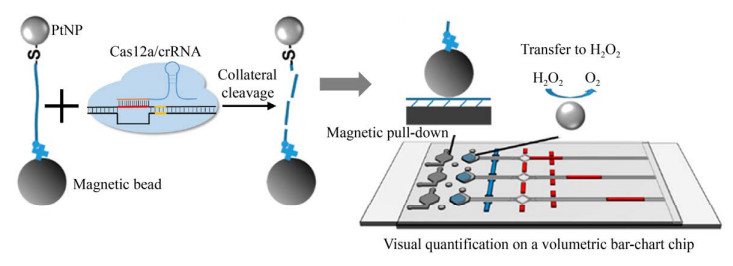
Ning等[51]基于之前开发的气动芯片(Volumetric bar-chart chip,V-chip) 检测平台,开发了一种磁介导的V-chip (Magnet-assisted V-chip, MAV-chip),结合金属铂报道子和CRISPR-Cas12a实现病原体核酸的可视化定量检测。在这个MAV-chip检测体系中,磁珠-单链DNA-金属铂纳米粒子(Magnetic bead-single-stranded DNA-PtNP,BDNP) 共轭化合物作为一种报道子添加到CRISPR-Cas12a体系中。一旦Cas12a/crRNA识别靶标DNA后,激发Cas12a非特异性切割ssDNA的活性,BDNPs中的ssDNA连接子就会被降解,从而金属铂从磁珠上被释放,释放的铂就会催化H2O2产生氧气,气体推动红墨水向前移动,从而直观可视化定量地检测靶标(图 5)。相较于荧光、比色和电化学生物传感器需要借助特定的仪器实现定量检测,这种新型的基于铂纳米报道子的MAV-chip不需要额外的仪器,仅凭肉眼就可实现定量检测,大大降低实验成本,提高了检测效率。
3 总结与展望随着COVID-19疫情暴发,已经开发和采用了许多检测方法,包括qRT-PCR、基因测序和等温扩增等。而基于CRISPR/Cas的新型生物传感器作为一种新型的检测方法,能够对新型冠状病毒进行精准高效的检测,引起了人们的广泛关注[19]。对比传统的检测方法,基于CRISPR/Cas的此类新型生物传感器并不需要昂贵的精密仪器,大大降低了检测成本;通过与恒温扩增技术相结合,提高了灵敏度;可与多种技术相结合,实现可视化检测;简化了实验步骤,缩短检测所需时间,提高了检测效率。
虽然基于CRISPR/Cas技术所建立的生物传感器在病原体检测上取得了巨大的突破,但在病原体核酸检测方面的应用仍然存在许多障碍和局限性:(1) 就很多已建立的基于CRISPR/Cas体系的生物传感器而言,都需要一个初始扩增方法去扩增病原体靶标序列,而CRISPR效应蛋白只是用来实现终点检测,换言之,这些生物传感器的灵敏度过度依赖初始扩增,CRISPR/Cas系统原有的这种超灵敏和严格的PAM识别却没有被完全利用。未来的工作应该放在如何充分利用Cas效应蛋白的“附带切割”活性,在保证检测灵敏度的同时,减少检测中的预扩增步骤。(2) sgRNA对靶标的识别依赖于PAM序列,这种序列限制致使可检测的序列范围缩小。虽然增加了检测体系的特异性,但同时也降低了sgRNA靶标选择和设计的灵活性。通过设计特异性的引物,可在扩增靶标时引入PAM序列,在一定程度上扩大可检范围。(3) RNA易被环境中的RNase降解,容易导致假阳性。通过设计集成的一步法检测体系,可大大避免外部环境中的RNase对实验结果产生的影响。(4) 试剂稳定性较差,不易保存。目前检测试剂可以通过冷冻干燥的形式进行保存,但由于检测组分比目前PCR体系更为复杂,如何将所有试剂集成还是一个需要创新解决的问题。
笔者课题组致力于HOLMES技术的开发和应用,旨在利用CRISPR/Cas12a实现对食源性致病菌的检测,目前在单核细胞增生李斯特菌和鼠伤寒沙门氏菌检测体系优化等方面进行了深入研究,以期能够为食源性致病菌的快速检测提供一份新的解决方案。
未来的工作需要将CRISPR检测体系集成在便携式的POCT生物传感器上,以溶液或者冻干的形式,将所需的所有试剂预装到检测装置中。同时,需要基于实验室的定量分析设备,例如基于微流控芯片的定量测试设备,在一个芯片上可同时进行同靶标不同样本或不同靶标同一样本的检测。利用CRISPR/Cas对靶标核酸的精准识别和高效的靶向切割活性,基于CRISPR/Cas体系的生物传感器在病原体核酸检测方面显示出了优良的性能,较传统的病原体检测方法而言,其拥有较高的检测灵敏度和准确性。但该技术正处于起步阶段,全面超越传统的检测方法仍存在一定距离。随着研究的不断深入有望开发出更多类型的CRISPR/Cas体系,并将它们应用在POCT中。
| [1] |
Broughton JP, Deng XD, Yu GX, et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol, 2020, 38(7): 870-874. DOI:10.1038/s41587-020-0513-4
|
| [2] |
Wang XJ, Zhong MT, Liu Y, et al. Rapid and sensitive detection of COVID-19 using CRISPR/Cas12a-based detection with naked eye readout, CRISPR/Cas12a-NER. Sci Bull, 2020, 65(17): 1436-1439. DOI:10.1016/j.scib.2020.04.041
|
| [3] |
Smyrlaki I, Ekman M, Lentini A, et al. Massive and rapid COVID-19 testing is feasible by extraction-free SARS-CoV-2 RT-PCR. Nat Commun, 2020, 11: 4812. DOI:10.1038/s41467-020-18611-5
|
| [4] |
Cho IH, Irudayaraj J. Lateral-flow enzyme immunoconcentration for rapid detection of Listeria monocytogenes. Anal Bioanal Chem, 2013, 405(10): 3313-3319. DOI:10.1007/s00216-013-6742-3
|
| [5] |
Sblattero D, Berti I, Trevisiol C, et al. Human recombinant tissue transglutaminase ELISA: an innovative diagnostic assay for celiac disease. Am J Gastroenterol, 2000, 95(5): 1253-1257. DOI:10.1111/j.1572-0241.2000.02018.x
|
| [6] |
Lequin RM. Enzyme immunoassay (EIA)/enzyme-linked immunosorbent assay (ELISA). Clin Chem, 2005, 51(12): 2415-2418. DOI:10.1373/clinchem.2005.051532
|
| [7] |
Li ZJ, Zhang XZ, Hu XL, et al. Development of an indirect ELISA assay for detecting antibodies against mammalian reovirus in pigs. J Virol Methods, 2018, 262: 61-64. DOI:10.1016/j.jviromet.2018.07.008
|
| [8] |
Geraldi A, Giri-Rachman EA. Synthetic biology-based portable in vitro diagnostic platforms. Alexand J Med, 2018, 54(4): 423-428. DOI:10.1016/j.ajme.2018.11.003
|
| [9] |
Ho SN, Hunt HD, Horton RM, et al. Site-directed mutagenesis by overlap extension using the polymerase chain reaction. Gene, 1989, 77(1): 51-59. DOI:10.1016/0378-1119(89)90358-2
|
| [10] |
Pang Y, Xia H, Zhang ZY, et al. Multicenter evaluation of genechip for detection of multidrug-resistant Mycobacterium tuberculosis. J Clin Microbiol, 2013, 51(6): 1707-1713. DOI:10.1128/JCM.03436-12
|
| [11] |
Culbreath K, Melanson S, Gale J, et al. Validation and retrospective clinical evaluation of a quantitative 16S rRNA gene metagenomic sequencing assay for bacterial pathogen detection in body fluids. J Mol Diagnost, 2019, 21(5): 913-923. DOI:10.1016/j.jmoldx.2019.05.002
|
| [12] |
Stals A, Van Coillie E, Uyttendaele M. Viral genes everywhere: public health implications of PCR-based testing of foods. Curr Opin Virol, 2013, 3(1): 69-73. DOI:10.1016/j.coviro.2012.11.003
|
| [13] |
Khwannimit B, Bhurayanontachai R. The direct costs of intensive care management and risk factors for financial burden of patients with severe sepsis and septic shock. J Crit Care, 2015, 30(5): 929-934. DOI:10.1016/j.jcrc.2015.05.011
|
| [14] |
Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes: zinc finger fusions to Fok Ⅰ cleavage domain. Proc Natl Acad Sci USA, 1996, 93(3): 1156-1160. DOI:10.1073/pnas.93.3.1156
|
| [15] |
Christian M, Cermak T, Doyle EL, et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics, 2010, 186(2): 757-761. DOI:10.1534/genetics.110.120717
|
| [16] |
Zhang J, Zong WM, Hong W, et al. Exploiting endogenous CRISPR-Cas system for multiplex genome editing in Clostridium tyrobutyricum and engineer the strain for high-level butanol production. Metabol Eng, 2018, 47: 49-59. DOI:10.1016/j.ymben.2018.03.007
|
| [17] |
Chen SY, Liu Y, Liang WJ, et al. Insertion sequences in the CRISPR-Cas system regulate horizontal antimicrobial resistance gene transfer in Shigella strains. Int J Antimicrob Agents, 2019, 53(2): 109-115. DOI:10.1016/j.ijantimicag.2018.09.020
|
| [18] |
Manglekar RR, Geng AL. CRISPR-Cas9-mediated seb1 disruption in Talaromyces pinophilus EMU for its enhanced cellulase production. Enzym Microb Technol, 2020, 140: 109646. DOI:10.1016/j.enzmictec.2020.109646
|
| [19] |
Wang M, Zhang R, Li JM. CRISPR/Cas systems redefine nucleic acid detection: principles and methods. Biosens Bioelectr, 2020, 165: 112430. DOI:10.1016/j.bios.2020.112430
|
| [20] |
Aman R, Mahas A, Mahfouz M. Nucleic acid detection using CRISPR/Cas biosensing technologies. ACS Synth Biol, 2020, 9(6): 1226-1233. DOI:10.1021/acssynbio.9b00507
|
| [21] |
Van Dongen JE, Berendsen JTW, Steenbergen RDM, et al. Point-of-care CRISPR/Cas nucleic acid detection: recent advances, challenges and opportunities. Biosens Bioelectr, 2020, 166: 112445. DOI:10.1016/j.bios.2020.112445
|
| [22] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829
|
| [23] |
Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140
|
| [24] |
Hsu DP, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010
|
| [25] |
Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096. DOI:10.1126/science.1258096
|
| [26] |
Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc Natl Acad Sci USA, 2012, 109(39): E2579-E2586. DOI:10.1073/pnas.1208507109
|
| [27] |
East-Seletsky A, O'Connell MR, Knight SC, et al. Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection. Nature, 2016, 538(7624): 270-273. DOI:10.1038/nature19802
|
| [28] |
Abudayyeh OO, Gootenberg JS, Konermann S, et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science, 2016, 353(6299): aaf5573. DOI:10.1126/science.aaf5573
|
| [29] |
Parmin NA, Hashim U, Gopinath SCB, et al. Human Papillomavirus E6 biosensing: current progression on early detection strategies for cervical cancer. Int J Biol Macromol, 2019, 126: 877-890. DOI:10.1016/j.ijbiomac.2018.12.235
|
| [30] |
Cesewski E, Johnson BN. Electrochemical biosensors for pathogen detection. Biosens Bioelectr, 2020, 159: 112214. DOI:10.1016/j.bios.2020.112214
|
| [31] |
Sun JD, Ji J, Sun YQ, et al. DNA biosensor-based on fluorescence detection of E. coli O157:H7 by Au@Ag nanorods. Biosens Bioelectr, 2015, 70: 239-245. DOI:10.1016/j.bios.2015.03.009
|
| [32] |
Ma Q, Li P, Gao ZQ, et al. Rapid, sensitive and highly specific label-free fluorescence biosensor for microRNA by branched rolling circle amplification. Sens Actuat B: Chem, 2019, 281: 424-431. DOI:10.1016/j.snb.2018.10.141
|
| [33] |
Li XH, Zhang XL, Wu J, et al. Hyperbranched rolling circle amplification (HRCA)-based fluorescence biosensor for ultrasensitive and specific detection of single-nucleotide polymorphism genotyping associated with the therapy of chronic hepatitis B virus infection. Talanta, 2019, 191: 277-282. DOI:10.1016/j.talanta.2018.08.064
|
| [34] |
Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038
|
| [35] |
Gootenberg JS, Abudayyeh OO, Lee JW, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science, 2017, 356(6336): 438-442. DOI:10.1126/science.aam9321
|
| [36] |
Li SY, Cheng QX, Wang JM, et al. CRISPR-Cas12a-assisted nucleic acid detection. Cell Discov, 2018, 4: 20.
|
| [37] |
Harrington LB, Burstein D, Chen JS, et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science, 2018, 362(6416): 839-842. DOI:10.1126/science.aav4294
|
| [38] |
Park M, Won J, Choi BY, et al. Optimization of primer sets and detection protocols for SARS-CoV-2 of coronavirus disease 2019 (COVID-19) using PCR and real-time PCR. Exp Mol Med, 2020, 52(6): 963-977. DOI:10.1038/s12276-020-0452-7
|
| [39] |
Chen JS, Ma EB, Harrington LB, et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science, 2018, 360(6387): 436-439. DOI:10.1126/science.aar6245
|
| [40] |
Li SY, Cheng QX, Liu JK, et al. CRISPR-Cas12a has both cis-and trans-cleavage activities on single-stranded DNA. Cell Res, 2018, 28(4): 491-493. DOI:10.1038/s41422-018-0022-x
|
| [41] |
Huang MQ, Zhou XM, Wang HY, et al. Clustered regularly interspaced short palindromic repeats/Cas9 triggered isothermal amplification for site-specific nucleic acid detection. Anal Chem, 2018, 90(3): 2193-2200. DOI:10.1021/acs.analchem.7b04542
|
| [42] |
Guk K, Keem JO, Hwang SG, et al. A facile, rapid and sensitive detection of MRSA using a CRISPR-mediated DNA FISH method, antibody-like dCas9/sgRNA complex. Biosens Bioelectr, 2017, 95: 67-71. DOI:10.1016/j.bios.2017.04.016
|
| [43] |
Suaifan GARY, Alhogail S, Zourob M. Rapid and low-cost biosensor for the detection of Staphylococcus aureus. Biosens Bioelectr, 2017, 90: 230-237. DOI:10.1016/j.bios.2016.11.047
|
| [44] |
Pardee K, Green AA, Takahashi MK, et al. Rapid, low-cost detection of Zika virus using programmable biomolecular components. Cell, 2016, 165(5): 1255-1266. DOI:10.1016/j.cell.2016.04.059
|
| [45] |
Wang XS, Xiong EH, Tian T, et al. Clustered regularly interspaced short palindromic repeats/Cas9-mediated lateral flow nucleic acid assay. ACS Nano, 2020, 14(2): 2497-2508. DOI:10.1021/acsnano.0c00022
|
| [46] |
Wang XJ, Ji PP, Fan HY, et al. CRISPR/Cas12a technology combined with immunochromatographic strips for portable detection of African swine fever virus. Commun Biol, 2020, 3: 62. DOI:10.1038/s42003-020-0796-5
|
| [47] |
Low KF, Zain ZM, Yean CY. A signal-amplified electrochemical DNA biosensor incorporated with a colorimetric internal control for Vibrio cholerae detection using shelf-ready reagents. Biosens Bioelectr, 2017, 87: 256-263. DOI:10.1016/j.bios.2016.08.064
|
| [48] |
Hajian R, Balderston S, Tran T, et al. Detection of unamplified target genes via CRISPR-Cas9 immobilized on a graphene field-effect transistor. Nat Biomed Eng, 2019, 3(6): 427-437. DOI:10.1038/s41551-019-0371-x
|
| [49] |
Dai YF, Somoza RA, Wang L, et al. Exploring the trans-cleavage activity of CRISPR-Cas12a (cpf1) for the development of a universal electrochemical biosensor. Angew Chem Int Ed, 2019, 58(48): 17399-17405. DOI:10.1002/anie.201910772
|
| [50] |
Zhang DC, Yan YR, Que HY, et al. CRISPR/Cas12a-mediated interfacial cleaving of hairpin DNA reporter for electrochemical nucleic acid sensing. ACS Sens, 2020, 5(2): 557-562. DOI:10.1021/acssensors.9b02461
|
| [51] |
Shao N, Han X, Song YN, et al. CRISPR-Cas12a coupled with platinum nanoreporter for visual quantification of SNVs on a volumetric bar-chart chip. Anal Chem, 2019, 91(19): 12384-12391. DOI:10.1021/acs.analchem.9b02925
|
| [52] |
Zhang YH, Qian L, Wei WJ, et al. Paired design of dCas9 as a systematic platform for the detection of featured nucleic acid sequences in pathogenic strains. ACS Synth Biol, 2017, 6(2): 211-216. DOI:10.1021/acssynbio.6b00215
|
| [53] |
Zhou WH, Hu L, Ying LM, et al. A CRISPR-Cas9-triggered strand displacement amplification method for ultrasensitive DNA detection. Nat Commun, 2018, 9(1): 5012. DOI:10.1038/s41467-018-07324-5
|
| [54] |
Wang T, Liu Y, Sun HH, et al. An RNA-guided Cas9 nickase-based method for universal isothermal DNA amplification. Angewand Chem Int Ed, 2019, 58(16): 5382-5386. DOI:10.1002/anie.201901292
|
| [55] |
Gootenberg JS, Abudayyeh OO, Kellner MJ, et al. Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6. Science, 2018, 360(6387): 439-444. DOI:10.1126/science.aaq0179
|
| [56] |
Li LX, Li SY, Wu N, et al. HOLMESv2: a CRISPR-Cas12b-assisted platform for nucleic acid detection and DNA methylation quantitation. ACS Synth Biol, 2019, 8(10): 2228-2237. DOI:10.1021/acssynbio.9b00209
|
| [57] |
Xu W, Jin T, Dai YF, et al. Surpassing the detection limit and accuracy of the electrochemical DNA sensor through the application of CRISPR-Cas systems. Biosens Bioelectr, 2020, 155: 112100. DOI:10.1016/j.bios.2020.112100
|
 2021, Vol. 37
2021, Vol. 37