中国科学院微生物研究所、中国微生物学会主办
文章信息
- 俞东红, 曹华, 王心睿
- Yu Donghong, Cao Hua, Wang Xinrui
- 类器官的研究进展及应用
- Advances and applications of organoids: a review
- 生物工程学报, 2021, 37(11): 3961-3974
- Chinese Journal of Biotechnology, 2021, 37(11): 3961-3974
- 10.13345/j.cjb.200764
-
文章历史
- Received: November 30, 2020
- Accepted: March 11, 2021
- Published: April 12, 2021
2. 福建医科大学附属福建省妇幼保健院医学研究中心,福建 福州 350001;
3. 福建省妇儿重大疾病研究重点实验室,福建 福州 350001;
4. 国家卫健委非人灵长类生育调节技术评价重点实验室,福建 福州 350013
2. Medical Research Center, Fujian Maternity and Child Health Hospital, Affiliated Hospital of Fujian Medical University, Fuzhou 350001, Fujian, China;
3. Fujian Key Laboratory of Women and Children's Critical Disease Research, Fuzhou 350001, Fujian, China;
4. Key Laboratory of Technical Evaluation of Fertility Regulation of Non Human Primate, National Health Commission, Fuzhou 350013, Fujian, China
在人体胚胎早期发育过程中,多能干细胞受到命运决定因子的调控,从而分化为多种细胞类型,随后这些细胞形成复杂的结构,并发展成具有特定生理功能的人体器官。几个世纪以来,科学家们一直在探究决定细胞定型和分化为特定器官的分子线索[1],而这个过程必须借助于许多模型系统作为研究工具。因此,不断开发和利用新的模型成为当前热点问题之一。
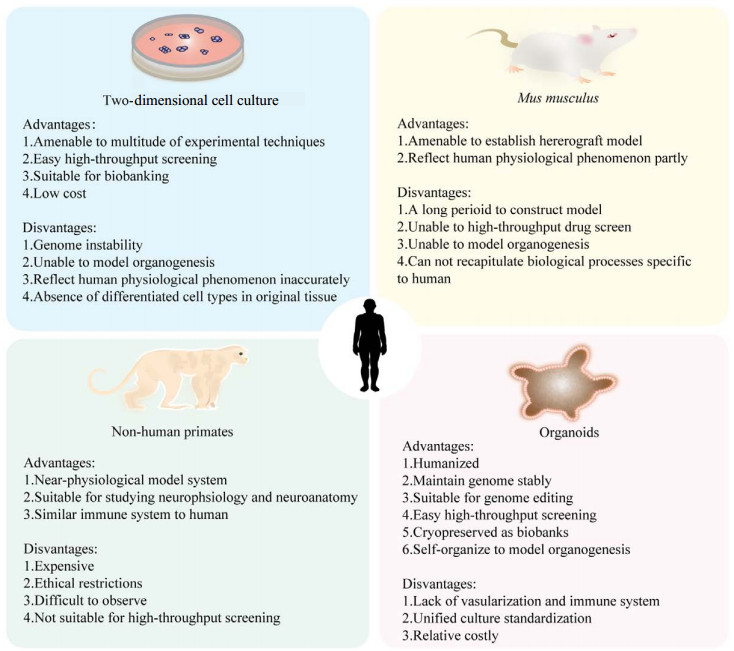
目前,最常见的模型是细胞系、酵母、秀丽隐杆线虫、黑腹果蝇、普通小鼠和斑马鱼,以及异种移植模型(Patient-derived xenograft,PDX) 等,已被广泛用于研究细胞信号通路、识别潜在的药物靶点、开发新药等方面,但也暴露出许多局限性[2]。2D (Two-dimensional) 细胞系虽操作简便,但通常只包含单一细胞类型,例如肿瘤细胞系,缺乏原始肿瘤的异质性并且在体外培养中基因组不稳定[2-3]。小鼠模型是大部分实验室最常用的研究模型,但它和人类特有的生物现象存在固有差异,如大脑发育、新陈代谢和药物疗效等[4]。非人灵长类(Non-human primate,NHP) 虽然在系统进化树上和人类最接近,具有高度相似的免疫系统、大脑结构和认知功能[5-7],但是价格昂贵,同时存在伦理限制、成像观察的局限性、不可控的复杂变量、难以进行药物高通量筛选(High-throughput screening) 等缺陷,限制其广泛应用。因此,根据“3R”原则(Reduce,reuse,recycle) 和物种保护原则,需要探索能够缩小与人体的差异又兼具稳定、易操作、可广泛应用的模型替代系统,来研究遗传多样性、疾病发病机制和预测药物反应。
1987年有研究者表明,乳腺上皮细胞在肿瘤细胞外基质上培养能够自组织形成乳腺导管、小管等3D结构[8]。2009年Sato等[9]利用含有表皮细胞生长因子(Epidermal growth factor,EGF)、Noggin、R-spondin的三维基质凝胶(Matrigel) 培养单个富含Lgr5 (Leucine rich repeat containing G protein-coupled receptor 5) 标记的小鼠肠干细胞,能够形成具有隐窝和绒毛样结构的小肠类器官,开创了类器官培养的时代。2014年Lancaster等[10]系统地提出了类器官的概念,即干细胞或器官祖细胞在体外培养中,能够发生与体内相似的细胞分化和谱系定向过程,并自组织形成具有人体器官部分的空间结构和特定功能的3D结构。随后越来越多组织来源的类器官,如肠[9]、视网膜[11]、大脑[12]、肾[13]等不断涌现,被Nature杂志评选为2017年生命科学领域的最佳技术。
与传统模型相比,类器官具有以下特性:(1) 自组装形成3D结构,在外源信号的调节作用下,多向分化并自我构建成类似其来源的组织或者器官,模拟体内器官的发育过程[10];(2) 具备器官的部分特定功能,如肺的呼吸功能、肠道的营养吸收、肾脏的滤过功能等[10];(3) 来源广泛,可大量扩增并保持基因组的稳定性[14],可基因编辑操作[15]和高通量筛选[16]。如图 1所示,根据类器官模型的特点,与传统模型互补应用而非取代,将为人类生物学研究开辟新的途径。下面围绕类器官模型当前的研究进展进行综述,并结合先进的技术,探讨类器官的发展前景及面临的挑战。
根据细胞来源的不同,类器官分为成体干细胞(Adult stem cell,ASC)、多能干细胞(Pluripotent stem cell,PSC) 或肿瘤类器官(Patient-derived organoid,POD),而PSC又分为胚胎干细胞(Embryonic stem cell,ESC) 和诱导多能干细胞(Induced pluripotent stem cell, iPSC)。不同来源的类器官具有不同的特性及用途,例如神经外胚层如视杯[18]、大脑[19]类器官仅来源于PSC,因而研究精神遗传疾病、发育生物学主要利用PSC类器官;而ASC来源于组织中具有再生能力的前体细胞,主要应用于研究成体组织生物学、组织再生和精准医疗[20-21]。
1.1 ESC和iPSC来源的类器官人类多能干细胞具有无限自我更新并分化为几乎所有器官特异性的细胞类型,其中胚胎干细胞ESC是来源于囊胚期内细胞团的全能干细胞,因涉及伦理问题使用受限[22]。与ESC相比,iPSC类器官的形成需要先将体细胞(通常是皮肤或者成纤维细胞) 重编程为具有多能干性的PSC[23],随后暴露于调控胚层发育和组织特异性定型因子中,激活或抑制关键信号通路以形成3D类器官[2]。PSC类器官形成过程如图 2所示,对于内胚层衍生的类器官,例如肠道类器官,转化生长因子(Transforming growth factor-β,TGF-β) 信号刺激PSC形成定形内胚层,然后成纤维细胞生长因子4 (Fibroblast growth factor 4,FGF4) 和Wnt3A (Wingless-type MMTV integration site family member 3A)以时间和剂量依赖的方式协同作用诱导后肠内皮层的形成[24]。对于外胚层衍生的类器官,如大脑类器官,由于人类ESC更类似于外胚层细胞,因此在没有外源诱导剂的情况下,ESC也可自主神经分化[25-26]。iPSC技术的出现,在干细胞领域具有里程碑式的意义,实现了人体特定类型细胞的无限获取,如神经元、心肌细胞和胰腺β细胞[27],为人类疾病建模和药物筛选等提供丰富资源。此外,PSC类器官形成过程遵循体内胚胎发育的命运轨迹,因而可用于重现组织或器官时空上的发育过程和内在变化[3, 28],对于大脑或肾小球等无再生能力的器官,是研究发育过程和遗传疾病很好的模型。2008年Eiraku等[26]率先用无血清培养胚状体样聚集物(Serum-free culture of embryoid body-like aggregates,SFEBq) 的方法将人类ESC自组织形成端脑类器官,在此基础上Lancaster等[12]使用旋转生物反应器,形成类似于胚胎9–10周的大脑皮质结构的类器官,反映人类大脑发育初期的部分特征。自2011年起美国NHLB1下一代基因研究联盟协会(The NextGen Genetic Association Studies Consortium) 开始收集和分析患者来源的iPSC,包括单基因病、多基因病,涉及肝、肺、心脏、血液等,并建立iPSC生物库(Biobank) 进行多项遗传病的队列研究[29]。未来,伴随类器官和基因工程技术的飞速发展,利用健康人体或者患者来源的iPSC类器官建立生物库,有望在疾病机制研究、药物潜在靶点分析等方面发挥越来越重要的作用。
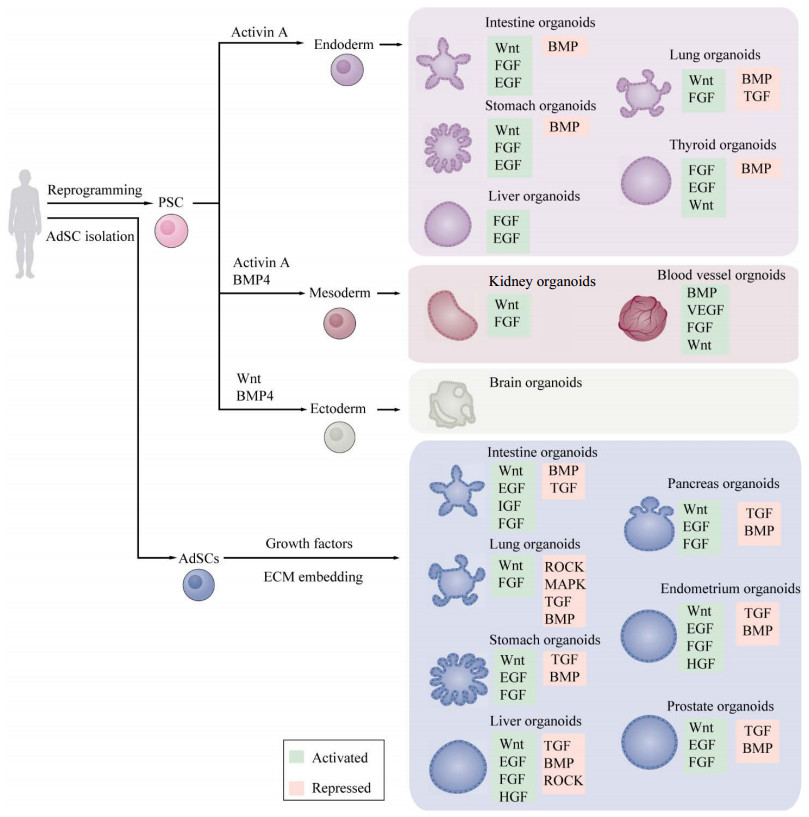
|
| 图 2 人PSC来源类器官和ASC来源类器官的形成过程[2] Fig. 2 Process for the establishment of human PSC-derived and ASC-derived organoids[2]. PSC-derived organoids are established following directed differentiation of PSCs, which requires germ-layer specification (endoderm, mesoderm or ectoderm), followed by induction, maturation, and culturing with specific growth and signaling factors to obtain the specific cell types that form the desired organ. Some PSC-derived organoids may contain cells from multiple germ layers to closely mimic the in vivo counterpart. ASC-derived organoid cultures require isolation of the tissue-specific stem cell population, which can then be embedded into an extracellular matrix (ECM) with defined, tissue-specific combinations of growth factors to allow propagation. ASC-derived organoids, as shown here, are of epithelial origin and lack a mesenchymal or immune component unless it is added separately. Signaling components that are important for guided differentiation and niche function are shown; activated signaling pathways are shown in green, and inhibited ones in red. BMP, bone morphogenetic protein; HGF, hepatocyte growth factor; IGF, insulin-like growth factor; ROCK, RHO-associated protein kinase; VEGF, vascular endothelial growth factor[2]. |
| |
成体组织干细胞ASC不仅在维持组织内稳态中发挥重要作用,而且在组织再生尤其是肝损伤后再生修复中的临床价值也备受瞩目[21]。与PSC具有多向分化的潜能相比,ASC通常来源于患者的活检组织,其分化能力十分有限,而且ASC类器官一般仅含有器官的上皮部分,缺乏基质、神经和血管系统[3],培养体系相对比较简单[2]。研究者们发现,Wnt信号通路的靶蛋白Lgr5是胃[30]、小肠、结肠[31]、肝脏[32]等许多ASC的标志物,基于此第一个小鼠肠道类器官培养系统成功建立,即R-Spondin培养法,包含EGF、WNT激活因子R-Spondin、BMP的抑制因子Noggin以及TGF-β等[9]。然而该培养体系并不能直接适用于人体类器官培养,Sato等[33]通过额外添加Wnt3A,以及胃泌素、FGF10、TGF-β、丝裂原活化蛋白激酶(Mitogen-activated protein kinase,MAPK) 抑制剂和烟酰胺等,首次从人体结肠和结肠腺瘤、巴雷特食管(Barrett esophagus,BE) 中建立人ASC类器官,这使得人体组织成体干细胞应用于组织再生和修复成为可能,在干细胞培养领域具有非常深远的意义。
历经10年的优化和条件摸索,如今已相继培养出一系列ASC类器官,包括几乎所有消化系统的类器官(肝脏[32]、胰腺[34]、结直肠、胃[30]、胆囊[35]等),以及部分非消化系统的类器官(前列腺[36]、乳腺[37]、肺[38]等)。研究者们发现,成功建立ASC类器官的前提是必须充分认识体内调控干细胞生态位因子和信号通路,培养体系中各因子细微的变化,都可能产生不同的培养结果。例如,人类Lgr5+肝干细胞为双潜能干细胞,在类器官标准培养条件下只生成导管上皮类器官,但在培养基中添加Notch抑制剂、FGF19、BMP7和地塞米松的条件下,具有生成成熟肝细胞的潜能[39]。此外,在肠道培养基中添加各种Wnt调节因子(CHIR99021,GSK3β抑制剂) 和Notch信号通路调节因子(丙戊酸,组蛋白去乙酰化酶抑制剂) 有助于丰富和维持干细胞的数量,同时促进其向成熟肠上皮细胞、杯状细胞分化[40]。目前人类对于ASC的认识还十分有限,随着体内ASC调控机制的深入研究,ASC类器官也许能够为组织再生和移植提供新的可能。
1.3 肿瘤来源的类器官当前肿瘤的主要研究模型仍然是肿瘤细胞系(Patient-derived cancer cell lines,PDC) 和PDX模型,但由于建立效率低、不能复制体内肿瘤异质性、基因组不稳定、培养周期长等原因,导致临床转化率极低。2011年Sato等[33]从结肠腺瘤、腺癌及BE中建立肿瘤类器官模型,开启了肿瘤个性化治疗的新纪元。研究们者们发现肿瘤类器官不仅能够实现和患者匹配,再现原始肿瘤的病理学特征、基因拷贝数变异和突变图谱[41-42],而且其培养高效,只需要非常小的活检组织,在短时间内(4–6周) 构建效率即可达30%,甚至大于90%[41-43],有望为癌症患者在时间窗内提供诊疗方案。Gao等[44]已成功地从前列腺癌液体活检组织及少量肿瘤循环细胞中长期培养前列腺癌类器官,弥补活检技术中难以富集少量循环肿瘤细胞的缺陷,为癌症个体化诊疗创造了新机会。Yao等[45]证明直肠癌类器官对放化疗的反应与临床上患者对药物的反应高度匹配,准确度为84.43%,灵敏度为78.01%,特异性为91.97%,这一体内外平行实验很有力地证实了来源于癌症患者的肿瘤类器官可有效地预测患者临床治疗结局。此外,从北美临床试验注册中心(ClinicalTrials.gov) 数据可见,截至2020年12月关于类器官相关的69项临床研究中,与肿瘤类器官相关的就有55项,如图 3所示,从乳腺癌、肺癌、前列腺癌、食管癌、直肠癌等建立肿瘤类器官生物库,随后进行抗癌药物筛选、药物毒性分析、寻找潜在治疗靶点已经成为肿瘤类器官研究的热点领域[41, 46-47]。
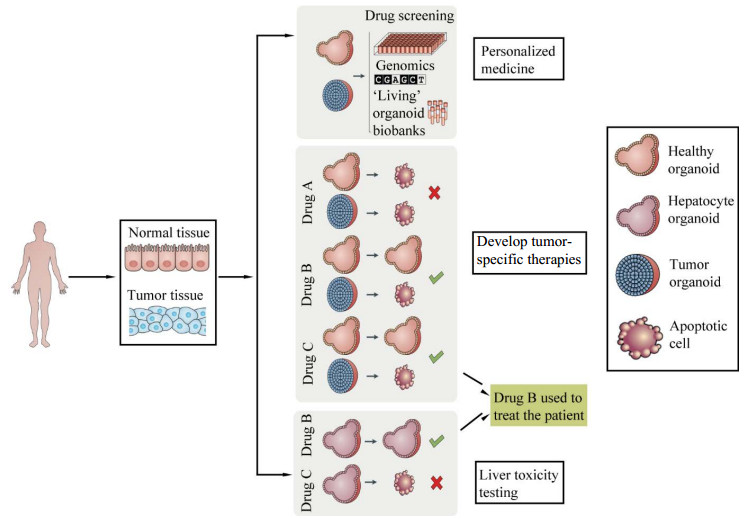
|
| 图 3 类器官在癌症个性化治疗和药物开发中的作用[48] Fig. 3 Organoid cultures for personalized cancer treatment and drug development[48]. Organoids can be established from patient-derived healthy and tumor tissue samples (upper panel). The organoid cultures can be genetically characterized and used for drug screening, which makes it possible to correlate the genetic background of a tumor with drug response. Organoids can be cryopreserved and stored in live organoid biobanks. The establishment of organoids from healthy tissue of the same patient (middle panel) gives the opportunity to develop less toxic drugs by screening for compounds that selectively kill tumor cells while leaving healthy cells unharmed. Self-renewing hepatocyte organoid cultures may be used to test for hepatotoxicity—one of the causes of drug failure in clinical trials — of a potential new drug (lower panel). In this example, drug B seems most suitable for treating the patient as it specifically kills tumor organoids and does not induce hepatotoxicity[48]. |
| |
此外,关于肿瘤类器官的另一个关注点是肿瘤细胞与肿瘤微环境的互作。众所周知,肿瘤微环境包括基质细胞、免疫细胞、趋化因子、细胞因子以及血管等,在肿瘤发生、发展和转移中都发挥着重要的作用[49]。而传统肿瘤培养方法只包含肿瘤上皮细胞,肿瘤微环境的研究还处于空白阶段。近年来,随着肿瘤类器官培养技术的优化,已经有重建肿瘤微环境法,如浸没基质胶培养法(Submerged matrigel culture) 和保留原始微环境的整体培养法,如3D微流控培养法(Microfluidic 3D culture)、气液界面培养法(Air-liquid interface,ALI)[50],培养方法见图 4。Neal等[51]利用气液界面法将肿瘤作为一个整体进行类器官培养,发现来自原发肿瘤碎片中的成纤维细胞和浸润免疫细胞群(T细胞、B细胞、巨噬细胞等) 能够持续增殖数周,保留原始肿瘤中T细胞克隆多样性。目前ALI培养法已初步应用于几种肿瘤免疫检查点疗法,包括黑色素瘤、肾细胞癌和非小细胞肺癌等[51],为研究肿瘤微环境与肿瘤细胞的互作提供一个更成熟的模型。
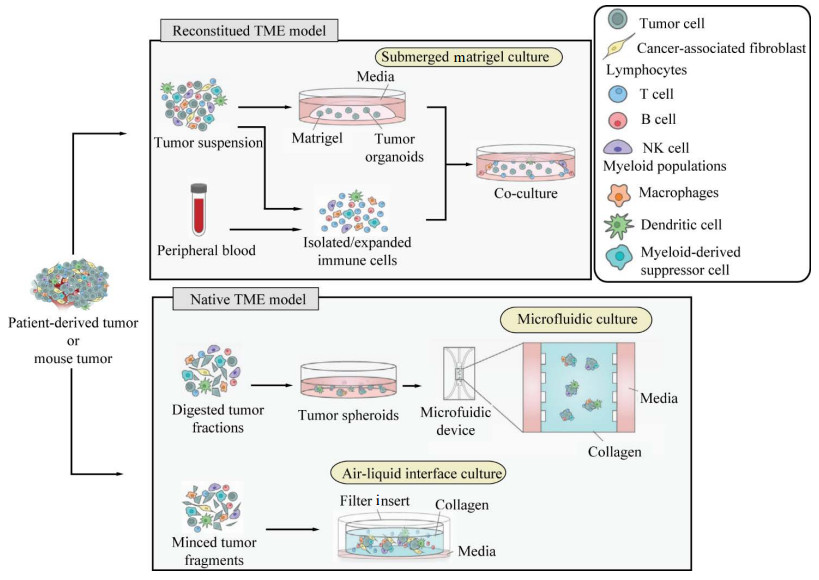
|
| 图 4 肿瘤类器官培养系统模拟肿瘤免疫微环境[50] Fig. 4 Tumor organoid culture systems modeling the tumor immune microenvironment[50]. The tumor immune microenvironment can be generated in organoids by two types of approaches. In reconstituted models, organoids containing exclusively tumor cells, often from physically and enzymatically dissociated tissues, are cultured in extracellular matrix domes (e.g., matrigel or BME-2) and submerged beneath tissue culture medium. Exogenous immune cells, such as those from autologous peripheral blood or tumor, are isolated and subsequently co-cultured with grown organoids. In holistic native TME models, the intrinsic immune microenvironment of tumor specimens is preserved along with tumor cells without reconstitution. Tumor spheroids from digested tumor tissues can be mixed with collagen and injected into microfluidic culture devices. Alternatively, in ALI culture, minced primary tissue fragments containing both tumor cells and immune components are embedded in collagen gels within an inner transwell dish. The top of the collagen gel is exposed to air, allowing cells access to a sufficient oxygen supply. NK: natural killer[50]. |
| |
类器官模型最显著的优势是人源性、近生理性,遗传病患者来源的类器官为研究复杂的多基因疾病、尚未阐明的风险基因位点和表型高度异质性的疾病机制提供可能[52]。例如,自闭症谱系障碍(Autism spectrum disorder,ASD) 是一种病因复杂的大脑发育障碍,缺乏明确的致病机制和可靠的研究模型[52]。Mariani等[53]利用ASD患者来源的端脑类器官,揭示叉头框G1 (Forkhead box G1,FOXG1) 基因及其下游基因表达水平异常增加,使γ-氨基丁酸能神经元前体细胞产生过量,抑制性神经元突触数量显著增加,从而导致ASD患儿皮质发育异常,这表明FOXG1可作为特发性ASD潜在的药物靶点。
类器官还可用于传染性疾病的研究,特别是针对仅感染人类或者特定细胞类型的病原体。2019冠状病毒病(COVID-19) 是由新型冠状病毒SARS-CoV-2 (Severe acute respiratory syndrome coronavirus 2) 引起的严重急性呼吸综合征,对全球公共卫生安全产生巨大影响[54]。Zhao等[55]首次利用人肝导管类器官模型,研究证实SARS-CoV-2感染后,COVID-19患者肝脏损伤的机制是紧密连接和胆汁酸运输的相关基因表达受干扰,最后破坏胆管细胞的屏障和胆汁酸运输功能。
此外,类器官可以实现宿主-病原生物体相互作用的研究。将艰难梭菌微注射到人类肠道类器官中,其释放毒素B与肠隐窝中的卷曲蛋白结合,引起潘氏细胞受损,最终导致肠道再生能力下降[56]。在此基础上,最新一项研究进展通过逆转肠道类器官的细胞极性,使根尖面朝外,让培养基中的微生物直接与肠类器官的根尖表面相互接触,该研究不仅能够解决微注射的技术难题,而且还能够更加直观地观察宿主-病原体的相互作用[57]。因此,研究者们一直在更新类器官技术、优化培养体系,旨在更加客观地认识人类疾病的机理。
2.2 药物研究绝大多数候选药物在Ⅰ期试验进行到临床批准过程中被中止[58],其中器官毒性是主要原因,最常见的是肝脏[59]和肾脏药物性损伤。目前常用的细胞和动物实验结果并不能顺利地转化到临床,而人源性类器官为药物毒性预测提供更精确的手段。一项研究分别利用肾母细胞瘤(Wilms瘤) 来源和正常肾组织来源的类器官对150种化合物库进行靶向药物筛选,发现两者对罗米地辛(Romidepsin) 同样敏感,正常肾组织来源的类器官对MEK抑制剂比较敏感,而Wilms瘤来源的类器官对帕比司他(Panobinostat) 更为敏感,这可能为Wilms瘤的治疗提供一个毒性较小的策略[41]。
此外,类器官还可用于药物筛选。目前针对中枢神经系统有效药物的需求量越来越大,然而大脑脉络丛上皮细胞(Choroid plexus epithelial cell,ChP) 有分泌脑脊液和构成血脑屏障两个主要功能[60],能够阻止绝大多数治疗药物进入大脑,常导致临床前试验的失败。Pellegrini等[61]研究发现ChP类器官能够分泌与脑脊液性质相似的无色液体,而且具有与体内的血脑屏障相似的强选择性,可用于中枢神经系统药物的预测和筛选。因此,在药物研究中,尤其是针对罕见病或缺乏大规模临床试验时,类器官能够为深度测序和功能测试、突变位点或表型分析提供足够资源,是药物毒性预测、新药筛选、个体化治疗的较好模型[3, 14, 48, 62]。
2.3 精准医疗近年来,精准医疗已成为公众关注的热点,其基本理念是整合个性化疾病信息,从而针对每个患者制定精准的诊断和治疗策略。对于癌症精准治疗而言,体外模型必须在生物学特性、基因突变谱上与体内肿瘤保持一致,而且能够维持高度异质性和基因型稳定性,如今肿瘤类器官的出现为癌症精准医疗创造了新的机遇。
当前肿瘤的异质性是导致抗肿瘤药物无效或耐药复发的主要原因之一。即使是基因突变谱相同的患者,其药物反应也各异。另外,一些罕见突变也需要进行药物疗效个体化测试。因此,越来越多的研究建立了肿瘤类器官生物库进行抗肿瘤药物高通量筛选和预测药物反应。Yan等[46]建立涵盖了绝大多数胃癌分子亚型和特异性突变谱的类器官生物库,对37种抗癌药物进行筛选,发现对常规化疗药物5-氟尿嘧啶(5-fluorouracil,5-FU) 和顺铂普遍存在耐药,而对奥沙利铂、表柔比星和紫杉醇反应较敏感。Vlachogiannis等[63]将来源于转移性胃肠癌患者的类器官生物库用于临床上预测患者对于靶向药物或者化疗的反应,发现敏感性可达100%,特异性达93%,阳性预测值达88%和阴性预测值达100%,因此肿瘤类器官生物库在精准医疗中逐渐得到广泛的应用,为癌症的个性化治疗提供大规模药物筛选平台和重要资源。
近年来新兴的基因编辑技术(Clustered regularly interspersed short palindromic repeats- caspase,CRISPR-Cas) 给人类生物学的研究带来巨大变革,也为精准医疗带来新希望。将CRISPR技术与肿瘤类器官相结合,能够实现精准研究单一基因突变对肿瘤细胞的影响,从而建立起基因型和表型的关联性[62]。Liu等[15]应用CRISPR-Cas9编辑技术失活APC基因,形成Wnt持续性激活的BE类器官模型,精准研究Wnt/β-catenin信号通路在BE向食管癌转化中的作用,避免了传统模型中其他信号噪声的干扰。最近一项研究使用CRISPR技术构建抗鼠科肉瘤病毒癌基因同源物B1 (The raf murine sarcoma viral oncogene homolog B,BRAF) 突变型锯齿状结肠癌类器官模型,随后顺序引入TGFβ2、P16/Ink4a失活突变,以确定特定基因对肿瘤生长转移的影响[64]。因此,CRISPR和类器官技术的碰撞产生了一个多功能工具箱,加速了人类癌症基因的研究,对精准医疗有重要的意义。
2.4 组织和器官再生再生医学的主要目标是在体外用健康组织替代某一功能或结构受损的器官,实现无免疫抑制、无并发症和毒性减少,避免因终生抗排斥治疗产生巨额的费用[65]。虽然现代医学已经能够实现异体移植,尤其在治疗终末期器官衰竭如心脏、肝脏或肾脏中,器官移植仍是临床主要采用的方法,但是存在供体数量严重短缺以及组织排斥等问题[66],因此寻找新的组织来源十分迫切。类器官能够同基因组织扩增从而用于自体移植,为器官替代策略提供可再生资源[28]。
肾是终末期衰竭率最高的器官,Taguchi等[67]已经成功将肾类器官移植到成年小鼠的肾包膜下,并血管化,有望进行器官替代。Yui等[31]将结肠类器官移植到硫酸葡聚糖诱导的急性结肠炎小鼠中能够再生结肠上皮组织,形成自我更新的隐窝,修复受损上皮细胞。虽然肝脏的类器官模型尚未建立,但2013年Takebe等[66]已成功将肝芽类器官移植到肝损伤小鼠体内,形成与人体肝脏类似的功能性血管系统和代谢系统,刺激肝脏器官的发生和再生,提高小鼠存活率。此后,研究者们在充氧旋转烧瓶中大规模培养人类肝脏类器官,使临床上大规模移植肝脏类器官成为可能[68]。尽管这些技术转化为临床可移植的器官仍需要漫长的过程,但类器官移植为再生医学的研究提供了一个新的希望。
3 类器官与其他技术结合的应用 3.1 类器官芯片类器官自组织的过程遵循体内器官发育程序,但是其培养系统不可控制且缺乏微环境的互作,因此需要开发能够精确产生、控制和分析类器官及其微环境的创新工程。器官芯片(Organ-on-a-chip) 指以微流控技术为核心,随后引入细胞、细胞外基质、检测细胞功能的传感器等元素构成的人体器官生理微系统,主要优势在于能够精确控制细胞及其微环境[69-70]。基于此,类器官芯片(Organoid-on-a-chip) 是将器官芯片和类器官技术相整合形成器官生理微系统[69],使两者优势互补,既能模拟器官的发育过程、生理状态和功能,又能弥补类器官培养中分子信号无法精确调控的缺陷。Shirure等[71]将乳腺癌类器官芯片形成微血管网络,模拟灌注血管向肿瘤细胞输送营养物质和抗肿瘤药物,可以实现实时监测肿瘤血管生成、增殖和迁移,并精确量化距离、药物流量和浓度等条件。因此,类器官芯片拓宽了类器官技术在基础生物学研究、药物毒性测试和临床前试验等多个领域的应用,为孕育出人体器官仿生芯片等高精尖的技术奠定基础。
3.2 单细胞RNA测序类器官模型是否能够保留原始器官的分子生物学特性和基因组信息,是研究者们评价一个新建模型系统必须关注的问题。近年来,单细胞RNA测序(Single-cell RNA sequencing,scRNA-seq) 已广泛用于研究类器官和原始器官细胞分化类型和转录组的差异性[72-74],以及量化类器官本身的异质性[75]。已有研究者利用scRNA-seq分析人体大脑和脑类器官的不同发育时期、不同皮层区域的转录组,结果显示,皮层类器官的放射状神经胶质细胞尚未成熟,缺乏人体特征性的细胞亚型和成熟的神经元[72]。Cowan等[73]通过scRNA-seq对视网膜类器官进行单细胞转录组分析,首次回答了视网膜类器官与成人视网膜基因表达是否以及何时相匹配,视网膜疾病是否具有细胞类型特异性,而视网膜类器官模型能否保留这些特异性的细胞类型等问题。
4 总结与展望类器官模型是人体组织实验研究的最新技术,然而与传统模型相比仍处于摸索阶段,其保真性、稳定性以及如何精确控制微环境条件等成为类器官技术发展中需要攻克的问题。
首先,类器官的生理特征和遗传等保真度和稳定性尚不清楚,在反映人类特定细胞的特征上存在局限性[76]。例如,脑类器官虽可以分化为多种细胞类型,但放射状胶质细胞、中间祖细胞、深层兴奋性神经元等与原始组织仍存在许多差异[72]。因此,除了多组学(蛋白组学、基因组学和转录组学) 分析外[75],还需要综合更精确、更全面的方法分析类器官的空间组织、表观遗传、代谢和电生理等功能参数。目前,人类细胞图谱计划(The human cell atlas project) 正在创建一个大型的数据库,用于收集来自不同人体组织和类器官的单细胞基因组数据[77]。这些资源对于全世界范围标准化类器官技术、评估类器官模型保真度具有非常深远的意义。
类器官研究面临的另一个挑战是成熟度有限,只能形成类似于胎儿的组织而非成人组织,并且缺乏体内重要的生理过程以及血管、淋巴管和神经功能,仍无法完全复制人体器官[76],只能称为简单的器官模型。已有研究利用3D反应器(3D bioreactor)[78]以及异种移植形成类器官——动物嵌合体[79]等技术促进类器官形成神经或血管网络。最近Rajasekar等[80]设计了一个微流控平台IFlowPlate,其内皮细胞可自组装成微血管网络,再与结肠类器官共培养,形成具有血管灌注和先天性免疫功能的结肠类器官。
此外,随着类器官体积和复杂性的增加,由于氧气和营养表面扩散的限制,常导致内部细胞缺氧和死亡,阻止类器官后期发育和成熟,限制了其在临床上的应用[76]。目前已有研究者开发类器官切片培养模型,将脑类器官切片成圆盘状,使其内部暴露于外部培养环境中,能够在长期培养中维持类器官神经发生和皮层结构的生长,模拟更复杂的人类胚胎晚期皮层发育特征[81]。随着这些创新的生物技术不断发展,将解决类器官成熟度的难题,拓宽其在研究中的应用。
总而言之,人类生物学的发展离不开借助有效的研究工具,从2D培养揭示细胞分子机制开始到3D结构重建人体器官,研究者们都试图探寻更加高度逼真,同时兼具能够广泛应用的模型系统,在这种背景条件下类器官技术应运而生并得到迅猛发展。虽然当前类器官系统只是初具雏形,并不能完全取代所有的传统模型,亦不能做到完全复制人体器官结构和功能,但类器官最大的特点是人源性、近生理性,能在器官水平上解决传统模型的一些困境,诸如在缺乏肿瘤异质性、不能进行药物高通量筛选、无法模拟器官形成和发育以及无法准确预测个体对药物反应等。因而,类器官模型与传统模型优势互补结合使用,将为人类生物学开辟新途径。目前,国内外学者聚焦于协同其他尖端技术(器官芯片、微移动阵列、scRNA-seq、CRISPR-Cas9、高通量筛选等) 分析和改造类器官,精细优化和标准化类器官培养系统中每一个实验步骤(培养基成分、细胞类型、数量、试剂添加顺序等),攻克细胞外微环境、血管、神经网络、成熟度等难题,旨在提高类器官的稳定性、保真性、重现性、可扩展性。我们有理由相信随着研究的不断深入和革新,类器官模型会逐渐趋于完善,甚至催生新的模型和新的研究工具,在转化医学和临床个体化治疗上扮演越来越重要的角色。
| [1] |
Frum T, Spence JR. hPSC-derived organoids: models of human development and disease. J Mol Med (Berl), 2020. DOI:10.1007/s00109-020-01969-w
|
| [2] |
Kim J, Koo BK, Knoblich JA. Human organoids: model systems for human biology and medicine. Nat Rev Mol Cell Biol, 2020, 21(10): 571-584. DOI:10.1038/s41580-020-0259-3
|
| [3] |
Schutgens F, Clevers H. Human organoids: tools for understanding biology and treating diseases. Annu Rev Pathol, 2020, 15: 211-234. DOI:10.1146/annurev-pathmechdis-012419-032611
|
| [4] |
Lui JH, Hansen DV, Kriegstein AR. Development and evolution of the human neocortex. Cell, 2011, 146(1): 18-36. DOI:10.1016/j.cell.2011.06.030
|
| [5] |
Estes JD, Wong SW, Brenchley JM. Nonhuman primate models of human viral infections. Nat Rev Immunol, 2018, 18(6): 390-404. DOI:10.1038/s41577-018-0005-7
|
| [6] |
Bliss-Moreau E, Rudebeck PH. Animal models of human mood. Neurosci Biobehav Rev, 2021, 120: 574-582. DOI:10.1016/j.neubiorev.2020.06.024
|
| [7] |
Li HW, Zhang L, Qin C. Current state of research on non-human primate models of Alzheimer's disease. Anim Models Exp Med, 2019, 2(4): 227-238. DOI:10.1002/ame2.12092
|
| [8] |
Li ML, Aggeler J, Farson DA, et al. Influence of a reconstituted basement membrane and its components on casein gene expression and secretion in mouse mammary epithelial cells. Proc Natl Acad Sci USA, 1987, 84(1): 136-140. DOI:10.1073/pnas.84.1.136
|
| [9] |
Sato T, Vries RG, Snippert HJ, et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature, 2009, 459(7244): 262-265. DOI:10.1038/nature07935
|
| [10] |
Lancaster MA, Knoblich JA. Organogenesis in a dish: modeling development and disease using organoid technologies. Science, 2014, 345(6194): 1247125. DOI:10.1126/science.1247125
|
| [11] |
Kruczek K, Swaroop A. Pluripotent stem cell-derived retinal organoids for disease modeling and development of therapies. Stem Cells, 2020, 38(10): 1206-1215.
|
| [12] |
Lancaster MA, Renner M, Martin CA, et al. Cerebral organoids model human brain development and microcephaly. Nature, 2013, 501(7467): 373-379. DOI:10.1038/nature12517
|
| [13] |
Takasato M, Er PX, Becroft M, et al. Directing human embryonic stem cell differentiation towards a renal lineage generates a self-organizing kidney. Nat Cell Biol, 2014, 16(1): 118-126. DOI:10.1038/ncb2894
|
| [14] |
Fatehullah A, Tan SH, Barker N. Organoids as an in vitro model of human development and disease. Nat Cell Biol, 2016, 18(3): 246-254. DOI:10.1038/ncb3312
|
| [15] |
Liu X, Cheng YL, Abraham JM, et al. Modeling Wnt signaling by CRISPR-Cas9 genome editing recapitulates neoplasia in human Barrett epithelial organoids. Cancer Lett, 2018, 436: 109-118. DOI:10.1016/j.canlet.2018.08.017
|
| [16] |
Czerniecki SM, Cruz NM, Harder JL, et al. High-throughput screening enhances kidney organoid differentiation from human pluripotent stem cells and enables automated multidimensional phenotyping. Cell Stem Cell, 2018, 22(6): 929-940.e4. DOI:10.1016/j.stem.2018.04.022
|
| [17] |
Li M, Izpisua Belmonte JC. Organoids-preclinical models of human disease. N Engl J Med, 2019, 380(6): 569-579. DOI:10.1056/NEJMra1806175
|
| [18] |
Eiraku M, Takata N, Ishibashi H, et al. Self-organizing optic-cup morphogenesis in three-dimensional culture. Nature, 2011, 472(7341): 51-56. DOI:10.1038/nature09941
|
| [19] |
Mansour AA, Goncalves JT, Bloyd CW, et al. An in vivo model of functional and vascularized human brain organoids. Nat Biotechnol, 2018, 36(5): 432-441. DOI:10.1038/nbt.4127
|
| [20] |
Jamieson PR, Dekkers JF, Rios AC, et al. Derivation of a robust mouse mammary organoid system for studying tissue dynamics. Development, 2017, 144(6): 1065-1071.
|
| [21] |
Maimets M, Rocchi C, Bron R, et al. Long-term in vitro expansion of salivary gland stem cells driven by Wnt signals. Stem Cell Rep, 2016, 6(1): 150-162. DOI:10.1016/j.stemcr.2015.11.009
|
| [22] |
De Wert G, Mummery C. Human embryonic stem cells: research, ethics and policy. Hum Reprod, 2003, 18(4): 672-682. DOI:10.1093/humrep/deg143
|
| [23] |
Takahashi K, Tanabe K, Ohnuki M, et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell, 2007, 131(5): 861-872. DOI:10.1016/j.cell.2007.11.019
|
| [24] |
Spence JR, Mayhew CN, Rankin SA, et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature, 2011, 470(7332): 105-109. DOI:10.1038/nature09691
|
| [25] |
Eiraku M, Sasai Y. Self-formation of layered neural structures in three-dimensional culture of ES cells. Curr Opin Neurobiol, 2012, 22(5): 768-777. DOI:10.1016/j.conb.2012.02.005
|
| [26] |
Eiraku M, Watanabe K, Matsuo-Takasaki M, et al. Self-organized formation of polarized cortical tissues from ESCs and its active manipulation by extrinsic signals. Cell Stem Cell, 2008, 3(5): 519-532. DOI:10.1016/j.stem.2008.09.002
|
| [27] |
Avior Y, Sagi I, Benvenisty N. Pluripotent stem cells in disease modelling and drug discovery. Nat Rev Mol Cell Biol, 2016, 17(3): 170-182. DOI:10.1038/nrm.2015.27
|
| [28] |
Rossi G, Manfrin A, Lutolf MP. Progress and potential in organoid research. Nat Rev Genet, 2018, 19(11): 671-687. DOI:10.1038/s41576-018-0051-9
|
| [29] |
Warren CR, Jaquish CE, Cowan CA. The NextGen genetic association studies consortium: a foray into in vitro population genetics. Cell Stem Cell, 2017, 20(4): 431-433. DOI:10.1016/j.stem.2017.03.021
|
| [30] |
Barker N, Huch M, Kujala P, et al. Lgr5+ve stem cells drive self-renewal in the stomach and build long-lived gastric units in vitro. Cell Stem Cell, 2010, 6(1): 25-36. DOI:10.1016/j.stem.2009.11.013
|
| [31] |
Yui S, Nakamura T, Sato T, et al. Functional engraftment of colon epithelium expanded in vitro from a single adult Lgr5+ stem cell. Nat Med, 2012, 18(4): 618-623. DOI:10.1038/nm.2695
|
| [32] |
Huch M, Dorrell C, Boj SF, et al. In vitro expansion of single Lgr5+ liver stem cells induced by Wnt-driven regeneration. Nature, 2013, 494(7436): 247-250. DOI:10.1038/nature11826
|
| [33] |
Sato T, Stange DE, Ferrante M, et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology, 2011, 141(5): 1762-1772. DOI:10.1053/j.gastro.2011.07.050
|
| [34] |
Boj SF, Hwang CI, Baker LA, et al. Organoid models of human and mouse ductal pancreatic cancer. Cell, 2015, 160(1-2): 324-338. DOI:10.1016/j.cell.2014.12.021
|
| [35] |
Lugli N, Kamileri I, Keogh A, et al. R-spondin 1 and noggin facilitate expansion of resident stem cells from non-damaged gallbladders. EMBO Rep, 2016, 17(5): 769-779. DOI:10.15252/embr.201642169
|
| [36] |
Karthaus WR, Iaquinta PJ, Drost J, et al. Identification of multipotent luminal progenitor cells in human prostate organoid cultures. Cell, 2014, 159(1): 163-175. DOI:10.1016/j.cell.2014.08.017
|
| [37] |
Linnemann JR, Miura H, Meixner LK, et al. Quantification of regenerative potential in primary human mammary epithelial cells. Development, 2015, 142(18): 3239-3251.
|
| [38] |
Rock JR, Onaitis MW, Rawlins EL, et al. Basal cells as stem cells of the mouse trachea and human airway epithelium. Proc Natl Acad Sci USA, 2009, 106(31): 12771-12775. DOI:10.1073/pnas.0906850106
|
| [39] |
Huch M, Gehart H, Van Boxtel R, et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell, 2015, 160(1/2): 299-312.
|
| [40] |
Yin X, Farin HF, Van Es JH, et al. Niche-independent high-purity cultures of Lgr5+ intestinal stem cells and their progeny. Nat Methods, 2014, 11(1): 106-112. DOI:10.1038/nmeth.2737
|
| [41] |
Calandrini C, Schutgens F, Oka R, et al. An organoid biobank for childhood kidney cancers that captures disease and tissue heterogeneity. Nat Commun, 2020, 11: 1310. DOI:10.1038/s41467-020-15155-6
|
| [42] |
Jacob F, Salinas RD, Zhang DY, et al. A patient-derived glioblastoma organoid model and biobank recapitulates inter- and intra-tumoral heterogeneity. Cell, 2020, 180(1): 188-204.e22. DOI:10.1016/j.cell.2019.11.036
|
| [43] |
Kim M, Mun H, Sung CO, et al. Patient-derived lung cancer organoids as in vitro cancer models for therapeutic screening. Nat Commun, 2019, 10: 3991. DOI:10.1038/s41467-019-11867-6
|
| [44] |
Gao D, Vela I, Sboner A, et al. Organoid cultures derived from patients with advanced prostate cancer. Cell, 2014, 159(1): 176-187. DOI:10.1016/j.cell.2014.08.016
|
| [45] |
Yao Y, Xu XY, Yang LF, et al. Patient-derived organoids predict chemoradiation responses of locally advanced rectal cancer. Cell Stem Cell, 2020, 26(1): 17-26.e6. DOI:10.1016/j.stem.2019.10.010
|
| [46] |
Yan HHN, Siu HC, Law S, et al. A comprehensive human gastric cancer organoid biobank captures tumor subtype heterogeneity and enables therapeutic screening. Cell Stem Cell, 2018, 23(6): 882-897.e11. DOI:10.1016/j.stem.2018.09.016
|
| [47] |
Van De Wetering M, Francies HE, Francis JM, et al. Prospective derivation of a living organoid biobank of colorectal cancer patients. Cell, 2015, 161(4): 933-945. DOI:10.1016/j.cell.2015.03.053
|
| [48] |
Drost J, Clevers H. Organoids in cancer research. Nat Rev Cancer, 2018, 18(7): 407-418. DOI:10.1038/s41568-018-0007-6
|
| [49] |
Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell, 2011, 144(5): 646-674. DOI:10.1016/j.cell.2011.02.013
|
| [50] |
Yuki K, Cheng N, Nakano M, et al. Organoid models of tumor immunology. Trends Immunol, 2020, 41(8): 652-664. DOI:10.1016/j.it.2020.06.010
|
| [51] |
Neal JT, Li XN, Zhu JJ, et al. Organoid modeling of the tumor immune microenvironment. Cell, 2018, 175(7): 1972-1988.e16. DOI:10.1016/j.cell.2018.11.021
|
| [52] |
Di Lullo E, Kriegstein AR. The use of brain organoids to investigate neural development and disease. Nat Rev Neurosci, 2017, 18(10): 573-584. DOI:10.1038/nrn.2017.107
|
| [53] |
Mariani J, Coppola G, Zhang P, et al. FOXG1-dependent dysregulation of GABA/glutamate neuron differentiation in autism spectrum disorders. Cell, 2015, 162(2): 375-390. DOI:10.1016/j.cell.2015.06.034
|
| [54] |
Shen B, Yi X, Sun YT, et al. Proteomic and metabolomic characterization of COVID-19 patient sera. Cell, 2020, 182(1): 59-72.e15. DOI:10.1016/j.cell.2020.05.032
|
| [55] |
Zhao B, Ni C, Gao R, et al. Recapitulation of SARS-CoV-2 infection and cholangiocyte damage with human liver ductal organoids. Protein Cell, 2020, 11(10): 771-775. DOI:10.1007/s13238-020-00718-6
|
| [56] |
Chen P, Tao L, Wang TY, et al. Structural basis for recognition of frizzled proteins by Clostridium difficile toxin B. Science, 2018, 360(6389): 664-669. DOI:10.1126/science.aar1999
|
| [57] |
Co JY, Margalef-Catala M, Li XN, et al. Controlling epithelial polarity: a human enteroid model for host-pathogen interactions. Cell Rep, 2019, 26(9): 2509-2520.e4. DOI:10.1016/j.celrep.2019.01.108
|
| [58] |
Horvath P, Aulner N, Bickle M, et al. Screening out irrelevant cell-based models of disease. Nat Rev Drug Discov, 2016, 15(11): 751-769. DOI:10.1038/nrd.2016.175
|
| [59] |
Ballet F. Hepatotoxicity in drug development: detection, significance and solutions. J Hepatol, 1997, 26(S2): 26-36.
|
| [60] |
Lun MP, Monuki ES, Lehtinen MK. Development and functions of the choroid plexus-cerebrospinal fluid system. Nat Rev Neurosci, 2015, 16(8): 445-457. DOI:10.1038/nrn3921
|
| [61] |
Pellegrini L, Bonfio C, Chadwick J, et al. Human CNS barrier-forming organoids with cerebrospinal fluid production. Science, 2020, 369(6500): eaaz5626. DOI:10.1126/science.aaz5626
|
| [62] |
Fujii M, Sato T. Somatic cell-derived organoids as prototypes of human epithelial tissues and diseases. Nat Mater, 2021, 20(2): 156-169. DOI:10.1038/s41563-020-0754-0
|
| [63] |
Vlachogiannis G, Hedayat S, Vatsiou A, et al. Patient-derived organoids model treatment response of metastatic gastrointestinal cancers. Science, 2018, 359(6378): 920-926. DOI:10.1126/science.aao2774
|
| [64] |
Lannagan TRM, Lee YK, Wang TT, et al. Genetic editing of colonic organoids provides a molecularly distinct and orthotopic preclinical model of serrated carcinogenesis. Gut, 2019, 68(4): gutjnl-2017-315920.
|
| [65] |
Edgar L, Pu T, Porter B, et al. Regenerative medicine, organ bioengineering and transplantation. Br J Surg, 2020, 107(7): 793-800. DOI:10.1002/bjs.11686
|
| [66] |
Takebe T, Sekine K, Enomura M, et al. Vascularized and functional human liver from an iPSC-derived organ bud transplant. Nature, 2013, 499(7459): 481-484. DOI:10.1038/nature12271
|
| [67] |
Taguchi A, Kaku Y, Ohmori T, et al. Redefining the in vivo origin of metanephric nephron progenitors enables generation of complex kidney structures from pluripotent stem cells. Cell Stem Cell, 2014, 14(1): 53-67. DOI:10.1016/j.stem.2013.11.010
|
| [68] |
Schneeberger K, Sánchez-Romero N, Ye SC, et al. Large-scale production of LGR5-positive bipotential human liver stem cells. Hepatology, 2020, 72(1): 257-270. DOI:10.1002/hep.31037
|
| [69] |
Park SE, Georgescu A, Huh D. Organoids-on-a-chip. Science, 2019, 364(6444): 960-965. DOI:10.1126/science.aaw7894
|
| [70] |
秦建华, 张敏, 于浩, 等. 人体器官芯片. 中国科学院院刊, 2017, 32(12): 1281-1289. Qin JH, Zhang M, Yu H, et al. Human organs-on-a-chip. Bull Chin Acad Sci, 2017, 32(12): 1281-1289 (in Chinese). |
| [71] |
Shirure VS, Bi Y, Curtis MB, et al. Tumor-on-a-chip platform to investigate progression and drug sensitivity in cell lines and patient-derived organoids. Lab a Chip, 2018, 18(23): 3687-3702. DOI:10.1039/C8LC00596F
|
| [72] |
Bhaduri A, Andrews MG, Leon WM, et al. Cell stress in cortical organoids impairs molecular subtype specification. Nature, 2020, 578(7793): 142-148. DOI:10.1038/s41586-020-1962-0
|
| [73] |
Cowan CS, Renner M, De Gennaro M, et al. Cell types of the human retina and its organoids at single-cell resolution. Cell, 2020, 182(6): 1623-1640.e34. DOI:10.1016/j.cell.2020.08.013
|
| [74] |
Van Den Brink SC, Alemany A, Van Batenburg V, et al. Single-cell and spatial transcriptomics reveal somitogenesis in gastruloids. Nature, 2020, 582(7812): 405-409. DOI:10.1038/s41586-020-2024-3
|
| [75] |
Brancati G, Treutlein B, Camp JG. Resolving neurodevelopmental and vision disorders using organoid single-cell multi-omics. Neuron, 2020, 107(6): 1000-1013. DOI:10.1016/j.neuron.2020.09.001
|
| [76] |
Bhaduri A, Andrews MG, Kriegstein AR, et al. Are organoids ready for prime time?. Cell Stem Cell, 2020, 27(3): 361-365. DOI:10.1016/j.stem.2020.08.013
|
| [77] |
The organoid cell atlas: a Rosetta stone for biomedical discovery and regenerative therapy[EB/OL]. [2020-12-20]. http://doi.org/10.5281/zenodo.4001718.
|
| [78] |
Qian X, Nguyen HN, Song MM, et al. Brain-region-specific organoids using mini-bioreactors for modeling ZIKV exposure. Cell, 2016, 165(5): 1238-1254. DOI:10.1016/j.cell.2016.04.032
|
| [79] |
Rowe RG, Daley GQ. Induced pluripotent stem cells in disease modelling and drug discovery. Nat Rev Genet, 2019, 20(7): 377-388. DOI:10.1038/s41576-019-0100-z
|
| [80] |
Rajasekar S, Lin DSY, Abdul L, et al. IFlowPlate-a customized 384-well plate for the culture of perfusable vascularized colon organoids. Adv Mater, 2020, 32(46): 2002974. DOI:10.1002/adma.202002974
|
| [81] |
Qian XY, Su YJ, Adam CD, et al. Sliced human cortical organoids for modeling distinct cortical layer formation. Cell Stem Cell, 2020, 26(5): 766-781.e9. DOI:10.1016/j.stem.2020.02.002
|
 2021, Vol. 37
2021, Vol. 37