中国科学院微生物研究所、中国微生物学会主办
文章信息
- 赖贞丽, 黄钢锋, 白丽萍
- Lai Zhenli, Huang Gangfeng, Bai Liping
- 甲基-辅酶M还原酶结构、功能及催化机制研究进展
- Advances of structure, function, and catalytic mechanism of methyl-coenzyme M reductase
- 生物工程学报, 2021, 37(12): 4147-4157
- Chinese Journal of Biotechnology, 2021, 37(12): 4147-4157
- 10.13345/j.cjb.200830
-
文章历史
- Received: December 31, 2020
- Accepted: March 10, 2021
- Published: March 25, 2021
甲烷对农业生产、生态环境及社会经济发展都有重要的影响[1]。一方面,它是一类重要的清洁能源[1-2];另一方面,它也是一类强力的温室气体[1-2]。产甲烷古菌是生物甲烷的主要生产者,也是地球微生物系统的重要组成部分,在碳素循环中扮演着重要的角色[3]。在已知的所有产甲烷或甲烷厌氧氧化代谢中,甲烷的生成或氧化都是由甲基-辅酶M还原酶(Methyl-coenzyme M reductase,Mcr) 催化的[4-7]。该酶的基因高度保守,被广泛应用于古菌的鉴定与系统发育研究[4-5, 8-9]。自2016年以来,研究者陆续发现了一些能够厌氧氧化非甲烷烷烃(如乙烷、丙烷、丁烷等) 未培养海洋古菌,又引起了人们对这类古菌新型烷烃厌氧氧化代谢的浓厚兴趣[10-15]。其中,类甲基-辅酶M还原酶(烷基-辅酶M还原酶,Mcr-like enzyme) 被认为是关键限速酶,催化烷烃碳氢(C-H) 键的裂解[10-15]。而认知和理解Mcr/Mcr-like对研究新型产甲烷代谢和烷烃厌氧氧化代谢都大有裨益。因此,本文从结构、功能及催化机制3个方面,结合本课题组当前的研究工作,概述了Mcr/Mcr-like的最新研究进展。
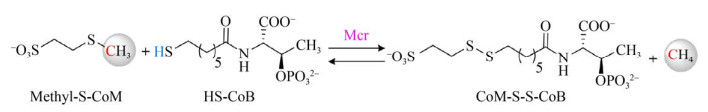
1 碳循环中的甲基-辅酶M还原酶甲烷是一类强力的温室气体,也是地球碳素循环中的关键节点[1]。全球每年产生约1×109 t甲烷,主要由产甲烷古菌代谢CO2/H2、乙酸和C1/C2甲基化合物生成;同时,约1×108 t甲烷又通过甲烷厌氧氧化代谢被氧化为CO2释放进入大气,这两个过程都涉及Mcr这一核心功能酶(图 1A)[2, 7, 16-18]。
|
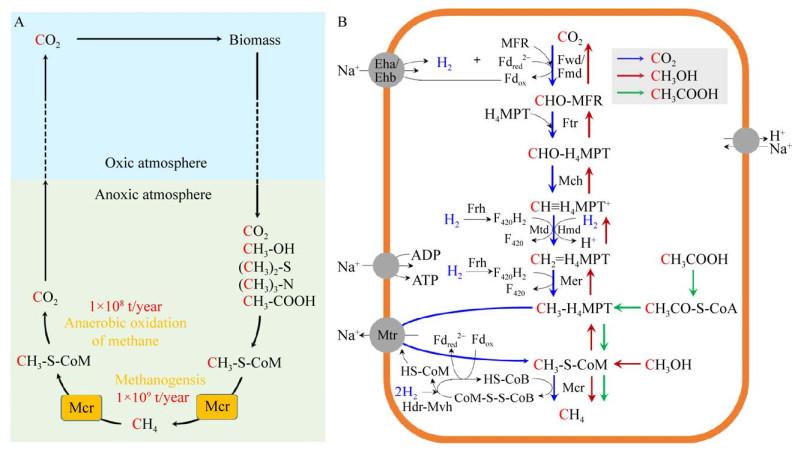
| 图 1 甲基-辅酶M还原酶(Mcr) 在全球碳循环及产甲烷代谢中的定位示意图[2, 7, 16-18, 26] Fig. 1 The location of methyl-coenzyme M reductase (Mcr) in the global carbon cycle and methanogenic pathways[2, 7, 16-18, 26]. (A) Mcr functions in the global carbon cycle as the key enzyme involved in methanogenesis and anaerobic oxidation of methane. (B) Mcr acts as the key enzyme in the final step of hydrogenotrophic, methylotrophic and acetoclastic methanogenesis. |
| |
产甲烷古菌主要以发酵细菌或互营细菌分解有机质产生的简单C1/C2化合物(如CO2、乙酸等) 作为底物进行产甲烷代谢来获取生长所需的能量(图 1B)[17-18]。根据底物的类型,通常将产甲烷代谢分为氢营养型、甲基营养型和乙酸营养型3大类[17]。氢营养型产甲烷古菌不含有细胞色素,以H2为电子供体,在一系列还原酶、转移酶和氢酶的催化下还原CO2生成CH4和H2O[18]。完整的代谢途径如图 1所示:(1) 甲酰基-甲基呋喃脱氢酶(Tungstopterin-dependent formylmethanofuran dehydrogenase,Fwd) 还原CO2为甲酰基(CHO-)并负载于一碳载体甲烷呋喃(Methanofuran, MFR) 生成CHO-MFR[19];(2) 甲酰基转移酶(Formyltransferase,Ftr) 转移甲酰基到一碳载体四氢甲基蝶呤(Tetrahydromethanopterin,H4MPT)生成CHO-H4MPT[20-21];(3) 环化水解酶(Methenyl- H4MPT cyclohydrolase,Mch)催化CHO-H4MPT脱水缩合形成次甲基-四氢甲基蝶呤(Methenyl-H4MPT+,CH≡H4MPT+)[22-23];(4) CH≡H4MPT+经过两次还原反应转化为甲基(Methyl-H4MPT,CH3-H4MPT),依次由F420依赖型亚甲基-四氢甲烷蝶呤脱氢酶(F420-dependent methylene-H4MPT dehydrogenase,Mtd)[24]和亚甲基-四氢甲基蝶呤还原酶(Methylene-H4MPT reductase,Mer)催化完成[25];(5) 甲基转移酶(Methyltransferase,Mtr)将CH3-H4MPT的甲基转移到一碳载体辅酶M (Coenzyme M,CoM-SH)形成甲基-辅酶M (CH3-S-CoM),并通过形成膜内外离子梯度来直接推动三磷酸腺苷(Adenosine triphosphate,ATP)的合成[26]。(6) Mcr催化CH3-S-CoM与辅酶B (Coenzyme B,CoB-SH) 生成终产物CH4和异二硫化合物(Heterodisulfide,CoB-S-S-CoM)[26]。后者在异二硫化合物还原酶/[NiFe]-氢酶复合体(Heterodisulide reductase/[NiFe]-hydrogenase,Hdr- Mvh) 的作用下通过电子歧化机制再次还原为CoM-SH与CoB-SH[27]。
甲基营养型和乙酸营养型途径都存在于含细胞色素的产甲烷古菌中[28]。甲基营养型主要存在于甲烷八叠球菌属(Methanosarcina)和甲烷球形菌属(Methanosphaera),利用甲醇、甲胺、甲硫醚等简单甲基化合物进行生长代谢[29-30]。其特点在于代谢的起始过程:甲基在底物特异性甲基转移酶的催化下从甲基化合物转移至辅酶M生成甲基-辅酶M (CH3-S-CoM),从而进入产甲烷代谢[29-30]。自然界中仅有Methanosarcina和Methanosaeta以乙酸盐作为底物进行乙酸营养型产甲烷代谢[29-30]。首先,乙酸被活化为乙酰辅酶A (Acetyl coenzyme A,Acetyl-CoA) 并消耗一个ATP[29-30];然后,由一氧化碳脱氢酶/乙酰辅酶A合成酶复合体(Carbon monoxide dehydrogenase/acetyl-CoA synthase,CODH/ ACS) 催化甲基从乙酰辅酶A转移至一碳载体四氢甲基萨尔奇纳蝶呤(Tetrahydrosarcinapterin,H4SPT) 生成CH3-H4SPT进入产甲烷代谢,CO基团被氧化生成CO2[31-32]。
2 甲基-辅酶M还原酶的生物学功能Mcr催化的甲烷合成反应是整个产甲烷代谢的核心限速步骤[33-36]。该酶以CoB-SH为电子供体,还原底物甲基-辅酶M (CH3-S-CoM) 生成CH4 (图 2),是产甲烷古菌的特征反应[33-36]。不同于产甲烷古菌代谢C1/C2化合物生产甲烷以获取能量,厌氧甲烷氧化古菌(Archaeal anaerobic methane oxidizers,ANMEs) 厌氧氧化甲烷进行生长代谢[37-41]。其代谢过程为产甲烷代谢的逆向途径[37-41],并通过与硫酸盐/硝酸盐还原菌进行互营生长以克服热力学能障(ΔG0′=+30 kJ/mol)[37-41]。Mcr也作用于甲烷厌氧氧化代谢的第一步激活反应,即催化甲烷生成的逆反应[37-41]。
Mcr是一个多亚基酶复合体,mcrA、mcrB和mcrG基因分别编码α-、β-和γ-亚基[42-43]。mcr基因簇中的mcrC和mcrD基因尚不清楚其功能[42-43]。其中,编码α-亚基的mcrA基因是高度保守的,将作为产甲烷古菌系统发育研究中的特征基因[42-43]。典型的Mcr酶复合体呈(αβγ)2构象,其活性中心含有特殊的镍卟啉F430辅因子[42-43]。或许是为了弥补Mcr的低催化活性,它在细胞中的表达量可达胞质总蛋白的10%左右[42-44]。Mcr一共有McrⅠ、McrⅡ和McrⅢ三类同工酶[45-49]。甲烷杆菌目(Methanobacteriales) 和甲烷球菌目(Methanococcales) 能够合成McrⅠ和McrⅡ两种同工酶,两者在结构上有60%–70%的相似度[45-46]。有研究发现,在马尔堡甲烷热杆菌Methanothermobacter marburgensis中,这两种同工酶的表达水平受温度、pH、生长阶段等生长条件的调控[45-46]。McrⅢ主要存在于Methanococcales,包括生长快速的嗜热和超嗜热的海洋产甲烷古菌(如甲烷热球菌Methanothermococcus thermolithotrophicus和詹氏甲烷球菌Methanocaldococcus jannaschii)[47-48]。系统发育分析表明,编码3类不同MCR的基因都来自同一祖先,其中编码McrⅠ和McrⅢ的基因簇都含有5个基因(mcrBDCGA),而MCRⅡ丢失了1–2个基因(mcrD或mcrC)[49],但这并不影响其催化活性[49]。
过去认为,Mcr只存在于广古菌门Methanobacteriales/Methanococcales/甲烷微菌目Methanomicrobiales/甲烷火菌目Methanopyrales/甲烷八叠球菌目Methanosarcinales五个目中,但近年来研究者在深古菌门和韦斯特古菌门中也发现了mcr基因簇[4, 11]。组学技术的发展与广泛应用,使得Mcr在更多的未培养古菌中被发现,尤其是在进行烷烃厌氧氧化代谢的海洋古菌中分布十分广泛[10-15]。虽然其生态分布不断拓展[4, 11, 36],但还未在古菌域以外的微生物中发现,也表明了其在进化上具有一定的特殊性[36]。
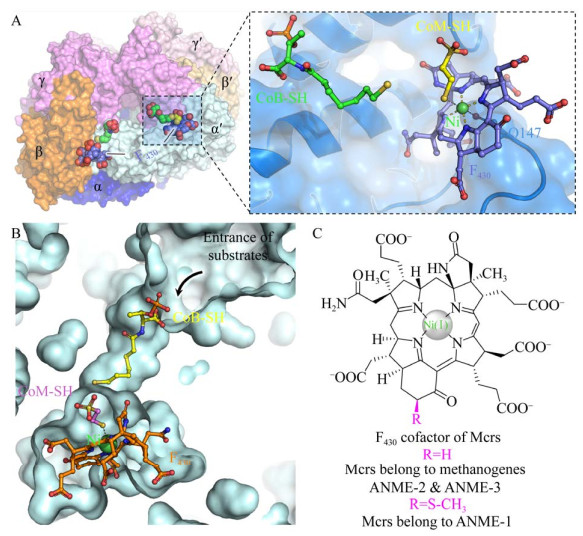
3 甲基-辅酶M还原酶的结构特性1997年,Ermler等首次解析了M. marburgensis McrⅠ复合体的三维结构,呈(αβγ)2构象,如图 3A所示[42]。在复合体中,α-/β-/γ-亚基和α’-/β’-/γ’-亚基分别构成一个催化单元,两个催化中心呈对称排布,相距约50 Å[42]。其中,构成每个催化单元的3个亚基紧密互联形成一个长约50 Å的疏水性通道(从复合体表面到活性中心)[42]。辅因子F430位于该狭长通道的底部,其金属中心镍(Ni) 呈6配位结构(图 3A)[42]。其中,α-谷氨酰胺(Glutamine,Q) 147和α’-Q147分别为(α’β’γ’) 和(αβγ) 中的Ni中心提供第5个配位(Ni的下方)[42]。Ni中心的第6个配位是底物的结合位点,位于Ni的上方,在非反应状态下该配位由水分子占据[42]。反应过程中,底物甲基-辅酶M (Methyl-coenzyme M) 经此疏水通道到达F430上方,硫(S) 结合于Ni位点[42]。如图 3B所示,由于整个疏水性通道呈上宽下窄的狭长孔道,当第2个底物CoB-SH进入反应活性中心后将牢牢锁住进入活性中心的通道[42, 50]。
|
| 图 3 Mcr (αβγ)2复合体、活性中心疏水性通道及辅因子F430的结构示意图[26, 36, 42-43, 54] Fig. 3 The structures of Mcr (αβγ)2 complex, hydrophobic channel within active center in Mcr and its cofactor F430[26, 36, 42-43, 54]. (A) Overview structure of Mcr (αβγ)2 from M. marburgensis. The hetero-hexamer consists of two active centers in which harbors F430 (purple), CoM-SH (yellow) and CoB-SH (green) (PDB code: 5A0Y). The only covalent bond of the Ni was Q147-O-Ni. The sixth coordination position of Ni was occupied by CoM-SH (yellow sticks). The electron donor CoM-SH (green stikcs) located on the upper of the F430 cofactor (purple sticks). (B) The hydrophobic channel (shown by light blue surface model) connected the protein surface and the deeply buried Ni site in which harbored F430 (orange), CoM-SH (pink) and CoB-SH (yellow) (PDB code: 5A0Y). (C) The chemical structure of Mcr cofactor F430 in methanogens/ANME-2/3 and ANME-1[36]. |
| |
由于Mcr活泼的金属Ni活性中心,目前尚未见活性状态的酶复合体晶体结构的报道[41]。Mcr催化中心金属Ni有Ni(Ⅰ)、Ni(Ⅱ)、Ni(Ⅲ) 三种氧化状态[42-43] (图 3C),但只有Ni(Ⅰ) 是活性价态[42-43]。Ni(Ⅰ) 极低的氧化还原电势(Ni(Ⅱ)/ Ni(Ⅰ)为–600至–700 mV) 极低[51-52],即使在严格厌氧的环境中(比如在95% N2/5% H2厌氧手套箱进行酶蛋白复合体结晶) Ni(Ⅰ)-F430也很容易被快速氧化为Ni(Ⅱ)-F430[36]。从目前所获得的Ni(Ⅱ)、Ni(Ⅲ) 状态的蛋白结构来看,该酶在整体构象及催化活性中心的结构均高度相似[36]。此外,研究者也在甲烷氧化古菌中获得了Mcr的蛋白晶体结构,发现其辅因子F430及活性位点的氨基酸存在部分修饰[26, 36, 53-54],可能与其执行甲烷氧化的功能相关[26, 36]。目前,已经鉴定的F430结构有9种,都是在Ni中心以外有部分的修饰,但是功能未知[36]。
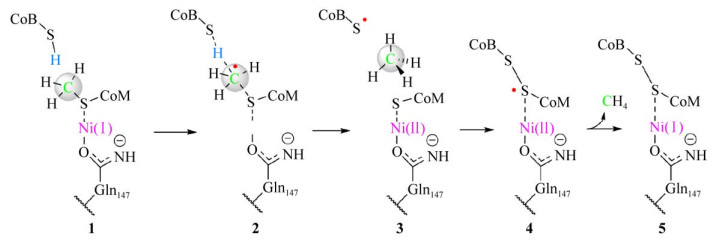
4 甲基-辅酶M还原酶的催化机制烷烃C-H键的裂解反应在化学上一直是一个很大的挑战[55],而Mcr能够克服热力学能障实现非活泼C-H键的厌氧裂解,因此其催化机制一直以来备受关注[34-36]。关于Mcr裂解C-H键的催化机理主要有两种假说[34-36]。第一种假说基于MCRox1-silent氧化态蛋白晶体结构的解析,该假说认为活性F430-Ni(Ⅰ) 亲核进攻CH3-S-CoM的甲基(CH3),从而形成CH3-Ni(Ⅲ)-F430中间过渡态复合体[42]。随后,中间过渡态复合体裂解产生甲烷(CH4) 和辅酶M-巯基自由基(CoM-S•)[42]。第二种假说在MCRox1-silent氧化态蛋白晶体结构的基础上结合了密度泛函理论计算(DFT),提出了甲基自由基催化机制[35]。如图 4所示,该假说认为活性F430-Ni(Ⅰ) 首先攻击CH3-S-CoM的硫(S) 导致甲基自由基(CH3•) 和CoM-S-Ni(Ⅱ)-F430过渡态复合体的形成[34-35, 56]。随后,CH3•自由基夺取电子供体辅酶B (CoB-SH) 巯基(-SH) 上的氢原子(H) 产生CH4和辅酶B巯基自由基(CoB-S•)[34-35]。伴随CH4的释放,CoB-S•与CoM-S-Ni(Ⅱ)-F430形成异二硫化物自由基阴离子([CoB-S-S-CoM]-)[34-35]。最后,CoB-S-S-CoM•的一个电子转移至Ni(Ⅱ)-F430重新形成Ni(Ⅰ)-F430,完成一个催化循环[34-35]。近年来,紫外/可见光谱(Ultraviolet-visible spectroscopy,UV-Vis)、电子磁共振波谱(Electron paramagnetic resonance spectroscopy,EPR)、圆二色谱(Circular dichroism spectroscopy,CD) 等都相继检测到了中间过渡态复合体CoM-S-Ni(Ⅱ)-F430,有力地支持了第二种假说[34-36]。另外,EPR和UV-Vis的检测数据显示,Mcr蛋白复合体的两个活性中心只有一个是处于活性状态,另一个是非活性的氧化状态[26, 35]。这一现象提示Mcr的两个活性中心可能是采用类似对置活塞式发动机的方式进行协同调控来完成催化反应[57-58]。
|
| 图 4 Mcr的甲基自由基催化机制示意图[26, 34-36, 59] Fig. 4 The proposed methyl radical catalytic mechanism of Mcr[26, 34-36, 59]. The reactive Ni(Ⅰ) attacked the S of methyl-S-CoM (1). Producing a methyl radical and a Ni(Ⅱ)-S-CoM intermediate (2). The methyl radical captured the H atom from the neighbored electron donor CoB-SH, generating a CH4 and a CoB thiyl radical (3). A heterodisulide thiyl radical was formed coupling with the release of CH4 (4). The Ni(Ⅰ) was regenerated by accepting one electron from the heterodisulide thiyl radical (5). The radical was marked as a closed-red cycle. The Ni-center was covalently adhered via the Gln147 in Mcr protein (PDB code: 5A0Y)[54]. |
| |
目前的催化机制研究主要是基于MCRox1-silent氧化态蛋白晶体结构分析和DFT计算,完整催化机制的进一步揭示还需要更多的实验性证据来支撑(比如获得MCRred1还原态蛋白晶体结构)[26, 36, 59]。当前,冷冻电子显微镜技术已经日渐成熟,特别是分辨率不断提升,为在近生理状态下捕捉Mcr复合体的活性状态提供了有力的技术手段[26, 36, 59]。同时,相比于传统的X-ray晶体技术,该技术将更是为捕捉Mcr催化过程的酶蛋白复合体构象的连续变化提供了契机[26, 36, 59]。
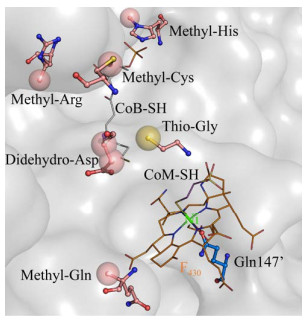
5 甲基-辅酶M还原酶的翻译后修饰Mcr在结构上的显著特点是活性中心附近的氨基酸位点出现不同程度的翻译后修饰,但是功能不详[26, 36, 60]。如图 5所示,目前获得的高分辨Mcr蛋白晶体结构及质谱数据显示,大部分产甲烷古菌的Mcr存在6种氨基酸位点的修饰,包括硫代甘氨酸(Thio-glycine)、1-N-甲基-组氨酸(1-N-methyl- histidine)、S-甲基-半胱氨酸(S-methyl-cysteine)、5-甲基-精氨酸(5-methyl-arginine)、2-甲基-谷氨酸(2-methyl-glutamine)、二氢天冬氨酸(Didehydro- aspartate)[26, 36, 54, 60-61]。特别是硫代甘氨酸和1-N-甲基-组氨酸这两种修饰高度保守,研究认为其与电子供体CoB-SH在催化活性中心的正确结合高度相关[60-61]。研究者还在Methanotorris formicicus和ANME-1中还发现了6-羟基色氨酸(6-hydroxy-tryptophane)和7-羟基色氨酸(7-hydroxy- tryptophane) 的修饰位点[26, 49, 53]。目前报道的氨基酸翻译后修饰位点都位于McrA亚基的活性中心区域,研究者推测它们很可能与催化反应的进行密切相关[26, 36, 54]。最近,有研究者对负责甲基精氨酸、甲基半胱氨酸、硫代甘氨酸修饰的基因做了敲除,但是实验显示这些修饰位点的去除并未显著地影响Mcr的反应活性[62-64]。当然,这并不排除其是否在特殊生理条件下发挥功能[36]。2018年,Lyu等首次成功地完成了来源于甲烷热球菌Methanothermococcus okinawensis的Mcr复合体在海藻甲烷球菌Methanococcus maripaludis中的异源表达,并发现其在体内能自组装为完整的Mcr复合体[65],这为将来通过定点突变来研究氨基酸位点修饰与催化机制包括进行定向改造提供了有力的技术手段。
|
| 图 5 Mcr活性中心氨基酸修饰示意图[26, 36, 54] Fig. 5 Modified amino acids in the active center of Mcr[26, 36, 54]. Mcr from M. marburgensis contains the thio-glycine, 1-N-methyl-histidine, S-methyl-cysteine, 5-methyl-arginine, 2-methyl-glutamine, and didehydro- aspartate (highlighted by spheres)[26, 36]. Additionally, Mcr from M. formicicus and an ANME-1 archaeon contains 6- and 7-hydroxy-tryptophane, respectively[26, 49, 53]. CoB-SH, CoM-SH and F430 were shown as lines. Ni site bond to the protein via Gln147' shown by sticks and balls. |
| |
近年来有研究发现一些未培养海洋古菌能够代谢一些非甲烷气态短链烷烃(包括乙烷、丙烷和丁烷),它们通过类似Mcr的Mcr-like催化烷烃的起始厌氧降解[10-15]。2016年,Laso-Pérez等在海底沉积层富集物中发现了未培养古菌Candidatus Syntrophoarchaeum能够厌氧降解丁烷[14]。Ca. Syntrophoarchaeum编码的Mcr-like,具有极高的表达量,代谢产物分析检测到关键中间代谢产物丁基-辅酶M[14]。研究者推测它是利用类似厌氧甲烷氧化的降解机制降解丁烷[14]。2019年,Chen等在海洋烃渗漏区域富集物中发现了高丰度的未培养古菌Candidatus Argoarchaeum ethanivorans,该菌能够厌氧降解乙烷,含有编码Mcr-like的基因簇,并在蛋白提取物中检测到该Mcr-like的所有蛋白亚基[15];同时,代谢产物分析也检测到关键中间代谢产物乙基-辅酶M,推测该菌也是通过Mcr-like介导的途径进行乙烷的厌氧降解[15]。随后,Laso-Pérez等在烃渗漏区富集物中发现未培养古菌Candidatus Methanoliparia独立附着于油滴而生存,并且在全球大量的油气藏都检测到该菌的存在[13];Ca. Methanoliparia的基因组信息显示该菌能够编码完整的产甲烷代谢途径和古菌的烷烃厌氧降解途径,包括负责甲烷产生的Mcr和负责烷烃激活的Mcr-like,推测该菌不仅能激活烷烃的厌氧降解,还能将中间产物用于产甲烷代谢产甲烷[13]。
目前,我们的研究团队也在胜利油田的深层地下油田环境中获得了具有厌氧石油烃降解能力的产甲烷古菌Candidatus Methanoliparum sp.高度富集物(未发表)。有意思的是该Ca. Methanoliparum sp.具备两个重要的特性:一是它能够厌氧降解长碳链的烷烃,如C16-C20系列的烷烃,这是继C2-C4烷烃厌氧降解后的首次发现(未发表);二是它具备独立降解长碳链的烷烃,并能将其完全转化为甲烷和CO2,不同于已发现的非甲烷气态短链烷烃降解型古菌需要硫酸盐/硝酸盐还原菌的协助共同完成烷烃的完全降解(未发表)。通过宏组学和代谢产物分析,我们发现该菌的长链烷烃的厌氧降解也是由Mcr-like激活并驱动的,具备Mcr-like依赖性的烷烃激活代谢模块、脂肪酸β氧化和产甲烷代谢模块(未发表),这使其能够克服热力学能障并平衡胞内还原力而独立完全降解烷烃。
近年来,研究者非常关注这些来自油污区的海洋古菌,它们能厌氧氧化乙烷、丙烷、丁烷等,其基因组均含有mcr和mcr-like基因簇[10-15]。遗憾的是这些新型海洋古菌的分离极具挑战性,目前还没有获得纯培养物,无法进行更为深入的研究。目前对Mcr-like的研究也仅仅停留在基因、生理水平,还未对其结构、功能、催化机理进行深入的解析。从报道来看,无论是甲烷氧化型古菌,还是新发现的乙烷/丙烷/丁烷氧化型古菌,包括我们正在研究的长链烷烃氧化型古菌Ca. Methanoliparum sp. (对于碳链的长度有很大的偏好性),都有极强的底物选择性[10-15, 36]。或许通过对新古菌来源的Mcr/Mcr-like蛋白结构及催化机制的研究能回答Mcr/Mcr-like是如何调控自身的双向催化功能(还原或氧化)?为何有如此强的底物特异性? 是如何调控的?
7 总结与展望从Mcr的首次发现至今已经50余年,但研究者对它的研究热情一直不减[26, 36]。特别是最新发现催化烷烃C-H键厌氧氧化的Mcr-like,更是掀起了对该类酶特殊C-H键裂解机理的研究热潮[10-15, 66]。为了能够将Mcr/Mcr-like应用于生物采油、油气转换或温室气体排放控制等,结合最新的生物物理、基因编辑、定向进化与量子计算等技术对其结构、功能和反应机理的进一步深入研究仍十分必要[26, 36]。就现阶段而言,以下几个方面的问题亟需解决:(1) Mcr/Mcr-like还原态(活性状态) 的蛋白结构的获取,这是研究其催化机制及底物偏好性(代谢不同碳链长度烷烃) 的基础。目前分辨率不断提升的冷冻电子显微镜技术[67-68]有望解决这一难题。(2) 高效且易操作的产甲烷古菌遗传操作系统的建立,这是研究其转录后修饰及人工改造的基础。目前的新型基因编辑技术[69-70]有望解决这一难点。(3) 从富集物中获得新型烷烃厌氧氧化古菌的纯培养物,这是研究这类新型古菌代谢途径及Mcr-like生理功能的基础。目前基于膜扩散、微流控及细胞分选的新型培养技术[71]有望解决这一问题。
| [1] |
Lyu Z, Shao NN, Akinyemi T, et al. Methanogenesis. Curr Biol, 2018, 28(13): R727-R732. DOI:10.1016/j.cub.2018.05.021
|
| [2] |
Haynes CA, Gonzalez R. Rethinking biological activation of methane and conversion to liquid fuels. Nat Chem Biol, 2014, 10(5): 331-339. DOI:10.1038/nchembio.1509
|
| [3] |
Offre P, Spang A, Schleper C. Archaea in biogeochemical cycles. Annu Rev Microbiol, 2013, 67: 437-457. DOI:10.1146/annurev-micro-092412-155614
|
| [4] |
Hua ZS, Wang YL, Evans PN, et al. Insights into the ecological roles and evolution of methyl-coenzyme M reductase-containing hot spring Archaea. Nat Commun, 2019, 10: 4574. DOI:10.1038/s41467-019-12574-y
|
| [5] |
Makogon YF. Natural gas hydrates—a promising source of energy. J Natll Gas Sci Eng, 2010, 2(1): 49-59. DOI:10.1016/j.jngse.2009.12.004
|
| [6] |
Timmers PHA, Welte CU, Koehorst JJ, et al. Reverse methanogenesis and respiration in methanotrophic archaea. Archaea, 2017, 2017: 1654237.
|
| [7] |
Thauer RK, Kaster AK, Seedorf H, et al. Methanogenic archaea: ecologically relevant differences in energy conservation. Nat Rev Microbiol, 2008, 6(8): 579-591. DOI:10.1038/nrmicro1931
|
| [8] |
Wagner A, Whitaker RJ, Krause DJ, et al. Mechanisms of gene flow in archaea. Nat Rev Microbiol, 2017, 15(8): 492-501. DOI:10.1038/nrmicro.2017.41
|
| [9] |
Evans PN, Boyd JA, Leu AO, et al. An evolving view of methane metabolism in the archaea. Nat Rev Microbiol, 2019, 17(4): 219-232. DOI:10.1038/s41579-018-0136-7
|
| [10] |
Wang YZ, Wegener G, Hou JL, et al. Expanding anaerobic alkane metabolism in the domain of archaea. Nat Microbiol, 2019, 4(4): 595-602. DOI:10.1038/s41564-019-0364-2
|
| [11] |
Wang YZ, Wegener G, Ruff SE, et al. Methyl/alkyl-coenzyme M reductase-based anaerobic alkane oxidation in archaea. Environ Microbiol, 2020, 23(2): 530-541.
|
| [12] |
Hahn CJ, Laso-Pérez R, Vulcano F, et al. "Candidatus Ethanoperedens" a thermophilic genus of archaea mediating the anaerobic oxidation of ethane. mBio, 2020, 11(2): e00600-20.
|
| [13] |
Laso-Pérez R, Hahn C, Van Vliet DM, et al. Anaerobic degradation of non-methane alkanes by "Candidatus Methanoliparia" in hydrocarbon seeps of the gulf of Mexico. mBio, 2019, 10(4): e01814-19.
|
| [14] |
Laso-Pérez R, Wegener G, Knittel K, et al. Thermophilic archaea activate butane via alkyl-coenzyme M formation. Nature, 2016, 539(7629): 396-401. DOI:10.1038/nature20152
|
| [15] |
Chen SC, Musat N, Lechtenfeld OJ, et al. Anaerobic oxidation of ethane by archaea from a marine hydrocarbon seep. Nature, 2019, 568(7750): 108-111. DOI:10.1038/s41586-019-1063-0
|
| [16] |
Enzmann F, Mayer F, Rother M, et al. Methanogens: biochemical background and biotechnological applications. AMB Expr, 2018, 8(1): 1-22. DOI:10.1186/s13568-017-0531-x
|
| [17] |
Ferry JG. Methanogenesis: ecology, physiology, biochemistry & genetics. Dordrecht: Springer Science & Business Media, 2012.
|
| [18] |
Thauer RK. The Wolfe cycle comes full circle. Proc Natl Acad Sci USA, 2012, 109(38): 15084-15085. DOI:10.1073/pnas.1213193109
|
| [19] |
Wagner T, Ermler U, Shima S. The methanogenic CO2 reducing-and-fixing enzyme is bifunctional and contains 46[4Fe-4S] clusters. Science, 2016, 354(6308): 114-117. DOI:10.1126/science.aaf9284
|
| [20] |
Donnelly MI, Wolfe RS. The role of formylmethanofuran: tetrahydromethanopterin formyltransferase in methanogenesis from carbon dioxide. J Biol Chem, 1986, 261(35): 16653-16659. DOI:10.1016/S0021-9258(18)66615-3
|
| [21] |
Breitung J, Thauer RK. Formylmethanofuran: tetrahydromethanopterin formyltransferase from Methanosarcina barkeri identification of N5-formyltetrahydromethanopterin as the product. FEBS Lett, 1990, 275(1/2): 226-230.
|
| [22] |
DiMarco AA, Donnelly MI, Wolfe RS. Purification and properties of the 5, 10-methenyltetrahydromethanopterin cyclohydrolase from Methanobacterium thermoautotrophicum. J Bacteriol, 1986, 168(3): 1372-1377. DOI:10.1128/jb.168.3.1372-1377.1986
|
| [23] |
Te Brömmelstroet B, Hensgens C, Geerts W, et al. Purification and properties of 5, 10-methenyltetrahydromethanopterin cyclohydrolase from Methanosarcina barkeri. J Bacteriol, 1990, 172(2): 564-571. DOI:10.1128/jb.172.2.564-571.1990
|
| [24] |
Hagemeier CH, Shima S, Thauer RK, et al. Coenzyme F420-dependent methylenetetrahydromethanopterin dehydrogenase (Mtd) from Methanopyrus kandleri: a methanogenic enzyme with an unusual quarternary structure. J Mol Biol, 2003, 332(5): 1047-1057. DOI:10.1016/S0022-2836(03)00949-5
|
| [25] |
Aufhammer SW, Warkentin E, Ermler U, et al. Crystal structure of methylenetetrahydromethanopterin reductase (Mer) in complex with coenzyme F420: architecture of the F420/FMN binding site of enzymes within the nonprolyl cis-peptide containing bacterial luciferase family. Protein Sci, 2005, 14(7): 1840-1849. DOI:10.1110/ps.041289805
|
| [26] |
Shima S, Huang GF, Wagner T, et al. Structural basis of hydrogenotrophic methanogenesis. Annu Rev Microbiol, 2020, 74: 713-733. DOI:10.1146/annurev-micro-011720-122807
|
| [27] |
Wagner T, Koch J, Ermler U, et al. Methanogenic heterodisulfide reductase (HdrABC-MvhAGD) uses two noncubane [4Fe-4S] clusters for reduction. Science, 2017, 357(6352): 699-703. DOI:10.1126/science.aan0425
|
| [28] |
Thauer RK, Kaster AK, Goenrich M, et al. Hydrogenases from methanogenic archaea, nickel, a novel cofactor, and H2 storage. Annu Rev Biochem, 2010, 79: 507-536. DOI:10.1146/annurev.biochem.030508.152103
|
| [29] |
Conrad R. Importance of hydrogeno-trophic, aceticlastic and methylotrophic methanogenesis for methane production in terrestrial, aquatic and other anoxic environments: a mini review. Pedosphere, 2020, 30(1): 25-39. DOI:10.1016/S1002-0160(18)60052-9
|
| [30] |
Söllinger A, Urich T. Methylotrophic methanogens everywhere—physiology and ecology of novel players in global methane cycling. Biochemical Society Transactions, 2019, 47(6): 1895-1907. DOI:10.1042/BST20180565
|
| [31] |
Adam PS, Borrel G, Gribaldo S. Evolutionary history of carbon monoxide dehydrogenase/acetyl-CoA synthase, one of the oldest enzymatic complexes. Proc Natl Acad Sci USA, 2018, 115(6): E1166-E1173. DOI:10.1073/pnas.1716667115
|
| [32] |
Cohen SE, Can M, Wittenborn EC, et al. Crystallographic characterization of the carbonylated a-cluster in carbon monoxide dehydrogenase/acetyl-CoA synthase. ACS Catalysis, 2020, 10(17): 9741-9746. DOI:10.1021/acscatal.0c03033
|
| [33] |
Ragsdale SW. Biochemistry of methyl-coenzyme M reductase: the nickel metalloenzyme that catalyzes the final step in synthesis and the first step in anaerobic oxidation of the greenhouse gas methane//Kroneck P, Torres M, Eds. The Metal-Driven Biogeochemistry of Gaseous Compounds in the Environment. Dordrecht: Springer, 2014: 125-145.
|
| [34] |
Shima S. The biological methane-forming reaction: mechanism confirmed through spectroscopic characterization of a key intermediate. Angew Chem Int Ed, 2016, 55(44): 13648-13649. DOI:10.1002/anie.201606269
|
| [35] |
Wongnate T, Sliwa D, Ginovska B, et al. The radical mechanism of biological methane synthesis by methyl-coenzyme M reductase. Science, 2016, 352(6288): 953-958. DOI:10.1126/science.aaf0616
|
| [36] |
Thauer RK. Methyl (Alkyl)-coenzyme M reductases: nickel F-430-containing enzymes involved in anaerobic methane formation and in anaerobic oxidation of methane or of short chain alkanes. Biochemistry, 2019, 58(52): 5198-5220. DOI:10.1021/acs.biochem.9b00164
|
| [37] |
Hallam SJ, Putnam N, Preston CM, et al. Reverse methanogenesis: testing the hypothesis with environmental genomics. Science, 2004, 305(5689): 1457-1462. DOI:10.1126/science.1100025
|
| [38] |
Krüger M, Meyerdierks A, Glöckner FO, et al. A conspicuous nickel protein in microbial mats that oxidize methane anaerobically. Nature, 2003, 426(6968): 878-881. DOI:10.1038/nature02207
|
| [39] |
Michaelis W, Seifert R, Nauhaus K, et al. Microbial reefs in the Black Sea fueled by anaerobic oxidation of methane. Science, 2002, 297(5583): 1013-1015. DOI:10.1126/science.1072502
|
| [40] |
Nauhaus K, Boetius A, Krüger M, et al. In vitro demonstration of anaerobic oxidation of methane coupled to sulphate reduction in sediment from a marine gas hydrate area. Environ Microbiol, 2002, 4(5): 296-305. DOI:10.1046/j.1462-2920.2002.00299.x
|
| [41] |
Hallam SJ, Girguis PR, Preston CM, et al. Identification of methyl coenzyme M reductase A (mcrA) genes associated with methane-oxidizing archaea. Appl Environ Microbiol, 2003, 69(9): 5483-5491. DOI:10.1128/AEM.69.9.5483-5491.2003
|
| [42] |
Ermler U, Grabarse W, Shima S, et al. Crystal structure of methyl-coenzyme M reductase: the key enzyme of biological methane formation. Science, 1997, 278(5342): 1457-1462. DOI:10.1126/science.278.5342.1457
|
| [43] |
Shima S, Goubeaud M, Vinzenz D, et al. Crystallization and preliminary X-ray diffraction studies of methyl-coenzyme M reductase from Methanobacterium thermoautotrophicum. J Biochem, 1997, 121(5): 829-830. DOI:10.1093/oxfordjournals.jbchem.a021660
|
| [44] |
Goubeaud M, Schreiner G, Thauer RK. Purified methyl-coenzyme-M reductase is activated when the enzyme-bound coenzyme F430 is reduced to the nickel(Ⅰ) oxidation state by titanium(Ⅲ) citrate. Eur J Biochem, 1997, 243(1/2): 110-114.
|
| [45] |
Bonacker LG, Baudner S, Thauer RK. Differential expression of the two methyl-coenzyme M reductases in Methanobacterium thermoautotrophicum as determined immunochemically via isoenzyme-specific antisera. Eur J Biochem, 1992, 206(1): 87-92. DOI:10.1111/j.1432-1033.1992.tb16904.x
|
| [46] |
Bonacker LG, Baudner S, Mörschel E, et al. Properties of the two isoenzymes of methyl-coenzyme M reductase in Methanobacterium thermoautotrophicum. Eur J Biochem, 1993, 217(2): 587-595. DOI:10.1111/j.1432-1033.1993.tb18281.x
|
| [47] |
Huber H, Thomm M, König H, et al. Methanococcus thermolithotrophicus, a novel thermophilic lithotrophic methanogen. Arch Microbiol, 1982, 132(1): 47-50. DOI:10.1007/BF00690816
|
| [48] |
Jones WJ, Leigh JA, Mayer F, et al. Methanococcus jannaschii sp. nov., an extremely thermophilic methanogen from a submarine hydrothermal vent. Arch Microbiol, 1983, 136(4): 254-261. DOI:10.1007/BF00425213
|
| [49] |
Wagner T, Wegner CE, Kahnt J, et al. Phylogenetic and structural comparisons of the three types of methyl coenzyme M reductase from Methanococcales and Methanobacteriales. J Bacteriol, 2017, 199(16): e00197-17.
|
| [50] |
Grabarse W, Mahlert F, Duin EC, et al. On the mechanism of biological methane formation: structural evidence for conformational changes in methyl-coenzyme M reductase upon substrate binding. J Mol Biol, 2001, 309(1): 315-330. DOI:10.1006/jmbi.2001.4647
|
| [51] |
Jaun B. Coenzyme F430 from methanogenic bacteria: oxidation of F430 pentamethyl ester to the Ni(Ⅲ) form. Helv Chim Acta, 1990, 73(8): 2209-2217. DOI:10.1002/hlca.19900730818
|
| [52] |
Jaun B, Pfaltz A. Coenzyme F430 from methanogenic bacteria: reversible one-electron reduction of F430 pentamethyl ester to the nickel (Ⅰ) form. J Chem Soc Chem Commun, 1986(17): 1327-1329. DOI:10.1039/c39860001327
|
| [53] |
Shima S, Krueger M, Weinert T, et al. Structure of a methyl-coenzyme M reductase from Black Sea mats that oxidize methane anaerobically. Nature, 2011, 481(7379): 98-101.
|
| [54] |
Wagner T, Kahnt J, Ermler U, et al. Didehydroaspartate modification in methyl-coenzyme M reductase catalyzing methane formation. Angew Chem Int Ed, 2016, 55(36): 10630-10633. DOI:10.1002/anie.201603882
|
| [55] |
Francis C, Robert G. Organic Chemistry. 11th ed. New York: McGraw-Hill Science, 2020.
|
| [56] |
Pelmenschikov V, Blomberg MRA, Siegbahn PEM, et al. A mechanism from quantum chemical studies for methane formation in methanogenesis. J Am Chem Soc, 2002, 124(15): 4039-4049. DOI:10.1021/ja011664r
|
| [57] |
Goenrich M, Duin EC, Mahlert F, et al. Temperature dependence of methyl-coenzyme M reductase activity and of the formation of the methyl-coenzyme M reductase red2 state induced by coenzyme B. J Biol Inorg Chem, 2005, 10(4): 333-342. DOI:10.1007/s00775-005-0636-6
|
| [58] |
Ebner S, Jaun B, Goenrich M, et al. Binding of coenzyme B induces a major conformational change in the active site of methyl-coenzyme M reductase. J Am Chem Soc, 2010, 132(2): 567-575. DOI:10.1021/ja906367h
|
| [59] |
Huang GF, Wagner T, Ermler U, et al. Methanogenesis involves direct hydride transfer from H2 to an organic substrate. Nat Rev Chem, 2020, 4(4): 213-221. DOI:10.1038/s41570-020-0167-2
|
| [60] |
Chen H, Gan QL, Fan CG. Methyl-coenzyme M reductase and its post-translational modifications. Front Microbiol, 2020, 11: 578356. DOI:10.3389/fmicb.2020.578356
|
| [61] |
Selmer T, Kahnt J, Goubeaud M, et al. The biosynthesis of methylated amino acids in the active site region of methyl-coenzyme M reductase. J Biol Chem, 2000, 275(6): 3755-3760. DOI:10.1074/jbc.275.6.3755
|
| [62] |
Deobald D, Adrian L, Schöne C, et al. Identification of a unique radical SAM methyltransferase required for the sp3-C-methylation of an arginine residue of methyl-coenzyme M reductase. Sci Rep, 2018, 8: 7404. DOI:10.1038/s41598-018-25716-x
|
| [63] |
Nayak DD, Liu AD, Agrawal N, et al. Functional interactions between posttranslationally modified amino acids of methyl-coenzyme M reductase in Methanosarcina acetivorans. PLoS Biol, 2020, 18(2): e3000507. DOI:10.1371/journal.pbio.3000507
|
| [64] |
Nayak DD, Mahanta N, Mitchell DA, et al. Post-translational thioamidation of methyl-coenzyme M reductase, a key enzyme in methanogenic and methanotrophic archaea. Elife, 2017, 6: e29218. DOI:10.7554/eLife.29218
|
| [65] |
Lyu Z, Chou CW, Shi H, et al. Assembly of methyl coenzyme M reductase in the methanogenic archaeon Methanococcus maripaludis. J Bacteriol, 2018, 200(7): e00746-17.
|
| [66] |
Singh R, Guzman MS, Bose A. Anaerobic oxidation of ethane, propane, and butane by marine microbes: a mini review. Front Microbiol, 2017, 8: 2056. DOI:10.3389/fmicb.2017.02056
|
| [67] |
De Oliveira TM, Van Beek L, Shilliday F, et al. Cryo-EM: the resolution revolution and drug discovery. SLAS Discov: Advanc Sci Drug Discov, 2020, 26(1): 17-31.
|
| [68] |
Nakane T, Kotecha A, Sente A, et al. Single-particle cryo-EM at atomic resolution. Nature, 2020, 587(7832): 152-156. DOI:10.1038/s41586-020-2829-0
|
| [69] |
Knott GJ, Doudna JA. CRISPR-Cas guides the future of genetic engineering. Science, 2018, 361(6405): 866-869. DOI:10.1126/science.aat5011
|
| [70] |
Gophna U, Allers T, Marchfelder A. Finally, archaea get their CRISPR-Cas toolbox. Trends Microbiol, 2017, 25(6): 430-432. DOI:10.1016/j.tim.2017.03.009
|
| [71] |
Lewis WH, Tahon G, Geesink P, et al. Innovations to culturing the uncultured microbial majority. Nat Rev Microbiol, 2020. DOI:10.1038/s41579-020-00458-8
|
 2021, Vol. 37
2021, Vol. 37