中国科学院微生物研究所、中国微生物学会主办
文章信息
- 范婷婷, 王慕瑶, 李俊, 王风楼, 章漳, 赵心清
- Fan Tingting, Wang Muyao, Li Jun, Wang Fenglou, Zhang Zhang, Zhao Xin-Qing
- 酵母生物多样性开发及工业应用
- Exploration of yeast biodiversity and development of industrial applications
- 生物工程学报, 2021, 37(3): 806-815
- Chinese Journal of Biotechnology, 2021, 37(3): 806-815
- 10.13345/j.cjb.200650
-
文章历史
- Received: October 11, 2020
- Accepted: January 5, 2021
- Published: March 15, 2021
2. 伽蓝(集团)股份有限公司,上海 200233
2. JALA Group. Co., Ltd., Shanghai 200233, China
酵母是一类可以通过出芽或者细胞分裂进行无性生殖的真核生物,其有性生殖状态没有子实体(Fruiting bodies)。截至目前,出版的酵母分类专著收录了149个属和将近1 500个种[1],酵母分常规酵母(包括酿酒酵母和裂殖酵母两个属) 和非常规酵母(除了常规酵母外的所有其他酵母)两大类。酿酒酵母是所有酵母中应用最广泛的,常见到把酿酒酵母等同于酵母来论述,但是酵母包含的种类要宽泛很多,因此应该注意严谨区分。
酿酒酵母广泛用于生物医药、食品、轻工业和生物能源等领域,生产重组蛋白、天然活性物质、有机酸和燃料乙醇等产品。非常规酵母包括毕赤酵母、假丝酵母、克鲁维酵母等,目前这些酵母也在不断被开发利用,具有越来越广泛的应用前景。不同酵母具有不同的生理特性和应用价值,比如酿酒酵母具有较好的乙醇发酵能力,有些酵母具备产生香气物质和活性化合物的能力,可用于食品和发酵产品风味改善等[2]。值得注意的是,虽然目前发现了非常多的酵母种,但是工业应用的酵母种类还很少,因此酵母的开发利用具有很大的潜力。
目前为止从植物或土壤等不同生态环境分离获得了很多天然酵母,但是很多植物和动物,包括人的肠道内部也生存有大量的酵母[3-4],对不同来源的天然酵母进行分离鉴定,不仅有利于生物资源调查和保护,以及研究自然进化和生态环境适应性,也有利于进一步开发利用这些酵母资源进行多种工业应用。此外,酵母是代谢工程改造的常用宿主[5],开发酵母遗传资源,包括关键酶基因、调控基因、启动子和信号肽编码基因等,对于提高菌株性能也非常重要。
酵母的天然生物多样性主要包括物种的多样性和遗传多样性,是适应环境和进化选择的结果,而利用突变、融合和定向设计等手段,可以产生或者筛选新的非天然多样性,包括具有所需要的生理代谢特征的新菌株和新功能的遗传元件,这些非天然多样性对提高酵母的工业应用效率具有重要意义。
本文综述了酵母菌株生物多样性的研究现状,以及天然多样性和非天然多样性的开发利用,为促进酵母在不同工业领域的应用提供借鉴,也为研究其他工业微生物菌株选育提供参考。
1 酵母天然生物多样性 1.1 酵母的种水平多样性随着微生物分类鉴定技术的不断推广和进步,酵母的新种不断被发现。根据笔者调查,2019年发表在国际权威微生物分类学期刊International Journal of Systematic and Evolutionary Microbiology上的酵母新种有28个,2020年我国学者在Studies in Mycology期刊上发表了107个分离自植物叶片和土壤的担子菌酵母新种,其中46个属于伞菌亚门Tremellomycetes (Agaricomycotina) 中的16个属,61个属于锈菌亚门Pucciniomycotina中的26个属[6]。可以预见,更多新的酵母将不断被分离鉴定。近期报道的新种包括Ke等[7]从中国云南省西双版纳热带雨林的腐烂木材中分离出的5种酵母,其中有两个是Kazachstania属的新种。Gao等[8]从中国河南省宝天满自然保护区分离出4种酵母菌,其中有两株是毕赤酵母属的新种。Avchar等[9]从一个酿酒工厂的废液中分离出来了一株Wickerhamiella属的酵母新种W. shivajii sp. nov.,该酵母可以在42 ℃条件下生长,是Wickerhamiella属中最耐热的酵母物种。随着越来越多的酵母新种被发现,酵母天然资源将被进一步利用,酵母的工业应用也将得到扩展。
1.2 酵母的菌株多样性在酵母的进化过程中,种群会随着生态环境的变化而进化以适应变化的生态环境,使其自身具有更强的生存能力。大规模的菌株基因组的挖掘和分析对认识菌株多样性和更好地利用开发菌株的潜力具有重要的意义。研究者对1 011个天然酿酒酵母菌株进行了基因组测序和分析,对其中971个菌株进行了不同生理条件下的表型分析,发现单核苷酸多态性的变异频率比较低,而且拷贝数变异比单核苷酸多态性的变异对表型的影响更大[10]。另外,对157个工业酿酒酵母(其中包含107个啤酒酵母) 的基因组和表型进行了分析,结果表明,工业酿酒酵母的表型和野生酿酒酵母相差甚远,相对于野生酵母,工业酿酒酵母对于糖的利用、耐受性以及风味物质的产生等方面表现出更多的进化,麦芽糖利用基因MAL的拷贝数增加,啤酒酵母在非发酵环境相关的胁迫条件下生存较差,而葡萄酒酵母耐性较好,体现了高糖和高乙醇环境在长期工业应用中对菌株驯化选择的结果[11]。以上对于天然和工业酵母大量基因图谱的研究可对我们进一步选择和利用酵母资源提供指导性的意义。我国中国科学院微生物研究所白逢彦团队的研究揭示了我国不仅是酿酒酵母野生群体的发源地,也是驯养酿酒酵母的起源中心,通过基因拷贝数扩增、删减、基因水平转移等方式,产生了显著的基因组变异,进一步实现发酵和对逆境环境抵抗能力的提高,以及对不同发酵环境的适应性进化[12]。纤维素乙醇生产过程中,发酵微生物经常面临很多环境胁迫条件的抑制,包括高温、乙醇、以及水解液中的乙酸等抑制物等。提高酵母菌对这些逆境环境的耐受性,有利于保持良好的发酵活性,提高发酵效率[13]。值得指出的是,同一个酵母菌种的不同菌株可能有很不相同的特征。笔者课题组研究发现,分离自甘蔗渣的酿酒酵母菌株YB-2625能很好利用混合糖中的木糖,而酿酒酵母长期以来普遍认为不具有木糖利用能力,这个研究结果启发研究者去发现天然菌株不同于模式菌株的代谢新潜力,包括碳源利用特性、逆境胁迫耐受性和新产品生产的性能等[14]。
2 酵母遗传和生理代谢多样性自然界中存在的野生酵母具有丰富的遗传多样性,例如,我国学者研究表明,野生酿酒酵母菌株的遗传组成存在较大的差异,在进化上,与人类关系密切和与人类活动关系较远的酵母存在比较鲜明的进化距离,同时,我国野生酵母的遗传多样性在全世界范围内是最丰富的[15]。该研究也提示,对我国丰富的野生酵母遗传资源进行深入分析,有利于更好利用这些菌株。例如,可通过基因组测序挖掘酵母中和蛋白分泌相关的信号肽[16],挖掘基因组中的代谢酶[17]和启动子[18]等。比利时学者对大量的非常规酵母进行多种纤维素乙醇发酵相关耐受性分析,选择了12个耐性好的非常规酵母进行发酵,发现5株酵母具有比较好的发酵特性,其中Pichia kudriavzevii和Wickerhamomyces anomalus具有非常好的性能[19]。该研究和其他相关研究都提示,应该重视非常规酵母在纤维素乙醇生产等多种领域的应用。对天然菌株中多样的遗传元件进行挖掘和分析,有利于更好地利用这些元件进行代谢工程改造,提高菌株的代谢效率。
3 酵母的生物多样性改造虽然天然菌株可能具备比较多样的代谢特征和比较好的抗逆性,但往往天然菌株的性能无法很好满足工业应用需要,因此需要通过不同手段进行菌株选育,获得突变菌株,人类也在利用各种方法对酵母菌株进行改造,使之具有更好的性能。
3.1 随机诱变酵母菌株的非天然多样性可以通过随机手段和定向改造的手段获得,其中随机诱变可使菌株产生多种表型,有利于大量筛选目的菌株。利用大气压和室温等离子体(Atmospheric room temperature plasma,ARTP) 进行菌株随机诱变,可很方便地大量获得多样的突变菌株,而且适用于多种微生物的选育[20]。利用全自动高通量微生物培养仪(Microbial microdroplet culture system,MMC) 可方便进行定向驯化[21],而高通量筛选技术的发展,有利于快速从随机诱变获得的大量突变体中获得所需要的表型[22]。
3.2 代谢工程改造代谢工程改造是理性育种的手段,其中酿酒酵母代谢工程改造近年来取得了飞速进展,包括启动子工程、终止子工程、酶工程、转录因子工程、蛋白质工程等[23]。非常规酵母的代谢工程也在不断成熟,其中研究比较多的非常规酵母包括解脂耶氏酵母和毕赤酵母等,其代谢工程改造技术都取得了明显进展[24-25]。通过代谢工程改造,可以赋予酵母水解纤维素的能力[26],和利用非天然碳源如木糖的能力[27],以及生产疫苗和异源合成天然活性产物的能力等[28-30],扩大了酵母的生理性能的多样性。
除了扩大酵母的代谢潜能,代谢工程也用于提高酵母对多种工业发酵条件的抗逆性[31]。乙酸是木质纤维素水解液中的主要毒性抑制物,高浓度乙酸会对酵母的生长和发酵能力产生抑制作用,因此,提高酵母的乙酸耐性,可以使酵母更好地用于木质纤维素类生物质的生物炼制。笔者课题组致力于酵母用于木质纤维素的水解以及提高酵母乙酸耐性的研究,并发现多个基因和蛋白与乙酸耐性有关,揭示了絮凝和发酵液中添加适当硫酸锌可以提高酵母的乙酸耐性[32],过表达组蛋白H4甲基化酶Set5p可以提高酵母的乙酸耐性[33],过表达酵母中的谷氧还蛋白编码基因GRX5可以促使细胞产生更多的海藻糖和甘油等保护物质以应对外界乙酸胁迫条件[34],过表达参与嘌呤从头合成的3个基因ADE1、ADE3和ADE17可以提高酿酒酵母在乙酸胁迫条件下的乙醇产率[35]。
3.3 合成生物学改造合成生物学设计为酵母菌株多样性的扩大提供了更多的可能性。合成生物学在酿酒酵母中的应用相对其他酵母中更成熟,开发了多样的工具箱[36]。我国研究者参加并作出重要贡献的酿酒酵母基因组合成计划是合成生物学代表性工作[37],在合成型酿酒酵母基因组基础上开展的基因组重排研究,即基于loxP位点特异性重组的合成型基因组重排系统(Synthetic chromosome rearrangement and modification by LoxPsym-mediated evolution,SCRaMbLE),大大加速了酿酒酵母进化速度,可用于获得具有更高活性物质合成能力或者更强抗逆性的菌株[38],也有利于对酵母细胞工厂高效运作机理的分析。
另外,通过对特定酶基因进行随机突变或者定点突变可以提高催化性能。例如,研究者对来自细菌的羧酸还原酶通过定向进化、结构指导的半理性设计及理性设计等优化获得了催化活性和底物特异性都提高的突变酶,表达突变酶的酿酒酵母合成中链脂肪醇的产量比表达未改造酶的出发菌株提高了2.8倍[39]。类似方法也可以用于改造酵母来源的酶基因等遗传元件,获得优良的突变遗传元件。基于天然的酵母遗传元件,也可以利用合成生物学设计新的元件,例如设计胁迫响应的启动子元件等[40]。图 1总结了酵母多样化改造的研究方法。
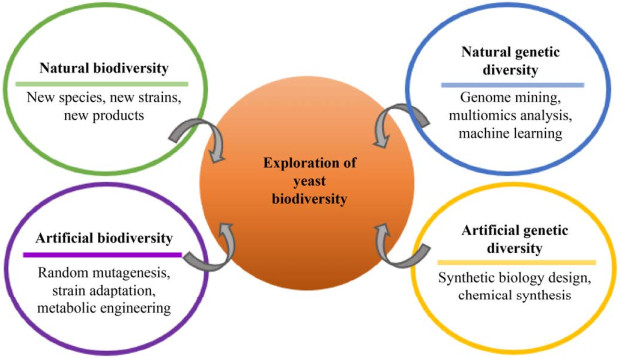
|
| 图 1 酵母菌生物及遗传多样性开发方法 Fig. 1 Methods to explore yeast biodiversity and genetic diversity. |
| |
以上综述了酵母菌种和菌株的多样性,以及进一步扩大多样性的方法,利用这些天然多样性和人工产生的非天然多样性,可生产多种活性物质及能源产品,拓宽酵母的工业应用范围。
4.1 酿酒酵母天然酿酒酵母菌株的应用不仅局限在发酵食品和啤酒、葡萄酒等酒精类产品的发酵,还报道了一些新的用途和特性。例如,日本学者从冲绳的芙蓉花中分离出的酿酒酵母新菌株具有良好的水果风味,经过诱变选育后,明显提高了菌株产生异戊醇和乙酸异戊酯的含量,即在酒精饮料的酿造过程中可以提高风味物质的产生[41]。酿酒酵母已知不能积累油脂,但是研究发现酿酒酵母D5A工业菌株在氮限制条件下可积累油脂,转录组分析表明,该菌株胞质乙酰辅酶A和NADPH以及氮吸收与油脂积累有关[42]。笔者课题组研究发现酿酒酵母YB-2625菌株具有比其他菌株更好的木糖利用性能[14],因此作为良好宿主,经过代谢工程改造后用于利用木糖生产燃料乙醇或化学品。
近年来很多研究者利用合成生物学技术对酿酒酵母进行改造合成天然产物,这些天然产物往往在植物中的积累量非常少,或者合成途径复杂,不容易调控,或者受到外界条件的限制较多因此产量不稳定,因此,利用合成生物学手段构建酿酒酵母细胞工厂,有利于实现稳定可持续的生产和调控合成。例如对酵母进行菌株改造生产阿片类药物[43],以及合成生物学改造生产大麻素等[44]。此外,我国学者利用酿酒酵母生产灯盏乙素,由于灯盏乙素在治疗缺血性脑血管疾病脑栓塞和脑溢血等疾病中有较好的效果,该工作将为工业化生产灯盏乙素相关药物提供新的途径[45]。在最近的研究中,我国浙江大学研究者利用酿酒酵母生产天然维生素E类化合物α-生育三烯酚获得了成功,解决了该类化合物生产困难的问题,实现了可持续生产,在利用酿酒酵母生产天然高附加值产物研究中又取得了重要的进展[46]。
4.2 非常规酵母一些非常规酵母具有特殊的生理特征或者良好的耐性[12, 47],可用于生产生物乙醇和化学品等,例如,马克斯克鲁维酵母Kluyveromyces marxianus具有很好的高温耐性,用于乙醇发酵或生产酶和风味物质[48],也可以作为宿主用于生产异源蛋白[49];解脂耶氏酵母Yarrowia lipolytica是良好的油脂生产酵母[50],同时也可以生产多种化学品,包括聚酮化合物、萜类化合物和类胡萝卜素等[51]。德巴利酵母Debaryomyces hansenii具有较好的高渗透压耐受性,可在高达4 mol/L的氯化钠中生长。除了在食品工业中的用途,该酵母也可用于生产油脂,并可生产水解酶和糖醇等精细化学品[52]。值得指出的是,很多非常规酵母都有非常好的抗逆性,例如,Mukherjee等[12]发现有23个非常规酵母菌株表现出良好的高渗透压耐性,可以在葡萄糖浓度为55% (W/V) 的平板上生长。其中有11个菌株可以在葡萄糖浓度为70% (W/V) 的平板上生长,包括Candida bombi、Torulaspora delbrueckii、Zygosaccharomyces rouxii等,这些菌株将有望用于高浓度底物发酵,具有较好的开发潜力。表 1列举了几种常见的非常规酵母应用于工业上的特性与优势。
| Species | Advantages | References |
| Debaryomyces hansenii | High osmo-tolera nce, produce lytic enzymes and fine chemicals | [52] |
| Candida bombi | Better osmotolerance and hydroxymethylfurfural tolerance | [62] |
| Hanseniaspora uvarum | Enhance wine flavor | [63] |
| Kluyveromyces lactis | Produce heterologous proteins; efficient expression and secretion of a variety of peptides and high molecular weight proteins | [64] |
| Kluyveromyces marxianus | High temperature tolerance; capability for fermentation of mixed sugars | [65] |
| Yarrowia lipolytica | Produce organic acid, lipids and squalene | [66] |
近年来利用代谢工程和合成生物学技术对非常规酵母进行改造合成化学品和高价值产品已经形成了热潮。虽然和酿酒酵母比较,非常规酵母遗传改造效率不高,但是目前一些非常规酵母,包括克鲁维酵母、解脂耶氏酵母、拜氏接合酵母等,已经建立起有效的改造方法[53-56],利用这些酵母的改造菌株高效生产有机酸、油脂、糖醇和萜类化合物等产品[57-61]。
5 展望随着菌种分离鉴定技术和高通量测序技术等相关技术的发展,酵母多样性将被更多揭示,大量酵母新种将被发现,其工业应用潜力也将不断被开发。笔者特别强调,除了对不同酵母种水平的开发,也需要注意对具有不同特性的酵母菌株的开发。预计未来酵母多样性开发和工业应用将着重从以下几个方向深入。
5.1 非常规酵母的改造及应用目前酿酒酵母的遗传操作相对成熟,其系统生物学和合成生物学技术方法已经被用于提高酵母细胞工厂的性能[67-68]。许多非常规酵母的应用价值有待开发,可以借助高通量测序技术获得非常规酵母的基因组序列,挖掘代谢潜能和工业应用价值。同时,很多非常规酵母很难被遗传改造,因此开发高效的非常规酵母遗传操作方法也非常必要。
5.2 人工智能辅助改造酵母菌株随着高通量测序的广泛应用,大量的组学数据有待深入挖掘应用。例如,利用机器学习研究了1亿个酿酒酵母的合成启动子,构建模型预测转录调控结果,发现转录因子特异性、活性、转录因子和染色质的相互作用等都影响表达水平[68]。在利用人工智能对大量数据进行快速分析基础上,可以提出有效的策略改造酵母菌株。
5.3 酵母细胞工厂合成生物学设计随着高通量筛选技术方法的进步,大量高效的酵母遗传元件将被开发利用[69],而不同非常规酵母底盘也将被更广泛应用,酵母基因组编辑技术和染色体工程技术[70]也日趋成熟,对酵母生理代谢调控的机理认识也不断深入,这些都将使利用合成生物学技术设计酵母工厂更加高效。
尽管目前人类对酵母的认知越来越深入,但是酵母的工业应用范围还有待进一步拓宽。未来将在利用酵母对农用废弃物等生物质进行生物炼制、利用多种酵母底盘生产高值产品、高效构建酵母细胞工厂等研究方向取得更多进展,更好地利用酵母为人类服务。
| [1] |
Kurtzman CP, Fell JW, Boekhout T. The Yeast-a Taxonomic Study. 5th Ed. Amsterdam: Elsevier, 2011.
|
| [2] |
彭立影, 刘功良, 费永涛, 等. 产酯酵母及其产酯关键酶的研究进展. 食品与发酵工业, 2020, 46(14): 275-282. Peng LY, Liu GL, Fei YT, et al. Research progress of ester-producing yeast and its key enzymes for esters synthesis. Food Fer Ind, 2020, 46(14): 275-282 (in Chinese). |
| [3] |
Chai CY, Huang LN, Cheng H, et al. Metschnikowia baotianmanensis f.a., sp. nov., a new yeast species isolated from the gut of the rhinoceros beetle Allomyrina dichotoma. Int J Syst Evol Microbiol, 2019, 69(10): 3087-3092. DOI:10.1099/ijsem.0.003593
|
| [4] |
Ramazzotti M, Stefanini I, Di Paola M, et al. Population genomics reveals evolution and variation of Saccharomyces cerevisiae in the human and insects gut. Environ Microbiol, 2019, 21(1): 50-71. DOI:10.1111/1462-2920.14422
|
| [5] |
Kavšček M, Stražar M, Curk T, et al. Yeast as a cell factory: current state and perspectives. Microb Cell Fact, 2015, 14: 94. DOI:10.1186/s12934-015-0281-x
|
| [6] |
Li AH, Yuan FX, Groenewald M, et al. Diversity and phylogeny of basidiomycetous yeasts from plant leaves and soil: proposal of two new orders, three new families, eight new genera and one hundred and seven new species. Stud Mycol, 2020, 96: 17-140. DOI:10.1016/j.simyco.2020.01.002
|
| [7] |
Ke T, Zhai YC, Yan ZL, et al. Kazachstania jinghongensis sp. nov. and Kazachstania menglunensis f. a., sp. nov., two yeast species isolated from rotting wood. Int J Syst Evol Microbiol, 2019, 69(11): 3623-3628. DOI:10.1099/ijsem.0.003670
|
| [8] |
Gao WL, Liu KF, Yao LG, et al. Pichia nanzhaoensis sp. nov. and Pichia paraexigua f. a., sp. nov., two yeast species isolated from rotting wood. Int J Syst Evol Microbiol, 2018, 68(10): 3311-3315. DOI:10.1099/ijsem.0.002989
|
| [9] |
Avchar R, Groenewald M, Baghela A. Wickerhamiella shivajii sp. nov., a thermotolerant yeast isolated from distillery effluent. Int J Syst Evol Microbiol, 2019, 69(10): 3262-3267. DOI:10.1099/ijsem.0.003616
|
| [10] |
Peter J, De Chiara M, Friedrich A, et al. Genome evolution across 1 011 Saccharomyces cerevisiae isolates. Nature, 2018, 556(7701): 339-344. DOI:10.1038/s41586-018-0030-5
|
| [11] |
Gallone B, Steensels J, Prahl T, et al. Domestication and divergence of Saccharomyces cerevisiae beer yeasts. Cell, 2016, 166(6): 1397-1410. DOI:10.1016/j.cell.2016.08.020
|
| [12] |
Duan SF, Han PJ, Wang QM, et al. The origin and adaptive evolution of domesticated populations of yeast from Far East Asia. Nat Commun, 2018, 9(1): 2690. DOI:10.1038/s41467-018-05106-7
|
| [13] |
Zhao XQ, Xiong L, Zhang MM, et al. Towards efficient bioethanol production from agricultural and forestry residues: exploration of unique natural microorganisms in combination with advanced strain engineering. Bioresour Technol, 2016, 215: 84-91. DOI:10.1016/j.biortech.2016.03.158
|
| [14] |
Cheng C, Tang RQ, Xiong L, et al. Association of improved oxidative stress tolerance and alleviation of glucose repression with superior xylose- utilization capability by a natural isolate of Saccharomyces cerevisiae. Biotechnol Biofuels, 2018, 11: 28. DOI:10.1186/s13068-018-1018-y
|
| [15] |
Wang QM, Liu WQ, Liti G, et al. Surprisingly diverged populations of Saccharomyces cerevisiae in natural environments remote from human activity. Mol Ecol, 2012, 21(22): 5404-5417. DOI:10.1111/j.1365-294X.2012.05732.x
|
| [16] |
Celińska E, Borkowska M, Białas W, et al. Robust signal peptides for protein secretion in Yarrowia lipolytica: identification and characterization of novel secretory tags. Appl Microbiol Biotechnol, 2018, 102(12): 5221-5233. DOI:10.1007/s00253-018-8966-9
|
| [17] |
Basak S, Sahoo NG, Pavanasam AK. Genome mining, in silico validation and phase selection of a novel aldo-keto reductase from Candida glabrata for biotransformation. Bioengineered, 2018, 9(1): 186-195. DOI:10.1080/21655979.2017.1342911
|
| [18] |
Nambu-Nishida Y, Sakihama Y, Ishii J, et al. Selection of yeast Saccharomyces cerevisiae promoters available for xylose cultivation and fermentation. J Biosci Bioeng, 2018, 125(1): 76-86. DOI:10.1016/j.jbiosc.2017.08.001
|
| [19] |
Mukherjee V, Radecka D, Aerts G, et al. Phenotypic landscape of non-conventional yeast species for different stress tolerance traits desirable in bioethanol fermentation. Biotechnol Biofuels, 2017, 10: 216. DOI:10.1186/s13068-017-0899-5
|
| [20] |
Zhang X, Zhang XF, Li HP, et al. Atmospheric and room temperature plasma (ARTP) as a new powerful mutagenesis tool. Appl Microbiol Biotechnol, 2014, 98(12): 5387-5396. DOI:10.1007/s00253-014-5755-y
|
| [21] |
Jian XJ, Guo XJ, Wang J, et al. Microbial microdroplet culture system (MMC): An integrated platform for automated, high-throughput microbial cultivation and adaptive evolution. Biotechnol Bioeng, 2020, 117(6): 1724-1737. DOI:10.1002/bit.27327
|
| [22] |
Zeng WZ, Guo LK, Xu S, et al. High-throughput screening technology in industrial biotechnology. Trends Biotechnol, 2020, 38(8): 888-906. DOI:10.1016/j.tibtech.2020.01.001
|
| [23] |
Lian JZ, Mishra S, Zhao HM. Recent advances in metabolic engineering of Saccharomyces cerevisiae: New tools and their applications. Mab Eng, 2018, 50: 85-108.
|
| [24] |
Abdel-Mawgoud AM, Markham KA, Palmer CM, et al. Metabolic engineering in the host Yarrowia lipolytica. Metab Eng, 2018, 50: 192-208. DOI:10.1016/j.ymben.2018.07.016
|
| [25] |
Peña DA, Gasser B, Zanghellini J, et al. Metabolic engineering of Pichia pastoris. Metabol Eng, 2018, 50: 2-15. DOI:10.1016/j.ymben.2018.04.017
|
| [26] |
Anandharaj M, Lin YJ, Rani RP, et al. Constructing a yeast to express the largest cellulosome complex on the cell surface. Proc Natl Acad Sci USA, 2020, 117(5): 2385-2394. DOI:10.1073/pnas.1916529117
|
| [27] |
Hou J, Jiao CL, Peng B, et al. Mutation of a regulator Ask10p improves xylose isomerase activity through up-regulation of molecular chaperones in Saccharomyces cerevisiae. Metab Eng, 2016, 38: 241-250. DOI:10.1016/j.ymben.2016.08.001
|
| [28] |
Ma T, Shi B, Ye ZL, et al. Lipid engineering combined with systematic metabolic engineering of Saccharomyces cerevisiae for high-yield production of lycopene. Metab Eng, 2019, 52: 134-142. DOI:10.1016/j.ymben.2018.11.009
|
| [29] |
Srinivasan P, Smolke CD. Biosynthesis of medicinal tropane alkaloids in yeast. Nature, 2020, 585(7826): 614-619.
|
| [30] |
Lei H, Xie BW, Gao T, et al. Yeast display platform technology to prepare oral vaccine against lethal H7N9 virus challenge in mice. Microb Cell Fact, 2020, 19(1): 907-932.
|
| [31] |
Deparis Q, Claes A, Foulquié-Moreno MR, et al. Engineering tolerance to industrially relevant stress factors in yeast cell factories. FEMS Yeast Res, 2017, 17(4): fox036.
|
| [32] |
Cheng C, Zhang MM, Xue C, et al. Development of stress tolerant Saccharomyces cerevisiae strains by metabolic engineering: new aspects from cell flocculation and zinc supplementation. J Biosci Bioeng, 2017, 123(2): 141-146. DOI:10.1016/j.jbiosc.2016.07.021
|
| [33] |
Zhang MM, Zhao XQ, Cheng C, et al. Improved growth and ethanol fermentation of Saccharomyces cerevisiae in the presence of acetic acid by overexpression of SET5 and PPR1. Biotechnol J, 2015, 10(12): 1903-1911. DOI:10.1002/biot.201500508
|
| [34] |
方青, 张明明, 熊亮, 等. 过表达谷氧还蛋白基因GRX5提高酿酒酵母乙酸耐性. 化工学报, 2015, 66(4): 1434-1439. Fang Q, Zhang MM, Xiong L, et al. Improvement of acetic acid tolerance of Saccharomyces cerevisiae by overexpressing glutaredoxin encoding gene GRX5. CIESC J, 2015, 66(4): 1434-1439 (in Chinese). |
| [35] |
Zhang MM, Xiong L, Tang YJ, et al. Enhanced acetic acid stress tolerance and ethanol production in Saccharomyces cerevisiae by modulating expression of the de novo purine biosynthesis genes. Biotechnol Biofuels, 2019, 12: 116. DOI:10.1186/s13068-019-1456-1
|
| [36] |
Chen BB, Lee HL, Heng YC, et al. Synthetic biology toolkits and applications in Saccharomyces cerevisiae. Biotechnol Adv, 2018, 36(7): 1870-1881. DOI:10.1016/j.biotechadv.2018.07.005
|
| [37] |
Xie ZX, Liu D, Li BZ, et al. Design and chemical synthesis of eukaryotic chromosomes. Chem Soc Rev, 2017, 46(23): 7191-7207. DOI:10.1039/C7CS00208D
|
| [38] |
柴梦哲, 贾斌, 李炳志, 等. 人工基因组合成与重排研究进展. 生命科学, 2019, 31(4): 364-371. Chai MZ, Jia B, Li BZ, et al. Advances in synthetic genome and genome rearrangement. Chin Bull Life Sci, 2019, 31(4): 364-371 (in Chinese). |
| [39] |
Hu YT, Zhu ZW, Gradischnig D, et al. Engineering carboxylic acid reductase for selective synthesis of medium-chain fatty alcohols in yeast. Proc Natl Acad Sci USA, 2020, 117(37): 22974-22983. DOI:10.1073/pnas.2010521117
|
| [40] |
Rajkumar AS, Liu GD, Bergenholm D, et al. Engineering of synthetic, stress-responsive yeast promoters. Nucleic Acids Res, 2016, 44(17): e136. DOI:10.1093/nar/gkw553
|
| [41] |
Abe T, Toyokawa Y, Sugimoto Y, et al. Characterization of a new Saccharomyces cerevisiae isolated from hibiscus flower and its mutant with L-leucine accumulation for awamori brewing. Front Genet, 2019, 10: 490. DOI:10.3389/fgene.2019.00490
|
| [42] |
He QN, Yang YF, Yang SH, et al. Oleaginicity of the yeast strain Saccharomyces cerevisiae D5A. Biotechnol Biofuels, 2018, 11(1): 258. DOI:10.1186/s13068-018-1256-z
|
| [43] |
Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast. Science, 2015, 349(6252): 1095-1100. DOI:10.1126/science.aac9373
|
| [44] |
Luo XZ, Reiter MA, D'Espaux L, et al. Complete biosynthesis of cannabinoids and their unnatural analogues in yeast. Nature, 2019, 567(7746): 123-126. DOI:10.1038/s41586-019-0978-9
|
| [45] |
Liu XN, Cheng J, Zhang GH, et al. Engineering yeast for the production of breviscapine by genomic analysis and synthetic biology approaches. Nat Commun, 2018, 9(1): 448. DOI:10.1038/s41467-018-02883-z
|
| [46] |
Shen B, Zhou PP, Jiao X, et al. Fermentative production of Vitamin E tocotrienols in Saccharomyces cerevisiae under cold-shock-triggered temperature control. Nat Commun, 2020, 11: 5155. DOI:10.1038/s41467-020-18958-9
|
| [47] |
Thorwall S, Schwartz C, Chartron JW, et al. Stress-tolerant non-conventional microbes enable next-generation chemical biosynthesis. Nat Chem Biol, 2020, 16(2): 113-121. DOI:10.1038/s41589-019-0452-x
|
| [48] |
Fonseca GG, Heinzle E, Wittmann C, et al. The yeast Kluyveromyces marxianus and its biotechnological potential. Appl Microbiol Biotechnol, 2008, 79(3): 339-354. DOI:10.1007/s00253-008-1458-6
|
| [49] |
Gombert AK, Madeira JV Jr, Cerdán ME, et al. Kluyveromyces marxianus as a host for heterologous protein synthesis. Appl Microbiol Biotechnol, 2016, 100(14): 6193-6208. DOI:10.1007/s00253-016-7645-y
|
| [50] |
Lazar Z, Liu N, Stephanopoulos G. Holistic approaches in lipid production by Yarrowia lipolytica. Trends Biotechnol, 2018, 36(11): 1157-1170. DOI:10.1016/j.tibtech.2018.06.007
|
| [51] |
Miller KK, Alper HS. Yarrowia lipolytica: more than an oleaginous workhorse. Appl Microbiol Biotechnol, 2019, 103(23/24): 9251-9262. DOI:10.1007/s00253-019-10200-x
|
| [52] |
Breuer U, Harms H. Debaryomyces hansenii—an extremophilic yeast with biotechnological potential. Yeast, 2006, 23(6): 415-437. DOI:10.1002/yea.1374
|
| [53] |
Wagner JM, Alper HS. Synthetic biology and molecular genetics in non-conventional yeasts: Current tools and future advances. Fungal Genet Biol, 2016, 89: 126-136. DOI:10.1016/j.fgb.2015.12.001
|
| [54] |
Löbs AK, Schwartz C, Thorwall S, et al. Highly multiplexed CRISPRi repression of respiratory functions enhances mitochondrial localized ethyl acetate biosynthesis in Kluyveromyces marxianus. ACS Synth Biol, 2018, 7(11): 2647-2655. DOI:10.1021/acssynbio.8b00331
|
| [55] |
Larroude M, Rossignol T, Nicaud JM, et al. Synthetic biology tools for engineering Yarrowia lipolytica. Biotechnol Adv, 2018, 36(8): 2150-2164. DOI:10.1016/j.biotechadv.2018.10.004
|
| [56] |
Kuanyshev N, Rao CV, Dien B, et al. Domesticating a food spoilage yeast into an organic acid-tolerant metabolic engineering host: Lactic acid production by engineered Zygosaccharomyces bailii. Biotechnol Bioeng, 2020. DOI:10.1002/bit.27576
|
| [57] |
Cernak P, Estrela R, Poddar S, et al. Engineering Kluyveromyces marxianus as a robust synthetic biology platform host. mBio, 2018, 9(5): e01410-18.
|
| [58] |
Yuzbasheva EY, Scarcia P, Yuzbashev TV, et al. Engineering Yarrowia lipolytica for the selective and high-level production of isocitric acid through manipulation of mitochondrial dicarboxylate- tricarboxylate carriers. Metab Eng, 2020. DOI:10.1016/j.ymben.2020.11.001
|
| [59] |
Wang N, Chi P, Zou YW, et al. Metabolic engineering of Yarrowia lipolytica for thermoresistance and enhanced erythritol productivity. Biotechnol Biofuels, 2020, 13: 176. DOI:10.1186/s13068-020-01815-8
|
| [60] |
Liu H, Wang F, Deng L, et al. Genetic and bioprocess engineering to improve squalene production in Yarrowia lipolytica. Bioresour Technol, 2020, 317: 123991. DOI:10.1016/j.biortech.2020.123991
|
| [61] |
Wang JP, Ledesma-Amaro R, Wei YJ, et al. Metabolic engineering for increased lipid accumulation in Yarrowia lipolytica — a review. Bioresour Technol, 2020, 313: 123707. DOI:10.1016/j.biortech.2020.123707
|
| [62] |
Ruyters S, Mukherjee V, Verstrepen KJ, et al. Assessing the potential of wild yeasts for bioethanol production. J Ind Microbiol Biotechnol, 2015, 42(1): 39-48. DOI:10.1007/s10295-014-1544-y
|
| [63] |
Guaragnella N, Chiara M, Capece A, et al. Genome sequencing and comparative analysis of three Hanseniaspora uvarum indigenous wine strains reveal remarkable biotechnological potential. Front Microbiol, 2020, 10: 3133. DOI:10.3389/fmicb.2019.03133
|
| [64] |
Johnson EA. Biotechnology of non-Saccharomyces yeasts — the ascomycetes. Appl Microbiol Biotechnol, 2013, 97(2): 503-517. DOI:10.1007/s00253-012-4497-y
|
| [65] |
Nurcholis M, Lertwattanasakul N, Rodrussamee N, et al. Integration of comprehensive data and biotechnological tools for industrial applications of Kluyveromyces marxianus. Appl Microbiol Biotechnol, 2020, 104(2): 475-488. DOI:10.1007/s00253-019-10224-3
|
| [66] |
Spagnuolo M, Yaguchi A, Blenner M. Oleaginous yeast for biofuel and oleochemical production. Curr Opin Biotechnol, 2019, 57: 73-81. DOI:10.1016/j.copbio.2019.02.011
|
| [67] |
Nielsen J. Yeast systems biology: Model organism and cell factory. Biotechnol J, 2019, 14(9): 1800421. DOI:10.1002/biot.201800421
|
| [68] |
De Boer CG, Vaishnav ED, Sadeh R, et al. Deciphering eukaryotic gene-regulatory logic with 100 million random promoters. Nat Biotechnol, 2020, 38(1): 56-65. DOI:10.1038/s41587-019-0315-8
|
| [69] |
Liu ZH, Zhang YP, Nielsen J. Synthetic biology of yeast. Biochemistry, 2019, 58(11): 1511-1520. DOI:10.1021/acs.biochem.8b01236
|
| [70] |
Jin J, Jia B, Yuan YJ. Yeast chromosomal engineering to improve industrially-relevant phenotypes. Curr Opin Biotechnol, 2020, 66: 165-170. DOI:10.1016/j.copbio.2020.07.003
|
 2021, Vol. 37
2021, Vol. 37




