中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张晨阳, 武耀康, 徐显皓, 吕雪芹, 李江华, 堵国成, 刘龙
- Zhang Chenyang, Wu Yaokang, Xu Xianhao, Lv Xueqin, Li Jianghua, Du Guocheng, Liu Long
- 工业微生物代谢网络模型的研究进展及应用
- Current status and future perspectives of metabolic network models of industrial microorganisms
- 生物工程学报, 2021, 37(3): 860-873
- Chinese Journal of Biotechnology, 2021, 37(3): 860-873
- 10.13345/j.cjb.200640
-
文章历史
- Received: October 8, 2020
- Accepted: January 18, 2021
2. 江南大学糖化学与生物技术教育部重点实验室,江苏 无锡 214122
2. Key Laboratory of Carbohydrate Chemistry and Biotechnology, Ministry of Education, Jiangnan University, Wuxi 214122, Jiangsu, China
在亿万年的自然选择和数千年的人类驯化过程中,为了适应环境的变化,一些微生物主动或者被动地进行了适应性进化,产生了某些可以满足人类需求的性状或产物,从而成为可应用的工业微生物[1]。随着社会的发展和技术的进步,人们对工业微生物提出了更高的要求,例如,对发酵过程具有更强的鲁棒性,可以利用更经济的底物以及生产更高价值、更高产量、更高得率的产物。
针对这一需求,国内外研究人员采用生化工程和代谢工程策略,通过对目标产物的相关合成途径基因进行强化,对其竞争途径相关基因进行弱化表达或敲除以及引入外源基因构建新途径等方法,提高工业微生物的产量或赋予微生物合成新产物的能力[2]。但是,微生物细胞是一个复杂的生命体,其自身在进化的过程中形成了精密且复杂的代谢网络[3]。虽然研究者已经对这一复杂的代谢调控网络进行了大量的研究,并且取得了卓有成效的进展,但是依然缺乏对微生物基因型与表型之间系统的生物学关联性分析的整体理解。因此,这可能会导致研究无法达到预期的效果,并且这种随机或者半理性的调控策略所消耗的时间、人力和经济成本较高,已经无法满足目前工业生物技术发展的需求。近年来,随着组学技术的发展,重要的模式工业微生物全基因组都已公布,研究者可以基于基因组数据构建基因组规模代谢网络模型(Genome-scale metabolic network model,GSMM),对基因组、代谢反应和蛋白三者之间的关系进行表征,使解析工业微生物复杂代谢网络和调控机制成为可能,为定向改造微生物奠定了基础。同时,基因组注释的不断深入、建模方法理论的逐渐丰富、模型求解工具和相关数据库[4]的日益完善,都为工业微生物GSMM的发展提供了强大的助力,更为工业生物技术的发展提供了前所未有的机遇。
文章首先简述了GSMM的发展历程,对GSMM的构建方法、评价标准以及分析算法进行介绍,就GSMM在4种重要的模式工业微生物枯草芽孢杆菌Bacillus subtilis、大肠杆菌Escherichia coli、谷氨酸棒杆菌Corynebacterium glutamicum和酿酒酵母Saccharomyces cerevisiae中的发展过程和相关应用进行了介绍,最后对GSMM未来的发展方向进行展望。
1 代谢网络模型的发展GSMM作为一种数学模型,用于表征生物体整个代谢途径中基因-蛋白-反应(Gene-Protein- Reaction,GPR) 之间的关系,基于化学计量学、能量平衡的所有的新陈代谢反应以及基因型-表型的关系对微生物细胞进行定量描述,可以用来模拟预测不同系统水平代谢反应的代谢通量[5]。自1999年报道的第一个用于流感嗜血杆菌Haemophilus influenzae的GSMM以来[6],随着生物技术和计算机工具的发展,通过整合组学数据[7-8]、添加约束条件[9-11]、融合生物模型[12]等方法,不断地提高GSMM模型预测的准确性,GSMM已经成为系统代谢水平建模研究的主要方法之一(图 1)。截至2020年10月,已经重构了6 285种生物(5 887种细菌、130种古菌和222种真核生物) 的GSMM,其中使用手动方法重构的GSMM包含了183种生物(113种细菌、10种古菌和60种原核生物)。
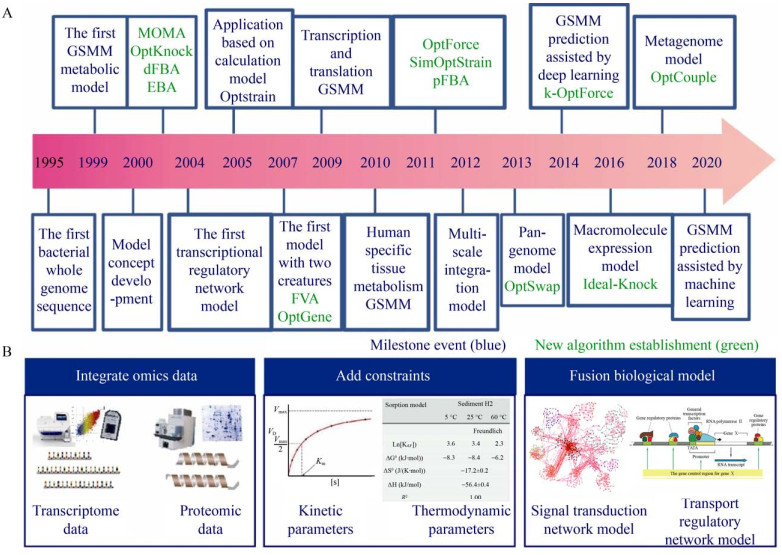
|
| 图 1 GSMM研究历程与策略(A:GSMM算法发展历程及里程碑事件;B:提高GSMM预测准确性的方法) Fig. 1 Research process and strategy of GSMM. (A) GSMM algorithm development process and milestone events. (B) the methods to improve the accuracy of GSMM prediction. |
| |
由于重构GSMM的数量快速增加,如何对GSMM的质量、覆盖范围和预测潜力进行统一成为急需解决的问题。Palsson课题组2010年在Nature Protocols发表了手动构建GSMM的教程,该教程分为5个部分(图 2),即重构模型草图的创建、重构模型的精炼、重构模型的数学形式转化、网络评估和数据汇总与发布,一共包含了96个具体的步骤[5]。手动重构GSMM依赖于基因组的注释结果,通过对代谢网络涉及的反应、蛋白和基因相关信息的收集和整理,并且需要研究者对于这一结果进行不断地迭代,直到GSMM达到模型构建者所需的性能,手动优化这种迭代过程使得手动重构GSMM过程变得非常耗时,但同时提高了GSMM的准确性和可靠性。随着生物技术的发展,大量的相关数据快速涌现,手动重构GSMM对于数据的收集与整理变得更加耗时,限制了部分研究者快速利用数据构建和应用GSMM。
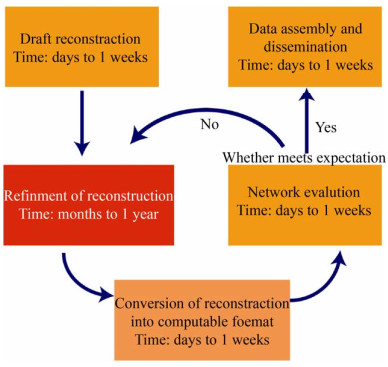
|
| 图 2 GSMM手动重构程序示意图 Fig. 2 Schematic diagram of manual reconstruction program of GSMM. |
| |
为了实现对于数据的快速挖掘和GSMM的重构,相关研究者已经开发了多个用于GSMM自动构建的工具(表 1)。这些工具旨在通过自动执行一些本应手动进行的工作,从而加快模型重构的过程。Merlin (Metabolic models reconstruction using genome-scale information)[13]包含了多个工具用于代谢网络重建和基因注释的整理,允许用户对注释算法中的几个参数进行更改,例如,期望值阈值和最大匹配数,使得这一工具的使用极为便利。Merlin可以进行蛋白质和代谢物的亚细胞定位预测,成为自动构建多腔室生物代谢模型的理想工具,已经被用于建立原核和真核微生物的代谢模型[14-15]。SuBliMinal Toolbox[16]对代谢网络模型自动构建的程序进行了模块化,允许研究者添加多种工具,为模型的重构提供多种选择。
| Tool | Language | Source database | Gap-filling | References |
| Merlin | JAVA | KEGG, TCDB | No | [13] |
| SuBliMinal toolbox | JAVA | KEGG, MetaCyc | No | [16] |
| Model SEED/KBase | Web | Model SEED database | Yes | [17, 20] |
| Pathway Tools | Python, Lisp | MetaCyc | Yes | [18] |
| RAVEN2.0 | Matlab | KEGG, MetaCyc | Yes | [19] |
| AutoKEGGRec | Matlab | KEGG | No | [21] |
| IMGMD | LAMP | MySQL | No | [22] |
| AuReMe | Python | KEGG, BIGG | YES | [23] |
以上这两种GSMM自动重构的方法虽然可以进行模型的自动重构,但是并未集成缺口填充功能,在使用过程中仍然需要人工对缺口进行进一步完善,这是一个较为耗时的过程。Model SEED[17]、Pathway Tools[18]、RAVEN2.0 (Reconstruction,Analysis and Visualization of Metabolic Networks)[19]这3个工具集成了自动填充缺口的算法,其中Model SEED和Pathway Tools允许完全自动填充的算法,而RAVEN2.0则需要用户为缺口添加适当的GPR。Model SEED是第一个将模型草图构建、模型完善、空缺填充、模型评价与表型数据集相集成的平台(http://modelseed.org)。Model SEED可以通过上传基因组序列进行代谢网络模型的构建与分析,是基于微生物或者植物基因组注释信息进行代谢模型构建的重要资源,目前已经包含了33 978种化合物和36 645种代谢反应,同时Model SEED的KBase[20]版本还可以进行批量代谢模型的重建,使大规模分析成为可能,但是,Model SEED无法进行手动优化及人为调控。RAVEN工具箱是一个MATLAB软件包,除了可以进行代谢网络自动重构之外,这一工具还侧重于网络可视化和分析,与Merlin相似的是,RAVEN也同样具有蛋白质亚细胞定位预测的功能。Pathway Tools是基于构建生物体特定途径的数据库工具发展而来的,目前已经成为代谢网络模型构建的一整套工具。
以上介绍了5个自动重构GSMM的工具,不同的工具之间各有优劣,每个工具之间功能的侧重是不同的,需要研究者根据自身的需要挑选合适的工具进行使用。虽然越来越多的自动构建模型的工具和策略迅速发展起来,但是代谢网络模型的自动构建仍然存在着许多挑战,例如,源数据库中很多固有的错误,所有的自动重构GSMM工具均不能使用Biolog表型数据矩阵、基因敲除实验和不同类型的组学数据等。
2.3 代谢网络模型评价指标随着GSMM构建工具的不断涌现和相关数据库的完善,每年都有越来越多的基于手动或自动构建的GSMM产生。每当新的GSMM产生时,都需要评估其是否具有比之前的模型更完整的覆盖范围或/和更好的预测性能。这些评估指标可以被分为两大类(描述性指标和生物行为预测指标) 和四小类(通用性指标、连接度指标、生长指标和基因敲除指标)[24]。
2.3.1 通用性指标对于一个新的GSMM评估的最基本的通用性指标是模型规模(基因、代谢物和代谢反应)。例如,S.cerevisiae的GSMM由Yeast7.6到目前最新的Yeast8.3中各项指标的变化(图 3)[25]。除此之外,还存在一些通用性指标,例如,不同模型中代谢物、反应或基因的相似性或异质性比较[26],是否包含替代途径[27],是否使用标准化的标识符[28]等。
任何GSMM的核心都是代谢网络,其可以简化为一个无向二分图。通过将代谢物和代谢反应进行连接,计算GSMM中的连接度指标,从而可以深刻地理解网络的拓扑属性。常用的计算指标有:任意节点的连接度、与某一代谢物的连接度和全局的连接度。
2.3.3 生长指标GSMM的主要用途之一是模型预测,是使用通量平衡(Flux balance analysis,FBA)[29]算法对微生物的生长速度、底物消耗和产物生成速率进行预测,使用实验数据验证模型的特定生长率或者其他代谢通量分布,已经成为模型验证的标准程序[27, 30-31]。另一个生长指标是对于GSMM内部代谢网络流量分布的评估,通过这一生长指标可以帮助研究者理解碳、氮源是如何被消耗或者微生物细胞在不同的环境中(有氧/无氧) 能量是如何进行分布的。
2.3.4 基因敲除指标对于新的GSMM评估的另一个生物行为预测指标是模型对于微生物基因敲除的预测能力。最常见的方法是单基因敲除分析[32]和双基因敲除分析[33],可以对微生物细胞中的必需基因和合成致死基因进行预测。最终研究者通过实验数据或文献报道进行验证,从而对模型的灵敏度、特异性和准确性等进行评估。
2020年3月,Lieven等[34]提出了一种基于Python开发的基因组规模代谢模型测试工具——代谢模型测定(Metabolic model tests,MEMOTE)。这一工具不仅包含了上述4类评估指标的功能,同时还具有结构验证功能,即需要研究者使用标准化的系统生物学标记语言对模型进行描述,这对于模型的重复性和再利用性具有重要的意义。除此之外,MEMOTE可以使用不同输入格式的实验数据对模型进行评价。MEMOTE还可以快速地对两个模型进行比较,并对模型中的功能指标进行量化,最终根据计算出的总分数可以直观地对新出现的GSMM的优劣进行评估[25]。
2.4 代谢网络模型分析算法目前代谢网络模型分析算法主要分为两类:一类是流量平衡分析类算法,即通过对模型中约束条件的整合,模拟流量在微生物代谢途径中的分布。FBA[29]是这类算法的基础,为了提高模型预测流量分布的准确性和拓展模型的应用,对代谢流量有额外约束的一系列算法被开发出来,例如Regulatory FBA (rFBA)[35]、Integrated FBA (iFBA)[36]、Dynamic FBA (dFBA)[37]和Community FBA (cFBA)[38]等。另一类算法是与分子改造预测相关的算法,可以分为基因敲除算法(OptKnock)[39]、基因添加算法(OptSwap)[40]、基因表达调控算法(OptForce和k-OptForce)[41-42]和异源途径表达算法(OptStrain和SimOptStrain)[43-44]等。通过对代谢途径中相关代谢通路中基因的最佳修饰进行模拟运算,以获得提高目标产物的最佳靶点,有助于实现对代谢流的精确调控,实现产物的高效合成。关于模型分析算法的全面评述请参见叶超等[45]的综述。
3 代谢网络模型在典型工业微生物中的发展与应用在过去的20年,随着研究的深入,一些重要的典型工业微生物菌株的GSMMs也在不断地进行修正和完善,研究者通过增加模型规模或者约束条件不断构建新的GSMM,下面我们将对4种典型工业微生物GSMM的发展过程(图 4) 以及相关应用进行介绍。
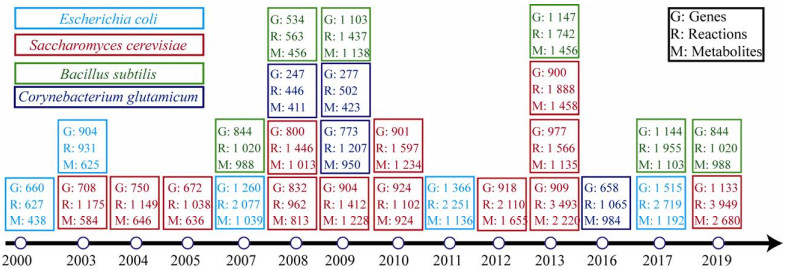
|
| 图 4 四种典型工业微生物的基因组规模代谢网络模型的发展过程 Fig. 4 The development of genome-scale metabolic network model of four typical industrial microorganisms. Sky blue represents Escherichia coli, red represents Saccharomyces cerevisiae, green represents Bacillus subtilis, and blue represents Corynebacterium glutamicum. 'G' stands for the number of genes incorporated in the model, 'R' for the number of reactions, and 'M' for the number of metabolites. |
| |
Bacillus subtilis是一种典型的革兰氏阳性菌,具有利用低成本的碳、氮源高效生长和有效分泌酶、维生素等产物的特性,是工业微生物领域中极具吸引力的一种底盘细胞[46-47]。研究者基于B.subtilis不同的基因注释进行了GSMM构建。Oh[48]、Goelzer[49]和Henry[50]等研究者基于Kunst等[51]对B.subtilis基因组的基因注释构建了不同的GSMM,分别应用于细胞生长最大化[48, 50]和评估细胞前体的合成预测细胞的生长[49]。Barbe等[52]对B.subtilis基因组进行了注释,研究者分别构建了iBsu1103V2[53]和iBsu1147[54]。Kocabas等[55]基于重新注释的基因组构建了GSMM—iBus1144,这一模型通过结合每个反应的标准摩尔吉布斯自由能变化的热力学信息来进行构建,以提高胞内可逆反应的准确性和一致性。Massaiu等[56]使用酶约束的方法基于枯草芽孢杆菌动力学和组学数据构建了ec-iY0844,这一模型可以更加准确地预测野生型和单基因缺失菌株的生长速率和代谢通量分布。
Kocabas等[55]通过iBus1144模型对丝氨酸碱性蛋白酶(Serine alkaline protease,SAP) 合成相应的代谢位点进行分析,发现不同的氧转移条件会影响SAP的合成,并对低、中、高3种氧转移条件进行模拟,预测了不同氧转移条件下的限速氨基酸(低:天冬酰胺(Asparagine,AsnL);中:AsnL,异亮氨酸(Isoleucine,IleL),苏氨酸(Threonine,ThrL) 和天冬氨酸(Aspartic acid,AspL);高:AsnL,IleL和丝氨酸(Serine,SerL);综合分析得出,生成SAP的最佳生物反应器为中氧转移条件,因此AsnL,IleL,ThrL和AspL被确定为主要的限速氨基酸,用于指导B.subtilis中的SAP合成。
3.2 大肠杆菌Escherichia coli是生物学中最重要的模式生物之一,具有可用于代谢工程和高密度培养的良好的工具和策略,以及快速生长等优点,是许多天然产物生物合成的细胞工厂[57]。E.coli K-12 MG1655基因组序列于1997年首次公布[58]。2000年,第一个E.coli GSMM——iJE660发表[59],之后研究者们为提高GSMM中GPR的覆盖范围和预测能力,进行了多次改进。Reed等[60]增加了GSMM对于替代碳源利用的预测能力,对代谢反应质量和电荷进行了平衡,重构了iJR904。Feist等[61]通过添加周质空间、扩展细胞壁代谢和增加热力学约束,进一步对GSMM的预测能力和覆盖范围进行提高,构建了iAF1260。Orth等[62]在GSMM中拓展了辅因子代谢预测和缺口填充功能,产生了iJO1366。目前E.coli K-12 MG1655中最新、最完整的GSMM是iML1515,其是在iJO1366的基础上通过增加蛋白质结构预测、活性氧代谢以及代谢物修复途径进行构建的[63]。iML1515包含了1 515个基因,2 719个代谢反应和1 192种代谢物,可以结合蛋白质结构信息,模拟感兴趣菌株在不同营养物质上的生长,并使用结构生物学方法评估菌株间的突变影响。
Yim等[64]基于iJR904使用OptKnock算法对E. coli高产1, 4-丁二醇的基因敲除靶点进行了预测,筛选出4个敲除位点,分别是乙醇脱氢酶(adhE)、丙酮酸甲酸裂解酶(pfl)、乳酸脱氢酶(ldh) 和苹果酸脱氢酶(mdh) 基因,同时通过模型预测得出在无氧条件时可以提高1, 4-丁二醇的产量,之后研究者基于预测的结果对菌株进行改造进行1, 4-丁二醇生产。Ranganathan等[65]利用OptForce算法对iAF1260模型中脂肪酸的合成进行了预测,鉴定出对fabZ基因和硫酯酶进行上调,同时敲除fadD基因,最终通过基因工程得到的菌株在最小M9培养基中C14-C16脂肪酸的产量达到了1.7 g/L。这些结果表明在设计重组E.coli生产目标产品中使用GSMM进行代谢调控是可行的。
3.3 谷氨酸棒杆菌Corynebacterium glutamicum是一种土壤中常见的革兰氏阳性细菌,是工业生物技术和环境修复的重要平台生物,多被应用于食品、饲料和制药工业中生产各类氨基酸[66-68]。2008年,Kieldsen等[69]基于C. glutamicum ATCC13032全基因组序列注释构建了第一个GSMM,该模型包含了247个基因,446个反应和411种代谢物。在这一GSMM的基础上,Shinfuku等[70]构建了一个包含了277个基因、502个反应和423种代谢物的GSMM。Zhang等[71]基于C. glutamicum ATCC13032全基因组序列注释构建了新的GSMM——iCW773,包含了773个基因、1 207个反应和950种代谢物。相较之前的GSMM,这一模型不仅提高了基因的覆盖率和优化了谷氨酸合成相关代谢通路,还限制代谢流的方向,并对代谢反应的质量和电荷进行了平衡,系统地解析了C.glutamicum的生理代谢特性,为C.glutamicum的代谢调控提供了策略和依据。由于之前的代谢模型缺乏对于谷氨酸合成代谢途径的模拟,Mei等[72]针对谷氨酸工业生产菌株C.glutamicum S9114,构建了一个新的GSMM——iJM658,该模型包含了658个基因、1 065个反应和984种代谢物。
Zhang等[71]基于iCW773利用OptForceMUST算法对L-脯氨酸生产过程中目标基因的修饰位点进行了预测,鉴定出糖酵解途径中的5个基因(pgi、pfKA、fda、gap和pgk),TCA循环中的3个基因(gltA、acn和icd) 以及L-脯氨酸合成基因proC需上调,下调丙酮酸到L-缬氨酸和L-丙氨酸的反应以及TCA循环中的kgd基因以及敲除putA基因。基于模型预测结果通过基因工程构建了一株从头合成L-脯氨酸的高产菌株,并且通过分批补料发酵,这株高产菌株在60 h生产了66.43 g/L的L-脯氨酸,产量为0.26 g/g,产率为1.11 g/(L·h)。这是目前报道的以葡萄糖为碳源的最小培养基中生产L-脯氨酸的最高产量和效价。Cheng等[73]通过向iCW773模型中添加透明质酸二糖缩合反应以及交换反应,并使用OptForceMUST算法对高产透明质酸代谢途径进行了预测。鉴定出高产透明质酸需要上调合成途径中的haS、hasB、hasD和glmM,下调糖酵解和磷酸戊糖途径,同时敲除丙酮酸转化为乳酸以及合成乙酸酯通路中的相关基因。之后通过基因工程手段对菌株进行改造,相应的工程菌株在5 L补料分批培养中透明质酸的滴度达到28.7 g/L,这是当时文献报道中最高的结果。综上所述,在C.glutamicum中利用GSMM指导菌株改造,对相关代谢位点进行调控从而提高目标代谢物产量是一种切实可行的策略。
3.4 酿酒酵母Saccharomyces cerevisiae是第一个进行基因组测序的重要真核微生物,目前已经在生物燃料、制药等方面被广泛的应用[74-75]。S.cerevisiae的GSMM都是基于S.cerevisiae S288c基因组序列注释进行构建的。自2003年,Försber等[31]建立了第一个GSMM——iFF708之后,不同的研究者对S.cerevisiae GSMM进行了更新,目前已经构建了13个GSMM。以iFF708为基础,Duarte等[76]构建了iND750,将GSMM的腔室数目扩展到8个,Kuepfer等[77]对基因预测进行了改进并构建了iLL672,Herrgard等[27]则是将脂质的预测包含在GSMM中重构了iLN800;iMM904是Mo等[78]通过优化iND750基因预测能力和整合代谢组学数据而产生的;Orth等[28]在iMM904和iLL672的基础上使用jumberree的方法构建了Yeast1的模型;Dobson等[79]以Yeast1作为初始模型,整合iLN800对模型的脂质代谢进行优化,同时还允许研究者修改参数对模型进行约束,从而构建了Yeast4模型;通过优化鞘脂代谢和改进厌氧预测,Heavner等构建了Yeast5[80]和Yeast6模型[81];Aung等[82]通过增强了脂肪酸、甘油酯和甘油磷脂代谢,构建了Yeast7模型。以Yeast7模型为基础,Becker等[25]通过对多个数据库中注释的功能基因进行收集,通过分析处理,对GPRS进行了更新,同时添加酶约束、利用蛋白质3D结构聚类分析和泛基因组,构建了Yeast8。Yeast8是S.cerevisiae目前最全面的GSMM,同时也为S.cerevisiae的全细胞建模奠定了基础。Yeast8可以在多尺度水平上解析S.cerevisiae代谢机制,从而可以预测单核苷酸变异是如何转化为表型性状。
在S.cerevisiae中,借助GSMM提高目标产物的代谢工程策略已经有了广泛的应用,例如,Bro等[83]使用GSMM——iFF708利用MOMO算法,以乙醇为目标产物进行代谢调控,通过使用LIGANS数据库中的生化反应,将每一个基因分别插入模型进行评估,最终筛选出56个可能的代谢反应,通过热力学限制分析,最终得到8种最佳策略。研究者选择引入外源酶gapN这一策略进行后续研究,模型模拟得出这一策略可以完全消除甘油的形成,并使乙醇的收率提高了10%。结合之前相关合成通路的文献分析,通过基因工程手段在S.cerevisiae表达非磷酸化gapN最终使工程菌乙醇的得率提高了3%,在厌氧条件下甘油的产量降低了40%。Ng等[84]基于iMM904使用OptKnock算法对生产2, 3-丁二醇的酿酒酵母菌株进行设计,OptKnock敲除策略认为需要破坏乙醇脱氢酶(ADH) 途径,研究者通过构建不同的基因缺失菌株并进行分批培养,发现在微氧条件下敲除ADH1、ADH3和ADH5基因的菌株2, 3-丁二醇的产量增加了55倍,但是这个菌株会过量合成甘油,最终通过引入Bacillus subtilis和E.aerogenes的2, 3-丁二醇生物合成途径,最终Δadh1 Δadh3 Δadh5菌株在厌氧条件下培养获得了2.29 g/L产量和0.113 g/g得率。综上所述,基于GSMM可以发掘合成代谢产物的潜在操作位点,并且可以用于优化外源基因表达,大大减少了实验所需的操作与时间,并提高产物产量。
基于上述4种工业微生物GSMM的发展过程可以发现,经过修正和完善之后的GSMMs,与之前的GSMMs相比,增加了模型规模及相关的约束,提高了模型预测的准确性,因此当研究者使用GSMMs指导菌株设计,进行代谢调控时应当使用最新的模型。
4 总结与展望GSMM经过20多年的发展,已经开发了一系列模型自动构建工具和分析工具,越来越多的工业微生物相关GSMM出现,有助于研究人员在全局水平上对工业微生物的生理代谢功能进行理解和调控,结合各种算法工具,研究者可以对多种可能性进行模拟研究,筛选可能的代谢策略用于指导实验设计,这将加快对工业微生物代谢途径的理性改造,并且节约成本,使工业微生物具有更强的鲁棒性、更广的底物谱和更高的产物得率,促进工业生物技术的快速发展。
虽然GSMM已经成为目前代谢工程领域中使用最为广泛的一种方法,但是在模型的自动构建和提高模型预测准确性方面仍然面临着巨大的挑战:1) 虽然目前已经可以实现GSMM的快速自动重构,但是,对于一些复杂的问题,如工业微生物特定生物量方程等,仍然需要人工辅助进行[85],并且相关的自动重构工具也无法利用已经发表的代谢组学等数据,因此开发用于检索、整合和应用生长数据、代谢组学数据的新一代自动重构工具将是未来的一种趋势。2) 目前模型算法只允许将代谢流作为输入参数,缺乏对于温度、pH等环境条件动态调控数据的约束,这限制了GSMM在工业规模方面的应用方面。而使用机器学习算法不需要对模型的约束参数进行设定,只需要研究者提供一系列的训练数据就可自动对模型进行修正,提高模型预测的准确性,Nandi等[86]使用支持向量机(Support vector machine,SVM) 对E.coli的必需基因进行预测,该策略整合了适当样品平衡训练集,独特的生物体特异性基因型,表型的非常规组合表征必需基因的属性和学习算法的最佳参数,以生成最佳的机器学习模型,提高了必需基因预测的准确性。Oyetunde等[87]以E.coli为研究对象,手动搜集了100篇文章中1 200个E.coli作为样本,然后使用与实验匹配的约束条件的GSMM (iML1515) 模拟数据,然后进行数据补充和机器学习(例如,SVM,梯度提升树和堆叠回归模型中的神经网络),解决数据不完整的问题,最终通过主成分分析(Principal components analysis,PCA) 对生物生产的影响因素进行排序。这一混合框架显示在假定的生物过程和途径条件下对E.coli产量,得率和生产速率预测具有较高的准确性。因此,利用机器学习的方法对代谢流和代谢途径进行精准预测,是未来GSMM发展的趋势之一。3) GSMM仅涉及具有催化功能的基因和代谢物的量化关系,缺少对转录调控、细胞复制和蛋白定位等遗传信息的精确刻画,导致遗传信息的不完整。全细胞模型是各种类型生物网络的集成,用来描述细胞内DNA、RNA、蛋白质和代谢物等所有分子形成过程及相互作用机制,将微生物体内的所有的生命活动模块化,系统地研究各个模块之间的作用机制,构建数字细胞。全细胞模型是目前最复杂的微生物模型,可以对微生物胞内活动进行动态模拟,精确预测细胞表型,对于合成生物学未来的发展极为重要。有助于提高研究者对于微生物细胞功能和进化的理解,并且可能在未来助力于合成基因组的设计。但是其在数据获取、数据精炼、模型构建与整合、快速计算、分析与可视化、模型验证、合作与社区发展等方面仍然存在巨大挑战,这将需要建模师、计算机科学家、统计学家、生物信息学家、软件工程师和实验学家进行跨领域合作[88]。毫无疑问,以GSMM框架为核心,构建基于完备数据的多层次全细胞模型将是GSMM未来的发展方向。
| [1] |
Steensels J, Gallone B, Voordeckers K, et al. Domestication of industrial microbes. Curr Biol, 2019, 29(10): R381-R393. DOI:10.1016/j.cub.2019.04.025
|
| [2] |
刘立明, 陈坚. 基因组规模代谢网络模型构建及其应用. 生物工程学报, 2010, 26(9): 1176-1186. Liu LM, Chen J. Reconstruction and application of genome-scale metabolic network model. Chin J Biotech, 2010, 26(9): 1176-1186 (in Chinese). |
| [3] |
蔺玉萍, 王钦宏. 工业微生物遗传和环境扰动的调控和适应进化. 生物工程学报, 2019, 35(10): 1925-1941. Lin YP, Wang QH. Regulation and adaptive evolution of industrial microorganisms towards genetic and environmental disturbances. Chin J Biotech, 2019, 35(10): 1925-1941 (in Chinese). |
| [4] |
Durot M, Bourguignon PY, Schachter V. Genome-scale models of bacterial metabolism: reconstruction and applications. FEMS Microbiol Rev, 2009, 33(1): 164-190. DOI:10.1111/j.1574-6976.2008.00146.x
|
| [5] |
Thiele I, Palsson BØ. A protocol for generating a high-quality genome-scale metabolic reconstruction. Nat Protocols, 2010, 5(1): 93-121. DOI:10.1038/nprot.2009.203
|
| [6] |
Edwards JS, Palsson BO. Systems properties of the Haemophilus influenzae Rd metabolic genotype. J Biol Chem, 1999, 274(25): 17410-17416. DOI:10.1074/jbc.274.25.17410
|
| [7] |
Yizhak K, Benyamini T, Liebermeister W, et al. Integrating quantitative proteomics and metabolomics with a genome-scale metabolic network model. Bioinformatics, 2010, 26(12): i255-i260. DOI:10.1093/bioinformatics/btq183
|
| [8] |
Feng XY, Zhao HM. Investigating xylose metabolism in recombinant Saccharomyces cerevisiae via 13C metabolic flux analysis. Microb Cell Fact, 2013, 12: 114. DOI:10.1186/1475-2859-12-114
|
| [9] |
Krumholz EW, Libourel IGL. Thermodynamic constraints improve metabolic networks. Biophys J, 2017, 113(3): 679-689. DOI:10.1016/j.bpj.2017.06.018
|
| [10] |
Sánchez BJ, Zhang C, Nilsson A, et al. Improving the phenotype predictions of a yeast genome-scale metabolic model by incorporating enzymatic constraints. Mol Syst Biol, 2017, 13(8): 935. DOI:10.15252/msb.20167411
|
| [11] |
Brunk E, Sahoo S, Zielinski DC, et al. Recon3D enables a three-dimensional view of gene variation in human metabolism. Nat Biotechnol, 2018, 36(3): 272-281. DOI:10.1038/nbt.4072
|
| [12] |
Ma SY, Minch KJ, Rustad TR, et al. Integrated modeling of gene regulatory and metabolic networks in Mycobacterium tuberculosis. PLoS Comput Biol, 2015, 11(11): e1004543. DOI:10.1371/journal.pcbi.1004543
|
| [13] |
Dias O, Rocha M, Ferreira EC, et al. Reconstructing genome-scale metabolic models with merlin. Nucleic Acids Res, 2015, 43(8): 3899-3910. DOI:10.1093/nar/gkv294
|
| [14] |
Resende T, Correia DM, Rocha M, et al. Re-annotation of the genome sequence of Helicobacter pylori 26695. J Integr Bioinform, 2013, 10(3): 233.
|
| [15] |
Gomes D, Aguiar TQ, Dias O, et al. Genome-wide metabolic re-annotation of Ashbya gossypii: new insights into its metabolism through a comparative analysis with Saccharomyces cerevisiae and Kluyveromyces lactis. BMC Genomics, 2014, 15(1): 810. DOI:10.1186/1471-2164-15-810
|
| [16] |
Swainston N, Smallbone K, Mendes P, et al. The SuBliMinaL Toolbox: automating steps in the reconstruction of metabolic networks. J Integr Bioinform, 2011, 8(2): 186.
|
| [17] |
Henry CS, DeJongh M, Best AA, et al. High-throughput generation, optimization and analysis of genome-scale metabolic models. Nat Biotechnol, 2010, 28(9): 977-982. DOI:10.1038/nbt.1672
|
| [18] |
Karp PD, Latendresse M, Paley SM, et al. Pathway Tools version 19.0 update: software for pathway/genome informatics and systems biology. Brief Bioinform, 2016, 17(5): 877-890. DOI:10.1093/bib/bbv079
|
| [19] |
Wang H, Marcišauskas S, Sánchez BJ, et al. RAVEN 2.0: a versatile toolbox for metabolic network reconstruction and a case study on Streptomyces coelicolor. PLoS Comput Biol, 2018, 14(10): e1006541. DOI:10.1371/journal.pcbi.1006541
|
| [20] |
Arkin AP, Cottingham R, Henry C, et al. KBase: the United States department of energy systems biology knowledgebase. Nat Biotechnol, 2018, 36(7): 566-569. DOI:10.1038/nbt.4163
|
| [21] |
Karlsen E, Schulz C, Almaas E. Automated generation of genome-scale metabolic draft reconstructions based on KEGG. BMC Bioinformatics, 2018, 19(1): 467. DOI:10.1186/s12859-018-2472-z
|
| [22] |
Ye C, Xu N, Dong C, et al. IMGMD: a platform for the integration and standardisation of in silico microbial genome-scale metabolic models. Sci Rep, 2017, 7: 727. DOI:10.1038/s41598-017-00820-6
|
| [23] |
Aite M, Chevallier M, Frioux C, et al. Traceability, reproducibility and wiki-exploration for "à-la-carte" reconstructions of genome-scale metabolic models. PLoS Comput Biol, 2018, 14(5): e1006146. DOI:10.1371/journal.pcbi.1006146
|
| [24] |
Sánchez BJ, Nielsen J. Genome scale models of yeast: towards standardized evaluation and consistent omic integration. Integrat Biol, 2015, 7(8): 846-858. DOI:10.1039/C5IB00083A
|
| [25] |
Lu HZ, Li FR, Sánchez BJ, et al. A consensus S.cerevisiae metabolic model Yeast8 and its ecosystem for comprehensively probing cellular metabolism. Nat Commun, 2019, 10(1): 3586. DOI:10.1038/s41467-019-11581-3
|
| [26] |
Ghaffari P, Mardinoglu A, Asplund A, et al. Identifying anti-growth factors for human cancer cell lines through genome-scale metabolic modeling. Sci Rep, 2015, 5: 8183. DOI:10.1038/srep08183
|
| [27] |
Nookaew I, Jewett MC, Meechai A, et al. The genome-scale metabolic model iIN800 of Saccharomyces cerevisiae and its validation: a scaffold to query lipid metabolism. BMC Syst Biol, 2008, 2: 71. DOI:10.1186/1752-0509-2-71
|
| [28] |
Herrgård MJ, Swainston N, Dobson P, et al. A consensus yeast metabolic network reconstruction obtained from a community approach to systems biology. Nat Biotechnol, 2008, 26(10): 1155-1160. DOI:10.1038/nbt1492
|
| [29] |
Orth JD, Thiele I, Palsson BØ. What is flux balance analysis?. Nat Biotechnol, 2010, 28(3): 245-248. DOI:10.1038/nbt.1614
|
| [30] |
Österlund T, Nookaew I, Bordel S, 等. Mapping condition-dependent regulation of metabolism in yeast through genome-scale modeling. BMC Syst Biol, 2013, 7: 36. |
| [31] |
Förster J, Famili I, Fu P, et al. Genome-scale reconstruction of the Saccharomyces cerevisiae metabolic network. Genome Res, 2003, 13(2): 244-253. DOI:10.1101/gr.234503
|
| [32] |
Förster J, Famili I, Palsson BØ, et al. Large-scale evaluation of in silico gene deletions in Saccharomyces cerevisiae. OMICS: A J Integrat Biol, 2003, 7(2): 193-202. DOI:10.1089/153623103322246584
|
| [33] |
Heavner BD, Price ND. Comparative analysis of yeast metabolic network models highlights progress, opportunities for metabolic reconstruction. PLoS Comput Biol, 2015, 11(11): e1004530. DOI:10.1371/journal.pcbi.1004530
|
| [34] |
Lieven C, Beber ME, Olivier BG, et al. MEMOTE for standardized genome-scale metabolic model testing. Nat Biotechnol, 2020, 38(3): 272-276. DOI:10.1038/s41587-020-0446-y
|
| [35] |
Covert MW, Schilling CH, Palsson B. Regulation of gene expression in flux balance models of metabolism. J Theoret Biol, 2001, 213(1): 73-88. DOI:10.1006/jtbi.2001.2405
|
| [36] |
Feng XY, Xu Y, Chen YX, et al. Integrating flux balance analysis into kinetic models to decipher the dynamic metabolism of Shewanella oneidensis MR-1. PLoS Comput Biol, 2012, 8(2): e1002376. DOI:10.1371/journal.pcbi.1002376
|
| [37] |
Mahadevan R, Edwards JS, Doyle Ⅲ FJ. Dynamic flux balance analysis of diauxic growth in Escherichia coli. Biophys J, 2002, 83(3): 1331-1340. DOI:10.1016/S0006-3495(02)73903-9
|
| [38] |
Khandelwal RA, Olivier BG, Röling WFM, et al. Community flux balance analysis for microbial consortia at balanced growth. PLoS ONE, 2013, 8(5): e64567. DOI:10.1371/journal.pone.0064567
|
| [39] |
Burgard AP, Pharkya P, Maranas CD. OptKnock: a bilevel programming framework for identifying gene knockout strategies for microbial strain optimization. Biotechnol Bioeng, 2003, 84(6): 647-657. DOI:10.1002/bit.10803
|
| [40] |
King ZA, Feist AM. Optimizing cofactor specificity of oxidoreductase enzymes for the generation of microbial production strains—OptSwap. Ind Biotechnol, 2013, 9(4): 236-246. DOI:10.1089/ind.2013.0005
|
| [41] |
Ranganathan S, Suthers PF, Maranas CD. OptForce: an optimization procedure for identifying all genetic manipulations leading to targeted overproductions. PLoS Comput Biol, 2010, 6(4): e1000744. DOI:10.1371/journal.pcbi.1000744
|
| [42] |
Chowdhury A, Zomorrodi AR, Maranas CD. k-OptForce: integrating kinetics with flux balance analysis for strain design. PLoS Comput Biol, 2014, 10(2): e1003487. DOI:10.1371/journal.pcbi.1003487
|
| [43] |
Pharkya P, Burgard AP, Maranas CD. OptStrain: a computational framework for redesign of microbial production systems. Genome Res, 2004, 14(11): 2367-2376. DOI:10.1101/gr.2872004
|
| [44] |
Kim J, Reed JL, Maravelias CT. Large-scale Bi-level strain design approaches and mixed-integer programming solution techniques. PLoS ONE, 2011, 6(9): e24162. DOI:10.1371/journal.pone.0024162
|
| [45] |
叶超, 徐楠, 陈修来, 等. 应用代谢网络模型解析工业微生物胞内代谢. 生物工程学报, 2019, 35(10): 1901-1913. Ye C, Xu N, Chen XL, et al. Application of metabolic network model to analyze intracellular metabolism of industrial microorganisms. Chin J Biotechnol, 2019, 35(10): 1901-1913 (in Chinese). |
| [46] |
Adams BL. The next generation of synthetic biology chassis: moving synthetic biology from the laboratory to the field. ACS Synth Biol, 2016, 5(12): 1328-1330. DOI:10.1021/acssynbio.6b00256
|
| [47] |
Guiziou S, Sauveplane V, Chang HJ, et al. A part toolbox to tune genetic expression in Bacillus subtilis. Nucleic Acids Res, 2016, 44(15): 7495-7508.
|
| [48] |
Oh YK, Palsson BO, Park SM, et al. Genome-scale reconstruction of metabolic network in Bacillus subtilis based on high-throughput phenotyping and gene essentiality data. J Biol Chem, 2007, 282(39): 28791-28799. DOI:10.1074/jbc.M703759200
|
| [49] |
Goelzer A, Brikci FB, Martin-Verstraete I, et al. Reconstruction and analysis of the genetic and metabolic regulatory networks of the central metabolism of Bacillus subtilis. BMC Syst Biol, 2008, 2: 20. DOI:10.1186/1752-0509-2-20
|
| [50] |
Henry CS, Zinner JF, Cohoon MP, et al. iBsu1103: a new genome-scale metabolic model of Bacillus subtilis based on SEED annotations. Genome Biol, 2009, 10(6): R69. DOI:10.1186/gb-2009-10-6-r69
|
| [51] |
Kunst F, Ogasawara N, Moszer I, et al. The complete genome sequence of the Gram-positive bacterium Bacillus subtilis. Nature, 1997, 390(6657): 249-256. DOI:10.1038/36786
|
| [52] |
Barbe V, Cruveiller S, Kunst F, et al. From a consortium sequence to a unified sequence: the Bacillus subtilis 168 reference genome a decade later. Microbiology, 2009, 155(6): 1758-1775. DOI:10.1099/mic.0.027839-0
|
| [53] |
Tanaka K, Henry CS, Zinner JF, et al. Building the repertoire of dispensable chromosome regions in Bacillus subtilis entails major refinement of cognate large-scale metabolic model. Nucleic Acids Res, 2013, 41(1): 687-699. DOI:10.1093/nar/gks963
|
| [54] |
Hao T, Han BB, Ma HW, et al. In silico metabolic engineering of Bacillus subtilis for improved production of riboflavin, Egl-237, (R, R)-2, 3-butanediol and isobutanol. Mol Biosyst, 2013, 9(8): 2034-2044. DOI:10.1039/c3mb25568a
|
| [55] |
Kocabaş P, Çalık P, Çalık G, et al. Analyses of extracellular protein production in Bacillus subtilis –Ⅰ: genome-scale metabolic model reconstruction based on updated gene-enzyme-reaction data. Biochem Eng J, 2017, 127: 229-241. DOI:10.1016/j.bej.2017.07.005
|
| [56] |
Massaiu I, Pasotti L, Sonnenschein N, et al. Integration of enzymatic data in Bacillus subtilis genome-scale metabolic model improves phenotype predictions and enables in silico design of poly-γ-glutamic acid production strains. Microb Cell Fact, 2019, 18(1): 3. DOI:10.1186/s12934-018-1052-2
|
| [57] |
Yang D, Park SY, Park YS, et al. Metabolic engineering of Escherichia coli for natural product biosynthesis. Trends Biotechnol, 2020, 38(7): 745-765. DOI:10.1016/j.tibtech.2019.11.007
|
| [58] |
Blattner FR, Plunkett Ⅲ G, Bloch CA, et al. The complete genome sequence of Escherichia coli K-12. Science, 1997, 277(5331): 1453-1462. DOI:10.1126/science.277.5331.1453
|
| [59] |
Edwards JS, Palsson BO. The Escherichia coli MG1655 in silico metabolic genotype: its definition, characteristics, and capabilities. Proc Natl Acad Sci USA, 2000, 97(10): 5528-5533. DOI:10.1073/pnas.97.10.5528
|
| [60] |
Reed JL, Vo TDTT, Schilling CH, et al. An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR). Genome Biol, 2003, 4(9): R54. DOI:10.1186/gb-2003-4-9-r54
|
| [61] |
Feist AM, Henry CS, Reed JL, et al. A genome-scale metabolic reconstruction for Escherichia coli K-12 MG1655 that accounts for 1260 ORFs and thermodynamic information. Mol Syst Biol, 2007, 3: 121. DOI:10.1038/msb4100155
|
| [62] |
Orth JD, Conrad TM, Na J, et al. A comprehensive genome-scale reconstruction of Escherichia coli metabolism-2011. Mol Syst Biol, 2011, 7: 535. DOI:10.1038/msb.2011.65
|
| [63] |
Monk JM, Lloyd CJ, Brunk E, et al. iML1515, a knowledgebase that computes Escherichia coli traits. Nat Biotechnol, 2017, 35(10): 904-908. DOI:10.1038/nbt.3956
|
| [64] |
Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1, 4-butanediol. Nat Chem Biol, 2011, 7(7): 445-452. DOI:10.1038/nchembio.580
|
| [65] |
Ranganathan S, Tee TW, Chowdhury A, et al. An integrated computational and experimental study for overproducing fatty acids in Escherichia coli. Metab Eng, 2012, 14(6): 687-704. DOI:10.1016/j.ymben.2012.08.008
|
| [66] |
Becker J, Gieẞelmann G, Hoffmann SL, et al. Corynebacterium glutamicum for sustainable bioproduction: from metabolic physiology to systems metabolic engineering, in Synthetic Biology//Zhao H, Zeng AP, Eds. Synthetic Biology-Metabolic Engineering. Cham: Springer, 2016: 217-263.
|
| [67] |
Villadangos AF, Ordóñez E, Pedre, B, et al. Engineered coryneform bacteria as a bio-tool for arsenic remediation. Appl Microbiol Biotechnol, 2014, 98(24): 10143-10152. DOI:10.1007/s00253-014-6055-2
|
| [68] |
Hirasawa T, Shimizu H. Recent advances in amino acid production by microbial cells. Curr Opin Biotechnol, 2016, 42: 133-146. DOI:10.1016/j.copbio.2016.04.017
|
| [69] |
Kieldsen KR, Nielsen J. In silico genome-scale reconstruction and validation of the Corynebacterium glutamicum metabolic network. Biotechnol Bioeng, 2009, 102(2): 583-597. DOI:10.1002/bit.22067
|
| [70] |
Shinfuku Y, Sorpitiporn N, Sono M, et al. Development and experimental verification of a genome-scale metabolic model for Corynebacterium glutamicum. Microb Cell Fact, 2009, 8: 43. DOI:10.1186/1475-2859-8-43
|
| [71] |
Zhang Y, Cai JY, Shang XL, et al. a new genome-scale metabolic model of corynebacterium glutamicum and its application. Biotechnol Biofuels, 2017, 10: 169. DOI:10.1186/s13068-017-0856-3
|
| [72] |
Mei J, Xu N, Ye C, et al. Reconstruction and analysis of a genome-scale metabolic network of Corynebacterium glutamicum S9114. Gene, 2016, 575(2): 615-622. DOI:10.1016/j.gene.2015.09.038
|
| [73] |
Cheng FY, Yu HM, Stephanopoulos G. Engineering Corynebacterium glutamicum for high-titer biosynthesis of hyaluronic acid. Metab Eng, 2019, 55: 276-289. DOI:10.1016/j.ymben.2019.07.003
|
| [74] |
Steen EJ, Chan R, Prasad N, et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microb Cell Fact, 2008, 7: 36. DOI:10.1186/1475-2859-7-36
|
| [75] |
Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 2006, 440(7086): 940-943. DOI:10.1038/nature04640
|
| [76] |
Duarte NC, Herrgård MJ, Palsson BØ. Reconstruction and validation of Saccharomyces cerevisiae iND750, a fully compartmentalized genome-scale metabolic model. Genome Res, 2004, 14(7): 1298-1309. DOI:10.1101/gr.2250904
|
| [77] |
Kuepfer L, Sauer U, Blank LM. Metabolic functions of duplicate genes in Saccharomyces cerevisiae. Genome Res, 2005, 15(10): 1421-1430. DOI:10.1101/gr.3992505
|
| [78] |
Mo ML, Palsson BO, Herrgård MJ. Connecting extracellular metabolomic measurements to intracellular flux states in yeast. BMC Syst Biol, 2009, 3: 37. DOI:10.1186/1752-0509-3-37
|
| [79] |
Dobson PD, Smallbone K, Jameson D, et al. Further developments towards a genome-scale metabolic model of yeast. BMC Syst Biol, 2010, 4: 145. DOI:10.1186/1752-0509-4-145
|
| [80] |
Heavner BD, Smallbone K, Barker B, et al. Yeast 5-an expanded reconstruction of the Saccharomyces cerevisiae metabolic network. BMC Syst Biol, 2012, 6: 55. DOI:10.1186/1752-0509-6-55
|
| [81] |
Heavner BD, Smallbone K, Price MD, et al. Version 6 of the consensus yeast metabolic network refines biochemical coverage and improves model performance. Database, 2013, 2013: bat059.
|
| [82] |
Aung HW, Henry SA, Walker LP. Revising the representation of fatty acid, glycerolipid, and glycerophospholipid metabolism in the consensus model of yeast metabolism. Ind Biotechnol, 2013, 9(4): 215-228. DOI:10.1089/ind.2013.0013
|
| [83] |
Bro C, Regenberg B, Förster J, et al. In silico aided metabolic engineering of Saccharomyces cerevisiae for improved bioethanol production. Metab Eng, 2006, 8(2): 102-111. DOI:10.1016/j.ymben.2005.09.007
|
| [84] |
Ng CY, Jung MY, Lee J, et al. Production of 2, 3-butanediol in Saccharomyces cerevisiae by in silico aided metabolic engineering. Microb Cell Fact, 2012, 11: 68. DOI:10.1186/1475-2859-11-68
|
| [85] |
Gu CD, Kim GB, Kim WJ, et al. Current status and applications of genome-scale metabolic models. Genome Biol, 2019, 20(1): 121. DOI:10.1186/s13059-019-1730-3
|
| [86] |
Nandi S, Subramanian A, Sarkar RR. An integrative machine learning strategy for improved prediction of essential genes in Escherichia coli metabolism using flux-coupled features. Mol Biosyst, 2017, 13(8): 1584-1596. DOI:10.1039/C7MB00234C
|
| [87] |
Oyetunde T, Liu D, Martin HG, et al. Machine learning framework for assessment of microbial factory performance. PLoS ONE, 2019, 14(1): e0210558. DOI:10.1371/journal.pone.0210558
|
| [88] |
Macklin DN, Ruggero NA, Covert MW. The future of whole-cell modeling. Curr Opin Biotechnol, 2014, 28: 111-115. DOI:10.1016/j.copbio.2014.01.012
|
 2021, Vol. 37
2021, Vol. 37





