中国科学院微生物研究所、中国微生物学会主办
文章信息
- 杨永富, 耿碧男, 宋皓月, 胡蜜蜜, 何桥宁, 陈守文, 白凤武, 杨世辉
- Yang Yongfu, Geng Binan, Song Haoyue, Hu Mimi, He Qiaoning, Chen Shouwen, Bai Fengwu, Yang Shihui
- 合成生物学时代基于非模式细菌的工业底盘细胞研究现状与展望
- Progress and perspective on development of non-model industrial bacteria as chassis cells for biochemical production in the synthetic biology era
- 生物工程学报, 2021, 37(3): 874-910
- Chinese Journal of Biotechnology, 2021, 37(3): 874-910
- 10.13345/j.cjb.200626
-
文章历史
- Received: October 1, 2020
- Accepted: December 29, 2020
- Published: January 19, 2021
2. 上海交通大学 生命科学技术学院 微生物代谢国家重点实验室,上海 200240
2. State Key Laboratory of Microbial Metabolism, School of Life Sciences and Biotechnology, Shanghai Jiao Tong University, Shanghai 200240, China
微生物菌株在工业、农业、环境及医学等不同领域的广泛应用有着悠久的历史和越来越重要的作用,是现代工业生物技术的核心。随着现代生物学技术方法的发展与应用,研究人员逐渐从利用自然界分离得到的自然菌株或菌群,过渡到结合以诱变或/和自然进化为主要手段的非理性手段及基于系统与合成生物学技术的底盘细胞(Chassis cell) 理性设计策略,以实现优良细胞工厂的筛选、构建与改造。
人类基因组计划(Human genome project,HGP) 的成功实施加速了遗传密码解读技术与“组学”概念的革新。在HGP的推动下,基于新一代测序(Next generation sequencing,NGS) 与质谱(Mass spectrometry,MS) 分析的“组学(Omics)”技术,已经广泛应用于生命科学研究的各个领域,使科学家得以使用系统生物学(Systems biology) 方法突破以往对单个基因或蛋白质研究的限制,在整个生命系统层面更系统、更全面地研究“基因-RNA-蛋白-代谢-表型”之间的相互关系,进而对生命现象的遗传机制有更全面深入的理解[1]。同时,通过基因组、转录组、蛋白质组、代谢组、代谢流组以及表型组等不同组学研究得到的大量系统生物学数据,除了帮助解析微生物细胞工厂在不同水平的变化规律与调控机制之外,还可用于生物元件的挖掘与数学建模,通过模拟分析更深入认识与理解生命过程。21世纪初,在系统生物学的研究基础上,引入工程学思想策略,并与现代生物学、系统科学及合成科学融合,将生物技术系统化和标准化,形成了在理性设计指导下重组或从头合成具有特定功能人造生命为目标的“合成生物学”[2-3]。
底盘细胞是代谢反应发生的宿主细胞,是将合成的功能化元件、线路和途径等系统置入其中达到理性设计目的的重要合成生物学反应平台。由于细胞的复杂性高,人工置入的生物元件、线路或系统会受到细胞内原有代谢与调控途径的影响。因此,对细胞工厂基本成分(如生物元件与线路) 的挖掘与鉴定,对生物元件和线路与底盘细胞在能量与物质代谢层面的适配与通用规律的理解,对相关生物元件设计理论与工具的开发,以及对高通量自动组装与测试方法的完善等影响底盘细胞设计与构建的各方面研究工作都需持续投入、不断完善,才能尽快实现合成生物学“建物致知”以及“建物致用”的目标。
本文简述了合成生物学“设计-构建-测试-学习(Design-Build-Test-Learn,DBTL)”策略中设计、构建、测试和学习各步骤中相关技术方法的发展;概述了底盘细胞构建的策略以及部分重要模式微生物的研究进展,如酿酒酵母Saccharomyces cerevisiae、大肠杆菌Escherichia coli、枯草芽孢杆菌Bacillus subtilis和谷氨酸棒杆菌Corynebacterium glutamicum。重点阐述了部分非模式工业底盘细菌工厂的研究进展,如需钠弧菌Vibrio natriegens、拜氏不动杆菌Acinetobacter baylyi ADP1、运动发酵单胞菌Zymomonas mobilis、扬氏梭菌Clostridium ljungdahlii、产乙醇梭菌Clostridium autoethanogenum、乳酸菌Lactic acid bacteria、真氧产碱杆菌Cupriavidus necator和盐单胞菌Halomonas bluephagenesis等,同时也对优良、安全的人工合成微生物细胞工厂的构建与应用策略进行了展望。
1 合成生物学研究策略及关键技术进展合成生物学DBTL的研究策略与技术手段已广为人知,相关进展及具体底盘细胞的详细内容可参阅Trends in Biotechnologya、Metabolic Engineeringb及《合成生物学》等期刊的专辑综述文章c。本文仅就底盘细胞构建涉及的主要技术(图 1) 作简要描述。

|
| 图 1 合成生物学“设计-构建-测试-认知”研究策略涉及的主要关键技术 Fig. 1 Key technologies involved in synthetic biology "Design-Build-Test-Learn" strategy. CasHRA: Cas9-facilitated homologous recombination assembly; COOL: codon optimization online; DOOR: database for prOkaryotic OpeRons; FACS: fluorescence-activated cell sorting; FADS: fluorescence activated droplet sorting; GSM: genome-scale model; MMC: microbial microdroplet culture system; RSBP: registry of standard biological parts; TdT: terminal deoxynucleotidyl transferase. |
| |
a https://www.sciencedirect.com/journal/trends-in-biotechnology/special-issue/10G0XCGV33B
b https://www.sciencedirect.com/journal/metabolic-engineering/vol/50/suppl/C
c http://www.synbioj.com/CN/2096-8280/home.shtml
1.1 设计(Design)设计是合成生物学DBTL策略的基础,即在前期已有知识的基础上遵循一定的规则,利用现有的标准化生物元件对基因、代谢通路或基因组进行理性设计。其中标准化的生物元件,尤其是DNA水平描述的生物元件,在设计的过程中尤为重要。虽然从2003年开始建立了虚拟化的标准生物元件注册库(Registry of Standard Biological Parts),且随后还建立了多个其他类型的生物元件数据库[4],但截止到目前,标准化元件的数量仍然较少,其中关于非模式生物的更为缺乏d。
d http://parts.igem.org/Catalog
为方便不同层次的设计,针对不同的非标准化生物元件,研究人员开发了相应的分析工具,如对启动子进行预测分析的Softberry软件及在线分析网站、对原核生物操纵子(Operon) 进行预测分析的DOOR (Database for prOkaryotic OpeRons)[5]、对终止子进行预测分析的TransTermHP等[6-7]。美国宾夕法尼亚州州立大学Howard Salis教授课题组开发了一系列生物元件算法工具与流程,实现对核糖体结合位点(Ribosome binding site,RBS)、RBS文库、Operon、非重复元件、核糖开关(Riboswitch) 与RNA传感器等的预测、优化及设计[8-9]。
在进行基因元件合成时,由于密码子在不同物种中的使用频率不尽相同,往往会根据所选底盘细胞的密码子偏好性利用相关软件程序进行密码子优化,但是密码子优化设计尚无法完全保证目的基因在所选底盘细胞的表达效果,这可能与体内tRNA丰度及翻译效率相关[10]。研究表明,有些同义突变虽然不影响活性酶的形成,但会显著改变体内蛋白质的折叠机制,进而加快其前体蛋白在体内的降解,影响生长[11]。这些可能造成蛋白表达与降解的影响因素在元件设计、基因组分析或细胞工厂设计中都需要综合考虑。随着人工智能(Artificial intelligence,AI) 技术在生物学研究中的应用,生物元件的从头设计也成为可能。DeepMind团队开发的基于机器学习的人工智能算法Alphafold,通过对已有知识进行训练后,能够基于蛋白质一级序列进行蛋白质结构预测[12],相关算法、模型的开发及应用将蛋白质的结构设计与功能预测相连接,加快生物元件的理性设计[13]。
途径设计方面,与基于实验结果的方法相比,基于代谢网络计算分析的策略以其快速、综合和系统的优势,逐渐受到新途径挖掘与设计的青睐。中国科学院天津工业生物技术研究所(天工所) 马红武团队通过整合天然反应数据库MetaCyc和非天然反应数据库ATLAS建立复合代谢反应集,利用组合计算算法进行一碳化合物利用途径的挖掘设计,提出了多条理论上碳利用率为100%的非天然途径,通过对途径进行动力学陷阱评估、新酶挖掘和热力学途径优化,最终在体外实现了一条碳利用率达88%的途径[14]。基因组尺度的代谢网络模型也是一种广泛使用的途径设计方法[15],计算模拟结果可以指导底盘细胞途径改造以提高相关产品的产率。韩国高级科学技术研究院(KAIST) Sang-Yup Lee课题组结合大肠杆菌iJO1366模型模拟分析的结果,对合成芳香型聚酯途径中的相关基因进行敲除,使聚苯乳酸酯的产量显著增加[16]。
除此之外,设计平台软件和设计原则在设计过程中也至关重要,它们可以使研究者从全局出发,快速高效地完成生物元件、逻辑线路、代谢途径以及基因组的设计。例如为全基因组合成设计的BioStudio软件已被应用于酵母染色体合成项目Syn2.0的设计中[17]。同时,国际遗传工程机器竞赛(International Genetically Engineered Machine Copetition,iGEM) 作为培养和发展合成生物学研究后备人才的全球竞技性赛事,也推动了合成生物学教育与研究的发展,涌现出了FLAME (Framework-based layout and metacircuit design engine)、Easy BBK (EASY Biobrick Blueprint’s pacKage)、S-Din和CRAFT (Community- based Retro-synthetic Analysis Functional plaTform) 等软件作品[18]。这类软件可以从不同方面指导实验方向及提升实验效率,受到研究者的广泛关注。但是,由于生命系统的复杂性及目前数据与算法等方面的局限性,理性设计平台的真正实现还有很大差距。
1.2 构建(Build)构建过程包括DNA合成、大片段组装以及基因编辑。DNA合成技术在合成生物学的发展过程中起着十分重要的支撑作用,其不同于体内扩增,不需要使用模板,可直接根据DNA序列进行化学合成。目前的主流技术为固相亚磷酰胺化学合成法,由于合成长度、错配率与生产成本之间的制约关系,该方法在合成长度为60-mer的寡核苷酸时效率最优,合成长度一般不超过200 bp[19]。微阵列芯片的发展将会进一步提高合成效率,降低合成成本[20]。此外,随着无模板酶促合成法的发展及相关瓶颈的突破,将可实现更长寡核苷酸Oligo的合成[21]。
针对DNA大片段的拼接和组装,研究人员也开发了BioBrick、BglBricks、In-Fusion、Gateway、Golden Gate、Start-Stop及寡核苷酸连接介导的DNA组装(Oligonucleotides linkers mediated DNA assembly,OLMA) 等多种组装方法(表 1)[22-23]。Gibson组装法利用核酸外切酶、DNA聚合酶和DNA连接酶3种酶实现大片段DNA的无缝拼接,由于其操作简单且可以无缝拼接而被广泛使用,实现了将4个大于100 kb的片段在体外组装成583 kb完整基因组的目标[24]。另外,基于同源重组原理的多个DNA大片段体内高效组装方法也在酿酒酵母、大肠杆菌和枯草芽孢杆菌等常用宿主中实现[25]。例如Cas9-facilitated homologous recombination assembly (CasHRA) 方法已成功用于E. coli总长为1.05 Mb的大片段组装,其中包括了449个必需基因和267个生长相关基因[26]。
| Method | Advantages | Disadvantages | Example | References | |
| Oligo DNA synthesis | Chemical synthesis by phosphoramidite | Efficient, stable initial reaction | Toxic chemical reagents, length < 200 nt | Commercial oligo synthesis | [35] |
| Microarray chip | Low cost, high-throughput | Limited length, quantity and accuracy | Construction of oligonucleotide library | [20] | |
| TdT | Mild conditions, less DNA damage and byproducts | Not commercialization yet | Long DNA synthesis | [21] | |
| Large fragment assembly | BioBrick | Easy to optimize and automate assembly | Protein expression affected by 8-bp insertion | Construction of circuits and devices | [36] |
| BglBricks | Translation not affected by the 6-bp linker | Scar sequence | Construction of devices and fusion proteins | [37] | |
| Golden Gate | Scarless and efficient assembly | No restriction enzyme site on final product | Multiple assembly (e.g. TALEs, TALENs) | [38] | |
| Gibson | Simple operation, scarless assembly of large DNA fragments | Not suitable for assembly of fragments less than 200 bp |
4 fragments larger than 100 kb assembled into a 583-kb genome | [24] | |
| Gene editing | CRISPR-Cas (Exogenous) | Precise targeting, rich gRNA design tools and plasmids, diverse hosts and applications | Cytotoxicity of heterologous Cas proteins, restriction of PAM sites | Deletion, insertion, replacement, tagging, CRISPRi/a | [39] |
| CRISPR-Cas (Endogenous) | Native system, low cytotoxicity | Difficulty to apply in other organisms, limited tools, relatively low efficiency | Deletion, insertion, replacement, tagging, CRISPRi/a | [40] |
基因编辑技术从第一代锌指核酸酶(Zinc finger nucleases,ZFN)、第二代转录激活样效应因子核酸酶(Transcription activator-like effector,TAL effector) 发展到目前的第三代CRISPR-Cas技术。CRISPR-Cas技术具有效率高、操作快捷、效果准确等优点,是目前基因编辑的主流技术,尤其是Ⅱ型的CRISPR-Cas9或CRISPR-Cas12a (Cpf1) 基因编辑技术,已在多种生物体系中得到广泛应用。但是大多数底盘细胞CRISPR-Cas技术的使用需要外源引入Cas蛋白,会引起细胞毒性,从而限制了CRISPR系统的应用。为了解决这个问题,研究人员在多个微生物中开发出了基于微生物自身内源CRISPR-Cas系统的基因组编辑技术,如硫化叶菌Sulfolobus islandicus、西班牙盐盒菌Haloarcula hispanica、酪丁酸梭菌Clostridium tyrobutyricum、巴斯德梭菌Clostridium pasteurianum、卷曲乳杆菌Lactobacillus crispatus和运动发酵单胞菌Zymomonas mobilis等[27],可快速实现基因插入、缺失和单碱基编辑等基因编辑,部分操作编辑效率高达100%,且不会受到外源Cas蛋白毒性的影响[28]。该部分涉及的代表技术方法及其优缺点和应用举例见表 1,在不同的工业底盘细胞中使用的方法存在一定差异。
1.3 测试(Test)无论是酶、报告基因或启动子、RBS等单个生物元件,还是逻辑线路及模块化的代谢途径,在通过理性或非理性设计后,都会存在大量的突变体或候选目标,因此高效、准确和经济的检测方法对最佳生物元件及组合的选择至关重要,如对酶元件的表达、纯化与酶活测试,转录或翻译元件以及非天然途径的体外或体内测试,细胞工厂改造后的时空调控及对生长和代谢的影响等。传统的检测方法无法满足合成生物学对大量定量化生物元件、逻辑线路及代谢与调控途径组合的需求,目前已经尝试开发利用多种高通量或自动化的筛选与检测技术来提高测试的效率。如用于DNA组装、基因线路和天然产物活性检测、菌株筛选和表型检测的微流控芯片(Microfluidics) 技术、荧光激活液滴分选系统(Fluorescence activated droplet sorting,FADS),以及基于拉曼光谱、傅里叶变换红外光谱或近红外光谱等电学和先进光谱传感器的筛选技术[29]、Biolog表型芯片、微孔板高通量筛选及全自动高通量微液滴培养(Microbial microdroplet culture system,MMC) 等技术平台[30-33]。对于工业底盘细胞而言,使用高通量筛选技术快速筛选到相关工业性能提升的细胞工厂至关重要。
除了对已构建的细胞工厂进行生长测试外,对合成生物学DBTL整个闭环的试错性检测也十分重要。快速高效的自动化和工程化平台将是有效的解决手段。工程化平台可以快速大量试错,高效验证DBTL过程并快速积累数据和经验。目前包括美国和英国等多个国家已构建或在建的重大工程化平台约有25个,我国的天工所、国家蛋白质科学中心(上海)、武汉生物技术研究院及江南大学等研究机构,也都已构建了相关合成生物学自动化平台。另外,中国科学院深圳先进技术研究院(深圳先进院)、天津大学化工学院、华大生命科学研究院等机构也在建或拟建相关平台[34]。目前天工所的平台已经实现了自动化单基因克隆,通量达到300–600个/d,多模块化复杂质粒组装也达到100个/d的通量,组装正确率 > 90%,同时还实现了多种模式生物的自动化基因操作,但工程化平台尚不能投入非模式细菌的使用,并且建设与维护平台费用昂贵,使用技术难度高,导致目前尚未普及使用。
1.4 学习(Learn)学习过程作为合成生物学DBTL中的重要一环,为下一个循环改进设计提供指导,如基于系统生物学方法的组学技术进行“基因-RNA-蛋白-代谢-表型”不同层面的分析,构建基因型-表型和代谢调控网络的知识图谱等。学习这一过程涉及数据收集整合、数据分析、结果可视化和建模分析等。当前组学数据大量积累以及过程检测数据剧增,专用的公共数据库为数据的收集整理提供了极大的便利(表 2)。同时,不同的数据库还提供了自动化的数据下载程序或脚本,极大方便了数据收集的过程。虽然由于研究积累的原因,非模式微生物相关的数据远少于模式微生物,但模式微生物相关数据的处理学习,也可以为非模式微生物提供很好的借鉴。
收集整理的大量数据可以利用生物信息学和人工智能机器学习等相关技术进行分析以及构建数学模型,如基因组尺度的代谢网络模型和全细胞模型等。同时,这些数据也给依赖大数据的机器学习尤其是深度学习提供了可能[41],如利用机器学习开发的Alphafold可用于蛋白结构的从头预测[12]。基因组尺度的代谢网络模型作为一种有效的系统生物学学习工具,近年来也被广泛用于快速理解工业微生物的体内代谢以寻找改造靶点[42],或应用于酶功能预测、多细胞或有机体间相互作用的模拟,以及对人类疾病的理解等[15],并且也有相关的数据库逐渐建立,如BioModels、BiGG Models及Kbase[43-45]。
相关数据结果通过关联、集中查询与可视化,可以促进合成生物学的进一步发展。基于Web的可交互式数据可视化以其方便性和实用性深受科学研究人员喜爱,目前许多数据库都提供基于Web端可视化的结果展示,例如提供上万种已测序微生物基因组及其代谢途径的BioCyc,将分析工具与结果展示集成在一起,是基于Web端的网页可视化的优秀实例[46]。目前已经发表的唯一全细胞模型——生殖支原体全细胞模型[47],也建立了专门的可视化平台WholeCellViz[48],以动态展示其模拟过程,直观地理解内部发生的过程,便于学习总结。“学习”这一阶段总结的规律与结论,可以指导合成生物学DBTL其他阶段的模块,优化建立更为高效精简的合成生物学工作流程,用于不同底盘细胞的构建和性能优化。
2 底盘细胞构建目前,微生物底盘细胞的构建大致分为“自上而下的基因组精简”以及“自下而上的基因组合成”两种可单独使用或互补使用的策略。基因组精简是自上而下的目标导向改造,对基因组中非必需的编码区域和非编码区域进行大规模的删减,得到“最小基因组”。在基因组精简之前,可通过生物信息学或代谢网络模型分析,并结合已有必需基因与非必需基因数据库进行对比分析[49-50],初步实现对必需基因与非必需基因的鉴定。同时也常结合转座子突变库法、RNA干扰法以及基因组CRISPRi等实验方法[51-52],确定必需基因与非必需基因。
在非必需基因的删除过程中,基因组编辑和大片段敲除等技术发挥了关键作用,常用的技术有λ-Red同源重组系统、Cre-loxP重组系统、FLP-FRT敲除系统以及CRISPR-Cas系统介导的基因敲除方法等。例如通过λ-Red重组系统和I-SceⅠ核酸内切酶结合,实现基因的敲除、插入、位点定向诱变、基因替换及基因组无缝删除等操作[53-54];通过CRISPR-Nickases则实现了细菌基因组的大片段定向删除[55];而通过将λ-Red和CRISPR-Cas方法结合建立的Scarless Cas Assisted Recombineering (no-SCAR) 方法,在大肠杆菌体系中实现了基因的一步敲除与敲入[56]。此外,对于多基因同时敲除的方法也在不断研究完善,例如在大肠杆菌中通过CRISPR-Nickases技术实现了多位点同时编辑删除133 kb片段的工作[55]。借助不同的方法,多种模式微生物基因组精简的底盘细胞也取得了可喜进展[3],如基因组分别精简了38.9%和36.0%的大肠杆菌和枯草芽孢杆菌[57-58] (表 3)。自上而下的基因组精简方法相对简单,但是这一策略需要对基因组组成与功能有深入理解,同时也存在耗时长和成本高等局限性问题。
| Chassis cell | Genome (Mb) | Strategy | Proportion minimized | References |
| E. coli | 4.6 | λ-Red based homologous recombination | 38.9% | [59] |
| B. subtilis | 4.2 | Homologous recombination | 36.0% | [60] |
| C. glutamicum | 3.3 | Homologous recombination | 7.9% 22.0% (unfinished) |
[61] |
| P. putida | 6.1 | Mini-Tn5 targeted FLP-FRT recombination system | 4.7% | [62] |
| S. avermitilis | 9.0 | Homologous recombination or Cre-loxP recombination | 18.5% | [63] |
| S. cerevisiae | 12.1 | De novo synthesis, Sc2.0 | 8.0% (unfinished) | [17] |
| M. mycoides | 1.1 | De novo synthesis, Syn3.0 | 50.8% | [64] |
基因组合成是自下而上的正向工程学策略,由生物元件到模块再到基因组合成组装与底盘细胞构建。在基因组合成的过程中,系统生物学手段在合成组装的基因组性能鉴定和表征上也发挥了重要作用。美国Craig Venter团队于2016年从头合成了最小人工基因组Syn 3.0,大小为531 kb,共有473个基因[64],这一成果在合成生物学发展史中具有里程碑的意义,标志着原核生物基因组的人工合成(表 3)。真核生物中,单条染色体酿酒酵母的人工构建为基因组合成的研究也提供了成功范例[65]。酿酒酵母12 Mb全基因组人工合成计划(Sc2.0 Project) 近期也取得了突破性进展[66-69],预期在2020年实现酿酒酵母的基因组全人工合成[3]。表 3扼要总结了基因组精简和基因组合成策略在构建代表性底盘细胞方面的研究进展。
2.1 常见模式微生物底盘细胞构建底盘细胞是合成生物学的“硬件”基础,常用的模式微生物有酿酒酵母、大肠杆菌、枯草芽孢杆菌、谷氨酸棒杆菌等。目前在这些模式微生物中利用合成生物学手段已经构建了有特定功能的底盘细胞,或者进行了一定程度的基因组精简优化,以达到“建物致知”的目的(表 3)。
酿酒酵母Saccharomyces cerevisiae具有培养条件简单、生长繁殖快、通常认为是安全的(Generally regarded as safe,GRAS)、遗传操作工具多样以及遗传背景清晰等优势,是第一个基因组被完全测序的真核生物及广泛使用的真核模式生物之一。酿酒酵母在合成生物学研究领域不断取得突破性进展,自2009年纽约大学Jef Boeke教授提出人工合成酵母基因组计划(Sc2.0计划)并由该课题组于2011年成功实现了酿酒酵母6号染色体左臂和9号染色体右臂的设计与合成以来[70],已相继完成了6条酿酒酵母人工染色体(2、3、5、6、10、12号) 的设计与合成工作[4]。2016年,中国科学院上海植物生理生态研究所(中科院上海植生所) 覃重军团队开发出酵母内源同源重组方法CasHRA,并将Gibson组装与CasHRA结合,在酿酒酵母中成功组装了1.03 Mb的大片段[26]。2018年该团队成功设计并完成了将酿酒酵母16条天然染色体人工创建为具有完整功能单条染色体的里程碑式研究成果,是合成生物学工程化精准设计、建物致知理念的生动体现,为探索真核生物的染色体结构和功能进化,研究端粒功能及细胞衰老提供了实验模型,并为理解生命本质开辟了新的研究方向[65]。2019年,北京化工大学刘子鹤课题组开发了gRNA-tRNA array CRISPR-Cas9 (GTR-CRISPR) 系统,用tRNA序列将多个gRNA串联表达,可快速实现基因组的多基因同时改造和产量的提高,同时还可省去在大肠杆菌中进行克隆的步骤,为合成生物学及自动化的发展提供了又一实用的工具[71]。
大肠杆菌Escherichia coli作为研究微生物遗传、生理和代谢的模式菌株,同样由于遗传操作工具多样以及遗传背景清晰等优势成为重要的底盘细胞之一。目前,大肠杆菌基因组已经从减少5.6%提升到了38.9%[72]。美国哈佛大学医学院George Church课题组重编码了大肠杆菌基因组,成功设计出一个包括57个密码子,基因组仅为3.97 Mb的重组大肠杆菌[73]。近期,英国剑桥大学Jason Chin课题组人工合成并替换了大肠杆菌的全基因组,其中替换了两个丝氨酸密码子和一个终止密码子,最终重编程的合成菌株只含有61个密码子[74]。2009年,Church课题组在大肠杆菌中开发一种多重自动化基因组工程(Multiplex automated genome engineering,MAGE) 方法,通过引入一个合成序列库,能够在细胞群的多个目标染色体位点快速持续地产生序列多样性,从而可用于大规模的细胞编程和进化[75]。随后,这一技术与CRISPR-Cas9整合形成CRMAGE技术,将重组效率提高到90%,极大方便了对大肠杆菌基因组的编辑[76]。匈牙利科学院生物研究中心Pósfai团队在大肠杆菌中将CRMAGE与基因组重排整合,删除了大肠杆菌中的9个前噬菌体(Prophage) 序列和50个插入元件(Insertion elements),提高了其作为底盘细胞的稳定性[77]。
枯草芽孢杆菌Bacillus subtilis是革兰氏阳性菌芽孢杆菌的模式菌株,具有强大的蛋白表达系统。枯草芽孢杆菌基因组大小约为4.2 Mb,随着其基因组编辑等遗传工具的完善,其基因组精简也从7.7%提高到了36.0%[60]。基因组精简后的菌株miniBacillus PG10,遗传背景更简单,无其他二级代谢产物的影响,可以提高分泌蛋白及羊毛硫抗生素的生产[78-79]。目前,已在枯草芽孢杆菌中建立了CRISPR-Cas9、CRISPR-dCas9-α/ω、CRISPR-Cas9n和CRISPR-Cpf1/dCpf1等系统[80-82],湖北大学张桂敏课题组将dCas9-ω与CRISPR辅助的基于寡核苷酸退火的启动子改组(Oligonucleotide annealing based promoter shuffling,OAPS) 策略相结合,在枯草芽孢杆菌中实现了淀粉酶BLA表达量提高260倍的成果[81]。江南大学刘龙和王淼课题组基于CRISPR-dCas9系统在枯草芽孢杆菌中开发了木糖诱导的CRISPRi策略,用于提升乳糖-N-新四糖(LNnT)产量;在该系统中,dCas9被整合到基因组上,并由木糖诱导型启动子PxylA控制,当设计的gRNA与木糖同时存在时,即可启动靶向基因沉默,经过系统性优化后,LNnT效价在摇瓶中达到了2.30 g/L[83]。近期,该课题组通过建立能够激活和抑制基因表达的双功能丙酮酸反应基因电路,首次在枯草芽孢杆菌中实现了对细胞中枢代谢进行动态调控,同时阻断副产物的生成,使葡萄糖酸的效价从207 mg/L增长到802 mg/L,该策略可实现中心代谢流的动态控制,有利于中心代谢衍生产物的高效合成[84]。
谷氨酸棒杆菌Corynebacterium glutamicum因其高产谷氨酸的特性而受到关注,目前已对53株谷氨酸棒杆菌进行了基因组测序,且开发了完备的基因组编辑操作系统,包括同源重组介导的基因编辑体系、CRISPR-Cas9和最新的CRISPR- Cpf1/dCpf1等技术[85-88]。2016年,美国麻省理工学院Timothy Lu课题组将基于dCas9的CRISPRi系统成功应用于谷氨酸棒杆菌中,为低效率重组手段带来了重大改革[89]。但由于Cas9的细胞毒性问题,无法用于基因编辑,这一问题随着中国科学院分子植物科学卓越创新中心杨晟团队在谷氨酸棒杆菌中成功建立CRISPR-Cpf1系统而解决[90]。北京理工大学霍毅欣课题组随后对CRISPR-Cpf1系统进行了系统研究,优化提升了该系统在谷氨酸棒杆菌中的基因编辑效率[87]。基于CRISPR-Cpf1系统,浙江大学徐志南课题组在谷氨酸棒杆菌中设计了RE-CRISPR系统,可实现基因组编辑和转录抑制的双功能[91]。最近,中国科学院天津工业生物技术研究所郑平和孙际宾团队开发了基于CRISPR- dCpf1的多基因表达调控技术,实现了多个目标基因的快速表达调控[86]。这些技术体系的建立推动了谷氨酸棒杆菌底盘细胞的优化改造,成功实现了赖氨酸、丝氨酸、缬氨酸等多种氨基酸的合成[92]。
其他重要的模式微生物还包括可直接利用光能和二氧化碳转化为生物化学品的自养模式微生物蓝细菌Cyanobacteria以及从重要的环境底盘微生物发展为一个重要的工业微生物底盘细胞的恶臭假单胞菌Pseudomonas putida,可参见近期综述[93-97]。通过对模式微生物底盘细胞的改造,这些细胞工厂在工业应用领域取得了巨大进展,构建了可以生产青蒿素前体青蒿酸、紫杉醇前体紫杉二烯、β-胡萝卜素、维生素B12、类鸦片、大麻素类及其非天然类似物生产的细胞工厂[3, 98-101]。此外,Dupont和Genomatica公司也分别利用大肠杆菌实现了1, 3-丙二醇(1, 3-propanediol,PDO)[102]和1, 4-丁二醇(1, 4-butanediol,1, 4-BDO)[103]的工业化生产。江南大学刘立明实验室工程化改造大肠杆菌的生命周期,以扩大细胞尺寸和延长细胞寿命,用来提高微生物细胞工厂的性能,实现乳-羟基丁酸共聚酯和丁酸的高产[104]。最近,研究人员取得了通过基于理性设计的系统改造将酵母从产乙醇菌株改造为完全产油脂菌株[105]、将大肠杆菌改造为利用CO2生长的自养微生物或利用甲醇生长的甲基营养菌的突破性成果[106-107]。
2.2 重要非模式微生物底盘细胞构建模式微生物由于研究历史长,研究人员多,遗传工具丰富而积累了大量的实验数据和菌株资源,无论在微生物学基础理论研究还是工业生物应用都一直发挥着重要的作用。在工业生产实践中,还有诸多非模式菌株由于天然具有理想细胞工厂所需的优异特性而在各个领域被广泛应用,是生物制造的核心组成部分。这些理想细胞工厂的特点包括GRAS安全菌株、可高效利用多种底物尤其是廉价原料、生长周期短、目标产物产量高、副产物少、性能稳定、耐受极端环境条件、鲁棒性强等[108] (图 2)。
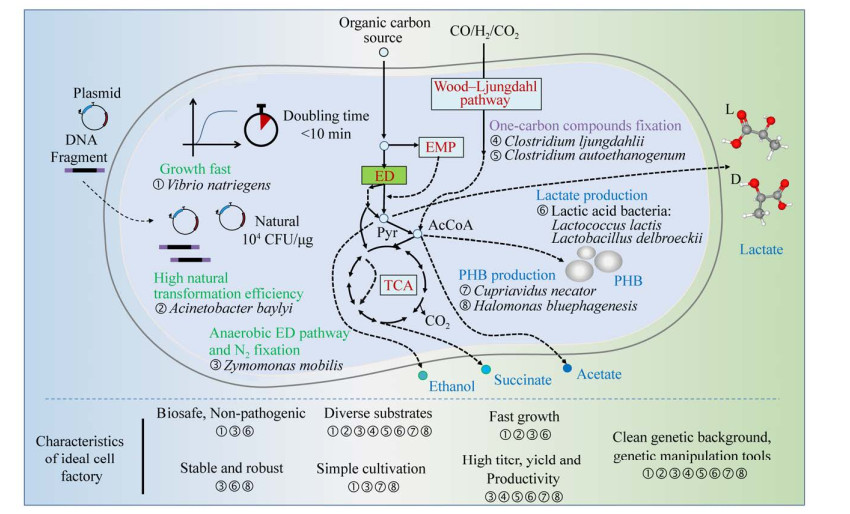
|
| 图 2 理想细胞工厂特点及部分非模式细菌细胞工厂主要特性 Fig. 2 Characteristics of ideal cell factories and a few non-model bacterial cell factories. AcCoA: acetyl-CoA; ED: entner-Doudoroff pathway; EMP: embden-Meyerhof-Parnas pathway; PHB: polyhydroxybutyrate; Pyr: pyruvate; TCA: tricarboxylic acid cycle. |
| |
工业非模式菌株由于早期遗传工具的匮乏而研究较少,随着测序与基因组编辑技术的发展、测序成本大幅降低、基因编辑效率不断提高,越来越多非模式微生物的基因组序列得到确定,相关基因表达载体和基因组编辑工具也不断建立完善,推动了重要非模式工业微生物的研究进展,并在短时间内发展成为重要的底盘细胞。如在工业生产上被广泛应用的异源蛋白表达优良宿主毕赤酵母Pichia pastoris以及活性天然产物和抗生素药物生产菌株放线菌Actinomyces等非模式微生物的遗传工具开发、基因组改造及工业应用研究都取得了重要突破[109-117]。基因组精简后的链霉菌Streptomycetes底盘细胞尽管与原始菌株相比无生长差异,但异源表达不同生物合成基因簇的产量高于原始菌株,表现了基因组精简后底盘细胞的优势及相关的工业应用价值[118]。
本文侧重比较工业生物技术领域具有特殊生理功能、利用一碳化合物及高效生产平台化合物的部分非模式细菌细胞工厂,包括生长迅速的需钠弧菌、高转化效率的拜氏不动杆菌和代谢途径特殊的乙醇生产模式菌株运动发酵单胞菌;具有一碳化合物利用能力的扬氏梭菌和产乙醇梭菌;生产平台化合物的重要工业菌株如乳酸生产菌株乳酸菌,聚3-羟基丁酸酯(PHB) 生产菌真氧产碱杆菌和盐单胞菌。这些菌株将具体分别综述,其特性比较参见图 2及表 4。
| Strain (Year discovered) | Physiology and growth conditions |
Genome (Mb) | Unique metabolic pathway | Genome manipulation tools | Products |
| V. natriegens (1958) | G–, facultative anaerobic, pH 5.5–9.5 (optimum 7.5), optimal temp 37 ℃ |
Chr1:3.24 Chr2:1.92 no plasmid | Anaerobic N2 fixation | CRISPR-Cas9 (CRISPRi), Cre-loxP system, λ-phage and SXT based recombination |
PHA, nano-selenium particles, alanine; β-carotene |
| A. baylyi ADP1 (1972) | G–, strictly aerobic, pH 3.0–9.0, 20–40 ℃ |
3.6 no plasmid | ED pathway, β-ketoadipate pathway | CRISPR-Cas9, Scarless HR based on sacB and tdk | Wax ester, triacylglycerol, tetrapyrrole, cyanophyte |
| Z. mobilis ZM4 (1924) | G–, facultative anaerobic, GRAS, pH 4.0–8.0, 24–45 ℃ |
2.2 4 plasmids | Anaerobic ED pathway, incomplete EMP pathway, incomplete TCA cycle, N2 fixation pathway |
CRISPR-Cas9, CRISPR-Cas12a, endogenous Type Ⅰ-F CRISPR system, HR |
Ethanol, succinate, levan, sorbitol; 2, 3-butanediol, isobutanol |
| C. ljungdahlii (1988) | G+, anaerobic, chemoautotrophs | 4.6 no plasmid | Wood-Ljungdahl pathway, Rnf-ATPase pathway | CRISPR-Cas9/dCas9, dCas9 based single base editing, CRISPR-Cas12a/ ddCas12a, Cre-loxP system |
Acetate, ethanol; butanol, isopropanol, mevalonate, butyrate |
| C. autoethanogenum (1994) | G+, strictly anaerobic, chemoautotrophs |
4.4 no plasmid | Wood-Ljungdahl pathway, Rnf-ATPase pathway | Endogenous Type I-B CRISPR cluster (undeveloped) | Acetate, ethanol, 2, 3-butanediol; PHB |
| L. lactis (1873) | G+, facultative anaerobic, optimal pH close to neutral, 10–40 ℃ |
2.5 4–7 plasmids | EMP pathway (homo-fermentation) | CRISPR-Cas9 (CRISPRi), Cre-loxP system, ssDNA oligonucleotide recombination |
Lactate; diacetyl, 2-butanol, acetoin, BDO, mannitol, acetate, ethanol |
| L. delbroeckii (1901) | G+, facultative anaerobic, pH 3.0–8.0 (optimum 5.5–6.2), optimum 40–45 ℃ |
1.86 1 or more plasmids | EMP pathway/PK pathway (homo-/hetero- fermentation) |
CRISPR-Cas9 | Lactate |
| C. necator H16 (1961) | G–, facultative, chemoautotrophs | Chr1:4.05 Chr2:2.91 1 plasmid |
ED pathway, incomplete EMP pathway, Double copy Calvin cycle |
CRISPR-Cas9 HR |
PHB; ethanol, acetoin, fatty acid, isoprenoid |
| H. bluephagenesis (2011) | G–, pH 5.0–11.0 (optimum 9.0) 0–45 ℃ (optimum 37 ℃) |
4.09 no plasmid | EMP pathway, methylcitrate cycle (artificially blocked to avoid competition for the use of propionyl-CoA) | CRISPR-Cas9 CRISPRi |
PHB; P34HB, PHBV, PhaR, acetoin, 5-aminolevulinic acid, threonine |
1) 需钠弧菌
需钠弧菌Vibrio natriegens为杆状海洋细菌,极端带有一条鞭毛,pH耐受范围为5.5–9.5,最适pH为7.5,最适生长温度为37 ℃,无致病性[119]。需钠弧菌是一种兼性厌氧菌,是目前已知生长周期最短的细菌(9.8 min),具备低氧环境下固定N2的能力[120]。需钠弧菌具有完整的糖酵解途径和三羧酸循环,除了生长迅速外,还具有在营养缺乏低代谢状态下长时间存活以及可利用广泛底物快速繁殖的能力,可以在各种碳源中生长,如糖蜜、蔗糖、麦芽糖、葡萄糖、果糖、阿拉伯糖、鼠李糖、甘露醇、甘油、葡萄糖酸、苹果酸、柠檬酸和苯乙酸等[120]。
需钠弧菌基因组大小约5.17 Mb,由大小约为3.24 Mb和1.92 Mb的两条染色体组成,无内源质粒[121-122]。目前多个需钠弧菌的基因组已被测序完成[121-124],并通过比较基因组学和CRISPRi全基因组范围的功能基因组研究,筛选出了基因组最小化改造可以删除的非必需区域和支持生长的核心基因[125],其中支持生长的基因主要富集在呼吸链通路[124]。近年来已经在需钠弧菌中构建了多种可稳定遗传的穿梭质粒作为菌株改造工具和异源蛋白表达载体,包括广泛宿主型复制子RSF1010、p15A和pMB1衍生质粒[125-126]。除此之外,需钠弧菌中已建立了3种DNA转化方法:接合转移、热击转化和电转。其中电转是使用最频繁且效率最高的转化方法,转化效率可达2×105–1×107 CFU/μg[124-125]。由于需钠弧菌具有天然转化能力,在对数期可有效摄取外源DNA,其转化可以不依赖于感受态制作[127-128]。
同时,在需钠弧菌体系中也开展了一系列生物元件开发和应用研究,检测了不同抗生素浓度对需钠弧菌的影响[125-126, 128],鉴定了一系列常用诱导型启动子,如Ptet、PBAD以及依赖IPTG或温度诱导的启动子[125-126]。研究发现在大肠杆菌中适用的多数生物元件如复制子、启动子、核糖体结合位点RBS、转录终止子、抗生素基因、报告基因和蛋白质降解标签等均适用于需钠弧菌[126]。基于需钠弧菌的无细胞蛋白合成体系也建立完成,可以完成体外转录和翻译过程[129-130]。中国科学院天工所王钦宏团队近期通过改变启动子及RBS区域成分,设计、构建并筛选了具有5个数量级范围不同转录和翻译强度的人工调控元件,为需钠弧菌合成生物学研究提供了简单、有效和标准化的组成型调控元件[131]。
需钠弧菌也建立了包括转座子突变、同源重组、CRISPR-(d)Cas9以及基因组多基因同时编辑(Multiplex genome editing by natural transformation,MuGENT) 等多种基因组编辑方法和工具[127-128],加快了菌株代谢工程改造和基因组精简优化进程。例如需钠弧菌噬菌体相关的整合性接合元件SXT的上调表达可以使同源重组效率提高约10 000倍,而ATCC 14048菌株中两个原噬菌体区域敲除的无原噬菌体突变体获得了比野生型对DNA损伤和低渗胁迫更好的鲁棒性[132]。2016年,美国合成基因组学有限公司(Synthetic Genomics,SGI) Matthew Weinstock等通过Dns核酸酶基因的敲除以及同源重组技术,得到了一株约194 kb DNA被敲除的菌株[125]。2020年SGI-DNA公司对需钠弧菌菌株进行了改造,获得了一株转化效率更高且基因组精简(敲除150个基因) 的菌株[133]。
上述遗传改造工具和系统生物学方法的建立和近期的研究,为开发需钠弧菌作为细胞工厂提供了良好的理论和技术基础。目前需钠弧菌已经被用于丙氨酸、聚羟基烷酸酯(Polyhydroxyalkanoate,PHA)、吲哚-3-乙酸和纳米硒等天然产品的生产;而经过代谢工程改造后的需钠弧菌也已用于生产重组蛋白、黑色素、β-胡萝卜素和紫罗兰等产品[120, 134-138]。综上所述,需钠弧菌底物利用范围广、底物摄取率高、生长快速、基因组改造工具丰富,具有成为像大肠杆菌一样的合成生物学研究底盘细胞和生物技术产业细胞工厂的潜力[125-126]。
2) 拜氏不动杆菌
拜氏不动杆菌Acinetobacter baylyi ADP1属于假单胞菌属,是一种严格需氧的革兰氏阴性细菌,含有一个约3.6 Mb的基因组,无内源质粒,G/C含量为40.3%。它最为突出的生理特点是具有很高的自然转化效率,同时能够在自然状态下摄取线性DNA并发生同源重组[139],被预测将会是下一代用于合成生物学和基因组工程的细菌宿主[140]。
拜氏不动杆菌可以利用多种碳源,如甘油、木糖、葡萄糖、醋酸、乙醇和丙酮酸等[141],在富营养培养基中生长迅速,倍增时间小于35 min[139]。拜氏不动杆菌具有独特的代谢途径,如降解各种长链二羧酸以及芳香族化合物的代谢途径,在自然状态下通过β-酮己二酸途径分解芳香族化合物,可以利用木质纤维素降解过程中衍生的芳香族化合物抑制物[139]。基因组测序发现ADP1缺失葡萄糖激酶、己糖激酶和葡萄糖转运蛋白磷酸转移酶系统的编码基因,推测ADP1不能直接磷酸化葡萄糖。同时ADP1还缺失编码6-磷酸果糖激酶和丙酮酸激酶的基因,因而推测ADP1可能使用Entner-Doudoroff (ED) 途径进行葡萄糖代谢[142]。
拜氏不动杆菌ADP1遗传操作技术已经成熟。除了其生长对数期可成功实现质粒DNA、连接产物、Gibson产物以及PCR产物的自然转化外,ADP1中已经构建使用了一系列调控生物元件,如广谱启动子、根据转录组数据选择的强启动子、人工合成启动子、针对强启动子Trc构建的强度不同的启动子库,以及利用双顺反子设计原则构建的强度不同的RBS突变库等[139]。拜氏不动杆菌ADP1也建立了多种基因编辑技术,包括基于sacB和tdk反筛选方法的无痕同源重组技术和编辑效率接近100%的CRISPR-Cas9基因编辑技术[139]。运用这些技术构建了转座子被敲除的无转座子菌株ADP1-Isx,这一菌株基因组更稳定、转化效率更高且细胞聚集和死亡率降低[140]。在这些技术的支持下,在ADP1菌株背景下也构建了单基因敲除菌株库[143]。
由于拜氏不动杆菌ADP1有天然积累脂质的趋势,使其成为脂质合成的有利底盘细胞,如通过异源还原酶复合物LuxCDE替换ADP1中的脂肪酰基辅酶A还原酶Acr1从而产生可修饰的蜡酯[144]。ADP1还具备天然生产三酰甘油的能力,通过代谢模型指导对其进行基因缺失编辑,进一步提高了三酰甘油在ADP1菌株中的产量[145]。通过敲除精氨酸代谢相关基因,从而改变ADP1菌株代谢流,使其蓝藻素产量提高[146]。
3) 运动发酵单胞菌
运动发酵单胞菌Zymomonas mobilis是一种兼性厌氧的革兰氏阴性菌,具有葡萄糖代谢速率快、乙醇得率高(可达最高理论得率的97%)、对高浓度乙醇和低pH耐受性好等优点,是目前已知唯一可在厌氧条件下利用ED途径的微生物。同时,运动发酵单胞菌为GRAS菌株,具有能够固定氮气,藿烷含量高(达总脂质50%),生长温度(24–45 ℃) 与pH (4.0–8.0) 范围广等生理特性。近年来被作为纤维素类生物质生物炼制的细胞工厂受到重视[147-148],是美国能源部国家可再生能源实验室(National Renewable Energy Laboratory,NREL) 及大湖生物能源研究中心(Great Lakes Bioenergy Research Center,GLBRC) 的主要底盘细胞之一。
运动发酵单胞菌基因组大小约为2.2 Mb,并含有4个内源质粒,全基因组共含有约2 000个编码基因[149]。运动发酵单胞菌的遗传操作工具也相对完备[149-151],除了常规的同源重组和CRISPR- Cas9基因编辑方法,已经成功开发了基于外源CRISPR-Cas12a以及内源I-F型CRISPR-Cas系统的基因编辑体系及基于dCas9的CRISPRi技术及gRNA设计网络工具CRISpy-pop[28, 152-155],为基因组编辑改造提供了多样化的遗传工具。同时,比较基因组学、转录组学、蛋白组学、代谢组学以及代谢网络模型等系统生物学研究也在运动发酵单胞菌中大量开展,解析了一系列突变菌株的基因型与表型之间的联系,并为开发生物元件及菌株理性设计改造提供了丰富的组学数据及遗传改造位点[148, 156-158]。
运动发酵单胞菌相关生物元件的预测与鉴定方法体系也在近期内不断建立完善,建立了sRNA预测鉴定方法以及生物调控元件双荧光报告基因检测系统,逐渐挖掘鉴定了一系列sRNA、启动子、RBS等相关生物元件。例如利用双荧光报告基因检测系统表征了基于系统生物学数据和生物信息学方法预测的38个启动子和4个RBS的强度,筛选确定了3个内源乙醇诱导性启动子[158-159],表征了sRNA与靶基因5′UTR之间的相互作用关系[160]。该系统可作为一种有效的工具,用于定量化鉴定运动发酵单胞菌非编码区域的相关调控元件与线路,增加可用于合成生物学研究的生物元件种类与数量。近期基于转录组、转录起始位点测序、蛋白组等多组学数据,研究确定了3 940个转录起始位点和2 091个转录终止位点,并确定了运动发酵单胞菌中–35和–10区域的保守序列,为进一步解析运动发酵单胞菌中的各种生物元件和理性设计改造提供了坚实的基础[156]。
运动发酵单胞菌野生型菌株可利用的碳源只有葡萄糖、果糖和蔗糖,研究人员利用代谢工程手段扩大了其底物利用谱,使其可以利用阿拉伯糖、木糖和甘露糖等多种碳源[148]。在应用方面,运动发酵单胞菌除了天然产乙醇外,还被改造用于PHB、D-乳酸、2, 3-丁二醇、山梨醇、乙醛、异丁醇和乳糖酸等的生物合成,此外,运动发酵单胞菌在食品、药品和作为益生菌的潜力也逐渐被挖掘,例如近期德国Farmako公司报道了利用运动发酵单胞菌开发的重组菌株Z. cannabinoidis®可进行大麻素生产,展现了运动发酵单胞菌有望在科研和应用的多个方向发挥作用的潜力[161-162]。
除了上述提到的有生长快速、转化效率高和底物代谢速率快等特殊生理性状的非模式细菌外,还有其他许多具有特殊生理性状的重要非模式细菌也被大量研究。例如革兰氏阳性菌丙丁梭菌Clostridium acetobutylicum在批量培养及磷限制时会出现产酸阶段和产溶剂阶段等几种不同的生理状态[163]。同时,由于其可天然生产丁醇、丙酮和丁酸等具有工业价值化学品的特性而受到研究人员的广泛关注,利用系统生物学和合成生物学方法对其特殊的生理性状进行了深入研究,为理解其不同生理条件下的调控机制及工业化应用奠定了基础[164-167]。革兰氏阴性菌假单胞菌Pseudomonas sp.也因其营养需求简单、环境适应性较强、繁殖速度快、内源代谢多样且能合成多种次生代谢物等优点而被开发为新的细胞工厂[168]。除了前文介绍的拜氏不动杆菌及提及的恶臭假单胞菌P. putida属于假单胞菌,铜绿假单胞菌P. aeruginosa、荧光假单胞菌P. fluorescens和绿针假单胞菌P. chlororaphis等假单胞菌也作为工业菌株被广为研究和应用[93, 169]。
2.2.2 一碳化合物利用非模式细菌虽然蓝藻等模式自养光合微生物具有固定CO2的能力,取得了以CO2为底物合成多种化学品的成果,但培养条件有限、整体固碳和转化效率尚低,不能满足工业应用的需求。随着合成生物学技术的发展,科学家们提出了第三代生物炼制的概念,其目标是将大气中的CO2固定转化为能源物质或其他化学品,形成绿色可循环的生产模式[170]。微生物固碳是第三代生物炼制的重要组成部分,天然可利用一碳气体(CO、CO2) 生产乙酸和乙醇的梭菌逐渐受到研究人员和产业界的关注,并致力于发挥其优势开发为生物技术生产的底盘细胞[171]。其中非模式细菌以扬氏梭菌和产乙醇梭菌为代表研究颇多,后者目前是美国朗泽公司(LanzaTech Inc.) 利用工业废气生产生物燃料的主要生产菌株[172]。
扬氏梭菌Clostridium ljungdahlii于1988年被发现,是一种典型的厌氧自养气体发酵产乙酸菌(Acetogen),能够将CO2和CO转化为生物质和多种代谢物[173],其基因组大小为4.63 Mb。扬氏梭菌进行CO和CO2固定的特殊代谢途径为Wood-Ljungdahl (WL) 途径,该途径是目前已知的6条天然固碳途径中最短且耗能最少的固碳途径,使用该途径使得扬氏梭菌可以将工业废气或来自家庭或农业废物的气化物主要成分CO、CO2和H2的混合气体转化为乙酸、乙醇、丁醇、丙酮、异丙醇、甲羟戊酸和丁酸等产品(表 4),展现了其工业化应用的潜力[171]。
产乙醇梭菌Clostridium autoethanogenum为严格厌氧革兰氏阳性细菌,与扬氏梭菌的基因组序列一致性高,二者具有基本相同的特性,均能够利用CO、CO2和H2进行乙醇和2, 3-丁二醇的生产。与扬氏梭菌最大的不同是,产乙醇梭菌含有6个脱氢酶和一个可能的甲酸转移酶[172]。另外,尽管目前尚未有内源基因编辑系统建立的报道,但实际上产乙醇梭菌含有内源Type Ⅰ-B型CRISPR-Cas基因簇[174],未来可利用该系统建立内源基因编辑体系。
上述两个梭菌的共同特点是拥有一种依赖Rnf复合物质子梯度保持能量的厌氧同质乙酸代谢模式,其Rnf-ATPase能量供应系统为产品的生产提供ATP来源,而细胞色素和钠离子都不参与能量生成这一过程[173]。该系统也可以作为研究质子转移、电子传递及Rnf复合物在供能过程中作用的模型系统[175]。两个梭菌中均报道了两条乙醇合成途径,一条为经典的AdhE途径,即从乙酰辅酶A出发,经过双功能醛/醇脱氢酶(AdhE) 两步氧化还原反应形成乙醇;另一条是以乙酸作为前体物质,经乙醛: 铁氧还蛋白氧化还原酶(Aldehyde oxidoreductase,Aor)催化形成乙醛,再由AdhE催化形成乙醇的AOR途径[171, 176]。
扬氏梭菌可以利用H2或CO作为质子供体,且发现当使用不同的质子供体时,其主要产物会有相应的变化。具体表现为,在H2供能时产乙酸,在CO供能时产乙醇[177]。当使用合成气进行生物乙醇生产时,乙醇的低产量制约了商业化的发展,尤其是在稳定期时,乙醇会被再次消耗利用。研究人员利用乙醇生成途径AdhE1和AdhE2单敲除和双敲除菌株,并结合13C和转录组数据,解析了扬氏梭菌体内的乙醇氧化途径。结果表明,扬氏梭菌中乙醇的合成是NADH依赖的,在以CO为碳源和能源时,生产和利用乙醇使用的途径相同,均为AOR途径。具体来说,其在对数期生成乙醇,在稳定期利用乙醇,乙醇的合成与能量生成及生物量的生成相偶联。这一调控机制的揭示说明了连续发酵生产技术将更适合梭菌的乙醇发酵生产,为利用梭菌高产乙醇奠定了理论基础[176]。
除了天然的乙醇和乙酸产品,研究人员开发了不同的遗传操作和基因编辑工具,以达到改造梭菌的目的(表 4)。研究人员引入了CRISPR-Cas9与CRISPR-Cas12a/ddCas12a系统,其中Cas12a的基因敲除效率可达80%–100%[178-179]。在此基础上,进一步开发的ddCas12a CRISPRi系统,基因抑制效率为80%[179]。研究人员还将丝氨酸位点特异性整合酶系统引入到了梭菌中,实现了8.5 kb产丁酸途径大片段的基因组的高效整合[180]。此外,还基于dCas9和胞嘧啶脱氨酶开发了单碱基编辑工具,可实现C到T的替换[181]。针对基因功能验证(敲除和回补),研究人员基于CRISPR-Cas9开发了24 nt的标签序列,使得基因原位回补的效率达91%[182]。该方法也可应用其他菌株和任何其他类型的CRISPR系统,提高基因功能验证的效率。除此之外,研究人员基于产乙醇梭菌成功开发了无细胞基因表达系统,表达量可达(236±24) μg/mL,为体外实验提供了技术支撑[183]。
上述两个梭菌的基因组均已测序发表,转录组、代谢组、代谢网络模型等系统生物学方法也已建立[172, 177, 184-186]。近期,基于在线气体分析和高分辨时间代谢组学的研究结果表明,在产乙醇梭菌体内气体吸收速率和细胞外副产物的振荡与生物量水平同步,并进一步通过热力学分析发现产乙醇梭菌体内的代谢振荡受热力学水平的调控[185]。这些方法将有助于研究人员从系统层面理解复杂的代谢网络和相关的生物过程,进一步解析梭菌利用一碳化合物的代谢途径及分子调控机制,为一碳化合物的工业化应用提供了坚实的技术手段及理论支撑。
上文以扬氏梭菌与产乙醇梭菌两个非模式细菌对一碳化合物的固定为例进行阐述。需要指出的是,除了上述两个非模式细菌可利用一碳化合物外,许多非模式微生物也可利用甲烷、甲醇和甲酸等其他一碳化合物;例如非模式细菌嗜甲烷菌对甲烷的利用及非模式真菌毕赤酵母对甲醇的利用等[109, 187]。毕赤酵母近期还被成功改造,实现了对CO2的固定利用[188]。相关固碳途径及一碳化合物利用微生物的介绍可参考近期综述文章[170, 189]。
2.2.3 生物塑料前体生产非模式细菌除了以上具有特殊生理特性和利用一碳化合物的非模式细菌外,还有一些工业微生物可以在特定环境生长时生产生物燃料或平台化合物产品。本文仅就生产生物塑料前体物质乳酸和PHA的几个菌株进行简要阐述。生物塑料的推广可帮助解决全球白色污染的问题,乳酸除了是一种用途广泛的重要化学品,可应用于食品、制药、皮革和纺织等工业领域,也是生产生物塑料的前体物质。
1) 乳酸生产菌
根据乳酸的手性(旋光性),可以分为L-乳酸和D-乳酸。由于D-乳酸不容易被人和动物利用,可能导致人体酸中毒等有害代谢,目前为止,市场仍然以L-乳酸为主。但在聚乳酸(Polylactic acid,PLA) 工业发展中,聚L-乳酸和聚D-乳酸混制得到的立体复合物比各自的单一聚合物耐热性能好,可大幅改善PLA的力学性能和物理性能。正因为D-乳酸在耐热聚乳酸工业中的重要地位,近些年D-乳酸的相关研究也备受关注[190-191]。微生物发酵是生产乳酸的首选方法,大约90%的乳酸是通过发酵产生;通过选择合适的乳酸发酵菌株还可生产具有高光学纯度的乳酸[192],其中生产乳酸最常用的野生型微生物是乳酸菌。
乳酸菌(Lactic acid bacteria,LAB) 是一组革兰氏阳性、低GC、耐酸性、大多不产芽孢的杆菌或球菌,是生产乳酸最常用的野生型微生物,有诸多代谢和生理学上的相似点。根据发酵最终产物,乳酸菌可分为同源发酵乳酸菌和异源发酵乳酸菌两类。同源发酵乳酸菌具有醛缩酶,并产生乳酸作为主要最终产物,主要有乳球菌、链球菌、小球菌、肠球菌及部分乳杆菌。而异源发酵乳酸菌除了产生乳酸外,还会产生其他副产物,如明串珠菌与一些乳杆菌。根据计算,同源和异源发酵时葡萄糖转化为乳酸的理论收率分别为1 g/g和0.5 g/g[193]。乳酸菌中已成功建立了多种转化方法(包括原生质体转化法、电转化法以及玻璃珠转化法等) 和基因敲除技术,并筛选鉴定了不同强度的启动子和终止子等生物元件,为其作为合成生物学底盘细胞奠定了坚实基础[194]。RecE/T辅助的CRISPR-Cas9高效基因编辑系统也在乳酸菌中成功建立[195]。
乳酸菌中进行乳酸发酵的通常是乳杆菌和乳球菌。乳杆菌不形成芽孢,一般厌氧并耐氧,耐酸或嗜酸,最适生长温度为30–40 ℃,最适pH为5.5–6.2[196]。乳杆菌中有的菌株含有一个或多个质粒。乳杆菌属是乳酸菌中最大和最多样化的属,乳杆菌基因组大小为1.23–4.91 Mb。德氏乳杆菌L. delbrueckii为该属的模式种,其基因组大小约为1.86 Mb,GC含量在49%–51%之间。部分德氏乳杆菌是嗜热乳酸菌,在50–55 ℃时仍能生长,在 < 10 ℃时仅有轻微生长。德氏乳杆菌分为4个亚种,各亚种发酵可利用的碳源不同。L. delbroeckii subsp. bulgaricus发酵范围最小,只能利用葡萄糖、乳糖和果糖;L. delbrueckii subsp. indicus所有菌株都能发酵葡萄糖、果糖、甘露糖和乳糖;L. delbroeckii subsp. delbrueckii的所有菌株发酵葡萄糖、果糖、甘露糖和蔗糖,一些菌株发酵纤维二糖、麦芽糖和海藻糖;L. delbroeckii subsp. lactis乳酸发酵的碳水化合物范围最广,可以利用葡萄糖、果糖、乳糖、麦芽糖、甘露糖、蔗糖和海藻糖,有些菌株还可以发酵纤维二糖和半乳糖[197]。
乳杆菌可通过噬菌体、转座子和其他可移动元件进行水平基因转移。另外,62.9%的乳杆菌编码CRISPR-Cas系统,其中Ⅱ型CRISPR-Cas系统出现的频率较高,但在乳酸乳球菌中很少出现CRISPR-Cas系统[198]。三种类型的CRISPR系统已在德氏乳杆菌中发现,并且在L. delbrueckii jakobsenii中发现了应用广泛的Ⅱ型CRISPR系统[199]。在德氏乳杆菌L. crispatus中,通过对内源Ⅰ型CRISPR-Cas系统的多种突变,使编辑效率达到了19%–100% [200]。
乳球菌为球形、无芽孢和兼性厌氧革兰氏阳性细菌,乳球菌生长温度通常在10–40 ℃,乳球菌基因组的大小为1.95–2.64 Mb[201]。大多数乳球菌携带4–7个3–130 kb的质粒。乳酸乳球菌Lactococcus lactis以其基因组小、遗传可操作性好、代谢简单以及基因工程工具齐全而成为乳球菌中的模式种。乳酸乳球菌的合成生物学工具也逐渐得到开发,如已广泛用于乳酸乳球菌同源和异源酶高效表达的乳链菌肽控制表达系统(Nisin-controlled gene expression system,NICE),锌调控表达系统(Zinc-regulated expression system,ZIREX),组成型天然启动子文库和基于Cre-loxP及CRISPR-Cas9/CRISPRi的多种基因组编辑技术[202-203]。近期,上海理工大学艾连中课题组在乳酸乳球菌中建立了受乳酸链球菌素诱导的CRISPR-dCas9基因干扰系统,该系统对单基因或多基因的沉默效率达99%[204]。诱导型CRISPRi系统的开发为在乳酸菌中筛选功能基因和代谢工程应用提供了高效的基因编辑工具。乳酸乳球菌不仅可以高产乳酸,通过代谢工程等方法改造还可以生产2-丁醇、双乙酰、丙氨酸、甘露醇、乙偶姻和BDO等产品[205-209]。
虽然其他细菌如芽孢杆菌、大肠杆菌、谷氨酸棒杆菌也可用于乳酸生产,但是由于大多数乳酸菌的高安全性、耐酸性及选择性生产D-或L-乳酸的能力使它们相对于其他细菌具有竞争优势[210],目前乳酸的工业化生产主要是基于乳酸菌的发酵[211]。乳酸工业生产遇到的挑战主要在光学纯度、耐酸性和底物成本几方面。乳酸的光学纯度对聚乳酸的物理性能有着重要影响,可通过代谢途径改造减少副产物生成、增加糖酵解途径代谢通量和诱变进化等方法进行改进。例如,通过过表达糖酵解途径和NADH脱氢酶基因以及异源ED代谢途径基因,使谷氨酸棒杆菌可生产光学纯度为99.9%的L-乳酸和D-乳酸[212]。耐酸性问题可以通过诱变、基因组改造和实验室适应性进化等多种方法增加乳酸菌耐酸性,获得在低pH下生产乳酸的突变株[213]。通过代谢工程使Lactobacillus plantarum成功地利用可再生木质纤维素生物质如玉米秸秆和高粱秸秆等生产D-乳酸,可低发酵生产的底物成本[190, 214]。
2) PHA生产菌
PHA是一种微生物在碳源充足而其他环境条件受限情况下在胞内积累的、可生物降解的聚合物[215]。PHA生产菌主要为革兰氏阴性菌,包括天然的产碱杆菌Cupriavidus、Alcaligenes、假单胞菌Pseudomonas、盐单胞菌Halomonas、芽孢杆菌Bacillus、气单胞菌Aeromonas以及非天然的重组大肠杆菌和谷氨酸棒杆菌等[216]。很多假单胞菌都能够合成PHA,且有许多都是高效合成PHA的优势菌株。恶臭假单胞菌相关基因组信息完备、遗传操作手段及工具丰富,主要利用脂肪酸从头合成途径或者脂肪酸β-氧化途径合成PHA (短链和长链PHA均可生产),其相关合成生物学进展也有较多综述[93-97, 217];同时该菌在环境及生物工程领域作用突出,能够代谢利用芳香化合物底物并整合进入聚合物链,还能利用多种底物生产定制的PHA[218]。PHB是最具代表性的PHA,有着较好的热塑性和生物可降解性,机械性质与聚丙烯、聚乙烯相似,在医疗应用、塑料包装、农业地膜等领域有着广阔的应用前景。真氧产碱杆菌和盐单胞菌主要通过乙酰辅酶A直接合成得到PHB。
真氧产碱杆菌Cupriavidus necator为一种兼性化能自养型细菌,能够利用碳水化合物、短链有机酸和醇类等广泛底物,在异养条件下能够达到极高的生长密度(OD600 > 200),使得其适合于许多生物技术应用,具备发展成各类生物化学品生产平台的潜力。当其处于氮源、磷酸盐或氧气缺乏的条件时,胞质内可积累超过85%干重的PHA,是合成短链PHA的首选菌种。
C. necator H16作为生产PHB的模式菌株,其基因组由4.05 Mb的染色体1和2.91 Mb的染色体2两条染色体及一个460 kb的大质粒3个部分组成,GC含量高达66.36%[219-221]。其中染色体1主要包含与细胞代谢和生命活动相关的必须基因,染色体2和大质粒主要包含从底物到末端电子受体(包括氢气氧化、二氧化碳固定和反硝化作用)相关的基因[222]。真氧产碱杆菌缺乏磷酸果糖激酶和6-磷酸葡萄糖激酶,在厌氧情况下通过ED途径进行糖酵解,在有氧情况下通过TCA循环提供生长需要的前体物及能量及产物的合成[223]。比较独特的是真氧产碱杆菌野生型菌株缺乏葡萄糖转运系统,无法代谢葡萄糖,不能直接利用几乎所有微生物偏好的葡萄糖。真氧产碱杆菌除了能利用有机物进行异养生长,也能通过基因组上双拷贝的卡尔文(Calvin-Benson-Bassham,CBB) 循环操纵子,在没有有机底物的情况下,利用CO2和H2作为碳和能源自养生长,使该菌逐渐成为研究CO2固定、解决CO2过量排放的模式菌株及一种有潜力的细胞工厂[216, 224]。
随着真氧产碱杆菌基因组测序的完成,系统生物学手段也得到广泛应用,为系统解析细胞内的代谢调控网络及其变化机制提供了手段。转录组、蛋白组学、代谢组学的应用揭示了不同培养条件下不同生长阶段脂质代谢、PHB合成等功能基因转录水平及代谢变化,有效揭示了PHB大量积累的原因[225-226]。同时,C. necator H16基因组尺度的代谢网络模型逐渐构建,可作为有效的工具预测不同情况下的代谢性能,其自养基因组规模的化学计量学模型RehMBEL1391由1 391个反应组成,包括229个输运反应和1 171个代谢物[227]。
真氧产碱杆菌相关的遗传改造、代谢工程与合成生物学研究方法也不断建立完善。目前已成功构建针对真氧产碱杆菌的自杀载体及pBBR1、pRP4、pSa、pKT230、RSF1010和pUC19等广谱质粒[228-229],建立了T7表达系统[230],筛选改造获取具有不同表达水平的内源启动子(PphaC、PacoD、PacoX、PacoE、PpdhE和PptR) 及异源诱导型启动子(如Plac和PBAD)[229-232],可对真氧产碱杆菌进行系统改造,构建生产高价值化合物的优良细胞工厂[216]。基于上述各类工具,研究人员进一步设计、构建并测试了一系列的启动子、RBS生物元件,同时优化了mRNA茎环结构以及A/U富集序列,以异戊二烯的生产为例,实现了对基因表达水平的量化调控,并在一定程度上分析了化合物的产量与产物合成酶的基因表达水平的相关性[233]。同年,研究人员通过对C. necator H16中应用最广泛的4个组成型启动子(PphaC1、PrrsC、Pj5和Pg25) 进行遗传修饰,构建了启动子文库,拓宽了启动子的应用范围[234]。
由于C. necator H16质粒骨架尺寸较大,克隆基因的大小通常会受到限制,近年来在该菌中也尝试建立了一些基因编辑的方法。其中基于Ⅱ型内含子的编辑技术,由于技术复杂而使用较少[228];而通过单交换重组整合自杀质粒的方法则耗时长,效率不确定[235]。基于电转的CRISPR-Cas9基因组编辑技术显著提高了真氧产碱杆菌遗传操作效率,其编辑效率达到78.3%–100%[236]。将C. necator H16作为底盘细胞的研究也在不断推进,研究人员利用还原性甘氨酸途径替代其自身的卡尔文循环,设计更有效的甘氨酸代谢,并利用短期进化实现途径活性优化,最终改造的菌株生物量和野生型大致相似,为进一步代谢改造和进化优化生产经济的产品打下了基础[237]。研究人员还以真氧产碱杆菌和大肠杆菌为底盘,利用异源I-CeuⅠ内切酶及内源核酸酶去除本体的染色体,得到无染色体的单核细胞(SimCells),这些SimCells具有宿主最小基因组作为核心遗传操作系统的潜力,可以作为简化的底盘细胞,编程实现目标产品的开发,而不受宿主基因组的干扰[238]。
同时,利用代谢工程改造、合成生物学手段开发真氧产碱杆菌生产其他高附加值生物化学品的研究也不断涌现,包括各类生物燃料和生物化学品产品[239-240]。近两年的研究进一步拓宽其产物谱,获得了异丙醇、乙偶姻、游离脂肪酸和异戊二烯等多元化产品[241-243]。
盐单胞菌Halomonas bluephagenesis是一类可以在高盐条件下生长的微生物,以嗜盐微生物作为工业生产菌株,其生长过程能够避免普通微生物污染,实现开放式连续发酵,具有低能耗、少淡水消耗、低固定资本投入、可连续生产等优势,从而大大降低了发酵生产成本。自1972年第一次报道在嗜盐微生物中发现PHA以来,越来越多的嗜盐微生物被发现可以合成PHA。清华大学陈国强课题组从新疆艾丁湖土壤样品中分离筛选得到的盐单胞菌Halomonas TD01 (后更名为Halomonas bluephagenesis TD01) 和盐单胞菌Halomonas campaniensis LS21可耐受高pH、高盐浓度,并有广泛的底物利用谱,在开放式连续发酵中表现出优异的PHA合成能力[244-246]。
合成生物学和代谢工程改造手段也逐渐应用到嗜盐微生物中[247]。越来越多的嗜盐微生物的全基因组序列得到测定,为其有效改造奠定了基础[248]。高表达proin构建的组成型染色体表达系统以及诱导表达各种基因的新型类T7诱导启动子生物元件已经在盐单胞菌H. bluephagenesis中成功开发[249-250]。CRISPR/Cas9和CRISPRi基因组编辑系统也已成功建立,可实现高效快捷的基因编辑与调控[251-252]。目前,陈国强课题组对嗜盐单胞菌通过启动子工程优化代谢途径表达[253],增加氧气利用率[254],调节控制NADH/NAD+比率[255],构建以葡萄糖为唯一碳源合成PHBV的代谢途径[256],利用TCA循环的染色体工程生产PHBV[257]等手段有效提高了多种PHA产量,取得了盐单胞菌混合碳源利用、基因组改造及超高PHA积累(92%)等成果。基于H. bluephagenesis的PHA已经实现了从实验室1-L发酵罐扩大到1 000-L中试的开放和连续化生产[215],以及多元化的PHA相关产品(如聚-3, 4羟基丁酸酯、聚3羟基丁酸-3羟基戊酸酯) 的生产[248, 258]。另外,基于该菌生产功能性PHA的工作仍在不断推进,得到可生产含12.5 mol% 3-羟基-5-己烯酸的功能性PHA的工程菌株H. bluephagenesis TDR4[259]。同时开发H. bluephagenesis作为下一代工业生物技术细胞工厂的研究也取得了阶段性进展,实现了生物表面活性剂蛋白PhaP、5-氨基乙酰丙酸、乙偶姻和苏氨酸等多种高附加值生物化学品在盐单胞菌细胞工厂的生产[260-262]。
除了上述细胞工厂主要生产单一平台化合物的非模式细胞工厂,具有多样代谢途径,生产多种产品的非模式工业微生物也得到发展。例如鲁棒性强及遗传工具丰富,已被用作多种蛋白质、化学品和抗生素生成的地衣芽孢杆菌Bacillus licheniformis[263-264];能够承受恶劣的环境条件和物理化学的压力、具有可高效降解多种芳香化合物和生物修复能力、无致病性的恶臭假单胞菌[93-94]等。同时,还有其他诸多特殊的非模式工业微生物也正在不断被发现,它们的进一步挖掘、开发和应用将推动对非模式工业微生物的深入理解、工业底盘细胞的理性设计及高效细胞工厂的构建与应用。
3 展望随着生物技术的不断革新飞跃,系统与合成生物学时代微生物细胞工厂的优化与构建拥有了更多的策略、方法与工具。尤其是通过合成生物学策略和手段对生物元件的挖掘与鉴定以及线路与系统的设计和测试,推动了通过“建物致知”对生命体系组成成分及调控机制的理解。然而,细胞工厂所处环境在不同时期对细胞工厂能量与物质代谢的时空影响及细胞工厂分子组成成分与调控网络的复杂性依然是制约动态生物元件及线路挖掘、设计与应用的瓶颈。在实现理性设计人工底盘细胞,构建具有不同特点的细胞工厂,实现“建物致用”目标的进程中需要结合模式与非模式微生物的优势,通过筛选改造更多具有不同特性的工业微生物细胞工厂,理解诸如生长速度快、底物利用效率高、环境耐受性强、产物产量高等优良表型关联的基因型及调控机制,才能更好地指导底盘细胞的理性设计。在这个过程中,可借鉴前期生物技术进步推动细胞工厂发展的成功经验,完善、发展系统与合成生物学方法技术,开发应用经济、高效的高通量智能装备,建立分子组学与表型组学研究平台,推动多层次系统生物学与表型组学大数据的解析、整合、模拟与可视化,建立高质量数字细胞,以指导基因组优化且具有生物安全特性的底盘细胞的理性设计与构建(图 3)。

|
| 图 3 底盘细胞理性设计及优异细胞工厂高效构建发展方向与策略 Fig. 3 Future direction and strategies for rational design and construction of microbial cell factories for diverse industrial applications. |
| |
对微生物细胞工厂的深入理解和应用与生物技术的发展相互促进:现代生物技术的快速发展提供了认识、改造及利用微生物细胞工厂的方法与工具,推动了人们对细胞工厂的理解,加快了工业微生物菌株的改造及在化工、医药、环境与农业等各个领域的应用。从文献报道的数量也可以看到,代谢工程和合成生物学研究在2010年之后稳步增加(图 4A)。与此同时,微生物细胞工厂和微生物底盘细胞的研究报道也同步不断增长(图 4B),尤其是微生物底盘细胞的报道与合成生物学的发展紧密相关(图 4)。从历年发表包括这些关键词的文章数量和趋势来看,微生物底盘细胞的研究依然严重滞后于微生物细胞工厂和合成生物学研究,到2019年底,一共只有196篇包括微生物底盘细胞的文章发表,仅分别为同时期微生物细胞工厂和合成生物学文章数量的8%和0.6% (图 4)。这在一定程度上说明了微生物底盘细胞的研究依然受到包括对细胞工厂理解不够全面和技术手段尚需进一步完善的制约。

|
| 图 4 PubMed数据库1980–2019年包括系统生物学、代谢工程与合成生物学技术(A),以及工业微生物、微生物细胞工厂与微生物底盘细胞(B) 关键词的文章发布数量 Fig. 4 The number of papers published from 1980 to 2019 including key words of systems biology, metabolic engineering and synthetic biology (A) or industrial microorganisms, microbial cell factories and microbial chassis (B) in PubMed database. |
| |
如上所述,目前在合成生物学DBTL策略中已经建立了一些革新技术手段和高通量自动化装备,加速了合成生物学研究的进展。但是仍需进一步开发完善经济高效的高通量智能装备,整合优化基因组学、转录组学、蛋白组学、代谢组学、表观组学等分子组学以及生长、形态、生理及环境适应性等表型组学的方法技术与研究平台,在不同环境条件下测试模式与非模式细胞工厂生物元件、线路与途径及其组合对细胞工厂物质与能量代谢的影响,解析控制相关特殊表型的分子元件及调控机制。同时,这些组学平台产生的海量数据也是理解细胞工厂优良特性分子机制的关键信息,如何解析、整合、模拟这些多层次大数据,并实现数据及结果的可视化展示是一个挑战。结合生物信息学与人工智能方法挖掘生物大数据背后的物理、数学规律,推动对复杂生命现象规律的认识,是实现合成生物学DBTL策略中理性设计的重要手段。
综合模式与非模式细胞工厂优势,推动工业微生物底盘细胞的理性设计与构建:由于众多研究人员的长期持续研究而使得模式微生物遗传工具齐全,菌株资源丰富,有系统的突变体或/和过表达菌株文库以及系统生物学与表型组学等实验数据,无论是在基础理论还是实践应用都一直发挥着重要的作用。模式微生物建立的方法和工具可以直接或改造后应用于非模式微生物,指导非模式微生物生物元件的挖掘及细胞工厂的优化。例如在模式微生物大肠杆菌和酿酒酵母中都建立了较高质量的全基因组尺度代谢网络模型并用于实际研究和应用工作中[265-266],这些代谢模型建立的工具和方法可以应用于非模式微生物高质量代谢模型的建立。除了代谢网络模型外,其他在模式微生物中快速发展的数学模型,如启动子的机器学习模型[267],也可被非模式微生物借鉴。
非模式微生物具有一些特殊的生长环境、代谢途径、生理性状及理想细胞工厂的优良特点,随着生物技术从早期的形态学观察发展到分子水平的分子生物学、系统生物学以及合成生物学,模式与非模式微生物之间研究的技术障碍正在逐步缩小。制约非模式微生物发展的基因组信息与遗传工具匮乏的瓶颈也随着高效经济的基因组测序、质谱技术、基因编辑工具及高通量检测设备的开发完善而逐步得以解决,推动了非模式微生物研究的进展。例如,盐单胞菌于2011年报道后,在不到10年的研究工作中,通过对基因组信息的解析及一系列基因组编辑工具的建立,成功改造了盐单胞菌细胞工厂,使盐单胞菌工程菌体内累积的生物塑料前体PHA产量达到细胞干重的92%,实现了PHA的1 000 L中试连续发酵生产[246, 248-249]。
在未来的研究中,可以从不同环境条件挖掘更多具有理想细胞工厂特性的微生物菌株,借鉴在模式微生物体系发展的工具、方法和数据,解析这些非模式细胞工厂的优良特性,结合模式与非模式细胞工厂的优势设计新的底盘细胞。另外,结合两者的优势同时在模式与非模式细胞工厂开展系统研究也有助于理解生物元件的通用性及其与底盘细胞的适配性等制约底盘细胞设计的因素,结合赋予它们不同优良特性的生物元件与调控线路,推动人工合成细胞工厂的理性设计与应用。例如中国台湾学者James Liao团队利用合成生物学的手段将来自非模式微生物的甲醇同化途径整合到模式微生物大肠杆菌中,结合模式和非模式细胞工厂的优势,使重组后的大肠杆菌能够以甲醇为唯一碳源进行生长[107]。清华大学陈国强课题组和北京大学欧阳颀课题组合作,通过对噬菌体基因组的挖掘,开发了一个可以在盐单胞菌和大肠杆菌中通用的类T7启动子系统,为模式与非模式细胞工厂的理性设计与构建提供了新思路[250]。
数字细胞及模式底盘细胞的构建与完善:多种细胞工厂分子与表型组学海量数据的积累及利用生物物理学与生物数学对复杂生命现象背后规律的确定也将推动数字细胞的构建。多组学数据的结合将提升数字细胞模型的质量,实现模型指导下底盘细胞构建的设计及其对内外环境因素变化反馈的预测,可以在高准确度模型预测的指导下减少实验工作。虽然目前已有相关自动化算法进行初始模型的构建[15],并且模式微生物大肠杆菌[265, 268]和酿酒酵母[266]等的模型也在逐渐更新完善,但是尚需进一步发展高通量检测、自然语言识别等方法技术,快速获取和挖掘实验和文献数据,实现模型的自动化修正及高质量数字细胞模型的快速自动化建立。
目前合成生物学正在生物元件、线路及设计语言平台等方面推动标准化。高质量数字细胞的建立可推动模式底盘细胞的建立,可与数字细胞的构建协调布局。麦肯锡在今年的《生物革命:创新将改变经济、社会和我们的生活》[269]报告中预计未来10–20年,4万亿美元的经济价值将由合成生物学主导。在合成生物学研究领域通过集成化、自动化和模块化策略对细胞工厂大规模理性设计改造的平台型公司一直受到投资公司的青睐。Ginkgo Bioworks目前的累积融资金额已达7.19亿美元,估值超过40亿美元。今年9月美国恩和生物(Bota Biosciences) 也完成了1 500万美元的A轮融资,将搭建生物铸造厂(Bio-foundry) 自动化高通量研究平台,实现细胞工厂的高效精准改造及高通量有效筛选。国内合成生物学公司蓝晶微生物2020年也完成了数千万元的A+轮融资,2021年又完成了近2亿元的B轮融资,该公司专注于利用合成生物学技术进行生物降解塑料PHA的生产,快速的融资推动了该合成生物学公司的产品研发和产业化落地。资本的动向一定程度上说明了社会的需求与发展方向,也说明了合成生物学技术对于未来的巨大作用,数字细胞模型和模式底盘细胞的构建与完善将进一步推动这一领域的发展。
人工合成底盘细胞的生物安全性控制与教育:合成生物学技术作为生命科学和生物技术的新兴前沿领域,将开启生物新经济(Bio-economy)时代的大门。在目前可以实现人工合成生命体的背景下,正确合理地利用这一科学技术将有利于全人类的共同生存与发展[270]。在不阻滞合成生物学的发展的同时,又保障生物安全性是一个需要关注、亟待解决并且不可跨越的关键问题,在国家、法律、技术和教育等不同层面都应该加以考虑。
除了在国家层面设立专门的研究与技术职责部门,制定一系列生物安全法律法规,完善监测、预警、报告与溯源等制度,提高应对生物安全风险的能力,保障国家安全和人民健康外,也需要将相关生物安全教育纳入学生培养教学大纲之中,将安全意识植入到新一代科技人才当中[271]。在学校教育的同时,也需要加强开展全民科普教育及科学传播,引导学生观看安全教育公开课,使公众正确认识新兴生物技术的两面性,理性对待生物安全问题,正确对待合成生物学的两面性[272]。
同时,人工细胞工厂的设计应考虑并提供安全性保障,如使用环境依赖型的复制子、依赖于小分子的自杀开关、或者引入无法与天然途径发生相互作用的系统等。美国哈佛医学院Pamela Silver课题组近期开发了一个pH敏感的自杀开关(Acidic termination of replicating population,acidTRP),可以在酸性条件下(pH 5.0) 终止种群复制,并与他们实验室前期开发的低温致死(Cryodeath) 开关[273]整合形成了一个双因素的防泄漏系统,该系统在保持进化稳定性的同时,可以保持小于千亿分之一(< 10–11) 的存活率[274]。哈佛医学院George Church课题组开发的基于CRISPR的“雏菊驱动” (Daisy-drive) 系统则是另一个很好的范例,该系统中的遗传元件像菊花链网络结构一样排列,每个遗传元件驱动下一个元件。该系统可以在本地复制使用等效自传播驱动系统实现的任何效果,但“雏菊驱动”的扩散能力因链条一端连续丢失非驱动元件而受到限制。这一系统保证了即使基因编辑的生物发生逃逸,它们也没有能力传播;并且当与阈值依赖相结合时,雏菊驱动可以简化地方社区的决策,可能成为地方生态工程的基础技术支撑[275]。此外,基于生物安全的底盘细胞进行研究也可以减少相关风险的产生,如人工设计的最小基因组底盘细胞只能在特定的环境中生存,其他适应不同环境的基因均被敲除,可在一定程度上防止相关基因及底盘细胞的扩散[276]。由于非模式微生物的多样性,从非模式微生物中挖掘不同的环境响应元件,将在开发合成生物学生物安全元件中发挥重要的作用。
| [1] |
Campbell K, Xia JY, Nielsen J. The impact of systems biology on bioprocessing. Trends Biotechnol, 2017, 35(12): 1156-1168. DOI:10.1016/j.tibtech.2017.08.011
|
| [2] |
赵国屏. 合成生物学: 开启生命科学"会聚"研究新时代. 中国科学院院刊, 2018, 33(11): 1135-1149. Zhao GP. Synthetic biology: unsealing the convergence era of life science research. Bull Chin Acad Sci, 2018, 33(11): 1135-1149 (in Chinese). |
| [3] |
张先恩. 中国合成生物学发展回顾与展望. 中国科学: 生命科学, 2019, 49(12): 1543-1572. Zhang XE. Synthetic biology in China: Review and prospects. Scientia Sinica Vitae, 2019, 49(12): 1543-1572 (in Chinese). |
| [4] |
温栾, 卢俊南, 钟伟, 等. 合成生物学设计技术. 中国细胞生物学学报, 2019, 41(11): 2060-2071. Wen L, Lu JN, Zhong W, et al. Design technology in synthetic biology. Chin J Cell Biol, 2019, 41(11): 2060-2071 (in Chinese). |
| [5] |
Mao XZ, Ma Q, Zhou C, et al. DOOR 2.0: presenting operons and their functions through dynamic and integrated views. Nucleic Acids Res, 2014, 42(D1): D654-659. DOI:10.1093/nar/gkt1048
|
| [6] |
Peters JM, Vangeloff AD, Landick R. Bacterial transcription terminators: the RNA 3'-end chronicles. J Mol Biol, 2011, 412(5): 793-813. DOI:10.1016/j.jmb.2011.03.036
|
| [7] |
Amarelle V, Sanches-Medeiros A, Silva-Rocha R, et al. Expanding the toolbox of broad host-range transcriptional terminators for proteobacteria through metagenomics. ACS Synth Biol, 2019, 8(4): 647-654. DOI:10.1021/acssynbio.8b00507
|
| [8] |
Borujeni AE, Cetnar D, Farasat I, et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Res, 2017, 45(9): 5437-5448. DOI:10.1093/nar/gkx061
|
| [9] |
Salis HM. The ribosome binding site calculator. Methods Enzymol, 2011, 498: 19-42.
|
| [10] |
赵晶, 张弓. 翻译组学: 方法及应用. 生命的化学, 2017, 37(1): 70-79. Zhao J, Zhang G. Translatomics: methods and applications. Chem Life, 2017, 37(1): 70-79 (in Chinese). |
| [11] |
Walsh IM, Bowman MA, Santarriaga IFS, et al. Synonymous codon substitutions perturb cotranslational protein folding in vivo and impair cell fitness. Proc Natl Acad Sci USA, 2020, 117(7): 3528-3534. DOI:10.1073/pnas.1907126117
|
| [12] |
Senior AW, Evans R, Jumper J, et al. Improved protein structure prediction using potentials from deep learning. Nature, 2020, 577(7792): 706-710. DOI:10.1038/s41586-019-1923-7
|
| [13] |
Zhou WJ, Šmidlehner T, Jerala R. Synthetic biology principles for the design of protein with novel structures and functions. FEBS Lett, 2020, 594(14): 2199-2212. DOI:10.1002/1873-3468.13796
|
| [14] |
Yang X, Yuan QQ, Luo H, et al. Systematic design and in vitro validation of novel one-carbon assimilation pathways. Metab Eng, 2019, 56: 142-153. DOI:10.1016/j.ymben.2019.09.001
|
| [15] |
Gu CD, Kim GB, Kim WJ, et al. Current status and applications of genome-scale metabolic models. Genome Biol, 2019, 20: 121. DOI:10.1186/s13059-019-1730-3
|
| [16] |
Yang JE, Park SJ, Kim WJ, et al. One-step fermentative production of aromatic polyesters from glucose by metabolically engineered Escherichia coli strains. Nat Commun, 2018, 9(1): 79. DOI:10.1038/s41467-017-02498-w
|
| [17] |
Richardson SM, Mitchell LA, Stracquadanio G, et al. Design of a synthetic yeast genome. Science, 2017, 355(6329): 1040-1044. DOI:10.1126/science.aaf4557
|
| [18] |
伍克煜, 刘峰江, 许浩, 等. 合成生物学基因设计软件: iGEM设计综述. 生物信息学, 2020, 18(1): 8-15. Wu KY, Liu FJ, Xu H, et al. Synthetic biology genetic design software: Review of iGEM software design. Chin J Bioinformat, 2020, 18(1): 8-15 (in Chinese). |
| [19] |
卢俊南, 罗周卿, 姜双英, 等. DNA的合成、组装及转移技术. 中国科学院院刊, 2018, 33(11): 1174-1183. Lu JN, Luo ZQ, Jiang SY, et al. Technologies for DNA synthesis, assembly, and transplantation. Bull Chin Acad Sci, 2018, 33(11): 1174-1183 (in Chinese). |
| [20] |
Kosuri S, Church GM. Large-scale de novo DNA synthesis: technologies and applications. Nat Methods, 2014, 11(5): 499-507. DOI:10.1038/nmeth.2918
|
| [21] |
Palluk S, Arlow DH, De Rond T, et al. De novo DNA synthesis using polymerase-nucleotide conjugates. Nat Biotechnol, 2018, 36(7): 645-650. DOI:10.1038/nbt.4173
|
| [22] |
李诗渊, 赵国屏, 王金. 合成生物学技术的研究进展——DNA合成、组装与基因组编辑. 生物工程学报, 2017, 33(3): 343-360. Li SY, Zhao GP, Wang J. Enabling technologies in synthetic biology—DNA synthesis, assembly and editing. Chin J Biotech, 2017, 33(3): 343-360 (in Chinese). |
| [23] |
Taylor GM, Mordaka PM, Heap JT. Start-Stop Assembly: a functionally scarless DNA assembly system optimized for metabolic engineering. Nucleic Acids Res, 2019, 47(3): e17. DOI:10.1093/nar/gky1182
|
| [24] |
Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods, 2009, 6(5): 343-345. DOI:10.1038/nmeth.1318
|
| [25] |
王培霞, 马渊, 吴毅. 大DNA体内组装技术进展. 生物加工过程, 2019, 17(1): 15-22. Wang PX, Ma Y, Wu Y. Advances in large DNA in vivo assembly. Chin J Bioproc Eng, 2019, 17(1): 15-22 (in Chinese). DOI:10.3969/j.issn.1672-3678.2019.01.003 |
| [26] |
Zhou JT, Wu RH, Xue XL, et al. CasHRA (Cas9-facilitated Homologous Recombination Assembly) method of constructing megabase-sized DNA. Nucleic Acids Res, 2016, 44(14): e124. DOI:10.1093/nar/gkw475
|
| [27] |
Zheng YL, Li J, Wang BY, et al. Endogenous type Ⅰ CRISPR-Cas: From foreign DNA defense to prokaryotic engineering. Front Bioeng Biotechnol, 2020, 8: 62. DOI:10.3389/fbioe.2020.00062
|
| [28] |
Zheng YL, Han JM, Wang BY, et al. Characterization and repurposing of the endogenous Type Ⅰ-F CRISPR-Cas system of Zymomonas mobilis for genome engineering. Nucleic Acids Res, 2019, 47(21): 11461-11475. DOI:10.1093/nar/gkz940
|
| [29] |
Ma X, Huo YX. The application of microfluidic-based technologies in the cycle of metabolic engineering. Synth Syst Biotechnol, 2016, 1(3): 137-142. DOI:10.1016/j.synbio.2016.09.004
|
| [30] |
崔金明, 刘陈立. 合成生物学中的高通量筛选与测量技术. 中国细胞生物学学报, 2019, 41(11): 2084-2090. Cui JM, Liu CL. High-throughput screening and measurement techniques in synthetic biology. Chin J Cell Biol, 2019, 41(11): 2084-2090 (in Chinese). |
| [31] |
Jian XJ, Guo XJ, Wang J, et al. Microbial microdroplet culture system (MMC): An integrated platform for automated, high-throughput microbial cultivation and adaptive evolution. Biotechnol Bioeng, 2020, 117(6): 1724-1737. DOI:10.1002/bit.27327
|
| [32] |
Sarnaik A, Liu A, Nielsen D, et al. High-throughput screening for efficient microbial biotechnology. Curr Opin Biotechnol, 2020, 64: 141-150. DOI:10.1016/j.copbio.2020.02.019
|
| [33] |
Zeng WZ, Guo LK, Xu S, et al. High-throughput screening technology in industrial biotechnology. Trends Biotechnol, 2020, 38(8): 888-906. DOI:10.1016/j.tibtech.2020.01.001
|
| [34] |
崔金明, 张炳照, 马迎飞, 等. 合成生物学研究的工程化平台. 中国科学院院刊, 2018, 33(11): 1249-1257. Cui JM, Zhang BZ, Ma YF, et al. Engineering platforms for synthetic biology research. Bull Chin Acad Sci, 2018, 33(11): 1249-1257 (in Chinese). |
| [35] |
Beaucage SL, Caruthers MH. Deoxynucleoside phosphoramidites—A new class of key intermediates for deoxypolynucleotide synthesis. Tetrahedron Letters, 1981, 22(20): 1859-1862. DOI:10.1016/S0040-4039(01)90461-7
|
| [36] |
Shetty RP, Endy D, Knight TF, J r. Engineering BioBrick vectors from BioBrick parts. J Biol Eng, 2008, 2: 5. DOI:10.1186/1754-1611-2-5
|
| [37] |
Anderson JC, Dueber JE, Leguia M, et al. BglBricks: A flexible standard for biological part assembly. J Biol Eng, 2010, 4: 1. DOI:10.1186/1754-1611-4-1
|
| [38] |
Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability. PLoS ONE, 2008, 3(11): e3647. DOI:10.1371/journal.pone.0003647
|
| [39] |
Pickar-Oliver A, Gersbach CA. The next generation of CRISPR-Cas technologies and applications. Nat Rev Mol Cell Biol, 2019, 20(8): 490-507. DOI:10.1038/s41580-019-0131-5
|
| [40] |
Liu ZQ, Dong HN, Cui YL, et al. Application of different types of CRISPR/Cas-based systems in bacteria. Microb Cell Fact, 2020, 19: 172. DOI:10.1186/s12934-020-01431-z
|
| [41] |
夏建业, 田锡炜, 刘娟, 等. 人工智能时代的智能生物制造. 生物加工过程, 2020, 18(1): 13-20. Xia JY, Tian XW, Liu J, et al. Intelligent biological manufacturing in the age of artificial intelligence. Chin J Bioproc Eng, 2020, 18(1): 13-20 (in Chinese). DOI:10.3969/j.issn.1672-3678.2020.01.002 |
| [42] |
叶超, 徐楠, 陈修来, 等. 应用代谢网络模型解析工业微生物胞内代谢. 生物工程学报, 2019, 35(10): 1901-1913. Ye C, Xu N, Chen XL, et al. Application of metabolic network model to analyze intracellular metabolism of industrial microorganisms. Chin J Biotech, 2019, 35(10): 1901-1913 (in Chinese). |
| [43] |
Li C, Donizelli M, Rodriguez N, et al. BioModels Database: An enhanced, curated and annotated resource for published quantitative kinetic models. BMC Syst Biol, 2010, 4: 92. DOI:10.1186/1752-0509-4-92
|
| [44] |
Arkin AP, Cottingham RW, Henry CS, et al. KBase: The united states department of energy systems biology knowledgebase. Nat Biotechnol, 2018, 36(7): 566-569. DOI:10.1038/nbt.4163
|
| [45] |
Norsigian CJ, Pusarla N, Mcconn JL, et al. BiGG Models 2020: multi-strain genome-scale models and expansion across the phylogenetic tree. Nucleic Acids Res, 2020, 48(D1): D402-D406.
|
| [46] |
Karp PD, Billington R, Caspi R, et al. The BioCyc collection of microbial genomes and metabolic pathways. Brief Bioinform, 2019, 20(4): 1085-1093. DOI:10.1093/bib/bbx085
|
| [47] |
Karr JR, Sanghvi JC, Macklin DN, et al. A whole-cell computational model predicts phenotype from genotype. Cell, 2012, 150(2): 389-401. DOI:10.1016/j.cell.2012.05.044
|
| [48] |
Lee R, Karr JR, Covert MW. WholeCellViz: data visualization for whole-cell models. BMC Bioinformatics, 2013, 14: 253. DOI:10.1186/1471-2105-14-253
|
| [49] |
Peng C, Lin Y, Luo H, et al. A comprehensive overview of online resources to identify and predict bacterial essential genes. Front Microbiol, 2017, 8: 2331. DOI:10.3389/fmicb.2017.02331
|
| [50] |
Juhas M, Reuß DR, Zhu BY, et al. Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering. Microbiology (Reading), 2014, 160(11): 2341-2351. DOI:10.1099/mic.0.079376-0
|
| [51] |
Goodall ECA, Robinson A, Johnston IG, et al. The essential genome of Escherichia coli K-12. mBio, 2018, 9(1): e02096-17.
|
| [52] |
Wang TM, Guan CG, Guo JH, et al. Pooled CRISPR interference screening enables genome-scale functional genomics study in bacteria with superior performance. Nat Commun, 2018, 9: 2475. DOI:10.1038/s41467-018-04899-x
|
| [53] |
Yang JJ, Sun BB, Huang H, et al. Multiple-site genetic modifications in Escherichia coli using lambda-Red recombination and I-SceⅠ cleavage. Biotechnol Lett, 2015, 37(10): 2011-2018. DOI:10.1007/s10529-015-1878-1
|
| [54] |
Kim J, Webb AM, Kershner JP, et al. A versatile and highly efficient method for scarless genome editing in Escherichia coli and Salmonella enterica. BMC Biotechnol, 2014, 14: 84. DOI:10.1186/1472-6750-14-84
|
| [55] |
Standage-Beier K, Zhang Q, Wang X. Targeted large-scale deletion of bacterial genomes using CRISPR-Nickases. ACS Synth Biol, 2015, 4(11): 1217-1225. DOI:10.1021/acssynbio.5b00132
|
| [56] |
Reisch CR, Prather KL. The no-SCAR (Scarless Cas9 Assisted Recombineering) system for genome editing in Escherichia coli. Sci Rep, 2015, 5: 15096. DOI:10.1038/srep15096
|
| [57] |
Hirokawa Y, Kawano H, Tanaka-Masuda K, et al. Genetic manipulations restored the growth fitness of reduced-genome Escherichia coli. J Biosci Bioeng, 2013, 116(1): 52-58. DOI:10.1016/j.jbiosc.2013.01.010
|
| [58] |
Ara K, Ozaki K, Nakamura K, et al. Bacillus minimum genome factory: effective utilization of microbial genome information. Biotechnol Appl Biochem, 2007, 46(3): 169-178. DOI:10.1042/BA20060111
|
| [59] |
Iwadate Y, Honda H, Sato H, et al. Oxidative stress sensitivity of engineered Escherichia coli cells with a reduced genome. FEMS Microbiol Lett, 2011, 322(1): 25-33. DOI:10.1111/j.1574-6968.2011.02331.x
|
| [60] |
Reuß DR, Altenbuchner J, Mäder U, et al. Large-scale reduction of the Bacillus subtilis genome: consequences for the transcriptional network, resource allocation, and metabolism. Genome Res, 2017, 27(2): 289-299. DOI:10.1101/gr.215293.116
|
| [61] |
Unthan S, Baumgart M, Radek A, et al. Chassis organism from Corynebacterium glutamicum—a top-down approach to identify and delete irrelevant gene clusters. Biotechnol J, 2015, 10(2): 290-301. DOI:10.1002/biot.201400041
|
| [62] |
Leprince A, Janus D, De Lorenzo V, et al. Streamlining of a Pseudomonas putida genome using a combinatorial deletion method based on minitransposon insertion and the Flp-FRT recombination system[M]//Weber W, E. F M. Synthetic Gene Networks. New York: Humana Press. 2012: 249-266.
|
| [63] |
Komatsu M, Uchiyama T, Ōmura S, et al. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism. Proc Natl Acad Sci USA, 2010, 107(6): 2646-2651. DOI:10.1073/pnas.0914833107
|
| [64] |
Hutchison ICA, Chuang RY, Noskov VN, et al. Design and synthesis of a minimal bacterial genome. Science, 2016, 351(6280): aad6253. DOI:10.1126/science.aad6253
|
| [65] |
Shao YY, Lu N, Wu ZF, et al. Creating a functional single-chromosome yeast. Nature, 2018, 560(7718): 331-335. DOI:10.1038/s41586-018-0382-x
|
| [66] |
Shen Y, Wang Y, Chen T, et al. Deep functional analysis of synⅡ, a 770-kilobase synthetic yeast chromosome. Science, 2017, 355(6329): eaaf4791. DOI:10.1126/science.aaf4791
|
| [67] |
Wu Y, Li BZ, Zhao M, et al. Bug mapping and fitness testing of chemically synthesized chromosome Ⅹ. Science, 2017, 355(6329): eaaf4706. DOI:10.1126/science.aaf4706
|
| [68] |
Xie ZX, Li BZ, Mitchell LA, et al. "Perfect" designer chromosome Ⅴ and behavior of a ring derivative. Science, 2017, 355(6329): eaaf4704. DOI:10.1126/science.aaf4704
|
| [69] |
Zhang WM, Zhao GH, Luo ZQ, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome. Science, 2017, 355(6329): eaaf3981. DOI:10.1126/science.aaf3981
|
| [70] |
Dymond JS, Richardson SM, Coombes CE, et al. Synthetic chromosome arms function in yeast and generate phenotypic diversity by design. Nature, 2011, 477(7365): 471-476. DOI:10.1038/nature10403
|
| [71] |
Zhang YP, Wang J, Wang ZB, et al. A gRNA-tRNA array for CRISPR-Cas9 based rapid multiplexed genome editing in Saccharomyces cerevisiae. Nat Commun, 2019, 10: 1053. DOI:10.1038/s41467-019-09005-3
|
| [72] |
Chi HT, Wang XL, Shao Y, et al. Engineering and modification of microbial chassis for systems and synthetic biology. Synth Syst Biotechnol, 2019, 4(1): 25-33. DOI:10.1016/j.synbio.2018.12.001
|
| [73] |
Ostrov N, Landon M, Guell M, et al. Design, synthesis, and testing toward a 57-codon genome. Science, 2016, 353(6301): 819-822. DOI:10.1126/science.aaf3639
|
| [74] |
Fredens J, Wang KH, De La Torre D, et al. Total synthesis of Escherichia coli with a recoded genome. Nature, 2019, 569(7757): 514-518. DOI:10.1038/s41586-019-1192-5
|
| [75] |
Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894-898. DOI:10.1038/nature08187
|
| [76] |
Ronda C, Pedersen LE, Sommer MO, et al. CRMAGE: CRISPR optimized MAGE recombineering. Sci Rep, 2016, 6: 19452. DOI:10.1038/srep19452
|
| [77] |
Umenhoffer K, Draskovits G, Nyerges Á, et al. Genome-wide abolishment of mobile genetic elements using genome shuffling and CRISPR/Cas-assisted MAGE allows the efficient stabilization of a bacterial chassis. ACS Synth Biol, 2017, 6(8): 1471-1483. DOI:10.1021/acssynbio.6b00378
|
| [78] |
Uárez RA, Stülke J, van Dijl JM. Less is more: toward a genome-reduced Bacillus cell factory for "difficult proteins". ACS Synth Biol, 2019, 8(1): 99-108. DOI:10.1021/acssynbio.8b00342
|
| [79] |
van Tilburg AY, van Heel AJ, Stülke J, et al. MiniBacillus PG10 as a convenient and effective production host for lantibiotics. ACS Synth Biol, 2020, 9(7): 1833-1842. DOI:10.1021/acssynbio.0c00194
|
| [80] |
Wu YK, Liu YF, Lv XQ, et al. CAMERS-B: CRISPR/Cpf1 assisted multiple-genes editing and regulation system for Bacillus subtilis. Biotechnol Bioeng, 2020, 117(6): 1817-1825. DOI:10.1002/bit.27322
|
| [81] |
Lu ZH, Yang SH, Yuan X, et al. CRISPR-assisted multi-dimensional regulation for fine-tuning gene expression in Bacillus subtilis. Nucleic Acids Res, 2019, 47(7): e40. DOI:10.1093/nar/gkz072
|
| [82] |
Liu DY, Huang C, Guo JX, et al. Development and characterization of a CRISPR/Cas9n-based multiplex genome editing system for Bacillus subtilis. Biotechnol Biofuels, 2019, 12: 197. DOI:10.1186/s13068-019-1537-1
|
| [83] |
Dong XM, Li N, Liu ZM, et al. CRISPRi-guided multiplexed fine-tuning of metabolic flux for enhanced lacto-N-neotetraose production in Bacillus subtilis. J Agric Food Chem, 2020, 68(8): 2477-2484. DOI:10.1021/acs.jafc.9b07642
|
| [84] |
Xu XH, Li XL, Liu YF, et al. Pyruvate-responsive genetic circuits for dynamic control of central metabolism. Nat Chem Biol, 2020, 16(11): 1261-1268. DOI:10.1038/s41589-020-0637-3
|
| [85] |
Wu M, Xu Y, Yang J, et al. Homing endonuclease I-SceⅠ-mediated Corynebacterium glutamicum ATCC 13032 genome engineering. Appl Microbiol Biotechnol, 2020, 104(8): 3597-3609. DOI:10.1007/s00253-020-10517-y
|
| [86] |
Li MY, Chen JZ, Wang Y, et al. Efficient multiplex gene repression by CRISPR-dCpf1 in Corynebacterium glutamicum. Front Bioeng Biotechnol, 2020, 8: 357. DOI:10.3389/fbioe.2020.00357
|
| [87] |
Zhang J, Yang FY, Yang YP, et al. Optimizing a CRISPR-Cpf1-based genome engineering system for Corynebacterium glutamicum. Microb Cell Fact, 2019, 18: 60. DOI:10.1186/s12934-019-1109-x
|
| [88] |
杨娟娟, 马晓雨, 王晓蕊, 等. 谷氨酸棒杆菌基因编辑的研究进展. 生物工程学报, 2020, 36(6). Yang JJ, Ma XY, Wang XR, et al. Advances in gene editing of Corynebacterium glutamate. Chin J Biotech, 2020, 36(6) (in Chinese). DOI:10.13345/j.cjb.190403.(inChinese) |
| [89] |
Cleto S, Jensen JVK, Wendisch VF, et al. Corynebacterium glutamicum metabolic engineering with CRISPR interference (CRISPRi). ACS Synth Biol, 2016, 5(5): 375-385. DOI:10.1021/acssynbio.5b00216
|
| [90] |
Jiang Y, Qian FH, Yang JJ, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun, 2017, 8: 15179. DOI:10.1038/ncomms15179
|
| [91] |
Liu W, Tang DD, Wang HJ, et al. Combined genome editing and transcriptional repression for metabolic pathway engineering in Corynebacterium glutamicum using a catalytically active Cas12a. Appl Microbiol Biotechnol, 2019, 103(21/22): 8911-8922. DOI:10.1007/s00253-019-10118-4
|
| [92] |
Becker J, Rohles CM, Wittmann C. Metabolically engineered Corynebacterium glutamicum for bio-based production of chemicals, fuels, materials, and healthcare products. Metab Eng, 2018, 50: 122-141. DOI:10.1016/j.ymben.2018.07.008
|
| [93] |
Weimer A, Kohlstedt M, Volke DC, et al. Industrial biotechnology of Pseudomonas putida: advances and prospects. Appl Microbiol Biotechnol, 2020, 104(18): 7745-7766.
|
| [94] |
Volke DC, Calero P, Nikel PI. Pseudomonas putida. Trends Microbiol, 2020, 28(6): 512-513. DOI:10.1016/j.tim.2020.02.015
|
| [95] |
Nikel PI, De Lorenzo V. Pseudomonas putida as a functional chassis for industrial biocatalysis: From native biochemistry to trans-metabolism. Metab Eng, 2018, 50: 142-155. DOI:10.1016/j.ymben.2018.05.005
|
| [96] |
Ng IS, Keskin BB, Tan SI. A critical review of genome editing and synthetic biology applications in metabolic engineering of microalgae and cyanobacteria. Biotechnol J, 2020, 15(8): 1900228. DOI:10.1002/biot.201900228
|
| [97] |
Santos-Merino M, Singh AK, Ducat DC. New applications of synthetic biology tools for cyanobacterial metabolic engineering. Front Bioeng Biotechnol, 2019, 7: 33. DOI:10.3389/fbioe.2019.00033
|
| [98] |
Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast. Science, 2015, 349(6252): 1095-1100. DOI:10.1126/science.aac9373
|
| [99] |
Luo X, Reiter MA, D'espaux L, et al. Complete biosynthesis of cannabinoids and their unnatural analogues in yeast. Nature, 2019, 567(7746): 123-126. DOI:10.1038/s41586-019-0978-9
|
| [100] |
Denby CM, Li RA, Vu VT, et al. Industrial brewing yeast engineered for the production of primary flavor determinants in hopped beer. Nat Commun, 2018, 9: 965. DOI:10.1038/s41467-018-03293-x
|
| [101] |
Wu T, Ye LJ, Zhao DD, et al. Membrane engineering — A novel strategy to enhance the production and accumulation of β-carotene in Escherichia coli. Metab Eng, 2017, 43: 85-91. DOI:10.1016/j.ymben.2017.07.001
|
| [102] |
Nakamura CE, Whited GM. Metabolic engineering for the microbial production of 1, 3-propanediol. Curr Opin Biotechnol, 2003, 14(5): 454-459. DOI:10.1016/j.copbio.2003.08.005
|
| [103] |
Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1, 4-butanediol. Nat Chem Biol, 2011, 7(7): 445-452. DOI:10.1038/nchembio.580
|
| [104] |
Guo L, Diao WW, Gao C, et al. Engineering Escherichia coli lifespan for enhancing chemical production. Nat Catal, 2020, 3(3): 307-318. DOI:10.1038/s41929-019-0411-7
|
| [105] |
Yu T, Zhou YJ, Huang MT, et al. Reprogramming yeast metabolism from alcoholic fermentation to lipogenesis. Cell, 2018, 174(6): 1549-1558. DOI:10.1016/j.cell.2018.07.013
|
| [106] |
Gleizer S, Ben-Nissan R, Bar-On YM, et al. Conversion of Escherichia coli to generate all biomass carbon from CO2. Cell, 2019, 179(6): 1255-1263. DOI:10.1016/j.cell.2019.11.009
|
| [107] |
Chen FY, Jung HW, Tsuei CY, et al. Converting Escherichia coli to a synthetic methylotroph growing solely on methanol. Cell, 2020, 182(4): 933-946e. DOI:10.1016/j.cell.2020.07.010
|
| [108] |
蔺玉萍, 王钦宏. 工业微生物遗传和环境扰动的调控和适应进化. 生物工程学报, 2019, 35(10): 1925-1941. Lin YP, Wang QH. Regulation and adaptive evolution of industrial microorganisms towards genetic and environmental disturbances. Chin J Biotech, 2019, 35(10): 1925-1941 (in Chinese). |
| [109] |
Peña DA, Gasser B, Zanghellini J, et al. Metabolic engineering of Pichia pastoris. Metab Eng, 2018, 50: 2-15. DOI:10.1016/j.ymben.2018.04.017
|
| [110] |
Obst U, Lu TK, Sieber V. A modular toolkit for generating Pichia pastoris secretion libraries. ACS Synth Biol, 2017, 6(6): 1016-1025. DOI:10.1021/acssynbio.6b00337
|
| [111] |
Tian Y, Li YL, Zhao FC, et al. Engineered Pichia pastoris production of fusaruside, a selective immunomodulator. BMC Biotechnol, 2019, 19: 37. DOI:10.1186/s12896-019-0532-8/fulltext.html
|
| [112] |
Zhang XY, Wang DG, Duan YH, et al. Production of lycopene by metabolically engineered Pichia pastoris. Biosci Biotechnol Biochem, 2020, 84(3): 463-470. DOI:10.1080/09168451.2019.1693250
|
| [113] |
Kong SJ, Pan H, Liu XY, et al. De novo biosynthesis of 2-phenylethanol in engineered Pichia pastoris. Enzyme Microb Technol, 2020, 133: 109459. DOI:10.1016/j.enzmictec.2019.109459
|
| [114] |
Zhao YW, Li L, Zheng GS, et al. CRISPR/dCas9-mediated multiplex gene repression in Streptomyces. Biotechnol J, 2018, 13(9): 1800121. DOI:10.1002/biot.201800121
|
| [115] |
Tao WX, Yang A, Deng ZX, et al. CRISPR/Cas9-based editing of Streptomyces for discovery, characterization, and production of natural products. Front Microbiol, 2018, 9: 1660. DOI:10.3389/fmicb.2018.01660
|
| [116] |
Li L, Wei KK, Zheng GS, et al. CRISPR-Cpf1-assisted multiplex genome editing and transcriptional repression in Streptomyces. Appl Environ Microbiol, 2018, 84(18): e00827-18.
|
| [117] |
Tong YJ, Whitford CM, Robertsen HL, et al. Highly efficient DSB-free base editing for streptomycetes with CRISPR-BEST. Proc Natl Acad Sci USA, 2019, 116(41): 20366-20375. DOI:10.1073/pnas.1913493116
|
| [118] |
卜庆廷, 毛旭明, 陈新爱, 等. 链霉菌较小基因组研究进展及展望. 生命科学, 2019, 31(4): 391-400. Bu QT, Mao XM, Chen XA, et al. Progress and prospect of genome-minimized Streptomyces hosts. Chin Bull Life Sci, 2019, 31(4): 391-400 (in Chinese). |
| [119] |
Hoff J, Daniel B, Stukenberg D, et al. Vibrio natriegens: an ultrafast-growing marine bacterium as emerging synthetic biology chassis. Environ Microbiol, 2020, 22(10): 4394-4408. DOI:10.1111/1462-2920.15128
|
| [120] |
Hoffart E, Grenz S, Lange J, et al. High substrate uptake rates empower Vibrio natriegens as production host for industrial biotechnology. Appl Environ Microbiol, 2017, 83(22): e01614-17.
|
| [121] |
Maida I, Bosi E, Perrin E, et al. Draft genome sequence of the fast-growing bacterium Vibrio natriegens strain DSMZ 759. Genome Announc, 2013, 1(4): e00648-13.
|
| [122] |
Wang Z, Lin BC, Hervey IWJ, et al. Draft genome sequence of the fast-growing marine bacterium Vibrio natriegens strain ATCC 14048. Genome Announc, 2013, 1(4): e00589-13.
|
| [123] |
Clark J, Awah A, Moreland R, et al. Complete genome sequence of Vibrio natriegens phage phriendly. Microbiol Resour Announc, 2019, 8(40): e01096-19.
|
| [124] |
Lee HH, Ostrov N, Wong BG, et al. Functional genomics of the rapidly replicating bacterium Vibrio natriegens by CRISPRi. Nat Microbiol, 2019, 4(7): 1105-1113. DOI:10.1038/s41564-019-0423-8
|
| [125] |
Weinstock MT, Hesek ED, Wilson CM, et al. Vibrio natriegens as a fast-growing host for molecular biology. Nat Methods, 2016, 13(10): 849-851. DOI:10.1038/nmeth.3970
|
| [126] |
Tschirhart T, Shukla V, Kelly EE, et al. Synthetic biology tools for the fast-growing marine bacterium Vibrio natriegens. ACS Synth Biol, 2019, 8(9): 2069-2079. DOI:10.1021/acssynbio.9b00176
|
| [127] |
Dalia TN, Yoon SH, Galli E, et al. Enhancing multiplex genome editing by natural transformation (MuGENT) via inactivation of ssDNA exonucleases. Nucleic Acids Res, 2017, 45(12): 7527-7537. DOI:10.1093/nar/gkx496
|
| [128] |
Dalia TN, Hayes CA, Stolyar S, et al. Multiplex genome editing by natural transformation (MuGENT) for synthetic biology in Vibrio natriegens. ACS Synth Biol, 2017, 6(9): 1650-1655. DOI:10.1021/acssynbio.7b00116
|
| [129] |
Wiegand DJ, Lee HH, Ostrov N, et al. Cell-free protein expression using the rapidly growing bacterium Vibrio natriegens. J Vis Exp, 2019(145). DOI:10.3791/59495
|
| [130] |
Failmezger J, Scholz S, Blombach B, et al. Cell-free protein synthesis from fast-growing Vibrio natriegens. Front Microbiol, 2018, 9: 1146. DOI:10.3389/fmicb.2018.01146
|
| [131] |
Wu FL, Chen WJ, Peng YF, et al. Design and reconstruction of regulatory parts for fast-frowing Vibrio natriegens synthetic biology. ACS Synth Biol, 2020, 9(9): 2399-2409. DOI:10.1021/acssynbio.0c00158
|
| [132] |
Pfeifer E, Michniewski S, Gätgens C, et al. Generation of a prophage-free variant of the fast-growing bacterium Vibrio natriegens. Appl Environ Microbiol, 2019, 85(17): e00853-19.
|
| [133] |
Weinstock MT, Wilson CM, Hesek ED. Genetically engineered Vibrio sp. and uses thereof: US, 20180057824[P]. 2018-03-01.
|
| [134] |
Kim JY, Oh JJ, Jeon MS, et al. Improvement of Euglena gracilis paramylon production through a cocultivation strategy with the indole-3-acetic acid-producing bacterium Vibrio natriegens. Appl Environ Microbiol, 2019, 85(19): e01548-19.
|
| [135] |
Fernández-Llamosas H, Castro L, Blázquez ML, et al. Speeding up bioproduction of selenium nanoparticles by using Vibrio natriegens as microbial factory. Sci Rep, 2017, 7: 16046. DOI:10.1038/s41598-017-16252-1
|
| [136] |
Wang Z, Tschirhart T, Schultzhaus Z, et al. Melanin produced by the fast-growing marine bacterium Vibrio natriegens through heterologous biosynthesis: characterization and application. Appl Environ Microbiol, 2020, 86(5): e02749-19.
|
| [137] |
Ellis GA, Tschirhart T, Spangler J, et al. Exploiting the feedstock flexibility of the emergent synthetic biology chassis Vibrio natriegens for engineered natural product production. Mar Drugs, 2019, 17(12): 679. DOI:10.3390/md17120679
|
| [138] |
Becker W, Wimberger F, Zangger K. Vibrio natriegens: An alternative expression system for the high-yield production of isotopically labeled proteins. Biochemistry, 2019, 58(25): 2799-2803. DOI:10.1021/acs.biochem.9b00403
|
| [139] |
Biggs BW, Bedore SR, Arvay E, et al. Development of a genetic toolset for the highly engineerable and metabolically versatile Acinetobacter baylyi ADP1. Nucleic Acids Res, 2020, 48(9): 5169-5182. DOI:10.1093/nar/gkaa167
|
| [140] |
Suárez GA, Renda BA, Dasgupta A, et al. Reduced mutation rate and increased transformability of transposon-free Acinetobacter baylyi ADP1-ISx. Appl Environ Microbiol, 2017, 83(17): e01025-17.
|
| [141] |
Salcedo-Vite K, Sigala JC, Segura D, et al. Acinetobacter baylyi ADP1 growth performance and lipid accumulation on different carbon sources. Appl Microbiol Biotechnol, 2019, 103(15): 6217-6229. DOI:10.1007/s00253-019-09910-z
|
| [142] |
Barbe V, Vallenet D, Fonknechten N, et al. Unique features revealed by the genome sequence of Acinetobacter sp. ADP1, a versatile and naturally transformation competent bacterium. Nucleic Acids Res, 2004, 32(19): 5766-5779. DOI:10.1093/nar/gkh910
|
| [143] |
Suárez GA, Dugan KR, Renda BA, et al. Rapid and assured genetic engineering methods applied to Acinetobacter baylyi ADP1 genome streamlining. Nucleic Acids Res, 2020, 48(8): 4585-4600. DOI:10.1093/nar/gkaa204
|
| [144] |
Lehtinen T, Efimova E, Santala S, et al. Improved fatty aldehyde and wax ester production by overexpression of fatty acyl-CoA reductases. Microb Cell Fact, 2018, 17: 19. DOI:10.1186/s12934-018-0869-z
|
| [145] |
Santala S, Efimova E, Kivinen V, et al. Improved triacylglycerol production in Acinetobacter baylyi ADP1 by metabolic engineering. Microb Cell Fact, 2011, 10: 36. DOI:10.1186/1475-2859-10-36
|
| [146] |
Elbahloul Y, Steinbuchel A. Engineering the genotype of Acinetobacter sp. strain ADP1 to enhance biosynthesis of cyanophycin. Appl Environ Microbiol, 2006, 72(2): 1410-1419. DOI:10.1128/AEM.72.2.1410-1419.2006
|
| [147] |
Xia J, Yang YF, Liu CG, et al. Engineering Zymomonas mobilis for robust cellulosic ethanol production. Trends Biotechnol, 2019, 37(9): 960-972. DOI:10.1016/j.tibtech.2019.02.002
|
| [148] |
Wang X, He QN, Yang YF, et al. Advances and prospects in metabolic engineering of Zymomonas mobilis. Metab Eng, 2018, 50: 57-73. DOI:10.1016/j.ymben.2018.04.001
|
| [149] |
Yang SH, Vera JM, Grass J, et al. Complete genome sequence and the expression pattern of plasmids of the model ethanologen Zymomonas mobilis ZM4 and its xylose-utilizing derivatives 8b and 2032. Biotechnol Biofuels, 2018, 11: 125. DOI:10.1186/s13068-018-1116-x
|
| [150] |
Yang SH, Pappas KM, Hauser LJ, et al. Improved genome annotation for Zymomonas mobilis. Nat Biotechnol, 2009, 27(10): 893-894. DOI:10.1038/nbt1009-893
|
| [151] |
Seo JS, Chong H, Park HS, et al. The genome sequence of the ethanologenic bacterium Zymomonas mobilis ZM4. Nat Biotechnol, 2005, 23(1): 63-68. DOI:10.1038/nbt1045
|
| [152] |
Shen W, Zhang J, Geng BN, et al. Establishment and application of a CRISPR-Cas12a assisted genome-editing system in Zymomonas mobilis. Microb Cell Fact, 2019, 18: 162. DOI:10.1186/s12934-019-1219-5
|
| [153] |
Cao QH, Shao HH, Qiu H, et al. Using the CRISPR/Cas9 system to eliminate native plasmids of Zymomonas mobilis ZM4. Biosci Biotechnol Biochem, 2017, 81(3): 453-459. DOI:10.1080/09168451.2016.1189312
|
| [154] |
Banta AB, Enright AL, Siletti C, et al. A high-efficacy CRISPR interference system for gene function discovery in Zymomonas mobilis. Appl Environ Microbiol, 2020, 86(23): AEM.01621-20.
|
| [155] |
Stoneman HR, Wrobel RL, Place M, et al. CRISpy-Pop: A web tool for designing CRISPR/Cas9-driven genetic modifications in diverse populations. G3 (Bethesda), 2020, 10(11): 4287-4294. DOI:10.1534/g3.120.401498
|
| [156] |
Vera JM, Ghosh IN, Zhang Y, et al. Genome-scale transcription-translation mapping reveals features of Zymomonas mobilis transcription units and promoters. mSystems, 2020, 5(4): e00250-20.
|
| [157] |
Ong WK, Courtney DK, Pan S, et al. Model-driven analysis of mutant fitness experiments improves genome-scale metabolic models of Zymomonas mobilis ZM4. PLoS Comput Biol, 2020, 16(8): e1008137. DOI:10.1371/journal.pcbi.1008137
|
| [158] |
Yang YF, Shen W, Huang J, et al. Prediction and characterization of promoters and ribosomal binding sites of Zymomonas mobilis in system biology era. Biotechnol Biofuels, 2019, 12: 52. DOI:10.1186/s13068-019-1399-6
|
| [159] |
Yang YF, Rong ZY, Song HY, et al. Identification and characterization of ethanol-inducible promoters of Zymomonas mobilis based on omics data and dual reporter-gene system. Biotechnol Appl Biochem, 2020, 67(1): 158-165. DOI:10.1002/bab.1838
|
| [160] |
Han RH, Haning K, Gonzalez-Rivera JC, et al. Multiple small RNAs interact to co-regulate ethanol tolerance in Zymomonas mobilis. Front Bioeng Biotechnol, 2020, 8: 155. DOI:10.3389/fbioe.2020.00155
|
| [161] |
Nissen L, Rollini M, Picozzi C, et al. Yeast-free doughs by Zymomonas mobilis: Evaluation of technological and fermentation performances by using a metabolomic approach. Microorganisms, 2020, 8(6).
|
| [162] |
De Aguiar Silva AT, Cavalcanti IDL, De Lima Fernandes MA, et al. Effect of zymomonas mobilis probiotic on cholesterol and its lipoprotein fractions and the intestinal regulation. Clin Nutr, 2020, 39(12): 3750-3755. DOI:10.1016/j.clnu.2020.04.002
|
| [163] |
Yoo M, Bestel-Corre G, Croux C, et al. A quantitative system-scale characterization of the metabolism of Clostridium acetobutylicum. mBio, 2015, 6(6): e01808-15.
|
| [164] |
Yoo M, Nguyen NP, Soucaille P. Trends in systems biology for the analysis and engineering of Clostridium acetobutylicum metabolism. Trends Microbiol, 2020, 28(2): 118-140. DOI:10.1016/j.tim.2019.09.003
|
| [165] |
Noh HJ, Woo JE, Lee SY, et al. Metabolic engineering of Clostridium acetobutylicum for the production of butyl butyrate. Appl Microbiol Biotechnol, 2018, 102(19): 8319-8327. DOI:10.1007/s00253-018-9267-z
|
| [166] |
Xue C, Zhao JB, Chen LJ, et al. Recent advances and state-of-the-art strategies in strain and process engineering for biobutanol production by Clostridium acetobutylicum. Biotechnol Adv, 2017, 35(2): 310-322. DOI:10.1016/j.biotechadv.2017.01.00
|
| [167] |
Joseph RC, Kim NM, Sandoval NR. Recent developments of the synthetic biology toolkit for Clostridium. Front Microbiol, 2018, 9: 154. DOI:10.3389/fmicb.2018.00154
|
| [168] |
Nikel PI, Martínez-García E, De Lorenzo V. Biotechnological domestication of pseudomonads using synthetic biology. Nat Rev Microbiol, 2014, 12(5): 368-379. DOI:10.1038/nrmicro3253
|
| [169] |
Wang SW, Cui JJ, Bilal M, et al. Pseudomonas spp. as cell factories (MCFs) for value-added products: from rational design to industrial applications. Crit Rev Biotechnol, 2020, 40(8): 1232-1249. DOI:10.1080/07388551.2020.1809990
|
| [170] |
Liu ZH, Wang K, Chen Y, et al. Third-generation biorefineries as the means to produce fuels and chemicals from CO2. Nat Catal, 2020, 3(3): 274-288. DOI:10.1038/s41929-019-0421-5
|
| [171] |
Zhang L, Zhao R, Jia D, et al. Engineering Clostridium ljungdahlii as the gas-fermenting cell factory for the production of biofuels and biochemicals. Curr Opin Chem Biol, 2020, 59: 54-61. DOI:10.1016/j.cbpa.2020.04.010
|
| [172] |
Norman ROJ, Millat T, Winzer K, et al. Progress towards platform chemical production using Clostridium autoethanogenum. Biochem Soc Trans, 2018, 46(3): 523-535. DOI:10.1042/BST20170259
|
| [173] |
Köpke M, Held C, Hujer S, et al. Clostridium ljungdahlii represents a microbial production platform based on syngas. Proc Natl Acad Sci USA, 2010, 107(29): 13087-13092. DOI:10.1073/pnas.1004716107
|
| [174] |
Brown SD, Nagaraju S, Utturkar S, et al. Comparison of single-molecule sequencing and hybrid approaches for finishing the genome of Clostridium autoethanogenum and analysis of CRISPR systems in industrial relevant Clostridia. Biotechnol Biofuels, 2014, 7: 40. DOI:10.1186/1754-6834-7-40
|
| [175] |
Tremblay PL, Zhang T, Dar SA, et al. The Rnf complex of Clostridium ljungdahlii is a proton-translocating ferredoxin: NAD+ oxidoreductase essential for autotrophic growth. mBio, 2012, 4(1): e00406-12.
|
| [176] |
Liu ZY, Jia DC, Zhang KD, et al. Ethanol metabolism dynamics in Clostridium ljungdahlii grown on carbon monoxide. Appl Environ Microbiol, 2020, 86(14): e00730-20.
|
| [177] |
Zhu HF, Liu ZY, Zhou X, et al. Energy conservation and carbon flux distribution during fermentation of CO or H2/CO2 by Clostridium ljungdahlii. Front Microbiol, 2020, 11: 416. DOI:10.3389/fmicb.2020.00416
|
| [178] |
Huang H, Chai CS, Li N, et al. CRISPR/Cas9-based efficient genome editing in Clostridium ljungdahlii, an autotrophic gas-fermenting bacterium. ACS Synth Biol, 2016, 5(12): 1355-1361. DOI:10.1021/acssynbio.6b00044
|
| [179] |
Zhao R, Liu YQ, Zhang H, et al. CRISPR-Cas12a-mediated gene deletion and regulation in Clostridium ljungdahlii and its application in carbon flux redirection in synthesis gas fermentation. ACS Synth Biol, 2019, 8(10): 2270-2279. DOI:10.1021/acssynbio.9b00033
|
| [180] |
Huang H, Chai CS, Yang S, et al. Phage serine integrase-mediated genome engineering for efficient expression of chemical biosynthetic pathway in gas-fermenting Clostridium ljungdahlii. Metab Eng, 2019, 52: 293-302. DOI:10.1016/j.ymben.2019.01.005
|
| [181] |
Xia PF, Casini I, Schulz S, et al. Reprogramming acetogenic bacteria with CRISPR-targeted base editing via deamination. ACS Synth Biol, 2020, 9(8): 2162-2171. DOI:10.1021/acssynbio.0c00226
|
| [182] |
Seys FM, Rowe P, Bolt EL, et al. A gold standard, CRISPR/Cas9-based complementation strategy reliant on 24 nucleotide bookmark sequences. Genes (Basel), 2020, 11(4): 458. DOI:10.3390/genes11040458
|
| [183] |
Krüger A, Mueller AP, Rybnicky GA, et al. Development of a clostridia-based cell-free system for prototyping genetic parts and metabolic pathways. Metab Eng, 2020, 62: 95-105. DOI:10.1016/j.ymben.2020.06.004
|
| [184] |
Heffernan JK, Valgepea K, De Souza Pinto Lemgruber R, et al. Enhancing CO2-valorization using Clostridium autoethanogenum for sustainable fuel and chemicals production. Front Bioeng Biotechnol, 2020, 8: 204. DOI:10.3389/fbioe.2020.00204
|
| [185] |
Mahamkali V, Valgepea K, De Souza Pinto Lemgruber R, et al. Redox controls metabolic robustness in the gas-fermenting acetogen Clostridium autoethanogenum. Proc Natl Acad Sci USA, 2020, 117(23): 13168-13175. DOI:10.1073/pnas.1919531117
|
| [186] |
Valgepea K, De Souza Pinto Lemgruber R, Meaghan K, et al. Maintenance of ATP homeostasis triggers metabolic shifts in gas-fermenting acetogens. Cell Syst, 2017, 4(5): 505-515. DOI:10.1016/j.cels.2017.04.008
|
| [187] |
Cantera S, Bordel S, Lebrero R, et al. Bio-conversion of methane into high profit margin compounds: an innovative, environmentally friendly and cost-effective platform for methane abatement. World J Microbiol Biotechnol, 2019, 35: 16. DOI:10.1007/s11274-018-2587-4
|
| [188] |
Gassler T, Sauer M, Gasser B, et al. The industrial yeast Pichia pastoris is converted from a heterotroph into an autotroph capable of growth on CO2. Nat Biotechnol, 2020, 38(2): 210-216. DOI:10.1038/s41587-019-0363-0
|
| [189] |
史硕博, 孟琼宇, 乔玮博, 等. 塑造低碳经济的第三代固碳生物炼制. 合成生物学, 2020, 1(1): 44-59. Shi SB, Meng QY, Qiao WB, et al. Establishing carbon dioxide-based third-generation biorefinery for a sustainable low-carbon economy. Syn Bio J, 2020, 1(1): 44-59 (in Chinese). |
| [190] |
Zhang Y, Kumar A, Hardwidge PR, et al. D-lactic acid production from renewable lignocellulosic biomass via genetically modified Lactobacillus plantarum. Biotechnol Prog, 2016, 32(2): 271-278. DOI:10.1002/btpr.2212
|
| [191] |
Fang H, Li D, Kang J, et al. Metabolic engineering of Escherichia coli for de novo biosynthesis of vitamin B12. Nat Commun, 2018, 9: 4917. DOI:10.1038/s41467-018-07412-6
|
| [192] |
Ali Abdel-Rahman M, Tashiro Y, Sonomoto K. Lactic acid production from lignocellulose-derived sugars using lactic acid bacteria: overview and limits. J Biotechnol, 2011, 156(4): 286-301. DOI:10.1016/j.jbiotec.2011.06.017
|
| [193] |
Ali Abdel-Rahman M, Tashiro Y, Sonomoto K. Recent advances in lactic acid production by microbial fermentation processes. Biotechnol Adv, 2013, 31(6): 877-902. DOI:10.1016/j.biotechadv.2013.04.002
|
| [194] |
刘洋儿, 郭明璋, 杜若曦, 等. 乳酸菌在合成生物学中的研究现状及展望. 生物技术通报, 2019, 35(8): 193-204. Liu YE, Guo MZ, Du RX, et al. Advances and prospects of synthetic biology in lactic acid bacteria. Biotechnol Bull, 2019, 35(8): 193-204 (in Chinese). |
| [195] |
Huang H, Song X, Yang S. Development of a RecE/T-assisted CRISPR-Cas9 toolbox for Lactobacillus. Biotechnol J, 2019, 14(7): e1800690. DOI:10.1002/biot.201800690
|
| [196] |
Sun Z, Yu J, Dan T, et al. Phylogenesis and evolution of lactic acid bacteria. .
|
| [197] |
Rizzello CG, De Angelis M. Lactobacillus spp. : Lactobacillus delbrueckii Group[M]//Fuquay J W. Encyclopedia of Dairy Sciences. 2nd ed. San Diego, Academic Press. 2011.
|
| [198] |
Hidalgo-Cantabrana C, O'flaherty S, Barrangou R. CRISPR-based engineering of next-generation lactic acid bacteria. Curr Opin Microbiol, 2017, 37: 79-87. DOI:10.1016/j.mib.2017.05.015
|
| [199] |
Sun ZH, Harris HM, Mccann A, et al. Expanding the biotechnology potential of lactobacilli through comparative genomics of 213 strains and associated genera. Nat Commun, 2015, 6: 8322. DOI:10.1038/ncomms9322
|
| [200] |
Hidalgo-Cantabrana C, Goh YJ, Pan MC, et al. Genome editing using the endogenous type Ⅰ CRISPR-Cas system in Lactobacillus crispatus. Proc Natl Acad Sci USA, 2019, 116(32): 15774-15783. DOI:10.1073/pnas.1905421116
|
| [201] |
Song AaL, In LLA, Lim SHE, et al. A review on Lactococcus lactis: from food to factory. Microb Cell Fact, 2017, 16: 55. DOI:10.1186/s12934-017-0669-x
|
| [202] |
樊斌, 陈欢, 宋婉莹, 等. 乳酸菌基因改造技术研究进展. 中国生物工程杂志, 2020, 40(6): 84-92. Fan B, Chen H, Song WY, et al. Advances in lactic acid bacteria gene modification. China Biotechnol, 2020, 40(6): 84-92 (in Chinese). |
| [203] |
Van Tilburg AY, Cao HJ, van der Meulen SB, et al. Metabolic engineering and synthetic biology employing Lactococcus lactis and Bacillus subtilis cell factories. Curr Opin Biotechnol, 2019, 59: 1-7.
|
| [204] |
Xiong ZQ, Wei YY, Kong LH, et al. Short communication: An inducible CRISPR/dCas9 gene repression system in Lactococcus lactis. J Dairy Sci, 2020, 103(1): 161-165. DOI:10.3168/jds.2019-17346
|
| [205] |
Mar MJ, Andersen JM, Kandasamy V, et al. Synergy at work: linking the metabolism of two lactic acid bacteria to achieve superior production of 2-butanol. Biotechnol Biofuels, 2020, 13: 45. DOI:10.1186/s13068-020-01689-w
|
| [206] |
Guo TT, Kong J, Zhang L, et al. Fine tuning of the lactate and diacetyl production through promoter engineering in Lactococcus lactis. PLoS One, 2012, 7(4): e36296. DOI:10.1371/journal.pone.0036296
|
| [207] |
Ye W, Huo GC, Chen JL, et al. Heterologous expression of the Bacillus subtilis (natto) alanine dehydrogenase in Escherichia coli and Lactococcus lactis. Microbiol Res, 2010, 165(4): 268-275. DOI:10.1016/j.micres.2009.05.008
|
| [208] |
Wisselink HW, Moers APHA, Mars AE, et al. Overproduction of heterologous mannitol 1-phosphatase: a key factor for engineering mannitol production by Lactococcus lactis. Appl Environ Microbiol, 2005, 71(3): 1507-1514. DOI:10.1128/AEM.71.3.1507-1514.2005
|
| [209] |
Liu JM, Wang ZH, Kandasamy V, et al. Harnessing the respiration machinery for high-yield production of chemicals in metabolically engineered Lactococcus lactis. Metab Eng, 2017, 44: 22-29. DOI:10.1016/j.ymben.2017.09.001
|
| [210] |
Krishna BS, Nikhilesh GSS, Tarun B, et al. Industrial production of lactic acid and its applications. Int J Biotechnol Res, 2018, 1(1): 42-54.
|
| [211] |
Mazzoli R. Metabolic engineering strategies for consolidated production of lactic acid from lignocellulosic biomass. Biotechnol Appl Biochem, 2020, 67(1): 61-72. DOI:10.1002/bab.1869
|
| [212] |
Tsuge Y, Kato N, Yamamoto S, et al. Metabolic engineering of Corynebacterium glutamicum for hyperproduction of polymer-grade L- and D-lactic acid. Appl Microbiol Biotechnol, 2019, 103(8): 3381-3391. DOI:10.1007/s00253-019-09737-8
|
| [213] |
Wang C, Cui YH, Qu XJ. Mechanisms and improvement of acid resistance in lactic acid bacteria. Arch Microbiol, 2018, 200(2): 195-201. DOI:10.1007/s00203-017-1446-2
|
| [214] |
Zhang YX, Vadlani PV, Kumar A, et al. Enhanced D-lactic acid production from renewable resources using engineered Lactobacillus plantarum. Appl Microbiol Biotechnol, 2016, 100(1): 279-288. DOI:10.1007/s00253-015-7016-0
|
| [215] |
Zheng Y, Chen JC, Ma YM, et al. Engineering biosynthesis of polyhydroxyalkanoates (PHA) for diversity and cost reduction. Metab Eng, 2020, 58: 82-93. DOI:10.1016/j.ymben.2019.07.004
|
| [216] |
Choi SY, Cho IJ, Lee Y, et al. Microbial polyhydroxyalkanoates and nonnatural polyesters. Adv Mater, 2020, 32(35): 1907138. DOI:10.1002/adma.201907138
|
| [217] |
Prieto A, Escapa IF, Martínez V, et al. A holistic view of polyhydroxyalkanoate metabolism in Pseudomonas putida. Environ Microbiol, 2016, 18(2): 341-357. DOI:10.1111/1462-2920.12760
|
| [218] |
Kumar P, Maharjan A, Jun HB, et al. Bioconversion of lignin and its derivatives into polyhydroxyalkanoates: Challenges and opportunities. Biotechnol Appl Biochem, 2019, 66(2): 153-162. DOI:10.1002/bab.1720
|
| [219] |
Little GT, Ehsaan M, Arenas-López C, et al. Complete genome sequence of Cupriavidus necator H16 (DSM 428). Microbiol Resour Announc, 2019, 8(37): e00814-19.
|
| [220] |
Moriuchi R, Dohra H, Kanesaki Y, et al. Complete genome sequence of 3-chlorobenzoate-degrading bacterium Cupriavidus necator NH9 and reclassification of the strains of the genera Cupriavidus and Ralstonia based on phylogenetic and whole-genome sequence analyses. Front Microbiol, 2019, 10: 133. DOI:10.3389/fmicb.2019.00133
|
| [221] |
Pohlmann A, Fricke WF, Reinecke F, et al. Genome sequence of the bioplastic-producing "Knallgas" bacterium Ralstonia eutropha H16. Nat Biotechnol, 2006, 24(10): 1257-1262. DOI:10.1038/nbt1244
|
| [222] |
Bowien B, Kusian B. Genetics and control of CO2 assimilation in the chemoautotroph Ralstonia eutropha. Arch Microbiol, 2002, 178(2): 85-93. DOI:10.1007/s00203-002-0441-3
|
| [223] |
Brigham C. Perspectives for the biotechnological production of biofuels from CO2 and H2 using Ralstonia eutropha and other 'Knallgas' bacteria. Appl Microbiol Biotechnol, 2019, 103(5): 2113-2120. DOI:10.1007/s00253-019-09636-y
|
| [224] |
Yu J. Fixation of carbon dioxide by a hydrogen-oxidizing bacterium for value-added products. World J Microbiol Biotechnol, 2018, 34(7): 89. DOI:10.1007/s11274-018-2473-0
|
| [225] |
Peplinski K, Ehrenreich A, Döring C, et al. Genome-wide transcriptome analyses of the 'Knallgas' bacterium Ralstonia eutropha H16 with regard to polyhydroxyalkanoate metabolism. Microbiology, 2010, 156(Pt 7): 2136-2152.
|
| [226] |
Raberg M, Peplinski K, Heiss S, et al. Proteomic and transcriptomic elucidation of the mutant Ralstonia eutropha G+1 with regard to glucose utilization. Appl Environ Microbiol, 2011, 77(6): 2058-2070. DOI:10.1128/AEM.02015-10
|
| [227] |
Park JM, Kim TY, Lee SY. Genome-scale reconstruction and in silico analysis of the Ralstonia eutropha H16 for polyhydroxyalkanoate synthesis, lithoautotrophic growth, and 2-methyl citric acid production. BMC Syst Biol, 2011, 5: 101. DOI:10.1186/1752-0509-5-101
|
| [228] |
Park JM, Jang YS, Kim TY, et al. Development of a gene knockout system for Ralstonia eutropha H16 based on the broad-host-range vector expressing a mobile group Ⅱ intron. FEMS Microbiol Lett, 2010, 309(2): 193-200.
|
| [229] |
Bi CH, Su P, Muller J, et al. Development of a broad-host synthetic biology toolbox for Ralstonia eutropha and its application to engineering hydrocarbon biofuel production. Microb Cell Fact, 2013, 12: 107. DOI:10.1186/1475-2859-12-107
|
| [230] |
Hu M, Xiong B, Li Z, et al. A novel gene expression system for Ralstonia eutropha based on the T7 promoter. BMC Microbiol, 2020, 20(1): 121. DOI:10.1186/s12866-020-01812-9
|
| [231] |
Gruber S, Hagen J, Schwab H, et al. Versatile and stable vectors for efficient gene expression in Ralstonia eutropha H16. J Biotechnol, 2014, 186: 74-82. DOI:10.1016/j.jbiotec.2014.06.030
|
| [232] |
Gruber S, Schwendenwein D, Magomedova Z, et al. Design of inducible expression vectors for improved protein production in Ralstonia eutropha H16 derived host strains. J Biotechnol, 2016, 235: 92-99. DOI:10.1016/j.jbiotec.2016.04.026
|
| [233] |
Alagesan S, Hanko EKR, Malys N, et al. Functional genetic elements for controlling gene expression in Cupriavidus necator H16. Appl Environ Microbiol, 2018, 84(19): e00878-18.
|
| [234] |
Johnson AO, Gonzalez-Villanueva M, Tee KL, et al. An engineered constitutive promoter set with broad activity range for Cupriavidus necator H16. ACS Synth Biol, 2018, 7(8): 1918-1928. DOI:10.1021/acssynbio.8b00136
|
| [235] |
Mifune J, Nakamura S, Fukui T. Targeted engineering of Cupriavidus necator chromosome for biosynthesis of poly(3-hydroxybutyrate-co-3- hydroxyhexanoate) from vegetable oil. Can J Chem, 2008, 86(6): 621-627. DOI:10.1139/v08-047
|
| [236] |
Xiong B, Li ZK, Liu L, et al. Genome editing of Ralstonia eutropha using an electroporation-based CRISPR-Cas9 technique. Biotechnol Biofuels, 2018, 11: 172. DOI:10.1186/s13068-018-1170-4
|
| [237] |
Claassens NJ, Bordanaba-Florit G, Cotton CaR, et al. Replacing the Calvin cycle with the reductive glycine pathway in Cupriavidus necator. Metab Eng, 2020, 62: 30-41. DOI:10.1016/j.ymben.2020.08.004
|
| [238] |
Fan C, Davison PA, Habgood R, et al. Chromosome-free bacterial cells are safe and programmable platforms for synthetic biology. Proc Natl Acad Sci USA, 2020, 117(12): 6752-6761. DOI:10.1073/pnas.1918859117
|
| [239] |
Chakravarty J, Brigham CJ. Solvent production by engineered Ralstonia eutropha: channeling carbon to biofuel. Appl Microbiol Biotechnol, 2018, 102(12): 5021-5031. DOI:10.1007/s00253-018-9026-1
|
| [240] |
Li ZK, Xiong B, Liu L, et al. Development of an autotrophic fermentation technique for the production of fatty acids using an engineered Ralstonia eutropha cell factory. J Ind Microbiol Biotechnol, 2019, 46(6): 783-790. DOI:10.1007/s10295-019-02156-8
|
| [241] |
Garrigues L, Maignien L, Lombard E, et al. Isopropanol production from carbon dioxide in Cupriavidus necator in a pressurized bioreactor. New Biotechnol, 2020, 56: 16-20. DOI:10.1016/j.nbt.2019.11.005
|
| [242] |
Windhorst C, Gescher J. Efficient biochemical production of acetoin from carbon dioxide using Cupriavidus necator H16. Biotechnol Biofuels, 2019, 12: 163. DOI:10.1186/s13068-019-1512-x
|
| [243] |
Lee HW, Park JH, Lee HS, et al. Production of bio-based isoprene by the mevalonate pathway cassette in Ralstonia eutropha. J Microbiol Biotechnol, 2019, 29(10): 1656-1664. DOI:10.4014/jmb.1909.09002
|
| [244] |
Tan D, Xue YS, Aibaidula G, et al. Unsterile and continuous production of polyhydroxybutyrate by Halomonas TD01. Bioresour Technol, 2011, 102(17): 8130-8136. DOI:10.1016/j.biortech.2011.05.068
|
| [245] |
Yue HT, Ling C, Yang T, et al. A seawater-based open and continuous process for polyhydroxyalkanoates production by recombinant Halomonas campaniensis LS21 grown in mixed substrates. Biotechnol Biofuels, 2014, 7: 108. DOI:10.1186/1754-6834-7-108/fulltext.html
|
| [246] |
尹进, 陈国强. 蓝水生物技术——聚羟基脂肪酸酯的产业发展. 新材料产业, 2016(2): 16-20. DOI:10.3969/j.issn.1008-892X.2016.02.005 |
| [247] |
Yu LP, Wu FQ, Chen GQ. Next-generation industrial biotechnology-transforming the current industrial biotechnology into competitive processes. Biotechnol J, 2019, 14(9): 1800437. DOI:10.1002/biot.201800437
|
| [248] |
杜鹤童, 赵倚晴, 陈金春, 等. 基于嗜盐微生物合成生物学的下一代工业生物技术. 生命科学, 2019, 31(4): 385-390. Du HT, Zhao YQ, Chen JC, et al. Next generation industrial biotechnology based on synthetic biology of halophiles. Chin Bull Life Sci, 2019, 31(4): 385-390 (in Chinese). |
| [249] |
Yin J, Fu XZ, Wu Q, et al. Development of an enhanced chromosomal expression system based on porin synthesis operon for halophile Halomonas sp. Appl Microbiol Biotechnol, 2014, 98(21): 8987-8997. DOI:10.1007/s00253-014-5959-1
|
| [250] |
Zhao H, Zhang HM, Chen XB, et al. Novel T7-like expression systems used for Halomonas. Metab Eng, 2017, 39: 128-140. DOI:10.1016/j.ymben.2016.11.007
|
| [251] |
Tao W, Lv L, Chen GQ. Engineering Halomonas species TD01 for enhanced polyhydroxyalkanoates synthesis via CRISPRi. Microb Cell Fact, 2017, 16(1): 48. DOI:10.1186/s12934-017-0655-3
|
| [252] |
Qin Q, Ling C, Zhao YQ, et al. CRISPR/Cas9 editing genome of extremophile Halomonas spp. Metab Eng, 2018, 47: 219-229. DOI:10.1016/j.ymben.2018.03.018
|
| [253] |
Shen R, Yin J, Ye JW, et al. Promoter engineering for enhanced P(3HB-co-4HB) production by Halomonas bluephagenesis. ACS Synth Biol, 2018, 7(8): 1897-1906. DOI:10.1021/acssynbio.8b00102
|
| [254] |
Ouyang PF, Wang H, Hajnal I, et al. Increasing oxygen availability for improving poly(3-hydroxybutyrate) production by Halomonas. Metab Eng, 2018, 45: 20-31. DOI:10.1016/j.ymben.2017.11.006
|
| [255] |
Ling C, Qiao GQ, Shuai BW, et al. Engineering NADH/NAD+ ratio in Halomonas bluephagenesis for enhanced production of polyhydroxyalkanoates (PHA). Metab Eng, 2018, 49: 275-286. DOI:10.1016/j.ymben.2018.09.007
|
| [256] |
Yin J, Wang H, Fu XZ, et al. Effects of chromosomal gene copy number and locations on polyhydroxyalkanoate synthesis by Escherichia coli and Halomonas sp. Appl Microbiol Biotechnol, 2015, 99(13): 5523-5534. DOI:10.1007/s00253-015-6510-8
|
| [257] |
Chen Y, Chen XY, Du HT, et al. Chromosome engineering of the TCA cycle in Halomonas bluephagenesis for production of copolymers of 3-hydroxybutyrate and 3-hydroxyvalerate (PHBV). Metab Eng, 2019, 54: 69-82. DOI:10.1016/j.ymben.2019.03.006
|
| [258] |
Yin J, Chen JC, Wu Q, et al. Halophiles, coming stars for industrial biotechnology. Biotechnol Adv, 2015, 33(7): 1433-1442. DOI:10.1016/j.biotechadv.2014.10.008
|
| [259] |
Yu LP, Yan X, Zhang X, et al. Biosynthesis of functional polyhydroxyalkanoates by engineered Halomonas bluephagenesis. Metab Eng, 2020, 59: 119-130. DOI:10.1016/j.ymben.2020.02.005
|
| [260] |
Ma H, Zhao YQ, Huang WZ, et al. Rational flux-tuning of Halomonas bluephagenesis for co-production of bioplastic PHB and ectoine. Nat Commun, 2020, 11(1): 3313. DOI:10.1038/s41467-020-17223-3
|
| [261] |
Du HT, Zhao YQ, Wu FQ, et al. Engineering Halomonas bluephagenesis for L-threonine production. Metab Eng, 2020, 60: 119-127. DOI:10.1016/j.ymben.2020.04.004
|
| [262] |
Chen XB, Yu LP, Qiao GQ, et al. Reprogramming Halomonas for industrial production of chemicals. J Ind Microbiol Biotechnol, 2018, 45(7): 545-554. DOI:10.1007/s10295-018-2055-z
|
| [263] |
Rao Y, Cai DB, Wang H, et al. Construction and application of a dual promoter system for efficient protein production and metabolic pathway enhancement in Bacillus licheniformis. J Biotechnol, 2020, 312: 1-10. DOI:10.1016/j.jbiotec.2020.02.015
|
| [264] |
Cai DB, Zhu J, Zhu S, et al. Metabolic engineering of main transcription factors in carbon, nitrogen, and phosphorus metabolisms for enhanced production of bacitracin in Bacillus licheniformis. ACS Synth Biol, 2019, 8(4): 866-875. DOI:10.1021/acssynbio.9b00005
|
| [265] |
Macklin DN, Ahn-Horst TA, Choi H, et al. Simultaneous cross-evaluation of heterogeneous E. coli datasets via mechanistic simulation. Science, 2020, 369(6502): aav3751.
|
| [266] |
Lu HZ, Li FR, Sánchez BJ, et al. A consensus S. cerevisiae metabolic model Yeast8 and its ecosystem for comprehensively probing cellular metabolism. Nat Commun, 2019, 10: 3586. DOI:10.1038/s41467-019-11581-3
|
| [267] |
Wang Y, Wang HC, Wei L, et al. Synthetic promoter design in Escherichia coli based on a deep generative network. Nucleic Acids Res, 2020, 48(12): 6403-6412. DOI:10.1093/nar/gkaa325
|
| [268] |
Monk JM, Lloyd CJ, Brunk E, et al. iML1515, a knowledgebase that computes Escherichia coli traits. Nat Biotechnol, 2017, 35(10): 904-908. DOI:10.1038/nbt.3956
|
| [269] |
Institute MG. The Bio Revolution: Innovations transforming economies, societies, and our lives. 2020.
|
| [270] |
Gómez-Tatay L, Hernández-Andreu JM. Biosafety and biosecurity in Synthetic Biology: A review. Crit Rev Env Sci Technol, 2019, 49(17): 1587-1621. DOI:10.1080/10643389.2019.1579628
|
| [271] |
曾小美, 苏莉, 刘亚丰, 等. 合成生物学底盘微生物细胞的应用及其生物安全在创新型本科生培养中的实践. 微生物学通报, 2020, 47(4): 1224-1229. Zeng XM, Su L, Liu YF, et al. The practice of application and biosafety of classis microbial cells in the training of innovative undergraduate students. Microbiol China, 2020, 47(4): 1224-1229 (in Chinese). |
| [272] |
李真真, 董永亮, 高旖蔚. 设计生命: 合成生物学的安全风险与伦理挑战. 中国科学院院刊, 2018, 33(11): 1269-1276. Li ZZ, Dong YL, Gao YW. Design life: Safety risks and ethical challenges in synthetic biology. Bull Chin Acad Sci, 2018, 33(11): 1269-1276 (in Chinese). |
| [273] |
Stirling F, Bitzan L, O'keefe S, et al. Rational design of evolutionarily stable microbial kill switches. Mol Cell, 2017, 68(4): 686-697e. DOI:10.1016/j.molcel.2017.10.033
|
| [274] |
Stirling F, Naydich A, Bramante J, et al. Synthetic cassettes for pH-mediated sensing, counting, and containment. Cell Rep, 2020, 30(9): 3139-3148e. DOI:10.1016/j.ce
|
| [275] |
Noble C, Min J, Olejarz J, et al. Daisy-chain gene drives for the alteration of local populations. Proc Natl Acad Sci USA, 2019, 116(17): 8275-8282. DOI:10.1073/pnas.1716358116
|
| [276] |
马延和, 江会峰, 娄春波, 等. 合成生物与生物安全. 中国科学院院刊, 2016, 31(4): 432-438. Ma YH, Jiang HF, Lou CB, et al. Synthetic life and biosecurity. Bull Chin Acad Sci, 2016, 31(4): 432-438 (in Chinese). |
 2021, Vol. 37
2021, Vol. 37




