中国科学院微生物研究所、中国微生物学会主办
文章信息
- 丁娜娜, 周胜虎, 邓禹
- Ding Nana, Zhou Shenghu, Deng Yu
- 基于转录因子的代谢物生物传感器的研究进展
- Progress in transcription factor-based metabolite biosensors
- 生物工程学报, 2021, 37(3): 911-922
- Chinese Journal of Biotechnology, 2021, 37(3): 911-922
- 10.13345/j.cjb.200641
-
文章历史
- Received: October 8, 2020
- Accepted: January 5, 2021
在生物体内广泛存在着基于蛋白质或核酸元件(例如:转录因子、核糖开关、适体酶) 的生物传感器[1]。当目标代谢物存在时,它们可以通过改变蛋白质或核酸的构象来实现响应特性且可以将目标代谢物浓度信号转换为荧光信号、生长速率信号或者代谢通路表达水平信号。近年来,代谢物生物传感器在微生物细胞工厂的化合物生产中受到了很大的关注[2-4]。然而,由于生物传感器难以精准定量检测目标代谢物浓度,且荧光蛋白的过表达往往给细胞带来生长负担,进而影响质粒的拷贝数,使得荧光检测出现偏差,因此,生物传感器在微生物细胞中的应用面临着巨大的挑战。为了解决这些挑战,已有很多研究通过替换启动子[5]、改造核糖体结合位点(Ribosome binding site,RBS)[6-7]和改变质粒拷贝数[8]等策略来构建可调节的代谢物生物传感器以缓解上述问题[9]。但是这些传统的调节策略是耗时费力的过程,有很大的限制。因此,借助数学模型是解决这一限制的重要方向之一。本文综述了基于转录因子的代谢物生物传感器的类别、作用原理及特点,介绍了其在微生物细胞中的应用,指出了其在应用中存在的限制,并介绍了调节代谢物生物传感器性能的研究进展。
1 基于转录因子的代谢物生物传感器的类别及其作用原理代谢物生物传感器主要由生物识别元件和信号输出元件组成。根据识别元件的不同可以分为蛋白质(即转录因子) 生物传感器和核酸(即核糖开关或适体酶) 生物传感器。在这里,我们主要介绍基于转录因子的生物传感器。根据转录因子类型和对诱导剂的应答效应可以将其分为4种类别,即基于抑制-抑制子、激活-抑制子、抑制-激活子和激活-激活子的生物传感器[10]。如图 1A和1B所示,基于抑制-抑制子或激活-抑制子的生物传感器能识别目标代谢物,使得抑制子构象发生改变进而从转录因子结合位点上释放或结合,促进或抑制RNA聚合酶结合到诱导启动子上,最终激活或抑制感兴趣基因的表达。相反地,如图 1C和1D所示,基于抑制-激活子或激活-激活子的生物传感器通过识别相应的代谢物,从转录因子结合位点上释放或结合,最终抑制或激活目标基因的表达。

|
| 图 1 基于转录因子生物传感器工作原理示意图 Fig. 1 Schematic diagram of transcription factor-based biosensor principle. Pconst: constitutive promoter; Pinduc.: inducible promoter. (A) Repressed-repressor biosensor. In the absence of the target metabolite, the repressor was bound to an inducible promoter, preventing the expression of downstream genes ("OFF"). In the presence of the target metabolite, they were bound to the repressor and altered its conformation, resulting in the release of the repressor from the promoter, initiating the expression of the downstream genes ("ON"). (B) Activated-repressor biosensor. In the absence of the target metabolite, the repressor was inactive. It caused the biosensor to be in the "ON" state. In the presence of the target metabolite, they were bound to the repressor and activated its repression, preventing the expression of downstream genes ("OFF"). (C) Repressed-activator biosensor. In the absence of the target metabolite, the activator was bound to an inducible promoter, turning on the expression of downstream genes ("ON"). In the presence of the target metabolite, they were bound to the activator and altered its conformation, repressing the activation, preventing the expression of the downstream genes ("OFF"). (D) Activated-activator biosensor. In the absence of the target metabolite, the activator was inactive. It caused the biosensor to be in the "OFF" state. In the presence of the target metabolite, the activator was activated and increased the expression of inducible promoter-controlled genes. It made the biosensor to be in the "ON" state. |
| |
根据输出信号的不同,基于转录因子的代谢物生物传感器可以分为两个应用领域,即代谢物检测和代谢物调节。其中可检测的荧光通常在代谢物定量检测和高通量筛选的应用中作为输出信号,而生长速率和代谢通路调节水平通常在自适应进化和动态控制系统中作为输出信号。
2.1 目标代谢物浓度的检测近年来,代谢物生物传感器可以实时监测目标代谢物生产水平,例如对脂肪酸生产的实时监测[11]。通过构建输入信号(目标代谢物浓度) 和输出信号(荧光强度) 的剂量应答曲线作为标准曲线可以计算目标代谢物生产水平。Baumann等在酿酒酵母(Saccharomyces cerevisiae,S. cerevisiae)中开发了快速检测短中链脂肪酸的生物传感器,代替了传统的气相色谱检测方法[12]。该生物传感器包含响应己酸、庚酸和辛酸的诱导型启动子PDR12,且分别在0.01–2.00 mmol/L (R2=0.98),0.01–1.50 mmol/L (R2=0.99) 和0.01–0.75 mmol/L (R2=0.99) 浓度范围内观察到生物传感器对3种酸的线性响应[12]。虽然在检测范围内对目标代谢物浓度的检测是可靠的,但是在实际生产中,当目标代谢物浓度不在检测范围内,就很难实现对目标代谢物浓度的实时定量监测。因此,设计具有期望性能的代谢物生物传感器对于各种代谢物浓度的检测至关重要。
2.2 高通量筛选基于转录因子的代谢物生物传感器的另一个重要的应用是结合荧光激活细胞分选仪(Fluorescence activated cell sorter,FACS) 建立代谢物响应的高通量筛选系统以从庞大的变异体中筛选出高效生产的菌株[13-15]。筛选流程如图 2A所示。对于具有很多工业应用价值的脂肪酸衍生物来说,由于脂肪酸代谢的高度动态性质,难以使用合理的方法鉴定调控脂肪酰基辅酶A水平的关键基因。而代谢物生物传感器可用于从庞大文库的基因中高通量筛选出关键基因。因此,Dabirian等利用来自大肠杆菌(Escherichia coli,E. coli) 的脂肪酰基辅酶A传感器转录因子FadR,结合基因过表达文库,在S. cerevisiae中建立并高通量筛选出能够提高脂酰CoA通量的关键基因[13]。利用PTEF1启动子调控来源于E. coli的FadR表达及其结合位点,最终可以控制输出信号GFP的表达[13]。当胞内存在脂酰CoA时,FadR会从其结合位点脱离,进而导致GFP的表达增强[13]。因此将S. cerevisiae的多基因过表达文库和传感器质粒进行共转化,就可以通过FACS分析,鉴定出S. cerevisiae中增加酰基辅酶A水平的关键基因。基于该高通量策略,发现RTC3、GGA2和LPP1的过表达导致脂肪醇水平增加约80%[13]。这表明使用这种酰基辅酶A生物传感器与基因过表达文库相结合,可以鉴定基因靶点,从而改善脂肪酸及其衍生产品的合成。然而,荧光蛋白的高输出通常会给细胞带来负担,影响细胞生长和荧光检测[16-18]。因此,使用抗性基因来代替荧光进而选择高产菌株也许会缓解这一现象。

|
| 图 2 基于转录因子生物传感器应用[21, 25] Fig. 2 The application of transcription factor-based biosensor[21, 25]. Pinduc.: inducible promoter; S: substrate; Ei: Enzyme i; P: product; GOI: gene of interest. (A) High-throughput screening system of high yield strains. Metabolite production pathway and biosensor were transformed into the target strain to screen high yield strains by FACS. (B) Nongenetic evolution system of high yield strains. Continuously screen high-performance variants using resistant selectors to increase their population and improve production efficiency. (C) The universal quorum-sensing circuit combined with CRISPRi effectively increased the production of target metabolites. |
| |
一般地,微生物在适应环境的过程中,菌体会发生变异、改变表型,这是进化的必然趋势。因此,微生物自适应实验室进化(Adaptive laboratory evolution,ALE) 系统应运而生。基于转录因子的代谢物生物传感器介导的自适应实验室进化是高效生产目标代谢物的强有力的策略[19]。通过这种方式,FACS或者以细胞为主的表型选择方案不断对代谢物生物传感器输出施加人工选择压力,以实现对特定高产菌株的选择。Mahr等使用人工选择压力结合基于转录调节子Lrp的生物传感器和FACS技术建立了自适应实验室进化系统,通过5轮进化,其中每一轮都通过FACS筛选并转移10%的最强荧光细胞,分离的谷氨酸棒状杆菌(Corynebacterium glutamicum,C. glutamicum) 变异体在仅有7个突变位点的情况下表现出比亲本菌株更高的生长速率,最终导致L-缬氨酸产量增加了25%,同时,副产物量减少了3–4倍[20]。然而,由于在细胞之间天然存在着非遗传因素使得其蛋白质表达和代谢物浓度水平有很大差异,因此,发酵异质性是影响代谢物生产的另一个重要因素[18]。
此外,Rugbjerg等的研究也表明遗传异质性在工业发酵过程中会由于代谢负担和毒性导致生产负荷,负荷在一定程度上影响产量、速率和效价,负荷的减少可以带来巨大的效益[18]。因此,研究者们通过深度DNA测序用于检测生产者群体中不良的遗传异质性,并诊断相关的遗传错误模式,即在工业发酵中,由于来自剪切、渗透压和饥饿的细胞外应力,逃逸率可能高于研究实验室条件[18]。遗传异质性的研究可以查明导致细胞负荷问题和产品毒性的机制。其中通过分离物质生长和生产的两阶段过程来延迟生产,可以减少细胞负荷的影响[18]。而平衡酶的浓度或效率,可以尽量减少有毒中间物。理解异质性将为代谢工程和合成生物学策略提供信息,以减少非生产突变体的出现,并最大限度地提高产品的质量和产量[18]。此外,遗传电路的构建将通过调节细胞代谢以响应细胞外信号或随机变化的单细胞胞内环境,以减少生产负荷或抑制表现不佳的种群来限制不利的异质性[18]。
为了降低发酵异质性对代谢物生产的影响,Xiao等使用基于FadR的生物传感器,开发了群体质量控制(Population quality control,PopQC)系统来连续地富集高产细胞和减少低效生产的细胞,因此,改善了群体细胞的性能[21]。如图 2B所示,PopQC通过生物传感器输出的四环素抗性有效地奖励具有竞争性细胞生长优势的高性能细胞。PopQC在酪氨酸和脂肪酸生产中的应用分别使其产量提高了2.6倍和3倍[21]。这些例子表明,代谢物生物传感器驱动的适应性进化在改善微生物代谢物生产和加速适应性进化方面发挥着至关重要的作用。
2.4 动态控制在目标代谢物生产过程中,通常会检测到很多副产物,这不仅会大量消耗细胞中的碳源和能源,还会给细胞造成生长负担[18]。因此,代谢工程一直朝着将代谢通量直接定位到目标代谢物合成途径且不会影响细胞的生长的方向而努力。然而,传统的代谢物改造策略如基因的敲除、沉默、下调或过表达容易使代谢通量失衡,虽然模块化代谢工程可以更合理地分配代谢通量,但是这些对代谢途径的改造手段是恒定的,属于静态调节,会影响细胞生长[22]。并且代谢通量是随着胞内代谢物水平和细胞环境的变化而进行着动态调节。动态控制的特点是在生长阶段全部的通量都用来生长,生产阶段全部的通量都用来生产[22]。因此,通过构建基于转录因子的代谢物生物传感器动态控制系统,根据细胞环境的变化对代谢通量进行实时调控,可以有效提高目标代谢物生产且不损害细胞生长。
当前,基因回路被广泛应用于代谢途径的动态控制,其可以有效缓解有毒中间代谢物的积累,平衡细胞生长与产物合成的代谢通量,从而提高产物的合成效率。因此,Xu等基于E. coli来源的丙酮酸响应转录因子PdhR,在枯草芽孢杆菌(Bacillus subtilis,B. subtilis) 中构建了丙酮酸激活型基因回路,并结合反义转录以及高通量筛选技术成功获得了丙酮酸抑制型的基因回路,从而获得受胞内丙酮酸响应的双功能基因回路[23]。丙酮酸是连接细胞糖酵解途径与三羧酸循环的关键中心代谢物,利用双功能丙酮酸生物传感器设计反馈控制系统,能够使细胞自发地响应胞内丙酮酸的浓度,动态调节B. subtilis中心碳代谢途径的代谢通量,使得B. subtilis中高效生产葡萄糖二酸,使其摇瓶产量提高了154%,而传统的静态控制方法仅提高了35%[23]。这表明碳中心代谢流的动态控制促进了中心代谢衍生产物的高效合成。
然而,目前可以使用的动态调控元件非常缺乏,很多代谢物没有发现其特异性响应的转录因子。从蛋白质全新合成角度考虑,虽然,Glasgow等报道了一种设计传感器-致动器蛋白的计算策略,但是尚无用于工程化任意新蛋白质传感器的通用方法[24]。因此,开发便捷、普适和自发驱动的动态控制系统为微生物天然产物有效合成具有重大意义。Tian等在链霉菌中开发了一种新型的动态调控系统EQCi (Endogenous quorum-sensing (QS) system with clustered regularly interspaced short palindromic repeats interference (CRISPRi)),使用γ-丁内酯(γ-butyrolactone,GBL) 信号分子响应的启动子驱动dCas9基因的表达,将群体感应系统与基因转录抑制技术(CRISPRi) 进行偶联[25]。如图 2C所示,能够同时对代谢途径中多个基因进行全自动精细动态控制。通过选择具有抗癌和免疫抑制等生物活性的雷帕霉素为目标产物,利用EQCi构建雷帕霉素重组菌株。分别通过下调三羧酸循环途径、脂肪酸合成途径和莽草酸途径这3个初级代谢途径中关键节点基因的转录,来增加雷帕霉素生物合成的前体供应,提高了生产效价[25]。然后使用EQCi系统对3个途径的代谢通量进行组合干预和精细微调每个节点的控制强度,获得了雷帕霉素效价为(1 836±191) mg/L的最优工程菌株,与天然菌株相比,提高了约6.6倍[25]。表明EQCi系统有效地平衡了初级代谢和产物合成(次级代谢) 的代谢通量分配,且为链霉菌来源的重要次级代谢天然产物细胞工厂的构建提供了高效通用的优化策略。
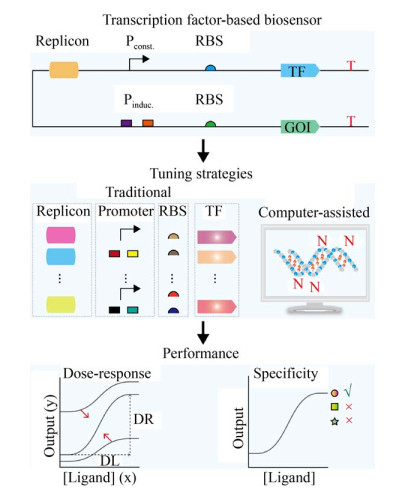
3 代谢物生物传感器的性能调节从代谢物生物传感器的各种应用中可以发现很多生物传感器的性能不足以发挥其在代谢工程和合成生物学中的应用潜力。因此,设计可调节的生物传感器性能可以发挥其最好的应用价值。生物传感器性能一般通过灵敏性、检测范围、动态范围和特异性来表征[9, 26]。其中,灵敏性表示与代谢物浓度和信号输出相关的拟合的希尔函数(Hill function) 的hill系数,描述了从低输出到高输出的陡峭程度[27]。检测范围表示可检测到的代谢物浓度下限和上限的范围[28]。动态范围表示在存在和缺乏代谢物时信号输出基因表达的倍数变化[29]。特异性表示与结合潜在的其他代谢物相比,结合靶代谢物后输出信号增加的相对差异[30]。此外,hill系数、检测范围和动态范围可以通过建立hill方程(剂量应答曲线) 来表征。因此,设计合适的调节策略来实现生物传感器的性能调节具有重要的意义。
然而,与原核生物不同,由于真核生物中转录和蛋白质表达的物理分离,转录因子跨核膜的核进出口机制在调节代谢物生物传感器性能中起关键作用[31]。因此,原核和真核生物传感器的设计机制不同。首先,真核传感器中的转录激活因子通过增加转录机制(介导激活因子和RNA聚合酶Ⅱ之间信号的蛋白质复合物) 的可及性来增强基因表达,从而结合转录起始位点并开启转录[32]。而原核生物传感器中的转录因子没有发现这样的转录机制。其次,真核传感器中的抑制子有多种机制阻止转录,包括基础转录机制的抑制、激活剂的失活和染色质或核小体3D结构的重塑[33]。然而,当前原核衍生的转录抑制子已被成功地工程化为酵母和哺乳动物细胞中的生物传感器[34]。例如,B. subtilis中转录抑制子FapR和同源DNA结合位点fapO已被工程化以在S. cerevisiae中构建丙二酸辅酶A传感器[35]。此外,与原核传感器不同,大多数转录调节发生在真核细胞的细胞核中,通常需要核定位信号(Nuclear localization signal,NLS)[35]才能使转录因子通过核膜,另外,代谢物或效应分子并不总是能自由渗透到细胞核膜,关闭和开启转录活性之间的转换要求将转录因子选择性地导入细胞核中并与靶启动子相互作用以控制基因表达。最后,由于真核细胞的复杂性,报告基因的选择决定了背景噪音以及如何表征真核生物传感器的方法。由于其简单性、高灵敏度和低背景噪音,非ATP依赖的荧光素酶(NanoLuc)[36]是鉴定真核转录系统的有前景的报告蛋白。当前的调节生物传感器性能的策略可以区分为传统调节策略和计算机辅助的调节。
3.1 传统调节策略一般地,可以通过改造生物传感器中启动子来调节生物传感器的性能,其中包括控制调节蛋白转录的组成型启动子[5, 37-38]和含有调节蛋白结合位点的诱导型启动子[23, 38-40]。如图 3所示,通过改造组成型启动子可以在转录水平上调控调节蛋白的转录进而影响生物传感器的剂量应答曲线。Kunjapur等在E. coli中使用新月柄杆菌(Caulobacter crescentus,C. crescentus) 的VanR-VanO系统构建了一种具有功能的香草酸生物传感器[37]。使用3种Anderson启动子(BBaJ23101,BBaJ23102,BBaJ23108) 代替C. crescentus的天然启动子来调控转录因子VanR的表达水平,并通过对VanR结合位点VanO的优化,获得了超过14倍的生物传感器动态范围,且构建的生物传感器具有显著的代谢物特异性,对副产物异香草酸没有响应,对结构类似的途径中间代谢物有很小的应答[37],表明在将来可用于改善香草醛途径。另外,用3种天然S. cerevisiae启动子TDH3、RNR2和REV1来控制benM的转录进而调节转录因子BenM的表达,最终提高了S. cerevisiae中基于BenM的生物传感器的动态范围[41]。
而改造诱导型启动子除了优化-35区和-10区之外,还可以通过调控调节蛋白和结合位点的亲和力进而影响下游报告基因的转录[26, 42-43]。Chen等在E. coli中通过设计亚砷酸响应的生物传感器调控启动子Pars的核心元件,即RNA聚合酶结合位点和转录因子ArsR结合位点(ArsR binding site,ABS),显著提高了生物传感器的动态范围[43]。通过对调控启动子的-10区进行定点突变,构建了7种响应亚砷酸的启动子变体(ParsA-ParsG),提高了启动子的活性[43]。此外,在-10区的下游额外添加了ArsR结合位点,这有效地降低了生物传感器响应亚砷酸的背景噪音[43]。在1 μmol/L亚砷酸诱导下,调控启动子变体ParsD-ABS-8形成的生物传感器具有179倍的动态范围,更重要的是,在0.1–4.0 μmol/L砷的检测范围内表现出良好的剂量响应(R2=0.992 8)[43]。此外,在真核生物传感器中的TATA序列上游插入转录因子结合位点,虽然启动子强度下调了,但实际上灵敏度和动态范围得到了相应地提高[44-45]。这表明调控启动子改造策略可以提高生物传感器的性能,从而促进其实际应用。
此外,调节蛋白和报告基因表达也可以通过改变质粒拷贝数进而调节生物传感器性能[46-47]。如图 3所示,Trabelsi等开发了一种基于调节子的响应生松素(Pinocembrin) 的新型生物传感器[47]。通过改变质粒拷贝数和响应柚皮素生物传感器中转录因子FdeR的浓度构建了组合文库,对不同的响应进行记录和建模,拟合的模型对于理解改变策略对生物传感器剂量应答曲线提供指导作用,可以用来构建具有强灵敏度和宽的线性检测范围的生物传感器[47]。启动子和质粒拷贝数工程主要在转录水平上调节蛋白质水平进而影响生物传感器性能。在翻译水平上,调节蛋白质翻译起始的关键因素之一是RBS。改造RBS可以通过控制调节蛋白和报告蛋白的翻译水平进而调节生物传感器的剂量应答曲线[38, 47-48]。Kasey等通过RBS饱和突变策略,筛选到了基于阻遏蛋白MphR对红霉素响应提高10倍的灵敏性,而动态范围却没有显著降低[49]。这表明RBS工程对于提高基于MphR生物传感器的灵敏性是一个有效的策略。
对于生物传感器的特异性性能来说,调节蛋白的结构决定了其特异性。因此,目前研究主要是通过蛋白质工程来理性或非理性地改变蛋白质的结构。SnoeK等提出来一种通用的高通量方法用于进化S. cerevisiae中的原核转录因子特异性和剂量应答曲线[50]。通过对效应物结合域(Effector-binding domain,EBD) 编码序列进行易错PCR引入随机突变,结合基于FACS的筛选机制,筛选到粘康酸诱导的转录因子BenM变体,进化到特异性响应己二酸的变体,并增加了动态范围和检测范围[50]。更重要的是,仅对EBD随机突变进化得到的生物传感器显示出与BenM相似的DNA结合亲和力,且在移植回原核生物底盘时仍然能发挥作用[50]。因此,此技术可以实现利用转录因子的可扩展性来开发具有用户定义的小分子特异性,同时不受宿主类型限制的新型生物传感器。
3.2 计算机辅助的调节虽然,传统调节方法可以在一定程度上调谐生物传感器的性能。然而,传统的调节策略一般需要不断地设计-构建-测试-学习循环,这循环过程是耗时而费力的。此外,生物传感器的内在调控机制是复杂的,不仅要考虑调节蛋白和结合位点的结合机制,还要考虑调节蛋白和报告蛋白的折叠态对生物传感器性能的影响。然而,研究这些机制仍然面临着很大的挑战[51]。如果借助数学模型比如机器学习或深度学习的方法可以不用考虑复杂的作用机理,可以快速精准地实现生物传感器性能的调节[52]。如图 3所示,深度学习是一种算法,它使用人工神经网络(例如,卷积神经网络(Convolutional neural network,CNN) 和循环神经网络(Recurrent neural network,RNN)) 作为框架来表征和学习数据集。机器学习使用贝叶斯、支持向量机和逻辑回归等算法挖掘事物背后隐藏的规则和本质,通过数据集训练获得模型[53-54]。通过开发机器学习或深度学习模型可以以DNA、RNA、氨基酸序列等任何生物学序列作为数据输入,来处理很多生物学问题。例如,通过大的生物数据库产生可视化图片、预测蛋白质折叠、分析染色体变异等。
Ding等在E. coli中基于RBS大数据文库开发了深度学习模型(Classification model between cRBSs and the average dynamic range of each sub-library,CLM-RDR),实现了RBS对生物传感器动态范围的智能化调控[55]。以葡萄糖二酸生物传感器为研究对象,通过随机选择不同来源的RBS替换调节基因cdaR和报告基因sfgfp的RBS,构建了81种cRBS生物传感器,经过方差分析发现RBS对改善生物传感器动态范围贡献最大[55]。此外,还揭示了RBS不仅影响蛋白质的翻译水平还影响蛋白质的折叠态[55]。然后,建立了RBS设计原则,基于该原则通过DNA芯片构建了12 000种cRBSs组合。随后,结合FACS分选技术和二代测序技术获得了7 053种cRBSs序列。最后,借助卷积神经网络CNN模型通过对RBS数据及其特征的不断学习建立了RBS和动态范围之间的分类模型,即CLM-RDR模型[55]。其中在受试者工作特征曲线(Receiver operating characteristic curve,ROC曲线) 下的平均面积为0.86,表明了此模型具有优越的预测性能[55],实现了生物传感器动态范围的智能化调控。
4 总结与展望基于转录因子的代谢物生物传感器在代谢工程中的应用取得了很大的进展[25, 56-58]。然而,对于在诱导剂存在下开启转录的生物传感器来说,在代谢物浓度定量检测和高产菌株高通量筛选方面,往往会出现高的荧光信号对应低的代谢物浓度,这会导致不准确的检测和筛选系统,因此,可以通过改造生物传感器性能或者使用抗性筛选基因替换荧光报告基因等方法来构建精准的、可扩展的检测和筛选系统;在自适应进化方面,虽然不需要考虑菌体复杂的代谢网络,只需根据目标设计对应的干扰因素来实现表型优化,但是也有其自身的缺陷,如连续培养过程中经过筛选得到的无效突变菌株的扩增以及对设备的高度依赖等,因此,通过借助计算机技术实现自适应自动化进化设计将有望使其潜力发挥出更大的作用;在代谢途径的动态控制方面,虽然可以有效避免静态改造策略造成的中间代谢物积累和细胞生长负担等问题,但是目前可以使用的调控元件非常缺乏,且很多元件对代谢途径的依赖性较强,因此,除了挖掘更多的动态调控元件,还需要进行适当的改造以拓展其适用范围,此外,也可以通过构建逻辑回路实现生物传感器性能更为精细的调节。
尽管对生物传感器性能的调节已经有了很大的改进,但是传统的改造方法存在着数据量少、通用性差以及理性设计弱等问题[37-38]。因此,为了更好更快地实现生物传感器性能的可预测精细调节而不受物种、环境条件等的限制,并改善通用性,将理性设计的大量的生物元件库与数学模型如深度学习或机器学习等相结合,可以实现一个生物元件(如启动子、RBS和氨基酸序列等)所对应的生物传感器性能的可预测性[54-55, 59]。另外,获得对应于数学模型训练的每个步骤的表型变化,将快速开发模型映射的生物传感器。这将为生物传感器在合成生物学和代谢工程中的应用发挥重要的指导作用。此外,借助生成模型还可以实现大量生物元件的全新合成,这极大地丰富了合成生物学的元件知识库。
然而,仍然存在许多需要克服的难题。首先,大量元件库的理性和鲁棒性的设计一般需要依赖诸如微阵列[55, 60]这样的技术来合成千万个调节元件,随后的分析也依赖于流式细胞仪或微流控技术,但是这些技术的成本较高。其次,分析和验证数学模型每一步产生的生物表型变化不仅需要大量的数据支持,还需要专业的人才,因此,交叉学科技术的融合是克服此问题的方法之一[55, 61]。最后,要了解生物元件如RBS对下游蛋白质折叠的影响,首先要确定与蛋白质折叠直接或间接相关的因素或参数,这是解决此问题的关键[51, 55, 62],这些因素或参数可以从现有文献中获得,也可以通过理性建模来定义。总之,应该致力于建立更为精细调节的生物传感器,以更加精准地检测代谢物浓度,并为建立更复杂的遗传电路提供广阔的数据资源。
| [1] |
Carpenter AC, Paulsen IT, Williams TC. Blueprints for biosensors: design, limitations, and applications. Genes, 2018, 9(8): 375. DOI:10.3390/genes9080375
|
| [2] |
Chen XL, Gao C, Guo L, et al. DCEO biotechnology: Tools to design, construct, evaluate, and optimize the metabolic pathway for biosynthesis of chemicals. Chem Rev, 2018, 118(1): 4-72. DOI:10.1021/acs.chemrev.6b00804
|
| [3] |
Ko YS, Kim JW, Lee JA, et al. Tools and strategies of systems metabolic engineering for the development of microbial cell factories for chemical production. Chem Soc Rev, 2020, 49(14): 4615-4636. DOI:10.1039/D0CS00155D
|
| [4] |
Lee S, Kim P. Current status and applications of adaptive laboratory evolution in industrial microorganisms. J Microbiol Biotechnol, 2020, 30(6): 793-803. DOI:10.4014/jmb.2003.03072
|
| [5] |
Thompson MG, Pearson AN, Barajas JF, et al. Identification, characterization, and application of a highly sensitive lactam biosensor from Pseudomonas putida. ACS Synth Biol, 2020, 9(1): 53-62. DOI:10.1021/acssynbio.9b00292
|
| [6] |
Yeom SJ, Kim M, Kwon KK, et al. A synthetic microbial biosensor for high-throughput screening of lactam biocatalysts. Nat Commun, 2018, 9(1): 5053. DOI:10.1038/s41467-018-07488-0
|
| [7] |
Kalkreuter E, Keeler AM, Malico AA, et al. Development of a genetically encoded biosensor for detection of polyketide synthase extender units in Escherichia coli. ACS Synth Biol, 2019, 8(6): 1391-1400. DOI:10.1021/acssynbio.9b00078
|
| [8] |
Lim HG, Jang S, Jang S, et al. Design and optimization of genetically encoded biosensors for high-throughput screening of chemicals. Curr Opin Biotechnol, 2018, 54: 18-25. DOI:10.1016/j.copbio.2018.01.011
|
| [9] |
Kent R, Dixon N. Contemporary tools for regulating gene expression in Bacteria. Trends Biotechnol, 2020, 38(3): 316-333. DOI:10.1016/j.tibtech.2019.09.007
|
| [10] |
Mannan AA, Liu D, Zhang FZ, et al. Fundamental design principles for transcription-factor-based metabolite biosensors. ACS Synth Biol, 2017, 6(10): 1851-1859. DOI:10.1021/acssynbio.7b00172
|
| [11] |
Rogers JK, Church GM. Genetically encoded sensors enable real-time observation of metabolite production. Proc the Natl Acad Sci USA, 2016, 113(9): 2388-2393. DOI:10.1073/pnas.1600375113
|
| [12] |
Baumann L, Rajkumar AS, Morrissey JP, et al. A Yeast-based biosensor for screening of short-and medium-chain fatty acid production. ACS Synth Biol, 2018, 7(11): 2640-2646. DOI:10.1021/acssynbio.8b00309
|
| [13] |
Dabirian Y, Teixeira PG, Nielsen J, et al. FadR-based biosensor-assisted screening for genes enhancing fatty acyl-CoA pools in Saccharomyces cerevisiae. ACS Synth Biol, 2019, 8(8): 1788-1800. DOI:10.1021/acssynbio.9b00118
|
| [14] |
Li LP, Tu R, Song GT, et al. Development of a synthetic 3-dehydroshikimate biosensor in Escherichia coli for metabolite monitoring and genetic screening. ACS Synth Biol, 2019, 8(2): 297-306. DOI:10.1021/acssynbio.8b00317
|
| [15] |
Lin JL, Wagner JM, Alper HS. Enabling tools for high-throughput detection of metabolites: Metabolic engineering and directed evolution applications. Biotechnol Adv, 2017, 35(8): 950-970. DOI:10.1016/j.biotechadv.2017.07.005
|
| [16] |
Zhou SH, Ding RP, Chen J, et al. Obtaining a panel of cascade promoter-5'-UTR complexes in Escherichia coli. ACS Synth Biol, 2017, 6(6): 1065-1075. DOI:10.1021/acssynbio.7b00006
|
| [17] |
Yang D, Park SY, Park YS, et al. Metabolic engineering of Escherichia coli for natural product biosynthesis. Trends Biotechnol, 2020, 38(7): 745-765. DOI:10.1016/j.tibtech.2019.11.007
|
| [18] |
Rugbjerg P, Sommer MOA. Overcoming genetic heterogeneity in industrial fermentations. Nat Biotechnol, 2019, 37(8): 869-876. DOI:10.1038/s41587-019-0171-6
|
| [19] |
Stella RG, Wiechert J, Noack S, et al. Evolutionary engineering of Corynebacterium glutamicum. Biotechnol J, 2019, 14(9): 1800444. DOI:10.1002/biot.201800444
|
| [20] |
Mahr R, Gätgens C, Gätgens J, et al. Biosensor-driven adaptive laboratory evolution of L-valine production in Corynebacterium glutamicum. Metab Eng, 2015, 32: 184-194. DOI:10.1016/j.ymben.2015.09.017
|
| [21] |
Xiao Y, Bowen CH, Liu D, et al. Exploiting nongenetic cell-to-cell variation for enhanced biosynthesis. Nat Chem Biol, 2016, 12(5): 339-344. DOI:10.1038/nchembio.2046
|
| [22] |
Venayak N, Anesiadis N, Cluett WR, et al. Engineering metabolism through dynamic control. Curr Opin Biotech, 2015, 34: 142-152. DOI:10.1016/j.copbio.2014.12.022
|
| [23] |
Xu XH, Li XL, Liu YF, et al. Pyruvate-responsive genetic circuits for dynamic control of central metabolism. Nat Chem Biol, 2020, 16(11): 1261-1268. DOI:10.1038/s41589-020-0637-3
|
| [24] |
Glasgow AA, Huang YM, Mandell DJ, et al. Computational design of a modular protein sense-response system. Science, 2019, 366(6468): 1024-1028. DOI:10.1126/science.aax8780
|
| [25] |
Tian JZ, Yang GH, Gu Y, et al. Developing an endogenous quorum-sensing based CRISPRi circuit for autonomous and tunable dynamic regulation of multiple targets in Streptomyces. Nucleic Acids Res, 2020, 48(14): 8188-8202. DOI:10.1093/nar/gkaa602
|
| [26] |
Hicks M, Bachmann TT, Wang BJ. Synthetic biology enables programmable cell-based biosensors. ChemPhysChem, 2020, 21(2): 132-144. DOI:10.1002/cphc.201900739
|
| [27] |
Thompson MG, Costello Z, Hummel NFC, et al. Robust characterization of two distinct glutarate sensing transcription factors of Pseudomonas putida L-lysine metabolism. ACS Synth Biol, 2019, 8(10): 2385-2396. DOI:10.1021/acssynbio.9b00255
|
| [28] |
Cheng F, Tang XL, Kardashliev T. Transcription factor-based biosensors in high-throughput screening: advances and applications. Biotechnol J, 2018, 13(7): 1700648. DOI:10.1002/biot.201700648
|
| [29] |
Zhang FZ, Carothers JM, Keasling JD. Design of a dynamic sensor-regulator system for production of chemicals and fuels derived from fatty acids. Nat Biotechnol, 2012, 30(4): 354-359. DOI:10.1038/nbt.2149
|
| [30] |
De Paepe B, Peters G, Coussement P, et al. Tailor-made transcriptional biosensors for optimizing microbial cell factories. J Ind Microbiol Biotechnol, 2017, 44(4/5): 623-645.
|
| [31] |
Wan X, Marsafari M, Xu P. Engineering metabolite-responsive transcriptional factors to sense small molecules in eukaryotes: current state and perspectives. Microb Cell Fact, 2019, 18: 61. DOI:10.1186/s12934-019-1111-3
|
| [32] |
Pia Cosma M. Ordered recruitment: Gene-specific mechanism of transcription activation. Mol Cell, 2002, 10(2): 227-236. DOI:10.1016/S1097-2765(02)00604-4
|
| [33] |
Gaston K, Jayaraman PS. Transcriptional repression in eukaryotes: repressors and repression mechanisms. Cell Mol Life Sci, 2003, 60(4): 721-741. DOI:10.1007/s00018-003-2260-3
|
| [34] |
Gossen M, Freundlieb S, Bender G, et al. Transcriptional activation by tetracyclines in mammalian-cells. Science, 1995, 268(5218): 1766-1769. DOI:10.1126/science.7792603
|
| [35] |
Li SJ, Si T, Wang M, et al. Development of a synthetic malonyl-CoA sensor in Saccharomyces cerevisiae for intracellular metabolite monitoring and genetic screening. ACS Synth Biol, 2015, 4(12): 1308-1315. DOI:10.1021/acssynbio.5b00069
|
| [36] |
England CG, Ehlerding EB, Cai WB. NanoLuc: a small luciferase is brightening up the field of bioluminescence. Bioconjugate Chem, 2016, 27(5): 1175-1187. DOI:10.1021/acs.bioconjchem.6b00112
|
| [37] |
Kunjapur AM, Prather KLJ. Development of a vanillate biosensor for the vanillin biosynthesis pathway in E. coli. ACS Synth Biol, 2019, 8(9): 1958-1967. DOI:10.1021/acssynbio.9b00071
|
| [38] |
Johnson AO, Gonzalez-Villanueva M, Wong L, et al. Design and application of genetically-encoded malonyl-CoA biosensors for metabolic engineering of microbial cell factories. Metab Eng, 2017, 44: 253-264. DOI:10.1016/j.ymben.2017.10.011
|
| [39] |
Liu XY, Silverman AD, Alam KK, et al. Design of a transcriptional biosensor for the portable, on-demand detection of cyanuric acid. ACS Synth Biol, 2020, 9(1): 84-94. DOI:10.1021/acssynbio.9b00348
|
| [40] |
Wu YK, Chen TC, Liu YF, et al. Design of a programmable biosensor-CRISPRi genetic circuits for dynamic and autonomous dual-control of metabolic flux in Bacillus subtilis. Nucleic Acids Res, 2020, 48(2): 996-1009. DOI:10.1093/nar/gkz1123
|
| [41] |
Skjoedt ML, Snoek T, Kildegaard KR, et al. Engineering prokaryotic transcriptional activators as metabolite biosensors in yeast. Nat Chem Biol, 2016, 12(11): 951-958. DOI:10.1038/nchembio.2177
|
| [42] |
Chen Y, Ho JML, Shis DL, et al. Tuning the dynamic range of bacterial promoters regulated by ligand-inducible transcription factors. Nat Commun, 2018, 9: 64. DOI:10.1038/s41467-017-02473-5
|
| [43] |
Chen SY, Wei WP, Yin BC, et al. Development of a highly sensitive whole-cell biosensor for arsenite detection through engineered promoter modifications. ACS Synth Biol, 2019, 8(10): 2295-2302. DOI:10.1021/acssynbio.9b00093
|
| [44] |
Feng J, Jester BW, Tinberg CE, et al. A general strategy to construct small molecule biosensors in eukaryotes. eLife, 2015, 4: e10606. DOI:10.7554/eLife.10606
|
| [45] |
Hector RE, Mertens JA. A Synthetic hybrid promoter for xylose-regulated control of gene expression in Saccharomyces yeasts. Mol Biotechnol, 2017, 59(1): 24-33. DOI:10.1007/s12033-016-9991-5
|
| [46] |
Thompson MG, Sedaghatian N, Barajas JF, et al. Isolation and characterization of novel mutations in the pSC101 origin that increase copy number. Sci Rep, 2018, 8: 1590. DOI:10.1038/s41598-018-20016-w
|
| [47] |
Trabelsi H, Koch M, Faulon JL. Building a minimal and generalizable model of transcription factor-based biosensors: Showcasing flavonoids. Biotechnol Bioeng, 2018, 115(9): 2292-2304. DOI:10.1002/bit.26726
|
| [48] |
Siedler S, Khatri NK, Zsohár A, et al. Development of a bacterial biosensor for rapid screening of yeast p-coumaric acid production. ACS Synth Biol, 2017, 6(10): 1860-1869. DOI:10.1021/acssynbio.7b00009
|
| [49] |
Kasey CM, Zerrad M, Li YW, et al. Development of transcription factor-based designer macrolide biosensors for metabolic engineering and synthetic biology. ACS Synth Biol, 2017, 7(1): 227-239.
|
| [50] |
Snoek T, Chaberski EK, Ambri F, et al. Evolution-guided engineering of small-molecule biosensors. Nucleic Acids Res, 2020, 48(1): e3. DOI:10.1093/nar/gkz954
|
| [51] |
Faure G, Ogurtsov AY, Shabalina SA, et al. Role of mRNA structure in the control of protein folding. Nucleic Acids Res, 2016, 44(22): 10898-10911. DOI:10.1093/nar/gkw671
|
| [52] |
Carbonell P, Radivojevic T, Martín HG. Opportunities at the intersection of synthetic biology, machine learning, and automation. ACS Synth Biol, 2019, 8(7): 1474-1477. DOI:10.1021/acssynbio.8b00540
|
| [53] |
Guo JH, Wang TM, Guan CG, et al. Improved sgRNA design in bacteria via genome-wide activity profiling. Nucleic Acids Res, 2018, 46(14): 7052-7069. DOI:10.1093/nar/gky572
|
| [54] |
Liu XY, Gupta STP, Bhimsaria D, et al. De novo design of programmable inducible promoters. Nucleic Acids Res, 2019, 47(19): 10452-10463. DOI:10.1093/nar/gkz772
|
| [55] |
Ding NN, Yuan ZQ, Zhang XJ, et al. Programmable cross-ribosome-binding sites to fine-tune the dynamic range of transcription factor-based biosensor. Nucleic Acids Res, 2020, 48(18): 10602-10613. DOI:10.1093/nar/gkaa786
|
| [56] |
Wan XY, Volpetti F, Petrova E, et al. Cascaded amplifying circuits enable ultrasensitive cellular sensors for toxic metals. Nat Chem Biol, 2019, 15(5): 540-548. DOI:10.1038/s41589-019-0244-3
|
| [57] |
Kimura Y, Kawai-Noma S, Saito K, et al. Directed evolution of the stringency of the LuxR Vibrio fischeri quorum sensor without off-state selection. ACS Synth Biol, 2020, 9(3): 567-575. DOI:10.1021/acssynbio.9b00444
|
| [58] |
Zeng WZ, Guo LK, Xu S, et al. High-throughput screening technology in industrial biotechnology. Trends Biotechnol, 2020, 38(8): 888-906. DOI:10.1016/j.tibtech.2020.01.001
|
| [59] |
Kim HK, Min S, Song M, et al. Deep learning improves prediction of CRISPR-Cpf1 guide RNA activity. Nat Biotechnol, 2018, 36(3): 239-241. DOI:10.1038/nbt.4061
|
| [60] |
Wang TM, Guan CG, Guo JH, et al. Pooled CRISPR interference screening enables genome-scale functional genomics study in bacteria with superior performance. Nat Commun, 2018, 9: 2475. DOI:10.1038/s41467-018-04899-x
|
| [61] |
Chen KM, Cofer EM, Zhou J, et al. Selene: a PyTorch-based deep learning library for sequence data. Nat Methods, 2019, 16(4): 315-318. DOI:10.1038/s41592-019-0360-8
|
| [62] |
Sauer C, Ver Loren Van Themaat E, Boender LGM, et al. Exploring the nonconserved sequence space of synthetic expression modules in Bacillus subtilis. ACS Synth Biol, 2018, 7(7): 1773-1784. DOI:10.1021/acssynbio.8b00110
|
 2021, Vol. 37
2021, Vol. 37