中国科学院微生物研究所、中国微生物学会主办
文章信息
- 康倩, 向梦洁, 张大伟
- Kang Qian, Xiang Mengjie, Zhang Dawei
- 枯草芽孢杆菌在系统与合成生物技术中研究进展及工业应用
- Research progress and industrial application of Bacillus subtilis in systematic and synthetic biotechnology
- 生物工程学报, 2021, 37(3): 923-938
- Chinese Journal of Biotechnology, 2021, 37(3): 923-938
- 10.13345/j.cjb.200638
-
文章历史
- Received: October 7, 2020
- Accepted: January 26, 2021
- Published: March 15, 2021
2. 中国科学院大学,北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
枯草芽孢杆菌是革兰氏阳性细菌的模式菌株之一,是常见的杆状细菌,能产生芽孢,广泛存在于土壤等自然环境中。因为其菌株基因组测序及必需基因解析较为清晰[1-2],枯草芽孢杆菌常被用作细菌遗传学及细胞代谢研究的模式菌株。除此之外,枯草芽孢杆菌菌株安全性强,被美国食品药品监督局(US Food and Drug Administration,FDA) 认定是公认的安全菌株(Generally recognized as safe,GRAS)。因为拥有生理生化特征清晰,遗传操作较为简单,分泌及表达能力强,培养发酵较为方便等优点,枯草芽孢杆菌还作为优良的底盘细胞,被改造为微生物细胞工厂,用于生产工业酶、维生素、功能糖、保健品及药物前体等目标产物,表现出了强大的工业生产应用能力[3-5]。
1 系统调控、代谢改造优化枯草芽孢杆菌底盘细胞枯草芽孢杆菌作为优秀的工业生产潜力菌株,被广泛地应用于生产工业、畜牧业酶、维生素、饲料添加剂、功能糖类产品、保健品等目标产物中。在以枯草芽孢杆菌为底盘细胞的代谢改造中,大多需要外源引入所需合成途径,所以需要系统地优化菌株自身全局代谢,协调菌株代谢网络与外源途径的平衡,减少途径副产物生成所造成的能量与资源的浪费。
系统生物学研究主要依托于多种组学(如基因组学、转录组学、蛋白质组学、代谢组学、相互作用组学等) 技术的实施与数据的整合,结合计算机模拟建模技术,构建预测模型,通过不断收集数据、构建信息网络,以完善模型。然后再以优化好的模型为工具进行生物体基本生理代谢功能的研究和改造的指导。旨在从全局上,系统性地对生物体进行研究及优化,以达到研究和应用的不同目的。合成生物学旨在针对唯一目的或产物,对自然界中不存在的途径或生产宿主进行创造,或对自然界中已经存在的生命系统进行改造,以满足合成和生产需求。主要结合了生物学、化学、热力学等多学科知识,对已发掘的生物元件进行一系列解析、组装及协调,创建生产宿主。结合系统生物学研究基础及理论指导,应用合成生物学手段,以枯草芽孢杆菌为底盘细胞,不断地加深对菌株的改造,以优化生产底盘细胞。该手段主要从对枯草芽孢杆菌各维度组学数据库的收集及标注,操控特定基因进行全局调控处理,多位点、多维度进行基因调控及动态调控,基因组精简,膜工程改造等方法方面有所体现。
1.1 枯草芽孢杆菌基因与代谢数据库枯草芽孢杆菌作为革兰氏阳性细菌的模式菌株,其基因组解析测序信息于1997年被发表在Nature杂志上[1],而后随着解析不断完善,其必需基因被详细解析注释[2],为枯草芽孢杆菌的菌株改造提供了良好的理论研究基础。在此基础上,多种数据库信息不断建立与完善,DBTBS[6] (http://dbtbs.hgc.jp/) 转录调控数据库,可查询枯草芽孢杆菌中转录调控信息;BioBrick Box[7]工具盒包含核糖体结合位点(Ribosome binding site,RBS)、免疫标签、报告基因和整合质粒等操作工具信息;SubtiWiki[8] (http://subtiwiki.uni-goettingen.de/) 基因数据库和MetaCyc[9] (https://metacyc.org/) 代谢数据库包含枯草芽孢杆菌中所有基因及基因相对应编码蛋白、基因表达、代谢网络及蛋白相互作用等信息。
1.2 全局调控枯草芽孢杆菌底盘细胞不同于在实验室中优良的培养条件,细菌在自然环境中能获得的碳、氮、磷和硫供给往往非常有限且不稳定,在此基础上,细菌自身进化出了一系列复杂且精确的调控系统,来调节自身营养代谢与生长,满足中央代谢途径(如糖酵解、戊糖磷酸途径、柠檬酸循环和酮戊二酸-谷氨酰胺-谷氨酸循环等),合成细胞微分子元件(如DNA、RNA、蛋白质、肽聚糖及磷脂等),以适应多变的环境[10]。
研究细菌自身代谢调控和系统生物学应用,挑战在于解析多种组织和调节网络的相互作用,其中最复杂的部分是在大量细胞元件之间的复杂的、非线性动态调控[11]。在枯草芽孢杆菌中有6个全局调控因子,可以调控枯草芽孢杆菌中基因的表达,其分别是:CcpA、CobY、TnrA、ComK、Spo0A和AbrB[12-13]。CcpA基因编码分解代谢控制蛋白A,分属于LacI/GalR转录调节因子家族,参与许多基因受制于葡萄糖调控下的表达,抑制分解代谢基因,且激活多余碳源的排出基因[12, 14-15]。CodY是枯草芽孢杆菌中的重要的多功能的转录多效抑制子,可以调控对支链氨基酸(BCAA) 限制响应的许多调节子(超过100个基因和操纵子)。在氮源限制的环境中,CodY也可以抑制遗传能力和调节产孢能力,使细胞适应环境[16-17]。CcpA可以激活合成乙酸、乳酸、乙偶姻的基因的表达,CodY也对乙酸、乳酸合成途径有激活作用,另外,CcpA和CodY可以抑制乙酸和乙酰苷的再利用酶[10]。TnrA属于MerR家族转录调节因子,参与全局氮调控,其可以响应氮源对多种基因进行激活和抑制调节。TnrA在细胞处于慢代谢氮源环境中被激活(如谷氨酸盐),通过结合TnrA box上调或下调多种基因的表达[18-19]。ComK是枯草芽孢杆菌中重要的转化转录因子,其在菌株感受态生长状态下能调节DNA结合、吸收和相关基因的转录[20]。Spo0A编码0期产孢蛋白A,是枯草芽孢杆菌在进入孢子形成时期主要的调节器,已经被证实在菌株发育早期直接、间接地影响超过500个基因[21],除此之外,Spo0A还在生物被膜的形成中起调节作用[22]。AbrB属于AbrB家族转录调节因子,是基因的转录多效性调节因子,可以在生长期和稳定期转化的过程中调节基因的表达,参与生物被膜形成的调节[23-24]。
在对枯草芽孢杆菌底盘细胞的代谢工程改造中,不仅仅单独优化目标代谢途径,还可以从菌株中心代谢着手,系统性地对菌株自身代谢与产物生产进行调节,以优化枯草芽孢杆菌底盘细胞的生产效率与生长平衡。通过敲除碳代谢阻遏的主要全局转录调控因子CcpA,并且针对敲除菌株抑制子进行进一步研究和分析,证明了基因表达的全局改变可以导致中心代谢的重组,从而回补了CcpA突变体导致的谷氨酸缺陷型,筛选出可以利用广泛碳源的枯草芽孢杆菌突变体[25]。2018年Cao等通过对全局调控因子CodY和CcpA进行随机突变并筛选,重新编辑了重组菌株中代谢调节网络,发现更高程度的抑制碳代谢和解除抑制氮代谢可以同样地提高报告基因β-半乳糖苷酶在枯草芽孢杆菌中的表达。进一步研究发现,报告蛋白的表达提升,始终和几个氮代谢操纵子的上调呈相关性关系,推测这一现象的产生是因为降低了在重组菌株中报告蛋白过表达而导致的代谢负担[26]。随后,又通过对全局调控因子CodY和CcpA的突变体进一步加以应用,提高了外源绿色荧光蛋白(Green fluorescent protein,GFP) 的产量,研究表明GFP蛋白表达的上调是因为在突变菌株中突变的全局调控因子的调节作用下,重组菌株略微地损失了生长速度,同时中央氮代谢重组导致的[27]。
通过调整底盘细胞中的全局调控因子,可以调节平衡菌株生长和生产之间的相互平衡,适当减轻因为异源生产而导致的代谢负担,从而优化底盘细胞。
1.3 多位点和多维调控、动态调控枯草芽孢杆菌底盘细胞近年来,在对枯草芽孢杆菌底盘细胞进行代谢改造时,对菌株内所需合成途径进行多位点、多维调控,或构件生物传感器,达成菌株自身动态调控等方法,逐渐成为研究的主流方法。区别于经典的直接对所需合成途径进行强化调控,对不需要的支路途经进行弱化、敲除等方法,构建多位点、多维共调控和动态调控的方法,可以更加有效地对菌株自身生长和生产情况进行平衡和协调,使得菌株生长和生产状态更优,从而保证底盘细胞可以持久、高效地进行所需产物的生产。
在对枯草芽孢杆菌进行多位点共调控时,优化和开发高效率的基因编辑工具是保证调控效果的根本方法。区别于应用双链断裂和同源修复的天然CRISPR-Cas9基因编辑方法,应用切口酶和同源定向修复(HDR) 的CRISPR-Cas9n对菌株造成的毒性和致死性程度更低,更适用于进行基因组大片段的改造和多位点的调控。2019年Liu等报道了应用优化后的CRISPR-Cas9n系统对枯草芽孢杆菌底盘细胞进行多位点的基因编辑,最终实现了双位点突变达91%和三位点突变达65%的编辑效率(图 1A)。并应用CRISPR-Cas9n系统对枯草芽孢杆菌中核黄素生产途径基因ribB、ribA、ribH进行RBS库的筛选,以三位点同时调节优化底盘细胞中核黄素的产量,经过一系列筛选和验证,对比原始菌株,核黄素的产量提升了59%,其生产中转化率达到了1.39 g/L和17.16 mg/g葡萄糖[28]。同样依托CRISPR系统进行改造,Lu等在枯草芽孢杆菌中将dCas9蛋白融合RNA聚合酶α和ω亚基,分别确定了以此设计为基础的CRISPRa (激活) 和CRISPRi (干扰) 的最适靶向窗口,通过特异性gRNA的指引,可以同时对不同的基因进行激活和抑制操作。并且在CRISPR-ω的应用上进行进一步扩充,创建了新的CRISPR辅助的寡核苷酸退火依赖的启动子筛选策略(Oligonucleotide annealing based promoter shuffling,OAPS),应用该策略可以在枯草芽孢杆菌中更有效地进行高效启动子文库的建立和启动子的筛选。文章报道,通过上述OAPS和dCas9-ω依赖的建库筛选方法,实现了在枯草芽孢杆菌中同时进行目标基因转录过程中启动子的筛选与调节,翻译修饰中分子伴侣的辅助和蛋白成熟后蛋白酶降解这3个阶段的精确调控,实现了目标蛋白表达在枯草芽孢杆菌中的多维、多层次精确调控(图 1C),并应用该种方法,精确调控在枯草芽孢杆菌中淀粉酶的表达,通过优化,对比简单用P43启动子启动表达,淀粉酶产量在枯草芽孢杆菌中提升了260倍[29]。
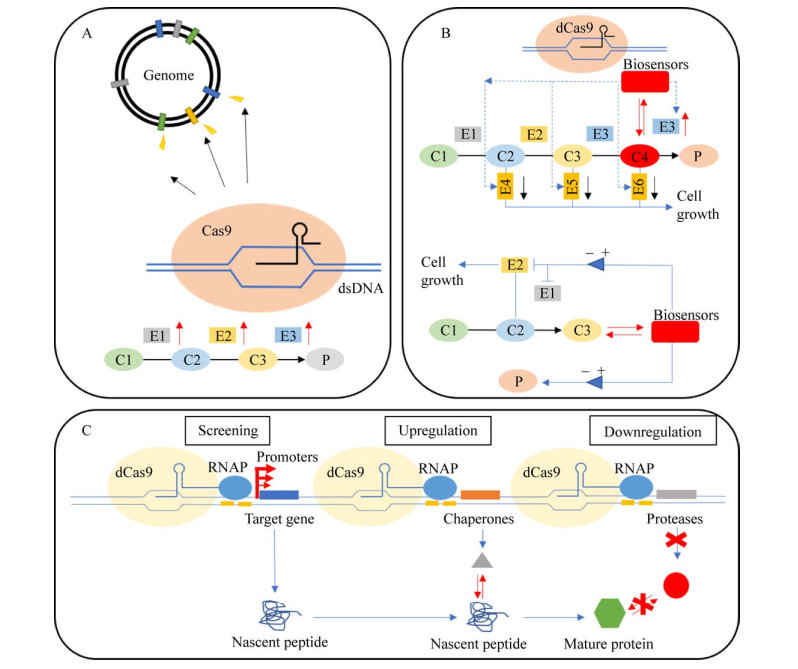
|
| 图 1 多位点、多维调控和动态调控枯草芽孢杆菌底盘细胞[28-31] Fig. 1 Multi-sites, multi-dimensional and dynamic regulation B. subtilis chassis cell[28-31]. (A) Multi-genes, multi-sites regulation in synthetic pathway by application optimized CRISPR-Cas system. By applying the optimized CRISPR-Cas9 gene editing system, the multi-sites gene editing in genome can be carry out. Up- and down-regulation the different genes in vivo, can regulating the chassis cells in multi-genes, multi reaction steps in catalyzing pathway, and optimizing the chassis cells roundly. (B) Establish the dynamic regulation in B. subtilis by using biosensors. By applying the biosensor aimed in micromolecular, or combining the biosensor and CRISPR-dCas9 gene editing system, regulating the downstream gene, can dynamically regulate the carbon flow in vivo and balance the grow and production in cell. (C) Multi-dimensional regulation by using CRISPR-dCas9 system. The optimizing the CRISPR-dCas9 gene editing system were used in screening the promoter library in protein expressing, upregulation the chaperone in protein folding of nascent peptide, and inhibiting and abolishing the protease expressing. Enhancing the protein producing by upregulation and protection in transcription, folding and modification and protein maturity. Abbreviation: C: compound; E: enzyme; P: production. |
| |
除了上述应用优化基因编辑方法进行多位点、多维调控枯草芽孢杆菌中基因表达之外,通过设计菌株内生物传感装置,进行动态调控的方法也逐渐在枯草芽孢杆菌的底盘细胞改造中被应用(图 1B)。近期Wu等发表的结果报道,将关键代谢物的生物传感器和CRISPRi结合使用,创建的自主双调节系统可以在枯草芽孢杆菌中进行调节应用,且有效地提升了N-乙酰氨基葡萄糖的生产。文章通过解析枯草芽孢杆菌中氨糖代谢途径,并从中筛选出六磷酸氨基葡萄糖(GlcN6P) 的生物传感蛋白GamR以应用,通过GamR蛋白响应细胞内GlcN6P的浓度,偶联CRISPRi干扰系统,设计反馈环调节系统。当菌株内GlcN6P达到响应浓度的时候,通过自主双调节系统中生物传感器进行响应,且将信号传递至CRISPRi系统,CRISPRi系统同时抑制N-乙酰氨基葡萄糖生产中的对中间代谢物进行消耗的zwf、pfkA、glmM基因表达,降低了支路途经对代谢流的消耗,同时增加了GlcN6P到N-乙酰氨基葡萄糖的合成代谢流,形成反馈环调节,有效增加了N-乙酰氨基葡萄糖的产量[30]。Xu等通过在枯草芽孢杆菌中引入并优化大肠杆菌丙酮酸响应蛋白PdhR,并结合反义转录的抑制机制,设计了枯草芽孢杆菌中生产葡萄糖二酸的动态调节机制。通过PdhR响应细胞内丙酮酸积累量动态切换代谢流走向,当丙酮酸积累量达到阈值时,丙酮酸激活的基因通路用于上调葡萄糖二酸的合成途径,反义转录依赖的丙酮酸抑制基因通路用于下调竞争途径(糖酵解途径、戊糖磷酸途径等);当丙酮酸浓度下降到一定浓度时,反馈调节环会激活糖酵解途径和戊糖磷酸途径,且抑制葡萄糖二酸合成途径,从而促进菌体生长。上述动态调控及结合敲除副产物合成途径,使得底盘细胞自身生长和目标产物生产达到动态平衡状态,大幅提升了目标产物葡萄糖二酸的产量(207 mg/L提升至802 mg/L)[31]。
通过上述多位点、多维和动态调控的方式在底盘细胞枯草芽孢杆菌中设计调节机制,可以更全面地对所需合成途径进行优化,并非一味的追求“最强”调节强度,而是进行更为精确、更为合适的调节强度优化,以达到底盘细胞的最优生产。
1.4 枯草芽孢杆菌基因组精简及优化为了达到最大的生产效率,优化获得最优的底盘细胞,对模式菌株基因组的大规模缩减是一个良好的优化策略。对菌株基因组进行精简和优化旨在不大幅度影响菌株生长状态的情况下,敲除非必需冗杂基因,维持菌株自身的生长,且使得目标产物生产最大化。
在微生物细胞工厂的构建中,常会因为引入的外源合成途径进入菌株自身合成途径,或通过中间产物和自身合成途径有交集,而导致改造菌株无法合成目标产物,或者因为底盘细胞对异源引入酶合成率较低,而造成了产量较低。基因组的精简和优化,不仅有利于对菌株自身代谢生长必需基因的探究,也是一个优良的菌株改造策略,有利于针对目标工业产品生产的合理设计和紧密调控。改造过程中可以通过敲除异源合成途径和菌株自身代谢相偶联步骤中的非必需基因,以分离菌株的生长和生产,提高底盘细胞合成效率。
对微生物基因组的精简策略,目前可以分为两种方向:自下向上的基因组精简和自上向下的基因组精简[32]。前者主要是应用一系列组装技术,将组成细胞所需的基本模块(如编码蛋白必需基因) 进行组装,以构成维持细胞生命及生存活力的最小生物单位,如2010年Gibson等设计组装合成了1.05 Mbp的Mycoplasma mycoides JCVI-syn1.0合成基因组,并成功使合成的宿主细胞拥有理想的表型和自复制能力[33],2014年首次报道了完整的设计和合成的真核生物基因组——酵母基因组SynⅢ[34]。自下向上地对基因组进行组装,受制于对底盘细胞自身遗传代谢研究的局限,操作方法复杂,工作量大等特点,目前还不适用于针对工业应用菌株的基因组缩减及改造。在工业生产菌株底盘细胞的改造中,目前常应用的是自上向下的基因组精简策略,主要应用删除基因组中大量的非必需基因部分,来简化现有细胞,从而达到按照目标产物的生产方向,来进行底盘细胞基因组的缩减作用。2002–2003年,首次报道了针对枯草芽孢杆菌基因组的缩减工作,敲除了原细胞中原噬菌体相关基因,敲除了332个非必需基因,缩减了7.7%的基因组,并未明显造成菌株生长缺陷,且中心碳代谢流和亲本菌株基本相同[35]。随后,在对枯草芽孢杆菌基因组必需基因的不断研究报道之下,删除了874 kbp (20.7%) 的枯草芽孢杆菌改造菌株MGB874,虽然对比野生型菌株生长速率略有下降,但是却能有效提升外源质粒表达的纤维素酶和蛋白酶的胞外产量[36]。随后,在删除了36%的基因组序列之后,构建的最小基因组枯草芽孢杆菌在丰富培养基中仍能以与野生型菌株相比几乎不变的生长状况进行生长,转录组数据证明,删减基因组后的枯草芽孢杆菌可以自身调节必需和非必需基因的转录效率,以达到生长的稳定,证明了大量删减底盘细胞基因组是一个良好的菌株优化策略[37]。应用减少底盘细胞基因组序列以优化枯草芽孢杆菌细胞,是以针对枯草芽孢杆菌自身生长代谢所需要的必需基因的研究为基础,在此基础上对菌株中可能存在的支路基因及非必需基因进行削减,2016年Daniel等总结了枯草芽孢杆菌生长所必需的最小代谢系统[38],近年来,有不少研究针对应用CRISPR基因编辑技术对枯草芽孢杆菌基因组精简及优化的报道,逐渐简化了基因组精简编辑的操作困难程度[39-40]。目前,枯草芽孢杆菌中已经分析获得的必需基因可以从SubtiWiki数据库进行查询[8]。
1.5 膜工程改造枯草芽孢杆菌底盘细胞在以枯草芽孢杆菌为底盘细胞生产蛋白质产物或其他化合物时,根据不同种类的产物对底盘细胞的细胞膜进行改造和优化,可以提高目标产物的生产效率。Westbrook等报道了在枯草芽孢杆菌中通过外源引入链球菌来源的透明质酸合成酶,并依次通过调整枯草芽孢杆菌内源基因优化细胞膜双磷脂酰甘油含量,调整细胞分裂抑制蛋白FtsZ影响细胞分裂形态,及移除细胞膜上磷脂酰乙醇胺和中性糖脂等措施,大幅优化了枯草芽孢杆菌中透明质酸的产量[41]。细胞内的功能性膜微室(FMMs) 可以给酶簇提供好的附着与催化平台,实验证明,在枯草芽孢杆菌中操作合成FMMs,可以提高细胞膜的有序性;将两个异源的N-乙酰葡萄糖的合成基因组装于FMMs上,对比未组装酶的对照菌株,枯草芽孢杆菌中异源合成N-乙酰葡萄糖的合成量提高了3倍,并且FMMs可以促进在细菌生长平稳期保证菌体活性,降低细菌生长后期的细胞裂解情况[42]。通过建库分析得知,枯草芽孢杆菌细胞表面阴离子膜磷脂(磷脂酰甘油和心磷脂) 的增加可以大幅提升枯草芽孢杆菌中异源表达α-淀粉酶的分泌,证明异源表达蛋白的等电点和细胞膜的磷脂层都在蛋白的分泌中起着重要的作用,这两个因素可以影响蛋白的分泌情况,在合成生物学改造中可以根据需要由此对异源蛋白产物的表达和分泌情况进行调整[43]。
2 枯草芽孢杆菌中的蛋白表达及分泌调控相较于大肠杆菌为代表的革兰氏阴性细菌来说,枯草芽孢杆菌的单层膜结构使得异源蛋白的分泌更加容易,又因为枯草芽孢杆菌的自身蛋白质表达和分泌系统研究资料丰富且较为全面,所以枯草芽孢杆菌被广泛地应用于异源蛋白质类目标产物的表达中。针对枯草芽孢杆菌对蛋白质类目标产物生产优化的方向和策略也逐渐被补充着。目前以枯草芽孢杆菌为底盘细胞,进行蛋白质的表达分泌优化主要在以下几个方面进行开展:表达过程中的启动子筛选与核糖体结合位点(RBS) 调控;蛋白分泌过程中的信号肽筛选与分泌系统的改造;枯草芽孢杆菌中新型非诱导剂表达系统的优化等。
枯草芽孢杆菌中可应用启动子的鉴定、设计改造及分类相对成熟且稳定。其中启动子根据其应用形式分为诱导型启动子和组成型启动子,其中诱导型启动子又有自诱导型、条件诱导型和诱导剂诱导型的区分及不同应用,也可根据启动子强度的不同对其进行分类[44]。近期有文献发表,通过对比4种枯草芽孢杆菌中生长时期依赖的启动子分类方式,对应用和筛选得到的启动子进行指数生长期、对数生长中期和早期稳定期、后对数期和稳定期,及稳定期启动子的分类,根据结果可对所需表达蛋白的时期进行调控[45]。
在对枯草芽孢杆菌底盘细胞的设计优化中,直接进行启动子的筛选和改造,可以对异源蛋白在枯草芽孢杆菌中的表达进行精确调控,优化所需异源蛋白产物在枯草芽孢杆菌中的表达。
2.1 枯草芽孢杆菌中无化学诱导剂表达系统在以枯草芽孢杆菌为底盘细胞的蛋白质表达系统中,一些不依赖化学诱导剂的表达系统被研究及应用[3]。依赖于枯草芽孢杆菌自身冷诱导的去饱和酶基因des进行的低温诱导表达体系,被证实可以在以枯草芽孢杆菌为底盘的外源基因表达中加以应用,以降低温度来诱导目标蛋白的表达,且有利于优化“难表达”蛋白质的表达[46]。枯草芽孢杆菌内源启动子PbltD、PydaD、PgerBC和PylnF也被报道属于低温诱导的启动子[47]。另外,依赖于pH诱导的启动子如PphrE、PcsbA、PyknW、PyqeZ、PywkA、PythP、PlytE、PptsI和PopuE显示可以在中性或碱性环境下进行目标蛋白的诱导表达[47]。将枯草芽孢杆菌自身群感调节基因srfA的启动子进行改造优化,构建了依赖于细胞密度的诱导表达系统,可以在枯草芽孢杆菌中进行无诱导剂诱导表达,应用氨基肽酶进行表达测试,证明其表达强度随着细胞密度的增加而增加,实现自诱导表达[48]。
2.2 枯草芽孢杆菌中信号肽的筛选与应用不同于以大肠杆菌为代表的革兰氏阴性细菌,枯草芽孢杆菌只有一层细胞膜,所以具有更优秀的蛋白分泌潜能。对异源蛋白直接进行细胞外的分泌优化,可以大幅减少工业应用中对于所需蛋白产物的纯化工作,简化产物分离步骤,极大程度地降低了生产成本,减弱了操作繁杂程度。利用融合表达枯草芽孢杆菌中蛋白质分泌信号肽,可引导目标蛋白进行细胞外分泌。枯草芽孢杆菌中分泌途径主要根据其应用分泌元件不同、翻译转运偶联程度不同,及切割信号肽酶不同,可大致分为Sec依赖的蛋白质分泌途径(经典分泌途径)、Tat分泌途径、ABC转运元件依赖的分泌途径和非经典分泌途径[49-50]。根据不同需求,可对异源表达蛋白产物进行信号肽的预测、筛选和改造[51-54],以优化目标蛋白的表达和生产,常用的信号肽预测网站有SignalP和LipoP等。
2.3 优化枯草芽孢杆菌中蛋白质分泌系统除了上述通过对信号肽筛选和改造,以优化目标蛋白产物的胞外分泌之外,对宿主细胞自身蛋白分泌系统的改造也同样可以提升蛋白质的分泌,优化目标蛋白的生产。枯草芽孢杆菌中主要的蛋白质分泌途径如Sec依赖的蛋白分泌途径[55-56]、Tat依赖的分泌途径[57-58]及ABC (ATP结合盒) 转运途径[59]的元件结构和分泌过程的解析已较为清晰,在此基础上,对蛋白质分泌系统中转运元件进行一定的过表达或改造,可以在一定程度上提高异源表达蛋白的分泌。研究表明,通过筛选优化启动子、信号肽,且共表达Sec蛋白分泌途径的分泌元件,可以明显地增加枯草芽孢杆菌中的脂肪酶分泌产量[60]。通过对枯草芽孢杆菌中SecA的序列进行分析,并删除其C端结构域,可以增加碱性纤维素酶、人源干扰素α在枯草芽孢杆菌中的大量分泌表达[61]。
3 枯草芽孢杆菌的遗传操作技术在对枯草芽孢杆菌作为底盘细胞改造中,除了需要对宿主菌株的生理代谢特点及调控有深入的研究,构建具有操作便捷、成功率高、技术成熟度高等特点的菌株遗传操作技术,也可大幅推动枯草芽孢杆菌底盘细胞的优化进程。其中遗传操作方法包括经典的基因交换方法、无标记重组技术、依赖于Cas9蛋白的CRISPR技术及在CRISPR/Cas系统上进化改进而来的碱基编辑器技术等。另外,增加菌株的转化效率,也是底盘细胞改造效率的良好保证。
3.1 枯草芽孢杆菌中基因交换及标记筛选基因交换及标记筛选是枯草芽孢杆菌中常用的遗传操作技术,反向选择盒通常由位于靶染色体基因座的两个同源区域,一个抗性基因,一个在诱导型启动子控制下的毒素基因以及两个直接重复的序列组成[62]。Jeong等构建了一个枯草芽孢杆菌基因工程的基因环作为可逆选择标记系统。该系统包含两个阻遏启动子(枯草芽孢杆菌xylA (Pxyl) 和spac (Pspac))、两个阻遏基因(lacI和xylR)和一个报告基因(氯霉素抗性基因(cat))[63]。Pxyl-lacI被整合到枯草芽孢杆菌基因组中,该基因组包含所需突变的靶基因。木糖作为抑制XylR的诱导剂,从而诱导lacI表达,而LacI抑制Pspac启动子,并且细胞对氯霉素敏感。因此,为了在氯霉素存在条件下存活,细胞必须通过野生型和突变靶基因之间的重组来删除Pxyl-lacI。在没有氯霉素的情况下,容易除去残留的辅助质粒。因此,该系统不会在染色体上保留任何外来DNA片段,这使其可用于食品级生物的基因组工程中[63]。除了上述的cat [63]基因外,还有upp[64]、blaI [65]、mazF [66]、ysbC [67]、hewl [62]等基因应用于枯草芽孢菌杆菌的可逆选择标记系统。
3.2 枯草芽孢杆菌中特定位点无标记重组位点特异性重组系统催化两个特异性识别位点之间的重组,从而产生所需的DNA整合、缺失或倒置[68]。目前在枯草芽孢杆菌上运用较多的是Cre/loxP系统和Xer/dif系统。Cre重组酶可催化两个loxP位点之间的特异性重组,而无需任何宿主辅因子或辅助蛋白[69]。Yan等将IPTG诱导的cre表达盒整合到枯草芽孢杆菌染色体中,以在2 d内完成突变过程[69]。因此,可以在枯草芽孢杆菌中有效地进行基因组规模的突变。对于Xer/dif系统,Xer重组酶将两个直接重复的dif位点重组为一个位点,从而切除了抗生素抗性盒或其他选择性标记基因[70]。
3.3 枯草芽孢杆菌中CRISPR-Cas系统及碱基编辑器簇状规则间隔的短回文重复序列(CRISPR)- Cas系统由于其可以切割特定的DNA序列而广泛用于介导各种生物体和细胞系中的基因组编辑[71-72]。CRISPR/Cas9蛋白-RNA复合物通过指导碱基与引导RNA (Single-guide RNA,sgRNA) 配对而定位于目标DNA序列,并在RNA靶向的位点自然产生DNA双链断裂(DNA double-strand break,DSB)。DSB可以刺激容易出错的非同源末端连接(Non-homologous end joining,NHEJ)或者可以在存在同源DNA模板的情况下在DSB位置进行同源直接修复(Homology directed repair,HDR)[73]。Westbrook等开发了一种用于枯草芽孢杆菌中整合基因工程的CRISPR-Cas9工具盒,该工具盒简化了CRISPR-Cas9介导的基因组编辑和转录干扰所需的遗传元件的构建,同时也避免了对潜在不稳定的多拷贝质粒的依赖以及随后的质粒稳定化[74]。Zhang等为工业生产菌株枯草芽孢杆菌ATCC 6051a开发了一种CRISPR/Cas9系统。该系统被用于破坏枯草芽孢杆菌ATCC 6051a基因组中的5个基因,所得突变体(BS5) 在发酵过程中形成的泡沫较少,显示出更强的抗孢子形成能力,并分泌了2.5倍的β-CGT酶[75]。然而,CRISPR-Cas9技术具有脱靶效应,目前已有一些报道来解决这一问题。例如,可以设计两个sgRNA来引导Cas9蛋白在相邻位点切割基因组,可以使用Cas9裂解酶的突变形式,并且该引导序列的最后12 bp具有高度特异性[76]。
碱基编辑器依赖于CRISPR/Cas9系统,也是一种基因组的编辑工具,且近年来逐渐被研究和开发。区别于传统的CRISPR/Cas9编辑系统,碱基编辑器不依赖于DNA双链断裂和同源重组,且可以更快地实现基因的失活和定点突变。通过应用胞嘧啶脱羧酶AID和CRISPR/dCas9,Yu等首次在枯草芽孢杆菌中构建了CRISPR-dCas9-AID碱基编辑系统。该碱基编辑系统应用单质粒同时表达dCas9-AID和sgRNA,在枯草芽孢杆菌中有5 nt的编辑窗口,与之前报道的在其他宿主细胞中建立的碱基编辑系统相同,可以实现C-T的碱基编辑来提前引入终止密码子,从而实现目标基因的失活。CRISPR-dCas9-AID系统可以实现在枯草芽孢杆菌中三位点和四位点同时编辑,其编辑效率分别达到100%和50%,可只通过简单两轮碱基编辑即实现枯草芽孢杆菌中8个蛋白酶的失活,相较传统的基因敲除技术在编辑效率和时间上有了极大的提升[77]。
3.4 遗传操作提升枯草芽孢杆菌转化率ComK是枯草芽孢杆菌中重要的转化转录因子(Competence transcription factor,CTF),调控着细胞进入感受态阶段的许多基因的表达和DNA的吸收。ComK在体内参与的代谢网络复杂,在ComK蛋白表达量高时,许多细胞进入转化生长阶段(K-state),在此阶段超过100个基因表达提升,其中DNA吸收的重要基因得到了强表达提升[78]。Zhang等利用上述调节途径,在枯草芽孢杆菌中利用木糖诱导启动子,过表达转化调节因子ComK蛋白,以增加枯草芽孢杆菌的转化效率,随着底盘细胞中comK基因的过表达,实现了在枯草芽孢杆菌中的较高转化效率(约107转化子/μg质粒和约104转化子/μg DNA片段)[79]。
4 以枯草芽孢杆菌为底盘细胞进行合成生产枯草芽孢杆菌作为革兰氏阳性菌的模式生物,也是重要的工业应用菌株,以其为底盘细胞合成小分子化合物、酶制剂等蛋白质产物和保健品、药品前体物质的研究不断被报道,菌株改造技术也不断优化以提高目标产物的产量。以枯草芽孢杆菌为底盘细胞进行合成的小分子化合物或酶制剂主要以核黄素、表面活性素、乙偶姻、植酸酶、木聚糖酶、L-天冬酰胺酶、软骨素、N-乙酰葡萄糖胺等为代表。
4.1 枯草芽孢杆菌合成小分子化合物核黄素(维生素B2) 是一种被应用于药物和动物饲料添加的重要的维生素品种。工业合成核黄素可以应用化学或生物手段,1999年,Perkins等应用基因工程技术以枯草芽孢杆菌为底盘细胞,构建了核黄素高产菌株,其发酵56 h核黄素产量可以达到15 g/L[80]。文献报道通过过表达葡萄糖-6-磷酸脱氢酶(GAPDH) 来改变戊糖磷酸途径的碳代谢流,可以增加核酮糖-5-磷酸(Ru5P) 的胞内积累,Ru5P作为核黄素的前体,增强其供应可以增加核黄素产量,使其增产25%[81]。通过基因编辑手段改造生产菌株,解除嘌呤途径[82]和糖酵解途径[83]的限制,都被证实可以增加枯草芽孢杆菌中核黄素的产量。另外,近期有文献报道,通过识别且敲除核黄素合成中转录核糖开关中的不影响其他二级、三级结构,唯一参与转录终止的关键核苷酸,有效地解除了核黄素合成菌株中嘌呤途径限制,使生产菌株的生物量增加了53%,进而增加了核黄素的产量[84]。
表面活性素(Surfactin) 是一种由脂肪酸和环状七肽构成的脂蛋白化合物,是一种强效的表面活性剂。表面活性素同时具有抗微生物、抗病毒和抗肿瘤的活性,在工业应用领域有很强的应用前景。在微生物中,表面活性素作为一种信号分子,参与细胞自身群感调节。枯草芽孢杆菌B. subtilis 168菌株天然不生产表面活性素,通过对菌株遗传改造,包括敲除潜在竞争途径,过表达表面活性素外排和抗体相关蛋白,理性改造支路脂肪酸合成途径增强前体供应,调整群感系统以增强srfA基因转录等手段,在枯草芽孢杆菌中建立了表面活性素合成途径,产量达到12.8 g/L[85]。
乙偶姻,又名3-羟基-2-丁酮,在微生物体内参与NADH/NAD+的调节反应,是食物加工产业中广泛使用的香精材料,也可作为前体参与合成许多重要化合物。在混合酸-丁二醇发酵时,枯草芽孢杆菌可以将丙酮酸转化为乳酸、乙醇、醋酸、乙偶姻和2, 3-丁二醇。为了增强枯草芽孢杆菌中乙偶姻的产量,生成2, 3-丁二醇、乳酸、乙醇等副产物的碳代谢流必须被削弱,上述副产物的合成途径被证实是NADH依赖的。通过敲除编码将乙偶姻转化为2, 3-丁二醇的乙偶姻还原酶编码基因bdhA,且筛选并引入NADH氧化酶编码基因,可以大幅地降低枯草芽孢杆菌底盘细胞中2, 3-丁二醇、乳酸、乙醇的产量,并且大幅提高乙偶姻的产量,使得枯草芽孢杆菌中乙偶姻的产量达到56.7 g/L[86]。
4.2 枯草芽孢杆菌合成酶制剂木聚糖酶是降解木聚糖的重要催化酶,可以协同其他木聚糖裂解酶,一起催化木聚糖降解生产艺术纸、木糖、木糖醇、生物乙醇等,也有潜力应用于降解纸浆造纸工业中污水废物的处理,还可应用于低聚木糖的生产,后者可用于功能性食品和医药产品的生产[87]。通过应用SigB依赖的通用压力调节基因的Pohr启动子构建枯草芽孢杆菌中木聚糖酶自诱导表达系统,免除了如IPTG诱导剂的添加诱导,实现了木聚糖酶在重组菌株中达到38 U/mL的酶活性[88]。
L-天冬酰胺酶(ASN) 可以催化水解L-天冬酰胺分解成天冬氨酸和氨气。因为可以抵抗高的L-天冬酰胺活性,ASN拥有较好的临床应用前景,特别是应用于治疗和诊断急性淋巴细胞白血病。另外,ASN还应用于食品产业,如通过移除天冬酰胺来生产无丙烯酰胺食物等。通过筛选和突变改造启动子、筛选信号肽、对蛋白编码序列进行截短等手段构建并优化ASN表达重组枯草芽孢杆菌菌株,另外通过优化发酵条件,其3 L发酵罐产量可以达到407.6 U/mL (2.5 g/L)[89]。
4.3 枯草芽孢杆菌生产保健品、药品前体软骨素和肝聚糖,是重要的多糖—硫酸软骨素和硫酸肝素合成的重要前体。在重组枯草芽孢杆菌中通过整合表达基因于基因组中、过表达枯草芽孢杆菌内源基因tuaD、补料发酵等优化手段,使得重组菌株中软骨素和肝聚糖的产量达到5.22 g/L和5.82 g/L (在3 L发酵罐中)[90]。N-乙酰葡萄糖胺(GlcNAc) 和葡萄糖胺(GlcN) 是葡萄糖胺聚糖生产合成的前体,长期被添加于药物和营养品中,以治疗骨关节和维持软骨和关节的健康。通过应用双启动子系统,敲除ldh、pta基因以阻断乳酸和醋酸合成,弱化糖酵解和肽聚糖合成模块等手段,优化重组枯草芽孢杆菌中N-乙酰葡萄糖胺的产量明显提高,在3 L发酵罐中达到了31.65 g/L[91]。通过重构枯草芽孢杆菌中心碳、氧代谢途径,降低乙酰CoA代谢流和消除副产物的生成,引入NAD(P) 不依赖的代谢途径以重启氧化还原代谢以缩小能量损失,优化N-乙酰葡萄糖胺的摇瓶产量达到24.5 g/L,产量达到0.468 g/g (N-乙酰葡萄糖胺/葡萄糖),达到了理论产率的98%[92]。
5 总结与展望枯草芽孢杆菌作为革兰氏阳性细菌的模式菌株,多年来,科学家们对其菌株的代谢研究、遗传操作手段开发和培养条件摸索等成果不断报道与积累,为其应用于实际工业生产小分子化合物或酶制剂提供了很好的理论研究基础。又因为其菌株的安全性较高,枯草芽孢杆菌逐渐变成系统与合成生物技术中研究的主要菌株。以其为底盘细胞可实现如维生素、乙偶姻等小分子化合物的生产,也可进行如植酸酶、木聚糖酶等工业酶制剂,N-乙酰葡萄糖胺等药物添加前体小分子的合成与生产。
虽然以枯草芽孢杆菌为底盘细胞进行目标产物生产的优化方法得到了一定系统性地开发与发展(图 2),但是在未来的发展与研究中,重点问题的针对性研究和新技术的结合或可更进一步优化枯草芽孢杆菌底盘细胞,推动其成为工业菌种中有力的竞争者,包括:①依赖于基因组、转录组等代谢网络解析的不断完善,生化合成新途径的设计和新酶的挖掘逐渐成为合成生物学研究的重点。不再依赖传统解析合成途径,而是选用更精简、热力学倾向性更高的新挖掘途径来进行目标产物的生产合成,辅加以筛选催化效率更高的途径酶进行应用,实现产物生产和转化率更优的优化。②虽然目前在枯草芽孢杆菌中已经可以初步实现多位点同时基因编辑及调控,但是针对较长且复杂的合成途径,基因编辑工具仍需继续开发与优化,实现多位点基因编辑效率的提高,同时结合高通量基因克隆组装与高通量筛选技术,从而实现自动化、高通量的底盘细胞改造流程,以加速底盘细胞的进化效率,得到更优的生产菌株。③基于胞内传感器的自调节系统仍然将会是枯草芽孢杆菌底盘细胞改造中的一项关键技术。旨在自主、动态调节底盘细胞自身生长和对目标产物的生产之间的平衡,一些关键中间产物的生物传感相应蛋白被不断地应用于菌株代谢动态调节中。基于实际工业生产发酵成本和操作的优化需求,摒除人工进行发酵条件的转化与调控,以菌株自身感应动态调节来取代发酵中复杂的调控操作,是一个良好的发展趋势与方向。未来在关键产物生产底盘细胞的构建中,应用几种生物传感器进行自身动态调节网络的调节,结合新兴基因编辑激活、干扰及失活方法,构建更为复杂且精细的自身动态调控,优化底盘细胞的产量及转化率,优化工业生产调节步骤。④除了菌株改造等生化技术,通过应用新兴人工智能、深度学习、机器学习等智能技术进行发酵工程的优化,构建智能发酵技术及设备,将有可能更科技、高效地对发酵工艺进行控制,在底盘细胞的优化基础上实现数量级上的产量优化与提升。
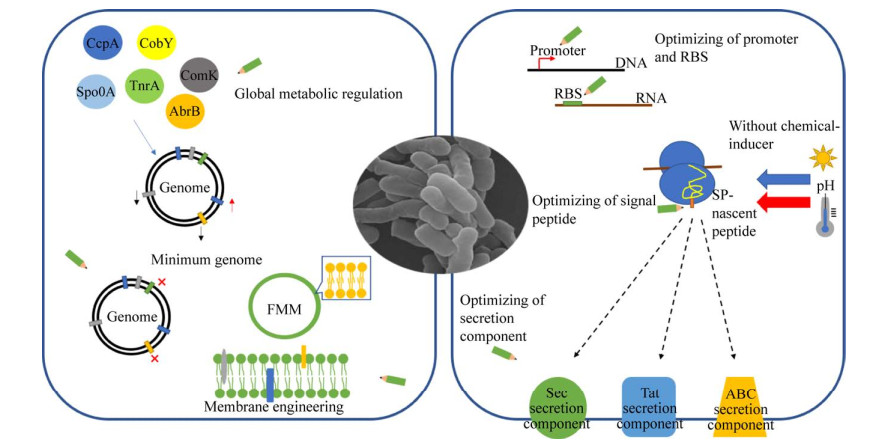
|
| 图 2 优化枯草芽孢杆菌作为底盘细胞的改造策略 Fig. 2 Modification strategy for optimizing the B. subtilis as chassis cell. |
| |
| [1] |
Kunst F, Ogasawara N, Moszer I, et al. The complete genome sequence of the gram-positive bacterium Bacillus subtilis. Nature, 1997, 390(6657): 249-256. DOI:10.1038/36786
|
| [2] |
Kobayashi K, Ehrlich SD, Albertini A, et al. Essential Bacillus subtilis genes. Proc Natl Acad Sci USA, 2003, 100(8): 4678-4683. DOI:10.1073/pnas.0730515100
|
| [3] |
Gu Y, Xu XH, Wu YK, et al. Advances and prospects of Bacillus subtilis cellular factories: From rational design to industrial applications. Metab Eng, 2018, 50: 109-121. DOI:10.1016/j.ymben.2018.05.006
|
| [4] |
Ÿztürk S, Ÿalik P, Ÿzdamar TH. Fed-batch biomolecule production by Bacillus subtilis: a state of the art review. Trends Biotechnol, 2016, 34(4): 329-345. DOI:10.1016/j.tibtech.2015.12.008
|
| [5] |
Zweers JC, Barák I, Becher D, et al. Towards the development of Bacillus subtilis as a cell factory for membrane proteins and protein complexes. Microb Cell Fact, 2008, 7: 10. DOI:10.1186/1475-2859-7-10
|
| [6] |
Sierro N, Makita Y, De Hoon M, et al. DBTBS: a database of transcriptional regulation in Bacillus subtilis containing upstream intergenic conservation information. Nucleic Acids Res, 2008, 36(Suppl.1): D93-D96.
|
| [7] |
Popp PF, Dotzler M, Radeck J, et al. The Bacillus BioBrick Box 2.0: expanding the genetic toolbox for the standardized work with Bacillus subtilis. Sci Rep, 2017, 7: 15058. DOI:10.1038/s41598-017-15107-z
|
| [8] |
Zhu BY, Stülke J. Subti Wiki in 2018: from genes and proteins to functional network annotation of the model organism Bacillus subtilis. Nucleic Acids Res, 2018, 46(D1): D743-D748. DOI:10.1093/nar/gkx908
|
| [9] |
Caspi R, Billington R, Fulcher CA, et al. The MetaCyc database of metabolic pathways and enzymes. Nucleic Acids Res, 2018, 46(D1): D633-D639. DOI:10.1093/nar/gkx935
|
| [10] |
Sonenshein AL. Control of key metabolic intersections in Bacillus subtilis. Nat Rev Microbiol, 2007, 5(12): 917-927. DOI:10.1038/nrmicro1772
|
| [11] |
Buescher JM, Liebermeister W, Jules M, et al. Global network reorganization during dynamic adaptations of Bacillus subtilis metabolism. Science, 2012, 335(6072): 1099-1103. DOI:10.1126/science.1206871
|
| [12] |
Moreno-Campuzano S, Janga SC, Pérez-Rueda E. Identification and analysis of DNA-binding transcription factors in Bacillus subtilis and other Firmicutes—a genomic approach. BMC Genomics, 2006, 7: 147. DOI:10.1186/1471-2164-7-147
|
| [13] |
Liu YF, Liu L, Li JH, et al. Synthetic biology toolbox and chassis development in Bacillus subtilis. Trends Biotechnol, 2019, 37(5): 548-562. DOI:10.1016/j.tibtech.2018.10.005
|
| [14] |
Grundy FJ, Waters DA, Allen SH, et al. Regulation of the Bacillus subtilis acetate kinase gene by CcpA. J Bacteriol, 1993, 175(22): 7348-7355. DOI:10.1128/JB.175.22.7348-7355.1993
|
| [15] |
Görke B, Stülke J. Carbon catabolite repression in bacteria: many ways to make the most out of nutrients. Nat Rev Microbiol, 2008, 6(8): 613-624. DOI:10.1038/nrmicro1932
|
| [16] |
Serror P, Sonenshein AL. CodY is required for nutritional repression of Bacillus subtilis genetic competence. J Bacteriol, 1996, 178(20): 5910-5915. DOI:10.1128/JB.178.20.5910-5915.1996
|
| [17] |
Ratnayake-Lecamwasam M, Serror P, Wong KW, et al. Bacillus subtilis CodY represses early-stationary- phase genes by sensing GTP levels. Genes Dev, 2001, 15(9): 1093-1103. DOI:10.1101/gad.874201
|
| [18] |
Belitsky BR, Wray LV Jr, Fisher SH, et al. Role of TnrA in nitrogen source-dependent repression of Bacillus subtilis glutamate synthase gene expression. J Bacteriol, 2000, 182(21): 5939-5947. DOI:10.1128/JB.182.21.5939-5947.2000
|
| [19] |
Yoshida KI, Yamaguchi H, Kinehara M, et al. Identification of additional TnrA-regulated genes of Bacillus subtilis associated with a TnrA box. Mol Microbiol, 2003, 49(1): 157-165. DOI:10.1046/j.1365-2958.2003.03567.x
|
| [20] |
Hamoen LW, Van Werkhoven AF, Bijlsma JJE, et al. The competence transcription factor of Bacillus subtilis recognizes short A/T-rich sequences arranged in a unique, flexible pattern along the DNA helix. Genes Dev, 1998, 12(10): 1539-1550. DOI:10.1101/gad.12.10.1539
|
| [21] |
Molle V, Fujita M, Jensen ST, et al. The Spo0A regulon of Bacillus subtilis. Mol Microbiol, 2003, 50(5): 1683-1701. DOI:10.1046/j.1365-2958.2003.03818.x
|
| [22] |
Vlamakis H, Chai YR, Beauregard P, et al. Sticking together: building a biofilm the Bacillus subtilis way. Nat Rev Microbiol, 2013, 11(3): 157-168. DOI:10.1038/nrmicro2960
|
| [23] |
Phillips ZE, Strauch MA. Bacillus subtilis sporulation and stationary phase gene expression. Cell Mol Life Sci, 2002, 59(3): 392-402. DOI:10.1007/s00018-002-8431-9
|
| [24] |
Hamon MA, Stanley NR, Britton RA, et al. Identification of AbrB-regulated genes involved in biofilm formation by Bacillus subtilis. Mol Microbiol, 2004, 52(3): 847-860. DOI:10.1111/j.1365-2958.2004.04023.x
|
| [25] |
Reuß DR, Rath H, Thürmer A, et al. Changes of DNA topology affect the global transcription landscape and allow rapid growth of a Bacillus subtilis mutant lacking carbon catabolite repression. Metab Eng, 2018, 45: 171-179. DOI:10.1016/j.ymben.2017.12.004
|
| [26] |
Cao HJ, Villatoro-Hernandez J, Weme RDO, et al. Boosting heterologous protein production yield by adjusting global nitrogen and carbon metabolic regulatory networks in Bacillus subtilis. Metab Eng, 2018, 49: 143-152. DOI:10.1016/j.ymben.2018.08.001
|
| [27] |
Cao HJ, Kuipers OP. Influence of global gene regulatory networks on single cell heterogeneity of green fluorescent protein production in Bacillus subtilis. Microb Cell Fact, 2018, 17(1): 134. DOI:10.1186/s12934-018-0985-9
|
| [28] |
Liu DY, Huang C, Guo JX, et al. Development and characterization of a CRISPR/Cas9n-based multiplex genome editing system for Bacillus subtilis. Biotechnol Biofuels, 2019, 12: 197. DOI:10.1186/s13068-019-1537-1
|
| [29] |
Lu ZH, Yang SH, Yuan X, et al. CRISPR-assisted multi-dimensional regulation for fine-tuning gene expression in Bacillus subtilis. Nucleic Acids Res, 2019, 47(7): e40. DOI:10.1093/nar/gkz072
|
| [30] |
Wu YK, Chen TC, Liu YF, et al. Design of a programmable biosensor-CRISPRi genetic circuits for dynamic and autonomous dual-control of metabolic flux in Bacillus subtilis. Nucleic Acids Res, 2020, 48(2): 996-1009. DOI:10.1093/nar/gkz1123
|
| [31] |
Xu XH, Li XL, Liu YF, et al. Pyruvate-responsive genetic circuits for dynamic control of central metabolism. Nat Chem Biol, 2020, 16(11): 1261-1268. DOI:10.1038/s41589-020-0637-3
|
| [32] |
Juhas M, Reuß DR, Zhu BY, et al. Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering. Microbiology, 2014, 160(11): 2341-2351. DOI:10.1099/mic.0.079376-0
|
| [33] |
Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome. Science, 2010, 329(5987): 52-56. DOI:10.1126/science.1190719
|
| [34] |
Annaluru N, Muller H, Mitchell LA, et al. Total synthesis of a functional designer eukaryotic chromosome. Science, 2014, 344(6179): 55-58. DOI:10.1126/science.1249252
|
| [35] |
Westers H, Dorenbos R, Van Dijl JM, et al. Genome engineering reveals large dispensable regions in Bacillus subtilis. Mol Biol Evol, 2003, 20(12): 2076-2090. DOI:10.1093/molbev/msg219
|
| [36] |
Morimoto T, Kadoya R, Endo K, et al. Enhanced recombinant protein productivity by genome reduction in Bacillus subtilis. DNA Res, 2008, 15(2): 73-81. DOI:10.1093/dnares/dsn002
|
| [37] |
Reuß DR, Altenbuchner J, Mäder U, et al. Large-scale reduction of the Bacillus subtilis genome: consequences for the transcriptional network, resource allocation, and metabolism. Genome Res, 2017, 27(2): 289-299. DOI:10.1101/gr.215293.116
|
| [38] |
Reuß DR, Commichau FM, Gundlach J, et al. The blueprint of a minimal cell: MiniBacillus. Microbiol Mol Biol Rev, 2016, 80(4): 955-987. DOI:10.1128/MMBR.00029-16
|
| [39] |
So Y, Park SY, Park EH, et al. A highly efficient CRISPR-Cas9-mediated large genomic deletion in Bacillus subtilis. Front Microbiol, 2017, 8: 1167. DOI:10.3389/fmicb.2017.01167
|
| [40] |
Lim H, Choi SK. Programmed gRNA removal system for CRISPR-Cas9-mediated multi-round genome editing in Bacillus subtilis. Front Microbiol, 2019, 10: 1140. DOI:10.3389/fmicb.2019.01140
|
| [41] |
Westbrook AW, Ren X, Moo-Young M, et al. Engineering of cell membrane to enhance heterologous production of hyaluronic acid in Bacillus subtilis. Biotechnol Bioeng, 2018, 115(1): 216-231. DOI:10.1002/bit.26459
|
| [42] |
Lv XQ, Zhang C, Cui SX, et al. Assembly of pathway enzymes by engineering functional membrane microdomain components for improved N-acetylglucosamine synthesis in Bacillus subtilis. Metabol Eng, 2020, 61: 96-105. DOI:10.1016/j.ymben.2020.05.011
|
| [43] |
Cao HJ, Van Heel AJ, Ahmed H, et al. Cell surface engineering of Bacillus subtilis improves production yields of heterologously expressed α-amylases. Microb Cell Fact, 2017, 16(1): 56. DOI:10.1186/s12934-017-0674-0
|
| [44] |
张大伟, 康倩. 枯草芽胞杆菌蛋白质表达分泌系统发展及展望. 微生物学杂志, 2019, 39(1): 1-10. Zhang DW, Kang Q. Development and prospect of protein expression and secretion systems in Bacillus subtilis. J Microbiol, 2019, 39(1): 1-10 (in Chinese). |
| [45] |
Kang XM, Cai X, Huang ZH, et al. Construction of a highly active secretory expression system in Bacillus subtilis of a recombinant amidase by promoter and signal peptide engineering. Int J Biol Macromol, 2020, 143: 833-841. DOI:10.1016/j.ijbiomac.2019.09.144
|
| [46] |
Welsch N, Homuth G, Schweder T. Stepwise optimization of a low-temperature Bacillus subtilis expression system for "difficult to express" proteins. Appl Microbiol Biotechnol, 2015, 99(15): 6363-6376. DOI:10.1007/s00253-015-6552-y
|
| [47] |
Yang S, Du GC, Chen J, et al. Characterization and application of endogenous phase-dependent promoters in Bacillus subtilis. Appl Microbiol Biotechnol, 2017, 101(10): 4151-4161. DOI:10.1007/s00253-017-8142-7
|
| [48] |
Guan CR, Cui WJ, Cheng JT, et al. Construction and development of an auto-regulatory gene expression system in Bacillus subtilis. Microb Cell Fact, 2015, 14: 150. DOI:10.1186/s12934-015-0341-2
|
| [49] |
Tjalsma H, Antelmann H, Jongbloed JDH, et al. Proteomics of protein secretion by Bacillus subtilis: separating the "secrets" of the secretome. Microbiol Mol Biol Rev, 2004, 68(2): 207-233. DOI:10.1128/MMBR.68.2.207-233.2004
|
| [50] |
Natale P, Brüser T, Driessen AJM. Sec- and Tat-mediated protein secretion across the bacterial cytoplasmic membrane—distinct translocases and mechanisms. Biochim Biophys Acta, 2008, 1778(9): 1735-1756. DOI:10.1016/j.bbamem.2007.07.015
|
| [51] |
Caspers M, Brockmeier U, Degering C, et al. Improvement of Sec-dependent secretion of a heterologous model protein in Bacillus subtilis by saturation mutagenesis of the N-domain of the AmyE signal peptide. Appl Microbiol Biotechnol, 2010, 86(6): 1877-1885. DOI:10.1007/s00253-009-2405-x
|
| [52] |
Kouwen TRHM, Nielsen AK, Denham EL, et al. Contributions of the pre- and pro-regions of a Staphylococcus hyicus lipase to secretion of a heterologous protein by Bacillus subtilis. Appl Environ Microbiol, 2010, 76(3): 659-669. DOI:10.1128/AEM.01671-09
|
| [53] |
Song YF, Nikoloff JM, Zhang DW. Improving protein production on the level of regulation of both expression and secretion pathways in Bacillus subtilis. J Microbiol Biotechnol, 2015, 25(7): 963-977. DOI:10.4014/jmb.1501.01028
|
| [54] |
Li DD, Fu G, Tu R, et al. High-efficiency expression and secretion of human FGF21 in Bacillus subtilis by intercalation of a mini-cistron cassette and combinatorial optimization of cell regulatory components. Microb Cell Fact, 2019, 18(1): 17. DOI:10.1186/s12934-019-1066-4
|
| [55] |
Tsirigotaki A, De Geyter J, Šoštaric' N, et al. Protein export through the bacterial Sec pathway. Nat Rev Microbiol, 2017, 15(1): 21-36. DOI:10.1038/nrmicro.2016.161
|
| [56] |
Akopian D, Shen K, Zhang X, et al. Signal recognition particle: an essential protein-targeting machine. Annu Rev Biochem, 2013, 82: 693-721. DOI:10.1146/annurev-biochem-072711-164732
|
| [57] |
Goosens VJ, Van Dijl JM. Twin-arginine protein translocation. Curr Top Microbiol Immunol, 2017, 404: 69-94.
|
| [58] |
Goosens VJ, Monteferrante CG, Van Dijl JM. The Tat system of Gram-positive bacteria. Biochim Biophys Acta, 2014, 1843(8): 1698-1706. DOI:10.1016/j.bbamcr.2013.10.008
|
| [59] |
Fu LL, Xu ZR, Li WF, et al. Protein secretion pathways in Bacillus subtilis: implication for optimization of heterologous protein secretion. Biotechnol Adv, 2007, 25(1): 1-12. DOI:10.1016/j.biotechadv.2006.08.002
|
| [60] |
Ma RJ, Wang YH, Liu L, et al. Production enhancement of the extracellular lipase LipA in Bacillus subtilis: effects of expression system and Sec pathway components. Protein Expr Purif, 2018, 142: 81-87. DOI:10.1016/j.pep.2017.09.011
|
| [61] |
Kakeshita H, Kageyama Y, Ara K, et al. Enhanced extracellular production of heterologous proteins in Bacillus subtilis by deleting the C-terminal region of the SecA secretory machinery. Mol Biotechnol, 2010, 46(3): 250-257. DOI:10.1007/s12033-010-9295-0
|
| [62] |
Wang Y, Weng J, Waseem R, et al. Bacillus subtilis genome editing using ssDNA with short homology regions. Nucleic Acids Res, 2012, 40(12): e91. DOI:10.1093/nar/gks248
|
| [63] |
Jeong DE, Park SH, Pan JG, et al. Genome engineering using a synthetic gene circuit in Bacillus subtilis. Nucleic Acids Res, 2015, 43(6): e42. DOI:10.1093/nar/gku1380
|
| [64] |
Fabret C, Ehrlich SD, Noirot P. A new mutation delivery system for genome-scale approaches in Bacillus subtilis. Mol Microbiol, 2002, 46(1): 25-36. DOI:10.1046/j.1365-2958.2002.03140.x
|
| [65] |
Brans A, Filée P, Chevigné A, et al. New integrative method to generate Bacillus subtilis recombinant strains free of selection markers. Appl Environ Microbiol, 2004, 70(12): 7241-7250. DOI:10.1128/AEM.70.12.7241-7250.2004
|
| [66] |
Morimoto T, Ara K, Ozaki K, et al. A new simple method to introduce marker-free deletions in the Bacillus subtilis genome. Genes Genet Syst, 2009, 84(4): 315-318. DOI:10.1266/ggs.84.315
|
| [67] |
Defoor E, Kryger MB, Martinussen J. The orotate transporter encoded by oroP from Lactococcus lactis is required for orotate utilization and has utility as a food-grade selectable marker. Microbiology (Reading), 2007, 153(11): 3645-3659. DOI:10.1099/mic.0.2007/005959-0
|
| [68] |
Dong HN, Zhang DW. Current development in genetic engineering strategies of Bacillus species. Microb Cell Fact, 2014, 13: 63. DOI:10.1186/1475-2859-13-63
|
| [69] |
Yan X, Yu HJ, Hong Q, et al. Cre/lox system and PCR-based genome engineering in Bacillus subtilis. Appl Environ Microbiol, 2008, 74(17): 5556-5562. DOI:10.1128/AEM.01156-08
|
| [70] |
Bloor AE, Cranenburgh RM. An efficient method of selectable marker gene excision by Xer recombination for gene replacement in bacterial chromosomes. Appl Environ Microbiol, 2006, 72(4): 2520-2525. DOI:10.1128/AEM.72.4.2520-2525.2006
|
| [71] |
Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems. Science, 2016, 353(6305): aaf8729. DOI:10.1126/science.aaf8729
|
| [72] |
Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol, 2014, 32(4): 347-355. DOI:10.1038/nbt.2842
|
| [73] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946
|
| [74] |
Westbrook AW, Moo-Young M, Chou CP. Development of a CRISPR-Cas9 tool kit for comprehensive engineering of Bacillus subtilis. Appl Environ Microbiol, 2016, 82(16): 4876-4895. DOI:10.1128/AEM.01159-16
|
| [75] |
Zhang K, Duan XG, Wu J. Multigene disruption in undomesticated Bacillus subtilis ATCC 6051a using the CRISPR/Cas9 system. Sci Rep, 2016, 6: 27943. DOI:10.1038/srep27943
|
| [76] |
Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380-1389. DOI:10.1016/j.cell.2013.08.021
|
| [77] |
Yu SL, Price MA, Wang Y, et al. CRISPR-dCas9 mediated cytosine deaminase base editing in Bacillus subtilis. ACS Synth Biol, 2020, 9(7): 1781-1789. DOI:10.1021/acssynbio.0c00151
|
| [78] |
Maier B. Competence and transformation in Bacillus subtilis. Curr Issues Mol Biol, 2020, 37: 57-76.
|
| [79] |
Zhang XZ, Zhang YHP. Simple, fast and high-efficiency transformation system for directed evolution of cellulase in Bacillus subtilis. Microb Biotechnol, 2011, 4(1): 98-105. DOI:10.1111/j.1751-7915.2010.00230.x
|
| [80] |
Perkins JB, Sloma A, Hermann T, et al. Genetic engineering of Bacillus subtilis for the commercial production of riboflavin. J Ind Microbiol Biotechnol, 1999, 22(1): 8-18. DOI:10.1038/sj.jim.2900587
|
| [81] |
Duan YX, Chen T, Chen X, et al. Overexpression of glucose-6-phosphate dehydrogenase enhances riboflavin production in Bacillus subtilis. Appl Microbiol Biotechnol, 2010, 85(6): 1907-1914. DOI:10.1007/s00253-009-2247-6
|
| [82] |
Shi T, Wang YC, Wang ZW, et al. Deregulation of purine pathway in Bacillus subtilis and its use in riboflavin biosynthesis. Microb Cell Fact, 2014, 13: 101. DOI:10.1186/s12934-014-0101-8/figures/2
|
| [83] |
Wang GL, Bai L, Wang ZW, et al. Enhancement of riboflavin production by deregulating gluconeogenesis in Bacillus subtilis. World J Microbiol Biotechnol, 2014, 30(6): 1893-1900. DOI:10.1007/s11274-014-1611-6
|
| [84] |
Boumezbeur AH, Bruer M, Stoecklin G, et al. Rational engineering of transcriptional riboswitches leads to enhanced metabolite levels in Bacillus subtilis. Metab Eng, 2020, 61: 58-68. DOI:10.1016/j.ymben.2020.05.002
|
| [85] |
Wu Q, Zhi Y, Xu Y. Systematically engineering the biosynthesis of a green biosurfactant surfactin by Bacillus subtilis 168. Metab Eng, 2019, 52: 87-97. DOI:10.1016/j.ymben.2018.11.004
|
| [86] |
Zhang X, Zhang RZ, Bao T, et al. The rebalanced pathway significantly enhances acetoin production by disruption of acetoin reductase gene and moderate-expression of a new water-forming NADH oxidase in Bacillus subtilis. Metab Eng, 2014, 23: 34-41. DOI:10.1016/j.ymben.2014.02.002
|
| [87] |
Helianti I, Nurhayati N, Ulfah M, et al. Constitutive high level expression of an endoxylanase gene from the newly isolated Bacillus subtilis AQ1 in Escherichia coli. J Biomed Biotechnol, 2010, 2010: 980567.
|
| [88] |
Panahi E, Vasheghani-Farahani RF, Shojaosadati SA, et al. Auto-inducible expression system based on the SigB-dependent ohrB promoter in Bacillus subtilis. Mol Biol, 2014, 48(6): 852-857. DOI:10.1134/S0026893314060132
|
| [89] |
Feng Y, Liu S, Jiao Y, et al. Enhanced extracellular production of L-asparaginase from Bacillus subtilis 168 by B. subtilis WB600 through a combined strategy. Appl Microbiol Biotechnol, 2017, 101(4): 1509-1520. DOI:10.1007/s00253-016-7816-x
|
| [90] |
Jin P, Zhang LP, Yuan PH, et al. Efficient biosynthesis of polysaccharides chondroitin and heparosan by metabolically engineered Bacillus subtilis. Carbohydrate Polym, 2016, 140: 424-432. DOI:10.1016/j.carbpol.2015.12.065
|
| [91] |
Liu YF, Zhu YQ, Li JH, et al. Modular pathway engineering of Bacillus subtilis for improved N-acetylglucosamine production. Metab Eng, 2014, 23: 42-52. DOI:10.1016/j.ymben.2014.02.005
|
| [92] |
Gu Y, Lv XQ, Liu YF, et al. Synthetic redesign of central carbon and redox metabolism for high yield production of N-acetylglucosamine in Bacillus subtilis. Metab Eng, 2019, 51: 59-69. DOI:10.1016/j.ymben.2018.10.002
|
 2021, Vol. 37
2021, Vol. 37




