中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈永安, 袁清焱, 李承, 梁书利, 林影
- Chen Yongan, Yuan Qingyan, Li Cheng, Liang Shuli, Lin Ying
- 快速筛选高效表达重组蛋白毕赤酵母菌株新方法的建立及评价
- Development and evaluation of a novel method for rapid screening of Pichia pastoris strains capable of efficiently expressing recombinant proteins
- 生物工程学报, 2021, 37(3): 939-949
- Chinese Journal of Biotechnology, 2021, 37(3): 939-949
- 10.13345/j.cjb.200629
-
文章历史
- Received: October 2, 2020
- Accepted: February 3, 2021
- Published: February 10, 2021
巴斯德毕赤酵母Pichia pastoris是当前应用最为广泛的重组蛋白表达系统之一,已有超过5 000种重组蛋白成功在毕赤酵母系统内实现表达[1]。这些酶既包括具有广泛应用范围的工业酶,如脂肪酶、植酸酶与甘露聚糖酶等,也包括具有重要应用价值的医药相关蛋白,如人血清白蛋白、胰岛素与乙肝表面抗原等[2]。毕赤酵母在表达重组蛋白方面的成功得益于其具有的多种优势,包括易实现高密度发酵、自身分泌蛋白少以及能够实现复杂的翻译后修饰等[3, 4]。但在分泌表达重组蛋白时,该表达系统仍会面临分泌表达效率低、外源基因不稳定、蛋白在胞内降解等问题[2, 5]。
为解决上述问题,研究人员通常从基因、转录、翻译、蛋白分泌等水平对出发菌株进行改造,并根据重组蛋白的性质选择特定的筛选方法来获得高效表达重组蛋白的重组菌株[2]。在筛选高表达工业酶类重组蛋白的重组菌株时,一般是对经过改造的不同转化子进行摇瓶培养并测定相应酶活力从而挑选得到高表达菌株。该筛选方法的缺点是通用性差,一种酶活力测定方法通常只适用于一种酶蛋白,而且操作繁琐,例如测定植酸酶酶活力时需要配制多种试剂,进行多步操作[6]。而对于不具有酶催化活性的医药相关蛋白,则需要建立更为复杂的免疫检测方法(如酶联免疫法或Western blotting等) 才能对重组菌株的蛋白表达水平进行分析[7]。近些年,也有研究人员根据在一定范围内毕赤酵母重组菌株的蛋白表达水平与基因剂量具有相关性的特性,通过体外法与体内法相结合的策略[8]或改变载体整合位点为在毕赤酵母基因组内具有高度重复性的核糖体DNA (Ribosomal DNA,rDNA)[9]从而实现高拷贝菌株的快速构建与筛选。但这类方法存在假阳性概率高的问题,有时鉴定得到的高拷贝菌株并不是高表达菌株[10]。综上,目前仍然欠缺一种通用、快速且准确地筛选高效表达重组蛋白毕赤酵母菌株的方法。
内质网是维持真核细胞生长与存活的重要细胞器,在蛋白质合成中发挥重要作用。内质网转膜蛋白Sec63广泛地分布在内质网上,是内质网的标签蛋白且经常融合表达增强型绿色荧光蛋白(Enhanced green fluorescent protein,EGFP) 而被用于观察内质网的形态[11-12]。当重组活性蛋白大量表达超出细胞处理能力范围时,会导致未折叠蛋白增多并激活未折叠蛋白响应(Unfolded protein response,UPR),从而启动UPR靶基因的转录并改变内质网的形态[13-15],从而回调重组蛋白的表达。在重组蛋白大量表达过程中内质网扩张与Sec63的表达量相关[12]。因此,重组蛋白表达量与Sec63的增量,以及Sec63的标记受到关注。在本研究中,利用Sec63融合表达EGFP的改造菌株确定了重组蛋白表达量与EGFP蛋白表达量之间的线性相关性,并以此为基础建立了混合菌群中高表达菌株的快速筛选方法,为筛选获得高效表达重组蛋白的毕赤酵母菌株提供了一种简便、快速的新途径。
1 材料与方法 1.1 材料 1.1.1 质粒和菌株携带植酸酶phy基因的重组质粒pPICZA- αE10-HKA/(Phy)6、pPICZA-αE10-phy-HKA、携带木聚糖酶xyn基因的重组质粒pHKa-4×xyn10与测定植酸酶phy基因拷贝数的重组质粒pPICZαA-phy-G均由本实验室保存[6, 16]。克隆载体宿主大肠杆菌Escherichia coli Top10、毕赤酵母表达宿主菌P. pastoris GS115购自美国Invitrogen公司;毕赤酵母表达宿主菌P. pastoris GS115-E由本实验室保存[17]。
1.1.2 培养基和溶液LB培养基:0.5%(W/V)酵母提取物,1%(W/V)蛋白胨,1%(W/V) NaCl,115 ℃灭菌20 min。
MD固体培养基:1.34%(W/V)YNB,2%(W/V)葡萄糖,2%(W/V)琼脂粉,115 ℃灭菌20 min后,摇匀后在超净台内倒平板。
1 mol/L磷酸钾缓冲液(pH 6.0):132 mL 1 mol/L K2HPO4,868 mL 1 mol/L KH2PO4,调节pH至6.0,121 ℃灭菌20 min。
BMGY液体培养基:1%(W/V)酵母提取物,2%(W/V)胰蛋白胨,1.34%(W/V)天氨基酵母氮源,1%(V/V)甘油,10%(V/V) 1 mol/L pH 6.0的磷酸钾缓冲液,115 ℃灭菌20 min。
BMMY液体培养基:将BMGY液体培养基中1%(V/V)甘油替换为甲醇,其余不变。
YPD培养基:1%(W/V) 酵母提取物,2%(W/V)蛋白胨,2%(W/V) 葡萄糖,115 ℃灭菌20 min。
YPD固体培养基:向YPD培养基中加入2% (W/V) 琼脂粉,115 ℃灭菌20 min后在超净台内倒平板。
0.25 mol/L醋酸钠溶液(pH 5.50):称取无水乙酸钠20.51 g,加900 mL蒸馏水溶解,用冰醋酸调节pH至5.5±0.01,用蒸馏水定容至1 L。
7.5 mmol/L植酸钠溶液(pH 5.50):称取植酸钠0.69 g,加90 mL 0.25 mol/L乙酸钠溶液溶解,用冰醋酸调节pH至5.5±0.01,用0.25 mol/L乙酸钠溶液定容至100 mL。
硝酸溶液:量取124 mL蒸馏水于烧杯中,搅拌并缓慢地加入76 mL 65%硝酸溶液。
100 g/L钼酸铵溶液:称取钼酸铵10 g,加90 mL蒸馏水溶解,待固体溶解后加入25%氨水1 mL,用蒸馏水定容至100 mL。
2.35 g/L钒酸铵溶液:称取钒酸铵0.235 g,加90 mL蒸馏水溶解,待固体溶解后加入65%硝酸溶液2 mL,用蒸馏水定容至100 mL。
50 mmol/L甘氨酸缓冲溶液(pH 9.0):称取3.75 g甘氨酸,加900 mL蒸馏水溶解,用NaOH调节pH至9.0±0.01,用蒸馏水定容至1 L。
3, 5-二硝基水杨酸溶液:用500 mL蒸馏水溶解3.15 g的3, 5-二硝基水杨酸,水浴至45 ℃。将20 g NaOH完全溶解于100 mL蒸馏水中并将NaOH溶液缓慢加入水杨酸溶液中,搅拌降温至溶液透明。然后依次加入91 g四水合酒石酸钾钠、2.50 g苯酚、2.50 g无水亚硫酸钠,不断搅拌,直至溶液清澈透明,用蒸馏水定容至1 L,保存在棕色瓶中,与二氧化碳隔绝,静置7 d后使用。
10 mg/mL木聚糖溶液:称取1.00 g木聚糖,加入适量50 mmol/L甘氨酸溶液并加热至其完全溶解,用甘氨酸溶液定容至100 mL。
50 mmol/L Tris-HCl缓冲溶液(pH 8.0):称取6.06 g Tris,加900 mL蒸馏水溶解,用HCl调节pH至8.0±0.01,用蒸馏水定容至1 L。
1.1.3 主要试剂与仪器试剂:限制性内切酶Kpn2Ⅰ购自美国Thermo Fisher Scientific公司。琼脂糖凝胶DNA回收试剂盒、质粒提取试剂盒、Bradford Protein Assay Kit购自上海捷瑞生物工程有限公司。Yeast DNAiso Kit、SYBR®Premix Ex TaqTM Ⅱ试剂盒购自大连宝生物工程(TaKaRa) 有限公司。酵母提取物、胰蛋白胨购自英国Oxoid公司。蛋白胨、无氨基酵母氮源(YNB) 购自美国BD-Difco公司。其余均为国产试剂。
仪器:酶标仪为美国Thermo公司的Multiskan Ascent酶标仪。荧光定量PCR仪为美国Applied Biosystems公司的ABI 7500型实时荧光定量PCR仪。流式细胞仪为美国BD公司的BD FACSAriaSORP分选型流式细胞仪。
1.2 方法 1.2.1 表达重组蛋白重组菌株的构建与摇瓶培养将重组质粒pPICZA-αE10-HKA/(Phy)6使用Kpn2Ⅰ线性化并胶回收后分别电转化GS115-E与GS115感受态细胞并在MD平板上进行阳性转化子筛选,得到的阳性转化子分别命名为GS115-E/ P6c、GS115/P6c。同样分别将重组质粒pPICZA- αE10-phy-HKA与pHKa-4×xyn10使用Kpn2Ⅰ线性化并胶回收后电转化GS115-E感受态细胞并在MD平板上进行阳性转化子筛选,得到的阳性转化子分别命名为GS115-E/P1c、GS115-E/X4c。
将得到的重组菌株分别接种于5 mL BMGY培养基,在30 ℃、250 r/min条件下培养20 h。8 000 r/min离心5 min收集适量细胞,然后将收集的细胞重悬到20 mL BMMY培养基至起始OD600为0.5,30 ℃、250 r/min振荡培养,每24 h取样并补加终浓度1% (V/V) 的甲醇。样品经6 000 r/min,4 ℃离心5 min分离菌体和发酵上清液,对于出发菌株是GS115-E的重组菌株检测菌体荧光、发酵上清液酶活与蛋白浓度,对于重组菌株GS115/P6c只检测发酵上清液酶活与蛋白浓度。
1.2.2 产酶分析植酸酶酶活测定参照Li等[6]的方法并稍有改变。具体过程如下:用pH为5.5的醋酸钠缓冲液稀释发酵上清液至合适的倍数并保持终体积为1 mL。37 ℃预热5 min后加入2 mL植酸钠溶液,混匀。37 ℃反应30 min后加入2 mL现配的显色/终止液(2.35 g/L钒酸铵溶液︰100 g/L钼酸铵溶液︰硝酸溶液=1︰1︰2),室温静止10 min后,于415 nm处测定吸光度。植酸酶酶活力定义:37 ℃,pH值为5.5条件下,每分钟水解植酸钠溶液释放1 μmol无机磷的酶量为l个酶活单位(U)。
木聚糖酶酶活力测定参照Lin等[18]的方法并有稍微调整。具体过程如下:用pH为9.0的甘氨酸缓冲液稀释发酵上清液至合适的倍数,混匀后取1 mL稀释液于25 mL比色管中。加入10 mg/mL的木聚糖溶液1 mL,混合,70 ℃反应30 min。之后马上加入3 mL的3, 5-二硝基水杨酸溶液,轻轻混匀后,沸水浴10 min,然后用蒸馏水定容至25 mL,取反应液于540 nm下测定其吸收值。木聚糖酶酶活力定义:70 ℃、pH 9.0条件下,每分钟水解10 mg/mL的木聚糖溶液释放1 μmol还原糖的酶量为l个酶活单位(U)。
发酵上清液蛋白浓度按照Bradford Protein Assay Kit说明书进行测定。
1.2.3 菌体EGFP荧光检测向收集得到的菌体中加入300 μL的Tris-HCl缓冲液,涡旋振荡洗涤菌体后,8 000 r/min、4 ℃离心1 min去上清。重复该过程3次,在最后一次洗涤结束后使用适量的Tris-HCl缓冲液重悬菌体,平行取3次相同体积菌悬液检测荧光强度(激发波长488 nm,发射波长520 nm),并测定菌悬液的OD600值。
1.2.4 利用流式细胞仪对GS115-E/P1c与GS115-E/P6c构成的混合菌群进行分选将重组菌株GS115-E/P1c与GS115-E/P6c接种到同一瓶BMGY培养基,参照1.2.1进行摇瓶培养,培养至96 h后取样。样品经6 000 r/min、4 ℃离心5 min并去上清。向收集得到的菌体中加入300 μL的Tris-HCl缓冲液洗涤3次,最后加入适量Tris-HCl缓冲液重悬菌体,并利用分选型流式细胞仪分选出高EGFP荧光值酵母细胞群落与低EGFP荧光值酵母细胞群落。将分选到的两种酵母细胞群落分别涂布于YPD平板,30 ℃培养48 h。在高荧光值菌群与低荧光值菌群涂布的平板上各挑选20个转化子并参照1.2.1进行摇瓶培养,每24 h取样并检测菌体荧光与发酵上清液酶活。
1.2.5 重组菌株基因组DNA提取挑取相应重组菌株单菌落于10 mL YPD培养基中,30 ℃、250 r/min培养20 h。取含有约1×108–2×108个细胞的菌液,12 000 r/min离心5 min收集菌体,弃上清。之后按照Yeast DNAiso Kit说明书提取毕赤酵母基因组DNA。
1.2.6 重组菌株phy基因拷贝数检测采用双标准曲线法测定phy基因拷贝数[6, 19],过程如下:重组质粒pPICZαA-phy-G包含单拷贝的phy基因与gapdh基因片段,以其作为基因拷贝数测定的标准质粒[6]。以一系列梯度稀释并具有合适浓度的pPICZαA-phy-G质粒为模板,分别利用引物P1/P2和P3/P4(表 1)使用SYBR®Premix Ex TaqTM Ⅱ (Tli RNaseH Plus) 试剂盒进行实时荧光定量PCR,将获得目的基因phy和内参基因gapdh的Ct值与相对的质粒浓度制作标准曲线。以提取的相关菌株的基因组DNA为模板,使用上述引物与条件进行实时荧光定量PCR,每个样品重复测定3次,得到的Ct值代入下列公式计算phy基因拷贝数:

|
| Primer name | Primer sequence (5′–3′) |
| P1 | TGGTTGGGGTAGAATCAC |
| P2 | TGCTTCTGAGGAGGATGA |
| P3 | GTCGGGACACGCCTGAAACT |
| P4 | CCACCTTTTGGACCCTATTGAC |
本研究中我们以内质网膜蛋白Sec63融合表达EGFP的重组菌株GS115-E为表达重组蛋白的宿主菌株,因其经过基因工程改造所以需要检测其表达重组蛋白的能力相比于最常用的表达宿主菌P. pastoris GS115是否发生改变。
将携带植酸酶phy基因的重组质粒pPICZA-αE10-HKA/(Phy)6分别电转化进入GS115-E与GS115的感受态细胞获得表达植酸酶的重组菌株GS115-E/P6c与GS115/P6c。对这两种重组菌株进行摇瓶培养并检测发酵上清液植酸酶酶活,结果发现在甲醇诱导培养的24–120 h期间,两种重组菌株表达重组蛋白植酸酶的水平均十分接近(图 1),说明对重组菌株GS115-E进行的基因工程改造并不影响其表达重组蛋白的能力,为之后的研究奠定了良好的基础。
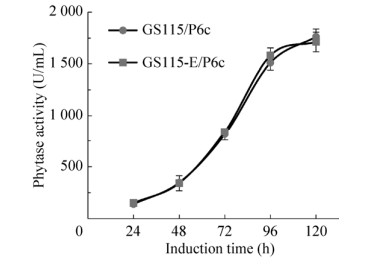
|
| 图 1 重组菌株GS115-E/P6c与GS115/P6c的产酶分析 Fig. 1 Analysis of enzyme production of recombinant strains GS115-E/P6c and GS115/P6c. |
| |
内质网转膜蛋白Sec63广泛地分布于内质网上,其表达量会随内质网形态的改变而变化,而与Sec63融合的EGFP的表达量也随之变化[12]。而重组蛋白大量表达时会导致内质网形态发生变化[13-15]。综合以上结果可推测Sec63融合的EGFP的表达量与重组蛋白的表达量之间具某种相关性,但这种相关性目前仍未得到确定。
在2.1中构建得到了表达植酸酶的重组菌株GS115-E/P6c,之后按照1.2.1中所述方法同样构建得到表达木聚糖酶的重组菌株GS115-E/X4c。对这两种重组菌株进行摇瓶培养并进行产酶分析与检测菌体EGFP荧光强度,结果显示,重组蛋白与Sec63融合的EGFP具有相似的表达趋势(图 2A),且重组蛋白与Sec63融合的EGFP的表达量之间具有良好的线性相关性(0.8 < |R| < 1) (图 2B,图 3)。另外,无论是对于植酸酶表达菌株还是木聚糖酶表达菌株,这种线性相关性均存在(图 2、3),说明重组蛋白与Sec63融合的EGFP的表达量之间的线性关系是普遍且通用的,也说明可以用检测菌体EGFP的荧光强度代替分析重组蛋白的表达水平,这是本研究实现高表达菌株快速筛选的基础。
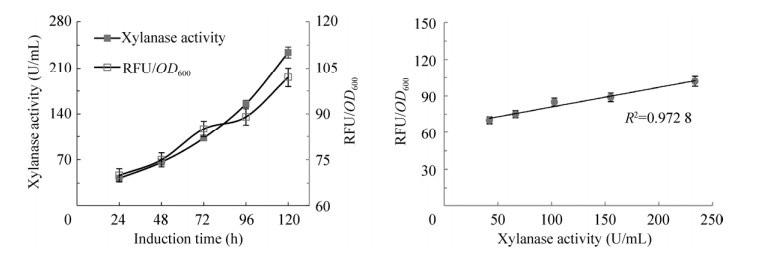
|
| 图 2 菌株GS115-E/X4c的木聚糖酶与EGFP荧光值之间的相关性 Fig. 2 The correlation between xylanase and EGFP expression of GS115-E/X4c. (A) Expression curve of xylanase and EGFP. (B) The correlation between xylanase activity and relative fluorescence units (RFU). |
| |
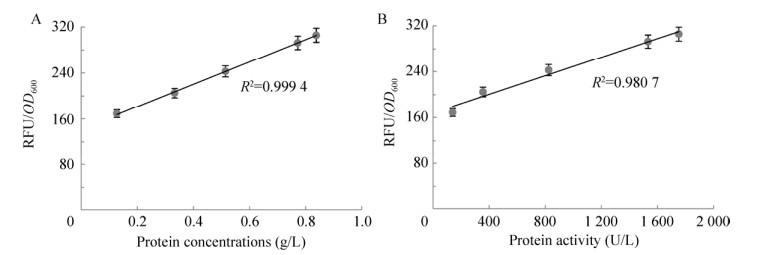
|
| 图 3 菌株GS115-E/P6c的植酸酶与EGFP荧光值之间的相关性 Fig. 3 The correlation between phytase and EGFP expression of GS115-E/P6c. (A) The correlation between supernatant protein concentration and RFU. (B) The correlation between phytase activity and RFU. |
| |
为了建立通过检测EGFP荧光强度进行高表达菌株快速筛选的方法,我们首先构建了具有不同重组蛋白表达水平的菌株混合形成的菌群。按照1.2.1中所述方法获得表达1拷贝phy基因的重组菌株GS115-E/P1c,而重组菌株GS115-E/P6c表达6拷贝phy基因。在摇瓶中将这两种重组菌株混合培养并利用流式细胞仪对混合菌群进行分析,发现混合菌群分为低EGFP荧光与高EGFP荧光的两种细胞群落(图 4A)。而且低荧光细胞群落是表达1个拷贝phy基因的重组菌株GS115-E/P1c (图 4B中蓝色散点图),高荧光细胞群落是表达6个拷贝phy基因的重组菌株GS115-E/P6c (图 4C中红色散点图)。

|
| 图 4 混合菌群的流式细胞仪分析图 Fig. 4 Analysis of mixed culture with flow cytometry. (A) Fluorescence-activated cell sorting (FACS) scatter diagram of mixed culture. (B) FACS scatter diagrams of mixed culture and GS115-E/P1c. (C) FACS scatter diagrams of mixed culture, GS115-E/P1c and GS115-E/P6c. |
| |
在发现混合菌群分为高EGFP荧光与低EGFP荧光两种细胞群落之后,利用分选型流式细胞仪根据EGFP荧光进行细胞分选。在分选得到的低荧光菌株与高荧光菌株中分别随机挑选20株进行摇瓶培养并进行产酶分析,结果显示,在甲醇诱导培养120 h时,高荧光菌株的重组蛋白植酸酶产量均显著高于低荧光菌株的植酸酶产量(图 5),高荧光菌株的植酸酶平均表达水平是低荧光菌株的4.09倍(图 6)。而且在诱导培养的48–120 h期间,高荧光菌株的重组蛋白植酸酶产量均远远高于低荧光菌株的植酸酶产量(图 6)。
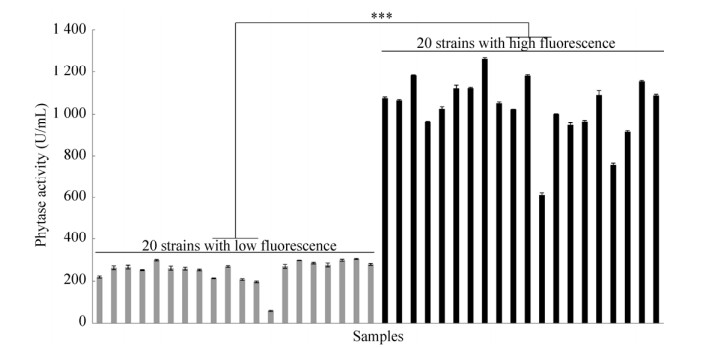
|
| 图 5 诱导培养120 h时高荧光与低荧光菌株的植酸酶表达情况 Fig. 5 Phytase expression of high-fluorescence and low-fluorescence strains in induced culture for 120 h. ***: P < 0.001. |
| |

|
| 图 6 高荧光与低荧光菌株的植酸酶酶活均值曲线 Fig. 6 Curve of mean values of phytase activity of high-fluorescence and low-fluorescence strains. |
| |
之后,从进行摇瓶培养的高荧光菌株和低荧光菌株中各随机选取2株进行phy基因拷贝数测定,发现低荧光菌株的phy基因拷贝数为1,而高荧光菌株的phy基因拷贝数为6 (图 7)。以上结果表明:已成功建立了通过检测EGFP荧光强度进行高表达菌株快速筛选的方法,该方法可以快速、准确地从混合菌群中筛选到高效表达重组蛋白的毕赤酵母菌株。
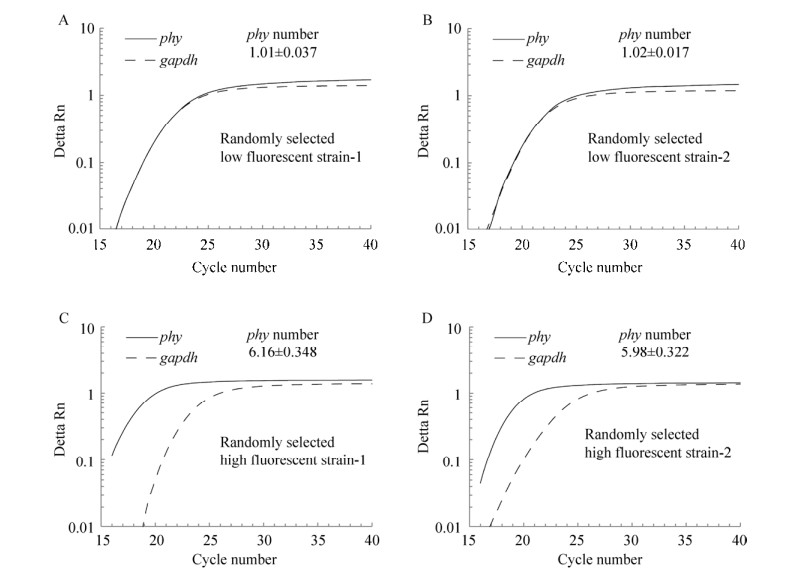
|
| 图 7 高荧光与低荧光菌株的phy基因拷贝数鉴定 Fig. 7 Quantitative PCR assay of the phy gene copy numbers in genomic DNA of low-fluorescence strain-1, 2 (A, B) and high-fluorescence strain-1, 2 (C, D). The values indicate the x±s from three independent qPCR experiments. |
| |
毕赤酵母从20世纪90年代早期开始被作为一种重组蛋白表达系统进行开发[1]。发展至今,研究人员已从外源基因表达盒、分泌途径、出发菌株、基因编辑与发酵过程控制等多个方面建立多种多样的方法来提高重组蛋白在毕赤酵母中的表达水平[1, 2, 4, 20, 21]。与之相比,如何从经过改造后的诸多转化子中快速筛选得到重组蛋白表达水平确定提高的菌株却鲜有报道。目前,在毕赤酵母系统中筛选高表达菌株主要还是采用传统的平板、多孔板或摇瓶培养的方法。这些方法的缺点之一是通用性差,一种表征平板或蛋白分析方法只适用于一种重组蛋白。另外这些方法操作繁琐、速度慢,如果重组蛋白是重组酶则需要采用较复杂的酶活检测方法,如检测植酸酶的酶活大约需要60 min的时间[6],而当重组蛋白是不具有酶催化活性的医药相关蛋白时则需要建立更为复杂的免疫检测方法[7]。近些年,也有研究人员利用毕赤酵母的蛋白表达水平与基因剂量具有相关性的特性,通过快速地构建并筛选高拷贝菌株来达到快速筛选得到高表达菌株的目的[8, 9]。这一类方法的问题是蛋白表达水平与基因剂量在超出一定范围后并不具有相关性,高拷贝菌株并不一定是高表达菌株。另外,外源基因拷贝数的增加还会导致基因整合稳定性的降低[10]。
在本研究中,首先验证了将EGFP与内质网转膜蛋白Sec63融合表达并不影响改造菌株表达重组蛋白的能力。之后,确定了与内质网转膜蛋白Sec63融合表达的EGFP的表达水平与重组蛋白的表达水平之间具有普遍且良好的线性相关性(0.8 < |R| < 1)。最后,利用分选型流式细胞仪成功地从混合菌群中筛选得到高表达植酸酶的重组菌株,分选得到的高表达菌株在摇瓶发酵120 h时植酸酶表达水平是低表达菌株的4.09倍。
本研究建立的通过检测EGFP荧光强度进行高表达菌株快速筛选的方法具有快速简便的优点,利用酶标仪检测EGFP荧光仅仅需要几分钟的时间,相比于复杂的酶活测定或蛋白免疫分析可大大节约时间与人工成本,与流式细胞仪或液滴微流控技术等高通量筛选仪器与技术结合则能进一步提高本方法筛选的速度与通量。液滴微流控技术是近年来发展迅速的高通量筛选新技术,具有通量高、速度快与检测成本低等优点[22-24]。最近,Ma等[25]研究人员将液滴微流控技术与常压室温等离子(Atmospheric and room temperature plasma,ARTP) 诱变技术结合,实现了高产木聚糖酶菌株的快速构建与高通量筛选,其筛选通量可达每天108个菌株。但是,该方法进行菌株筛选时需要添加特定的木聚糖酶荧光底物,如要进行对表达其他重组蛋白菌株的筛选则需要更换荧光底物甚至无法实现筛选,例如不具有酶催化活性的医药蛋白,应用条件的严格与单一限制了该方法的进一步应用。与此相比,本研究建立方法的应用条件更为宽松和广泛,本方法实现菌株筛选的基础是内质网膜蛋白融合表达的EGFP的表达水平与重组蛋白的表达水平之间具有良好的线性相关性,虽然该现象背后的分子机制仍待进一步确定,但利用本方法进行菌株筛选时对重组蛋白的种类及性质没有限制,对于分泌表达工业酶或不具有酶催化活性的重组蛋白的毕赤酵母菌株均可应用本方法进行筛选。综上,本研究建立的方法为筛选获得高效表达重组蛋白的毕赤酵母菌株提供了一种简便、快捷的新途径。
| [1] |
García-Ortega X, Cámara E, Ferrer P, et al. Rational development of bioprocess engineering strategies for recombinant protein production in Pichia pastoris (Komagataella phaffii) using the methanol-free GAP promoter. Where do we stand?. New Biotechnol, 2019, 53(August 2018): 24-34.
|
| [2] |
Zhu TC, Sun HB, Wang MY, et al. Pichia pastoris as a versatile cell factory for the production of industrial enzymes and chemicals: current status and future perspectives. Biotechnol J, 2019, 14(6): 1800694. DOI:10.1002/biot.201800694
|
| [3] |
Yang ZL, Zhang ZS. Engineering strategies for enhanced production of protein and bio-products in Pichia pastoris: a review. Biotechnol Adv, 2018, 36(1): 182-195. DOI:10.1016/j.biotechadv.2017.11.002
|
| [4] |
Fischer J E, Glieder A. Current advances in engineering tools for Pichia pastoris. Curr Opin Biotechnol, 2019, 59: 175-181. DOI:10.1016/j.copbio.2019.06.002
|
| [5] |
Ahmad M, Hirz M, Pichler H, et al. Protein expression in Pichia pastoris: Recent achievements and perspectives for heterologous protein production. Appl Microbiol Biotechnol, 2014, 98(12): 5301-5317. DOI:10.1007/s00253-014-5732-5
|
| [6] |
Li C, Lin Y, Zheng XY, et al. Combined strategies for improving expression of Citrobacter amalonaticus phytase in Pichia pastoris. BMC Biotechnol, 2015, 15(1): 1-11. DOI:10.1186/s12896-015-0115-2
|
| [7] |
Knapp A, Ripphahn M, Volkenborn K, et al. Activity-independent screening of secreted proteins using split GFP. J Biotechnol, 2017, 258(May): 110-116.
|
| [8] |
Zhu T, Guo M, Tang Z, et al. Efficient generation of multi-copy strains for optimizing secretory expression of porcine insulin precursor in yeast Pichia pastoris. J Appl Microbiol, 2009, 107(3): 954-963. DOI:10.1111/j.1365-2672.2009.04279.x
|
| [9] |
Marx H, Mecklenbräuker A, Gasser B, et al. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Res, 2009, 9(8): 1260-1270. DOI:10.1111/j.1567-1364.2009.00561.x
|
| [10] |
Aw R, Polizzi KM. Can too many copies spoil the broth?. Microb Cell Fact, 2013, 12(1): 1-9. DOI:10.1186/1475-2859-12-1
|
| [11] |
Chen S, Novick P, Ferro-Novick S. ER structure and function. Curr Opin Cell Biol, 2013, 25(4): 428-433. DOI:10.1016/j.ceb.2013.02.006
|
| [12] |
Schuck S, Prinz WA, Thorn KS, et al. Membrane expansion alleviates endoplasmic reticulum stress independently of the unfolded protein response. J Cell Biol, 2009, 187(4): 525-536. DOI:10.1083/jcb.200907074
|
| [13] |
Zhong YJ, Yang L, Guo YG, et al. High-temperature cultivation of recombinant Pichia pastorisincreases endoplasmic reticulum stress and decreases production of human interleukin-10. Microb Cell Fact, 2014, 13(1): 163. DOI:10.1186/s12934-014-0163-7
|
| [14] |
Guerfal M, Ryckaert S, Jacobs P P, et al. The HAC1 gene from Pichia pastoris: characterization and effect of its overexpression on the production of secreted, surface displayed and membrane proteins. Microb Cell Fact, 2010, 9(1): 49. DOI:10.1186/1475-2859-9-49
|
| [15] |
Vogl T, Thallinger G G, Zellnig G, et al. Towards improved membrane protein production in Pichia pastoris: general and specific transcriptional response to membrane protein overexpression. N Biotechnol, 2014, 31(6): 538-552. DOI:10.1016/j.nbt.2014.02.009
|
| [16] |
林小琼. 基于iTRAQ技术的高效表达木聚糖酶重组毕赤酵母细胞的蛋白组学研究[D]. 广州: 华南理工大学, 2013. Lin XQ. iTRAQ-based quantitative proteomic analysis of high-level expression of xylanase in Pichia pastoris[D]. Guangzhou: South China University of Technoiogy, 2013. |
| [17] |
李承. 高表达植酸酶毕赤酵母内质网稳态的形成及Wsc1p的调控[D]. 广州: 华南理工大学, 2018. Li C. Reestablishment of endoplasmic reticulum homeostasis and regulation of Wsclp during phytase overproduction in Pichia pastoris[D]. Guangzhou: South China University of Technoiogy, 2018. |
| [18] |
Lin XQ, Liang SL, Han SY, et al. Quantitative iTRAQ LC–MS/MS proteomics reveals the cellular response to heterologous protein overexpression and the regulation of HAC1 in Pichia pastoris. J Proteomics, 2013, 91: 58-72. DOI:10.1016/j.jprot.2013.06.031
|
| [19] |
Lin XQ, Han SY, Zhang N, et al. Bleach boosting effect of xylanase A from Bacillus halodurans C-125 in ECF bleaching of wheat straw pulp. Enzyme Microb Technol, 2013, 52(2): 91-98. DOI:10.1016/j.enzmictec.2012.10.011
|
| [20] |
Peña DA, Gasser B, Zanghellini J, et al. Metabolic engineering of Pichia pastoris. Metab Eng, 2018, 50(February): 2-15.
|
| [21] |
Juturu V, Wu JC. Heterologous protein expression in Pichia pastoris: latest research progress and applications. ChemBioChem, 2018, 19(1): 7-21. DOI:10.1002/cbic.201700460
|
| [22] |
Chiu FWY, Stavrakis S. High-throughput droplet-based microfluidics for directed evolution of enzymes. Electrophoresis, 2019, 40(21): 2860-2872. DOI:10.1002/elps.201900222
|
| [23] |
Autour A, Ryckelynck M. Ultrahigh-throughput improvement and discovery of enzymes using droplet-based microfluidic screening. Micromachines, 2017, 8(4).
|
| [24] |
Beneyton T, Thomas S, Griffiths AD, et al. Droplet-based microfluidic high-throughput screening of heterologous enzymes secreted by the yeast Yarrowia lipolytica. Microb Cell Fact, 2017, 16(1): 18. DOI:10.1186/s12934-017-0629-5
|
| [25] |
Ma CX, Tan ZL, Lin Y, et al. Gel microdroplet-based high-throughput screening for directed evolution of xylanase-producing Pichia pastoris. J Biosci Bioeng, 2019, 128(6): 662-668. DOI:10.1016/j.jbiosc.2019.05.008
|
 2021, Vol. 37
2021, Vol. 37




