
中国科学院微生物研究所、中国微生物学会主办
文章信息
- 崔佳佳, 张雪洪
- Cui Jiajia, Zhang Xuehong
- 微生物源农用抗生素的研发与高产策略
- Development and high yield strategies of microbial-derived antibiotics in agriculture
- 生物工程学报, 2021, 37(3): 1032-1041
- Chinese Journal of Biotechnology, 2021, 37(3): 1032-1041
- 10.13345/j.cjb.200643
-
文章历史
- Received: October 10, 2020
- Accepted: January 23, 2021
- Published: February 3, 2021
近年来随着生态保护、食品安全、绿色生产、可持续发展等理念的深入人心,化学农药的监管也越来越严格。开发高效、低毒、低残留、无污染的生物农药成为未来农药研发的发展方向。微生物源农用抗生素在生物农药中占据重要地位,它们是由微生物合成的具有生物可降解性的次级代谢产物,因其高效、安全、环境友好的特性成为了化学农药的绿色替代品,是植物病虫害防治和绿色可持续发展农业领域的研究热点。
目前微生物源农用抗生素已经广泛应用于农业害虫和植物病害的防治,为农业生产做出了重要贡献。随着微生物育种技术和发酵技术的提升,各种农用抗生素的发酵效价不断提高、发酵规模不断扩大、生产成本逐步降低,其在农业生产应用的竞争力日益增强,如井冈霉素、阿维菌素、中生菌素、南昌霉素、春雷霉素、浏阳霉素、多抗霉素、申嗪霉素、武夷霉素、宁南霉素、多杀菌素、灭瘟素等。
同时,通过筛选活性先导化合物,采用组合技术和基因工程方法,对基因簇及代谢途径进行修饰和改造、研究新型的抗生素和提高有效组分的产量;在微生物全基因组测序和功能基因研究的基础上,克隆和发掘新的具有杀虫和杀菌功能的基因,研究基因的高效表达和调控;建立生物农药大规模、高通量筛选技术平台,筛选新型农用抗生素;基于高通量筛选技术利用体外分子诱变进化技术、基因随机诱变或重组等,筛选和改造农用抗生素高产菌株;利用现代发酵工程技术、生化工程技术以及工程化系统集成加快发酵工艺优化改进,大幅度提高农用抗生素发酵技术水平。新的农用抗生素得到持续研发、传统农用抗生素的产品质量进一步稳定、生产效率大大提高。本文简要总结了近10年来研发的新型微生物源农用抗生素的种类,农用抗生素产生菌株的高产育种研究策略等,为未来农用抗生素的研发提供参考。
1 近10年来研发的微生物源农用抗生素微生物源农用抗生素因高效、绿色环保等特性而成为生物农药的主力品种,积极筛选和开发新型微生物源农用抗生素对于推动生物农药的发展、促进我国农业生产具有十分重要的意义。本文简要总结了近10年来研发的新型微生物源农用抗生素(表 1)。
| Antibiotics | Strains | Structure | Biological activity | References |
| Phenazine-1-carboxylic acid, PCA | Pseudomonas aeruginosa M18 | 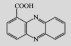 |
Antagonistic to fungal phytopathogens | [1] |
| Phenazine-1-carboxamide, PCN | P. chlororaphis HT66, P. aeruginosa |  |
Antagonistic to fungal phytopathogens | [2] |
| 2-hydroxyphenazines, 2-OH-PHZ | P. chlororaphis 30-84, P. chlororaphis GP72 |  |
Antagonistic to fungal phytopathogens | [2] |
| Borrelidin | Streptomyces spp. | 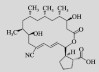 |
Antagonistic to fungal phytopathogens | [3] |
| Milbemycins | S. hygroscopicus subsp. Aureolacrimosus, S. bingchenggensis |  |
Acaricidal, insecticidal, and anthelmintic activities | [4] |
| Pyoluteorin | P. aeruginosa, Pseudomonas spp. |  |
Antifungal and antibacterial activities | [5] |
| Anisomycin | S. roseochromogenes, S. griseolus, S. hygrospinosus var. beijingensis |  |
Antagonistic to fungal phytopathogens | [6] |
| Bacillomycin D | Bacillus amyloliquefaciens, B. subtitles | 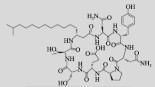 |
Antagonistic to fungal phytopathogens | [7] |
| Xinaomycin | Streptomyces spp. | 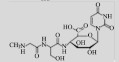 |
Antibacterial, antifungal and antiviral activities | [8] |
| Xenocoumacin 1 | Xenorhabdus nematophila | 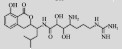 |
Antifungal activity | [9] |
| Enduracidins | S. fungicidicus |  |
Inhibits Gram-positive bacteria | [10] |
吩嗪类抗生素是一类具有含氮杂环结构的次级代谢产物,假单胞菌属和链霉菌属是天然吩嗪类抗生素的主要生产者,吩嗪类抗生素因其广谱抑菌活性受到研究人员的广泛关注[11]。目前作为农用抗生素研究的主要有吩嗪-1-羧酸、吩嗪-1-甲酰胺、2-羟基吩嗪等。
1.1.1 吩嗪-1-羧酸(Phenazine-1-carboxylic acid,PCA)PCA是一种黄色的吩嗪类化合物,在2011年由铜绿假单胞菌Pseudomonas aeruginosa M18产生的PCA被中国农业部批准为一种新型微生物源杀菌剂“申嗪霉素”,具有自主知识产权。PCA对于水稻纹枯病、西瓜枯萎病、甜椒疫病、辣椒疫病以及黄瓜枯萎病等具有良好的防治效果,但是PCA的抗真菌活性受环境pH的影响较大[12]。
1.1.2 吩嗪-1-甲酰胺(Phenazine-1-carboxamide,PCN)PCN可由绿针假单胞菌P. chlororaphis和P. aeruginosa等合成,PCN对于尖孢镰刀菌、水稻黄单胞菌、立枯丝核菌[13]、番茄镰刀菌以及终极腐霉等多种植物病原真菌具有显著的拮抗作用。在中性条件下,PCN的抗真菌活性比PCA高得多,因此PCN能够适应不断变化的环境[14-15]。
1.1.3 2-羟基吩嗪(2-hydroxyphenazines,2-OH-PHZ)2-OH-PHZ是由P. chlororaphis 30–84和P. chlororaphis GP72等绿针假单胞菌产生的一种吩嗪衍生物,2-OH-PHZ具有广谱杀菌性,能够有效防治小麦全蚀病,对于疫霉、腐霉等植物病原真菌也具有很好的抑制作用[16]。
1.2 聚酮类抗生素 1.2.1 疏螺体素(Borrelidin)疏螺体素为浅黄色晶体,是一种大环内酯类抗生素,分离自链霉菌属。疏螺体素对于大豆疫霉菌、瓜果腐霉、终极腐霉以及辣椒疫霉等植物病原真菌具有显著的拮抗作用,其中疏螺体素对大豆疫霉菌具有很高的特异性抗真菌活性,10 mg/L的疏螺体素对大豆疫霉菌的防治效果达到了94.72%,其防治效果显著高于甲霜灵[3]。
1.2.2 米尔贝霉素(Milbemycins)米尔贝霉素是由链霉菌产生的一类十六元大环内酯类化合物,具有强效的抗虫和杀虫活性。由冰城链霉菌Streptomyces bingchenggensis产生的米尔贝霉素A3/A4已被开发为防治农业螨虫的杀螨剂,米尔贝霉素A3/A4的半合成衍生物米尔比霉素肟化物已经用于线虫、丝虫等害虫的防治,其他米尔贝霉素A3/A4的衍生物如雷皮菌素和拉替菌素也已经广泛应用于农业领域[17]。
1.2.3 藤黄绿脓菌素(Pyoluteorin,Plt)Plt是一种最早从P. aeruginosa中分离出来的聚酮合酶(Polyketide synthase,PKS)/非核糖体肽合酶(Non-ribosomal peptide synthetase,NRPS)杂合抗生素,对于真菌(特别是腐霉属) 和细菌具有广谱抗菌活性,可通过采用薄层色谱法以及飞行时间质谱法对藤黄绿脓菌素进行相应的鉴定[18-19]。
1.2.4 茴香霉素(Anisomycin)茴香霉素是由玫瑰产色链霉菌S. roseochromogenes、刺孢吸水链霉菌北京变种S. hygrospinosus var. beijingensis、浅灰色链霉菌S. griseolus等产生的一种吡咯烷类抗生素,是农抗120中重要的有效成分之一,对其生物合成基因簇进行鉴定后通过发酵分离得到。茴香霉素主要用于农作物真菌病害的防治,比如农作物白粉病、西瓜腐烂病、水稻纹枯病等[6, 20]。
1.2.5 Xenocoumacin 1 (Xcn1)Xenocoumacin 1 (Xcn1) 是从嗜线虫致病杆菌Xenorhabdus nematophila的培养物中分离出的主要抗菌化合物,对革兰氏阳性细菌表现出广泛的抗菌活性,并且对于互隔交链孢霉、灰霉菌、立枯丝核菌、疫霉菌等具有很强的抑制活性,具有成为新型生物农药的巨大潜力[21-22]。
1.3 多肽类抗生素 1.3.1 杆菌霉素D (Bacillomycin D)杆菌霉素D是由解淀粉芽孢杆菌Bacillus amyloliquefaciens、枯草芽孢杆菌B. subtitles、贝莱斯芽孢杆菌B. velezensis等产生的一种环状抗真菌脂肽。杆菌霉素D可以有效抑制孢子萌发和菌丝体生长[23],对于黄曲霉、禾谷镰刀菌、炭疽菌、灰葡萄孢菌、匍枝根霉等具有很强的抑制活性[24-25]。杆菌霉素D安全且易于降解,土壤中的杆菌霉素D通过分解为氨基酸残基,可以防止其积累达到有害水平[26]。
1.3.2 恩拉霉素(Enduracidins)恩拉霉素最初是从杀真菌素链霉菌S. fungicidicus中分离出来的一组脂肽抗生素,可以甲醇为溶剂并利用超声辅助进行分离提取[27]。恩拉霉素由17个氨基酸组成,根据脂肪链长度的不同分为恩拉霉素A和恩拉霉素B。恩拉霉素是一种碱性抗生素,由于对于大多数革兰氏阳性菌具有很好的抑制活性[10, 27-28],因此有望在农业中广泛应用。
1.4 核苷类抗生素—新奥霉素(Xinaomycin)新奥霉素是由中国科学院成都生物研究所用遗传改良菌株“诺尔斯链霉菌Xi Ao-3”发酵生产的一种新型尿嘧啶核苷肽类抗生素,对疫霉菌、炭疽病菌、棉花黄萎病菌、番茄花叶病毒以及西瓜花叶病毒具有很强的抑制活性[29-30]。新奥霉素作为一种新型广谱生物杀菌剂存在一定的缺陷,一是新奥霉素产品呈酸性,不适宜与碱性药剂混合使用;二是新奥霉素对紫外光线敏感,不宜在强日照下使用[8]。
2 微生物源农用抗生素的高产育种新策略不同用途的微生物源抗生素的高产育种方法基本相似,相关方法同样应用于农用抗生素的研发。从自然界分离出来的野生型菌株所产生的抗生素含量一般很低,无法满足商业需求。工业微生物高产菌株选育的常用手段是采用化学或物理诱变、基因工程育种等。传统的化学诱变、UV诱变、放射诱变等仍是生物企业常用的育种手段,近年来常压室温等离子体(Atmospheric and room temperature plasma,ARTP) 因安全、简便得到广泛应用。随着微生物基因组学的发展,代谢工程技术及基因编辑技术的进步,基因工程育种已成为抗生素高产菌株获得的主要途径。
2.1 新型诱变育种近年来,ARTP技术和重离子诱变技术被认为是有效和新颖的微生物诱变育种技术。ARTP的核心组件是RF ARGD等离子体发生器,它产生的等离子体射流可以改变细胞壁和细胞质膜的结构和通透性,导致DNA损伤,包括错义突变、核苷酸缺失或核苷酸移码突变等,与传统诱变育种方法相比,ARTP具有突变速度快,操作灵活度高、安全高效等特点[30-31]。目前,ARTP技术已经成功应用于假单胞菌属和链霉菌属等高产菌株的选育,使得吩嗪-1-甲酰胺、阿维菌素和米尔贝霉素A3/A4的产量有了较大的提升[32-34]。
重离子诱变技术也是一种产生微生物突变体的有效方法,相比传统的诱变方法它可以产生更高的突变率和突变谱。重离子诱变通过热和电离效应等直接造成微生物菌株的DNA损伤,这些DNA损伤更加有利于微生物突变体的产生。如通过利用兰州重离子加速器(Heavy ion research facility in Lanzhou,HIRFL) 对链霉菌进行诱变育种,提高了恩拉霉素以及阿维菌素的产量[35]。
微生物进行诱变育种后需要对大量的菌株进行测试与筛选,传统的琼脂平板初步筛选、摇瓶发酵二次筛选和高效液相色谱(High performance liquid chromatography,HPLC) 验证产量的筛选过程非常耗时,因此在育种过程中需要建立简单快速的高通量筛选方法以有效率地获得阳性菌株。在P. chlororaphis GP72中将2-OH-PHZ合成途径中的限制性酶PhzO替换为绿色荧光蛋白(Green fluorescent protein,GFP),以GFP作为筛选标记进行诱变育种,通过酶标仪检测荧光强度进行高通量筛选,在筛选出的突变株中通过PhzO的回替,使2-OH-PHZ的产量比野生型菌株提高了4.62倍[36];在对S. lomondensis S015进行ARTP技术和紫外复合诱变育种后,建立了基于24孔深孔板发酵和酶标仪快速检测的高通量筛选方法[37]。
新型诱变育种操作简单、突变率高,可在较短时间内获得优良的突变类型,但是诱变育种具有一定的盲目性与随机性,一般情况下产生的有益突变体频率低,而且后期突变菌株的筛选比较困难。
2.2 基因工程育种 2.2.1 利用代谢工程技术改造生产菌株负调控基因的敲除、正调控基因的过表达、分支途径的改造是基因工程育种的常用方法。微生物代谢网络是一个复杂的系统,基因组学技术、生物信息学的快速发展和基因编辑技术的建立为微生物源农用抗生素的工业化生产创造了新的发展空间。通过系统性筛选和代谢网络分析,抑制负调控途径或因子、扩增正调控途径或因子、加强产物运输系统、增强前体途径、操纵产物反馈机制以及消除竞争性代谢途径等方法可以使微生物源农用抗生素的产量有较大的提升[19, 38]。
在P. chlororaphis GP72中敲除4个负调控基因pykF、rpeA、rsmE和lon,过表达6个基因ppsA、tktA、phzC、aroB、aroD和aroE (提高莽草酸途径) 后使得2-OH-PHZ的产量由4.5 mg/L提高到450.4 mg/L,比原始菌株提高了99倍[16];通过在P. chlororaphis HT66中敲除3个负调控基因lon、parS和prsA,成功构建了高产菌株HT66LSP,高产菌的PCN产量达到了4.10 g/L,比野生菌株提高了8.6倍[39];在S. bingchenggensis中,通过敲除cyp41基因和过表达milE基因使得米尔贝霉素A3/A4的产量提高了53.1%[40]。在P. aeruginosa PA12O1中通过对毒力因子、PCA生物合成途径、PCA流出泵系统以及次级代谢途径进行组合基因工程,获得了一株遗传稳定、低毒性的高产菌株PA-Ⅳ,其PCA产量为9 882 mg/L,与原始菌株相比提高了54倍[41]。
随着大片段DNA克隆技术的发展,在优化的宿主菌株中进行整个合成基因簇的复制或者异源表达,有望大幅度提高抗生素的产量[39]。目前已有许多抗生素实现了异源表达,这也为抗生素的高产提供了有利条件。通过利用Red/ET同源重组技术在B. amyloliquefaciens FZB42中克隆了杆菌霉素的合成基因簇(37.2 kb),然后将克隆的基因簇整合到枯草芽孢杆菌的染色体上实现了杆菌霉素的异源表达[42];以S. coelicolor M145的衍生物菌株M1152和M1154作为宿主,通过在φC31 att位点分别引入来自S. venezuelae ATCC 10712的氯霉素基因簇和S. ambofaciens ATCC 23877的杀刚果锥虫素基因簇,使氯霉素和杀刚果锥虫素的产量提高了20–40倍[43]。
2.2.2 利用底盘细胞构建高产菌株由于缺乏高效的遗传操作技术、生长速度慢、产量低或易受环境干扰等原因,许多天然微生物并不是抗生素等代谢产物的理想生产对象。因此,在微生物底盘细胞中异源表达天然产物合成途径引起了越来越多的关注。随着系统生物学和合成生物学工具在途径识别、预测和重构方面的发展,一些模式微生物,如大肠杆菌、酿酒酵母、枯草芽孢杆菌、链霉菌等,已被确定为异源表达和大规模生产高价值天然产物的理想底盘。这些底盘细胞的优势在于生长速度较快,其基因组和代谢网络具有很透彻的研究,因此通过不同的合成生物学技术对其进行工程化处理,可以实现天然产物的高效表达[44]。
基因组的精简和优化是构建底盘细胞的重要策略,删减非必需基因不仅可以提高基因组的产物合成稳定性,还可以简化目标代谢网络。例如,通过敲除S. avermiltilis线性染色体9.02 Mb中总长度超过1.4 Mb的非必需基因,构建了阿维链霉菌的“最小基因组”,将这个最小基因组作为底盘对链霉素、头孢霉素C和寡霉素的外源基因簇分别进行了异源表达,使得链霉素和头孢霉素C的产量明显高于原始菌株,也使得寡霉素的产量提高了10倍以上[45]。另外,在P. chlororaphis GP72中通过采用同源重组方法从染色体上敲除685 750 bp的片段(占基因组的10.3%,包括5个非必要的次级代谢基因簇和17个菌株特异性大片段),构建了小基因组菌株MDS22,并使得MDS22的2-羟基吩嗪产量提高了3.4倍[46]。基因组规模的代谢重建以及调控网络分析将会提供更多信息,为构建具有系统简单性和生物技术应用可行性的理想底盘细胞奠定基础。
2.2.3 核糖体工程技术核糖体工程技术是利用核糖体蛋白结构上的突变对微生物次级代谢调控产生影响,进而进行微生物育种的一项技术,目前已成为激活与提高微生物多种重要次生代谢产物的主要技术。通过向核糖体或RNA聚合酶引入特定的抗生素抗性突变,使该菌株核糖体的特定基因发生突变,赋予目的菌株某种抗生素抗性,从而激活次级代谢产物合成,迅速提高抗生素产量。相比经典的微生物育种方法,核糖体工程技术具有产量提高迅速、耗时少、基因突变位点明确且易于检测等优点,可以大幅度提高正向突变菌株的获得率[47]。另外,核糖体工程技术还适用于那些没有明确遗传背景的菌株改造。
利用核糖体工程技术可以有效地提高抗生素的产量。在S. viridochromogenes Co γ-316中,通过基因组重排与核糖体工程相结合的技术(核糖体蛋白S12突变),使得阿维菌素的产量从0.24 g/L增加到1.4 g/L,比原始菌株提高了4.85倍[48];通过基因组重排与核糖体工程相结合对S. actuosus AW7进行改造,使得那西肽的产量增加到1.2 g/L,比亲本菌株AW7高7.0倍[49];通过ARTP和核糖体工程技术相结合的育种方法对S. albus S12进行改造,使得沙利霉素的产量达到了34.7 g/L,比原始菌株提高了1倍多[30]。
基因工程育种可以定向改造微生物,提高目标产物产量,育种周期相对较短;但是该育种方法需要对微生物的代谢网络进行透彻的研究,操作难度较诱变育种大。人们可以根据菌株的遗传背景了解程度和改造难度,选择合适的育种方法。
3 发酵优化提高菌株高产能力微生物源农用抗生素的筛选以及高产菌株生产能力的鉴定一般都是通过发酵实现的,发酵过程会受到各种因素(如营养因素、氧气供应、温度、pH值等) 的影响,进而影响微生物次级代谢产物的合成水平。如在S. hygroscopicus 5008发酵过程中,采用碱性pH冲击使得井冈霉素A的产量提高了27.43%[50];在微生物发酵过程中通过添加适当的氧化剂、还原剂等调节活性氧(Reactive oxygen species,ROS) 水平,也可以起到增加次级代谢物产量的作用。通过在S. hygroscopicus 5008的发酵培养基中添加H2O2,并对其添加浓度和添加时间进行优化,使得井冈霉素A的产量获得了显著的提升[51];在假单胞菌中通过在不同的时间添加二硫苏糖醇(Dithiothreitol,DTT) 和H2O2,运用两步发酵策略使得2-OH-PHZ的产量提高了1.6倍,这些可为其他抗生素的发酵提供借鉴[52]。
传统的发酵优化试验多以单因素试验和正交试验为主,研究耗时、模型指导性较差。近年来,优秀试验设计软件不断推广,大大提高了高产条件的优化研究速度。目前通过使用Plackett-Burman (PB) 设计、Box-Behnken实验设计(Box-Behnken design,BBD)、中心复合设计(Central composite design,CCD)、响应曲面法(Response surface method,RSM) 等统计方法进行发酵参数的优化,使得次级代谢物产量提升的同时也有效地降低了发酵成本,同时还可以采用数学模型指导优化过程。通过对PCA高产菌株铜绿假单胞菌M18MSU1采用部分析因设计(Fractional factorial design,FFD)、最陡爬坡法和CCD进行试验设计并确定优化条件,使得PCA产量达到4 771 mg/L,约是优化前的2倍[53];通过采用PB设计和CCD对绿针假单胞菌P3Δlon菌株进行培养基参数的优化,使得PCN产量提高了3倍左右,其PCN产量达到了9 174 mg/L[15];通过对井冈霉素A的发酵培养基进行优化,并采用RSM进行最优化选择和验证,使得井冈霉素A的产量比优化前提高了近2倍[54]。
随着代谢组学的发展,影响抗生素合成的前体不断得到确定,添加前体或者关键代谢物也是提高抗生素产量的常用手段,在束丝放线菌Actinosynnema pretiosum发酵过程中通过添加前体异丁醇,使得安丝菌素P-3的产量提高了4倍左右[55];S. nodosus可以产生一种多烯大环内酯类抗生素-两性霉素B,在24 h时通过向发酵培养基中添加异丙醇、丙氨酸、丙酮酸和烟酰胺使得两性霉素B的产量提高了28.5%[56]。
4 总结与展望农作物病虫害一直以来都是影响农业生产的重要因素,而化学农药的广泛应用对农业可持续发展和人类健康等产生了一系列的负面影响,高效、低毒、无残留的微生物源农用抗生素具有很好的市场发展前景,能够促进绿色生产,提升农业生产品质。微生物源农用抗生素经过20世纪70年代开始的辉煌,在21世纪初进入发展瓶颈,但基因组学、代谢工程、合成生物学等技术的发展将推动微生物源农用抗生素的再次发展,并推进传统农药产业的结构调整和技术提升,将农业微生物产业带上一个更高的台阶。
| [1] |
Zhang T, Zhang L, Su W, et al. The direct electrocatalysis of phenazine-1-carboxylic acid excreted by Pseudomonas alcaliphila under alkaline condition in microbial fuel cells. Bioresour Technol, 2011, 102(14): 7099-7102. DOI:10.1016/j.biortech.2011.04.093
|
| [2] |
Arseneault T, Filion M. Phenazine-producing Pseudomonas spp. as biocontrol agents of plant pathogens. In microbial inoculants in sustainable agricultural productivity; Springer: New Delhi, 2016: 53-68.
|
| [3] |
Liu CX, Zhang J, Wang XJ, et al. Antifungal activity of borrelidin produced by a Streptomyces strain isolated from soybean. J Agric Food Chem, 2012, 60(5): 1251-1257. DOI:10.1021/jf2044982
|
| [4] |
Kim MS, Cho WJ, Song MC, et al. Engineered biosynthesis of milbemycins in the avermectin high-producing strain Streptomyces avermitilis. Microbial Cell Factories, 2017, 16(1): 9. DOI:10.1186/s12934-017-0626-8
|
| [5] |
Chen J, Wang W, Xu Y, et al. Slow-release formulation of a new biological pesticide, pyoluteorin, with mesoporous silica. J Agric Food Chem, 2011, 59(1): 307-311. DOI:10.1021/jf103640t
|
| [6] |
Zheng X, Cheng Q, Yao F, et al. Biosynthesis of the pyrrolidine protein synthesis inhibitor anisomycin involves novel gene ensemble and cryptic biosynthetic steps. Proc Natl Acad Sci USA, 2017, 114(16): 4135-4140. DOI:10.1073/pnas.1701361114
|
| [7] |
Jin P, Wang H, Tan Z, et al. Antifungal mechanism of bacillomycin D from Bacillus velezensis HN-2 against Colletotrichum gloeosporioides Penz. Pestic Biochem Physiol, 2020, 163: 102-107. DOI:10.1016/j.pestbp.2019.11.004
|
| [8] |
沈艳, 孙如意, 李蕾, 等. 新奥霉素母药对5种非靶标生物的急性毒性. 农药学学报, 2018, 20(4): 535-539. Shen Y, Sun RY, Li L, et al. Acute toxicity study of xinaomycin TK to five non-target organisms. Journal of Pesticide Science, 2018, 20(4): 535-539 (in Chinese). |
| [9] |
Zhou T, Zeng H, Qiu D, et al. Global transcriptional responses of Bacillus subtilis to xenocoumacin 1. J Appl Microbiol, 2011, 111(3): 652-662. DOI:10.1111/j.1365-2672.2011.05086.x
|
| [10] |
Chen YW, Liu XC, Lv FX, et al. Characterization of three regulatory genes involved in enduracidin biosynthesis and improvement of enduracidin production in Streptomyces fungicidicus. J Appl Microbiol, 2019, 127(6): 1698-1705. DOI:10.1111/jam.14417
|
| [11] |
Xu S, Pan X, Luo J, et al. Effects of phenazine-1-carboxylic acid on the biology of the plant-pathogenic bacterium Xanthomonas oryzae pv. oryzae. Pestic Biochem Physiol, 2015, 117(1): 39-46.
|
| [12] |
Zhou L, Jiang HX, Sun S, et al. Biotechnological potential of a rhizosphere Pseudomonas aeruginosa strain producing phenazine-1-carboxylic acid and phenazine-1-carboxamide. World J Microbiol Biotechnol, 2016, 32(3): 50. DOI:10.1007/s11274-015-1987-y
|
| [13] |
Jin XJ, Peng HS, Hu HB, et al. iTRAQ-based quantitative proteomic analysis reveals potential factors associated with the enhancement of phenazine-1-carboxamide production in Pseudomonas chlororaphis P3. Sci Rep, 2016, 6(7): 27393.
|
| [14] |
Peng H, Zhang P, Bilal M, et al. Enhanced biosynthesis of phenazine-1-carboxamide by engineered Pseudomonas chlororaphis HT66. Microb Cell Fact, 2018, 17(1): 117. DOI:10.1186/s12934-018-0962-3
|
| [15] |
Peng H, Tan J, Bilal M, et al. Enhanced biosynthesis of phenazine-1-carboxamide by Pseudomonas chlororaphis strains using statistical experimental designs. World J Microbiol Biotechnol, 2018, 34(9): 129. DOI:10.1007/s11274-018-2501-0
|
| [16] |
Liu K, Hu H, Wang W, et al. Genetic engineering of Pseudomonas chlororaphis GP72 for the enhanced production of 2-hydroxyphenazine. Microb Cell Fact, 2016, 15(1): 131. DOI:10.1186/s12934-016-0529-0
|
| [17] |
He H, Ye L, Li C, et al. SbbR/SbbA, an important ArpA/AfsA-like system, regulates milbemycin production in Streptomyces bingchenggensis. Front Microbiol, 2018, 9: 1064. DOI:10.3389/fmicb.2018.01064
|
| [18] |
Vinay JU, Naik MK, Rangeshwaran R, et al. Detection of antimicrobial traits in fluorescent pseudomonads and molecular characterization of an antibiotic pyoluteorin. 3 Biotech, 2016, 6(2): 1-11.
|
| [19] |
Shi H, Huang X, Wang Z, et al. Improvement of pyoluteorin production in Pseudomonas protegens H78 through engineering its biosynthetic and regulatory pathways. Appl Microbiol Biotechnol, 2019, 103(8): 3465-3476. DOI:10.1007/s00253-019-09732-z
|
| [20] |
Shen J, Kong L, Li Y, et al. A LuxR family transcriptional regulator AniF promotes the production of anisomycin and its derivatives in Streptomyces hygrospinosus var. beijingensis. Synth Syst Biotechnol, 2019, 4(1): 40-48. DOI:10.1016/j.synbio.2018.12.004
|
| [21] |
Zhang S, Fang X, Tang Q, et al. CpxR negatively regulates the production of xenocoumacin 1, a dihydroisocoumarin derivative produced by Xenorhabdus nematophila. MicrobiologyOpen, 2019, 8(2): e00674. DOI:10.1002/mbo3.674
|
| [22] |
Zhou T, Yang X, Qiu D, et al. Inhibitory effects of xenocoumacin 1 on the different stages of Phytophthora capsiciand its control effect on Phytophthora blight of pepper. Bio Control, 2017, 62(2): 151-160. DOI:10.1007/s10526-016-9779-3
|
| [23] |
Balderas-Ruíz KA, Bustos P, et al. Bacillus velezensis 83 a bacterial strain from mango phyllosphere, useful for biological control and plant growth promotion. AMB Express, 2020, 10(1): 1-19. DOI:10.1186/s13568-019-0926-y
|
| [24] |
Gu Q, Yang Y, Yuan Q, et al. Bacillomycin D produced by Bacillus amyloliquefaciens is involved in the antagonistic interaction with the plant-pathogenic fungus Fusarium graminearum. Appl Environ Microbiol, 2017, 83(19).
|
| [25] |
Lin F, Xue Y, Huang Z, et al. Bacillomycin D inhibits growth of Rhizopus stolonifer and induces defense-related mechanism in cherry tomato. Appl Microbiol Biotechnol, 2019, 103: 7663-7674. DOI:10.1007/s00253-019-09991-w
|
| [26] |
Lin F, Huang Z, et al. Effect of combined Bacillomycin D and chitosan on growth of Rhizopus stolonifer and Botrytis cinerea and cherry tomato preservation. J Sci Food Agric, 2020, 101(1): 229-239.
|
| [27] |
Hu Y, Yang W, Wan W, et al. Investigation on ultrasound-assisted extraction and separation of enduracidin from streptomyces sp. NJWGY3665. Appl Biochem Biotechnol, 2012, 166(4): 830-838. DOI:10.1007/s12010-011-9473-y
|
| [28] |
Zhong TH, Zeng XM, Zhang YH, et al. Discovery, gene modification, and optimization of fermentation of an enduracidin-producing strain. J Asian Nat Prod Res, 2018, 20(7): 633-648. DOI:10.1080/10286020.2018.1451517
|
| [29] |
Sun FH, Luo D, Shu D, et al. Development of an intergeneric conjugal transfer system for xinaomycins-producing Streptomyces noursei Xinao-4. Int J Mol Sci, 2014, 15(7): 12217-12230. DOI:10.3390/ijms150712217
|
| [30] |
Zhang K, Mohsin A, Dai Y, et al. Combinatorial effect of ARTP mutagenesis and ribosome engineering on an industrial strain of Streptomyces albus S12 for enhanced biosynthesis of salinomycin. Front Bioeng Biotechnol, 2019, 7: 212. DOI:10.3389/fbioe.2019.00212
|
| [31] |
Zhang X, Zhang XF, Li HP, et al. Atmospheric and room temperature plasma (ARTP) as a new powerful mutagenesis tool. Appl Microbiol Biotechnol, 2014, 98(12): 5387-96. DOI:10.1007/s00253-014-5755-y
|
| [32] |
谭剑, 熊欣, 梁万利, 等. ARTP技术选育吩嗪-1-甲酰胺高产菌株及发酵优化. 生物技术通报, 2016, 32(1): 174-179. Tan J, Xiong X, Liang WL, et al. Breeding of a phenazine-1-carboxamid-producing strain by ARTP mutation and its optimization of fermentation. Biotechnol Info, 2016, 32(1): 174-179 (in Chinese). |
| [33] |
Wang LY, Huang ZL, Li G, et al. Novel mutation breeding method for Streptomyces avermitilis using an atmospheric pressure glow discharge plasma. J Appl Microbiol, 2010, 108(3): 851-858. DOI:10.1111/j.1365-2672.2009.04483.x
|
| [34] |
Wang HY, Zhang J, Zhang YJ, et al. Combined application of plasma mutagenesis and gene engineering leads to 5-oxomilbemycins A3/A4 as main components from Streptomyces bingchenggensis. Appl Microbiol Biotechnol, 2014, 98(23): 9703-9712. DOI:10.1007/s00253-014-5970-6
|
| [35] |
Liu L, Hu W, Li W, et al. Heavy-ion mutagenesis significantly enhances enduracidin production by Streptomyces fungicidicus. Eng Life Sci, 2019, 19(2): 112-120. DOI:10.1002/elsc.201800109
|
| [36] |
江耀祖, 彭华松, 张雪洪. 基于ARTP诱变诱变和高通量筛选的绿针假单胞菌GP72育种方法. 微生物学通报, 2017, 44(10): 2421-2427. Jiang YZ, Peng HS, Zhang XH. Breeding of Pseudomonas chlororaphis GP72 based on ARTP mutagenesis and high throughput screening. Microbiol China, 2017, 44(10): 2421-2427 (in Chinese). |
| [37] |
金鸣, 王威, 张雪洪. 高产洛蒙真菌素洛蒙德链霉菌高通量筛选方法的建立与复合育种. 微生物学通报, 2020, 47(1): 200-209. Jin M, Wang W, Zhang XH. High throughput screening and combined breeding of Streptomyces lomondensis for high production of lomofungin. Microbiol China, 2020, 47(1): 200-209 (in Chinese). |
| [38] |
Li D, Zhang J, Tian Y, et al. Enhancement of salinomycin production by ribosome engineering in Streptomyces albus. Sci China Life Sci, 2019, 62(2): 276-279. DOI:10.1007/s11427-018-9474-7
|
| [39] |
Yao R, Pan K, Peng H, et al. Engineering and systems-level analysis of Pseudomonas chlororaphis for production of phenazine-1-carboxamide using glycerol as the cost-effective carbon source. Biotechnol Biofuels, 2018, 11(1): 130. DOI:10.1186/s13068-018-1123-y
|
| [40] |
Wang H, Cheng X, Liu Y, et al. Improved milbemycin production by engineering two Cytochromes P450 in Streptomyces bingchenggensis. Appl Microbiol Biotechnol, 2020, 104: 2935-2946. DOI:10.1007/s00253-020-10410-8
|
| [41] |
Jin K, Zhou L, Jiang H, et al. Engineering the central biosynthetic and secondary metabolic pathways of Pseudomonas aeruginosa strain PA1201 to improve phenazine-1-carboxylic acid production. Metab Eng, 2015, 32: 30-38. DOI:10.1016/j.ymben.2015.09.003
|
| [42] |
Liu Q, Shen Q, Bian X, et al. Simple and rapid direct cloning and heterologous expression of natural product biosynthetic gene cluster in Bacillus subtilis via Red/ET recombineering. Sci Rep, 2016, 6: 34623. DOI:10.1038/srep34623
|
| [43] |
Gomez-Escribano JP, Bibb MJ. Engineering Streptomyces coelicolorfor heterologous expression of secondary metabolite gene clusters. Microb Biotechnol, 2011, 4(2): 207-215. DOI:10.1111/j.1751-7915.2010.00219.x
|
| [44] |
Xu X, Liu Y, Du G, et al. Microbial chassis development for natural product biosynthesis. Trends Biotechnol, 2020, 38(7): 779-796. DOI:10.1016/j.tibtech.2020.01.002
|
| [45] |
Komatsu M, Uchiyama T, Ōmura S, et al. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism. Proc Nat Acad Sci USA, 2010, 107(6): 2646-2651. DOI:10.1073/pnas.0914833107
|
| [46] |
Shen X, Wang Z, Huang X, et al. Developing genome-reduced Pseudomonas chlororaphis strains for the production of secondary metabolites. BMC Genomics, 2017, 18(1): 715. DOI:10.1186/s12864-017-4127-2
|
| [47] |
Zhu S, Duan Y, Huang Y. The application of ribosome engineering to natural product discovery and yield improvement in Streptomyces. Antibiotics, 2019, 8(3): 133. DOI:10.3390/antibiotics8030133
|
| [48] |
Lv XA, Jin YY, Li YD, et al. Genome shuffling of Streptomyces viridochromogenes for improved production of avilamycin. Appl Microbiol Biotechnol, 2013, 97(2): 641-648. DOI:10.1007/s00253-012-4322-7
|
| [49] |
Wang Q, Zhang D, Li Y, et al. Genome shuffling and ribosome engineering of Streptomyces actuosus for high-yield nosiheptide production. Appl Biochem Biotechnol, 2014, 173(6): 1553-1563. DOI:10.1007/s12010-014-0948-5
|
| [50] |
Jiang J, Sun YF, Tang X, et al. Alkaline pH shock enhanced production of validamycin A in fermentation of Streptomyces hygroscopicus. Bioresour Technol, 2018, 249: 234-240. DOI:10.1016/j.biortech.2017.10.012
|
| [51] |
Wei ZH, Bai L, Deng Z, et al. Enhanced production of validamycin A by H2O2-induced reactive oxygen species in fermentation of Streptomyces hygroscopicus 5008. Bioresour Technol, 2011, 102(2): 1783-1787. DOI:10.1016/j.biortech.2010.08.114
|
| [52] |
Yue SJ, Huang P, Li S, et al. Enhanced production of 2-hydroxyphenazine from glycerol by a two-stage fermentation strategy in Pseudomonas chlororaphis GP72AN. J Agric Food Chem, 2019, 68(2): 561-566.
|
| [53] |
Du X, Li Y, Zhou W, et al. Phenazine-1-carboxylic acid production in a chromosomally non-scar triple-deleted mutant Pseudomonas aeruginosa using statistical experimental designs to optimize yield. Appl Microbiol Biotechnol, 2013, 97(17): 7767-7778. DOI:10.1007/s00253-013-4921-y
|
| [54] |
Fan Y, Yu Y, Jia X, et al. Cloning, expression and medium optimization of validamycin glycosyltransferase from Streptomyces hygroscopicus var. jinggangensis for the biotransformation of validoxylamine A to produce validamycin A using free resting cells. Bioresour Technol, 2013, 131: 13-20. DOI:10.1016/j.biortech.2012.12.021
|
| [55] |
Jia Y, Zhong JJ. Enhanced production of ansamitocin P-3 by addition of isobutanol in fermentation of Actinosynnema pretiosum. Bioresour Technol, 2011, 102(21): 10147-10150. DOI:10.1016/j.biortech.2011.08.031
|
| [56] |
Zhang B, Zhang YH, Chen Y, et al. Enhanced AmB Production in Streptomyces nodosus by fermentation regulation and rational combined feeding strategy. Front Bioeng Biotechnol, 2020, 8: 597. DOI:10.3389/fbioe.2020.00597
|
 2021, Vol. 37
2021, Vol. 37




