中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈涛, 崔真真, 户文亚, 王智文, 赵学明
- Chen Tao, Cui Zhenzhen, Hu Wenya, Wang Zhiwen, Zhao Xueming
- 代谢工程发展30年
- Thirty years development of metabolic engineering: a review
- 生物工程学报, 2021, 37(5): 1477-1493
- Chinese Journal of Biotechnology, 2021, 37(5): 1477-1493
- 10.13345/j.cjb.200739
-
文章历史
- Received: November 22, 2020
- Accepted: February 19, 2021
- Published: February 23, 2021
2. 系统生物工程教育部重点实验室 合成生物学前沿科学中心,天津 300072;
3. 天津化学化工协同创新中心,天津 300072
2. Frontier Science Center for Synthetic Biology, Key Laboratory of Systems Bioengineering, Ministry of Education, Tianjin 300072, China;
3. Collaborative Innovation Center of Chemical Science and Engineering, Tianjin 300072, China
1991年Bailey[1]和Stephanopoulos等[2]提出代谢工程的概念,标志着代谢工程学科的诞生,其研究过程通常包括分析、合成和表征3个循环步骤。代谢工程近30年的发展先后与分子生物学、系统生物学和合成生物学发生了深度的交叉融合。代谢工程在诞生之初就是一个与分子生物学交叉的学科,但它不仅仅是分子生物学的技术体现,在随后的发展中,其重点聚焦于通过代谢控制分析和代谢通量分析确定产物合成途径的关键节点和优化靶点,然后通过重组DNA技术针对靶点定向改进菌株[3]。20个世纪90年代中期,随着功能基因组学和系统生物学的发展,大量微生物全基因组序列的测定和基因功能的解析注释为基因组尺度代谢网络的构建奠定了基础,研究者可以在整体代谢的系统水平上研究微生物的代谢网络特征、模拟优化代谢途径。与此同时,转录组、蛋白质组、代谢物组和通量组等各种高通量组学分析技术的涌现使得研究者从多个层次系统地解析微生物的代谢特征成为可能。这些系统生物学的研究工具显著提高了代谢工程循环步骤中的分析、表征能力和确定改进靶点的准确度[4]。近10年来,代谢工程与合成生物学的深度交叉融合又为其发展提供了新的推动力。DNA组装、基因组编辑、基因元件和基因调控线路的设计、蛋白支架等合成生物技术的涌现极大地丰富了代谢工程改造微生物细胞的策略和工具,尤其是显著提高了代谢工程循环步骤中的合成能力。代谢工程正以前所未有的深度和广度促进生物技术产业的升级和进步。本文从SCI论文发表的角度总结了代谢工程成立30年以来的学术研究情况,重点介绍了近10年来系统生物学方法和DNA组装、基因组编辑、基因表达调控等重要使能技术在代谢工程中的应用,并探讨了目前代谢工程发展中存在的主要问题和今后的发展趋势。
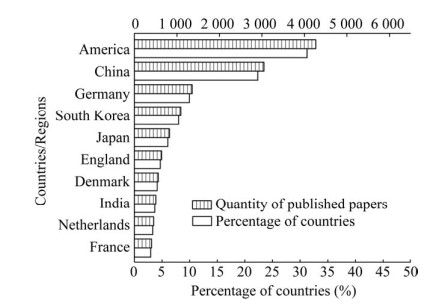
1 代谢工程30年的学术研究现状图 1显示了代谢工程学科成立30年以来本领域SCI论文的年度发文量和中美两国的论文占比情况,从中可以看出代谢工程研究一直处于持续活跃与发展之中。1991–2000年期间,年平均发文增长量约10篇/年,而在2001–2010年和2011–2019年期间,这一数据分别增加为约28篇/年和108篇/年,增长速率显著加快。近30年来代谢工程研究活跃的国家包括美国、中国、德国、韩国、日本等国家,其中美国的发文量占比高达31.3%,是本领域研究贡献度最大的国家,我国发文量排名第二,占比达到22.3% (图 2)。从年度发文量来看,美国长期位居首位,但其发文量占比从1993年的91.7%持续下降至2020年的22.6%,而我国在1991–1998年的发文量为0,发文量占比从1999年的约1%持续攀升为2020年的38.9%,发文量在2018年首次超过美国而位居第一(图 1),由此可见我国科研工作者已经成为代谢工程领域研究的主力军。

|
|
图 1 1991–2020年代谢工程领域SCI论文发表情况
Fig. 1 Papers published in SCI-indexed journals in the field of metabolic engineering from 1991 to 2020.
|
| |
|
|
图 2 1991–2020年代谢工程领域SCI论文发表主要国家
Fig. 2 Ranking of major countries according to the number of papers published in the field of metabolic engineering from 1991 to 2020.
|
| |
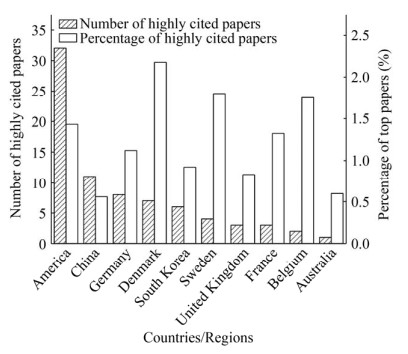
图 3显示了2010–2020年代谢工程领域高被引研究论文(不包括综述论文) 的分布情况,虽然我国的高被引研究论文数量在国际上排名第二,但是和排名第一的美国相比,仍然有明显差距。值得注意的是,这一时期我国发表的SCI论文中高被引研究论文仅占0.56%,在高被引研究论文发文量前十的国家中排名末位,而欧美等国家这一数据普遍在1%–2%之间(图 3),这表明我国代谢工程研究相对缺乏高影响力的原创性研究成果,研究的总体水平仍然有很大的提升空间。
|
|
图 3 2010–2020年高被引论文发表情况
Fig. 3 Number and percentage of highly cited papers of major countries from 2010 to 2020.
|
| |
系统生物学方法主要包括基因组尺度代谢网络模型(Genome-scale metabolic models,GEMs)的构建与模拟分析以及各种组学方法,这些方法既可以单独应用也可以相互整合后应用于代谢工程,不仅能进一步拓展研究者对生物系统的认知,还能分析确定一些和代谢途径没有直接关联或难以通过直觉发现的潜在改进靶点。
2.1 基因组尺度代谢网络模型GEMs是指导代谢工程进行理性设计的有力工具,在细胞生理和表型预测、生物合成途径优化设计、代谢工程靶点预测等方面发挥着重要的作用[5]。近10年来,ModelSEED、RAVEN (Reconstruction,analysis and visualization of metabolic networks)、CarveME (Carve metabolic engineering) 等一系列模型构建工具的开发有力推动了GEMs的构建,目前已发表了100多个物种的近400个GEMs[6],但这些模型的构建标准和质量参差不齐,亟需建立统一的模型构建和质量评价标准。为此,来自包括中国在内的15个国家的研究者于2020年提出了一套由社区维护的标准化代谢模型质量自动测试流程MEMOTE,用户可利用该工具提交模型并生成质量测试报告,根据MEMOTE检测结果修正模型,最终得到高质量的GEMs[7]。仅仅由化学反应计量关系约束的GEMs由于缺少细胞内酶量、热力学、动力学、应急响应等约束信息,无法预测出如生长延迟、代谢溢流等特定的生物学现象[8]。为了扩展模型的功能,研究者在计量学模型的基础上进行了很多有益的改进:2017年,Nielsen团队报道了在酿酒酵母Yeast7模型中引入酶约束的GECKO方法,构建的酶约束ecYeast7模型可成功预测乙醇代谢溢流现象,对于不同培养基中的生长模拟也明显优于Yeast7模型[9]。为了进一步通过增加约束条件来更精准地描述细胞过程,研究者还开发了整合代谢、转录、信号转导和多组学数据等多层次的全细胞模型,目前构建全细胞模型的菌株有海栖热袍菌[10]、大肠杆菌[11]和永达尔梭菌[12]。此外,代谢工程研究中常采用扩展模型解空间的方式来预测新途径,通过在某物种的代谢网络模型中引入异源反应来扩展模型,从而预测新途径。如Chatsurachai等[13]将7 769个异源反应分别引入大肠杆菌、谷氨酸棒杆菌和酿酒酵母的模型中构建了扩展模型,利用这些模型可以预测和评估任意异源产品在不同宿主中的合成能力。
高质量GEMs及开发一系列模拟算法已成功用于指导代谢工程改造微生物细胞合成聚-3-羟基丁酸酯[14]、丙酮[15]、苯甲酸[16]和赖氨酸[17]等众多生物基产品。其用途之一是途径分析,以确定产物合成效率最优或次优的生物合成途径。例如:Borodina等利用酿酒酵母iTO977模型分析确定通过β-丙氨酸途径合成3-羟基丙酸(3-hydroxypropionic acid,3-HP) 的效率高于丙二酰辅酶A途径[18]。随后的实验表明通过β-丙氨酸途径合成3-HP的工程菌在碳源限制流加发酵中的产量和得率分别为21 g/L和15.9% C-mol[19],均优于通过丙二酰辅酶A途径合成3-HP的工程菌(9.8 g/L和13% C-mol)[20]。2020年,Luo等[16]利用大肠杆菌iML1515模型分析了5条不同苯甲酸合成途径的理论得率分别为0.451、0.441和0.419 g苯甲酸/g葡萄糖(其中次优途径有3条),由于缺乏最优途径的相关基因信息,他们在大肠杆菌中分别导入了一条得率次优和得率最低的途径,得到的工程菌株分别可产2.37 g/L和0.18 g/L的苯甲酸表明高得率的途径在苯甲酸合成方面的优势。高质量GEMs及模拟算法的另一个主要用途是预测有利于产物合成的基因敲除或过表达靶点。近期,Ye等利用GECKO方法在大肠杆菌iML1515模型基础上构建了酶约束模型ec_iML1515,通过该模型模拟预测出提高赖氨酸合成效率时需求最多的20种酶,随后的基因过表达证实其中9个靶点具有显著效果(产量均提升约30%以上),对效果最好的lysC和fre进行协调共表达后,赖氨酸产量提升了169.1%,达到95.7 g/L[17]。
2.2 转录组学转录组学是分析测定特定条件下细胞内所有基因的转录水平,从而在全局水平上得到细胞对环境或基因扰动的转录响应信息。早期的转录组研究主要基于DNA芯片技术,近年来随着高通量深度测序技术的飞速发展,成本低、精度高及RNA分析种类多的各种RNA测序技术(RNA-seq) 已经成为转录组研究的主要方法[21]。
通过转录组的比较分析可以帮助研究者确定和特定表型或功能相关的未知基因以及重要基因的转录调控或变化规律,并由此确定潜在的代谢工程操作靶点。例如,Chung等[22]发现在培养基里添加0.25 mol/L琥珀酸时导致谷氨酸棒杆菌的琥珀酸生产速度和底物消耗速度分别降低了98.2%和66.4%,显示了严重的产物抑制现象。他们通过比较分析转录组数据发现:琥珀酸的添加导致9个转录调控基因以及中心碳代谢的4个基因转录水平显著下调。随后分别过表达这些基因以鉴定它们和产物抑制现象的相关性,结果发现过表达转录调控基因NCgl0275后几乎完全消除了产物抑制现象,相应菌株的琥珀酸产量达到55.4 g/L,比对照菌株提高了37.7%,琥珀酸的得率和产率也均提高了40%左右。需要注意的是,由于细胞内存在多种转录后的调控过程,转录组数据不一定显示出与细胞代谢活性的相关性,单独基于转录组数据确定代谢工程的修饰靶点时需要慎重的评估和解释[23]。
2.3 蛋白质组学蛋白质组学是测定特定条件下细胞的全部蛋白质表达水平,并能提供蛋白质的翻译后修饰、蛋白质之间的相互作用等信息。其分析技术主要包括二维凝胶电泳、毛细管电泳和高效液相色谱等分离技术和串联质谱等鉴定技术,其中多维液相分离技术不仅能和质谱在线串联进行高通量分析,还能显著提高分离峰的峰容量及对低丰度蛋白质的分析能力,因此近年来已成为蛋白质组研究中的主要分离技术[24]。Ren等[25]开发了一种将1根强阳离子交换柱(Ion-Exchange,IEX,第一维) 和12根并行的反向色谱柱(Reverse-phase liquid chromatography,RPLC,第二维) 串联装配的多维色谱分离技术,利用该技术分离大肠杆菌裂解液里的完整蛋白质,可得到900多个分离峰。Huang等[26]开发一种基于阵列的二维SAX-RPLC系统,利用该系统从人血浆里鉴定出浓度范围在0.01 ng/mL到41 mg/mL之间的1 332种蛋白质。此外,近期一些三维或四维分离技术也相继开发出来,在蛋白质糖基化、磷酸化修饰位点分析中显示了强大的功能[27-28]。
在代谢工程的应用方面,借助蛋白质组研究可以确定产物合成途径中众多关键酶的表达量,并从中找出影响产物合成的限速反应,这种策略已经成功应用于提高菌株生产青蒿素前体紫穗槐二烯[29]、聚3-羟基丁酸酯[30]、柠檬烯和没药烯[31]、银松素[32]和l-鸟氨酸[33]等产物的能力中。此外,通过蛋白质组研究还能发现环境或基因扰动对蛋白质翻译后修饰的影响,并确定相应的代谢工程改造策略。其中一个典型的例子是近期Xu等[32]的工作:他们发现在培养基中添加浅蓝菌素(脂肪酸合成的抑制剂),虽然可以增加银松素合成前体物丙二酰辅酶A的供给,但工程菌株的银松素产量反而下降;随后通过蛋白质组分析发现浅蓝菌素的添加导致胞内丙二酰辅酶A的浓度增加,因此激发了胞内众多蛋白质的丙二酰化,银松素合成途径中的4-香豆酸连接酶和对二苯乙烯合成酶酰基化后对酶活产生了显著的抑制,从而不利于其合成;随后,他们将对二苯乙烯合成酶中的两个酰基化位点(均为赖氨酸残基) 突变为精氨酸以避免酰基化,使得突变株的银松素产量提高了1.2倍。
2.4 代谢物组学代谢物组学是对特定条件下细胞产生的低分子量代谢物全面的定量分析,其分析技术通常涉及气相色谱、液相色谱和毛细管电泳等分离技术和质谱鉴定技术的联用。通常,单根色谱柱很难分离所有的代谢物种类,而不同色谱柱的并联或连续串联使用能大幅度提升对代谢物组的覆盖度。此外,近来开发的包括超高液相色谱分析(通常作为第二维) 在内的二维液相、二维气相和超临界流体色谱技术都可以显著提高代谢组分析的解析度和灵敏度[34]。
代谢物组研究揭示各种代谢物响应环境或遗传扰动的规律,由于代谢物浓度和细胞的代谢活性直接关联,因此通过分析菌株中某一途径的代谢物水平的变化趋势可以确定途径中的限速反应。目前,代谢组学研究已经在醇类[35-36]、琥珀酸[37]、酪氨酸[38]等生物基产品的菌株改进、增强菌株的底物利用能力[39]以及对醇类和酸类的耐受性[40-41]等方面得到了成功的应用。例如,Ohtake等[35]对产丁醇工程菌株进行了代谢组分析,发现pta基因敲除虽然可减少副产物乙酸的积累,但会造成细胞内正丁醇合成途径中辅酶A类代谢物的积累及游离辅酶A浓度的降低,同时检测到副产物丙酮酸和丁酸甲酯产量显著提高,这些信息表明胞内辅酶A循环的不平衡限制了丁醇的生产,他们据此提高丁酰辅酶A还原酶的表达水平以增加辅酶A循环的效率,使得丁醇的积累量从3.9 g/L提高到7 g/L以上。此外,基于代谢组分析的结果,可以调整培养基成分以补偿胞内缺乏的代谢物,从而增加产物的合成。例如上述Ohtake等的研究中也采取了在培养基中添加l-甲硫氨酸以增加胞内辅酶A供给的策略,丁醇的产量得到进一步的提高[35]。Hasunuma等[40]通过代谢组分析,发现虽然在集胞藻中过表达了磷酸烯醇式丙酮酸羧化酶,但其生产琥珀酸的限速反应仍然在C3到C4的回补反应,他们通过提高培养基中碳酸氢钠的浓度提高回补反应效率,使其琥珀酸的产量从约0.22 g/L提高至1.8 g/L。
2.5 通量组学通量组学是通过代谢通量分析(Metabolic flux analysis,MFA) 确定特定条件下细胞内的通量分布特征。精确的通量定量分析需要13C同位素标记实验、质谱和核磁共振分析技术以及相应的计算分析软件包的综合运用,通常能获得100–150个中心碳代谢反应的通量信息。近期已有研究者开发了机器人系统用于高通量的自动化通量组分析[42]。在各种组学研究方法中,通量组和细胞表型的紧密程度最高,已经发展成为指导代谢工程中一个关键方法[23]。除了分析限定培养基中稳态生长的生物系统之外,研究者还相继开发了动态MFA (Dynamic MFA,DMFA)、非生长态的MFA、复杂培养体系的MFA等方法,研究对象也从异养微生物扩展到自养微生物和微生物菌群[43]。
在代谢工程应用方面,通量组数据可以为基因组尺度代谢模型的模拟预测提供更多的约束条件,以提高模型模拟的精度。例如,Gonzalez等[44]分析了大肠杆菌分别利用葡萄糖和木糖在好氧和厌氧条件下生长时的通量组,不仅精确解析了大肠杆菌在4种培养条件下的不同代谢特征,还为代谢通量分析工具包COBRA的改进提供了重要参考数据。通量组研究还能发现新的反应、途径和酶功能,并为菌株的代谢工程改进提供重要参考依据。一个典型的研究案例是近期Lange等[45]的工作,他们分析了产琥珀酸的巴斯夫菌利用蔗糖发酵时的代谢通量,发现蔗糖通过蔗糖磷酸转移系统(Phosphotransferase system,PTS) 进入胞内后分解成6-磷酸-葡萄糖和果糖,其中约60%的果糖被分泌到周质空间后经PTS系统重新进入细胞内代谢(变为1-磷酸-果糖),约40%的果糖被一种新发现的果糖激酶RbsK催化成6-磷酸-果糖后进行代谢,通量数据表明蔗糖和大部分果糖通过PTS系统进入细胞,导致大量丙酮酸衍生副产物的生成而琥珀酸的得率仅有1 mol/mol。随后,他们采取了过表达RbsK和敲除果糖PTS系统的策略,大幅度减少了副产物积累,其中果糖PTS系统敲除菌株在流加发酵中琥珀酸产量达71 g/L,得率显著提高至2.5 mol/mol。
3 合成生物学的主要使能技术 3.1 CRISPR/Cas基因编辑技术基因编辑技术对后基因组时代的基因功能研究和菌株的代谢工程改造起着关键作用。在众多基因组编辑技术中,CRISPR/Cas系统在操作便捷性、编辑效率、成本和通用性等方面具有明显的优势,已成为目前主流的基因编辑方法[46]。
3.1.1 CRISPR/Cas基因编辑系统的研究进展目前已经在众多模式微生物和一些非模式微生物中成功开发了CRISPR/Cas基因编辑系统,其中典型模式微生物的单位点编辑效率几乎都达到了90%以上[47-48],但是也存在一些亟需解决的问题。酿脓链球菌Streptococcus pyogenes Cas9蛋白(SpCas9) 在某些宿主中断裂基因组后会由于重组修复效率较低而产生毒性,而使用仅切割单链DNA的Cas9切口酶(Cas9 nickase,Cas9n) 可以避免该问题,其在编辑过程中产生的DNA切口更容易修复而减少宿主细胞的生长压力,并能降低脱靶几率[49],目前利用Cas9n在多个物种中实现了高效的基因编辑[50-52]。CRISPR-Cas系统的脱靶效应可能导致意外的DNA突变,从而影响编辑的效率和准确度,除了使用Cas9n之外,通过生物信息学工具理性设计的gRNA[53-54]、使用截短的sgRNA[55]以及在5′端引入两个未配对的鸟嘌呤核苷酸[56]都可以显著弱化脱靶效应。基因组的多位点共编辑是高效调控多基因的表达、构建多基因靶点组合库的重要手段。近期,酿酒酵母中的多位点编辑取得了进一步的突破,Zhang等利用tRNA序列将多个gRNA串联后构建gRNA表达质粒,可在7 d内实现8个基因的共编辑,效率达到87%。将质粒组装反应液和修复DNA共转化酿酒酵母,则可在3 d内共编辑6个基因,效率达到60%,为目前报道的最高水平[57]。但在其他物种中,多位点的共编辑效率偏低仍然是一个需要解决的问题。在CRISPR/Cas系统中,还有另一种Type Ⅴ-A型Cas12a蛋白也在基因编辑中发挥着重要的作用,近几年研究者先后针对大肠杆菌、鼠伤寒耶尔森菌和耻垢分枝杆菌[58]、谷氨酸棒状杆菌[59]、链霉菌[60]、解脂耶氏酵母[61]成功开发了CRISPR/Cas12a编辑系统,表明了Cas12a在原核微生物中同样具有相对广泛的适用范围。在链霉菌[60]、谷氨酸棒状杆菌[59]等菌株中表达Cas9时会带来较大的毒性而影响编辑效果,而表达Cas12a蛋白可以避免这种影响。另外,由于Cas12a是由单一的CRISPR RNA (crRNA) 引导,不需要反式激活crRNA (tracrRNA),对pre-crRNA处理具有独特的RNase活性,且Cas12a的crRNA比Cas9的sgRNA更短[62],这些优势使得Cas12a在多基因调控上的表现优于Cas9,已经在细菌、植物和哺乳动物中得到了成功的应用[61, 63-64]。此外,CRISPR/Cas系统经过改造也可用于单碱基编辑[65-67],Zhao等近期通过改造现有的单碱基编辑工具胞嘧啶核苷碱基编辑(Cytidine base editors,CBE),构建出适用于哺乳动物细胞的介导C-G碱基颠换的新工具糖苷酶碱基编辑(Glycosylase base editors,GBE),以及适用于微生物的介导C-A碱基对颠换的新工具[67]。由于通过适应性进化或诱变育种获得的优良菌株中含有的突变大多为单碱基突变,这一系列新工具的出现为工业微生物的基因型-表型关系分析和反向代谢工程研究提供了新的助力。
3.1.2 CRISPR/Cas基因编辑系统的应用CRISPR/Cas基因编辑系统在代谢工程方面的应用通过以下几个典型的案例说明。(1) 组合代谢工程:2015年,Jakounas等利用开发的CRISPR/Cas9系统针对甲羟戊酸合成途径中的5个相关基因进行共编辑(4个敲除和1个下调表达),一次性得到含有5个修饰位点全排列组合的31个菌株,并从中筛选到甲羟戊酸产量比野生型提高41倍的突变株[68]。(2) 大规模基因修饰迭代组合和模块优化:Li等优化CRISPR/Cas9系统后在大肠杆菌中针对β-胡萝卜素合成途径、MEP途径和中心碳代谢途径3个模块中的33个基因靶点进行了迭代组合测试,从构建的103株具有不同靶点组合的菌株中得到的最优菌株,在流加发酵中可产2.0 g/L的β-胡萝卜素[69]。(3) 高通量基因型-表型关联分析:Gill团队开发了可追踪基因型-表型关系的CRISPR可追踪基因组工程(CRISPR Enabled Trackable Genome Engineering,CREATE)系统[70],该系统利用CRISPR/Cas针对基因组上的众多位点构建菌株的饱和突变库,通过适应性进化或高通量筛选富集具有优良表型的菌株群体,随后通过对群体中CREATE质粒的深度测序来追踪与优良表型相关的基因突变。他们利用改进的迭代CREATE (Iterative CREATE,iCREATE) 在大肠杆菌中针对115个基因制备突变库,从162 000个突变株中鉴定出有利于3-羟基丙酸生产的主要突变,将这些突变依次导入野生菌株中后可产30 g/L的3-羟基丙酸,比出发菌株高约60倍[71]。这些研究实例充分展现CRISPR/Cas系统在代谢工程菌株构建中的技术优势。
3.2 DNA组装技术DNA组装是合成生物学和代谢工程学最重要的基础技术之一。高效、高保真、模块化、流程简单、成本低廉的DNA组装技术在快速构建DNA元件、模块或途径库及长合成途径的组装方面具有重要用途。
3.2.1 DNA组装技术的研究进展根据组装的机制不同,DNA组装方法可分为以下几类:基于限制酶的方法(BioBrickTM、BglBrick、epathBrick、MASTER和Golden Gate等),基于体内(DNA assembler、TAR和CasHRA等) 和体外(OE-PCR、SLiCE、CPEC、Gibson、SIRA和TPA等) 序列同源性的方法,以及基于寡核苷酸桥接的方法(Ligase cycling reaction,LCR等)[72-73]。其中的一步组装技术大都能高效将10个以内的DNA片段一次性无痕组装在一起(总长一般小于10 kb)。近期Liang等[74]开发TPA (Twin-primer assembly) 技术能以80%的保真度一步或二步无痕组装10个小DNA片段(总长7 kb),以50%的保真度无痕组装5个较大的DNA片段(总长达31 kb)。Taylor等[75]开发了一种无痕且适合多元件组装的起始-终止组装技术(Start-stop assembly),该技术可将60种元件和质粒载体组装成含15个表达单元(各单元均由启动子、RBS序列、CDS和终止子4类元件组成) 的表达质粒,可实现超长途径的构建及表达单元的组合优化。
3.2.2 DNA组装技术在代谢工程中的应用DNA组装技术在代谢工程中通常用于长途径的基因组装和组合途径优化。例如:Galanie等[76]将阿片类药物蒂巴因和氢可酮合成途径的相关基因(21个异源基因和2个酵母基因) 用Gibson和DNA assembler技术组装成数个模块后插入酵母染色体上或质粒上表达,成功构建了合成上述两种药物的工程菌。Fang等[77]采用传统酶切连接和Gibson、Golden Gate等组装法将维生素B12合成途径中的28个异源基因组装成5个模块后导入大肠杆菌中表达,首次实现了维生素B12在大肠杆菌中的从头合成。Taylor等[75]将起始-终止组装技术应用到虾青素合成途径的组合优化中,他们将启动子和RBS元件(各6种) 以及途径中8个基因的CDS元件按照4基因操纵子-单顺反子-3基因操纵子的结构组装,得到含611种组合的途径库(可在3个位点组装不同的启动子以及8个位点组装不同的RBS),将该库导入大肠杆菌后以番茄红素含量为标准进行表征,显示了良好的多样性分布(含量在0–108 mg/g DCW之间),充分显示了该DNA组装技术在途径组合中的高效性。
3.3 基因表达调控技术反义RNA (Antisense RNA,asRNAs)[78]、小RNA (Small RNA,sRNAs)[79]以及CRISPR-dCas9[80]等基因表达调控技术可以同时调控代谢途径中多个基因的表达水平以实现通量优化,并通过对调控序列的设计实现表达水平的微调,这比直接更换启动子和RBS等表达元件要更为便捷和高效。
3.3.1 asRNA技术及应用asRNA是指能与特定DNA或RNA互补结合的RNA片段,最早是在大肠杆菌产肠杆菌素的ColE1质粒中发现的[81]。天然的asRNA广泛存在于原核和真核生物中,在包括转录、RNA编辑、转录后调节、翻译等多个水平上调控基因的表达。asRNA技术是借助基因重组技术,根据碱基互补原理,用人工合成或生物体合成的特定RNA片段抑制或封闭目标基因表达的技术,可以通过优化asRNA的长度、结构和浓度等参数来调节基因的表达效率。近年来,asRNA技术在大肠杆菌[82]、丙酮丁醇梭菌[83]、鼠李糖乳杆菌[84]和丝状真菌[85]等多种微生物中得到运用。Papoutsakis等[83]利用asRNA技术调控丙酮丁醇梭菌中基因的表达,通过构建不同长度的asRNA来下调磷酸转丁基酶和丁酸激酶的表达,最后使得丙酮和丁醇的浓度分别降低了96%和75%。Wang等[85]利用asRNA技术对丝状真菌的木糖代谢途径进行改造,有效地抑制了木糖醇代谢的关键酶(木糖醇脱氢酶) 的表达,抑制率达到65%,最终木糖醇的积累量达到2.37 g/L,比初始菌株高5倍。大量的数据表明,asRNA技术能够有效地抑制基因表达,甚至可以达到基因敲除的效果。
3.3.2 小RNA技术及应用小RNA (sRNA) 是一种天然存在于细菌中的基因表达调控元件,通过靶标mRNA的碱基互补配对结合来抑制或激活其靶标基因的表达。研究者基于天然sRNA的结构设计构建了合成的sRNA用于抑制靶标基因的表达水平,其序列包括靶标mRNA结合域和伴侣蛋白Hfq的结合域,Hfq和sRNA结合后进一步促使RNase E降解与sRNA结合的靶标mRNA。Yoo等[79]首次详细介绍了大肠杆菌中合成sRNAs的设计原则和构建步骤,随后人工合成的sRNAs调控技术在谷氨酸棒杆菌、枯草芽孢杆菌等多种模式微生物中得到运用,成功优化了氨基酸[86-87]、琥珀酸[88]、甲羟戊酸[89]、N-乙酰葡糖胺[90]、丁醇[91]和苯酚[92]等生物基产品的合成途径。Noh等[93]针对腐胺合成途径中的15个靶标基因构建了sRNA的表达质粒库(每个靶基因的sRNA圴用5种启动子分别控制转录,共75种质粒),将这些质粒依次导入产腐胺的大肠杆菌基础菌中表征,发现分别抑制argF和glnA表达时的效果最好,腐胺的产量分别提高了61.3%和25%,随后通过更换不同启动子实现了上述两种sRNA共表达的组合优化,得到的最佳菌株在流加发酵中生产了43 g/L的腐胺,比基础菌株提高了77.7%。采用同样的sRNA调控技术,他们还构建了脯氨酸产量提高6倍的工程菌株,流加发酵产量达到大肠杆菌中的最高纪录(33.8 g/L)。该研究实例充分展示了sRNA技术在大规模筛选有效靶基因并精细控制表达水平上的高效率。
3.3.3 CRISPRa/i技术及应用基于CRISPR/Cas9系统开发的CRISPRa (CRISPR activation) 和CRISPRi (CRISPR interference) 技术分别用于基因表达的激活和抑制[94]。它们使用的是失去了剪切DNA能力但仍然被sgRNA精确地招募到靶DNA上的dCas9,通过空间位阻效应抑制基因表达,或将dCas9与转录激活因子融合后靶向启动子和增强子区域激活基因的表达。最近,改良版的CRISPRi在dCas9上连接了一个来自转录沉默子(Krüppel-associated box,KRAB)的结构域,从而使DNA难以转录,大大改善了CRISPRi在人体细胞中的抑制效率[95]。同时,一些研究者正在利用KRAB结构域让dCas9结合得更加稳定,以实现永久抑制特定位点的转录。
CRISPRa/i在l-赖氨酸、l-谷氨酸[96]、莽草酸[97]、聚(3-羟基丁酸酯-co-4-羟基丁酸酯)[98]和番茄红素[89]等生物基产品的代谢工程菌株构建中均有成功的应用。Lv等[98]在大肠杆菌中构建了一条由葡萄糖生成聚(3-羟基丁酸酯-co-4-羟基丁酸酯) [Poly(3-hydroxybutyrate-co-4-hydroxybutyrate),P(3HB-co-4HB)] 的途径,利用CRISPRi技术调控主要前体物4HB的合成途径中必需基因的表达,从而获得各种具有柔性性质的PHA材料。当合成途径中单基因如sdhA、sdhB、sucC、sucD或sad的表达受到负调控时,重组大肠杆菌菌株可产生含1 mol%–9 mol% 4HB的P(3HB-co-4HB)。当共抑制2个基因时,共聚物中4HB的含量最高增加到12 mol%。当共抑制5个基因时,4HB含量可超过18 mol%。结果表明,CRISPRi技术能够有效地微调单个或多个基因的表达,展示了其对代谢通量的灵活调控。
3.3.4 动态调控及应用Mahadevan等[99]在2008年首先提出动态代谢工程的概念,即根据胞内状态和环境条件的变化对基因表达进行动态调控,缓解静态调控所造成的生长阻滞和代谢流失衡,显著提高目标产品产量。根据响应机制的不同,动态调控可以分为代谢物依赖型和非代谢物依赖型动态调控。近年来,除单向动态调控外,双向动态调控也在代谢工程中发挥越来越重要的作用[100]。另外,动态调控与其他代谢工程改造方法的组合也有利于细胞工厂在产物合成中产量、转化率和产率的进一步提升[101]。
动态调控策略在番茄红素[102]、脂肪酸[103]、N-乙酰葡糖胺(N-acetylglucosamine,GlcNAc)[100]、衣康酸[104]、肌醇[105]和5-氨基乙酰丙酸[106]等生物基产品的代谢工程中得到了成功应用。Farmer等[102]利用乙酰磷酸响应的转录因子-启动子构建了调节番茄红素合成的动态调控开关。当胞内乙酰磷酸异常累积时,会上调磷酸烯醇丙酮酸合酶和异戊烯基焦磷酸异构酶的表达水平,将丙酮酸转化成磷酸烯醇丙酮酸,进而合成番茄红素,最终其产率提高了约50%。Liu等[100]将N-乙酰葡糖胺响应的生物传感器与基于CRISPRi的NOT门耦合,构建了一种可同时动态激活和抑制靶基因表达的自主双向动态调控(Autonomous dual-control,ADC)系统。其中,构建的遗传反馈电路可动态上调c合成模块,同时下调竞争途径模块,从而将GlcNAc产量从59.9 g/L显著提高到97.1 g/L。该系统可以在没有复杂过程控制的情况下实现高水平的生产性能,对其他微生物细胞工厂的动态调控具有重要参考价值。目前,动态调控元件种类少、特异性强、响应阈值窄和调控范围有限等问题仍然制约着其在代谢工程中的应用,但随着合成生物学、计算机模拟技术和蛋白质工程等学科的发展,一定会极大地促进动态调控策略的应用。
3.4 支架组装技术支架组装是一种将途径中的多个酶固定在支架上而形成人工多酶体系的方法。借助该技术可以形成代谢物的通道效应,增强酶和底物之间的相互作用,从而提高途径的代谢通量。另外,还可以通过改变支架结构调整不同酶的结合比例或数量,达到优化途径通量的目的。
3.4.1 支架种类目前已经建立的支架结构有蛋白支架和核酸支架[107]。蛋白支架是利用蛋白质间的相互作用将途经中的酶固定在同一蛋白支架上[108]。构建蛋白支架的关键是筛选和开发蛋白质-蛋白质相互作用对(域和配体)。目前已经报道的有SH3域[109]、PDZ域[110]、GBD域[111]、亮氨酸拉链[112]、phyB/pif3光可切换结合域[113]、亲合体affibody分子及相应的配体。含有单个域的支架蛋白及其配体分别和两种不同的酶融合后,通过域和配体的相互作用可以实现双酶组装。对于两种以上的多酶组装,则需要开发多个蛋白相互作用域融合的合成支架,不同域的配体分别和不同的酶融合表达,从而进一步实现组装。其中应用最成熟也最成功是(GBD)x-(SH3)y-(PDZ)z融合蛋白支架。核酸支架包括DNA支架[114]和RNA支架[115]两种类型,同蛋白支架相比,核酸支架具有独特的优势:短链核酸分子可折叠成各种结构,甚至可折叠成二聚体或多聚体[116],还可利用计算机模拟对核酸结构进行理性设计[117]。目前DNA支架技术较为成熟,包括以下两类:一类是用与DNA支架互补的核苷酸序列对途径酶进行化学修饰,再通过碱基互补配对将酶固定在DNA支架上,主要应用于体外环境。另一类是途径酶与含有DNA结合结构域的蛋白进行融合表达,借助结构域和特定DNA序列的相互作用将酶固定在DNA支架上,这种支架适合在体内环境中应用。
3.4.2 支架组装技术在代谢工程中的应用近年来,支架组装技术已经在萜烯类、衣康酸、甲羟戊酸、苏氨酸、吲哚-3-乙酸等生物基产品合成途径的通量优化中得到了应用[118-122]。例如,Dueber等[120]将甲羟戊酸合成途径中的乙酰乙酰-辅酶A硫解酶、羟甲基戊二酸-辅酶A合成酶和羟基-甲基戊二酰-辅酶A还原酶分别与GBD、SH3、PDZ的配体融合,并将可与它们特异性结合的蛋白支架在大肠杆菌中共同表达。在对蛋白支架的比例和酶的化学计量数进行优化时发现,当x、y、z分别是1、2、2时,甲羟戊酸的产量比无支架时提高了77倍。此支架还成功应用于丁酸、葡萄糖酸和白藜芦醇的生产中[123-125]。Lee等[121]报告了使用DNA支架系统在大肠杆菌中提高l-苏氨酸的生产速率,其中锌指蛋白充当适配器,将参与l-苏氨酸生产的高丝氨酸脱氢酶、高丝氨酸激酶和苏氨酸合酶的位点特异性结合到精确有序的位置。这个DNA支架系统显著提高了苏氨酸生物合成的效率,将发酵时间缩短了50%以上,而且该DNA支架系统通过降低毒性中间体的细胞内浓度,使宿主菌株的生长速率得到了提高。锌指蛋白与核酸的特异性结合也成功使白藜芦醇、1, 2-丙二醇、甲羟戊酸的产量分别提高了5倍、4.6倍、2.5倍[114]。
除了上述的主要使能技术之外,近年来,研究者通过理性或半理性设计方法构建了启动子、RBS、生物传感器等众多基因表达和调控元件(库)。这些多样性的元件在高性能菌株的构建和筛选中发挥着不可替代的重要作用,具体可以参考国内学者最近发表的一些典型综述[126-129],本文不再赘述。
4 总结与展望近10年来,系统生物学和合成生物学方法在代谢工程中不乏成功应用的例子,但也面临一些重要的问题或挑战:1) 在系统生物学的应用方面,虽然各种组学的研究产生了大量的数据集,但是大部分应用还是描述性的表征,精确预测靶点进而改进细胞性能的研究相对而言较少。其中主要原因是多组学数据整合及有效数据挖掘仍然是一个难题,通过数据驱动的机器学习算法从数据集中挖掘重要代谢信息是今后应对该挑战的主要方法[130]。2) 系统生物学方法预测出的潜在靶点仍然需要大量的鉴定测试(例如测试转录组学分析得到的数目众多的差异表达基因、通过微调优化靶点的表达水平等),除了少数几种模式微生物之外,在其他物种中进行大规模的遗传扰动测试仍然费时费力,而随着合成生物技术的发展,开发适合于更多微生物物种的表达元件和使能技术将在很大程度上解决这一问题。3) 目前,成功应用于商业化生产的代谢工程菌株仍然不多,其中工程菌株的鲁棒性是影响其放大生产的一个重要因素。因此,一方面需要开发更多天然适合于某类化学品生产的非模式底盘菌株及其合成生物技术[131]。另一方面,在菌株构建策略的选择中要避免其对鲁棒性的负面影响,通过基因表达的精细调控平衡细胞代谢、通过进化工程等方法提高细胞的抗逆性能、通过基因组编辑技术提高遗传稳定性等策略都可以显著改善工程菌株的鲁棒性[132],这些策略在代谢工程中的应用将促进更多的微生物细胞工厂走向实用化。
| [1] |
Bailey JE. Toward a science of metabolic engineering. Science, 1991, 252(5013): 1668-1675. DOI:10.1126/science.2047876
|
| [2] |
Stephanopoulos G, Vallino JJ. Network rigidity and metabolic engineering in metabolite overproduction. Science, 1991, 252(5013): 1675-1681. DOI:10.1126/science.1904627
|
| [3] |
Nielsen J, Villadsen J, Lidén G. Bioreaction engineering principles. 2nd ed. New York: Kluwer Academic, 2003.
|
| [4] |
张学礼. 代谢工程发展20年. 生物工程学报, 2009, 25(9): 1285-1295. Zhang XL. Twenty years development of metabolic engineering: a review. Chin J Biotech, 2009, 25(9): 1285-1295 (in Chinese). DOI:10.3321/j.issn:1000-3061.2009.09.002 |
| [5] |
Fang X, Lloyd CJ, Palsson BO. Reconstructing organisms in silico: genome-scale models and their emerging applications. Nat Rev Microbiol, 2020, 18(12): 731-743.
|
| [6] |
叶超, 徐楠, 陈修来, 等. 应用代谢网络模型解析工业微生物胞内代谢. 生物工程学报, 2019, 35(10): 1901-1913. Ye C, Xu N, Chen XL, et al. Application of metabolic network model to analyze intracellular metabolism of industrial microorganisms. Chin J Biotech, 2019, 35(10): 1901-1913 (in Chinese). |
| [7] |
Lieven C, Beber ME, Olivier BG, et al. MEMOTE for standardized genome-scale metabolic model testing. Nat Biotechnol, 2020, 38(3): 272-276. DOI:10.1038/s41587-020-0446-y
|
| [8] |
赵欣, 杨雪, 毛志涛, 等. 基于酶约束的代谢网络模型研究进展及其应用. 生物工程学报, 2019, 35(10): 1914-1924. Zhao X, Yang X, Mao ZT, et al. Progress and application of metabolic network model based on enzyme constraints. Chin J Biotech, 2019, 35(10): 1914-1924 (in Chinese). |
| [9] |
Sánchez BJ, Zhang C, Nilsson A, et al. Improving the phenotype predictions of a yeast genome-scale metabolic model by incorporating enzymatic constraints. Mol Syst Biol, 2017, 13(8): 935-935. DOI:10.15252/msb.20167411
|
| [10] |
Lerman JA, Hyduke DR, Latif H, et al. In silico method for modelling metabolism and gene product expression at genome scale. Nat Commun, 2012, 3: 929. DOI:10.1038/ncomms1928
|
| [11] |
O'Brien EJ, Lerman JA, Chang RL, et al. Genome-scale models of metabolism and gene expression extend and refine growth phenotype prediction. Mol Syst Biol, 2013, 9(1): 693. DOI:10.1038/msb.2013.52
|
| [12] |
Liu JK, Lloyd C, Al-Bassam MM, et al. Predicting proteome allocation, overflow metabolism, and metal requirements in a model acetogen. PLoS Comp Biol, 2019, 15(3): e1006848. DOI:10.1371/journal.pcbi.1006848
|
| [13] |
Chatsurachai S, Furusawa C, Shimizu H. An in silico platform for the design of heterologous pathways in nonnative metabolite production. BMC Bioinformatics, 2012, 13: 93. DOI:10.1186/1471-2105-13-93
|
| [14] |
Lin ZQ, Zhang Y, Yuan QQ, et al. Metabolic engineering of Escherichia coli for poly(3-hydroxybutyrate) production via threonine bypass. Microb Cell Fact, 2015, 14: 185. DOI:10.1186/s12934-015-0369-3
|
| [15] |
Yang XY, Yuan QQ, Zheng YY, et al. An engineered non-oxidative glycolysis pathway for acetone production in Escherichia coli. Biotechnol Lett, 2016, 38(8): 1359-1365. DOI:10.1007/s10529-016-2115-2
|
| [16] |
Luo ZW, Lee SY. Metabolic engineering of Escherichia coli for the production of benzoic acid from glucose. Metab Eng, 2020, 62: 298-311. DOI:10.1016/j.ymben.2020.10.002
|
| [17] |
Ye C, Luo QL, Guo L, et al. Improving lysine production through construction of an Escherichia coli enzyme-constrained model. Biotechnol Bioeng, 2020, 117(11): 3533-3544. DOI:10.1002/bit.27485
|
| [18] |
Borodina I, Kildegaard KR, Jensen NB, et al. Establishing a synthetic pathway for high-level production of 3-hydroxypropionic acid in Saccharomyces cerevisiae via β-alanine. Metab Eng, 2015, 27(5): 57-64.
|
| [19] |
Lis AV, Schneider K, Weber J, et al. Exploring small-scale chemostats to scale up microbial processes: 3-hydroxypropionic acid production in S. cerevisiae. Microb Cell Fact, 2019, 18: 50. DOI:10.1186/s12934-019-1101-5
|
| [20] |
Kildegaard KR, Jensen NB, Schneider K, et al. Engineering and systems-level analysis of Saccharomyces cerevisiae for production of 3-hydroxypropionic acid via malonyl-CoA reductase-dependent pathway. Microb Cell Fact, 2016, 15: 53. DOI:10.1186/s12934-016-0451-5
|
| [21] |
Hrdlickova R, Toloue M, Tian B. RNA-Seq methods for transcriptome analysis. Wiley Interdiscip Rev RNA, 2017, 8(1): e1364. DOI:10.1002/wrna.1364
|
| [22] |
Chung SC, Park JS, Yun JE, et al. Improvement of succinate production by release of end-product inhibition in Corynebacterium glutamicum. Metab Eng, 2017, 40: 157-164. DOI:10.1016/j.ymben.2017.02.004
|
| [23] |
Becker J, Wittmann C. From systems biology to metabolically engineered cells—an omics perspective on the development of industrial microbes. Curr Opin Microbiol, 2018, 45: 180-188. DOI:10.1016/j.mib.2018.06.001
|
| [24] |
Yuan HM, Jiang B, Zhao BF, et al. Recent advances in multidimensional separation for proteome analysis. Anal Chem, 2019, 91(1): 264-276. DOI:10.1021/acs.analchem.8b04894
|
| [25] |
Ren JT, Beckner MA, Lynch KB, et al. Two-dimensional liquid chromatography consisting of twelve second-dimension columns for comprehensive analysis of intact proteins. Talanta, 2018, 182: 225-229. DOI:10.1016/j.talanta.2018.01.072
|
| [26] |
Huang Z, Yan GQ, Gao MX, et al. Array-based online two dimensional liquid chromatography system applied to effective depletion of high-abundance proteins in human plasma. Anal Chem, 2016, 88(4): 2440-2445. DOI:10.1021/acs.analchem.5b04553
|
| [27] |
Quan Q, Feng JW, Lui LT, et al. Phosphoproteome of crab-eating macaque cerebral cortex characterized through multidimensional reversed-phase liquid chromatography/mass spectrometry with tandem anion/cation exchange columns. J Chromatogr A, 2017, 1498: 196-206. DOI:10.1016/j.chroma.2017.01.048
|
| [28] |
Qu YY, Sun LL, Zhang ZB, et al. Site-specific glycan heterogeneity characterization by hydrophilic interaction liquid chromatography solid-phase extraction, reversed-phase liquid chromatography fractionation, and capillary zone electrophoresis-electrospray ionization-tandem mass spectrometry. Anal Chem, 2018, 90(2): 1223-1233. DOI:10.1021/acs.analchem.7b03912
|
| [29] |
Ma SM, Garcia DE, Redding-Johanson AM, et al. Optimization of a heterologous mevalonate pathway through the use of variant HMG-CoA reductases. Metab Eng, 2011, 13(5): 588-597. DOI:10.1016/j.ymben.2011.07.001
|
| [30] |
Lee SH, Kang KH, Kim EY, et al. Metabolic engineering of Escherichia coli for enhanced biosynthesis of poly(3-hydroxybutyrate) based on proteome analysis. Biotechnol Lett, 2013, 35(10): 1631-1637. DOI:10.1007/s10529-013-1246-y
|
| [31] |
Alonso-Gutierrez J, Kim EM, Batth TS, et al. Principal component analysis of proteomics (PCAP) as a tool to direct metabolic engineering. Meta Eng, 2015, 28: 123-133. DOI:10.1016/j.ymben.2014.11.011
|
| [32] |
Xu JY, Xu Y, Chu XH, et al. Protein acylation affects the artificial biosynthetic pathway for pinosylvin production in engineered E. coli. ACS Chem Biol, 2018, 13(5): 1200-1208. DOI:10.1021/acschembio.7b01068
|
| [33] |
Jiang Y, Huang MZ, Chen XL, et al. Proteome analysis guided genetic engineering of Corynebacterium glutamicum S9114 for tween 40-triggered improvement in l-ornithine production. Microb Cell Fact, 2020, 19: 2. DOI:10.1186/s12934-019-1272-0
|
| [34] |
Haggarty J, Burgess KEV. Recent advances in liquid and gas chromatography methodology for extending coverage of the metabolome. Curr Opin Biotechnol, 2017, 43: 77-85. DOI:10.1016/j.copbio.2016.09.006
|
| [35] |
George KW, Thompson MG, Kang A, et al. Metabolic engineering for the high-yield production of isoprenoid-based C5 alcohols in E. coli. Sci Rep, 2015, 5: 11128. DOI:10.1038/srep11128
|
| [36] |
Ohtake T, Pontrelli S, Laviña WA, et al. Metabolomics-driven approach to solving a CoA imbalance for improved 1-butanol production in Escherichia coli. Metab Eng, 2017, 41: 135-143. DOI:10.1016/j.ymben.2017.04.003
|
| [37] |
Hasunuma T, Matsuda M, Kato Y, et al. Temperature enhanced succinate production concurrent with increased central metabolism turnover in the cyanobacterium Synechocystis sp. PCC 6803. Metab En, 2018, 48: 109-120. DOI:10.1016/j.ymben.2018.05.013
|
| [38] |
Gold ND, Gowen CM, Lussier FX, et al. Metabolic engineering of a tyrosine-overproducing yeast platform using targeted metabolomics. Microb Cell Fact, 2015, 14: 73. DOI:10.1186/s12934-015-0252-2
|
| [39] |
Kawaguchi H, Yoshihara K, Hara KY, et al. Metabolome analysis-based design and engineering of a metabolic pathway in Corynebacterium glutamicum to match rates of simultaneous utilization of D-glucose and L-arabinose. Microb Cell Fact, 2018, 17: 76. DOI:10.1186/s12934-018-0927-6
|
| [40] |
Hasunuma T, Sanda T, Yamada R, et al. Metabolic pathway engineering based on metabolomics confers acetic and formic acid tolerance to a recombinant xylose-fermenting strain of Saccharomyces cerevisiae. Microb Cell Fact, 2011, 10: 2. DOI:10.1186/1475-2859-10-2
|
| [41] |
Teoh ST, Putri S, Mukai Y, et al. A metabolomics-based strategy for identification of gene targets for phenotype improvement and its application to 1-butanol tolerance in Saccharomyces cerevisiae. Biotechnol Biofuels, 2015, 8: 144. DOI:10.1186/s13068-015-0330-z
|
| [42] |
Heux S, Juliette P, Stéphane M, et al. A novel platform for automated high-throughput fluxome profiling of metabolic variants. Metab Eng, 2014, 25: 8-19. DOI:10.1016/j.ymben.2014.06.001
|
| [43] |
Antoniewicz MR. A guide to metabolic flux analysis in metabolic engineering: methods, tools and applications. Metab Eng, 2021, 63: 2-12. DOI:10.1016/j.ymben.2020.11.002
|
| [44] |
Gonzalez JE, Long CP, Antoniewicz MR. Comprehensive analysis of glucose and xylose metabolism in Escherichia coli under aerobic and anaerobic conditions by 13C metabolic flux analysis. Metab Eng, 2017, 39: 9-18. DOI:10.1016/j.ymben.2016.11.003
|
| [45] |
Lange A, Becker J, Schulze D, et al. Bio-based succinate from sucrose: high-resolution 13C metabolic flux analysis and metabolic engineering of the rumen bacterium Basfia succiniciproducens. Metab Eng, 2017, 44: 198-212. DOI:10.1016/j.ymben.2017.10.003
|
| [46] |
孟娇, 刘丁玉, 黄灿, 等. CRISPR/Cas基因编辑系统在原核微生物细胞工厂构建中的开发与应用. 微生物学通报, 2019, 46(10): 2730-2742. Meng J, Liu DY, Huang C, et al. Development and application of CRISPR/Cas genome editing system in the construction of prokaryotic microbial cell factories. Microbiol China, 2019, 46(10): 2730-2742 (in Chinese). |
| [47] |
Liu ZQ, Dong HN, Cui YL, et al. Application of different types of CRISPR/Cas-based systems in bacteria. Microb Cell Fact, 2020, 19: 172. DOI:10.1186/s12934-020-01431-z
|
| [48] |
Stovicek V, Holkenbrink C, Borodina I. CRISPR/Cas system for yeast genome engineering: advances and applications. FEMS Yeast Res, 2017, 17(5): fox030.
|
| [49] |
Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033
|
| [50] |
Xu T, Li YC, He ZL, et al. Cas9 nickase-assisted RNA repression enables stable and efficient manipulation of essential metabolic genes in Clostridium cellulolyticum. Front Microbiol, 2017, 8: 1744. DOI:10.3389/fmicb.2017.01744
|
| [51] |
Liu DY, Huang C, Guo JX, et al. Development and characterization of a CRISPR/Cas9n-based multiplex genome editing system for Bacillus subtilis. Biotechnol Biofuels, 2019, 12: 197. DOI:10.1186/s13068-019-1537-1
|
| [52] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-Guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829
|
| [53] |
Bae S, Park J, Kim JS. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics, 2014, 30(10): 1473-1475. DOI:10.1093/bioinformatics/btu048
|
| [54] |
Stemmer M, Thumberger T, Keyer MD, et al. CCTop: an intuitive, flexible and reliable CRISPR/Cas9 target prediction tool. PLoS ONE, 2015, 10(4): e0124633. DOI:10.1371/journal.pone.0124633
|
| [55] |
Fu YF, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol, 2014, 32(3): 279-284. DOI:10.1038/nbt.2808
|
| [56] |
Kim D, Bae S, Park J, et al. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods, 2015, 12(3): 237-243. DOI:10.1038/nmeth.3284
|
| [57] |
Zhang YP, Wang J, Wang ZB, et al. A gRNA-tRNA array for CRISPR-Cas9 based rapid multiplexed genome editing in Saccharomyces cerevisiae. Nat Commun, 2019, 10: 1053. DOI:10.1038/s41467-019-09005-3
|
| [58] |
Yan MY, Yan HQ, Ren GX, et al. CRISPR-Cas12a-assisted recombineering in bacteria. Appl Environ Microbiol, 2017, 83(17): e00947-17.
|
| [59] |
Jiang Y, Qian FH, Yang JJ, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun, 2017, 8: 15179. DOI:10.1038/ncomms15179
|
| [60] |
Li L, Wei KK, Zheng GS, et al. CRISPR-Cpf1-assisted multiplex genome editing and transcriptional repression in Streptomyces. Appl Environ Microbiol, 2018, 84(18): e0082-18.
|
| [61] |
Yang ZL, Edwards H, Xu P. CRISPR-Cas12a/Cpf1-assisted precise, efficient and multiplexed genome-editing in Yarrowia lipolytica. Metab Eng Commun, 2020, 10: e00112. DOI:10.1016/j.mec.2019.e00112
|
| [62] |
Fonfara I, Richter H, Bratovič M, et al. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature, 2016, 532(7600): 517-521. DOI:10.1038/nature17945
|
| [63] |
Zhang XC, Wang JM, Cheng QX, et al. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discov, 2017, 3: 17018.
|
| [64] |
Zetsche B, Heidenreich M, Mohanraju P, et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nat Biotechnol, 2017, 35(1): 31-34. DOI:10.1038/nbt.3737
|
| [65] |
Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644
|
| [66] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946
|
| [67] |
Zhao DD, Li J, Li SW, et al. Glycosylase base editors enable C-to-A and C-to-G base changes. Nat Biotechnol, 2020, 39(1): 35-40.
|
| [68] |
Jakočiūnas T, Sonde I, Herrgård M, et al. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae. Metab Eng, 2015, 28: 213-222. DOI:10.1016/j.ymben.2015.01.008
|
| [69] |
Li YF, Lin ZQ, Huang C, et al. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing. Metab Eng, 2015, 31: 13-21. DOI:10.1016/j.ymben.2015.06.006
|
| [70] |
Garst AD, Bassalo MC, Pines G, et al. Genome-wide mapping of mutations at single-nucleotide resolution for protein, metabolic and genome engineering. Nat Biotechnol, 2017, 35(1): 48-55. DOI:10.1038/nbt.3718
|
| [71] |
Liu RM, Liang LY, Choudhury A, et al. Iterative genome editing of Escherichia coli for 3-hydroxypropionic acid production. Metab Eng, 2018, 47: 303-313. DOI:10.1016/j.ymben.2018.04.007
|
| [72] |
常汉臣, 王琛, 王培霞, 等. DNA组装技术. 生物工程学报, 2019, 35(12): 2215-2226. Chang HC, Wang C, Wang PX, et al. DNA assembly technologies: a review. Chin J Biotech, 2019, 35(12): 2215-2226 (in Chinese). |
| [73] |
Chao R, Yuan YB, Zhao HM. Recent advances in DNA assembly technologies. FEMS Yeast Res, 2015, 15(1): 1-9. DOI:10.1093/femsyr/fou003
|
| [74] |
Liang J, Liu ZH, Low XZ, et al. Twin-primer non-enzymatic DNA assembly: an efficient and accurate multi-part DNA assembly method. Nucleic Acids Res, 2017, 45(11): e94. DOI:10.1093/nar/gkx132
|
| [75] |
Taylor GM, Mordaka PM, Heap JT. Start-stop assembly: a functionally scarless DNA assembly system optimized for metabolic engineering. Nucleic Acids Res, 2019, 47(3): e17. DOI:10.1093/nar/gky1182
|
| [76] |
Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast. Science, 2015, 349(6252): 1095-1100. DOI:10.1126/science.aac9373
|
| [77] |
Fang H, Li D, Kang J, et al. Metabolic engineering of Escherichia coli for de novo biosynthesis of vitamin B12. Nat Commun, 2018, 9: 4917. DOI:10.1038/s41467-018-07412-6
|
| [78] |
Brantl S. Regulatory mechanisms employed by cis-encoded antisense RNAs. Curr Opin Microbiol, 2007, 10(2): 102-109. DOI:10.1016/j.mib.2007.03.012
|
| [79] |
Yoo SM, Na D, Lee SY. Design and use of synthetic regulatory small RNAs to control gene expression in Escherichia coli. Nat Protoc, 2013, 8(9): 1694-1707. DOI:10.1038/nprot.2013.105
|
| [80] |
Gilbert LA, Horlbeck MA, Adamson B, et al. Genome-scale CRISPR-mediated control of gene repression and activation. Cell, 2014, 159(3): 647-661. DOI:10.1016/j.cell.2014.09.029
|
| [81] |
Tomizawa J, Itoh T, Selzer G, et al. Inhibition of ColE1 RNA primer formation by a plasmid-specified small RNA. Proc Natl Acad Sci USA, 1981, 78(3): 1421-1425. DOI:10.1073/pnas.78.3.1421
|
| [82] |
Kubo T, Bakalova R, Ohba H, et al. Antisense effects of DNA-peptide conjugates. Nucleic Acids Res Suppl, 2003(3): 179-180.
|
| [83] |
Desai RP, Papoutsakis ET. Antisense RNA strategies for metabolic engineering of Clostridium acetobutylicum. Appl Environ Microbiol, 1999, 65(3): 936-945. DOI:10.1128/AEM.65.3.936-945.1999
|
| [84] |
Bouazzaoui K, LaPointe G. Use of antisense RNA to modulate glycosyltransferase gene expression and exopolysaccharide molecular mass in Lactobacillus rhamnosus. J Microbiol Methods, 2006, 65(2): 216-225. DOI:10.1016/j.mimet.2005.07.011
|
| [85] |
Wang TH, Zhong YH, Huang W, et al. Antisense inhibition of xylitol dehydrogenase gene, xdh1 from Trichoderma reesei. Lett Appl Microbiol, 2005, 40(6): 424-429. DOI:10.1111/j.1472-765X.2005.01685.x
|
| [86] |
Na D, Yoo SM, Chung H, et al. Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs. Nat Biotechnol, 2013, 31(2): 170-174. DOI:10.1038/nbt.2461
|
| [87] |
Sun DH, Chen JZ, Wang Y, et al. Metabolic engineering of Corynebacterium glutamicum by synthetic small regulatory RNAs. J Ind Microbiol Biotechnol, 2019, 46(2): 203-208. DOI:10.1007/s10295-018-02128-4
|
| [88] |
Kang Z, Wang XR, Li YK, et al. Small RNA RyhB as a potential tool used for metabolic engineering in Escherichia coli. Biotechnol Lett, 2012, 34(3): 527-531. DOI:10.1007/s10529-011-0794-2
|
| [89] |
Kim SK, Han GH, Seong W, et al. CRISPR interference-guided balancing of a biosynthetic mevalonate pathway increases terpenoid production. Metab Eng, 2016, 38: 228-240. DOI:10.1016/j.ymben.2016.08.006
|
| [90] |
Liu YF, Zhu YQ, Li JH, et al. Modular pathway engineering of Bacillus subtilis for improved N-acetylglucosamine production. Metab Eng, 2014, 23: 42-52. DOI:10.1016/j.ymben.2014.02.005
|
| [91] |
Cho C, Lee SY. Efficient gene knockdown in Clostridium acetobutylicum by synthetic small regulatory RNAs. Biotechnol Bioeng, 2017, 114(2): 374-383. DOI:10.1002/bit.26077
|
| [92] |
Kim B, Park H, Na D, et al. Metabolic engineering of Escherichia coli for the production of phenol from glucose. Biotechnol J, 2014, 9(5): 621-629. DOI:10.1002/biot.201300263
|
| [93] |
Noh M, Yoo SM, Kim WJ, et al. Gene expression knockdown by modulating synthetic small RNA expression in Escherichia coli. Cell Syst, 2017, 5(4): 418-426.e4. DOI:10.1016/j.cels.2017.08.016
|
| [94] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022
|
| [95] |
Alerasool N, Segal D, Lee H, et al. An efficient KRAB domain for CRISPRi applications in human cells. Nat Methods, 2020, 17(11): 1093-1096. DOI:10.1038/s41592-020-0966-x
|
| [96] |
Cleto S, Jensen JVK, Wendisch VF, et al. Corynebacterium glutamicum metabolic engineering with CRISPR interference (CRISPRi). ACS Synth Biol, 2016, 5(5): 375-385. DOI:10.1021/acssynbio.5b00216
|
| [97] |
Zhang B, Liu ZQ, Liu C, et al. Application of CRISPRi in Corynebacterium glutamicum for shikimic acid production. Biotechnol Lett, 2016, 38(12): 2153-2161. DOI:10.1007/s10529-016-2207-z
|
| [98] |
Lv L, Ren YL, Chen JC, et al. Application of CRISPRi for prokaryotic metabolic engineering involving multiple genes, a case study: controllable P(3HB-co-4HB) biosynthesis. Metab Eng, 2015, 29: 160-168. DOI:10.1016/j.ymben.2015.03.013
|
| [99] |
Anesiadis N, Cluett WR, Mahadevan R. Dynamic metabolic engineering for increasing bioprocess productivity. Metab Eng, 2008, 10(5): 255-266. DOI:10.1016/j.ymben.2008.06.004
|
| [100] |
Wu YK, Chen TC, Liu YF, et al. Design of a programmable biosensor-CRISPRi genetic circuits for dynamic and autonomous dual-control of metabolic flux in Bacillus subtilis. Nucleic Acids Res, 2020, 48(2): 996-1009. DOI:10.1093/nar/gkz1123
|
| [101] |
Tan SZ, Prather KL. Dynamic pathway regulation: recent advances and methods of construction. Curr Opin Chem Biol, 2017, 41: 28-35. DOI:10.1016/j.cbpa.2017.10.004
|
| [102] |
Farmer WR, Liao JC. Improving lycopene production in Escherichia coli by engineering metabolic control. Nat Biotechnol, 2000, 18(5): 533-537. DOI:10.1038/75398
|
| [103] |
Xu P, Li LY, Zhang FM, et al. Improving fatty acids production by engineering dynamic pathway regulation and metabolic control. Proc Natl Acad Sci USA, 2014, 111(31): 11299-11304. DOI:10.1073/pnas.1406401111
|
| [104] |
Harder BJ, Bettenbrock K, Klamt S. Temperature-dependent dynamic control of the TCA cycle increases volumetric productivity of itaconic acid production by Escherichia coli. Biotechnol Bioeng, 2018, 115(1): 156-164. DOI:10.1002/bit.26446
|
| [105] |
Brockman IM, Prather KLJ. Dynamic knockdown of E. coli central metabolism for redirecting fluxes of primary metabolites. Metab Eng, 2015, 28: 104-113. DOI:10.1016/j.ymben.2014.12.005
|
| [106] |
Zhou LB, Ren J, Li ZD, et al. Characterization and engineering of a clostridium glycine riboswitch and its use to control a novel metabolic pathway for 5-aminolevulinic acid production in Escherichia coli. ACS Synth Biol, 2019, 8(10): 2327-2335. DOI:10.1021/acssynbio.9b00137
|
| [107] |
Lv XQ, Cui SX, Gu Y, et al. Enzyme assembly for compartmentalized metabolic flux control. Metabolites, 2020, 10(4): 125. DOI:10.3390/metabo10040125
|
| [108] |
Siu KH, Chen RP, Sun Q, et al. Synthetic scaffolds for pathway enhancement. Curr Opin Biotechnol, 2015, 36: 98-106. DOI:10.1016/j.copbio.2015.08.009
|
| [109] |
Zarrinpar A, Park SH, Lim WA. Optimization of specificity in a cellular protein interaction network by negative selection. Nature, 2003, 426(6967): 676-680. DOI:10.1038/nature02178
|
| [110] |
Harris BZ, Hillier BJ, Lim WA. Energetic determinants of internal motif recognition by PDZ domains. Biochemistry, 2001, 40(20): 5921-5930. DOI:10.1021/bi010142l
|
| [111] |
Kim AS, Kakalis LT, Abdul-Manan N, et al. Autoinhibition and activation mechanisms of the Wiskott-Aldrich syndrome protein. Nature, 2000, 404(6774): 151-158. DOI:10.1038/35004513
|
| [112] |
Reinke AW, Grant RA, Keating AE. A synthetic coiled-coil interactome provides heterospecific modules for molecular engineering. J Am Chem Soc, 2010, 132(17): 6025-6031. DOI:10.1021/ja907617a
|
| [113] |
Levskaya A, Weiner OD, Lim WA, et al. Spatiotemporal control of cell signalling using a light-switchable protein interaction. Nature, 2009, 461(7266): 997-1001. DOI:10.1038/nature08446
|
| [114] |
Conrado RJ, Wu GC, Boock JT, et al. DNA-guided assembly of biosynthetic pathways promotes improved catalytic efficiency. Nucleic Acids Res, 2011, 40(4): 1879-1889.
|
| [115] |
Sachdeva G, Garg A, Godding D, et al. In vivo co-localization of enzymes on RNA scaffolds increases metabolic production in a geometrically dependent manner. Nucleic Acids Res, 2014, 42(14): 9493-9503. DOI:10.1093/nar/gku617
|
| [116] |
Rothemund PW. Folding DNA to create nanoscale shapes and patterns. Nature, 2006, 440(7082): 297-302. DOI:10.1038/nature04586
|
| [117] |
Zadeh JN, Steenberg CD, Bois JS, et al. NUPACK: analysis and design of nucleic acid systems. J Comput Chem, 2011, 32(1): 170-173. DOI:10.1002/jcc.21596
|
| [118] |
Tippmann S, Anfelt J, David F, et al. Affibody scaffolds improve sesquiterpene production in Saccharomyces cerevisiae. ACS Synth Biol, 2017, 6(1): 19-28. DOI:10.1021/acssynbio.6b00109
|
| [119] |
Yang ZW, Gao X, Xie H, et al. Enhanced itaconic acid production by self-assembly of two biosynthetic enzymes in Escherichia coli. Biotechnol Bioeng, 2017, 114(2): 457-462. DOI:10.1002/bit.26081
|
| [120] |
Dueber JE, Wu GC, Malmirchegini GR, et al. Synthetic protein scaffolds provide modular control over metabolic flux. Nat Biotechnol, 2009, 27(8): 753-759. DOI:10.1038/nbt.1557
|
| [121] |
Lee JH, Jung SC, Bui LM, et al. Improved production of L-threonine in Escherichia coli by use of a DNA scaffold system. Appl Environ Microbiol, 2013, 79(3): 774-782. DOI:10.1128/AEM.02578-12
|
| [122] |
Zhu LY, Qiu XY, Zhu LY, et al. Spatial organization of heterologous metabolic system in vivo based on TALE. Sci Rep, 2016, 6: 26065. DOI:10.1038/srep26065
|
| [123] |
Moon TS, Dueber JE, Shiue E, et al. Use of modular, synthetic scaffolds for improved production of glucaric acid in engineered E. coli. Metab Eng, 2010, 12(3): 298-305. DOI:10.1016/j.ymben.2010.01.003
|
| [124] |
Baek JM, Mazumdar S, Lee SW, et al. Butyrate production in engineered Escherichia coli with synthetic scaffolds. Biotechnol Bioeng, 2013, 110(10): 2790-2794. DOI:10.1002/bit.24925
|
| [125] |
Wang YC, Yu O. Synthetic scaffolds increased resveratrol biosynthesis in engineered yeast cells. J Biotechnol, 2012, 157(1): 258-260. DOI:10.1016/j.jbiotec.2011.11.003
|
| [126] |
田荣臻, 刘延峰, 李江华, 等. 典型模式微生物基因表达精细调控工具的研究进展. 合成生物学, 2020, 1(4): 454-469. Tian RZ, Liu YF, Li JH, et al. Progress in the regulatory tools of gene expression for model microorganisms. Synth Biol J, 2020, 1(4): 454-469 (in Chinese). |
| [127] |
Qiu CX, Zhai HT, Hou J. Biosensors design in yeast and applications in metabolic engineering. FEMS Yeast Res, 2019, 19(8): foz082. DOI:10.1093/femsyr/foz082
|
| [128] |
Jin LQ, Jin WR, Ma ZC, et al. Promoter engineering strategies for the overproduction of valuable metabolites in microbes. Appl Microbiol Biotechnol, 2019, 103(21/22): 8725-8736. DOI:10.1007/s00253-019-10172-y
|
| [129] |
Gao C, Xu P, Ye C, et al. Genetic circuit-assisted smart microbial engineering. Trends Microbiol, 2019, 27(12): 1011-1024. DOI:10.1016/j.tim.2019.07.005
|
| [130] |
Presnell KV, Alper HS. Systems metabolic engineering meets machine learning: a new era for data-driven metabolic engineering. Biotechnol J, 2019, 14(9): 1800416. DOI:10.1002/biot.201800416
|
| [131] |
Fatma Z, Schultz JC, Zhao HM. Recent advances in domesticating non-model microorganisms. Biotechnol Prog, 2020, 36(5): e3008.
|
| [132] |
Jiang T, Li CY, Teng YX, et al. Recent advances in improving metabolic robustness of microbial cell factories. Curr Opin Biotechnol, 2020, 66: 69-77. DOI:10.1016/j.copbio.2020.06.006
|
 2021, Vol. 37
2021, Vol. 37




