
中国科学院微生物研究所、中国微生物学会主办
文章信息
- 于勇, 朱欣娜, 毕昌昊, 张学礼
- Yu Yong, Zhu Xinna, Bi Changhao, Zhang Xueli
- 大肠杆菌细胞工厂的创建技术
- Construction of Escherichia coli cell factories
- 生物工程学报, 2021, 37(5): 1564-1577
- Chinese Journal of Biotechnology, 2021, 37(5): 1564-1577
- 10.13345/j.cjb.200716
-
文章历史
- Received: November 7, 2020
- Accepted: January 15, 2021
代谢工程是以基因工程为基础发展起来的一个新的学科领域,是指利用基因工程技术对细胞代谢途径进行有目的的修饰与改造,改变细胞特性,生产出特定的目标产物。
大肠杆菌因研究历史悠久、遗传背景清晰、厌氧生长速度快、能利用简单的无机盐培养基并可利用多种底物(葡萄糖、木糖、甘油) 等特点,成为了一种优秀的工业模式微生物。代谢工程改造大肠杆菌,可用于生产有机醇、氨基酸、有机酸、有机胺、维生素、天然产物及聚羟基脂肪酸酯(Polyhydroxyalkanoates,PHA) 等多种产品,并应用于l-丙氨酸[1]、l-赖氨酸[2]、l-苏氨酸[3]、1, 3-丙二醇[4]、d-乳酸[5]、丁二酸[6]、戊二胺[7]等大宗化学品的绿色生物合成。
代谢工程发展至今已有30年的历史(图 1),从早期的敲除副产物生成途径、解除产物合成抑制、过表达合成途径中的关键酶等传统改造技术,发展出了代谢反应数据库建立与网络模型计算、代谢途径组装、基因表达动态调控等新的代谢改造技术(图 2)。本文将从代谢途径设计、合成途径创建与优化、细胞全局优化三方面,介绍代谢工程发展的30年间对大肠杆菌代谢改造起重要推动作用的技术,并对大肠杆菌代谢工程中关键技术的应用进行了展望。
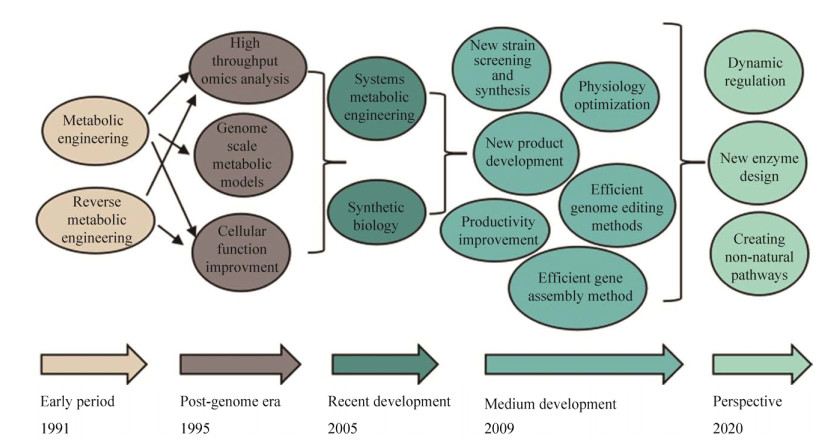
|
|
图 1 大肠杆菌代谢工程30年发展历程
Fig. 1 30-year development of metabolic engineering in Escherichia coli.
|
| |

|
|
图 2 大肠杆菌代谢工程技术
Fig. 2 Technology of metabolic engineering in Escherichia coli.
|
| |
随着基因组测序技术的发展,越来越多的生物全基因组序列公布,提高了研究人员从自然界中挖掘新功能基因的速度,积累了大量的代谢反应数据,开发和建立了代谢网络模型[8]和代谢反应数据库包括KEGG、MetaCyc、Brenda等,它们为大肠杆菌的代谢途径设计提供了改造策略和参考。
以KEGG中存储的大肠杆菌代谢数据为基础,Zhang等[9]构建了包含7 316个反应的大肠杆菌代谢网络模型,再结合大肠杆菌外源的代谢反应数据,发现了1 777种以葡萄糖为底物发酵合成的异源发酵产品。这些研究提高了研究人员设计新反应、新途径的效率。
设计的大肠杆菌代谢途径依据来源可分为3种情况:(1) 仅利用大肠杆菌自身代谢途径。如丁二酸、丙酮酸、l-苏氨酸、l-缬氨酸等代谢产物的合成途径本就存在于天然的大肠杆菌代谢途径中。对大肠杆菌自身的代谢途径进行适当改造与调控,如解除关键位点的反馈抑制、提高限速酶的表达量、调控辅因子代谢平衡等,将代谢流最大程度地引向目标产品,得到相应的大肠杆菌细胞工厂。(2) 引入外源代谢途径。大肠杆菌自身拥有的基因有限,常常需要利用外源基因将代谢途径补充完整或提高原有代谢途径的效率。例如在产1, 3-丙二醇大肠杆菌细胞工厂中引入了来源于酿酒酵母的甘油-3-磷酸脱氢酶、甘油-3-磷酸酶以及肺炎克雷伯氏菌的甘油脱水酶[4];在l-丙氨酸细胞工厂中引入了来自嗜热脂肪地芽孢杆菌Geobacillus stearothermophilus的丙氨酸脱氢酶[1];1, 4-丁二醇大肠杆菌细胞工厂中则整合了5个不同来源的外源基因[10]。(3) 创建自然界中不存在的代谢途径。在蛋白质理性改造与从头设计等技术的加持下,代谢途径的设计也突破了天然途径的限制,逐步发展出了非天然的合成途径。例如,中国科学院天津工业生物技术研究所、中国科学院微生物研究所和山东大学设计并制造了乙醇醛合酶,继而创造了一个合成乙酰辅酶A的碳一(甲醛) 代谢途径[11]。
随着人工智能等先进技术的不断发展,未来代谢途径的设计范围会更广泛,可以像化学合成一样基本实现所有化学品的生物合成,不断降低生物制造的成本,实现化学品的绿色生产。
2 大肠杆菌合成途径的创建与优化 2.1 DNA片段组装技术DNA片段组装技术是实现大肠杆菌途径创建的重要前提。传统的DNA片段组装主要依靠限制性核酸内切酶和DNA连接酶,但这种组装方式有一定的局限性,如接头处会留下酶切位点“疤痕”,需组装的基因片段内部不能存在相应的酶切位点,使可选择的内切酶范围变少。近年来,研究人员开发出新的DNA片段组装技术,这些技术简单、可操作性强,能实现DNA的高效无痕组装。
(1) Golden Gate组装技术[12]:需要使用ⅡS类限制性核酸内切酶,这类酶具有识别和切割双链DNA位置不一致的特性。这种特性使其在识别位点之外切开DNA,产生的粘性末端在连接酶的作用下按照顺序连接,组装成不含酶切位点的DNA片段,实现多个片段的无缝连接。
(2) Gibson组装技术[13]:不需使用限制性内切酶,但各DNA片段末端上互含15–20 bp的同源序列,据此,按照同源序列的顺序进行组装。组装由多个酶协同参与,DNA 5′核酸外切酶首先切出一个粘性末端,各粘性末端间根据同源关系互补,再由DNA聚合酶将缺失的碱基重新复制合成,最后由DNA连接酶连接粘性末端。该方法可以将一个或多个DNA片段按照预定的方向快速、高效、精确地插入到线性化载体中,实现“无缝”组装。
(3) CPEC技术[14]:CPEC (Circular polymerase extension cloning) 是环形聚合酶延伸克隆的简称。其对DNA片段的要求类似于Gibson,末端互含同源序列。CPEC是一种通过PCR实现DNA片段组装的技术:DNA片段及载体经过变性解链,退火时末端同源序列互补,序列间互为模板和引物在DNA聚合酶的作用下延伸为含有缺口的环状DNA分子,该缺口可在大肠杆菌中得到修复从而得到完整的质粒。总的来说,CPEC是一种更为简单、高效且经济的DNA组装技术,应用更加广泛,但不适合高GC含量DNA片段的组装。
除此以外,为了方便引物设计,基于Golden Gate、Gibson等组装方法,新一代DNA组装的辅助设计软件J5 Device Editor也逐渐得到普及[15]。
2.2 大肠杆菌基因组编辑技术在基因组上创建代谢途径,获得遗传性状稳定的大肠杆菌,离不开基因组编辑技术。通过基因编辑,实现外源基因的插入、内源基因敲除以及关键基因的表达调控。
同源重组系统是大肠杆菌基因组编辑技术的基础。大肠杆菌中最为常用的系统是Red同源重组系统[16],它来源于λ噬菌体,包括3个蛋白Exo、Beta、Gam[17],在这3种蛋白的协同下,实现外源片段的同源重组。其同源重组效率较高,同源臂可短至50 bp,操作简单,广泛地应用于大肠杆菌基因编辑中。基于Red同源重组系统,结合不同的策略,研究人员开发出了无抗性的基因组编辑技术,常用的有以下3种:(1) 基于Flp重组酶的两步同源重组[18]:Flp重组酶能够识别短翻转酶识别位点序列,并能切除位于两个翻转酶识别位点(Flippase recognition target,FRT)之间的序列。结合Red同源重组系统,先将两端带有FRT序列的抗性基因整合进基因组上的目标位点,再在Flp重组酶的介导下将抗性基因敲除。该技术的缺点是会在目标位点上留下一段FRT序列,并非无痕编辑,在多轮编辑后会影响基因的编辑效率。(2) 基于sacB基因的两步正负筛选法[19]:sacB基因编码分泌性蔗糖果聚糖酶,能催化蔗糖水解成葡萄糖和果糖,并将果糖合成为高分子量的果聚糖,对大肠杆菌产生致死作用,是一种负筛选标记。在Red同源重组系统介导下,先将带有抗性基因和sacB基因的片段整合进目标位点,再将靶片段整合进目标位点,替换掉带有抗性基因和sacB基因的基因片段,在含有蔗糖的培养基中培养,通过反向筛选策略,获得替换掉抗性基因和sacB基因片段的正确转化子。(3) 基于CRISPR/Cas9的一步同源重组:Cas9蛋白在gRNA介导下在靶位点发生剪切,使大肠杆菌基因组双链断裂,产生致死效果。Red同源重组系统可将供体片段整合进靶位点,修复断裂的DNA双链,或是在Cas9蛋白剪切前发生同源重组,使Cas9丢失靶位点而不发生剪切。2013年张锋课题组[20]证实了CRISPR/Cas9对大肠杆菌进行基因编辑的可行性。2015年中国科学院上海植物生理生态研究所在大肠杆菌中建立了完善的CRISPR/Cas9双质粒编辑系统[21],将Cas9、Red建立在一个质粒上,将N20-gRNA、供体DNA片段构建在另一个质粒上,利用该系统,大肠杆菌单基因编辑的效率可达100%。
除同源重组外,研究人员开发出了对碱基直接实现编辑的技术。David Liu团队[22]通过将CRISPR/Cas9和脱氨酶融合表达,实现了C·T、A·G的碱基转换;新型GBE碱基编辑器还能在大肠杆菌中实现C·A的颠换[23],从而实现大肠杆菌中碱基的任意编辑。这些新技术拓展了大肠杆菌基因编辑的类型,提高了基因编辑的效率。
2.3 基因表达调控技术基因表达调控是优化代谢途径的重要手段,是代谢工程改造的核心。在大肠杆菌中创建途径之后,需要对关键节点的基因进行表达强度的调控,包括增强产物合成途径和辅因子合成途径,或弱化一些敲除后会导致菌体不生长但对产品生产有不利影响的基因。根据技术层次,分为单基因调控、多基因调控以及基因动态调控技术。
(1) 单基因调控:单基因调控是在染色体上对代谢途径某个特定基因的表达进行调控。代谢工程早期,常用强启动子进行单基因调控,如IPTG诱导型Tac启动子、T7启动子等。但对于代谢途径,单基因的强表达可能不是最优方式,会导致代谢负荷。为实现单基因的最优表达,Lu等[24]在大肠杆菌中构建了启动子文库,获得了不同强度的启动子,用于葡萄糖转运蛋白基因galP和葡萄糖激酶基因glk的调控,组合调控菌株GalP93-Glk37显著提高了葡萄糖的消耗速率[25]。除了启动子文库,Chen等[26]利用RBS文库对丁二酸转运蛋白DcuB和DcuC单基因调控,提高了向细胞外转运丁二酸的能力和产量。
随着人工智能的发展,Wang等[27]探索了大量的潜在有效序列,开发了一种新型的基于AI的大肠杆菌启动子从头设计框架。通过深度学习技术建立启动子产生模型,创建新的人工启动子。这一技术的应用将提高单基因调控的效率,降低成本。
(2) 多基因调控:单基因调控只能在单基因维度上操作,即使组合调控,其文库的容量也大大减小,很难筛选出多个基因的最优组合。在代谢途径的优化中,为实现目标化学品的高效生产,常需要多个基因的协同表达才能达到代谢途径的优化。Keasling团队[28]开发了一种可调控基因间区域(Tunable intergenic region,TIGRs) 文库技术。该技术基于基因间序列改变会对基因表达强度产生影响的原理,实现了在一个操纵子内同时对多个基因的表达强度调控的目的。使用这一技术,大肠杆菌中甲羟戊酸途径的多个基因实现了协同表达,甲羟戊酸产量提高了7倍。Isaacs等[29]开发了多重自动基因组编辑技术(Multiplex automated genome engineering,MAGE),该技术利用Red同源重组系统,通过自动化循环设备,对多个基因位点进行循环编辑,提高了基因编辑效率。基于CRISPR/Cas9技术,Zhu等[30]在染色体上实现对木糖代谢途径3个基因文库调控,筛选到的最优菌株,其木糖代谢速率提高了3倍。
(3) 基因动态调控:动态调控是代谢途径优化中最有效的策略之一。虽然静态调控如单基因和多基因调控易操作、效果显著,但细胞内代谢物是动态的,若相关基因表达量过高会浪费细胞资源,过低表达又会限制代谢通路,最终目标产物的产率和产量受细胞代谢失衡而难以提升。为了适时地平衡产物合成所需的基因表达与全局代谢水平的关系,基因动态调控的引入能够实时响应代谢信号,并及时进行反馈调节,适应细胞内部代谢和环境的变化。例如,研究人员开发了一种动态感知-调控系统(Dynamic sensor-regulator system,DSPS),能够在大肠杆菌中根据宿主的代谢状态调节代谢途径,进而控制脂肪酸乙酯(Fatty acid ethyl ester,FAEE) 的生产。这一策略使用了一个检测胞内代谢中间产物乙酰辅酶A的检测器,并且它的同源调控因子——响应传感器的启动子被设计用来控制FAEE生物合成途径相关基因的表达。该系统根据胞内乙酰辅酶A的浓度动态调控整条代谢途径的生产与消耗,最终不仅将FAEE的产量提高了3倍,还降低了有毒代谢中产物的浓度,并且提高了菌株的遗传稳定性[31]。除脂肪酸外,动态调控策略已经应用于番茄红素、氨基酸、肌醇等[32]高附加值生物化学品的合成,实现了目标产物的大幅度增产。
3 细胞全局优化大肠杆菌经过途径改造,还需要通过全细胞优化来实现产量、生产速率及转化率的提高,这些技术手段包括辅因子工程、全局转录因子工程、生物传感器、适应性进化和生理改造。全细胞优化后的菌株产业化优势更显著:菌株环境耐受性更强,生产的稳定性更好,产品的经济性更优(图 3)。

|
|
图 3 细胞全局优化
Fig. 3 Optimization of global cellular performance.
|
| |
辅因子包括还原力NADH/NAD+、NADPH/ NADP+及能量ATP/ADP等。它们为生物合成与分解反应提供氧化还原载体,是细胞内能量传递的重要代谢因子[33]。辅因子在胞内的形式及浓度,将影响代谢网络、信号转导和物质转运,进而影响细胞全局的生理功能。控制辅因子所采用的分子生物学手段称为辅因子工程,它通过改造细胞内辅因子的再生途径,调控辅因子的形式和浓度,定向改变和优化微生物细胞代谢功能,最终实现目标产物碳代谢流的最大化[34]。常用的辅因子工程技术策略如表 1所示。
| Cofactor | Strategy | Case | Reference |
| NAD(P)H | Increase | Activate PPP pathway to synthesize succinate | [36] |
| Activate the ED pathway to synthesize terpenoids | [37] | ||
| Balance | Activate pntAB+yfjB(NADH→NADPH) to synthesize isobutanol | [38] | |
| Activate udhA(NADPH→NADH) to synthesize butanol | [39] | ||
| Change the bias | 2, 5-diketo-D-gulonic acid reductase(NADPH→NADH) | [41] | |
| Ketate reductase(KARI) (NADPH→NADH) | [42] | ||
| Computational simulation CSR-SALAD | [44] | ||
| ATP | Proton-motive force | Synthesis of succinate from glycerol | [45] |
在代谢途径中,作为底物或产物,还原力NADH和NADPH参与了化合物的分解或合成反应。研究表明,还原力供给与需求的平衡是维持细胞正常代谢的一项基本需求[34]。实现代谢途径还原力的平衡,是提高终产物的产量和转化率的先决条件。
(1) 利用磷酸戊糖途径(PPP) 和2-酮-3-脱氧-6-磷酸葡萄糖酸途径(ED) 供给还原力
丁二酸是一种重要的平台化合物,在大肠杆菌中创建丁二酸合成途径,利用还原性三羧酸循环,厌氧条件下合成1 mol丁二酸需要消耗2 mol NADH,尽管糖酵解能提供2 mol NADH,但糖酸转化率只有1 mol/mol[35]。若用C5磷酸戊糖途径代替C6糖酵解途径,使85.7%的碳流向PPP途径,理论上每1 mol葡萄糖通过PPP途径产生2 mol NADPH,结合转氢酶SthA,将葡萄糖代谢产生的还原力从2 mol提高到3.67 mol,丁二酸理论转化率可达到1.71 mol/mol [6]。Tan等[36]在大肠杆菌中激活PPP途径,结合转氢酶SthA的转化,将丁二酸的糖酸转化率从1.12 mol/mol提高到1.61 mol/mol (理论最大转化率的94%),实现了PPP途径作为还原力供给合成丁二酸的新途径。
ED途径,将1 mol葡萄糖代谢生成2 mol丙酮酸时,能产生1 mol ATP、1 mol NADH和1 mol NADPH。相比于糖酵解途径,其途径短、效率高;相比于PPP途径,其没有碳损失。因此,ED途径也常作为NADPH的来源。Ng等[37]将来源于运动假单胞菌的ED途径在大肠杆菌中组装,并利用了RBS库调控ED途径基因的表达,结果显示,使用了ED途径的大肠杆菌,产生的NADPH比野生型提高了25倍,用于类异戊二烯化合物合成途径MEP生产萜类化合物,链孢红素(一种萜类化合物) 的产量提高了97%。
(2) 利用转氢酶实现代谢途径还原力需求类型的转化和平衡
Shi等[38]在大肠杆菌中创建了异丁醇的合成途径,该途径需要使用NADPH作为还原力,但细胞在糖酵解的过程中产生的还原力类型是NADH形式,导致还原力的供给类型与需求类型之间不平衡,影响了异丁醇的最终产率。为解决这个问题,作者对转氢酶基因pntAB (将质子H从NADH转到NADP+上生成NADPH) 和NAD激酶基因yfjB (将NAD+转化为NADP+) 用人工调控元件和组合方式增强表达,实现还原力类型从NADH到NADPH的高效转化。经过辅因子工程改造的菌株,在厌氧条件下,异丁醇产量提高了80%,产率提高了39%,达到0.92 mol/mol,接近理论最大值。类似地,在大肠杆菌中构建的梭菌丁醇合成途径需要还原力NADH的参与,且合成1 mol丁醇需要消耗4 mol NADH,但糖酵解分解1 mol葡萄糖仅仅产生2 mol NADH,导致还原力的供给和需求的不平衡。为产生更多的NADH来驱动丁醇的合成,Saini等[39]过表达转氢酶基因udhA (催化NADPH转换为NADH),实现还原力类型从NADPH到NADH的转化,胞内的NADH的含量增加了36%,丁醇产量增加了25.6%。
(3) 利用酶工程改变辅因子的偏好性
代谢途径中,还原力供给或需要类型的转化除了通过转氢酶来实现,还可以通过酶工程的手段实现。即对相关的氧化还原酶进行定点或随机突变,改变其对辅因子的偏好性(如从NADH到NADPH或反之),从而实现代谢途径还原力的平衡[40]。例如,2, 5-二酮基-d-古龙酸还原酶(2, 5-DKG) 是维生素C合成途径中的关键酶,辅因子为NADPH,但途径供给的还原力类型为NADH。为实现辅因子供给和需求类型的平衡,Banta等[41]利用酶工程进行定点突变,获得的四突变F22Y/K232G/R238H/A272G 2, 5-DKG对NADH的亲和力提高了2倍,成功实现了2, 5-DKG辅因子偏好性由NADPH向NADH的转变,解决了胞内辅因子的平衡问题,提高了维生素C的生产。类似的改造如异丁醇合成途径中的关键酶酮酸还原酶(KARI) 辅因子偏好性从NADPH改变为NADH,实现了途径还原力平衡,使大肠杆菌厌氧异丁醇合成达到100%的理论转化率[42]。
随着计算机科学的发展,酶工程辅因子改造的效率得以极大地提升。利用计算机程序化的模拟、计算和分析,自动化高通量地设计出辅因子偏好性改变的酶。Cui等利用计算机程序,评估酶和辅因子相互作用的氢键强弱,结合分子动力学参数,获得需要改造的蛋白质位点实现辅因子偏好性的转化[43]。近来一个新的用于辅因子偏好性改变的计算工具,称为辅因子特异性逆转-结构分析和库设计(CSR-SALAD),计算网站为http://www.che.caltech.edu/groups/fha/CSRSALAD/index.html,其可操作性强,为代谢途径中酶辅因子偏好性改造提供了良好的工具,极大地减少了酶辅因子改造的工作量[44]。
3.1.2 能量ATP在代谢途径中除了考虑还原力的因素外,还需要考虑能量ATP的合成。细胞需产生足够的ATP供细胞生长和产物合成。甘油是一种高还原力碳源,用于丁二酸生产有较大优势:1 mol甘油与1 mol CO2理论上可合成1 mol丁二酸。但在厌氧条件下,从甘油到丁二酸的合成途径不能产生足够的ATP,导致丁二酸产量和生产速率相当低。Yu等[45]使用来自克雷伯氏菌的ATP依赖型二羟基丙酮激酶(DhaK) 替代了大肠杆菌原有的PEP依赖型二羟基丙酮激酶(DhaKLM),在大肠杆菌中创建出一条新的甘油厌氧代谢途径。和原有途径相比,新途径能产生质子动力势为细胞生长和丁二酸外排提供额外能量。丁二酸产量、比生产速率和胞内ATP含量分别增加了282%、63%和338%。利用质子动力势提高ATP供给也为途径中能量代谢调控提供了思路。
3.2 全局转录因子工程在优化细胞生理性能时,很多情况下无法确定明确的靶基因和调控因子。过去科学家们大多是通过随机诱变的方法获得生理性能优化了的突变菌株。然而这种方法会在细胞基因组上造成随机突变,其中很多对细胞生长代谢不利。突变菌株虽然提高了特定的生理性能,但同时也伴随了很多负面的作用。细胞全局转录因子工程(Global transcription machinery engineering,gTME) 通过对转录复合体成分,尤其是负责DNA序列识别从而决定RNA聚合酶结合偏好性的转录因子进行定性进化,对细胞整体的转录发生扰动,筛选相应细胞性状的策略。该策略可以有针对性地在细胞基因组上造成改变,筛选目标生理性能,获得遗传背景清晰的代谢工程菌。Alper等[46]利用易错PCR,对大肠杆菌关键转录机器组分sigma因子(rpoD基因编码) 突变,获得乙醇的耐受突变株。在50 g/L乙醇条件下倍增时间为3.5 h,而前人所得到的乙醇耐性最好的E. coli菌株在同等条件下倍增时间为4–6 h。转录水平分析显示,在20 g/L乙醇条件下,培养突变株的354个基因的转录水平有变化,这些基因都与逆境胁迫响应有关,其中也包括乙醇胁迫。同遗传改造相比,细胞全局转录因子工程不需要制备感受态,操作方法简单,着眼于调控细胞整体基因转录,定向性强,具有较强的工业菌株应用前景。
3.3 生物传感器(Biosensor)经过内源性代谢途径优化或异源性途径的整合和调控,大肠杆菌细胞内的物质和能量重新分配,最大限度地生产特定化合物。但同时也会出现细胞“逃逸”现象:代谢途径中基因的过表达会对细胞造成代谢负荷,造成代谢失衡;中间代谢物的积累或耗竭可能会损害细胞活力[47];过表达的异源蛋白会消耗额外的能量,引起细胞应激反应[48];实验室工程菌株无法适应工业生产环境维持代谢稳态,发生变异[49-50]。解决的方案之一就是设计生物传感器(感应-传导-执行),让细胞能接收反馈信号,自主调整通路的表达,对抗代谢异质性和环境压力,实现全细胞优化。
代谢负荷生物传感器:Ceroni等[51]利用转录组分析,得到了细胞负荷感应启动子htpG,它能对细胞的非天然状态(如基因过表达) 发生信号响应。利用该启动子控制sgRNA (靶向需要控制的基因启动子) 的表达,当细胞中基因过表达产物过多,对细胞造成负荷,htpG启动子接收信号,启动sgRNA的表达,在dCas9的协助下,负控制基因启动子的表达,解除基因过表达对细胞产生的代谢负荷。
群体质量控制生物传感器(PopQC):群体质量控制系统包括一个基于转录调控因子(FadR/TryR) 的生物传感器,该生物传感器可以正向响应中间代谢物或终产物(游离脂肪酸/酪氨酸) 的浓度。在低产细胞或非生产细胞内,转录调控因子可以结合在启动子(PAR/PT1/2) 上,抑制抗性基因tetA的表达,从而清除低产菌株;而在高产细胞内,转录因子解除对启动子的阻遏,抗性基因表达,高产菌株富集。Xiao等[52]将该策略用于酪氨酸合成,使酪氨酸的产量提高了2.6倍。
除了上述的生物传感器,还有代谢物-成瘾生物传感器[53],以及基于群体感应系统建立的多层级生物传感器[54]等,这些生物传感器的开发和应用,为大肠杆菌细胞工厂发酵性能优化提供了更多的思路。
3.4 适应性进化对细胞生理性能的优化,当通过代谢工程的理性改造达不到目标时,可通过非理性的适应性进化实现。即通过在发酵反应器中连续传代培养微生物,在一定压力条件或生长偶联方式累积目标突变,获得生理性能显著提高或代谢途径更加优化的菌株。
通过适应性进化,可以获得适合工业化环境的大肠杆菌工程菌株,如在低pH、高浓度盐和高温等压力环境条件下进行适应性进化,可获得耐酸[55]、耐高渗[6, 56]和耐高温[57]等优良性能的菌株。同时,结合突变技术,如化学诱变[58]、紫外诱变[59-60]和改变细胞的DNA修复系统[61-62]等,可加快适应性进化的效率。
组学(重测序、转录组、蛋白质组和代谢组)技术的发展为适应进化菌株的深度表征提供了方法[63-65]。对适应进化菌株,通过基因组重测序对突变进行基因型表征;对突变基因进行转录组和酶活分析,建立表型和基因型合理的联系。在研究过程中,多种分析策略组合应用,将有助于合理解释观察到表型的潜在机制[65],也有助于用理性的方式对细胞生理进行改造。
3.5 生理改造改变细胞膜或细胞形态的大小等生理性状将有效提高目标代谢产物的装载量,是提高微生物细胞工厂效率的有效策略之一。
Wu等[66]研究了大肠杆菌细胞膜改造对提高萜类化合物合成能力的影响。过表达与膜小泡合成相关的单葡萄糖甘油二酯合成酶Almgs,将β-胡萝卜素的单位细胞含量提高了70%,进一步增强了细胞膜主要成分甘油磷脂的合成途径,将β-胡萝卜素的单位细胞含量提高了2.9倍(由6.7 mg/g CDW提高至19.6 mg/g CDW)。经过膜工程改造的大肠杆菌细胞,其电镜图像有明显的膜堆积,膜的形态改变与β-胡萝卜素储存容量相关。为提高β-胡萝卜素的外排能力,作者进一步对膜囊泡系统进行了改造,构建了人工膜囊泡系统(Artificial membrane vesicle trafficking system,AMVTS)。含有AMVTS系统的菌株,使β-胡萝卜素的分泌量提高13.7%,产量与对照菌株相比提高35.6%;使高产菌株CAR025中β-胡萝卜素产量提高61%[67]。膜形态的改变和AMVTS膜囊泡转运系统提高了疏水萜类化合物在细胞内的产量和外排能力,为创建高产疏水化合物的细胞工厂提供了新的思路,有广泛的应用前景。
Chen等[68]研究了大肠杆菌细胞形态的大小对提高聚羟基脂肪酸酯(Polyhydroxyalkanoates,PHA) 产量的影响。大肠杆菌通常的直径大小为0.5–2 μm,通过降低mreB基因(参与细胞骨架的形成)的表达,细胞的大小增加,直径约为10 μm;进一步诱导sulA基因(参与抑制细胞的分裂) 的表达,细胞成为细长型,聚羟基丁酸(Poly-hydroxybutrate,PHB) 积累增加了100%。与此类似,Ding等[69]通过调控大肠杆菌的细胞分裂实现了化学品的生产:过表达与分裂相关的蛋白(FtsZA) 和核糖核苷还原酶(NrdAB),有效缩短细胞分裂周期,缩小细胞形态,提高比表面积(SSA),用于大肠杆菌乙偶姻发酵,产量增加到67.2 g/L,比野生型提高了39.19%;核糖核苷还原酶的弱表达或分裂蛋白抑制剂SulA、MinC、MinD、MinE和FtsH的过表达,可延长细胞分裂周期,扩大细胞形态,提高平均细胞体积(Mean cell volume,MCV),用于聚乳酸-co-3-羟基丁酸(Polylactate-co-3-hydroxybutrate,PLH) 合成,大肠杆菌的PLH含量增加至53.8 wt%,是出发菌株DQ0的2.01倍。改变细胞形态提高细胞目标产物的策略,为细胞工厂的构建提供了新的方法。
4 总结与展望自然界中单一生命已有合成途径的化学品,相对得到了比较深入的开发,工业化应用案例较多。随着基因测序与解析技术的发展,越来越多的未知代谢途径得到解析,更多的功能基因也得到发掘。对不同来源的代谢途径进行组合,可以合成一系列自然界中没有的化学品生物合成途径。在计算生物学、化学生物学、蛋白质科学的交叉融合下,甚至可以获得一系列催化非天然反应的酶,从而进一步拓展代谢工程的应用范围。大肠杆菌由于遗传背景清晰,又具备极为丰富的代谢元件库和完备的基因组编辑技术,成为了最适合进行代谢工程改造实验的底盘菌株之一。近年来,大肠杆菌在CO2固定和甲醇利用方面取得了一系列的新进展[11],经过代谢工程改造的大肠杆菌甚至能够以甲醇为唯一碳源生长,在碳的循环利用方面作出了突出贡献[70]。相信随着人工智能以及工程化理性设计的发展,结合日益丰富先进的生物技术,大肠杆菌代谢工程将实现理性化、工程化、智能化的革命,化学品的生物制造成本更低,具有更强的竞争力。但大肠杆菌亦存在诸多不足,如不具有某些关键辅因子如维生素B12的合成途径;糖基化和磷酸化修饰能力弱、不易形成复杂的二硫键导致外源基因表达困难等。随着CRISPR/Cas9等基因组编辑技术的发展,酵母菌、丝状真菌以及其他原核生物的代谢改造工具也日趋完备,未来将有更多的宿主菌可供选择,便于构建更具竞争力的细胞工厂。
| [1] |
Zhang XL, Jantama K, Moore JC, et al. Production of L-alanine by metabolically engineered Escherichia coli. Appl Microbiol Biotechnol, 2007, 77(2): 355-366. DOI:10.1007/s00253-007-1170-y
|
| [2] |
Becker J, Zelder O, Häfner S, et al. From zero to hero—Design-based systems metabolic engineering of Corynebacterium glutamicum for L-lysine production. Metab Eng, 2011, 13(2): 159-168. DOI:10.1016/j.ymben.2011.01.003
|
| [3] |
Lee KH, Park JH, Kim TY, et al. Systems metabolic engineering of Escherichia coli for L-threonine production. Mol Syst Biol, 2007, 3: 149. DOI:10.1038/msb4100196
|
| [4] |
Nakamura CE, Whited GM. Metabolic engineering for the microbial production of 1, 3-propanediol. Curr Opin Biotechnol, 2003, 14(5): 454-459. DOI:10.1016/j.copbio.2003.08.005
|
| [5] |
Zhou SD, Causey TB, Hasona A, et al. Production of optically pure D-lactic acid in mineral salts medium by metabolically engineered Escherichia coli W3110. Appl Environ Microbiol, 2003, 69(1): 399-407. DOI:10.1128/AEM.69.1.399-407.2003
|
| [6] |
Zhu XN, Tan ZG, Xu HT, et al. Metabolic evolution of two reducing equivalent-conserving pathways for high-yield succinate production in Escherichia coli. Metab Eng, 2014, 24: 87-96. DOI:10.1016/j.ymben.2014.05.003
|
| [7] |
Ma WC, Chen KQ, Li Y, et al. Advances in cadaverine bacterial production and its applications. Engineering, 2017, 3(3): 308-317. DOI:10.1016/J.ENG.2017.03.012
|
| [8] |
Thiele I, Palsson BØ. A protocol for generating a high-quality genome-scale metabolic reconstruction. Nat Protoc, 2010, 5(1): 93-121. DOI:10.1038/nprot.2009.203
|
| [9] |
Zhang XL, Tervo CJ, Reed JL. Reed JL. Metabolic assessment of E. coli as a biofactory for commercial products. Metab Eng, 2016, 35: 64-74. DOI:10.1016/j.ymben.2016.01.007
|
| [10] |
Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1, 4-butanediol. Nat Chem Biol, 2011, 7(7): 445-452. DOI:10.1038/nchembio.580
|
| [11] |
Lu XY, Liu YW, Yang YQ, et al. Constructing a synthetic pathway for acetyl-coenzyme A from one-carbon through enzyme design. Nat Commun, 2019, 10(1): 1378. DOI:10.1038/s41467-019-09095-z
|
| [12] |
Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability. PLoS ONE, 2008, 3(11): e3647. DOI:10.1371/journal.pone.0003647
|
| [13] |
Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods, 2009, 6(5): 343-345. DOI:10.1038/nmeth.1318
|
| [14] |
Quan JY, Tian JD. Circular polymerase extension cloning for high-throughput cloning of complex and combinatorial DNA libraries. Nat Protoc, 2011, 6(2): 242-251. DOI:10.1038/nprot.2010.181
|
| [15] |
Hillson NJ, Rosengarten RD, Keasling JD. j5 DNA assembly design automation software. ACS Synth Biol, 2012, 1(1): 14-21. DOI:10.1021/sb2000116
|
| [16] |
Murphy KC. Use of bacteriophage λ recombination functions to promote gene replacement in Escherichia coli. J Bacteriol, 1998, 180(8): 2063-2071. DOI:10.1128/JB.180.8.2063-2071.1998
|
| [17] |
Sharan SK, Thomason LC, Kuznetsov SG, et al. Recombineering: a homologous recombination-based method of genetic engineering. Nat Protoc, 2009, 4(2): 206-223. DOI:10.1038/nprot.2008.227
|
| [18] |
Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci USA, 2000, 97(12): 6640-6645. DOI:10.1073/pnas.120163297
|
| [19] |
Zhang YM, Muyrers JPP, Testa G, et al. DNA cloning by homologous recombination in Escherichia coli. Nat Biotechnol, 2000, 18(12): 1314-1317. DOI:10.1038/82449
|
| [20] |
Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508
|
| [21] |
Li YF, Lin ZQ, Huang C, et al. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing. Metab Eng, 2015, 31: 13-21. DOI:10.1016/j.ymben.2015.06.006
|
| [22] |
Anzalone AV, Randolph PB, Davis JR, et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature, 2019, 576(7785): 149-157. DOI:10.1038/s41586-019-1711-4
|
| [23] |
Zhao DD, Li J, Li SW, et al. Glycosylase base editors enable C-to-A and C-to-G base changes. Nat Biotechnol, 2020. DOI:10.1038/s41587-020-0592-2
|
| [24] |
Lu J, Tang JL, Liu Y, et al. Combinatorial modulation of galP and glk gene expression for improved alternative glucose utilization. Appl Microbol Biotechnol, 2012, 93(6): 2455-2462. DOI:10.1007/s00253-011-3752-y
|
| [25] |
Tang JL, Zhu XN, Lu J, et al. Recruiting alternative glucose utilization pathways for improving succinate production. Appl Microbiol Biotechnol, 2013, 97(6): 2513-2520. DOI:10.1007/s00253-012-4344-1
|
| [26] |
Chen J, Zhu XN, Tan ZG, et al. Activating C4-dicarboxylate transporters DcuB and DcuC for improving succinate production. Appl Microbiol Biotechnol, 2014, 98(5): 2197-2205. DOI:10.1007/s00253-013-5387-7
|
| [27] |
Wang Y, Wang HC, Wei L, et al. Synthetic promoter design in Escherichia coli based on a deep generative network. Nucleic Acids Res, 2020, 48(12): 6403-6412. DOI:10.1093/nar/gkaa325
|
| [28] |
Pfleger BF, Pitera DJ, Smolke CD, et al. Combinatorial engineering of intergenic regions in operons tunes expression of multiple genes. Nat Biotechnol, 2006, 24(8): 1027-1032. DOI:10.1038/nbt1226
|
| [29] |
Isaacs FJ, Carr PA, Wang HH, et al. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement. Science, 2011, 333(6040): 348-353. DOI:10.1126/science.1205822
|
| [30] |
Zhu XN, Zhao DD, Qiu HN, et al. The CRISPR/Cas9-facilitated multiplex pathway optimization (CFPO) technique and its application to improve the Escherichia coli xylose utilization pathway. Metab Eng, 2017, 43: 37-45. DOI:10.1016/j.ymben.2017.08.003
|
| [31] |
Zhang FZ, Carothers JM, Keasling JD. Design of a dynamic sensor-regulator system for production of chemicals and fuels derived from fatty acids. Nat Biotechnol, 2012, 30(4): 354-359. DOI:10.1038/nbt.2149
|
| [32] |
Gao C, Xu P, Ye C, et al. Genetic circuit-assisted smart microbial engineering. Trends Microbiol, 2019, 27(12): 1011-1024. DOI:10.1016/j.tim.2019.07.005
|
| [33] |
Wang YP, San KY, Bennett GN. Cofactor engineering for advancing chemical biotechnology. Curr Opin Biotechnol, 2013, 24(6): 994-999. DOI:10.1016/j.copbio.2013.03.022
|
| [34] |
Wang M, Chen BQ, Fang YM, et al. Cofactor engineering for more efficient production of chemicals and biofuels. Biotechnol Adv, 2017, 35(8): 1032-1039. DOI:10.1016/j.biotechadv.2017.09.008
|
| [35] |
Chatterjee R, Millard CS, Champion K, et al. Mutation of the ptsG gene results in increased production of succinate in fermentation of glucose by Escherichia coli. Appl Environ Microbiol, 2001, 67(1): 148-154. DOI:10.1128/AEM.67.1.148-154.2001
|
| [36] |
Tan ZG, Chen J, Zhang XL. Systematic engineering of pentose phosphate pathway improves Escherichia coli succinate production. Biotechnol Biofuels, 2016, 9: 262. DOI:10.1186/s13068-016-0675-y
|
| [37] |
Ng CY, Farasat I, Maranas CD, et al. Rational design of a synthetic Entner-Doudoroff pathway for improved and controllable NADPH regeneration. Metab Eng, 2015, 29: 86-96. DOI:10.1016/j.ymben.2015.03.001
|
| [38] |
Shi AQ, Zhu XN, Lu J, et al. Activating transhydrogenase and NAD kinase in combination for improving isobutanol production. Metab Eng, 2013, 16: 1-10. DOI:10.1016/j.ymben.2012.11.008
|
| [39] |
Saini M, Li SY, Wang ZW, et al. Systematic engineering of the central metabolism in Escherichia coli for effective production of n-butanol. Biotechnol Biofuels, 2016, 9: 69. DOI:10.1186/s13068-016-0467-4
|
| [40] |
Zhao HM, Van Der Donk WA. Regeneration of cofactors for use in biocatalysis. Curr Opin Biotechnol, 2003, 14(6): 583-589. DOI:10.1016/j.copbio.2003.09.007
|
| [41] |
Banta S, Swanson BA, Wu S, et al. Optimizing an artificial metabolic pathway: engineering the cofactor specificity of Corynebacterium 2, 5-diketo-D-gluconic acid reductase for use in vitamin C biosynthesis. Biochemistry, 2002, 41(20): 6226-6236. DOI:10.1021/bi015987b
|
| [42] |
Brinkmann-Chen S, Flock T, Cahn JKB, et al. General approach to reversing ketol-acid reductoisomerase cofactor dependence from NADPH to NADH. Proc Natl Acad Sci USA, 2013, 110(27): 10946-10951. DOI:10.1073/pnas.1306073110
|
| [43] |
Cui DB, Zhang LJ, Jiang SQ, et al. A computational strategy for altering an enzyme in its cofactor preference to NAD(H) and/or NADP(H). FEBS J, 2015, 282(12): 2339-2351. DOI:10.1111/febs.13282
|
| [44] |
Cahn JKB, Werlang CA, Baumschlager A, et al. A general tool for engineering the NAD/NADP cofactor preference of oxidoreductases. ACS Synth Biol, 2017, 6(2): 326-333. DOI:10.1021/acssynbio.6b00188
|
| [45] |
Yu Y, Zhu XN, Xu HT, et al. Construction of an energy-conserving glycerol utilization pathways for improving anaerobic succinate production in Escherichia coli. Metab Eng, 2019, 56: 181-189. DOI:10.1016/j.ymben.2019.10.002
|
| [46] |
Alper H, Stephanopoulos G. Global transcription machinery engineering: a new approach for improving cellular phenotype. Metab Eng, 2007, 9(3): 258-267. DOI:10.1016/j.ymben.2006.12.002
|
| [47] |
Leonard E, Ajikumar PK, Thayer K, et al. Combining metabolic and protein engineering of a terpenoid biosynthetic pathway for overproduction and selectivity control. Proc Natl Acad Sci USA, 2010, 107(31): 13654-13659. DOI:10.1073/pnas.1006138107
|
| [48] |
Zelcbuch L, Antonovsky N, Bar-Even A, et al. Spanning high-dimensional expression space using ribosome-binding site combinatorics. Nucleic Acids Res, 2013, 41(9): e98. DOI:10.1093/nar/gkt151
|
| [49] |
Rugbjerg P, Sarup-Lytzen K, Nagy M, et al. Synthetic addiction extends the productive life time of engineered Escherichia coli populations. Proc Natl Acad Sci USA, 2018, 115(10): 2347-2352. DOI:10.1073/pnas.1718622115
|
| [50] |
Rugbjerg P, Myling-Petersen N, Porse A, et al. Diverse genetic error modes constrain large-scale bio-based production. Nat Commun, 2018, 9: 787. DOI:10.1038/s41467-018-03232-w
|
| [51] |
Ceroni F, Boo A, Furini S, et al. Burden-driven feedback control of gene expression. Nat Methods, 2018, 15(5): 387-393. DOI:10.1038/nmeth.4635
|
| [52] |
Xiao Y, Bowen CH, Liu D, et al. Exploiting nongenetic cell-to-cell variation for enhanced biosynthesis. Nat Chem Biol, 2016, 12(5): 339-344. DOI:10.1038/nchembio.2046
|
| [53] |
Xiong MY, Schneiderman DK, Bates FS, et al. Scalable production of mechanically tunable block polymers from sugar. Proc Natl Acad Sci USA, 2014, 111(23): 8357-8362. DOI:10.1073/pnas.1404596111
|
| [54] |
Doong SJ, Gupta A, Prather KLJ. Layered dynamic regulation for improving metabolic pathway productivity in Escherichia coli. Proc Natl Acad Sci USA, 2018, 115(12): 2964-2969. DOI:10.1073/pnas.1716920115
|
| [55] |
Hughes BS, Cullum AJ, Bennett AF. Evolutionary adaptation to environmental pH in experimental lineages of Escherichia coli. Evolution, 2007, 61(7): 1725-1734. DOI:10.1111/j.1558-5646.2007.00139.x
|
| [56] |
Xiao MY, Zhu XN, Fan FY, et al. Osmotolerance in Escherichia coli is improved by activation of copper efflux genes or supplementation with sulfur-containing amino acids. Appl Environ Microbiol, 2017, 83(7): e03050-16.
|
| [57] |
Tenaillon O, Rodríguez-Verdugo A, Gaut RL, et al. The molecular diversity of adaptive convergence. Science, 2012, 335(6067): 457-461. DOI:10.1126/science.1212986
|
| [58] |
Lee CH, Gilbertson DL, Novella IS, et al. Negative effects of chemical mutagenesis on the adaptive behavior of vesicular stomatitis virus. J Virol, 1997, 71(5): 3636-3640. DOI:10.1128/JVI.71.5.3636-3640.1997
|
| [59] |
Mundhada H, Schneider K, Christensen HB, et al. Engineering of high yield production of L-serine in Escherichia coli. Biotechnol Bioeng, 2016, 113(4): 807-816. DOI:10.1002/bit.25844
|
| [60] |
Mundhada H, Seoane JM, Schneider K, et al. Increased production of L-serine in Escherichia coli through adaptive laboratory evolution. Metab Eng, 2017, 39: 141-150. DOI:10.1016/j.ymben.2016.11.008
|
| [61] |
Antonovsky N, Gleizer S, Noor E, et al. Sugar synthesis from CO2 in Escherichia coli. Cell, 2016, 166(1): 115-125. DOI:10.1016/j.cell.2016.05.064
|
| [62] |
Chou HH, Keasling JD. Programming adaptive control to evolve increased metabolite production. Nat Commun, 2013, 4: 2595. DOI:10.1038/ncomms3595
|
| [63] |
Long CP, Gonzalez JE, Feist AM, et al. Fast growth phenotype of E. coli K-12 from adaptive laboratory evolution does not require intracellular flux rewiring. Metab En, 2017, 44: 100-107. DOI:10.1016/j.ymben.2017.09.012
|
| [64] |
Sandberg TE, Lloyd CJ, Palsson BO, et al. Laboratory evolution to alternating substrate environments yields distinct phenotypic and genetic adaptive strategies. Appl Environ Microbiol, 2017, 83(13): e00410-17.
|
| [65] |
Long CP, Gonzalez JE, Feist AM, et al. Dissecting the genetic and metabolic mechanisms of adaptation to the knockout of a major metabolic enzyme in Escherichia coli. Proc Natl Acad Sci USA, 2018, 115(1): 222-227. DOI:10.1073/pnas.1716056115
|
| [66] |
Wu T, Ye LJ, Zhao DD, et al. Membrane engineering —A novel strategy to enhance the production and accumulation of β-carotene in Escherichia coli. Metab Eng, 2017, 43: 85-91. DOI:10.1016/j.ymben.2017.07.001
|
| [67] |
Wu T, Li SW, Ye LJ, et al. Engineering an artificial membrane vesicle trafficking system (AMVTS) for the excretion of β-carotene in Escherichia coli. ACS Synth Biol, 2019, 8(5): 1037-1046. DOI:10.1021/acssynbio.8b00472
|
| [68] |
Chen GQ, Jiang XR, Guo YY. Synthetic biology of microbes synthesizing polyhydroxyalkanoates (PHA). Synth Syst Biotechnol, 2016, 1(4): 236-242. DOI:10.1016/j.synbio.2016.09.006
|
| [69] |
Ding Q, Ma DL, Liu GQ, et al. Light-powered Escherichia coli cell division for chemical production. Nat Commun, 2020, 11: 2262. DOI:10.1038/s41467-020-16154-3
|
| [70] |
Chen FYH, Jung HW, Tusei CY, et al. Converting Escherichia coli to a synthetic methylotroph growing solely on methanol. Cell, 2020, 182(4): 933-946.e14. DOI:10.1016/j.cell.2020.07.010
|
 2021, Vol. 37
2021, Vol. 37




