中国科学院微生物研究所、中国微生物学会主办
文章信息
- 江丽红, 董昌, 黄磊, 徐志南, 连佳长
- Jiang Lihong, Dong Chang, Huang Lei, Xu Zhinan, Lian Jiazhang
- 酿酒酵母代谢工程技术
- Metabolic engineering tools for Saccharomyces cerevisiae
- 生物工程学报, 2021, 37(5): 1578-1602
- Chinese Journal of Biotechnology, 2021, 37(5): 1578-1602
- 10.13345/j.cjb.200690
-
文章历史
- Received: October 26, 2020
- Accepted: January 11, 2021
- Published: February 1, 2021
2. 浙江大学 化学工程与生物工程学院 合成生物学研究中心,浙江 杭州 310027
2. Center for Synthetic Biology, College of Chemical and Biological Engineering, Zhejiang University, Hangzhou 310027, Zhejiang, China
面临日益严峻的资源、能源和环境问题,生物制造是人类获得新能源、新化学品、新药物和新材料的重要方向。生物制造能够以可再生生物资源作为原料,利用生物催化和转化高效合成高附加值的化合物。整个过程绿色可循环、环境友好,是未来制造的新趋势。例如通过发酵由生物质转化而来的生物乙醇,不仅具有与矿物燃料相当的燃烧性能,而且无污染、可再生,可以取代矿石燃料。同时,在各种重要化学品和药品的制造中,生物制造的占比也越来越大。《生物质技术路线图》和《发展和推进生物质基产品和生物能源》报告中指出,预计到2030年,超过25%的有机化学品将被生物基产品取代[1]。我国近年来生物产业也一直保持年均近12%的快速增长,有望在2025年突破10万亿元的产值规模[2]。
针对生物制造的重大需求,Jay Bailey和Gregory Stephanopoulos于1991年率先提出了代谢工程的概念。代谢工程(Metabolic engineering)是通过优化细胞内遗传和调控过程以提高细胞目标产物产量的方法。代谢工程改造通常基于以下几个目标[3-6]:拓宽和增强细胞多种底物利用能力;提高产物合成能力/底物转化率;增强菌株鲁棒性等。从微观上说,代谢工程的出发点是构建可能需要的新合成途径,然后探究合成途径的潜在影响因素,在此基础上进一步增强目标途径、降低或完全去除旁路途径,增强目标产物积累,从而获得尽可能高的底物到产物的转换效率。
酿酒酵母作为第一个全基因组测序的模式真核生物,也是最早被应用于酿酒、食品等方面的微生物。酿酒酵母具有遗传背景清楚、遗传操作性强、发酵性能好等优点,自代谢工程诞生以来,一直都是首选的代谢工程底盘细胞之一。随着酿酒酵母代谢工程技术的不断发展,对其代谢网络的理解日渐加深,酿酒酵母作为平台细胞工厂的生产效率也得到了极大提升,如今已经被广泛应用于各种食品、化工产品和药品的大规模生产,如生物乙醇、生物丁醇、2, 3-丁二醇、青蒿酸、白藜芦醇和法尼烯等[7-11]。从早期着眼于单个基因或者局部代谢网络的经典代谢工程,过渡到基于多组学分析技术和基因组水平代谢网络模型进行全局代谢网络调控的系统代谢工程,再到如今合成生物学驱动的高通量、可预测代谢工程,本文以时间和技术发展脉络,整理分析了近30年来酿酒酵母代谢工程技术的发展和趋势,并总结归纳了代谢工程技术发展过程中的应用实例(图 1)。

|
| 图 1 酿酒酵母代谢工程技术 Fig. 1 Metabolic engineering technologies of S. cerevisiae, including classical metabolic engineering, systems biology-guided metabolic engineering, and synthetic biology-driven metabolic engineering. |
| |
代谢工程早期在启动子强度、终止子强度、基因拷贝数、辅因子平衡以及调控蛋白等方面对酿酒酵母进行遗传改造。这些经典的代谢工程技术在早期表征了大量表达和调控元件,同时发展了适用于酿酒酵母遗传操作的技术方法,提升了酿酒酵母作为细胞工厂的能力,也为后续新技术的发展奠定了基础。本节归纳总结了酿酒酵母中经典的代谢工程方法和技术。
1.1 基因表达和调控技术 1.1.1 启动子工程代谢工程中经常需要选择合适的启动子来表达或调控目标元件,以构建新的代谢途径,并将基因表达强度调整到适当的水平。不同的代谢工程应用需要不同的启动子特性,选定合适的启动子来驱动途径中的基因转录,合理调整转录强度是提升目标产物含量的第一步。酿酒酵母的启动子比原核生物的启动子更加复杂,序列不保守且大部分元件已经退化[12]。因此在对酿酒酵母启动子的表征中,通常只关注于启动子类型(如组成型、诱导型、双向启动子等)和启动子强度。
针对酿酒酵母内源性启动子的研究较为透彻,多个课题组使用不同的方法对其进行了表征。例如Lee等克隆选取了起始密码子前700 bp作为启动子,一共表征了19个组成型、2个配体特殊型和2个诱导型启动子,其表达强度范围可达1 000倍[13]。Sun等则克隆了14种组成型启动子,并通过绿色荧光蛋白(Green fluorescent protein,GFP) 荧光强度和mRNA转录水平检测其相对强度[14]。
对现有启动子进行工程改造是获得不同强度启动子文库和提高启动子活性的重要手段。Alper等利用定向进化与流式细胞分选(Fluorescence- activated cell sorting,FACS)偶联,通过易错PCR (Polymerase chain reaction) 构建了酿酒酵母TEF1启动子的突变文库,启动子强度可提升2倍以上[15]。Nevoigt等也使用了相同的策略对TEF1启动子进行了突变文库的构建,并筛选得到11个突变体,活性为野生型的8%–120%[16]。采用人工合成启动子的方法来建立强启动子文库以调节基因表达是最新的发展趋势,例如杂交合成启动子文库[17]、依据核小体亲和度极小化原则构建的合成启动子文库[18]、最小合成启动子文库[19]等。这些研究为酿酒酵母的代谢工程提供了不同来源、不同强度、不同大小的启动子元件库,使得研究者可以根据代谢工程的需求选择最优启动子。
1.1.2 终止子工程尽管调控基因表达的大部分研究都是基于启动子特性的,但也有部分研究探究了终止子在酵母中的作用并阐明了其作用机理。Curran等表征了超过30种用于酿酒酵母代谢工程的终止子,并确定mRNA半衰期的变化是导致蛋白质和转录表达水平变化的主要原因[20]。对于强启动子,使用不同终止子时的转录水平差异可以超过6.5倍。当与弱启动子偶联时,终止子选择的影响会进一步放大,表达增强的终止子与CYC1终止子之间的蛋白质表达最大差异为11倍,与无终止子相比,差异超过35倍。在另一项研究中,Yamanishi等表征了酿酒酵母中5 880个基因的5 302个终止子区域(基于TDH3启动子驱动的GFP基因表达),以PGK1终止子为参照,荧光强度分布在0.036–2.52倍之间[21]。上述两个研究均表明终止子能够通过控制mRNA半衰期来精准调节基因的表达水平,从而应用于酿酒酵母的代谢工程改造。
与合成启动子类似,合成终止子在最小终止子和核小体占用最少终止子方面也有相关研究,例如,Curran等设计构建了38–75 bp的一系列元件作为最小终止子,与野生型CYC1终止子相比,效果最好的最小终止子能将荧光强度提高3.7倍[22]。Morse等则根据一个隐藏的Markov模型假定减少核小体占用来提高终止子效率,设计构建了可以将蛋白表达提升4倍的终止子[23]。
1.1.3 拷贝数除了合适的启动子和终止子强度,表达框的拷贝数在很大程度上也能影响基因的表达水平。高拷贝数可以提升途径中关键酶的表达量,因而提高表达框的拷贝数是一种常用而且有效的代谢工程手段。例如,提升质粒的拷贝数可以较为简单快速地应用于游离质粒的表达体系,高拷贝的基因组整合则适用于工业化生产等相关的高稳定性需求的应用场景。
酿酒酵母中有两种常用的质粒类型:低拷贝质粒(CEN/ARS复制子,约1–4个拷贝/细胞)和高拷贝质粒(2μ复制子,约20–30个拷贝/细胞),从而可以在不同的应用场景中选择合适拷贝数的质粒进行代谢工程研究[24]。通过截短驱动标签蛋白表达的启动子[25]和在标签蛋白上融合蛋白降解序列[26]可以显著提高游离质粒的拷贝数。例如,Lian等系统地表征了标签基因启动子截短的效应,获得了一系列不同拷贝数提高的质粒,最高可以超过100个拷贝/细胞[25]。高拷贝质粒已经应用于很多产物的合成,例如番茄红素[25]、广藿香醇[26]和青蒿素前体[10]等,能有效地解决代谢通路中的瓶颈。
由于质粒在酿酒酵母中存在容易丢失和需要维持筛选压力等缺点,基因组整合特别是高拷贝整合更利于实现代谢工程的工业化应用。基因组高拷贝整合通常需要借助高浓度的抗性标签压力筛选,例如Zhao等通过多轮迭代、逐步提升G418的浓度,在多倍体酿酒酵母中实现了SAM2基因的高拷贝整合,获得了能够合成8.81 g/L S-腺苷-L-甲硫氨酸(S-adenosyl-L-methionine,SAM)的工程酵母[27]。与游离质粒类似,也可以使用部分失活的筛选标签来提高基因组整合拷贝数,例如Lian等使用活性降低的G418标签实现了30个拷贝的荧光蛋白基因的整合[25]。近些年高速发展的成簇规律间隔短回文重复序列及其关联基因(Clustered regularly interspaced short palindromic repeats/ CRISPR-associated,CRISPR/Cas)系统也成功应用于酿酒酵母δ位点高拷贝整合或基因组多位点定点整合,这部分内容将在后文详述。
1.1.4 辅因子平衡辅因子(Co-factor)是酶中非蛋白质的部分,例如NADH、NADPH和辅酶Q等,在酶促反应中主要起着传递电子和某些化学基团的作用,参与胞内许多重要的催化和生化反应。代谢工程中,辅因子不平衡或者供应不足也是限制产物合成的关键因素。所以,酿酒酵母代谢工程中也经常通过增强辅因子平衡和调整辅因子专一性等方法来改善辅因子供应进而提升目标化合物的产量。在经典的无氧发酵过程中,酿酒酵母通过体内的甘油合成途径来消耗胞内过量的NADH从而维持胞内辅因子平衡,但是副产物甘油的合成和积累会造成目标产物得率下降。引入以氧气为电子受体的NADH氧化酶可以降低酿酒酵母胞内NADH/NAD+的比例[28],但不适用于大规模的工业发酵过程。Guadalupe等通过引入NAD+依赖的乙醛脱氢酶(例如来自大肠杆菌的mhpF)将乙酸还原为乙醇,消耗胞内多余的NADH[29]。由于乙酸大量存在于木质纤维的水解物中,在以木质纤维素水解物发酵合成乙醇的发酵中不仅可以消耗培养基中有毒的乙酸,还能进一步增加乙醇的产量。纤维素乙醇发酵过程中,木糖还原酶使用NAD(P)H将木糖还原为木糖醇,而木糖醇脱氢酶偏好NAD+,Bengttsson以及Matsusshika等通过调整辅因子专一性的方式,如将木糖还原酶的辅酶调整至偏好利用NADH或将木糖醇脱氢酶调整为偏好利用NADP+,均可以降低木糖醇的积累,进而提高乙醇的得率[30-31]。
1.1.5 转运子工程转运子是一类可以在膜内外转运小分子的膜蛋白,可以将细胞所需要的底物转移至胞内,也能把部分目标产物转移至胞外。所以合理运用转运子工程可以提高底物消耗速率并降低产物在胞内积累,增加产物的总得率。例如以木糖和纤维二糖作为发酵的碳源时,酿酒酵母自身的转运蛋白对这些碳源的专一性差且转运效率低下,严重影响菌株的生长。Wang等进化了一个木糖特异性转运蛋白,大大提升了木糖的转运效率,使工程菌株具备在葡萄糖和木糖培养基中高效利用木糖的能力[32]。相似地,Lian等进化了纤维糊精转运蛋白2,工程菌株的纤维二糖消耗速率提升了4.0倍,乙醇合成速率提升了4.4倍[33]。Zelle等和Hu等则通过引入转运蛋白将目标产物转运到胞外,来降低产物分泌到胞外所需的能量并解除产物积累带来的生长抑制,最终他们分别将苹果酸和脂肪醇的产量提升了6倍和5倍[34-35]。
1.1.6 调控蛋白改造细胞中的各种中间代谢产物的浓度受到各种代谢调控系统的高度调控,以维持最适生长速度,所以在代谢工程中也需要对调控系统(转录调控和蛋白修饰调控等)进行改造,以突破细胞自身的限制来获得更高的产物得率。翻译后修饰的蛋白质存在抑制或调控结合位点从而受到调控,通过移除这些位点来解除调控以提升蛋白活性是一种很有效的策略。例如甲羟戊酸途径中含有一个跨膜调控区域的3-羟基-3-甲基戊二酰辅酶A还原酶(Hydroxy-3-methyl glutaryl coenzyme A reductase,HMGR) 往往是后续途径的限速步骤[36],通过截短这部分区域使之变成可溶蛋白(Truncated HMG-CoA reductase,tHMGR),可以大幅度提升角鲨烯[36-37]、类胡萝卜素[38]、青蒿素[10]等萜烯类化合物的产量。此外,对转录因子的改造也是代谢工程中的常用策略。例如转录因子UPC2可以对固醇代谢和甲羟戊酸途径进行调控,其点突变体(UPC2-1) 可以提升其在胞内的含量进而大幅度提升甲羟戊酸途径下游产物例如青蒿素前体的产量[10, 39]。全局转录调控工程(Global transcription machinery engineering,gTME) 则通过转录因子的定向进化对细胞代谢网络进行全局调控。Alper等通过对通用转录因子SPT15的定向进化影响了3种RNA酶的转录过程从而实现了全基因组水平的扰动,随后通过富集筛选获得了可以显著提高乙醇耐受性的SPT15突变体[40]。
1.2 酵母基因组靶向改造技术除了通过启动子和拷贝数等对外源基因进行优化之外,还需要对酵母基因组进行靶向改造,以提高宿主细胞代谢网络与外源基因的适配性,进而实现目标产物的高效合成。基于同源重组替换和Cre-loxP的系统是最常用的酿酒酵母基因组靶向改造方法。通过缺陷型标签或者抗性标签可以直接以同源重组的方式在酿酒酵母内实现基因敲除或者插入,但受限于菌株可用标签数目。Cre-loxP系统是来源于噬菌体的重组酶系统,Cre酶通过识别loxP位点介导DNA重组[41-42],可以实现基因组靶向改造后筛选标签的回收利用,使得多位点的多轮迭代改造成为可能。每轮操作后会在改造位点残留一个loxP位点,可能会造成未知的基因组扰动,如基因丢失等[43]。最新发展的酿酒酵母Syn2.0计划,即在合成酿酒酵母染色体时在全基因组范围设计添加了loxP位点,可以很简单地实现全基因组规模重组扰动(Synthetic chromosome rearrangement and modification by loxP-mediated evolution,SCRaMbLE)[44-45]。另外一种来自噬菌体的丝氨酸整合酶(Serine integrase) 也被成功应用于酿酒酵母[46]。相比于Cre-loxP系统,丝氨酸整合酶系统在改造位点不会留下残余的重复序列,属于无痕编辑系统。
转录激活因子样效应物核酸酶(Transcription activator-like effector nucleases,TALENs) 利用模块化、可识别特定碱基的蛋白序列结合至基因组特定位点,然后利用连接的核酸酶实现基因组的剪切和靶向改造[47]。Li等利用模块化组装策略实现了酿酒酵母中URA3、ADE2和LYS2基因的高效敲除[48]。Zhang等进一步发展了基于TALENs的多重编辑技术,利用GAL启动子诱导的TALENs系统多轮迭代诱导整合目标基因,最后筛选获得了葡萄糖消耗速率更快、乙醇得率更高的菌株[49]。
CRISPR/Cas系统是最新一代的基因组编辑工具,具有设计简单、操作便利、效率高等优点,是目前酿酒酵母基因组靶向改造的不二之选[50]。DiCarlo等在2013年首次利用CRISPR/Cas系统在酿酒酵母实现了高效基因组编辑,CAN1和ADE2基因的敲除效率接近100%[51]。得益于CRISPR/Cas9系统的高切割效率和酿酒酵母的高同源重组效率,基因组多位点敲除和整合成为可能。Jakociunas等通过共转化多个gRNA和双链oligo修复模板实现了代谢通路上5个基因的100%同时高效改造[52]。Lian等则实现了多倍体酿酒酵母工业菌株中的基因组多重改造,实现了4个基因在双倍体菌株(8个位点) 以及三倍体菌株(12个位点) 的同时敲除[53]。Randa等利用CRISPR/Cas系统在酿酒酵母中一次整合了3个长度为5.1–6.6 kb的胡萝卜素合成途径基因,3个基因同时整合的效率高达85%[54]。Lian等和Shi等则借助CRISPR/Cas系统实现了代谢途径的多拷贝整合[25, 55],其中Shi等在酿酒酵母基因组整合了多达18拷贝的木糖利用和2, 3-丁二醇(Butanediol,BDO)合成途径(总长达24 kb),从而实现木糖到BDO的高效转化。这些工程应用阐述了CRISPR/ Cas技术在基因敲除和代谢通路整合等基因组定向改造方面的强大能力和广泛的应用前景。
2 系统代谢工程方法传统代谢工程是利用已知的细胞代谢网络背景和重组DNA技术,通过对启动子和终止子强度、基因拷贝数、辅因子等的优化,有效调控酿酒酵母的细胞代谢网络,从而改善细胞性能以实现目标产物高效合成的目的。然而,酿酒酵母细胞代谢网络复杂,大多数基因的功能尚未得到清晰的阐释。因此传统代谢工程通常仅仅是局部调控而无法实现全局调控。系统代谢工程在传统代谢工程基础上,结合系统生物学和进化工程,真正从“系统意义”上对菌株进行改造,加速工业优良菌株的开发[56]。随着测序技术的发展以及包括转录组学、蛋白质组学和代谢组学在内的多种系统代谢分析工具的支持,通过基因组规模的计算机代谢模拟,可对细胞进行多种调节,例如转录调节、翻译调节、反馈抑制以及代谢通量调节,有助于从整体上、多层次、更系统地认识酿酒酵母复杂的遗传背景和代谢网络[57-59]。
2.1 系统代谢分析工具 2.1.1 基因组学分析基因组学分析是以全基因组测序为基础,定量生物体内所有基因并表征其功能的技术[60]。酿酒酵母作为第一个完成全基因组测序的真核生物,一直是基因组学研究的重要模式生物[61]。工业上已经成功利用酿酒酵母生产乙醇,但是为了进一步提高乙醇的产量,需要提高细胞耐高温和耐高浓度乙醇的能力。Caspeta等在培养温度≥40 ℃时,利用适应性进化策略,获得了细胞生长和乙醇产量显著提高的酵母菌株,并通过对整个基因组测序分析发现,ERG3基因突变导致甾醇组成变化,从而提高酵母的耐热性[62]。Hou等通过代谢工程构建了木糖利用酿酒酵母菌株,并在自适应性进化之后获得木糖利用能力提高的酵母菌株。利用全基因组测序对该菌株做进一步分析发现,ASK10M475R突变改善了细胞的生长,并通过增强分子伴侣提高了木糖异构酶的活性,揭示了分子伴侣在木糖异构酶活性调节中的重要性[63]。
2.1.2 转录组学分析转录组学分析是从RNA水平研究细胞中整体基因转录情况及转录调控规律[64]。随着芯片技术及测序技术的发展,各种转录组学研究技术也被应用到酿酒酵母的转录规律和转录调控的分析中,包括微阵列技术(Microarray)[64]、表达序列标签技术(Expressed sequence tags,EST)[65]、基因表达序列分析技术(Serial analysis of gene expression,SAGE)[66]和转录组RNA测序技术(RNA-seq) 等[67]。对具有特殊表型的酿酒酵母菌株和野生型菌株进行转录组学分析,比较并鉴定出转录水平变化的基因,从而为下一步菌株遗传改造提供指导。Wahlbom等通过经典化学诱变筛选得到木糖利用能力提升的酿酒酵母菌株,并利用微阵列技术,将该改良菌株与亲本菌株的全基因组mRNA转录情况进行比较分析。结果显示,多种基因可能参与酿酒酵母细胞木糖的代谢,如编码己糖转运蛋白基因HXT5,编码木酮糖激酶基因XKS,以及编码磷酸戊糖途径相关基因GND1、TAL1和TKL1的表达显著提高[68]。真核细胞特别是酿酒酵母细胞通常是生产药物蛋白的首选细胞工厂,然而由于对其蛋白质分泌机制与调控缺乏了解,在真核细胞中生产重组蛋白存在诸多瓶颈。Huang等利用紫外诱变和微流体液滴分选技术筛分得到几种不同的α-淀粉酶产量提高的酿酒酵母菌株,并利用RNA-seq对突变株进行全基因组转录分析,发现α-淀粉酶的产量提高与酿酒酵母基因表达的整体调节有关,包括31种基因的不同程度的上调和下调,例如ANB1、TIR3和AAC3等在厌氧条件下表达的基因显著上调,参与磷酸盐反应的基因PHO12、PHO84和SPL2显著下调[69]。
2.1.3 蛋白质组学分析蛋白质组学分析是研究细胞的蛋白质组成及其变化规律的技术,能够阐明无法单独使用基因组或转录组分析才能发现的生物系统特性,有效地从蛋白质水平对基因组/转录组分析数据进行补充[70]。酿酒酵母菌株KAY446在高葡萄糖浓度(120–300 g/L)下,仍然具备高葡萄糖利用能力,并进行乙醇的高效发酵。Pham等对KAY446菌株在不同葡萄糖条件下进行蛋白质组学分析,并鉴定出413种表型相关蛋白,其中25种参与氨基酸代谢的蛋白质表达水平降低,12种热休克相关蛋白表达增强。并且在高葡萄糖浓度下,除了乙醇产量提高之外,葡萄糖和许多副产物的同化作用也得到增强。该结果说明在维持正确的细胞氧化或渗透状态的同时,需要抑制其他许多不必要的途径,以提高乙醇的产量[71]。Chen等对脂肪酸积累酿酒酵母菌株进行蛋白质组学分析,鉴定出500多种蛋白质,其中82种蛋白质的表达出现显著变化,包括与糖酵解、乙酸代谢、脂肪酸合成、三羧酸循环、乙醛酸循环、磷酸戊糖途径、呼吸以及运输和应激反应相关蛋白表达的上调,参与甘油、麦角固醇和细胞壁合成相关蛋白表达的下调。这些结果有助于阐明工程菌株中脂肪酸产量增加的机制,进一步提高酿酒酵母中脂肪酸的产量[72]。
2.1.4 代谢组学分析代谢组学分析是利用气质联用、液质联用、核磁共振等多种质谱分析手段,研究某一时刻特定条件下生物体内所有低分子量代谢物的技术,是转录组和蛋白质组的“放大”,是系统生物学的重要组成部分[73-75]。尽管代谢组学分析的使用不如基因组分析、转录组分析和蛋白质组分析普遍,但代谢组学分析有望通过对细胞代谢途径的分析为了解和改造细胞作出重要贡献。Hayakawa等在不同的生产条件下对SAM高产酿酒酵母菌株进行代谢组学分析。结果表明,ATP供应是SAM生产的限速步骤,并且在补充L-蛋氨酸的情况下,SAM高产菌株中腺苷核苷酸的降解产物水平显著高于对照菌株;同时ATP含量分析表明,SAM高产菌株的细胞内ATP水平高于对照菌株,表明高SAM产量归因于增强的ATP供应和更快的ATP消耗速率[76]。Ming等采用代谢组学分析比较了不同浓度乙醇培养的酿酒酵母细胞中的代谢物组成和含量的变化,发现总共有36种代谢物发生了显著变化,包括氨基酸、有机酸和脂肪酸等,其中柠檬酸循环以及丙氨酸、天冬氨酸和谷氨酸代谢途径受到明显的干扰。这些结果表明,代谢组学分析有助于揭示酿酒酵母中乙醇耐受的作用机理[77]。
2.1.5 代谢通量组学分析代谢通量组学是分析细胞内部代谢通量的技术。基于约束的通量平衡分析和基于同位素的通量分析是确定通量的常用方法[78-80]。近年来,酿酒酵母利用木糖进行木质纤维素燃料生产的研究已经取得重大进展。然而,由于对木糖利用机制尚未充分理解,酿酒酵母合成乙醇的效率仍然较低。为了研究可能限制酵母细胞木糖利用的因素,Wasylenko等在有氧和厌氧条件下,分别以葡萄糖和木糖作为碳源,对木糖高效利用酿酒酵母菌株的中央碳代谢物库的大小和代谢通量进行了分析;结果发现,在木糖利用过程中,非氧化戊糖磷酸途径(Pentose phosphate pathway,PPP) 的通量较高,而氧化PPP的通量较低;此外,木糖不能引起酿酒酵母葡萄糖发酵中特征性的全碳分解代谢物阻遏效应;该研究间接表明,木糖发酵的不完全激活会导致糖酵解速率降低,使糖酵解产生的NADH的再氧化效率低下,从而影响酿酒酵母乙醇的合成[81]。
2.1.6 多组学分析如上所述,每种组学分析方法都有其自身的优势与不足,因此,将不同的组学分析方法整合以进行酿酒酵母细胞代谢分析成为未来研究的趋势。多组学分析可以更完整、更系统地掌握细胞的代谢网络。为了改善酿酒酵母生产脂肪醇的工艺,Dahlin等对产脂肪醇酿酒酵母菌株进行了包括转录组学、代谢组学和代谢通量的多组学分析。结果显示,脂肪醇的生产会诱发细胞的细胞壁压力反应,同时也发现脂肪醇的合成会对酵母细胞的代谢产生影响,推测充足的NADPH可能是脂肪醇高效合成的关键因素,即有必要在酿酒酵母细胞中去除乙醇合成途径和增强NADPH的再生能力。通过多组学分析得到的这些信息可以为进一步改善菌株脂肪醇合成能力提供指导[82]。
2.2 基因组代谢模型指导的理性代谢工程基因组规模的代谢模型基于全基因组测序和基因组注释,通过将模型预测与实验数据进行比较,系统地改善我们当前对微生物代谢网络的认识。自1999年Palsson课题组构建的第一个微生物基因组代谢模型后,已成功建立其他多种微生物的基因组代谢模型,包括大肠杆菌、酿酒酵母和幽门螺杆菌等[83-86]。酿酒酵母作为模式真核生物,已经发布了多种酵母基因组规模代谢组模型,能够帮助我们更系统地认识酿酒酵母复杂的代谢网络系统。Nielsen和Palsson课题组基于基因组708个结构开放阅读框和相对应的1 035个代谢反应,首次建立了酿酒酵母全基因组模型iFF708。该模型形成了一个由1 175个代谢反应和584种代谢物组成的代谢网络,可应用于分析不同遗传背景和生理条件下的表型行为[87]。之后,Dobson等通过结合文献中的新反应,重建了酵母基因组代谢网络,开发了Yeast 4.0模型。该模型包括1 102个独特的代谢反应,涉及924个独特的代谢产物,比此前建立的代谢网络要大得多,并且允许执行基于约束的分析[88]。随着测序技术和组学分析技术的飞速发展,酿酒酵母代谢模型也在不断发展和完善。最近建立的Yeast 8.0模型提高了基因组覆盖率,进一步扩大了酵母代谢网络范围,并通过使用GECKO框架考虑了酶限制,改善了基因组代谢模型对细胞表型的预测能力。此外,在Yeast 8.0的基础上通过公共数据库收集和评估酵母代谢蛋白的3D结构开发了proYeast8DB模型,该模型能鉴定与特定表型相关的突变热点[89]。为了将维持氧化还原平衡的甘油代谢流重新定向到乙醇的生产,Bro等利用酿酒酵母全基因组模型iFF708,对氧化还原代谢改造的不同策略进行评分,其最佳策略(表达非磷酸化且NADP+依赖性的甘油醛-3-磷酸脱氢酶) 使得在厌氧发酵条件下,甘油的产率下降了40%,乙醇的产率提高了3%[90]。类似地,为了建立酿酒酵母大规模生产倍半萜平台,Asadollahi等使用iFF708模型和OptGene算法[91]来鉴定新的代谢工程靶点,以增强酿酒酵母中倍半萜的生物合成。该模型评估了基因缺失对酿酒酵母代谢模型中通量分布的影响,并确定GDH1基因(编码NADPH依赖性谷氨酸脱氢酶)的缺失是提高酵母倍半萜生物合成水平的最佳基因靶点。虽然GDH1基因的删除使得倍半萜产量增加了85%,但是细胞生长也受到了严重影响。鉴于谷氨酸脱氢酶活性对氨代谢的重要性,在GDH1缺失的工程细胞中过表达GDH2 (编码NADH依赖性谷氨酸脱氢酶),能够在提高倍半萜合成水平的同时维持细胞的正常生长速率[92]。
3 合成生物学驱动代谢工程方法合成生物学以工程学理论为指导,设计构建模块化、标准化的生物元件,并组装成具有新功能的人工细胞[93]。近年来,由合成生物学驱动的酿酒酵母代谢工程也受到了广泛的关注和研究,包括组合代谢工程(如组合途径优化和组合基因组工程)、途径共定位、动态调控代谢工程以及全基因组进化等。相比于传统代谢过程,由合成生物学驱动的代谢工程将标准化和模块化的元件组装成完整的代谢途径和网络,具有可预测性、可重复性和可靠性等优点[56]。
3.1 组合途径优化传统的代谢工程先从酿酒酵母的代谢途径出发,寻找影响整个代谢途径的限速步骤,再进行遗传改造以优化整个代谢途径。但往往一个限制性因素刚被解决,由此引发的代谢通量不平衡可能会带来新的代谢瓶颈。因此,能同时优化多个基因的组合途径优化策略可实现代谢网络的全局平衡与优化。Du等在酿酒酵母中开发了基于组合转录工程的代谢途径定制优化(Customized optimization of metabolic pathways by combinatorial transcriptional engineering,COMPACTER) 方法,即通过创建一系列不同强度的启动子突变体,并将得到的启动子突变体用于同时调控目标途径中多个基因表达水平,通过DNA组装产生具有多种表达水平的途径突变文库,进而实现组合途径优化(图 2)。为避免在组装过程中由重复序列引起的错误重组,途径中每个基因均单独设计了一对独特的启动子和终止子。利用COMPACTER方法,构建了由11个ENO2启动子突变体控制的纤维二糖转运蛋白基因和10个PDC1启动子突变体控制的β-葡萄糖苷酶基因组成的纤维二糖利用途径突变文库,基于菌落大小的高通量筛选方法,最优突变菌株的纤维二糖消耗速率提高了5.4倍,乙醇生产率提高了5.3倍[94]。类似地,Song等对11个不同的启动子进行了10种组合,以调控融合基因BTS1- ERG20、GGPPSsa和tHMGR的表达,并优化酿酒酵母中香叶基香叶醇合成途径,结合发酵条件优化,香叶基香叶醇的产量可达到1 315.44 mg/L[95]。
随着分子生物学和酿酒酵母代谢网络模型的发展,越来越多的异源途径成功在酿酒酵母细胞中进行组装。然而,很多异源途径需要多个酶共同催化,这可能会给细胞造成负担,从而影响整个代谢途径的合成效率。因此,一些代谢途径酶共定位的策略被应用于代谢途径的优化,包括蛋白融合表达、蛋白支架组装和亚细胞结构定位等(图 3)。这些策略可以对代谢途径中的关键酶进行空间分布的优化,从而提高底物和酶的相互作用效率,降低中间体向竞争途径的代谢流。

|
| 图 3 代谢途径酶共定位 Fig. 3 Co-localization of metabolic pathway enzymes. (A) Two or more proteins were connected by a flexible linker for fusion expression. (B) Protein scaffolds are used for the co-localization of two or more proteins. (C) Compartmentalization of multiple enzymes of the metabolic pathway into the same subcellular organelle. Enz: Enzyme. |
| |
蛋白融合表达策略利用柔性接头(Linker) 将两个或多个蛋白连接融合表达,产生一个多功能的融合蛋白(图 3A)[96]。融合蛋白的不同结构域互相独立,而在空间上相互靠近,既能使蛋白之间的折叠表达不互相干扰,又能提高底物和酶的相互作用效率[97]。因此,蛋白融合表达策略已被广泛应用于代谢过程的优化。为了提高酿酒酵母合成白藜芦醇的产量,Zhang等将白藜芦醇合成途径中的两个酶4-香豆酸CoA连接酶(4-coumarate coenzyme A ligase,4CL) 和二苯乙烯合酶(Stilbene synthase,STS) 进行融合表达。相比单独表达这两个酶,融合表达策略使白藜芦醇的产量提高15倍,说明4CL和STS共定位有助于提高白藜芦醇合成途径的效率[97]。Ma等将催化生成法尼焦磷酸(Farnesyl pyrophosphate,FPP) 酶(FPP合酶) 和以FPP作为底物的酶(广藿香醇合酶) 进行融合表达,以提高广藿香醇前体FPP的利用率,同时通过增加前体的供应、抑制竞争途径以及优化发酵工艺,广藿香醇的产量达到(466.8±0.5) mg/L,是之前报道最高产量的7.8倍[98]。
3.2.2 蛋白支架组装尽管蛋白融合表达是进行多酶组装的一种简单而有效的策略,但在实际应用中也存在着很多限制,尤其是多种蛋白融合表达可能会出现蛋白的错误折叠而影响蛋白活性和催化效率。蛋白支架组装是将代谢途径相关酶通过蛋白质-蛋白质相互作用固定在蛋白支架上,是实现多酶共定位的有效策略(图 3B)。进一步通过调整蛋白支架的受体结构域还可以调节途径酶的顺序和比例,从而优化目标途径的代谢效率。Wang等通过蛋白支架组装策略募集白藜芦醇生物合成途径的相关酶4-香豆酸CoA连接酶(4CL) 和二苯乙烯合酶(STS),以改善酿酒酵母细胞中白藜芦醇的生产。与没有蛋白支架的对照相比,白藜芦醇的产量提高了5.0倍,比4CL-STS融合表达提高了2.7倍[99]。Tippmann等利用抗体构建了蛋白支架,并在酿酒酵母中首次实现法呢基焦磷酸合酶和法呢烯合酶的共定位。通过不同参数的优化,法呢烯的产率提高了135%[100]。除此之外,Li等在酿酒酵母中设计了标准化的组装方案,并开发了一套强大的基于Golden-Gate的合成生物学工具包,用于人工支架蛋白的构建[101]。
3.2.3 亚细胞结构定位酿酒酵母细胞中存在很多亚细胞器,其pH值、氧浓度、氧化还原电位和辅因子等都可能与细胞质存在差异,这些差异可能更适合某些酶促反应的进行。并且将代谢物整合到这些亚细胞器中,可以减少中间代谢产物对细胞的可能毒害作用,增加底物和酶的局部浓度,以及抑制竞争代谢流等(图 3C)。因此利用亚细胞结构定位策略以实现代谢途径相关酶的共定位是提高途径代谢效率的更为复杂且有效的途径共定位法,其中线粒体、内质网、过氧化物酶体和液泡是酿酒酵母中被选择定位的几个亚细胞器[102-106]。Farhi等将异源FPP合酶和倍半萜合酶(如朱栾倍半萜合酶或紫穗槐二烯合酶) 共定位于线粒体中,使朱栾倍半萜和紫穗槐二烯的产量分别提高了8倍和20倍[107]。Avalos等将完整的异丁醇合成途径定位在线粒体内,使酿酒酵母合成异丁醇的产量提高了260%[108]。类似地,Zhou等将脂肪酸衍生物(脂肪醇、烷烃和烯烃) 的生物合成途径定位到过氧化物酶体中,使脂肪酸衍生物的产量提高了700%[109]。
3.2.4 人工合成亚细胞器共定位通过人工合成亚细胞器共定位是在酿酒酵母中实现代谢途径共定位的一种新方法。人工合成亚细胞器是一系列被包裹在远离细胞质的酶簇,类似于细胞器,但不是细胞的天然结构。除了具有天然亚细胞器的优点如体积小、与细胞质分离等之外,人工合成亚细胞器还可以避免天然亚细胞器存在的竞争或干扰。由60个或180个相同的亚基(20–24 nm或30–32 nm) 通过自组装而构建的胶囊蛋白被应用于酿酒酵母中的人工合成亚细胞器,并成功实现了分段的mVenus的共定位[110]。在酵母中构建人工合成亚细胞器的另一种方法是通过光诱导酶聚类技术。光诱导的光簇和光抑制的PixELL系统产生刚性酶簇,该酶簇使得两种或多种酶紧密接近。利用这两种系统,成功将脱氧紫胶素途径中一个分支点的通量引导至脱氧紫胶素。这些光诱导的合成细胞器可以迅速形成和解离,表明将来有望用作生产各种产品的动态合成细胞器[111-112]。
3.3 动态调控工程在自然界中,微生物本身具有监测外界变化的能力,从而紧密控制自身基因的表达水平,以适应环境的变化。基于酵母细胞代谢物或者外界输入信号如温度、光照等的人工动态调控系统被广泛应用于代谢工程[113]。丙二酰辅酶A是脂肪酸生物合成的关键中间体,也是很多工业化合物如聚酮化合物的前体。David等通过分层动态途径调控实现了酿酒酵母中丙二酰辅酶A和脂肪酸的通量控制,成功提高了异源3-羟基丙酸(3-hydroxypropionic acid,3-HP) 的产量。首先在酿酒酵母表达来源于枯草芽孢杆菌的丙二酰辅酶A转录响应因子FapR,并在组成型启动子TEF1中引入FapR结合位点(34 bp操纵子fapO),在酿酒酵母中成功开发了丙二酰辅酶A生物传感器。另一方面,通过葡萄糖响应的HXT1启动子控制3-HP竞争途径即脂肪酸合成途径中的FAS1基因的表达,在葡萄糖充足的培养前期,FAS1表达以合成脂肪酸进行细胞生长;在细胞培养后期,HXT1启动子受葡萄糖限制,脂肪酸生物合成途径下调,丙二酰辅酶A积累,从而激活由启动子TEF1 (包含FapR结合位点) 驱动的CaMCR基因的表达,以进行3-HP的合成。该动态调控策略将细胞生长和产物合成两阶段分开,成功将3-HP的产量提高了10倍[114]。类似地,Zhou等基于GAL调控系统,通过敲除GAL80基因以及定向进化产生温度敏感Gal4突变体GalM9,成功构建了温度响应的动态调控系统,通过温度变化使细胞生长和产物积累分段进行。通过温度响应的动态调控,酿酒酵母细胞的生物量提高了44%,番茄红素的产量提高了144%[113]。
3.4 组合基因组工程利用酿酒酵母进行目标产物的合成经常需要对细胞代谢重新布局以提高产物产量,这就需要对不同的靶标进行不同程度的表达调控,如提高编码限速酶基因的表达,降低或者删除竞争途径相关基因的表达等。同时,由于酿酒酵母细胞代谢调控网络复杂,需要利用组合基因组工程对多个,特别是有协同作用的靶标基因进行组合优化,以有效提高酵母细胞中目标途径的代谢效率。Lian等开发了CRISPR-AID三功能正交体系,利用SaCas9 (Cas9 from Staphylococcus aureus)、核酸酶失活的dLbCpf1 (Nuclease-deficient Cpf1 from Lachnospiraceae bacterium ND2006) (融合激活结构域)和dSpCas9 (Nuclease-deficient Cas9 from Streptococcus pyogenes) (融合抑制结构域) 同时实现基因敲除、转录激活和转录抑制,并将CRISPR-AID应用于酿酒酵母表面展示表达重组蛋白内切葡聚糖酶Ⅱ(EGⅡ) 的组合优化。首先选择基因组上14个基因靶点进行转录激活、17个靶点进行转录抑制以及5个靶点进行基因敲除,并生成了(15×18×6) 种组合突变文库。通过富集高荧光细胞,获得了EGⅡ表达水平显著提升的细胞,结合DNA测序,发现PDI1上调、MNN9下调和PMR1缺失的组合最大程度地提高了EGⅡ的展示水平[115]。需要特别指出的是,PDI1上调和MNN9下调存在显著的协同效应,该组合无法通过传统的代谢工程方法获得,凸显组合基因组工程在代谢工程应用的重要性。
3.5 可追踪的全基因组进化技术近年来,随着CRISPR技术、寡核苷酸芯片技术以及二代测序技术的迅速发展,除了能够在酿酒酵母实现高效精准的基因组编辑外,还可以基于酿酒酵母基因组文库创建大规模的突变文库,并确定基因型和表型的对应关系,以实现对酿酒酵母细胞的全基因组进化。目前已经开发的酿酒酵母全基因组进化方法包括寡核苷酸整合技术(如YOGE (Yeast oligo-mediated genome engineering)[116]和eMAGE (Eukaryotic multiplex automated genome engineering)[117])、基于RNAi的基因组进化方法(如RAGE (RNAi-assisted genome evolution)[118]) 以及基于CRISPR的全基因组方法(如CHAnGE (CRISPR/Cas9- and homology-directed-repair-assisted genome-scale engineering)[119]和MAGIC (Multi-functional genome-wide CRISPR system)[120])。
酿酒酵母强大的同源重组机制使其具备高效的基因编辑能力,但是其编辑效率对同源片段的长度有很大的依赖性,即同源臂长度的降低,会显著影响其编辑效率。因此,研究者希望在酿酒酵母中利用较短的单链寡核苷酸片段就能实现高效的基因编辑。Church课题组成功开发了YOGE (酵母寡核苷酸介导的基因组工程) 技术,由单链寡核苷酸介导重组以在酿酒酵母进行基因组编辑。在对基因MLH1和MSH2 (错配修复相关基因) 敲除,Rad51p (K342E) 和Rad54p蛋白(过表达DNA重组酶) 过表达,以及其他参数优化后,利用YOGE技术成功对3种不同的酿酒酵母细胞进行了基因组修饰。然而,YOGE的等位基因替换频率仍旧较低,只达到0.2%–2%[116]。与YOGE相比,Isaacs教授开发的eMAGE是一种更高效、更精确的寡核苷酸整合技术。eMAGE技术基于在DNA复制过程中由ssDNA退火蛋白介导在滞后链上退火合成寡核苷酸的原理。同时,他们通过添加羟基脲减慢复制叉速度以及优化寡核苷酸浓度、寡核苷酸长度等参数,同时实现了60个靶点的突变[117]。然而,eMAGE技术需要依赖URA3标记的选择和靶标序列与复制起始点的紧密接近,在酵母基因组的应用上有一定的局限性。
RAGE是由Si等开发的一种基于RNA干扰(RNAi)的全基因组进化技术。通过引入来源于芽殖酵母的Ago1p和Dcr1p蛋白在酿酒酵母重构RNAi途径,并根据基因组基因构建了双链RNA文库,以对酿酒酵母细胞实现全基因组进化。利用RAGE技术,对PTC6、YPRP84W和tRNAVal (AAC) 3个基因表达水平的下调,显著提高了酿酒酵母细胞对乙酸的耐受性[118]。随后,基于RAGE技术,Si等又开发了通过全长cDNA表达实现基因过表达、全长反义RNA实现基因表达水平下调的酿酒酵母自动化多重基因组进化技术。利用该技术,在一个月内经过多轮迭代筛选,酿酒酵母菌株对乙酸的耐受性显著提高,在1.1%乙酸浓度下可以实现葡萄糖到乙醇的高效转化[121]。
CHAnGE是一种基于CRISPR/Cas9和同源定性修复的、可追踪的酿酒酵母全基因组进化技术。针对酿酒酵母6 000多个开放阅读框构建了包括guide RNA和同源重组模板的CHAnGE表达框。同时,CHAnGE表达框含有独特的DNA条形码,可以追踪基因组的突变基因。将CHAnGE表达框在芯片上合成并构建到质粒载体上,转入酿酒酵母细胞中即可实现酵母全基因组进化。利用CHAnGE技术,经过在5 mmol/L糠醛和10 mmol/L糠醛浓度条件下的两轮迭代进化,SIZ1和LCB3基因的敲除能显著提供糠醛的耐受性。类似地,利用CHAnGE技术也使酿酒酵母的乙酸耐受性提高了20倍[119]。
Lian等在多功能CRISPR-AID体系的基础上开发了多功能全基因组进化技术技术(MAGIC)。MAGIC技术根据酵母基因组构建包括转录激活、转录抑制和基因敲除的不同gRNA的最全面的基因组文库,同时可以利用gRNA作为独特的条形码进行追踪。在MAGIC全基因组进化技术,经过3轮迭代,发现SIZ1基因表达水平下调、NAT1基因表达水平上调以及PDR1基因表达水平下调可以使酿酒酵母细胞在17.5 mmol/L糠醛浓度下利用大部分葡萄糖生产乙醇,显著提高了糠醛的耐受性[120]。
4 代谢工程技术应用实例 4.1 酿酒酵母中从头合成青蒿素前体青蒿酸疟疾是由疟原虫引起的严重疾病,每年造成超过100万人死亡。屠呦呦等[10]从中药古方中发现了青蒿素作为治疗疟疾的特效药,然而青蒿素是一种需要从菊科植物中提取的倍半萜内酯过氧化物,获得方式困难,价格高昂,大规模应用挑战较大。2006年Ro等利用各种代谢工程技术实现了青蒿素前体青蒿酸的高效从头合成(图 4)[10],青蒿酸可以后续经由化学反应转化为青蒿素,因此奠定了青蒿素大规模商业应用的基础。青蒿酸细胞工厂构建过程中使用了各种代谢工程技术。首先,为了构建法尼焦磷酸(FPP,青蒿酸前体)高产菌株,他们截短表达了可溶形式的3-羟基-3-甲基戊二酰辅酶A还原酶(tHMGR)基因,降低了旁路角鲨烯途径ERG9的启动子强度,使用了调控蛋白Upc2p的点突变体UPC2-1,过表达了FPP合酶基因ERG20,并外源表达了来自黄花蒿的紫穗槐-4, 11-二烯合酶(Amorpha-4, 11-diene Synthase,ADS)、细胞色素P450 (CYP71AV1) 及其电子受体CPR (细胞色素P450还原酶)。FPP经由ADS的催化生成紫穗槐二烯,而后经过P450的三步催化合成青蒿酸。同时,为了确保宿主菌株的遗传稳定性,这些改造都是通过基因组整合进行的。重组菌株的青蒿酸摇瓶发酵产量为115 mg/L,而后通过发酵优化进一步提高到2.5 g/L。Paddon等通过进一步的基因挖掘找到了另外3个关键基因(细胞色素b5基因CYB5、青蒿醇脱氢酶基因ADH1、青蒿醛脱氢酶基因ALDH1)。在合成青蒿二烯酿酒酵母菌株中表达这3个基因,并优化CYP71AV1辅助还原酶CPR1的表达量和发酵过程,使得青蒿酸产量提高到25 g/L[122]。
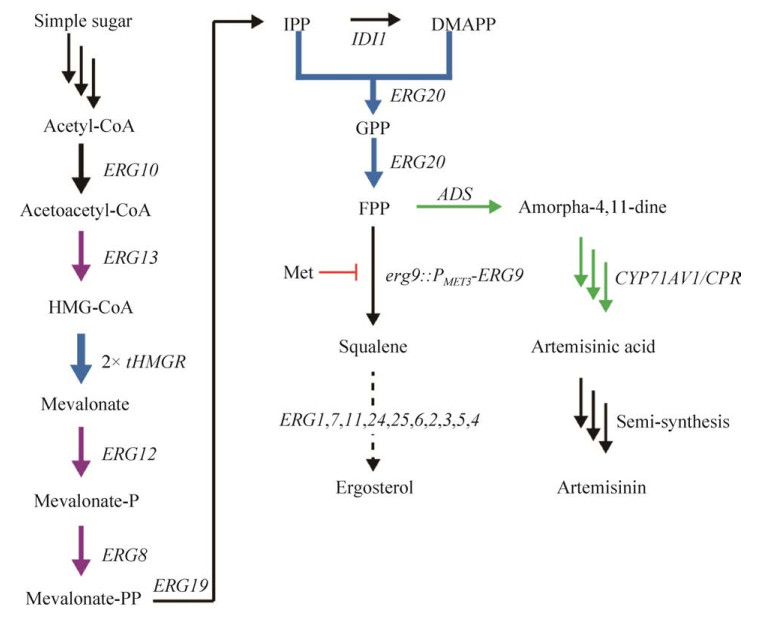
|
| 图 4 酿酒酵母中从头合成青蒿素前体青蒿酸[10] Fig. 4 De novo biosynthesis of the artemisinin precursor artemisinic acid in S. cerevisiae[10]. Overexpressed genes are shown in blue, genes up-regulated by editing the regulatory protein UPC2-1 are shown in purple, ERG9 down-regulated by replacing the endogenous promoter with the MET3 promoter is shown in red, and the foreign genes, including ADS (amorphadiene synthase), cytochrome P450 CYP71AV1 and the electron receptor CPR from Artemisia annua are shown in green. HMG-CoA: 3-hydroxy-3-methylglutaryl-CoA; IPP: isopentenyl pyrophosphate; DMAPP: dimethylallyl pyrophosphate; GPP: geranyl pyrophosphate; FPP, farnesyl pyrophosphate. |
| |
微生物脂肪酸的生物合成因其在产生油性化学品和生物燃料中的潜在用途而备受关注。其中,游离脂肪酸(Free fatty acid,FFA)是制造洗涤剂、润滑剂、化妆品和药物成分的理想原料。因此,利用微生物合成FFA的产量需要进一步提高以实现工业规模的生产。Nielsen课题组模拟含油酵母的代谢,对酿酒酵母进行了代谢重排,以实现将传统的乙醇发酵定向为高效的脂肪酸代谢(图 5)。首先,选择FAA产量为7 g/L的菌株YJZ45建立了FAA生产的有效途径。鉴于丙酮酸转化为乙酰辅酶A过程可能是FAA生产的限速步骤,并且乙酰辅酶A无法穿过线粒体膜,需要柠檬酸穿梭将乙酰辅酶A运送到细胞质,因此利用强启动子TEF1表达PCY1和ACC1以确保柠檬酸穿梭需要的草酰乙酸的有效合成和提高丙二酰辅酶A的供应,使FFA的产量提高14%。随后,又通过增强线粒体丙酮酸载体基因MPC1和MPC3的表达以增强线粒体丙酮酸的积累,过表达异源柠檬酸合酶基因以增强线粒体柠檬酸的合成,过表达反转运蛋白基因YHM2、异柠檬酸脱氢酶基因IDP2和柠檬酸裂解酶基因AnACL以增强柠檬酸向细胞质的运输和乙酰辅酶A的合成,使摇瓶中FAA产量达到1 166 mg/L。其次,通过上调磷酸戊糖(PPP) 途径来满足NADPH需求,下调三羧酸循环来微调ATP的供应,以确保途径中间产物的平衡,结合补料分批发酵,使FAA的产量达到33.6 g/L。最后,通过删除乙醇合成关键基因PDC1/PDC5/ PDC6,希望将乙醇合成代谢流定向到脂肪酸合成。但是PDC基因的敲除会显著影响细胞生长,因此采用实验室适应性进化(Adaptive laboratory evolution,ALE),恢复细胞在葡萄糖下的生长,FAA的产量可达25 g/L。对ALE实验中分离的3个单克隆细胞进行全基因组测序,鉴定出30个突变位点,其中在3个单克隆中都发现了丙酮酸激酶Pyk1p的突变,并且在进化过程中Pyk1p几乎没有活性,从而确保了糖酵解通量与FFA合成以及呼吸通量之间的平衡。从乙醇发酵到脂肪生成的酵母代谢重排实验说明酿酒酵母细胞的代谢具有可塑性,并且通过系统代谢工程和适应性实验室进化的结合,有可能完全重新编程酵母细胞代谢网络的功能[123]。

|
| 图 5 通过代谢流重排和实验室适应性进化改造得到的产油酿酒酵母[123] Fig. 5 Oil-producing yeast generated by metabolic engineering and adaptive laboratory evolution[123]. The main carbon metabolism flow is shown in red, arrows indicate the carbon metabolism flow for cell growth, the energy supply pathway is in green, and the central metabolites are shown in yellow. In order to transform S. cerevisiae from ethanol fermentation to oil production, ATP citrate lyase (ACL) was introduced to synthesize acetyl-CoA in the cytoplasm, and then glycolysis, pentose phosphate pathway (PPP), and tricarboxylic acid cycle (TCA) were finely tuned to provide three substrates (NADPH, ATP, and acetyl-CoA) for the synthesis of FFA. Finally, the ethanol fermentation pathway was knocked out and adaptive evolution was carried out to completely reprogram S. cerevisiae into an oil-producing yeast. |
| |
可待因、氢可酮和吗啡是来自罂粟的一类重要的、具有止疼作用的阿片类药物。然而,由于罂粟种植环境的不稳定性和多变性,以及市场上的巨大需求,阿片类药物的供应面临巨大的风险。在这种情况下,Galanie等对单倍体酿酒酵母CEN.PK2进行了工程改造,以实现阿片类化合物的完全从头生物合成(图 6)[124]。首先,他们在酵母细胞中构建了阿片类化合物重要中间体(S)-牛心果碱的生物合成途径。包括17种酶的(S)-牛心果碱合成途径被分为4个基因调控模块。模块Ⅰ过表达酵母内源的酪氨酸途径相关基因,以提高L-酪氨酸和4-羟苯基乙醛(4-hydroxyphenylacetaldehyde,4-HPAA)的积累;模块Ⅱ旨在合成并回收哺乳动物氧化还原辅助因子四氢生物蝶呤(Tetrahydrobiopterin,BH4);模块Ⅲ过表达不同来源的从L-酪氨酸合成(S)-去甲乌药碱途径的相关基因,以合成(S)-去甲乌药碱;模块Ⅳ过表达了去甲乌药碱6-O-甲基转移酶(Norcoclaurine 6-O-methyltransferase,6OMT)、乌药碱-N-甲基转移酶(Coclaurine N-methyltransferase,CNMT)、4′-O-甲基转移酶(3′-hydroxy-N- methylcoclaurine 4′-O-methyltransferase,4′OMT)、罂粟来源的细胞色素P450还原酶和来自花菱草的N-甲基乌药碱羟化酶(N-methylcoclaurine hydroxylase,NMCH),实现了罂粟(S)-牛心果碱的合成。同时,通过敲除ZWF1基因和过表达TyrHWR (酪氨酸羟化酶的突变体)、4′OMT和NCS ((S)-norcoclaurine Synthase,(S)-去甲乌药碱合酶),增加了通向(S)-牛心果碱的代谢流。随后,从罂粟中鉴定出1, 2-脱氢牛心果碱合酶和1, 2-脱氢牛心果碱还原酶(1, 2-dehydroreticuline synthase-1, 2- dehydroreticuline reductase,DRS-DRR酶),可以高效地进行(S)-牛心果碱到(R)-牛心果碱的转化。然后,对多花罂粟碱合酶SalSyn (Salutaridine synthase)进行蛋白质改造,使(R)-牛心果碱的产量提高了6倍。最后,通过表达多花罂粟碱还原酶基因PbSalR和salutaridinol-7-O-乙酰基转移酶基因PsSalAT,成功在酿酒酵母中实现了蒂巴因的从头合成,合成水平达到(6.4±0.3) mg/L。蒂巴因在酵母中的生物合成途径由21个异源基因和2个天然基因组成,该途径的成功创建激发了通过合成生物学方法驱动的代谢工程在酵母中合成更高价值的化合物。
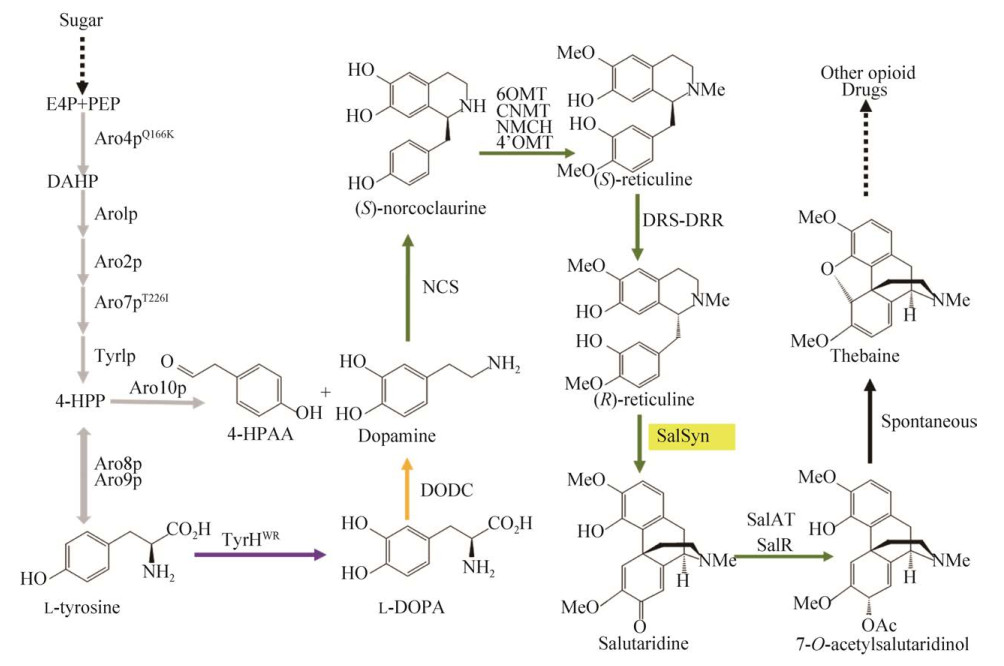
|
| 图 6 酿酒酵母中蒂巴因的从头生物合成途径[124] Fig. 6 The complete biosynthetic pathway of thebaine from sugar in S. cerevisiae[124]. E4P: erythrose 4-phosphate; PEP: phosphoenolpyruvate; DAHP: 3-deoxy-d-arabino-2-heptulosonic acid 7-phosphate; 4-HPP: 4-hydroxyphenylpyruvate; 4-HPAA: 4-hydroxyphenylacetaldehyde; Aro4pQ166K: DAHP synthase; Aro1p: pentafunctional arom enzyme; Aro2p: bifunctional chorismate synthase and flavin reductase; Aro7pT226Ⅰ: chorismate mutase; Tyr1p: prephenate dehydrogenase; Aro8p, aromatic aminotransferase Ⅰ; Aro9p: aromatic aminotransferase Ⅱ; Aro10p: phenylpyruvate decarboxylase; TyrHWR: feedback inhibition-resistant tyrosine hydroxylase (mutations R37E, R38E, W166Y); DODC: l-DOPA decarboxylase; NCS: (S)-norcoclaurine synthase; 6OMT: norcoclaurine 6-O-methyltransferase; CNMT: coclaurine N-methyltransferase; NMCH: N-methylcoclaurine hydroxylase; 4'OMT: 3'-hydroxy-N-methylcoclaurine 4'-O-methyltransferase; DRS-DRR: 1, 2-dehydroreticuline synthase-1, 2-dehydroreticuline reductase; SalSyn: salutaridine synthase; SalR: salutaridine reductase; SalAT: salutaridinol 7-O-acetyltransferase. |
| |
β-法尼烯是一种具有多种工业应用的植物倍半萜。由于酿酒酵母中类异戊二烯合成途径效率低,限制了β-法尼烯在酿酒酵母中的高效合成,无法满足工业需求。通过系统生物学和合成生物学方法结合,Adam等利用4个非天然代谢反应重排酿酒酵母的中心碳代谢,能够以较低的ATP需求合成异戊二烯前体——乙酰辅酶A,从而减少碳向二氧化碳排放的损失反应;同时通过改善途径的氧化还原平衡,成功构建能够在细胞质中高产乙酰辅酶A的酿酒酵母平台细胞工厂,可用于不同的乙酰辅酶A衍生物的高效合成(图 7)[125]。首先,将与乙酰辅酶A、氧化还原和糖异化相关的异源反应化学计量添加到天然酿酒酵母基因组规模模型中,设计了改良的法尼烯生物合成途径,并重点研究了木酮糖-5-磷酸特异性磷酸酮醇酶(D-xylulose-5-phosphate-using phosphoketolase,xPK)、磷酸转乙酰基酶(Phosphotransacetylase,PTA)、酰化乙醛脱氢酶(Aldehyde dehydrogenase acylating,ADA) 和NADH依赖的HMG-CoA还原酶(NADH-HMGr)对法尼烯合成效率的影响。基于此,利用acs2ΔACS1菌株(由于ACS1受葡萄糖抑制,该菌株能在乙醇条件下生长而不能在葡萄糖条件下生长) 的生长互补性,鉴定了功能性ADA和PK-PTA,并测试其合成胞质乙酰辅酶A的能力。同时鉴定了来自海洋菌的对NADH高度特异性的NADH-HMGr。最后,以法尼烯生产菌株AMR4作为出发菌株,通过消除其胞质乙酰辅酶A的天然合成途径,敲除RHR2基因,并过表达ADA、PK和NADH-HMGr,成功将β-法尼烯的产量提高到130 g/L,并使所需的氧气减少了75%,为法尼烯的工业化生产奠定了基础。

|
| 图 7 酿酒酵母中乙酰辅酶A途径改造实现法尼烯的高效生物合成[125] Fig. 7 Acetyl-CoA pool engineering for highly efficient biosynthesis of farnesene in S. cerevisiae[125]. Blue arrows represent native enzymes, and green arrows represent the heterologous enzymes for the biosynthesis of farnesene. G6P: glucose-6-phosphate; F6P: fructose-6-phosphate; FBP: fructose-1, 6-bisphosphate; GAP: glyceraldehyde 3-phosphate; E4P: erythrose 4-phosphate; S7P: sedoheptulose 7-phosphate; R5P: ribose-5-phosphate; X5P: xylulose-5-phosphate; RL5P: ribulose-5-phosphate; Acetyl P: acetyl phosphate; HMG-CoA: 3-hydroxy-3-methylglutaryl-CoA; xPK: X5P-using phosphoketolase; PTA: phosphotransacetylase; ADA: aldehyde dehydrogenase acylating; NADH-HMGr, NADH-specific HMG CoA reductase. |
| |
随着能源短缺和全球变暖等环境问题日益严重,工业生物技术特别是微生物细胞工厂成为了研究热点,而代谢工程迅速推动了工业生物技术的发展。经典代谢工程探索并表征了酿酒酵母中的一些表达元件和代谢调控机制,发展了稳定可靠的基因组操作方法,成功实现了一系列高附加值化合物在酿酒酵母中的从头合成。然而由于酿酒酵母细胞复杂的遗传背景和代谢网络,传统代谢工程只能实现局部调控,往往达不到预期的效果。而系统代谢工程在传统代谢工程基础上,结合系统生物学和进化工程,能够实现对细胞的全局调控。同时,随着各种计算工具和测序技术的发展,可以对酿酒酵母细胞进行基因组/转录组分析、蛋白质组分析和代谢组分析,还可以通过基因组规模的计算机代谢模拟,帮助我们更深入地了解酿酒酵母复杂的遗传背景和代谢网络。此外,由合成生物学驱动的代谢工程方法,包括组合途径优化、途径共定位、组合基因组工程等可以有效优化代谢途径,提高目标途径的代谢效率。近年来,利用CRISPR技术进行基因编辑成为研究热潮,由之开发的各种酿酒酵母全基因组进化技术,可以对酿酒酵母基因组实现精确有效的表达调控,使得代谢工程改造的深度和广度都得到了极大的提升。
高通量筛选是合成生物学驱动的代谢工程研究的核心技术。除了常规的利用产物的颜色和荧光建立高通量筛选方法之外,利用微流体液滴系统和机器人平台已成为筛选目标表型的有效手段[126-127]。基质辅助激光解吸/电离飞行时间质谱(Matrix- assisted laser desorption/ionization time-of-flight mass spectrometry,MALDI-ToF)也已成功应用于包含多步酶促反应的微生物菌落的高通量分析,提供了简单有效的高通量筛选方法[128]。此外Li等使用结构和数据驱动的方法,即将用于蛋白质重新设计的软件程序Rosetta与可以计算氨基酸进化信息的GREMLIN结合,从而改变植物P450的底物特异性并提高其活性。Rosetta和GREMLIN的组合可有效减少突变文库的大小并避免无用的筛选工作。这种由结构和数据驱动的方法,包括计算蛋白设计、进化信息以及实验数据驱动的优化,为代谢工程提供了新的方向[129]。近年发现的一些新型的表达元件和调控元件,光遗传学调控系统[130]和磁感应蛋白系统[131]等也都可能帮助发展应用于酿酒酵母的新型传感或调控系统。
总之,在过去30年,代谢工程伴随着合成生物学以及系统生物学工具的快速发展,对酿酒酵母细胞的遗传背景和代谢网络有着越来越清晰的认识,利用酿酒酵母进行更多大宗化学品以及天然代谢产物的生产已成为新的研究热点与发展趋势。结合合成生物学、系统生物学、生物信息学、基因编辑技术以及人工智能的代谢工程将在未来的工业应用等多个领域不断扩展。
| [1] |
Santos MFRFD, Borschiver S. The technology roadmap for biorefinery lignin in the U.S. . Energy Power Eng, 2013, v. 3: 51-60.
|
| [2] |
吴晓燕, 陈方, 丁陈君, 等. 全球生物制造产业市场与融资现状分析. 中国生物工程杂志, 2000, 40(5): 117-124. Wu XY, Chen F, Ding CJ, et al. Analysis of the global biomanufacturing industry market and financing status. China Biotechnol, 2000, 40(5): 117-124 (in Chinese). |
| [3] |
Ostergaard S, Olsson L, Nielsen J. Metabolic engineering of Saccharomyces cerevisiae. Microbiol Mol Biol Rev, 2000, 64(1): 34-50. DOI:10.1128/MMBR.64.1.34-50.2000
|
| [4] |
Hong KK, Nielsen J. Metabolic engineering of Saccharomyces cerevisiae: a key cell factory platform for future biorefineries. Cell Mol Life Sci, 2012, 69(16): 2671-2690. DOI:10.1007/s00018-012-0945-1
|
| [5] |
Nevoigt E. Progress in metabolic engineering of Saccharomyces cerevisiae. Microbiol Mol Biol Rev, 2008, 72(3): 379-412. DOI:10.1128/MMBR.00025-07
|
| [6] |
Nielsen J, Keasling JD. Engineering cellular metabolism. Cell, 2016, 164(6): 1185-1197. DOI:10.1016/j.cell.2016.02.004
|
| [7] |
Azhar SHM, Abdulla R, Jambo SA, et al. Yeasts in sustainable bioethanol production: a review. Biochem Biophys Rep, 2017, 10: 52-61.
|
| [8] |
Schadeweg V, Boles E. n-butanol production in Saccharomyces cerevisiae is limited by the availability of coenzyme A and cytosolic acetyl-CoA. Biotechnol Biofuels, 2016, 9: 44. DOI:10.1186/s13068-016-0456-7
|
| [9] |
Lian JZ, Chao R, Zhao HM. Metabolic engineering of a Saccharomyces cerevisiae strain capable of simultaneously utilizing glucose and galactose to produce enantiopure (2R, 3R)-butanediol. Metabol Eng, 2014, 23: 92-99. DOI:10.1016/j.ymben.2014.02.003
|
| [10] |
Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast. Nature, 2006, 440(7086): 940-943. DOI:10.1038/nature04640
|
| [11] |
Shin SY, Jung SM, Kim MD, et al. Production of resveratrol from tyrosine in metabolically engineered Saccharomyces cerevisiae. Enzyme Microb Technol, 2012, 51(4): 211-216. DOI:10.1016/j.enzmictec.2012.06.005
|
| [12] |
Blazeck J, Alper HS. Promoter engineering: recent advances in controlling transcription at the most fundamental level. Biotechnol J, 2013, 8(1): 46-58. DOI:10.1002/biot.201200120
|
| [13] |
Lee ME, DeLoache WC, Cervantes B, et al. A highly characterized yeast toolkit for modular, multipart assembly. ACS Synth Biol, 2015, 4(9): 975-986. DOI:10.1021/sb500366v
|
| [14] |
Sun J, Shao ZY, Zhao H, et al. Cloning and characterization of a panel of constitutive promoters for applications in pathway engineering in Saccharomyces cerevisiae. Biotechnol Bioeng, 2012, 109(8): 2082-2092. DOI:10.1002/bit.24481
|
| [15] |
Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter engineering. Proc Natl Acad Sci USA, 2005, 102(36): 12678-12683. DOI:10.1073/pnas.0504604102
|
| [16] |
Nevoigt E, Kohnke J, Fischer CR, et al. Engineering of promoter replacement cassettes for fine-tuning of gene expression in Saccharomyces cerevisiae. Appl Environ Microbiol, 2006, 72(8): 5266-5273. DOI:10.1128/AEM.00530-06
|
| [17] |
Blazeck J, Garg R, Reed B, et al. Controlling promoter strength and regulation in Saccharomyces cerevisiae using synthetic hybrid promoters. Biotechnol Bioeng, 2012, 109(11): 2884-2895. DOI:10.1002/bit.24552
|
| [18] |
Curran KA, Crook NC, Karim AS, et al. Design of synthetic yeast promoters via tuning of nucleosome architecture. Nat Commun, 2014, 5: 4002. DOI:10.1038/ncomms5002
|
| [19] |
Redden H, Alper HS. The development and characterization of synthetic minimal yeast promoters. Nat Commun, 2015, 6: 7810. DOI:10.1038/ncomms8810
|
| [20] |
Curran KA, Karim AS, Gupta A, et al. Use of expression-enhancing terminators in Saccharomyces cerevisiae to increase mRNA half-life and improve gene expression control for metabolic engineering applications. Metab Eng, 2013, 19: 88-97. DOI:10.1016/j.ymben.2013.07.001
|
| [21] |
Yamanishi M, Ito Y, Kintaka R, et al. A genome-wide activity assessment of terminator regions in Saccharomyces cerevisiae provides a "terminatome" toolbox. ACS Synth Biol, 2013, 2(6): 337-347. DOI:10.1021/sb300116y
|
| [22] |
Curran KA, Morse NJ, Markham KA, et al. Short synthetic terminators for improved heterologous gene expression in yeast. ACS Synth Biol, 2015, 4(7): 824-832. DOI:10.1021/sb5003357
|
| [23] |
Morse NJ, Gopal MR, Wagner JM, et al. Yeast terminator function can be modulated and designed on the basis of predictions of nucleosome occupancy. ACS Synth Biol, 2017, 6(11): 2086-2095. DOI:10.1021/acssynbio.7b00138
|
| [24] |
Karim AS, Curran KA, Alper HS. Characterization of plasmid burden and copy number in Saccharomyces cerevisiae for optimization of metabolic engineering applications. FEMS Yeast Res, 2013, 13(1): 107-116. DOI:10.1111/1567-1364.12016
|
| [25] |
Lian JZ, Jin R, Zhao HM. Construction of plasmids with tunable copy numbers in Saccharomyces cerevisiae and their applications in pathway optimization and multiplex genome integration. Biotechnol Bioeng, 2016, 113(11): 2462-2473. DOI:10.1002/bit.26004
|
| [26] |
Chen Y, Partow S, Scalcinati G, et al. Enhancing the copy number of episomal plasmids in Saccharomyces cerevisiae for improved protein production. FEMS Yeast Res, 2012, 12(5): 598-607. DOI:10.1111/j.1567-1364.2012.00809.x
|
| [27] |
Zhao WJ, Shi F, Hang BJ, et al. The improvement of sam accumulation by integrating the endogenous methionine adenosyltransferase gene SAM2 in genome of the industrial Saccharomyces cerevisiae strain. Appl Biochem Biotechnol, 2016, 178(6): 1263-1272. DOI:10.1007/s12010-015-1943-1
|
| [28] |
Kim JW, Kim J, Seo SO, et al. Enhanced production of 2, 3-butanediol by engineered Saccharomyces cerevisiae through fine-tuning of pyruvate decarboxylase and NADH oxidase activities. Biotechnol Biofuels, 2016, 9: 265. DOI:10.1186/s13068-016-0677-9
|
| [29] |
Medina VG, Almering MJH, Van Maris AJA, et al. Elimination of glycerol production in anaerobic cultures of a Saccharomyces cerevisiae strain engineered to use acetic acid as an electron acceptor. Appl Environ Microbiol, 2010, 76(1): 190-195. DOI:10.1128/AEM.01772-09
|
| [30] |
Bengtsson O, Hahn-Hägerdal B, Gorwa-Grauslund MF. Xylose reductase from Pichia stipitis with altered coenzyme preference improves ethanolic xylose fermentation by recombinant Saccharomyces cerevisiae. Biotechnol Biofuels, 2009, 2: 9. DOI:10.1186/1754-6834-2-9
|
| [31] |
Matsushika A, Watanabe S, Kodaki T, et al. Expression of protein engineered NADP+-dependent xylitol dehydrogenase increases ethanol production from xylose in recombinant Saccharomyces cerevisiae. Appl Microbiol Biotechnol, 2008, 81(2): 243-255. DOI:10.1007/s00253-008-1649-1
|
| [32] |
Wang M, Yu CZ, Zhao HM. Directed evolution of xylose specific transporters to facilitate glucose-xylose co-utilization. Biotechnol Bioeng, 2016, 113(3): 484-491. DOI:10.1002/bit.25724
|
| [33] |
Lian JZ, Li YL, HamediRad M, et al. Directed evolution of a cellodextrin transporter for improved biofuel production under anaerobic conditions in Saccharomyces cerevisiae. Biotechnol Bioeng, 2014, 111(8): 1521-1531. DOI:10.1002/bit.25214
|
| [34] |
Zelle RM, De Hulster E, Van Winden WA, et al. Malic acid production by Saccharomyces cerevisiae: engineering of pyruvate carboxylation, oxaloacetate reduction, and malate export. Appl Environ Microbiol, 2008, 74(9): 2766-2777. DOI:10.1128/AEM.02591-07
|
| [35] |
Hu YT, Zhu ZW, Nielsen J, et al. Heterologous transporter expression for improved fatty alcohol secretion in yeast. Metab Eng, 2018, 45: 51-58. DOI:10.1016/j.ymben.2017.11.008
|
| [36] |
Donald KA, Hampton RY, Fritz IB. Effects of overproduction of the catalytic domain of 3-hydroxy-3-methylglutaryl coenzyme A reductase on squalene synthesis in Saccharomyces cerevisiae. Appl Environ Microbiol, 1997, 63(9): 3341-3344. DOI:10.1128/AEM.63.9.3341-3344.1997
|
| [37] |
Rico J, Pardo E, Orejas M. Enhanced production of a plant monoterpene by overexpression of the 3-hydroxy-3-methylglutaryl coenzyme A reductase catalytic domain in Saccharomyces cerevisiae. Appl Environ Microbiol, 2010, 76(19): 6449-6454. DOI:10.1128/AEM.02987-09
|
| [38] |
Xie WP, Ye LD, Lv XM, et al. Sequential control of biosynthetic pathways for balanced utilization of metabolic intermediates in Saccharomyces cerevisiae. Metab Eng, 2015, 28: 8-18. DOI:10.1016/j.ymben.2014.11.007
|
| [39] |
Paradise EM, Kirby J, Chan R, et al. Redirection of flux through the FPP branch-point in Saccharomyces cerevisiae by down-regulating squalene synthase. Biotechnol Bioeng, 2008, 100(2): 371-378. DOI:10.1002/bit.21766
|
| [40] |
Alper H, Moxley J, Nevoigt E, et al. Engineering yeast transcription machinery for improved ethanol tolerance and production. Science, 2006, 314(5805): 1565-1568. DOI:10.1126/science.1131969
|
| [41] |
Nagy A. Cre recombinase: the universal reagent for genome tailoring. Genesis, 2000, 26(2): 99-109. DOI:10.1002/(SICI)1526-968X(200002)26:2<99::AID-GENE1>3.0.CO;2-B
|
| [42] |
Turan S, Bode J. Site-specific recombinases: from tag-and-target- to tag-and-exchange-based genomic modifications. FASEB J, 2011, 25(12): 4088-4107. DOI:10.1096/fj.11-186940
|
| [43] |
Solis-Escalante D, Van Den Broek M, Kuijpers NGA, et al. The genome sequence of the popular hexose-transport-deficient Saccharomyces cerevisiae strain EBY..VW4000 reveals LoxP/Cre-induced translocations and gene loss. FEMS Yeast Res, 2015, 15(2): fou004.
|
| [44] |
Dymond JS, Richardson SM, Coombes CE, et al. Synthetic chromosome arms function in yeast and generate phenotypic diversity by design. Nature, 2011, 477(7365): 471-476. DOI:10.1038/nature10403
|
| [45] |
Jia B, Wu Y, Li BZ, et al. Precise control of SCRaMbLE in synthetic haploid and diploid yeast. Nat Commun, 2018, 9: 1933. DOI:10.1038/s41467-018-03084-4
|
| [46] |
Xu ZY, Brown WRA. Comparison and optimization of ten phage encoded serine integrases for genome engineering in Saccharomyces cerevisiae. BMC Biotechnol, 2016, 16: 13. DOI:10.1186/s12896-016-0241-5
|
| [47] |
Bogdanove AJ, Voytas DF. TAL effectors: customizable proteins for DNA targeting. Science, 2011, 333(6051): 1843-1846. DOI:10.1126/science.1204094
|
| [48] |
Li T, Huang S, Zhao XF, et al. Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes. Nucleic Acids Res, 2011, 39(14): 6315-6325. DOI:10.1093/nar/gkr188
|
| [49] |
Zhang GQ, Lin YP, Qi XN, et al. TALENs-assisted multiplex editing for accelerated genome evolution to improve yeast phenotypes. ACS Synth Biol, 2015, 4(10): 1101-1111. DOI:10.1021/acssynbio.5b00074
|
| [50] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143
|
| [51] |
DiCarlo JE, Norville JE, Mali P, et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucl Acids Res, 2013, 41(7): 4336-5343. DOI:10.1093/nar/gkt135
|
| [52] |
Jakočiūnas T, Bonde I, Herrgård M, et al. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae. Metab Eng, 2015, 28: 213-222. DOI:10.1016/j.ymben.2015.01.008
|
| [53] |
Lian JZ, Bao ZH, Hu SM, et al. Engineered CRISPR/Cas9 system for multiplex genome engineering of polyploid industrial yeast strains. Biotechnol Bioeng, 2018, 115(6): 1630-1635. DOI:10.1002/bit.26569
|
| [54] |
Ronda C, Maury J, Jakočiūnas T, et al. CrEdit: CRISPR mediated multi-loci gene integration in Saccharomyces cerevisiae. Microb Cell Fact, 2015, 14: 97. DOI:10.1186/s12934-015-0288-3
|
| [55] |
Shi SB, Liang YY, Zhang MM, et al. A highly efficient single-step, markerless strategy for multi-copy chromosomal integration of large biochemical pathways in Saccharomyces cerevisiae. Metab Eng, 2016, 33: 19-27. DOI:10.1016/j.ymben.2015.10.011
|
| [56] |
Choi KR, Jang WD, Yang D, et al. Systems metabolic engineering strategies: integrating systems and synthetic biology with metabolic engineering. Trends Biotechnol, 2019, 37(8): 817-837. DOI:10.1016/j.tibtech.2019.01.003
|
| [57] |
Park JH, Lee SY, Kim TY, et al. Application of systems biology for bioprocess development. Trends Biotechnol, 2008, 26(8): 404-412. DOI:10.1016/j.tibtech.2008.05.001
|
| [58] |
Bro C, Nielsen J. Impact of 'ome' analyses on inverse metabolic engineering. Metab Eng, 2004, 6(3): 204-211. DOI:10.1016/j.ymben.2003.11.005
|
| [59] |
Mülleder M, Calvani E, Alam MT, et al. Functional metabolomics describes the yeast biosynthetic regulome. Cell, 2016, 167(2): 553-565. DOI:10.1016/j.cell.2016.09.007
|
| [60] |
Hieter P, Boguski M. Functional genomics: it's all how you read it. Science, 1997, 278(5338): 601-602. DOI:10.1126/science.278.5338.601
|
| [61] |
Clayton RA, White O, Ketchum KA, et al. The first genome from the third domain of life. Nature, 1997, 387(6632): 459-462. DOI:10.1038/387459a0
|
| [62] |
Caspeta L, Chen Y, Ghiaci P, et al. Altered sterol composition renders yeast thermotolerant. Science, 2014, 346(6205): 75-78. DOI:10.1126/science.1258137
|
| [63] |
Hou J, Jiao CL, Peng B, et al. Mutation of a regulator Ask10p improves xylose isomerase activity through up-regulation of molecular chaperones in Saccharomyces cerevisiae. Metab Eng, 2016, 38: 241-250. DOI:10.1016/j.ymben.2016.08.001
|
| [64] |
Heller MJ. DNA microarray technology: devices, systems, and applications. Annu Rev Biomed Eng, 2002, 4: 129-153. DOI:10.1146/annurev.bioeng.4.020702.153438
|
| [65] |
Marra MA, Hillier L, Waterston RH. Expressed sequence tags——establishing bridges between genomes. Trends Genet, 1998, 14(1): 4-7. DOI:10.1016/S0168-9525(97)01355-3
|
| [66] |
Velculescu VE, Zhang L, Vogelstein B, et al. Serial analysis of gene expression. Science, 1995, 270(5235): 484-487. DOI:10.1126/science.270.5235.484
|
| [67] |
Qian X, Ba Y, Zhuang QF, et al. RNA-seq technology and its application in fish transcriptomics. OMICS, 2014, 18(2): 98-110. DOI:10.1089/omi.2013.0110
|
| [68] |
Wahlbom CF, Otero RRC, Van Zyl WH, et al. Molecular analysis of a Saccharomyces cerevisiae mutant with improved ability to utilize xylose shows enhanced expression of proteins involved in transport, initial xylose metabolism, and the pentose phosphate pathway. Appl Environ Microbiol, 2003, 69(2): 740-746. DOI:10.1128/AEM.69.2.740-746.2003
|
| [69] |
Huang MT, Bao JC, Hallström BM, et al. Efficient protein production by yeast requires global tuning of metabolism. Nat Commun, 2017, 8: 1131. DOI:10.1038/s41467-017-00999-2
|
| [70] |
Wasinger VC, Cordwell SJ, Cerpa-Poljak A, et al. Progress with gene-product mapping of the Mollicutes: Mycoplasma genitalium. Electrophoresis, 1995, 16(7): 1090-1094.
|
| [71] |
Pham TK, Chong PK, Gan CS, et al. Proteomic analysis of Saccharomyces cerevisiae under high gravity fermentation conditions. J Proteome Res, 2006, 5(12): 3411-3419. DOI:10.1021/pr060377p
|
| [72] |
Chen LW, Lee JJL, Zhang JH, et al. Comparative proteomic analysis of engineered Saccharomyces cerevisiae with enhanced free fatty acid accumulation. Appl Microbiol Biotechnol, 2016, 100(3): 1407-1420. DOI:10.1007/s00253-015-7028-9
|
| [73] |
Oldiges M, Lütz S, Pflug S, et al. Metabolomics: current state and evolving methodologies and tools. Appl Microbiol Biotechnol, 2007, 76(3): 495-511. DOI:10.1007/s00253-007-1029-2
|
| [74] |
Kell DB, Brown M, Davey HM, et al. Metabolic footprinting and systems biology: the medium is the message. Nat Rev Microbiol, 2005, 3(7): 557-565. DOI:10.1038/nrmicro1177
|
| [75] |
Nobeli I, Thornton JM. A bioinformatician's view of the metabolome. BioEssays, 2006, 28(5): 534-545. DOI:10.1002/bies.20414
|
| [76] |
Hayakawa K, Matsuda F, Shimizu H. Metabolome analysis of Saccharomyces cerevisiae and optimization of culture medium for S-adenosyl-L-methionine production. AMB Expr, 2016, 6: 38. DOI:10.1186/s13568-016-0210-3
|
| [77] |
Ming M, Wang XY, Lian LL, et al. Metabolic responses of Saccharomyces cerevisiae to ethanol stress using gas chromatography-mass spectrometry. Mol Omics, 2019, 15(3): 216-221. DOI:10.1039/C9MO00055K
|
| [78] |
Price ND, Reed JL, Palsson BØ. Genome-scale models of microbial cells: evaluating the consequences of constraints. Nat Rev Microbiol, 2004, 2(11): 886-897. DOI:10.1038/nrmicro1023
|
| [79] |
Sauer U. Metabolic networks in motion: 13C-based flux analysis. Mol Syst Biol, 2006, 2(1): 62. DOI:10.1038/msb4100109
|
| [80] |
Uk Kim H, Kim TY, Lee SY. Metabolic flux analysis and metabolic engineering of microorganisms. Mol Biosyst, 2008, 4(2): 113-120. DOI:10.1039/B712395G
|
| [81] |
Wasylenko TM, Stephanopoulos G. Metabolomic and 13C-metabolic flux analysis of a xylose-consuming Saccharomyces cerevisiae strain expressing xylose isomerase. Biotechnol Bioeng, 2015, 112(3): 470-483. DOI:10.1002/bit.25447
|
| [82] |
Dahlin J, Holkenbrink C, Marella ER, et al. Multi-omics analysis of fatty alcohol production in engineered yeasts Saccharomyces cerevisiae and Yarrowia lipolytica. Front Genet, 2019, 10: 747. DOI:10.3389/fgene.2019.00747
|
| [83] |
Edwards JS, Palsson BO. Systems properties of the Haemophilus influenzae Rd metabolic genotype. J Biol Chem, 1999, 274(25): 17410-17416. DOI:10.1074/jbc.274.25.17410
|
| [84] |
Edwards JS, Palsson BO. The Escherichia coli MG1655 in silico metabolic genotype: its definition, characteristics, and capabilities. Proc Nat Acad Sci USA, 2000, 97(10): 5528-5533. DOI:10.1073/pnas.97.10.5528
|
| [85] |
Van Gulik WM, Heijnen JJ. A metabolic network stoichiometry analysis of microbial growth and product formation. Biotech Bioeng, 1995, 48(6): 681-698. DOI:10.1002/bit.260480617
|
| [86] |
Schilling CH, Covert MW, Famili I, et al. Genome-scale metabolic model of Helicobacter pylori 26695. J Bacteriol, 2002, 184(16): 4582-4593. DOI:10.1128/JB.184.16.4582-4593.2002
|
| [87] |
Förster J, Famili I, Fu P, et al. Genome-scale reconstruction of the Saccharomyces cerevisiae metabolic network. Genome Res, 2003, 13(2): 244-253. DOI:10.1101/gr.234503
|
| [88] |
Dobson PD, Smallbone K, Jameson D, et al. Further developments towards a genome-scale metabolic model of yeast. BMC Syst Biol, 2010, 4: 145. DOI:10.1186/1752-0509-4-145
|
| [89] |
Lu HZ, Li FR, Sánchez BJ, et al. A consensus S. cerevisiae metabolic model Yeast8 and its ecosystem for comprehensively probing cellular metabolism. Nat Commun, 2019, 10(1): 3586. DOI:10.1038/s41467-019-11581-3
|
| [90] |
Bro C, Regenberg B, Förster J, et al. In silico aided metabolic engineering of Saccharomyces cerevisiae for improved bioethanol production. Metab Eng, 2006, 8(2): 102-111. DOI:10.1016/j.ymben.2005.09.007
|
| [91] |
Patil KR, Rocha I, Förster J, et al. Evolutionary programming as a platform for in silico metabolic engineering. BMC Bioinf, 2005, 6: 308. DOI:10.1186/1471-2105-6-308
|
| [92] |
Asadollahi MA, Maury J, Patil KR, et al. Enhancing sesquiterpene production in Saccharomyces cerevisiae through in silico driven metabolic engineering. Metab Eng, 2009, 11(6): 328-334. DOI:10.1016/j.ymben.2009.07.001
|
| [93] |
Ramzi AB. Metabolic engineering and synthetic biology//Aizat W, Goh HH, Baharum S, Eds. Omics Applications for Systems Biology. Cham: Springer, 2018, 1102: 81-95.
|
| [94] |
Du J, Yuan YB, Si T, et al. Customized optimization of metabolic pathways by combinatorial transcriptional engineering. Nucleic Acids Res, 2012, 40(18): e142. DOI:10.1093/nar/gks549
|
| [95] |
Song TQ, Ding MZ, Zhai F, et al. Engineering Saccharomyces cerevisiae for geranylgeraniol overproduction by combinatorial design. Sci Rep, 2017, 7: 14991. DOI:10.1038/s41598-017-15005-4
|
| [96] |
Chen XY, Zaro JL, Shen WC. Fusion protein linkers: property, design and functionality. Adv Drug Deliv Rev, 2013, 65(10): 1357-1369. DOI:10.1016/j.addr.2012.09.039
|
| [97] |
Chichili VPR, Kumar V, Sivaraman J. Linkers in the structural biology of protein-protein interactions. Prot Sci, 2013, 22(2): 153-167. DOI:10.1002/pro.2206
|
| [98] |
Ma B, Liu M, Li ZH, et al. Significantly enhanced production of patchoulol in metabolically engineered Saccharomyces cerevisiae. J Agric Food Chem, 2019, 67(31): 8590-8598. DOI:10.1021/acs.jafc.9b03456
|
| [99] |
Wang YC, Yu O. Synthetic scaffolds increased resveratrol biosynthesis in engineered yeast cells. J Biotechnol, 2012, 157(1): 258-260. DOI:10.1016/j.jbiotec.2011.11.003
|
| [100] |
Tippmann S, Anfelt J, David F, et al. Affibody scaffolds improve sesquiterpene production in Saccharomyces cerevisiae. ACS Synth Biol, 2017, 6(1): 19-28. DOI:10.1021/acssynbio.6b00109
|
| [101] |
Li TY, Chen XQ, Cai YZ, et al. Artificial protein scaffold system (AProSS): an efficient method to optimize exogenous metabolic pathways in Saccharomyces cerevisiae. Metab Eng, 2018, 49: 13-20. DOI:10.1016/j.ymben.2018.07.006
|
| [102] |
Szczebara FM, Chandelier C, Villeret C, et al. Total biosynthesis of hydrocortisone from a simple carbon source in yeast. Nat Biotechnol, 2003, 21(2): 143-149. DOI:10.1038/nbt775
|
| [103] |
Lv XM, Wang F, Zhou PP, et al. Dual regulation of cytoplasmic and mitochondrial acetyl-CoA utilization for improved isoprene production in Saccharomyces cerevisiae. Nat Commun, 2016, 7: 12851. DOI:10.1038/ncomms12851
|
| [104] |
Chen LW, Zhang JH, Chen WN. Engineering the Saccharomyces cerevisiae β-oxidation pathway to increase medium chain fatty acid production as potential biofuel. PLoS ONE, 2014, 9(1): e84853. DOI:10.1371/journal.pone.0084853
|
| [105] |
Thodey K, Galanie S, Smolke CD. A microbial biomanufacturing platform for natural and semisynthetic opioids. Nat Chem Biol, 2014, 10(10): 837-844. DOI:10.1038/nchembio.1613
|
| [106] |
Bayer TS, Widmaier DM, Temme K, et al. Synthesis of methyl halides from biomass using engineered microbes. J Am Chem Soc, 2009, 131(18): 6508-6515. DOI:10.1021/ja809461u
|
| [107] |
Farhi M, Marhevka E, Masci T, et al. Harnessing yeast subcellular compartments for the production of plant terpenoids. Metab Eng, 2011, 13(5): 474-481. DOI:10.1016/j.ymben.2011.05.001
|
| [108] |
Avalos JL, Fink GR, Stephanopoulos G. Compartmentalization of metabolic pathways in yeast mitochondria improves the production of branched-chain alcohols. Nat Biotechnol, 2013, 31(4): 335-341. DOI:10.1038/nbt.2509
|
| [109] |
Zhou YJ, Buijs NA, Zhu ZW, et al. Harnessing yeast peroxisomes for biosynthesis of fatty-acid-derived biofuels and chemicals with relieved side-pathway competition. J Am Chem Soc, 2016, 138(47): 15368-15377. DOI:10.1021/jacs.6b07394
|
| [110] |
Lau YH, Giessen TW, Altenburg WJ, et al. Prokaryotic nanocompartments form synthetic organelles in a eukaryote. Nat Commun, 2018, 9: 1311. DOI:10.1038/s41467-018-03768-x
|
| [111] |
Zhao EM, Zhang YF, Mehl J, et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature, 2018, 555(7698): 683-687. DOI:10.1038/nature26141
|
| [112] |
Zhao EM, Suek N, Wilson MZ, et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nat Chem Biol, 2019, 15(6): 589-597. DOI:10.1038/s41589-019-0284-8
|
| [113] |
Zhou PP, Xie WP, Yao Z, et al. Development of a temperature-responsive yeast cell factory using engineered Gal4 as a protein switch. Biotech Bioeng, 2018, 115(5): 1321-1330. DOI:10.1002/bit.26544
|
| [114] |
David F, Nielsen J, Siewers V. Flux control at the malonyl-coa node through hierarchical dynamic pathway regulation in Saccharomyces cerevisiae. ACS Synth Biol, 2016, 5(3): 224-233. DOI:10.1021/acssynbio.5b00161
|
| [115] |
Lian JZ, HamediRad M, Hu SM, et al. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nat Commun, 2017, 8(1): 1688. DOI:10.1038/s41467-017-01695-x
|
| [116] |
DiCarlo JE, Conley AJ, Penttilä M, et al. Yeast oligo-mediated genome engineering (YOGE). ACS Synth Biol, 2013, 2(12): 741-749. DOI:10.1021/sb400117c
|
| [117] |
Barbieri EM, Muir P, Akhuetie-Oni BO, et al. Precise editing at DNA replication forks enables multiplex genome engineering in eukaryotes. Cell, 2017, 171(6): 1453-1467. DOI:10.1016/j.cell.2017.10.034
|
| [118] |
Si T, Luo YZ, Bao ZH, et al. RNAi-assisted genome evolution in Saccharomyces cerevisiae for complex phenotype engineering. ACS Synth Biol, 2015, 4(3): 283-291. DOI:10.1021/sb500074a
|
| [119] |
Bao Z, HamediRad M, Xue P, et al. Genome-scale engineering of Saccharomyces cerevisiae with single-nucleotide precision. Nat Biotechnol, 2018, 36(6): 505-508. DOI:10.1038/nbt.4132
|
| [120] |
Lian JZ, Schultz C, Cao MF, et al. Multi-functional genome-wide CRISPR system for high throughput genotype-phenotype mapping. Nat Commun, 2019, 10: 5794. DOI:10.1038/s41467-019-13621-4
|
| [121] |
Si T, Chao R, Min YH, et al. Automated multiplex genome-scale engineering in yeast. Nat Commun, 2017, 8: 15187. DOI:10.1038/ncomms15187
|
| [122] |
Paddon CJ, Westfall PJ, Pitera DJ, et al. High-level semi-synthetic production of the potent antimalarial artemisinin. Nature, 2013, 496(7446): 528-532. DOI:10.1038/nature12051
|
| [123] |
Yu T, Zhou YJ, Huang MT, et al. Reprogramming yeast metabolism from alcoholic fermentation to lipogenesis. Cell, 2018, 174(6): 1549-1558. DOI:10.1016/j.cell.2018.07.013
|
| [124] |
Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast. Science, 2015, 349(6252): 1095-1100. DOI:10.1126/science.aac9373
|
| [125] |
Meadows AL, Hawkins KM, Tsegaye Y, et al. Rewriting yeast central carbon metabolism for industrial isoprenoid production. Nature, 2016, 537(7622): 694-697. DOI:10.1038/nature19769
|
| [126] |
Dörr M, Fibinger MPC, Last D, et al. Fully automatized high-throughput enzyme library screening using a robotic platform. Biotechnol Bioeng, 2016, 113(7): 1421-1432. DOI:10.1002/bit.25925
|
| [127] |
Mair P, Gielen F, Hollfelder F. Exploring sequence space in search of functional enzymes using microfluidic droplets. Curr Opin Chem Biol, 2017, 37: 137-144. DOI:10.1016/j.cbpa.2017.02.018
|
| [128] |
Si T, Li B, Comi TJ, et al. Profiling of microbial colonies for high-throughput engineering of multistep enzymatic reactions via optically guided matrix-assisted laser desorption/Ionization mass spectrometry. J Am Chem Soc, 2017, 139(36): 12466-12473. DOI:10.1021/jacs.7b04641
|
| [129] |
Li DW, Ma YS, Zhou Y, et al. A structural and data-driven approach to engineering a plant cytochrome P450 enzyme. Sci China Life Sci, 2019, 62(7): 873-882. DOI:10.1007/s11427-019-9538-3
|
| [130] |
An-Adirekkun JM, Stewart CJ, Geller SH, et al. A yeast optogenetic toolkit (yOTK) for gene expression control in Saccharomyces cerevisiae. Biotechnol Bioeng, 2020, 117(3): 886-893. DOI:10.1002/bit.27234
|
| [131] |
Lohmann KJ. Protein complexes: A candidate magnetoreceptor. Nat Mater, 2016, 15(2): 136-138. DOI:10.1038/nmat4550
|
 2021, Vol. 37
2021, Vol. 37





