中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王颖珊, 郭峰, 严伟, 信丰学, 章文明, 姜岷
- Wang Yingshan, Guo Feng, Yan Wei, Xin Fengxue, Zhang Wenming, Jiang Min
- 四碳有机酸生物合成的代谢工程研究进展
- Advances in the metabolic engineering for the production of tetracarbon organic acids
- 生物工程学报, 2021, 37(5): 1697-1720
- Chinese Journal of Biotechnology, 2021, 37(5): 1697-1720
- 10.13345/j.cjb.200727
-
文章历史
- Received: November 13, 2020
- Accepted: February 22, 2021
- Published: February 25, 2021
2. 南京工业大学 江苏先进生物与化学制造协同创新中心 (SICAM),江苏 南京 211816
2. Jiangsu National Synergetic Innovation Center for Advanced Materials (SICAM), Nanjing Tech University, Nanjing 211816, Jiangsu, China
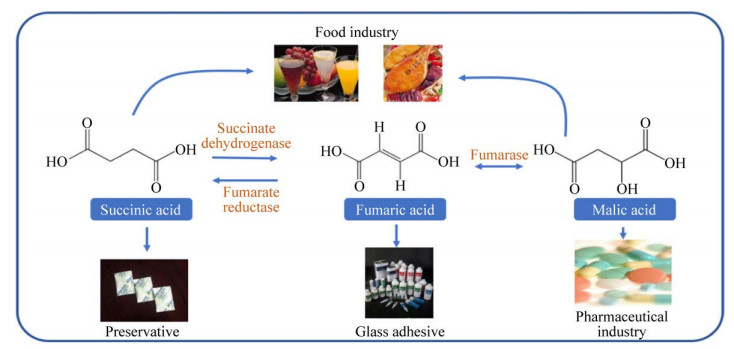
四碳有机酸是一类含有4个碳原子且具有一个或两个酸性官能团的低分子量有机化合物。以富马酸、苹果酸和琥珀酸等为代表的四碳有机酸已经被广泛应用于食品、制药、化妆品、洗涤剂、聚合物和纺织等领域(图 1)[1]。美国能源部已经将包括富马酸、苹果酸和琥珀酸在内的多种四碳有机酸列为重要的平台化学品。传统的四碳有机酸的生产方法是依赖以石油为原料的化学合成法。然而化学合成过程通常发生在高温下,会导致副产物的形成以及造成巨大的能量消耗。此外,石油作为一种不可再生的资源,它的使用还会造成温室气体排放等问题。除化学合成法外,可以用酶促转化的方法生产纯的四碳有机酸,然而,所使用的酶对温度的敏感性和底物抑制作用会限制四碳有机酸的大规模生产。因此,与化学合成和酶促转化相比,利用可再生生物质原料,依靠生物发酵法生产四碳有机酸具有发酵效率高、成本低廉等优点,被认为是更有前途的路线[2]。在过去的几十年里,已经开展了大量关于四碳有机酸生物合成的研究,发掘了大量的各种类型的四碳有机酸生产的底盘微生物[3]。在此基础上,通过随机诱变和筛选、合理的代谢工程以及近年来兴起的系统代谢工程手段,获得了一批性状优良,生产性能和鲁棒性更强的改良菌株。
|
| 图 1 四碳有机酸的转化与应用 Fig. 1 Interconversion of tetracarbon organic acids and their applications. |
| |
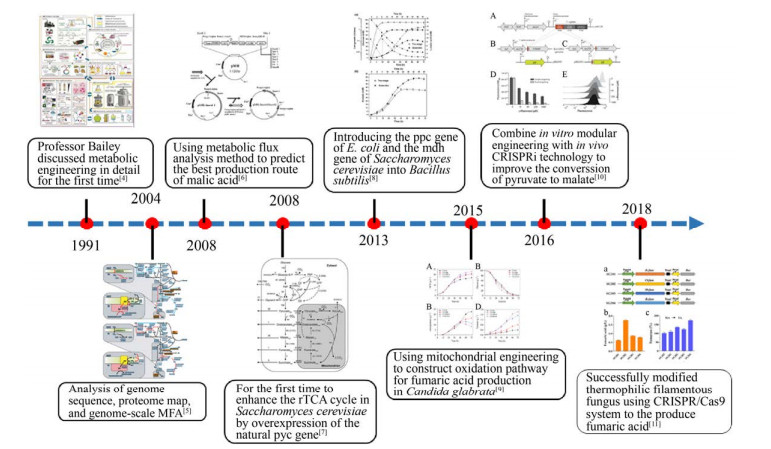
1991年,美国加州理工学院Bailey教授首次详细论述了代谢工程,标志代谢工程新学科的诞生[4],在这短短30年里,它与细胞生物工程、基因工程等技术的联合应用已成为研究和控制微生物生长代谢的一个必不可少的手段。2004年,随着代谢通量分析方法的不断发展,Lee等尝试对基因组序列、蛋白质组图和基因组规模的MFA进行分析,确定了负责琥珀酸生产的目标蛋白并开始对代谢特性有了深入的了解[5]。为了利用代谢工程手段提升苹果酸的产量,2008年,研究人员进一步使用代谢通量分析方法,预测最佳的苹果酸生产途径,最终将pck基因引入了pta基因突变的大肠杆菌中,从而提高了大肠杆菌发酵产苹果酸的产量[6]。除了应用代谢通量分析等方法外,Zelle等首次通过天然pyc基因的过表达增强酿酒酵母中的还原三羧酸(rTCA) 循环,生物反应器的进一步放大和优化使苹果酸产量大幅度提高[7]。几年后,研究人员逐渐发现不同微生物来源的关键酶对有机酸的产生有很大影响,于是尝试将大肠杆菌的ppc基因和酿酒酵母的mdh基因组成的高效异源生物合成途径引入枯草芽孢杆菌,这代表了第一个成功设计的用于生产苹果酸的枯草芽孢杆菌,为进一步提高枯草芽孢杆菌中苹果酸的产量铺平了道路[8]。近年来,除了基因的引入或过表达外,通过开发DNA支架、RNA支架和蛋白质支架合成生物技术,以提高途径转运效率,也已被用于生产许多化学品。2015年,研究人员应用合成生物学策略进一步优化代谢平衡,利用线粒体工程学构建光滑念珠菌中富马酸生产的氧化途径,最终得到高产量富马酸[9]。随着CRISPRi技术的发展,2016年研究人员通过将体外模块工程与体内CRISPRi技术相结合,提高丙酮酸至苹果酸的转化,同时发现,使用CRISPRi技术还可以减少操纵子环境中多个基因异源表达引起的代谢负担[10]。两年后,Liu等通过使用CRISPR/Cas9系统成功地改造了嗜热丝状真菌,用于富马酸的生产[11] (图 2)。随着合成生物学的飞速发展,基因表达的微调应用于许多生物和异源系统中。同时,新型的核糖调节器开关成功应用于调节大肠杆菌中的基因表达并调节代谢通量,从而提高琥珀酸的产量。通过代谢工程途径生产四碳有机酸,不仅有助于实现资源替代,同时可以为四碳有机酸产业的发展和提升提供技术支撑。本文介绍了四碳有机酸的生产途径和代谢机制,综述了微生物发酵法生产富马酸、苹果酸和琥珀酸的代表性成果,并探讨了生物发酵法高效合成四碳有机酸的可行策略及前景展望。
一般来说,在微生物体内,四碳有机酸主要通过以下3种途径生成(图 3):位于胞质的还原三羧酸循环途径、位于线粒体内的氧化三羧酸循环途径和乙醛酸分流途径。

|
| 图 3 四碳有机酸合成路线(Red line:四碳有机酸合成路径;PYR:丙酮酸;OAA:草酰乙酸;MAL:苹果酸;FUM:富马酸;SUC:琥珀酸;SUCC:琥珀酰辅酶;α-AKG:α-酮戊二酸;ICI:异柠檬酸;CIT:柠檬酸;AcCoA:乙酰辅酶A;Blue line:三羧酸循环) Fig. 3 Biosynthetic routes for tetracarbon organic acids. |
| |
还原三羧酸循环以丙酮酸的羧化作为起点,通常被认为是真核微生物中有机酸积累的主要途径[12]。在该途径中,丙酮酸首先在ATP和CO2的参与下通过位于胞质中的丙酮酸羧化酶(PYC) 转化为草酰乙酸[13]。随后,草酰乙酸在苹果酸脱氢酶(MDH)、富马酸酶(FUM) 和富马酸还原酶(FRD) 的作用下被依次转化为苹果酸、富马酸和琥珀酸。由于第一步中丙酮酸的羧化固定了一分子的CO2,该途径的最大理论产率为2 mol/mol葡萄糖。另一方面,相比于氧化TCA循环,还原三羧酸途径由于其是定位于胞质的线性的代谢途径,除目标产物外几乎不积累其余有机酸,产物的转化率更高。因此,还原三羧酸循环通常被认为是最为高效的四碳有机酸生产途径。通过引入来源于天然四碳有机酸生产菌株中该途径的相关基因,已经在包括大肠杆菌和酿酒酵母在内的多种模式微生物中实现了各种四碳有机酸的生产。除了在PYC和MDH的作用下由丙酮酸转化为苹果酸外,苹果酸酶(ME) 可以在NADP+/NADPH或NAD+/NADH的参与下,一步催化丙酮酸和苹果酸的相互转化。尽管经过了几十年的研究,ME在大多数微生物中的生理功能仍然没有得到明确的阐明。目前的研究发现,大多数的微生物都具有NADP+依赖的ME;同时也有一些细菌,如大肠杆菌、枯草芽孢杆菌、根瘤菌、假单胞菌等,同时具有NADP+和NAD+依赖型的ME。
四碳有机酸生产的第2个主要途径是氧化三羧酸循环,它在四碳有机酸的积累中也发挥了重要的作用[14]。在此途径中,草酰乙酸和乙酰辅酶A在柠檬酸合酶的作用下被转化成柠檬酸,随后在好氧条件下在线粒体中发生两次氧化反应,生成琥珀酸,继而通过琥珀酸脱氢酶和富马酸酶的作用生成富马酸和苹果酸。该循环由于涉及CO2的排放,最高理论产率被限制在1 mol/mol葡萄糖。由于富马酸酶的高活性,通过氧化三羧酸循环通常难以得到高效的富马酸积累,富马酸更容易被进一步地转化为苹果酸。此外,对于原核生物,一些特异的调节因子的删除已经被证明能够增强这一氧化途径的活性,如DNA结合相应调节子arcA,乳糖操纵子的转录抑制子lacⅠ等。
除了上述两种主要的生产途径,乙醛酸途径也是生产四碳有机酸的潜在途径[15]。在该途径中,通过氧化三羧酸循环形成的异柠檬酸在异柠檬酸裂解酶的催化下分解为琥珀酸和乙醛酸。随后乙醛酸与乙酰辅酶A结合,在苹果酸合成酶的催化下合成苹果酸[16]。虽然其理论收率不如还原三羧酸循环,但乙醛酸旁路的代谢途径相对较短,反应简单,因而具有很大的开发潜力。然而,由于以葡萄糖为底物时形成的中间体磷酸烯醇式丙酮酸会抑制该途径中关键酶异柠檬酸裂解酶的作用,乙醛酸途径在高糖环境中往往会受到强烈的抑制作用而难以激活[17]。
在以上3种途径中,还原三羧酸途径产物的转化率最高,且副产物有机酸的积累量更少,通常被认为是最高效的四碳有机酸积累途径。氧化三羧酸循环在好氧条件下线粒体内进行,最大理论产率低于还原三羧酸途径。乙醛酸途径作为一种潜在途径,虽然其理论产率低于还原三羧酸途径,但具有代谢途径短、反应简单等优势,因此乙醛酸途径是具有开发潜力的代谢途径。
除了以上3种途径外,近年来的研究证明氨基酸和脂肪酸代谢也参与了真核生物四碳有机酸的产生。例如,Yu等分析了经紫外诱变的富马酸高产菌株与原始菌株的代谢物丰度,发现高产菌株中碳和氨基酸代谢水平较高[18]。与原始菌株相比,4-氨基丁酸和5-氨基乙酰丙酸含量分别提高了10.33倍和7.22倍。这些发现证明,调节氨基酸和脂肪酸的代谢或许也是提高有机酸生产的一个可行策略。近年来,一系列通过改造尿素和嘌呤核苷酸循环来生产四碳有机酸的研究也进一步证实了这一假设[19]。
除了合成路径,高效的转运系统也是提高四碳有机酸积累的关键因素之一。目前对于四碳有机酸的转运机制依旧缺乏系统的报道。在酿酒酵母中,苹果酸、琥珀酸和富马酸已被证明很可能以被动扩散的方式进入细胞。目前的研究表明,这些四碳二羧酸很可能是通过共用一套转运系统进出细胞的。大量的研究已经证明,通过引入来源于粟酒裂殖酵母Schizosaccharomyces pombe四碳二羧酸转运蛋白SpMAE能够显著地提高苹果酸、富马酸和琥珀酸的转运[20]。另一方面,在胞质与线粒体中分布有大量的线粒体转运蛋白,近年来的研究逐渐解析了其中相当一部分转运蛋白的功能,其中的绝大多数具有双向的转运活性。例如线粒体二羧酸转运蛋白(Mitochondrial dicarboxylate carrier,DIC1p) 被鉴定为一种负责二元羧酸交换的转运蛋白,通过强制性反交换机制转运二元羧酸,如苹果酸、丁二酸或丙二酸以及无机磷酸盐。DIC1p的转运特性与其他线粒体中的二羧酸载体非常相似,转运方程为malate-cyt+pi-mit malate-mit+pi-cyt。而酿酒酵母中的另一种转运蛋白ODC1p (Mitochondrial oxodicarboxylate carrier),通过反交换机制可高效转运α-酮戊二酸和氧代己二酸,并在较小程度上运输相应的二羧酸和苹果酸。ODC1p在酵母中的重要生理作用是将α-酮戊二酸输出到胞质溶液中,参与胞质酶的氮同化反应,如谷氨酸脱氢酶、谷氨酸合酶和谷氨酰胺合酶,同时换取苹果酸转至线粒体中,转运方程为malate-cyt+oxoglutarate-mit malate-mit+oxoglutarate- cyt。遗憾的是,目前针对线粒体转运蛋白的改造是缺乏的,或许这是未来代谢工程或是酶工程提高四碳有机酸产量的一个可行的方向。
2 富马酸生物生产富马酸作为一种四碳二羧酸,由于其化学结构易于酯化和聚合[21],被广泛应用于食品、化工、农业、医药等行业。主要可作为化工原料的不饱和聚酯树脂、醇酸树脂、增塑剂以及用作生产电泳漆的原料[22]。此外,富马酸因其无毒特性和独特的风味,纯度高于99%的食品级富马酸也被广泛用作食品酸剂和饮料配料[23]。医药上富马酸可以用于生产解毒药二巯基丁二酸钠等,富马酸钠与硫酸亚铁的置换产物富马酸铁则广泛应用于医学上治疗人体的小红血球贫血病[24]。基于这些广泛的用途,富马酸已经被美国能源部列为优先发展的12种平台化合物之一[21]。随着其应用领域的不断拓展,目前全球对富马酸的需求量提升速度加快,2012年全球对富马酸的需求为225.2 kt,到2020年需求量已超过300 kt。富马酸可通过马来酸在矿物酸、过氧化合物或硫脲的催化作用下异构化合成。该方法因产率较高,是目前应用最广泛的富马酸生产方法。然而,异构化过程通常需要提供一个高温、高压的条件,这不仅意味着巨大的能量消耗,还往往伴随着副产物的积累。化学合成法制备富马酸所面临的主要问题是化石资源的短缺、生产成本的提高以及对环境造成的污染,石油作为一种不可再生的资源,其储存量是有限的,石油资源的短缺使得世界石油市场供需严重失衡[25],因此,寻找新的资源和新的合成路径已经刻不容缓。与石化方法相对的微生物发酵法由于其可持续性和环境友好性近年来愈来愈引起人们的兴趣。通过菌株筛选、条件诱变、代谢工程改造以及优化发酵条件手段,微生物发酵法在产量、收率以及生产强度上都已经达到了相当可观的水平,有的生产方法已经实现了产业化。
2.1 利用野生型和诱变菌株生产富马酸一些细菌,如运动发酵单胞菌Zymomonas mobilis、蜡样芽孢杆菌Bacillus macerans、嗜热厌氧杆菌Thermoanaerobacter ethanolicus、菊欧文氏菌Erwinia chrysanthemi等,和酵母菌包括树干毕赤酵母Scheffersomyces stipitis、嗜鞣管囊酵母Pachysolen tannophilus等均已被发现能够天然地积累富马酸。然而,丝状真菌仍然是富马酸主要的生产菌株,尤其是丝状毛霉菌,例如黑根霉Rhizopus nigricans、少根根酶Rhizopus arrhizus、米根霉Rhizopus oryzae等[25]。
一般认为,根霉属Rhizopus sp.菌株是最为理想的富马酸生产菌株。其中,少根根酶曾被认为是最适的富马酸生产菌株,是由于它能够达到更高的富马酸产量。然而,少根根酶对营养的需求更为严格,这提高了整个生物发酵过程的成本。另一方面,尽管具有相对更高的产量,少根根酶生产富马酸的收率并不高,这也进一步提高了底物成本。例如,Lee等以少根根酶生产富马酸,尽管获得了121.0 g/L的产量,但是其收率仅为0.37 g/g[26]。因此,营养需求较低、底物转化率更高的米根霉已经成为富马酸生产的主要菌株。据报道,通过与下游分离装置组合,米根霉ATCC 20344能够在旋转生物膜接触器中生产0.91 g/g的富马酸,且生产强度可达到4.25 g/(L·h)[27]。与诱变型菌株相比,根霉作为野生型菌株具有在自然界分布较广、易于获得、不需经过处理,生产条件相对简单等优势,是最为理想的富马酸生产菌株。尽管野生型的根霉已经具有良好的生产性能,但相比于石化方法依旧缺乏足够的竞争力,其生产能力仍不能满足商业化的需求。因此通过一些物理、化学或两者结合的随机诱变手段改造野生型菌株,是获取性状优良的高产、抗逆菌株的一条捷径,尤其是对于根霉这样目前缺少遗传改造工具的微生物。常见的物理诱变方法包括紫外线或γ-射线诱变,常见的化学诱变方法则主要是通过添加烷化剂、亚硝基化合物或氯化锂进行诱变。对于随机诱变,高效的筛选方式是获取目的菌株的关键,在这方面,近年来的研究开发了一系列新的,高效的筛选方法。例如,Xu等发现以少根根霉为出发菌种,通过紫外线和氯化锂诱变处理,发现当紫外线照射时间为3 min,并在质量分数4%的氯化锂的平板中培养,可诱变出富马酸高产菌[28]。在此基础上,用溴甲酚绿加塑料小管的平板进行对诱变菌株进行初筛,其生成的富马酸会通过塑料小管底部渗透到指示培养基中产生变色圈。根据变色圈大小就可以初步判断诱变菌株的产酸能力,从而大大缩短了筛选时间。通过这一筛选方法筛选获得的菌株在发酵72 h后能产生55.02 g/L的富马酸,相比于原始菌种提高了2.49倍[28]。随机诱变除了用于筛选富马酸高产的菌株,还可用于筛选副产物产量更低的突变体。乙醇是富马酸发酵过程中的主要副产物,它的积累降低了碳通量向富马酸的转化。Fu等采用紫外与亚硝基胍结合的诱变方法,通过烯丙醇抗性筛选,分离出了低乙醇产量的突变体R. oryzae ME-F01;与亲本相比,突变体的富马酸产量提高了21.1%,而乙醇产量则降低了83.7%[29]。
相对于随机诱变,适应性进化是一种更有针对性的菌株选育策略,可以根据特定的需求和目的确定菌株进化方向。近年来,这一策略也被用于筛选特异的富马酸生产菌株,尤其是用于筛选能够利用其他碳源的突变体。例如,Deng等通过适应性进化的方式获得了一批米根霉突变体,其中表现最好的菌株可以以玉米淀粉为底物生产39.8 g/L的富马酸[30]。具体来说,由于淀粉利用能力可以通过葡萄糖淀粉酶的活性来表征,研究人员以葡萄糖类似物2-脱氧葡萄糖作为进化选育压力,高效地筛选了具有高葡萄糖淀粉酶活性的突变体。类似的方法也被用于筛选以木糖为碳源生产富马酸的少根根霉。通过不断提高培养基中木糖的浓度,Wen等最终获得了一株能利用木糖生产28.5 g/L的富马酸的突变菌株R. arrhizus RH-7-13,而亲本菌株在相同条件下只能积累13.2 g/L的富马酸[31]。
2.2 利用代谢工程菌株生产富马酸近年来,代谢工程的迅猛发展使我们能够在分子层面上有目的地对细胞代谢途径进行修饰、改造,改变细胞特性。通过代谢构建的菌株能够具有较高的富马酸产量、较少的副产物形成或者更有效的底物利用率。通过代谢工程提高富马酸生产的策略大致可归纳为:1) 过量表达关键限速酶;2) 引入新的代谢途径;3) 敲除副产物积累相关的途径;4) 平衡能量代谢及辅因子生成;5) 消除反馈抑制;6) 相关转运蛋白的增强或抑制等方面。
2.2.1 原核生物生产富马酸的代谢工程改造大肠杆菌Escherichia coli凭借其较快的生长速度、清晰的遗传背景以及较广的底物利用谱,被广泛的应用到富马酸的合成中。作为最为常见的模式菌株,通过代谢工程策略改造大肠杆菌生产富马酸的研究是开展得最早,同时也是研究得最深入的。对于原核生物,常见的提高富马酸生产能力的策略是过量表达参与氧化三羧酸循环的基因。除了这些常见的策略,近年来一些调节因子已被证实在氧化三羧酸循环中发挥重要的作用。例如,arcA是一个DNA结合反应调节因子,敲除arcA会导致三羧酸循环加剧。缺失arcA的大肠杆菌被发现能够具有更好的生长能力,富马酸产量会提高1.5倍[15]。草酰乙酸作为糖酵解途径的前体,在富马酸积累过程中起着重要作用。因此,促进草酰乙酸的合成可以作为一种可行的策略以提高富马酸产量。在E. coli CWF41中通过强启动子tac过量表达PPC,PPC的酶活相比于原始菌株提高了4.7倍,富马酸产量则提高2.8倍。进一步地敲除副产物合成相关的基因后,最终菌株在分批培养条件下的富马酸产量为28.2 g/L,生产强度为0.448 g/(L·h)[32]。另一种常见的增加大肠杆菌中富马酸积累的方法是增强乙醛酸分流的碳通量。具体的方法包括敲除编码异柠檬酸裂解酶抑制因子的基因iclR、乳酸脱氢酶LDH以及丙酮酸甲酸裂解酶PFL等。减少苹果酸和琥珀酸等主要副产物的积累也是常见的代谢工程改造菌株的思路。例如,在一株可以生产富马酸的原始菌株E. coli JM109(DE3) 中敲除基因fumABC、frdABCD和iclR后,重组菌能产生2.12 mmol/L的富马酸[33]。除了苹果酸与琥珀酸这两大主要的副产物,其他副产物如乙酸、乳酸和天冬氨酸积累的抑制也可以提高富马酸的产量。例如,乙酰辅酶A合成酶(ACS) 是乙酸分泌和同化的关键酶之一。为了减少乙酸的积累,在E. coli ABCDIA-ptsG−-mdh−-glk-galp-ppc中过量表达ACS,重组菌株比的富马酸产量比原始菌株提高了1.5倍[34]。
2.2.2 真核生物生产富马酸的代谢工程改造与大肠杆菌相比,酵母具有更高的耐酸能力,发酵过程副产物种类更少,具有较强的工业化潜能,是良好的工业生产有机酸的底盘微生物[35]。然而据报道,酵母中内源的富马酸酶对富马酸的亲和力是对l-苹果酸亲和力的17倍[36],导致胞质富马酸酶主要催化富马酸向苹果酸的转化,而非富马酸的积累。Xu等将来源于米根霉的富马酸合成的rTCA途径引入酿酒酵母中,通过过量表达RoMDH、RoFUM1以及内源的PYC基因,减少富马酸向苹果酸的转化,从而增加富马酸的积累,重组酿酒酵母能够生产3.2 g/L的富马酸[37]。除了酿酒酵母,一些其他非常规酵母同样也被开发用作富马酸的生物合成。最近的研究发现,调控精氨酸琥珀酸裂解酶(ASL)、腺苷琥珀酸裂解酶(ADSL)、延胡索酰乙酰乙酸酶(FAA) 和富马酸酶4种胞质酶的表达水平已经能够调节尿素和嘌呤核苷酸循环,这类酶参与了光滑念珠菌富马酸的生物合成。例如,通过控制ASL的高水平表达和ADSL的低水平表达,可以获得5.6 g/L的富马酸,在此基础上进一步过量表达四碳二羧酸转运蛋白SpMAE可使富马酸的产量提高到8.8 g/L。
近年来合成生物学和系统生物学的发展使得对于代谢途径的调控不再着眼于单一途径的表达或敲除,而能够通过模块化、区室化的划分合理地分配和调控整体的代谢网络。例如,在酿酒酵母中同时引入富马酸合成的氧化和还原途径后,重组菌株FMME004-6的富马酸产量仅为5.6 g/L,这说明简单地引入外源的代谢途径往往难以达到良好的效果。因此,近年来许多的合成生物学策略被用以优化代谢平衡或提高中间代谢物的运输效率,例如线粒体工程、支架工程、辅因子工程和模块化组装[38]。例如,辅因子再生已被证明是通过改变细胞内辅因子池来维持细胞氧化还原平衡的有效途径,许多酶已被证明能够直接影响NADH/NAD+或ATP/ADP的比率,例如磷酸烯醇式丙酮酸羧激酶(PCK) 和甲酸脱氢酶(FDH)。因此,通过过量表达PCK产生ATP,以促进RoPYC催化的丙酮酸羧化为草酰乙酸,过量表达FDH产生NADH,驱动RoMDH催化的草酰乙酸还原为苹果酸。通过这一设计,改变NADH/NAD+和ATP/ADP的比率后重组菌株相比于对照菌株的富马酸产量提高了63.7%,终产量达到了21.6 g/L。
米根霉作为最常用的富马酸生产菌株,已经成功地通过代谢工程的策略实现富马酸产量的提高。磷酸烯醇式丙酮酸羧化酶(PPC) 和丙酮酸羧化酶(PYC) 分别催化磷酸烯醇式丙酮酸和丙酮酸转化为草酰乙酸,因此过量表达PPC和PYC被认为能够增加菌株中草酰乙酸的通量,从而提高富马酸的产量。Zhang等在R. oryzae M16中分别过量表达了内源的PYC和PPC;与原始菌株相比,过量表达PPC的菌株富马酸的产量提高了26%,收率达到了0.78 g/g;而过量表达PYC的菌株生长受到显著影响,产量极低(< 0.05 g/g)[39]。除了过表达PPC和PYC之外,研究人员发现CRISPR/Cas9是一种能够通过DNA剪接技术实现基因编辑的方法。最近,利用CRISPR/Cas9,Gu等成功地在嗜热丝状真菌Myceliophthora thermophila实现了基因编辑,用于富马酸的生产。研究人员首先将来源于克鲁斯假丝酵母的编码富马酸酶的基因fum导入嗜热丝状真菌中,发现富马酸的产量提高了3倍[40]。为了进一步提高富马酸的产量,借助于开发的CRISPR/Cas9系统,两个胞质富马酸还原酶FRD1和FRD2,线粒体富马酸酶FUM被敲除。此外,由于苹果酸是富马酸合成的前体,线粒体苹果酸转运载体也被敲除以促进l-苹果酸的合成。最终菌株在补料分批发酵中获得了17.0 g/L的富马酸。
传统的根霉菌发酵有机酸主要以葡萄糖和淀粉等为原料,成本相对较高,近几年粮食价格的上涨,以粮食类原料为底物发酵生产有机酸面临着巨大的挑战。采用廉价易得的木质纤维素生产有机酸,不仅能降低原料成本,还对解决粮食危机及环境污染问题具有重要意义[41]。目前关于木质纤维素研究主要集中在纤维素乙醇方面,但乙醇对糖的转化率较低,如葡萄糖转化为乙醇的理论转化率仅为51%,而同样的葡萄糖对乳酸的理论转化率为75%,对富马酸的理论转化率达129%,故用木质纤维素生产有机酸理论上较乙醇具有更高的原料利用率[41]。
3 苹果酸的生物生产苹果酸作为一种四碳有机酸,同样具有广泛而强大的功能。生理上,苹果酸被证明对心脏具有保护作用,可以促进线粒体呼吸作用和促进细胞内ATP产生,强化体内的能量代谢。外源补充苹果酸能够影响机体氧化还原状态,从而提高机体的抗氧化能力。此外苹果酸对正常体力劳动及紧张劳动后的体力恢复有显著影响。在食品行业中,苹果酸可以作为酸味调节剂、保鲜剂、面食强化剂,且具有减盐作用和护色作用;在医药行业中,苹果酸可以降低血氨浓度,对肝脏有保护作用,是治疗肝衰竭、肝癌的良药,同时作为心脏病基础液成分之一,对心肌梗塞的缺血性心肌层起到保护作用,也可作为抗癌药的前体以及动物生长促进剂[45];在化学工业方面,苹果酸可以作为牙膏和烟草的调味剂、皮肤清洁剂、洗涤剂、废气脱硫剂,也可以代替柠檬酸作为各种金属表面的除锈剂。美国能源部已将苹果酸列为12大基本化工原料之一,预计苹果酸的市场潜力将超过每年20万t[3]。苹果酸包括D-、l-和对称碳原子的光学异构体的混合3种类型,在实际应用中3种类型的苹果酸发挥不同的作用。化学合成路线是合成苹果酸的主要方法,以石油衍生的马来酸或富马酸为底物,通过水化反应生成DL-混合的苹果酸外消旋物。化学合成法主要包括反丁烯二酸或顺丁烯二酸经高温高压催化加水等几种方法。虽然化学合成法生产成本较低,但其生产的苹果酸不易被人体吸收且具有一定的毒副作用,限制了其在食品、医药行业的广泛应用。
| Strain | Description | Substrate | Titer (g/L) | Yield (g/g) | Productivity (g/(L·h)) | References |
| E. coli EF02 | Expression of ppc; deletion of fumB, fumAC and aspA | Glycerol | 41.5 | 0.88 | 0.51 | [17] |
| R. oryzae FM19 | Femtosecond laser | Glucose | 49.4 | 0.56 | 0.29 | [18] |
| R. oryzae ZJU11 | UV | Glucose | 57.4 | – | – | [28] |
| R. oryzae ME-UN-8 | UV, NTG | Glucose | 52.7 | – | – | [29] |
| E. coli CWF812 | Expression of ppc; deletion of iclR, fumC, fumA, fumB, arcA, ptsG, aspA and lacI | Glucose | 28.2 | 0.39 | 0.45 | [32] |
| R. oryzae pyc | Expression of pyc | Glucose | 0.7 | < 0.5 | – | [39] |
| R. oryzae ppc | Expression of ppc | Glucose | 0.25 | 0.78 | 0.26 | [39] |
| M.thermophila SG515 | Expression of SpMAE1, Ckfum and Mtsfc; deletion of Mtfrd1, Mtfrd2, Mtfum and moc | Glucose | 17.0 | 0.24 | – | [40] |
| R. oryzae RUR709 | UV, γ-rays | Glucose | 26.2 | 0.32 | 0.22 | [42] |
| R. oryzae fumR2 | Expression of fumR2 | Glucose | 21.5 | 0.65 | – | [43] |
| S. stipitis PSYPMFfS | Expression of Ymae1; deletion of ura3, leu2, Psfum1 and Psfum | Xylose | 4.7 | 0.10 | – | [44] |
除了化学合成法,利用固定化或分离的富马酸酶对富马酸进行酶促转化,也可用于l-苹果酸生产。酶转化法生产l-苹果酸主要以富马酸酶转化富马酸为主,因此原料依然依赖于高纯度化学合成的富马酸,其生产过程存在原料价格昂贵、生产污染大、产品中杂酸含量偏高等缺点。且富马酸酶对温度的敏感性和底物抑制效应限制了该方法的大规模应用[46]。相对于化学合成或酶促转化,利用木质纤维素等可再生的原料底物取代石化原料合成l-苹果酸是一条较为经济环保的路线。特别是随着化石燃料的消耗和对环境问题的关注,微生物发酵生产l-苹果酸具有反应条件温和、选择性高和产品分布可控等优点。微生物发酵法已经日益成为人们关注的焦点。
3.1 利用野生型菌株生产苹果酸最初,l-苹果酸主要是从苹果酸含量不超过1%的苹果汁或蛋壳中提取[47]。随着化学合成路线、酶转化路线以及微生物发酵路线的迅速发展,传统的合成方法已被完全取代。
曲霉菌包括黄曲霉、黑曲霉和米曲霉等,都是天然的l-苹果酸生产菌株,被广泛用于生产l-苹果酸[48] (表 2)。早在20世纪60年代,研究人员利用一株分离的黄曲霉菌株获得了苹果酸生产工艺的专利,该菌株的苹果酸产量为58 g/L,收率和生产强度分别为0.58 g/g和0.27 g/(L·h)。通过一系列的工艺优化,利用Aflatoxin flavus ATCC 13697在16 L发酵罐中发酵6–8 d,以葡萄糖为底物时获得113 g/L苹果酸,生产强度为0.59 g/(L·h)。然而,由于黄曲霉发酵过程中会产生致癌的黄曲霉素,黄曲霉并不适合用于发酵生产苹果酸。由于黄曲霉和米曲霉之间的高度同源性,Knuf等以A. oryzae NRRL 3488为出发菌株进行苹果酸生产,在高糖限氮的条件下,能够在47.5 h内生产30.27 g/L的苹果酸,收率为0.67 g/g[49]。进一步研究发现,不同碳源对米曲霉的发酵性能影响很大。例如,当使用木糖或甘油作为底物时,A. oryzae DSM 1863仅积累45.43 g/L的苹果酸,明显低于以葡萄糖作为底物时的产量(58.16 g/L)。曲霉菌广泛分布于土壤、灰尘、植物及其果实上,生长温度范围广,易于获得和培养,相比于需要改造的代谢工程菌株,是一类良好的野生型产苹果酸菌株。
| Strain | Description | Substrate | Titer (g/L) | Yield (g/g) | Productivity (g/(L·h)) | References |
| Penicillium viticola 152 | – | Glucose | 131 | 1.00 | 1.36 | [50] |
| Ustilago trichophora TZ1 | – | Glycerol | 196 | 0.82 | 0.39 | [54] |
| Escherichia coli | Expression of Afpyc, Scms, cs, can and icl | Glucose | 36.05 | 0.55 | 0.58 | [60] |
| Aspergillus oryzae NRRL 3488 | – | Glucose | 30.27 | 0.64 | 0.67 | [65] |
| Aspergilus oryzae DSM 1863 | – | Syngas | 1.83 | 0.22 | 0.02 | [66] |
| Aspergillus niger ATCC 9142 | – | Glucose, glycerol, lactic acid | 17 | 0.80 | 0.09 | [67] |
| Aspergillus niger ATCC 10577 | – | Glucose, glycerol, lactic acid | 19 | – | 0.10 | [67] |
| Rhizopus delemar | – | Corn straw hydrolyte | 120 | 0.96 | 2.00 | [68] |
| Aspergillus oryzae NRRL 3488 | Erexpression of pyc, mdh and Spmae | Glucose | 154 | 1.03 | 0.94 | [69] |
| Saccharomyces cerevisiae | Expression of Afpyc, Ropyc, Afmdh, Romdh and Spmae1 | Glucose | 30.25 | 0.30 | 0.32 | [70] |
| Pichia pastoris | expression of pyc, mdh1 | Glucose | 8.50 | 0.14 | 0.18 | [71] |
| Thermobifida fusca muC-16 | Expression of Cgpyc | Cellulose | 62.76 | 0.63 | 0.53 | [72] |
青霉菌Penicillium spp.是第二大类苹果酸生产菌株,已被报道有多种菌株能够天然地积累苹果酸。例如,从海藻中分离得到的Penicillium viticola 152能够利用富含生物素的玉米浆作为氮源生产苹果酸;在优化条件下,该菌株可通过摇瓶发酵获得103 g/L的苹果酸,在10 L发酵罐中产量可达131 g/L,是以青霉菌发酵时获得的最高苹果酸产量[50]。Wang等也从海洋环境中筛选获得了菌核青霉Penicillium sclerotiorum K302,能够高水平地积累苹果酸;通过优化生产条件,摇瓶发酵的产量达到69 g/L;在10 L发酵罐中,72 h内苹果酸产量可达71.67 g/L,收率为0.69 g/g[51]。
一些其他的真核微生物也可以在代谢过程中产生苹果酸。例如,裂褶菌Schizophyllum commun IFO-4928可在27 ℃条件下,以50 g/L葡萄糖为底物生产18 g/L苹果酸,生产强度为0.18 g/(L·h)[52]。鲁氏酵母Zygosaccharomyces rouxii作为一种耐糖酵母,可在葡萄糖浓度30%,初始pH 5.0,培养温度25 ℃的优化条件下,15 d内从193 g/L葡萄糖中可以生产74.90 g/L苹果酸,收率为0.39 g/g。此外,谷氨酸作为苹果酸和琥珀酸的前体,对苹果酸的积累有明显的促进作用。研究发现,在培养基中添加0.5%谷氨酸时,苹果酸的产量显著增加[53]。
最近,Geiser等筛选出68株黑粉菌科菌株。其中,稗黑粉菌Ustilago trichophora TZ1具有从葡萄糖和甘油中合成苹果酸的能力[54]。一般来说,菌株发酵时葡萄糖作为底物的产率高于甘油,是生产苹果酸的首选底物。然而,U. trichophora TZ1却倾向利用甘油来生产苹果酸。基于这一特性,将适应性进化与发酵优化相结合,U. trichophora TZ1可以以甘油为底物生产196 g/L苹果酸,产量和生产强度分别为0.82 g/g和0.39 g/(L·h)[54]。
3.2 利用代谢工程菌株生产苹果酸 3.2.1 原核生物生产苹果酸的代谢工程改造大肠杆菌作为研究最久、遗传操作最为成熟的模式菌株,是代谢工程改造生产苹果酸的良好底盘(图 4A)。最为常见的代谢工程策略是删除或削弱竞争途径,以及表达或过度表达关键酶。例如,Moon等根据代谢通量分析预测最佳的苹果酸产生途径,发现PCK的过表达有利于苹果酸的积累,将来源于曼氏产琥珀酸菌Mannheimia succiniciproducens MBEL55E的磷酸烯醇式丙酮酸羧激酶PCK引入pta基因突变的大肠杆菌后,最终菌株E. coli W3110可在12.5 h内产生9.25 g/L的苹果酸,收率为0.42 g/g;由于副产物的积累在大多数情况下会影响有机酸的产量,尽管没有在发酵液中检测到副产物,研究人员依旧敲除了两个编码副产物合成的相关基因ldhA和adhE以试图增加苹果酸的积累;然而与设想相反的是,苹果酸的产量并没有得到进一步的提高,反而生产强度略有下降[55]。大肠杆菌的一大优势在于能够较为容易地进行多基因的敲除和表达以提高苹果酸的产量。例如,通过敲除6个中心发酵基因,E. coli KJ071能够在含有100 g/L葡萄糖的无机盐培养基中通过简单的分批发酵合成83.88 g/L的苹果酸[56]。研究人员进一步对富马酸还原酶合成相关的基因进行了敲除,重组菌株的琥珀酸产量降低了90%,从而促进了苹果酸的积累。与此同时,丙酮酸的积累增加,而乙酸的产量减少。因此,为了进一步减少副产物的产生,针对性地对丙酮酸积累相关的基因scfA和maeB进行了敲除,减少了副产物丙酮酸的积累。最终菌株E. coli XZ658在此基础上进一步地敲除了一系列基因(丙酮醛合酶mgsA、乳酸脱氢酶ldhA、丙酮酸激酶pkyA和pykF) 后通过分批发酵从50 g/L葡萄糖中获得了34 g/L苹果酸,且收率与生产强度分别为1.06 g/g和0.47 g(L·h)[57]。

|
| 图 4 四碳有机酸的代谢途径 Fig. 4 Metabolic pathways for the synthesis of tetracarbon organic acids in: (A) eukaryotes, (B) prokaryotes. |
| |
为了实现更简单高效的发酵模式,对E. coli W3110进行了基因改造和优化,通过一步合成途径生产苹果酸。首先,敲除ldhA、poxB、pflB、pta、ackA基因,以积累大量的丙酮酸。然后,依赖于NADP+的苹果酶和NADH激酶的过表达以及敲除与琥珀酸生物合成相关的基因使丙酮酸转化为苹果酸。最终在5 L生物反应器中进行发酵,苹果酸产量为21.65 g/L,产率为0.36 g/g,生产强度为0.30 g/(L·h)。副产物是16.54 g/L的丙酮酸,0.98 g/L的琥珀酸和0.19 g/L的富马酸[58]。同时,需要注意的是NADPH在苹果酸生产中的重要性,因为1 mol的L-苹果酸的生物合成需要1 mol的NADPH,因此NADPH的有效性对于提高目标产品的产量至关重要[59]。
乙醛酸途径也是苹果酸合成的一条可行路径。最近,Kim等报道了将体外模块化工程与体内CRISPRi技术相结合,对乙醛酸途径中的五酶级联反应进行多重调节生产苹果酸的研究[60]。CRISPRi技术可以最大限度地减少多个基因异源表达造成的代谢负担,以及解决不同模块之间的代谢通量失衡的问题,从而减少柠檬酸和α-酮戊二酸的积累。在优化的条件下,最终菌株在3.6 L补料分批发酵条件下苹果酸效价为36.05 g/L,收率为0.55 g/g[60]。
枯草芽孢杆菌Bacillus subtilis是一种严格需氧或兼性厌氧的杆菌,由于其成熟的遗传工具和发酵技术以及生物安全性,在发酵工业中也得到了广泛的应用。Mu等研究了富马酸酶对枯草芽孢杆菌生长和苹果酸生产的影响,首次实现了以枯草芽孢杆菌为底盘细胞生产苹果酸。通过将来源于大肠杆菌的PPC和来源于酿酒酵母的MDH导入枯草芽孢杆菌中,构建了异源的苹果酸生物合成途径,重组菌株能够生产0.81 g/L苹果酸。进一步地,通过敲除乳酸脱氢酶基因,苹果酸产量提高到了2.01 g/L,收率与生产强度分别为0.11 g/g和0.03 g/(L·h)[61]。
3.2.2 真核生物生产苹果酸的代谢工程改造米曲霉作为天然的苹果酸生产宿主,已经被开发并通过代谢工程的手段进一步地提高其生产能力。通过过量表达PYC、MDH和四碳二羧酸转运蛋白,优化苹果酸产生途径,改造后的米曲霉A. oryzae NRRL 3488在164 h内的苹果酸产量达到了154 g/L,且收率和生产强度分别为1.03 g/g和0.94 g/(L·h)。值得注意的是,尽管重组菌株中不存在柠檬酸积累的途径,发酵液中依然检测出了少量的柠檬酸。研究人员推测可能是细胞内存在的线粒体三羧酸转运蛋白介导的过程,导致积累的苹果酸被转化为柠檬酸并排出胞外。基于这样的假设,Liu等设计了一个草酰乙酸回补策略,减少苹果酸向柠檬酸的转化,通过强化氧化TCA循环以提高苹果酸的合成。磷酸烯醇式丙酮酸可以通过PCK或PPC的作用被转化为草酰乙酸,这被认为是固定CO2合成苹果酸的限速步骤。因此,研究人员首先通过异源表达来源于大肠杆菌的PCK和PPC,以及来源于粟酒裂殖酵母的四碳二羧酸转运蛋白基因SpMAE构建苹果酸合成途径。进一步的,由pfk基因编码的调节糖酵解途径的6-磷酸果糖激酶被认为是苹果酸合成的潜在限速步骤,并在强启动子SodM的作用下进一步过表达。最终得到的改良米曲霉的苹果酸浓度为165g/L,在3 L生物反应器中,苹果酸的收率和生产强度分别为0.68 g/g和1.38 g/(L·h)[62]。
除A. oryzae NRRL3488外,另一个由其衍生的野生菌株A. oryzae 2103a-68也被优化用于生产苹果酸。通过过量表达PYC、MDH和SpMAE,该菌株最终可以积累66.30 g/L苹果酸,收率为1.11 g/g,生产强度为0.86 g/(L·h)。然而除苹果酸外,该菌株还积累了其他副产物,如9.03 g/L的琥珀酸,这表明胞质苹果酸池的增加会使一部分通量转移到琥珀酸的积累[55]。
与丝状真菌相比,酿酒酵母在苹果酸生产中表现出一系列独特的优势,如:1) 对高底物浓度的耐受性强;2) 单细胞真菌可以更好地控制过程;3) 能够利用更广泛的碳源;4) 在食品工业中更安全等。因此,通过基因工程改造的酿酒酵母是极具潜力的苹果酸生产宿主(图 4B)。通过过量表达PYC、MDH3和SpMAE,重组酿酒酵母能够在摇瓶中生产59 g/L的苹果酸,收率为0.31 g/g,生产效率为0.19 g/(L·h)。通过生物反应器的进一步放大和优化,苹果酸的产率被提高到了0.36 g/g,比摇瓶提高了19%[20]。近年来,为了进一步提升酿酒酵母的苹果酸产量,结合途径优化和转运蛋白修饰的通用策略被应用于重建或强化酿酒酵母苹果酸生物合成途径和运输系统。研究发现,不同微生物来源的关键酶对苹果酸的产生有很大影响。通过对比不同来源的PYC和MDH,发现黄曲霉来源的PYC和米曲霉的MDH组合的苹果酸产量最高,进一步地过量表达转运蛋白SpMAE使重组菌株能够积累30.25 g/L的苹果酸,收率和生产强度分别为0.30 g/g和0.32 g/(L·h)[63]。
与酿酒酵母相比,巴斯德毕赤酵母因其高效的异源基因表达能力而具有更好的生物转化特性。此外,巴斯德毕赤酵母的优势在于甲醇异化途径可以产生两个NADH分子,能够为苹果酸的生产提供充足的NADH。Zhang等分别为内源的PYC,胞质MDH和FUM构建了3个表达框,并整合到P. pastoris GS115的基因组中以获得稳定的表达,由此构建的重组菌株能够在96 h内积累42.28 g/L的苹果酸[64]。
除了酿酒酵母和毕赤酵母,光滑球拟酵母由于具有耐受性相对较强等优势也被开发用于苹果酸的生产。光滑球拟酵母Torulopsis glabrata CCTCC M202019是一株产丙酮酸的营养缺陷型酵母,通过调节其从丙酮酸到苹果酸的碳通量已被成功地用于苹果酸的生产。在通过基因组模型确定苹果酸生产的瓶颈后,来源于米根霉的PYC和MDH,以及SpMAE被整合到菌株的基因组上。在最佳维生素浓度下,60 h内苹果酸的最终产量为8.5 g/L。值得注意的是,苹果酸对葡萄糖的收率仅为0.14 g/g,远低于途径的理论产率以及以酿酒酵母为生产菌株时的实际收率(0.30 g/g)。这说明在苹果酸生物合成途径中存在动力学瓶颈,丙酮酸的通量不能有效地转化为苹果酸[73]。
4 琥珀酸的生物生产琥珀酸是一种常见的天然有机酸,广泛存在于人体、动物、植物和微生物中,在工业上有着广泛的应用[74]。在工业上,它可以作为中间体用于生产多种工业化学品,如己酸、1, 4-丁二醇、四氢呋喃、n-甲基吡咯烷酮、2-吡咯烷酮和丁内酯[75-76]。在农业领域,琥珀酸是一种常用的生长调节剂,可用于种子处理和植物生根。在食品工业中,琥珀酸可作为饮料中的调味增强剂、面包中的软化剂和食品调味品制备的催化剂[77]。在医药领域,琥珀酸可以用于生产磺胺药、维生素A、维生素B等抗痉挛剂、利尿剂和止血药物,还可用作制备润滑剂和表面活性剂。此外,琥珀酸也可以作为合成可生物降解材料的平台化合物。由于琥珀酸在食品、化工、医药等领域的广泛应用,它被认为是继柠檬酸之后最有前途的有机酸。广泛的用途使近年来琥珀酸的需求量日益增加,根据对欧洲和北美建造的制造设施的研究,生物基琥珀酸的年产量占整个市场的49%。2020年生物基琥珀酸市场将达到60万t。目前已经开发了多种生产琥珀酸的化学路线,包括石蜡氧化、催化加氢和马来酸或马来酸酐的电解还原[78]等。石蜡氧化法是生产琥珀酸的原始方法,采用钙或锰作为催化剂,将石蜡完全转化为二羧酸的混合物,随后,通过蒸馏、结晶和干燥来提纯琥珀酸。虽然该工艺已经建立,但通过该途径获得的琥珀酸收率和纯度相对较低,且会造成环境的污染[79]。与石化法合成琥珀酸相比,生物技术生产具有转化效率高、成本低、底物可再生等优点[80]。最近,由于石化燃料的消耗和对环境问题的关切,使用可再生原料生产琥珀酸日益引起研究人员的关注。
4.1 利用野生型菌株生产琥珀酸迄今为止,已经筛选和鉴定了多种具有琥珀酸生产能力的微生物,细菌以琥珀酸放线杆菌Actinobacillus succinogenes、产琥珀酸厌氧螺菌Anaerobiospirillum succiniciproducens、产琥珀酸曼海姆菌M. succiniciproducens、谷氨酸棒杆菌Corynebacterium glutamicum、大肠杆菌E. coli为代表;真菌则主要包括酿酒酵母Saccharomyces cerevisiae、解脂耶氏酵母Yarrowia lipolytica以及一些青霉属的菌株(表 3)[81]。
| Strain | Description | Substrate | Titer (g/L) | Yield (g/g) | Productivity (g/(L·h)) | References |
| A. succinogenes FZ53 | – | Glucose | 105.8 | 0.82 | 1.36 | [85] |
| A. succinogenes 130Z | – | Xylose | 29.0 | 0.70 | 3.40 | [86] |
| M. succiniciproducens MBEL55E | – | Glucose | – | – | 3.90 | [87] |
| A. succinogenes 130Z | Expression of mdh | Glucose, xylose | 27.5 | 0.19 | 0.23 | [94] |
| A. succinogenes CGMCC1593 | – | Glucose | 60.2 | 0.75 | 1.30 | [112] |
| A. succiniciproducens ATCC53488 | – | Glucose | 32.20 | 0.90 | 1.19 | [113] |
| M. succiniciproducens LPK7 | Deletion of ldhA, pflB and pta-ackA | Glucose | 52.40 | 0.76 | 1.80 | [114] |
| M. succiniciproducens PALFK | Deletion of ldhA, fruA and pta-ackA | Sucrose, glycerol | 78.40 | 1.07 | 6.02 | [115] |
| Y. lipolytica PGC01003 | Deletion of sdh5 | Glycerol | 51.9 | 0.42 | 1.46 | [116] |
| Y. lipolytica Y -3314 | Expression of pck, scs2; deletion of ach | Glycerol | 110.7 | 0.53 | 0.80 | [117] |
琥珀酸放线杆菌和产琥珀酸厌氧螺菌是最早被鉴定得到的天然琥珀酸生产菌株,它们分别是从牛瘤胃和粪便中分离得到的[82]。琥珀酸放线杆菌是革兰氏阴性兼性厌氧菌,由于其生产能力高以及不需要曝气的优点,是最有前途的琥珀酸生产菌株之一[83]。此外,琥珀酸放线杆菌可以利用广泛的五碳糖(如木糖、阿拉伯糖、甘露糖)、以葡萄糖为代表的六碳糖以及各种双糖和其他碳源(包括半乳糖、甘露醇、淀粉、烘焙废物、甘蔗糖蜜、甘油) 和农业残留物来合成琥珀酸[84]。例如,以酵母提取物和玉米浆为氮源时,A. succinogenes FZ53突变体能够利用葡萄糖生产105.8 g/L的琥珀酸,收率为0.82 g/g,生产强度1.36 g/(L·h)[85]。以木糖作为底物时,A. succinogenes 130Z能够在8.5 h的发酵过程中积累29.0 g/L的琥珀酸[86]。然而,尽管琥珀酸放线杆菌在高效生产琥珀酸方面具有上述这些优势,在其发酵过程中的副产物积累、产物抑制以及对氨基酸(如谷氨酸盐和甲硫氨酸) 和维生素(如烟酸和硫胺素) 的苛求等缺点影响了其工业化的价值。产琥珀酸厌氧螺菌同样可以利用多种底物作为碳源。在以葡萄糖为唯一碳源的条件下,A. succiniciproducens ATCC 53488能够积累32.2 g/L琥珀酸,而以半乳糖作为碳源同样可以积累15.3 g/L的琥珀酸。但琥珀酸厌氧螺菌的主要问题是副产物积累较多,产品分离成本较高。为了去除发酵液中的最终产物,采用了耦合单极电渗析以提高菌体生长、琥珀酸产量和生产强度。最终得到的琥珀酸生产强度为10.40 g/(L·h),最终浓度为83.0 g/L,产率为0.89 g/g[81]。琥珀酸放线杆菌作为一种野生型产琥珀酸菌株,相比于代谢工程菌株,有广泛的碳源利用能力,同时具有较强的底物耐受性等优势,是目前公认的最具潜力的产琥珀酸优势菌株之一。
产琥珀酸曼海姆菌Mannheimia succiniciproducens也是一种天然的琥珀酸生产菌株,是兼性厌氧的革兰氏阴性菌。它同样是从牛瘤胃中分离出来的,具有固定CO2的能力,并生产包含高浓度的琥珀酸以及甲酸、乙酸和乳酸等在内的混合酸。Mannheimia succiniciproducens MBEL55E是氨基酸和维生素的营养缺陷型,在厌氧条件下以玉米浆作为底物,最高获得了3.90 g/(L·h) 的生产强度[87]。该菌株还能有效地从甘油、木糖、蔗糖、果糖、麦芽糖、乳糖、甘露醇和阿拉伯糖醇中生产琥珀酸。在这些底物中,甘露醇是一种更具有还原性的碳源,可以转化为额外的产物乙醇,以平衡细胞的还原力。
谷氨酸棒状杆菌是一种生长快速的革兰氏阳性菌,也可以利用广泛的碳源。在厌氧条件下,谷氨酸棒状杆菌能够在添加碳酸氢盐和生物素的培养基上生产琥珀酸、乙酸和乳酸。Okino等利用谷氨酸棒状杆菌发酵获得了23.0 g/L的琥珀酸,收率为0.19 g/g,生产强度为3.83 g/(L·h)[88]。对于谷氨酸棒状杆菌,深入的基因组分析和成熟的基因操作工具正致力于开发更高效的菌株和更新颖的生物处理方法,这使其成为极具潜力的琥珀酸生产宿主。
4.2 利用代谢工程菌株生产琥珀酸 4.2.1 原核生物生产琥珀酸的代谢工程改造在原核生物中,磷酸烯醇式丙酮酸一方面可以通过三羧酸循环产生琥珀酸,另一方面也可以转化为丙酮酸,进而导致副产物的生成。因此,抑制丙酮酸的积累是减少副产物生成和PEP消耗的有效策略。为了提高琥珀酸的产量,首先要减少副产物生成,一个常见的策略是通过敲除编码磷酸转移酶系统中葡萄糖特异性渗透酶蛋白的ptsG基因来抑制丙酮酸的合成进而提高琥珀酸产量[89]。另一方面,也可以通过敲除编码乳酸脱氢酶、醋酸激酶和醇脱氢酶的基因ldhA、ackA和adhE/C等来减少副产物乳酸、乙酸和乙醇的积累[90-91]。
此外,过去的代谢通量分析结果显示,从磷酸烯醇式丙酮酸到琥珀酸的主要途径包含一系列基因,如ppc、pck、pyc、mdh、fum、frd,对这些基因的过量表达均已被证明能够强化琥珀酸的合成。为了增加磷酸烯醇式丙酮酸到琥珀酸的转化效率,研究人员采取了一系列方法。例如,过量表达大肠杆菌ppc基因可以使琥珀酸产量从3.0 g/L增加到10.7 g/L,且收率从0.08 g/g增加到0.29 g/g。除此之外,在构建的大肠杆菌菌株中过量表达琥珀酸放线杆菌来源的pck可以使琥珀酸产量增加6.5倍[92]。
为了清楚地阐明琥珀酸放线杆菌的代谢机制,近年来研究人员陆续采用了13C标记的代谢通量分析、中心代谢模型、主要生物量组分模型等手段进行了分析。在此基础上,已经提出了若干高效的表达方法如pLGZ920质粒,并建立了电穿孔基因敲除的手段。例如,在A. succinogenes 130Z的基础上敲除丙酮酸甲酸裂解酶(PFL) 和甲酸脱氢酶(FDH),同时提供外部电子供体如中性红或H2,重组菌株能够在没有副产物的情况下生产92.1 g/L的琥珀酸[93]。
rTCA循环是琥珀酸产生的主要途径,因此,在模型预测的基础上,研究人员过量表达了琥珀酸放线杆菌中参与rTCA循环的基因,并敲除了副产物的形成途径。然而,与野生型菌株(30.6 g/L) 相比,最终的重组菌株表现出更低的琥珀酸积累(27.5 g/L),生长速率与葡萄糖消耗速率也均有所下降[94]。总而言之,尽管对琥珀酸放线杆菌的代谢模型和基因操作工具正在不断地完善,但是不可否认的是这些工作尚处于初级阶段,有待进一步地开发和完善。
由于清晰的遗传原理、简单的基因操作、高生长速率和最少的营养需求,大肠杆菌已引起研究人员的广泛关注并进行了深入研究。为了解决野生型大肠杆菌在厌氧条件下除了合成琥珀酸之外还会合成大量的乳酸、甲酸、乙酸和乙醇等副产物的问题[95],研究人员在一株ptsG突变大肠菌株中敲除副产物甲酸和乳酸积累相关的基因pflB和ldhA,构建了一株琥珀酸高产菌株E. coli AFP111,在双相发酵条件下,琥珀酸的收率为1.07 g/g,生产强度为1.89 g/(L·h)[96]。然而,仅仅通过阻断副产物合成途径并不足以高效生产琥珀酸。因此,在敲除ptsG、pflB和ldhA的基础上,进一步的过量表达ppc基因,重组菌株能够在75 h内获得产量为116.2 g/L的琥珀酸[97]。另一方面,研究发现,ldhA和pflB基因的敲除会阻碍NAD+的再生,影响菌株在厌氧条件下在葡萄糖上生产的能力。为了实现细胞内的氧化还原平衡,提出了一种改进的两阶段发酵策略。首先在好氧阶段以乙酸作为碳源诱导糖异生、rTCA途径和乙醛酸分流中具有氧化还原调节功能的基因;然后在第二阶段以葡萄糖作为碳源进行厌氧发酵,最终得到了60 g/L的琥珀酸,且生产强度为1.00 g/(L·h)[98]。另一方面,通过优化乙酸的同化途径及ATP的有氧供给,也报道了以乙酸作为碳源直接生产3.6 g/L琥珀酸,且实际收率达到了理论最大收率[99-100]。近几十年来,随着合成生物学的迅速发展,基因表达调控已经在许多微生物中得到了应用,有多种方法被广泛应用于设计和优化生物系统。例如,在E. coli JW1021中,一种新的核糖体调节开关已经被成功地应用于调控基因表达和代谢通量。通过这种特殊设计的核糖体调节开关,几个关键基因的表达能够根据菌株的生长阶段而自发地开启和关闭。引入这一核糖体调节开关的菌株最终能够积累114.0 g/L的琥珀酸,且收率与生产强度分别为0.91 g/g和3.25 g/(L·h),相比于原始菌株均有明显的提升。Zhang等则通过表达一个全局调节因子IrrE提高了菌株对高盐胁迫的耐受性,从而能够从海水中直接生产24.5 g/L的琥珀酸[101]。最近有研究发现,在大肠杆菌内引入甲醇代谢模块,大肠杆菌厌氧代谢合成丁二酸的产量会有所增加,但甲醇的利用能力仍然偏低。为提高甲醇的利用效率,开展了甲醇异化途径敲除,甲醇同化代谢前体核酮糖-5-磷酸再生途径增强等研究内容。通过过量表达rpe,tkt,强化核酮糖-5-磷酸再生,增加甲醛的同化代谢。发酵结果显示,重组菌株Suc260/optpTrc99a-msi-rpe-tkt的生长速率比原始菌株提高了7.1%,甲醇的消耗速率比Suc460增加了2.24 mmol/L;此外,琥珀酸的产量和收率分别比Suc460提高了3.9%、3.4%。
谷氨酸假单胞菌不仅能够天然地积累琥珀酸,较为成熟的基因改造工具也被用于进一步提高琥珀酸产量或减少副产物的积累。例如,通过敲除乳酸产生途径的几个基因(pqo、cat、ackA-PTA) 和乙酸产生途径(ldhA),成功地消除了乳酸和乙酸两种副产物的积累。进一步地表达pyc、fdh和gapA基因,加强PEP到琥珀酸的代谢途径之后,最终的工程菌株能够生产133.8 g/L琥珀酸,且收率可达1.09 g/g[102]。另一方面,为了增强糖酵解的速率和NADH的供应,Kim等构建了一株H+-ATP酶点突变菌株C. glutamicum NC-3-1,能够在5 L生物反应器中生产113.0 g/L琥珀酸,相比于原始菌提高39%[103]。
除了上述两种常见的底盘细胞,近年来藻类也被开发作为“细胞工厂”用于固定二氧化碳并生产常见的化学品如琥珀酸。例如,在嗜热蓝藻中,通过过量表达琥珀酸合成途径相关的基因ppc、gltA、kgd、gabD和sdhAB以及敲除glgc,将琥珀酸的产量从0.44 mg/L提高到0.43 g/L。在此基础上,给予适宜的环境条件,将温度从30 ℃升高到37 ℃,以及补充NaHCO3,可进一步将产量提高到1.8 g/L[104]。
4.2.2 真核生物生产琥珀酸的代谢工程改造虽然细菌利用葡萄糖可以获得高产量的琥珀酸,但潜在的致病性、低渗透压和低pH耐受性等原因限制了它们的应用[5]。因此,一部分研究人员将注意力转移到了开发能够在低pH条件下生产琥珀酸的基因工程酵母上。例如,酿酒酵母和解脂耶氏酵母可以在很宽的pH范围内代谢生产琥珀酸,这能够显著降低中下游分离纯化过程的成本。
酿酒酵母具有明确的遗传背景,研究人员设计了多种代谢工程策略来提高酿酒酵母的琥珀酸产量。以酿酒酵母作为底盘细胞生产琥珀酸的一大问题在于发酵过程中会产生大量的副产物乙醇。为了减少乙醇的产生,常用的策略是敲除编码乙醇脱氢酶的基因adh和丙酮酸脱羧酶基因。例如,在酿酒酵母中敲除了pdc1、pdc5和pdc6消除了乙醇的积累,进一步地敲除其余副产物相关基因(his3、fum1和gpd1) 以及引入胞质rTCA途径,在pH为3.8的环境下生产13.0 g/L琥珀酸,收率为0.13 g/L[105]。这一结果表明,乙醇生产途径的敲除对琥珀酸合成有积极影响。另一方面,一旦琥珀酸的生成量增加,乙酸的积累量就会相应地降低,这表明这两种酸地产生途径之间存在竞争机制[106]。基于计算机的分析方法为提高琥珀酸产量提供指导,通过分析线粒体和胞浆之间的交换通量,研究人员用13C标记的方法发现胞外琥珀酸会通过细胞膜重新进入线粒体[107]。基于这个发现,Agren等敲除了相关的线粒体转运蛋白DIC1,实现了琥珀酸产量的提升[108]。尽管有上述这些进展,总的来说,以酿酒酵母作为底盘生产琥珀酸的产量,收率和生产强度相比于其他琥珀酸生产菌株依旧较低。
库德毕赤酵母Pichia kudriavzevii能够在高浓度琥珀酸和低pH条件下生长良好,是生产琥珀酸的潜在底盘。为了提高琥珀酸的产量,Zhu等敲除了编码乳酸细胞色素C氧化还原酶的Cyb2a,并引入了来源于克鲁维假丝酵母Candida krusei的pyc1和fum1,S. cerevisiae的frd1,Zygoaccharomyces rouxii的mdh基因,和布氏锥虫Trypanosoma brucei的frd1基因。最终菌株P. kudriavzevii 13171能够生产23.0 g/L的琥珀酸。另一株菌株P. kudriavzevii 13723通过敲除ura和pdc基因,以及过量表达pyc1、fum、mae、frd和mdh基因,可以产生48.2 g/L的琥珀酸[109]。
解脂耶氏酵母也是生产琥珀酸的潜在菌株。例如,通过敲除基因sdh1、sdh2和suc2,重组菌株能够以甘油为碳源生产45.4 g/L的琥珀酸[110]。Cui等通过敲除编码辅酶A-转移酶的基因Ach,以及过量表达来源于酿酒酵母的pck和内源的琥珀酰辅酶A合成酶基因scs2,重组菌株无需控制pH,能够从200 g/L甘油中生产110.7 g/L琥珀酸[111]。最近,Li等开发了一个原位纤维床生物反应器,在120 g/L初始甘油和3 L/min的通气率条件下,通过分批补料策略获得了198.2 g/L琥珀酸,这是目前获得的最高产量。值得一提的是,通过稳定性研究发现,琥珀酸的生产强度在460 h,12个重复的循环内没有显示出明显的下降,并且在故意中断后的两个重复批次中甚至恢复了细胞活力[51]。这些研究证明了解脂耶氏酵母在琥珀酸生产上的潜力。
5 总结与展望迄今为止,已经开发出了多种发酵策略用于发酵法生产四碳有机酸。尽管通过随机诱变、定向进化和代谢工程改造等手段已经在提高和优化四碳有机酸合成效率的方面取得了显著的成果,利用发酵法生产这类产品的经济竞争力仍然低于化学合成。因此,进一步降低发酵生产的成本对于产业化十分重要,可以通过低成本原料的利用、二氧化碳的直接利用、微生物共培养和工艺优化等手段。为了提升发酵法生产四碳有机酸的竞争力,首先需要筛选天然的四碳有机酸高产菌株,野生型菌株营养需求量相对较低,生产条件相对简单,野生型菌株的筛选及诱导是代谢工程菌株生产四碳有机酸的基础。由于能够利用二氧化碳和阳光作为唯一的碳源和能源,蓝藻在降低生产成本方面显示出巨大的潜力,作为生产四碳有机酸的底盘而受到越来越多的关注。另外,微生物共培养体系以其广泛的底物利用率和对复杂环境的稳健性,在生物制品生产中显示出巨大的潜力。例如,木质纤维素降解菌和琥珀酸产生菌的组合可以实现木质纤维素到琥珀酸的直接转化。与通过代谢工程同时构建底物降解和产物合成途径相比,这种劳动分工、协同互作的方式显得更为方便经济,尤其是对于一些非模式微生物。另一方面,尽管在本文中并没有涉及讨论,但是由于四碳有机酸生产的很大一部分成本在于下游产品回收,因此发酵过程的设计和操作是否有利于下游工艺也应予以考虑。最后,生物基四碳有机酸的生产应注意与现有的市场需求相结合。例如,在当前全国范围内大力推动垃圾分类的背景下,丁二酸作为生产可生物降解塑料聚丁二酸丁二醇酯(PBS) 的前体,其市场需求有望进一步扩大。
总之,随着生物合成技术的不断探索和发展,生物发酵法在四碳有机酸生产的领域必定会扮演一个越来越重要的角色,最终成为一条环境友好且具有经济竞争力的四碳有机酸合成路线。
| [1] |
Becker J, Lange A, Fabarius J, et al. Top value platform chemicals: bio-based production of organic acids. Curr Opin Biotechnol, 2015, 36: 168-175. DOI:10.1016/j.copbio.2015.08.022
|
| [2] |
Sauer M, Porro D, Mattanovich D, et al. Microbial production of organic acids: expanding the markets. Trends Biotechnol, 2008, 26(2): 100-108. DOI:10.1016/j.tibtech.2007.11.006
|
| [3] |
Alonso S, Rendueles M, Díaz M. Microbial production of specialty organic acids from renewable and waste materials. Crit Rev Biotechnol, 2015, 35(4): 497-513. DOI:10.3109/07388551.2014.904269
|
| [4] |
Bailey JE. Toward a science of metabolic engineering. Science, 1991, 252(5013): 1668-1675. DOI:10.1126/science.2047876
|
| [5] |
Hong SH, Kim JS, Lee SY, et al. The genome sequence of the capnophilic rumen bacterium Mannheimia succiniciproducens. Nat Biotechnol, 2004, 22(10): 1275-1281. DOI:10.1038/nbt1010
|
| [6] |
Moon SY, Hong SH, Kim TY, et al. Metabolic engineering of Escherichia coli for the production of malic acid. Biochem Eng J, 2008, 40(2): 312-320. DOI:10.1016/j.bej.2008.01.001
|
| [7] |
Zelle RM, De Hulster E, Van Winden WA, et al. Malic acid production by Saccharomyces cerevisiae: engineering of pyruvate carboxylation, oxaloacetate reduction, and malate export. Appl Environ Microbiol, 2008, 74(9): 2766-2777. DOI:10.1128/AEM.02591-07
|
| [8] |
Mu L, Wen JP. Engineered Bacillus subtilis 168 produces L-malate by heterologous biosynthesis pathway construction and lactate dehydrogenase deletion. World J Microbiol Biotechnol, 2013, 29(1): 33-41. DOI:10.1007/s11274-012-1155-6
|
| [9] |
Chen XL, Dong XX, Wang YC, et al. Mitochondrial engineering of the TCA cycle for fumarate production. Metab Eng, 2015, 31: 62-73. DOI:10.1016/j.ymben.2015.02.002
|
| [10] |
Kim SK, Han GH, Seong W, et al. CRISPR interference-guided balancing of a biosynthetic mevalonate pathway increases terpenoid production. Metab Eng, 2016, 38: 228-240. DOI:10.1016/j.ymben.2016.08.006
|
| [11] |
Gu SY, Li JG, Chen BC, et al. Metabolic engineering of the thermophilic filamentous fungus Myceliophthora thermophila to produce fumaric acid. Biotechnol Biofuels, 2018, 11.
|
| [12] |
Liu X, Gallay C, Kjos M, et al. High-throughput CRISPRi phenotyping identifies new essential genes in Streptococcus pneumoniae. Mol Syst Bio, 2017, 13(5): 931. DOI:10.15252/msb.20167449
|
| [13] |
Song H, Lee SY. Production of succinic acid by bacterial fermentation. Enzyme Microb Technol, 2006, 39(3): 352-361. DOI:10.1016/j.enzmictec.2005.11.043
|
| [14] |
Xu Q, Li S, Huang H, et al. Key technologies for the industrial production of fumaric acid by fermentation. Biotechnol Adv, 2012, 30(6): 1685-1696. DOI:10.1016/j.biotechadv.2012.08.007
|
| [15] |
Liu H, Zhao S, Jin Y, et al. Production of fumaric acid by immobilized Rhizopus arrhizus RH 7-13-9# on loofah fiber in a stirred-tank reactor. Bioresour Technol, 2017, 244(pt 1): 929-933.
|
| [16] |
Zhang T, Wang ZN, Deng L, et al. Pull-in urea cycle for the production of fumaric acid in Escherichia coli. Appl Microbiol Biot, 2015, 99(12): 5033-5044. DOI:10.1007/s00253-015-6556-7
|
| [17] |
Regev-Rudzki N, Battat E, Goldberg I, et al. Dual localization of fumarase is dependent on the integrity of the glyoxylate shunt. Mol Microbiol, 2009, 72(2): 297-306. DOI:10.1111/j.1365-2958.2009.06659.x
|
| [18] |
Li N, Zhang B, Wang Z, et al. Engineering Escherichia coli for fumaric acid production from glycerol. Bioresour Technol, 2014, 174: 81-87. DOI:10.1016/j.biortech.2014.09.147
|
| [19] |
Yu S, Huang D, Wen J, et al. Metabolic profiling of a Rhizopus oryzae fumaric acid production mutant generated by femtosecond laser irradiation. Bioresour Technol, 2012, 114: 610-615. DOI:10.1016/j.biortech.2012.03.087
|
| [20] |
Chen X, Wu J, Song W, et al. Fumaric acid production by Torulopsis glabrata: engineering the urea cycle and the purine nucleotide cycle. Biotechnol Bioeng, 2015, 112(1): 156-167. DOI:10.1002/bit.25334
|
| [21] |
Zelle RM, De Hulster E, Van Winden WA, et al. Malic acid production by Saccharomyces cerevisiae: engineering of pyruvate carboxylation, oxaloacetate reduction, and malate export. Appl Environ Microb, 2008, 74(9): 2766-2777. DOI:10.1128/AEM.02591-07
|
| [22] |
Roa Engel CA, Straathof AJ, Zijlmans TW, et al. Fumaric acid production by fermentation. Appl Microbiol Biotechnol, 2008, 78(3): 379-389. DOI:10.1007/s00253-007-1341-x
|
| [23] |
Koutinas AA, Vlysidis A, Pleissner D, et al. Valorization of industrial waste and by-product streams via fermentation for the production of chemicals and biopolymers. Chem Soc Rev, 2014, 43(8): 2587-2627. DOI:10.1039/c3cs60293a
|
| [24] |
McGinn SM, Beauchemin KA, Coates T, et al. Methane emissions from beef cattle: effects of monensin, sunflower oil, enzymes, yeast, and fumaric acid. J Anim Sci, 2004, 82(11): 3346-3356. DOI:10.2527/2004.82113346x
|
| [25] |
李学坤, 张昆, 高振, 等. 富马酸的合成及应用. 现代化工, 2005, 25: 81-85. Lee XK, Zhang K, Gao Z, et al. Synthesis and application of fumaric acid. Modern Chemical Industry, 2005, 25: 81-85 (in Chinese). DOI:10.3321/j.issn:0253-4320.2005.z1.021 |
| [26] |
Xu Q, He SL, Jiang L, et al. Extractive fermentation for fumaric acid production by Rhizopus oryzae. Sep Sci Technol, 2017, 52(9): 1512-1520.
|
| [27] |
Lee SY, Kim HU, Chae TU, et al. A comprehensive metabolic map for production of bio-based chemicals. Nat Catal, 2019, 2(1): 18-33. DOI:10.1038/s41929-018-0212-4
|
| [28] |
Cao N, Du J, Gong CS, et al. Simultaneous production and recovery of fumaric acid from immobilized Rhizopus oryzae with a rotary biofilm contactor and an adsorption column. Appl Environ Microbiol, 1996, 62(8): 2926-2931. DOI:10.1128/AEM.62.8.2926-2931.1996
|
| [29] |
Huang L, Wei PL, Zang R, et al. High-throughput screening of high-yield colonies of Rhizopus oryzae for enhanced production of fumaric acid. Ann Microbiol, 2010, 60(2): 287-292. DOI:10.1007/s13213-010-0039-y
|
| [30] |
Fu YQ, Xu Q, Li S, et al. Strain improvement of Rhizopus oryzae for over-production of fumaric acid by reducing ethanol synthesis pathway. Korean J Chem Eng, 2010, 27(1): 183-186. DOI:10.1007/s11814-009-0323-3
|
| [31] |
Deng YF, Li S, Xu Q, et al. Production of fumaric acid by simultaneous saccharification and fermentation of starchy materials with 2-deoxyglucose-resistant mutant strains of Rhizopus oryzae. Bioresour Technol, 2012, 107: 363-367. DOI:10.1016/j.biortech.2011.11.117
|
| [32] |
Wen S, Liu L, Nie KL, et al. Enhanced fumaric acid production by fermentation of xylose using a modified strain of Rhizopus arrhizus. BioResources, 2013, 8(2): 2186-2194. DOI:10.15376/biores.8.2.2186-2194
|
| [33] |
Moon SY, Hong SH, Kim TY, et al. Metabolic engineering of Escherichia coli for the production of malic acid. Biochem Eng J, 2008, 40(2): 312-320. DOI:10.1016/j.bej.2008.01.001
|
| [34] |
Zhang T, Wang ZN, Deng L, et al. Pull-in urea cycle for the production of fumaric acid in Escherichia coli. Appl Microbiol Biotechnol, 2015, 99(12): 5033-5044. DOI:10.1007/s00253-015-6556-7
|
| [35] |
Liu H, Song R, Liang Y, et al. Genetic manipulation of Escherichia coli central carbon metabolism for efficient production of fumaric acid. Bioresour Technol, 2018, 270: 96-102. DOI:10.1016/j.biortech.2018.08.024
|
| [36] |
Yadav VG, De Mey M, Giaw Lim C, et al. The future of metabolic engineering and synthetic biology: towards a systematic practice. Metab Eng, 2012, 14(3): 233-241. DOI:10.1016/j.ymben.2012.02.001
|
| [37] |
Pines O, Even-Ram S, Elnathan N, et al. The cytosolic pathway of L-malic acid synthesis in Saccharomyces cerevisiae: the role of fumarase. Appl Microbiol Biotechnol, 1996, 46(4): 393-399.
|
| [38] |
Xu G, Liu L, Chen J. Reconstruction of cytosolic fumaric acid biosynthetic pathways in Saccharomyces cerevisiae. Microb Cell Fact, 2012, 11: 24. DOI:10.1186/1475-2859-11-24
|
| [39] |
Chen X L, Li Y, Tong T, et al. Spatial modulation and cofactor engineering of key pathway enzymes for fumarate production in Candida glabrata. Biotechnol Bioeng, 2019, 116(3): 622-630. DOI:10.1002/bit.26906
|
| [40] |
Zhang B, Skory CD, Yang ST. Metabolic engineering of Rhizopus oryzae: effects of overexpressing pyc and pepc genes on fumaric acid biosynthesis from glucose. Metab Eng, 2012, 14(5): 512-520. DOI:10.1016/j.ymben.2012.07.001
|
| [41] |
Gu S, Li J, Chen B, et al. Metabolic engineering of the thermophilic filamentous fungus Myceliophthora thermophila to produce fumaric acid. Biotechnol Biofuels, 2018, 11: 323. DOI:10.1186/s13068-018-1319-1
|
| [42] |
Kang SW, Lee H, Kim D, et al. Strain development and medium optimization for fumaric acid production. Biotechnol Bioprocess Eng, 2010, 15(5): 761-769. DOI:10.1007/s12257-010-0081-4
|
| [43] |
Zhang BH, Yang ST. Metabolic engineering of Rhizopus oryzae: effects of overexpressing fumR gene on cell growth and fumaric acid biosynthesis from glucose. Process Biochem, 2012, 47(12): 2159-2165. DOI:10.1016/j.procbio.2012.08.009
|
| [44] |
Wei L, Liu J, Qi H, et al. Engineering Scheffersomyces stipitis for fumaric acid production from xylose. Bioresour Technol, 2015, 187: 246-254. DOI:10.1016/j.biortech.2015.03.122
|
| [45] |
徐晴, 马小琛, 付永前, 等. 根霉菌利用木质纤维素发酵生产有机酸的研究进展. 中国生物工程杂志, 2009, 29(9): 114-118. Xu Q, Ma XS, Fu YQ, et al. Research progress on Rhizopus fermentative production of organic acids from lignocellulose. China Biotechnol, 2009, 29(9): 114-118 (in Chinese). |
| [46] |
Liu G, Zhou Y, Luo H, et al. A comparative evaluation of different types of microbial electrolysis desalination cells for malic acid production. Bioresour Technol, 2015, 198: 87-93. DOI:10.1016/j.biortech.2015.08.149
|
| [47] |
Giorno L, Drioli E, Carvoli G, et al. Study of an enzyme membrane reactor with immobilized fumarase for production of L-malic acid. Biotechnol Bioeng, 2001, 72(1): 77-84. DOI:10.1002/1097-0290(20010105)72:1<77::AID-BIT11>3.0.CO;2-L
|
| [48] |
Goldberg I, Rokem JS, Pines O. Organic acids: old metabolites, new themes. J Chem Technol Biotechnol, 2006, 81(10): 1601-1611. DOI:10.1002/jctb.1590
|
| [49] |
Knuf C, Nookaew I, Remmers I, et al. Physiological characterization of the high malic acid-producing Aspergillus oryzae strain 2103a-68. Appl Microbiol Biotechnol, 2014, 98(8): 3517-3527. DOI:10.1007/s00253-013-5465-x
|
| [50] |
Khan I, Nazir K, Wang ZP, et al. Calcium malate overproduction by Penicillium viticola 152 using the medium containing corn steep liquor. Appl Microbiol Biotechnol, 2014, 98(4): 1539-1546. DOI:10.1007/s00253-013-5326-7
|
| [51] |
Wang ZP, Wang GY, Khan I, et al. High-level production of calcium malate from glucose by Penicillium sclerotiorum K302. Bioresour Technol, 2013, 143: 674-677. DOI:10.1016/j.biortech.2013.06.051
|
| [52] |
Kawagoe M, Hyakumura K, Suye SI, et al. Application of bubble column fermentors to submerged culture of Schizophyllum commune for production of L-malic acid. J Ferment Bioeng, 1997, 84(4): 333-336. DOI:10.1016/S0922-338X(97)89254-9
|
| [53] |
Taing O, Taing K. Production of malic and succinic acids by sugar-tolerant yeast Zygosaccharomyces rouxii. Eur Food Res Technol, 2007, 224(3): 343-347.
|
| [54] |
Zambanini T, Sarikaya E, Kleineberg W, et al. Efficient malic acid production from glycerol with Ustilago trichophora TZ1. Biotechnol Biofuels, 2016, 9: 67. DOI:10.1186/s13068-016-0483-4
|
| [55] |
Moon SY, Hong SH, Kim TY, et al. Metabolic engineering of Escherichia coli for the production of malic acid. Biochem Eng J, 2008, 40(2): 312-320. DOI:10.1016/j.bej.2008.01.001
|
| [56] |
Jantama K, Haupt MJ, Svoronos SA, et al. Combining metabolic engineering and metabolic evolution to develop nonrecombinant strains of Escherichia coli C that produce succinate and malate. Biotechnol Bioeng, 2008, 99(5): 1140-1153. DOI:10.1002/bit.21694
|
| [57] |
Zhang X, Wang X, Shanmugam KT, et al. L-malate production by metabolically engineered Escherichia coli. Appl Environ Microbiol, 2011, 77(2): 427-434. DOI:10.1128/AEM.01971-10
|
| [58] |
Dong XX, Chen XL, Qian YY, et al. Metabolic engineering of Escherichia coli W3110 to produce L-malate. Biotechnol Bioeng, 2017, 114(3): 656-664. DOI:10.1002/bit.26190
|
| [59] |
Chemler JA, Fowler ZL, McHugh KP, et al. Improving NADPH availability for natural product biosynthesis in Escherichia coli by metabolic engineering. Metab Eng, 2010, 12(2): 96-104. DOI:10.1016/j.ymben.2009.07.003
|
| [60] |
Gao C, Wang S, Hu G, et al. Engineering Escherichia coli for malate production by integrating modular pathway characterization with CRISPRi-guided multiplexed metabolic tuning. Biotechnol Bioeng, 2018, 115(3): 661-672. DOI:10.1002/bit.26486
|
| [61] |
Mu L, Wen JP. Engineered Bacillus subtilis 168 produces L-malate by heterologous biosynthesis pathway construction and lactate dehydrogenase deletion. World J Microbiol Biotechnol, 2013, 29(1): 33-41. DOI:10.1007/s11274-012-1155-6
|
| [62] |
Liu J, Xie Z, Shin HD, et al. Rewiring the reductive tricarboxylic acid pathway and L-malate transport pathway of Aspergillus oryzae for overproduction of L-malate. J Biotechnol, 2017, 253: 1-9. DOI:10.1016/j.jbiotec.2017.05.011
|
| [63] |
Chen XL, Wang YC, Dong XX, et al. Engineering rTCA pathway and C4-dicarboxylate transporter for L-malic acid production. Appl Microbiol Biotechnol, 2017, 101(10): 4041-4052. DOI:10.1007/s00253-017-8141-8
|
| [64] |
Knuf C, Nookaew I, Remmers I, et al. Physiological characterization of the high malic acid-producing Aspergillus oryzae strain 2103a-68. Appl Microbiol Biotechnol, 2014, 98(8): 3517-3527. DOI:10.1007/s00253-013-5465-x
|
| [65] |
Zambanini T, Kleineberg W, Sarikaya E, et al. Enhanced malic acid production from glycerol with high-cell density Ustilago trichophora TZ1 cultivations. Biotechnol Biofuels, 2016, 9: 135. DOI:10.1186/s13068-016-0553-7
|
| [66] |
West TP. Malic acid production from thin stillage by Aspergillus species. Biotechnol Lett, 2011, 33(12): 2463-2467. DOI:10.1007/s10529-011-0720-7
|
| [67] |
Li XJ, Liu Y, Yang Y, et al. High levels of malic acid production by the bioconversion of corn straw hydrolyte using an isolated Rhizopus delemar strain. Biotechnol Bioprocess Eng, 2014, 19(3): 478-492. DOI:10.1007/s12257-014-0047-z
|
| [68] |
Brown SH, Bashkirova L, Berka R, et al. Metabolic engineering of Aspergillus oryzae NRRL 3488 for increased production of L-malic acid. Appl Microbiol Biotechnol, 2013, 97(20): 8903-8912. DOI:10.1007/s00253-013-5132-2
|
| [69] |
Chen XL, Wang YC, Dong XX, et al. Engineering rTCA pathway and C4-dicarboxylate transporter for L-malic acid production. Appl Microbiol Biot, 2017, 101(10): 4041-4052. DOI:10.1007/s00253-017-8141-8
|
| [70] |
Yin X, Li J, Shin HD, et al. Metabolic engineering in the biotechnological production of organic acids in the tricarboxylic acid cycle of microorganisms: advances and prospects. Biotechnol Adv, 2015, 33(6 pt 1): 830-841.
|
| [71] |
Deng Y, Mao Y, Zhang X. Metabolic engineering of a laboratory-evolved Thermobifida fusca muC strain for malic acid production on cellulose and minimal treated lignocellulosic biomass. Biotechnol Prog, 2016, 32(1): 14-20. DOI:10.1002/btpr.2180
|
| [72] |
Zhang T, Ge CY, Deng L, et al. C4-dicarboxylic acid production by overexpressing the reductive TCA pathway. FEMS Microbiol Lett, 2015, 362(9): fnv052.
|
| [73] |
Xu N, Liu L, Zou W, et al. Reconstruction and analysis of the genome-scale metabolic network of Candida glabrata. Mol Biosyst, 2013, 9(2): 205-216. DOI:10.1039/C2MB25311A
|
| [74] |
Li C, Yang XF, Gao S, et al. Hydrolysis of fruit and vegetable waste for efficient succinic acid production with engineered Yarrowia lipolytica. J Clean Prod, 2018, 179: 151-159. DOI:10.1016/j.jclepro.2018.01.081
|
| [75] |
Thangarasu S, Siva V, Athimoolam S, et al. Molecular structure, spectroscopic and quantum chemical studies on benzoic acid and succinic acid co-crystals of 2-aminopyrimidine. J Theor Comput Chem, 2018, 17(4): 1850021. DOI:10.1142/S0219633618500219
|
| [76] |
Klein BC, Silva JFL, Junqueira TL, et al. Process development and techno-economic analysis of bio-based succinic acid derived from pentoses integrated to a sugarcane biorefinery. Biofuels, Bioprod Bioref, 2017, 11(6): 1051-1064. DOI:10.1002/bbb.1813
|
| [77] |
Altuna L, Herrera ML, Foresti ML. Synthesis and characterization of octenyl succinic anhydride modified starches for food applications. A review of recent literature. Food Hydrocoll, 2018, 80: 97-110. DOI:10.1016/j.foodhyd.2018.01.032
|
| [78] |
Li RR, Zhao J, Han DM, et al. Pd/C modified with Sn catalyst for liquid-phase selective hydrogenation of maleic anhydride to gamma-butyrolactone. Chin Chem Lett, 2017, 28(6): 1330-1335. DOI:10.1016/j.cclet.2017.04.028
|
| [79] |
Gu BH, Zheng P, Yan Q, et al. Aqueous two-phase system: an alternative process for recovery of succinic acid from fermentation broth. Sep Purif Technol, 2014, 138: 47-54. DOI:10.1016/j.seppur.2014.09.034
|
| [80] |
Dessie W, Xin F, Zhang W, et al. Opportunities, challenges, and future perspectives of succinic acid production by Actinobacillus succinogenes. Appl Microbiol Biotechnol, 2018, 102(23): 9893-9910. DOI:10.1007/s00253-018-9379-5
|
| [81] |
Meynial-Salles I, Dorotyn S, Soucaille P. A new process for the continuous production of succinic acid from glucose at high yield, titer, and productivity. Biotechnol Bioeng, 2008, 99(1): 129-135. DOI:10.1002/bit.21521
|
| [82] |
Lee PC, Lee SY, Hong SH, et al. Biological conversion of wood hydrolysate to succinic acid by Anaerobiospirillum succiniciproducens. Biotechnol Lett, 2003, 25(2): 111-114. DOI:10.1023/A:1021907116361
|
| [83] |
Der Werf MJV, Guettler MV, Jain MK, et al. Environmental and physiological factors affecting the succinate product ratio during carbohydrate fermentation by Actinobacillus sp. 130Z. Arch Microbiol, 1997, 167(6): 332-342. DOI:10.1007/s002030050452
|
| [84] |
Carvalho M, Matos M, Roca C, et al. Succinic acid production from glycerol by Actinobacillus succinogenes using dimethylsulfoxide as electron acceptor. Nat Biotechnol, 2014, 31(1): 133-139.
|
| [85] |
Pateraki C, Patsalou M, Vlysidis A, et al. Actinobacillus succinogenes: Advances on succinic acid production and prospects for development of integrated biorefineries. Biochem Eng J, 2016, 112: 285-303. DOI:10.1016/j.bej.2016.04.005
|
| [86] |
Bradfield MFA, Nicol W. Continuous succinic acid production from xylose by Actinobacillus succinogenes. Bioprocess Biosyst Eng, 2016, 39(2): 233-244. DOI:10.1007/s00449-015-1507-3
|
| [87] |
Lee PC, Lee SY, Hong SH, et al. Batch and continuous cultures of Mannheimia succiniciproducens MBEL55E for the production of succinic acid from whey and corn steep liquor. Bioprocess Biosyst Eng, 2003, 26(1): 63-67. DOI:10.1007/s00449-003-0341-1
|
| [88] |
Okino S, Inui M, Yukawa H. Production of organic acids by Corynebacterium glutamicum under oxygen deprivation. Appl Microbiol Biotechnol, 2005, 68(4): 475-480. DOI:10.1007/s00253-005-1900-y
|
| [89] |
Ma JF, Li F, Liu RM, et al. Succinic acid production from sucrose and molasses by metabolically engineered E. coli using a cell surface display system. Biochem Eng J, 2014, 91: 240-249. DOI:10.1016/j.bej.2014.08.014
|
| [90] |
Zhu LW, Li XH, Zhang L, et al. Activation of glyoxylate pathway without the activation of its related gene in succinate-producing engineered Escherichia coli. Metab Eng, 2013, 20: 9-19. DOI:10.1016/j.ymben.2013.07.004
|
| [91] |
De Mey M, De Maeseneire S, Soetaert W, et al. Minimizing acetate formation in E. coli fermentations. J Ind Microbiol Biotechnol, 2007, 34(11): 689-700. DOI:10.1007/s10295-007-0244-2
|
| [92] |
Kim P, Laivenieks M, Vieille C, et al. Effect of overexpression of Actinobacillus succinogenes phosphoenolpyruvate carboxykinase on succinate production in Escherichia coli. Appl Environ Microbiol, 2004, 70(2): 1238-1241. DOI:10.1128/AEM.70.2.1238-1241.2004
|
| [93] |
Park DH, Laivenieks M, Guettler MV, et al. Microbial utilization of electrically reduced neutral red as the sole electron donor for growth and metabolite production. Appl Environ Microbiol, 1999, 65(7): 2912-2917. DOI:10.1128/AEM.65.7.2912-2917.1999
|
| [94] |
Guarnieri MT, Chou YC, Salvachúa D, et al. Metabolic engineering of Actinobacillus succinogenes provides insights into succinic acid biosynthesis. Appl Environ Microbiol, 2017, 83(17). DOI:10.1128/aem.00996-17
|
| [95] |
Zhang X, Jantama K, Shanmugam KT, et al. Reengineering Escherichia coli for succinate production in mineral salts medium. Appl Environ Microbiol, 2009, 75(24): 7807-7813. DOI:10.1128/AEM.01758-09
|
| [96] |
Jiang M, Liu SW, Ma JF, et al. Effect of growth phase feeding strategies on succinate production by metabolically engineered Escherichia coli. Appl Environ Microbiol, 2010, 76(4): 1298-1300. DOI:10.1128/AEM.02190-09
|
| [97] |
Wang D, Li Q, Song ZY, et al. High cell density fermentation via a metabolically engineered Escherichia coli for the enhanced production of succinic acid. J Chem Technol Biotechnol, 2011, 86(4): 512-518. DOI:10.1002/jctb.2543
|
| [98] |
Liu Y, Wu H, Li Q, et al. Process development of succinic acid production by Escherichia coli NZN111 using acetate as an aerobic carbon source. Enzyme Microb Technol, 2011, 49(5): 459-464. DOI:10.1016/j.enzmictec.2011.07.006
|
| [99] |
Huang B, Yang H, Fang GC, et al. Central pathway engineering for enhanced succinate biosynthesis from acetate in Escherichia coli. Biotechnol Bioeng, 2018, 115(4): 943-954. DOI:10.1002/bit.26528
|
| [100] |
Li YJ, Huang B, Wu H, et al. Production of succinate from acetate by metabolically engineered Escherichia coli. ACS Synth Biol, 2016, 5(11): 1299-1307. DOI:10.1021/acssynbio.6b00052
|
| [101] |
Zhang W, Zhu J, Zhu X, et al. Expression of global regulator IrrE for improved succinate production under high salt stress by Escherichia coli. Bioresour Technol, 2018, 254: 151-156. DOI:10.1016/j.biortech.2018.01.091
|
| [102] |
Litsanov B, Brocker M, Bott M. Toward homosuccinate fermentation: metabolic engineering of Corynebacterium glutamicum for anaerobic production of succinate from glucose and formate. Appl Environ Microbiol, 2012, 78(9): 3325-3337. DOI:10.1128/AEM.07790-11
|
| [103] |
Xu H, Zhou Z, Wang C, et al. Enhanced succinic acid production in Corynebacterium glutamicum with increasing the available NADH supply and glucose consumption rate by decreasing H(+)-ATPase activity. Biotechnol Lett, 2016, 38(7): 1181-1186. DOI:10.1007/s10529-016-2093-4
|
| [104] |
Hasunuma T, Matsuda M, Kato Y, et al. Temperature enhanced succinate production concurrent with increased central metabolism turnover in the cyanobacterium Synechocystis sp. PCC 6803. Metab Eng, 2018, 48: 109-120. DOI:10.1016/j.ymben.2018.05.013
|
| [105] |
Yan DJ, Wang CX, Zhou JM, et al. Construction of reductive pathway in Saccharomyces cerevisiae for effective succinic acid fermentation at low pH value. Bioresour Technol, 2014, 156: 232-239. DOI:10.1016/j.biortech.2014.01.053
|
| [106] |
Franco-Duarte R, Bessa D, Gonçalves F, et al. Genomic and transcriptomic analysis of Saccharomyces cerevisiae isolates with focus in succinic acid production. FEMS Yeast Res, 2017, 17(6). DOI:10.1093/femsyr/fox057
|
| [107] |
Wahl SA, Bernal Martinez C, Zhao Z, et al. Intracellular product recycling in high succinic acid producing yeast at low pH. Microb Cell Fact, 2017, 16(1): 90. DOI:10.1186/s12934-017-0702-0
|
| [108] |
Agren R, Otero JM, Nielsen J. Genome-scale modeling enables metabolic engineering of Saccharomyces cerevisiae for succinic acid production. J Ind Microbiol Biotechnol, 2013, 40(7): 735-747. DOI:10.1007/s10295-013-1269-3
|
| [109] |
Dai ZX, Guo F, Zhang SJ, et al. Bio-based succinic acid: an overview of strain development, substrate utilization, and downstream purification. Biofuels, Bioprod Bioref, 2020, 14(5): 965-985. DOI:10.1002/bbb.2063
|
| [110] |
Yuzbashev TV, Yuzbasheva EY, Sobolevskaya TI, et al. Production of succinic acid at low pH by a recombinant strain of the aerobic yeast Yarrowia lipolytica. Biotechnol Bioeng, 2010, 107(4): 673-682. DOI:10.1002/bit.22859
|
| [111] |
Cui Z, Gao C, Li J, et al. Engineering of unconventional yeast Yarrowia lipolytica for efficient succinic acid production from glycerol at low pH. Metab Eng, 2017, 42: 126-133. DOI:10.1016/j.ymben.2017.06.007
|
| [112] |
Liu YP, Zheng P, Sun ZH, et al. Economical succinic acid production from cane molasses by Actinobacillus succinogenes. Bioresour Technol, 2008, 99(6): 1736-1742. DOI:10.1016/j.biortech.2007.03.044
|
| [113] |
Lee PC, Lee WG, Lee SY, et al. Succinic acid production with reduced by-product formation in the fermentation of Anaerobiospirillum succiniciproducens using glycerol as a carbon source. Biotechnol Bioeng, 2001, 72(1): 41-48. DOI:10.1002/1097-0290(20010105)72:1<41::AID-BIT6>3.0.CO;2-N
|
| [114] |
Lee SJ, Song H, Lee SY. Genome-based metabolic engineering of Mannheimia succiniciproducens for succinic acid production. Appl Environ Microbiol, 2006, 72(3): 1939-1948. DOI:10.1128/AEM.72.3.1939-1948.2006
|
| [115] |
Lee JW, Yi J, Kim TY, et al. Homo-succinic acid production by metabolically engineered Mannheimia succiniciproducens. Metab Eng, 2016, 38: 409-417. DOI:10.1016/j.ymben.2016.10.004
|
| [116] |
Li C, Yang X, Gao S, et al. High efficiency succinic acid production from glycerol via in situ fibrous bed bioreactor with an engineered Yarrowia lipolytica. Bioresour Technol, 2017, 225: 9-16. DOI:10.1016/j.biortech.2016.11.016
|
| [117] |
Cui Z, Gao C, Li J, et al. Engineering of unconventional yeast Yarrowia lipolytica for efficient succinic acid production from glycerol at low pH. Metab Eng, 2017, 42: 126-133. DOI:10.1016/j.ymben.2017.06.007
|
 2021, Vol. 37
2021, Vol. 37