中国科学院微生物研究所、中国微生物学会主办
文章信息
- 马晓焉, 王雪芹, 马炼杰, 霍毅欣
- Ma Xiaoyan, Wang Xueqin, Ma Lianjie, Huo Yi-Xin
- 高级醇的微生物绿色制造
- Microbial green manufacturing of higher alcohols
- 生物工程学报, 2021, 37(5): 1721-1736
- Chinese Journal of Biotechnology, 2021, 37(5): 1721-1736
- 10.13345/j.cjb.200700
-
文章历史
- Received: October 31, 2020
- Accepted: February 22, 2021
煤、石油和天然气等化石资源是目前人类使用的主要燃料,也是重要的化工原料。随着工业化进程的加速,不可再生的石油能源已不能满足社会发展的需要,引发了日益凸显的能源危机。同时,化石燃料燃烧产生的CO2等温室气体加剧了气候变化,对全球生态造成了严重冲击。为满足日益增长的全球能源需求并降低对环境的不利影响,源自可再生资源的生物燃料开始成为传统燃料的补充与替代品。当今最常见的生物燃料是源自玉米淀粉或蔗糖的乙醇,其在20世纪70年代就被作为汽车燃油的主要添加剂。然而,相比于汽油,乙醇的能量密度低、吸湿性高,且具有腐蚀性,不利于长期储存和大量添加,因而无法作为理想的替代燃料。相比之下,碳原子数大于2的高级醇,特别是丁醇、戊醇等长链高级醇具有与汽油接近的能量密度,同时兼备不吸湿与腐蚀性低的特性,可以与汽油以任意比例混合甚至完全取代汽油,因而被认为是最具前景的燃油替代品[1] (表 1)。这类高级醇包括具有3个碳原子(C3) 的正丙醇和异丙醇,4个碳原子(C4) 的异丁醇、正丁醇和2-丁醇,5个碳原子(C5) 的正戊醇、2-甲基-1-丁醇和3-甲基-1-丁醇,以及大于6个碳原子(> C6) 的正己醇、正庚醇等(表 2)。其中C4的正丁醇可由梭状芽孢杆菌Clostridium通过丙酮-丁醇-乙醇(Acetone-butanol-ethanol,ABE) 发酵产生,并在20世纪20年代开始规模化发酵生产,一度成为最受关注的高级醇种类。然而,这一传统方法在合成正丁醇的同时也产生了丁酸、丙酮、乙醇等副产物,目标产物的产率严重受限。同时,梭状芽孢杆菌生长缓慢、生理特性复杂、遗传操作困难、存在芽孢期等问题也极大地限制了发酵菌株的改良优化,正丁醇的产量已难以满足当前对高级醇的迫切需求[2]。而其同分异构体异丁醇甚至不存在天然的高产菌株,仅伴随酿酒酵母Saccharomyces cerevisiae等少数微生物的乙醇发酵过程少量产生,远未达到规模化产量水平。因此,利用代谢工程手段,在常用的模式工业菌株中重构高级醇的合成途径,是克服高级醇生产瓶颈的必要手段[3-4]。随着2007年起国际油价的持续走高,高级醇的代谢工程研究开始升温(图 1A),并在原油价格增至顶峰的2008年取得了里程碑式突破,相继搭建了正丁醇、异丁醇以及C6–C8长链醇的人工合成途径,并在异源微生物中实现了上述醇类的高效合成[3-5],摆脱了高级醇生产长期以来对原生宿主的依赖。在接下来的5年中,针对高级醇合成各个环节的工程研究百花齐放。部分研究着眼于合成途径的优化,如通过酶挖掘与定向进化改造限速酶的辅因子偏好性,实现了厌氧条件下C4醇的高效生产;相当一部分研究着眼于原料的替换,将高级醇合成的物料来源由简单的糖类扩展至纤维素、蛋白质以及CO2等一碳(C1) 化合物,并同步开发了梭菌Clostridia[6]、枯草芽孢杆菌Bacillus subtilis[7]、罗氏真养产碱菌Ralstonia eutropha[8]、蓝细菌[9]等一系列底盘宿主;另有部分研究集中于菌株的鲁棒性提升,获得了耐受高浓度高级醇的优良个体[10]。上述重大突破在2011年前后集中发表,将高级醇的生物合成推向快速发展时期并转入产业化轨道。近年来,随着传统代谢工程与合成生物学等新兴技术的集成,高级醇的代谢工程迎来了新的发展阶段,CRISPR[11]、生物传感[12]等技术开始被用于生产菌株的升级改造,有望进一步拉动高级醇当前产率向理论值的提升(图 1B)。自高级醇代谢工程兴起至今,美国科学院与工程院双院院士James C. Liao教授、加州大学伯克利分校的美国工程院院士Jay D. Keasling教授、加州理工大学的诺贝尔奖得主Frances H. Arnold教授等知名团队都争先投身于这一研究领域。特别是以James C. Liao教授为代表的研究团队更是取得了数个开创性成果,并因此获得了2010年的美国总统绿色化学挑战奖。作为James C. Liao教授当年的团队成员之一,本文作者亲历了高级醇异源微生物合成从零到一,以及实现产量跨越的各项里程碑式工作。在代谢工程诞生30周年之际,本文聚焦高级醇这一代谢工程领域的明星化合物,详细梳理了其异源合成技术的开发历史,从合成途径的创建、原料的扩展、底盘的开发、产量的优化等方面进行了全方位回顾总结(表 3),并对未来发展趋势进行了提炼,希望以此激发对高级醇微生物合成技术的进一步开发,助力我国生物能源领域的开拓创新。
| Ethanol | n-butanol | Isobutanol | Gasoline | |
| Energy density (MJ/L) | 21.4 | 26.9 | 26.6 | 30–33 |
| Gasoline energy (%) | 64.8 | 81.5 | 80.6 | 100.0 |
| Octane number | 100.0 | 90.0 | 98.0 | 88–93 |
| Boiling point (℃) | 78.4 | 117.8 | 107.9 | 27–225 |
| Vapor pressure (kPa) | 16.0 | 2.2 | 3.3 | 54–103 |
| Water solubility (%/20 ℃) | 100 | 7.7 | 8.5 | – |
| Oxygen content (weight percentage %) | 35.0 | 22.0 | 22.0 | – |
| Types of higher alcohols | Structural formulas | Native producers |
| n-propanol |  |
No |
| Isopropanol |  |
Yes (e.g. E. coli) |
| n-butanol |  |
Yes (e.g. Clostridium spp.) |
| Isobutanol |  |
Yes (e.g. S. cerevisiae) |
| 2-butanol |  |
Yes (e.g. Lactobacillus spp.) |
| n-pentanol |  |
No |
| 2-methyl-1-butanol |  |
No |
| 3-methyl-1-butanol |  |
No |
| n-hexanol |  |
No |
| n-heptanol |  |
No |

|
| 图 1 高级醇代谢工程的发展历程 Fig. 1 Development course for metabolic engineering of higher alcohols. (A) Trends in academic citations derived from Dimensions (dimensions.ai) using the key words "higher alcohols" and "biosynthesis". (B) Milestones in the biosynthesis of higher alcohols. |
| |
| Products | Hosts | Genotypes (knockout; overexpression) | Substrates | Fermentation conditions (extraction methods) | Titer (g/L) | References |
| n-propanol | E. coli | ΔilvA ΔilvB; kivD adh2 cimAmut leuABCD | Glucose | Shake flask | 2.78 | [16] |
| E. coli | ΔlacI ΔlysA ΔmetA ΔtdhA ΔiclR ΔilvIH ΔilvBNΔrpoS thrAC1034T lysCC1005T | Glycerol | Bioreactor | 10.3 | [17] | |
| Isopropanol | E. coli | None; lacIq thl atoDA adc adhB-593 | Glucose | Stirred flask (fed- batch with gas stripping) | 143 | [18] |
| E. coli | None; thl atoDA adc adhB-593 bgl-blc | Cellobiose | Shake flask (gas stripping) | 4.1 | [19] | |
| E. coli | None; thl ctfAB adc adhB-593 | Glucose | Shake baffled flask | 13.6 | [20] | |
| Alcohol mixture | C. acetobutylicum | Δbuk: : ermC; adc ctfAB adhB-593 | Glucose | Bioreactor (gas stripping) | 35 | [21] |
| C. acetobutylicm | Δbuk ΔC1502; adc ctfAB adhB-593 | Glucose | Bioreactor (gas stripping) | 20.4 | [22] | |
| n-butanol | S. elongatus | None; atoB, hbd, crt, ter, AdhE2 | CO2 | Flask and bioreactor | 0.015 | [9] |
| S. elongatus | None; nphT7 phaB aphaJ ter bld yqhD | CO2 | Static capped flask | 0.029 9 | [23] | |
| C. acetobutylicm | Δpta Δbuk; adhED485G | Glucose | Bioreactor | 130 | [24] | |
| Clostridium tyrobutyricum | None; adhE2 | Mannitol | Bioreactor; anaerobic | 20.5 | [25] | |
| S. cerevisiae | None; thl, hbd, crt, bcd, etfAB, adhE2 | Galactose | Shake flask | 0.002 5 | [26] | |
| B. subtilis | None; thl, hbd, crt, bcd, etfAB, adhE2 | Glycerol | Shake flask | 0.024 | [27] | |
| P. putida | None; thl, hbd, crt, bcd, etfAB, adhE1 | Glycerol | Shake flask | 0.122 | [27] | |
| L. brevis | None; thl, hbd, crt, bcd, etfAB | Glucose | Vial | 0.3 | [28] | |
| Clostridium ljungdahlii | None; thlA, hbd, crt, bcd, adhE, and bdhA | Syngas | Shake flask | 0.148 | [29] | |
| E. coli | ΔldhA ΔadhE ΔfrdBC Δpta; atoB hbd crt adhE2 fdh ter | Glucose | Bioreactor; anaerobic (gas stripping) | 30 | [30] | |
| Isobutanol | E. coli | ΔadhE ΔldhA ΔfrdBC Δfnr Δpta ΔpflB; alsS ilvCD kivD adh2 | Glucose | Static capped flask | 22 | [4] |
| E. coli | None; alsS ilvCD kivD adhA | Glucose | Bioreactor; | 56 | [31] | |
| S. elongatus | None; alsS, ilvCD, kivd, yqhD | CO2 | Bottle | 0.450 | [32] | |
| C. crenatium | None; ILV2, ILV3, ILV5, kivD, ADH6 | Lignocellulose | Bottle | 5.61 | [33] | |
| Bacillus megaterium | None; kivd, yqhD | Glucose | Test tube (extracted with solvent supercritical CO2) | 0.3 | [34] | |
| B. subtilis | Δldh; ilvCD alsS kivD adh2 | Glucose | Bioreactor | 3.83 | [35] | |
| C. cellulolyticum | None; kivD yqhD alsS ilvCD | Cellulose | Not specified | 0.66 | [6] | |
| Corynebacterium glutamicum | ΔaceE Δpqo ΔilvE ΔldhA Δmdh; ilvBNCD pntAB kivD adhA | Glucose | Bioreactor | 13 | [36] | |
| R. eutropha | ΔphaB2C2 ΔphaC1AB1; alsS ilvCD kivD yqhD | CO2 | Bioreactor with electrodes | 0.09 | [8] | |
| R. eutropha | ΔphaCAB ΔilvE ΔbkdAB ΔaceE; adh ilvBHCD kivD | Fructose | Shake flask | 0.27 | [37] | |
| S. cerevisiae | Δlpd; kivD ADH6 ILV2 ILV5c ILV3c ILV2C MAE1 | Glucose | Shake flask | 1.62 | [38] | |
| 2-butanol | L. diolivorans | None; pduQ, PDO-DH (NADPH) | meso-2, 3-butanediol/glycerol | Bioreactor | 13.4 | [39] |
| Klebsiella pneumoniae | ΔldhA; pduCDEGHQ337A or F375I, adh | Glucose | Shake flask | 1.03 | [40] | |
| L. brevis | None; none | meso-2, 3-butanediol | Test tube | 0.88 | [41] | |
| Lactobacillus buchneri | None; none | butanone | Test tube | 0.04 | [41] | |
| S. cerevisiae | None; pduCDEGH, adh | meso-2, 3‐butanediol | Shake flask | 0.004 | [42] | |
| Lactobacillus spp. | None; None | meso-2, 3‐butanediol | Shake flask | 0.41 | [43] | |
| 2-methyl-1-butanol | E. coli | ΔmetA Δtdh; ilvGM ilvCD ilvA kivD adh2 thrABC | Glucose | Shake baffled flask | 1.25 | [44] |
| 3-methyl-1-butanol | E. coli | None; alsS ilvCD kivD adh2 leuAG462D leuBCD | Glucose | Shake flask | 9.5 | [45] |
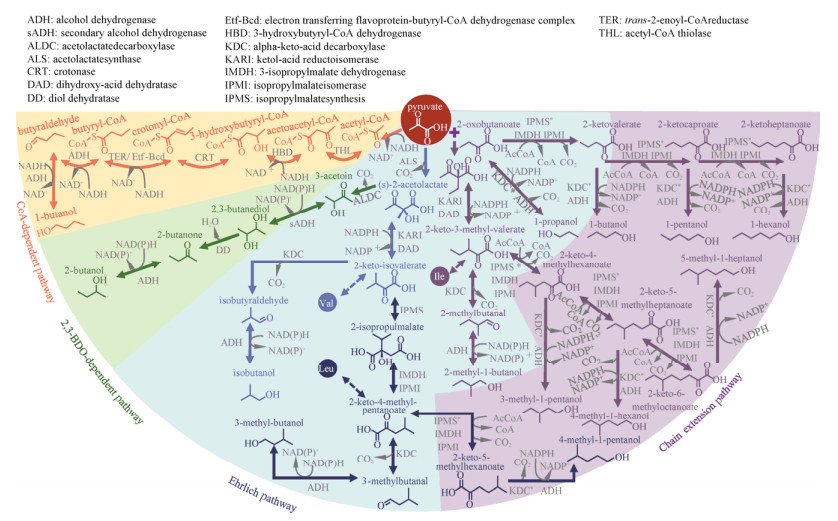
异丁醇在性质上非常接近汽油,拥有与汽油相似的能量密度、辛烷值和蒸汽压(表 1),是理想的燃油替代品。2008年,James C. Liao教授团队在大肠杆菌Escherichia coli中成功构建了异丁醇的合成途径(图 1B),实现了高达86%理论转化率的异丁醇合成[4]。该设计劫持了大肠杆菌的分支氨基酸合成途径,在缬氨酸合成的最后一步,即α-酮异戊酸经转氨生成缬氨酸的反应步骤中插入了Ehrlich途径,催化α-酮异戊酸发生脱羧反应生成异丁醛,随后经一步还原反应转化为终产物异丁醇(图 2)。在以上反应中,α-酮酸脱羧酶催化的酮酸脱羧反应是整个合成途径中唯一的不可逆步骤,为产物合成提供了持续驱动力,迫使本应用于缬氨酸合成的α-酮异戊酸分配至异丁醇的合成支路。在尝试了3个物种的5个α-酮酸脱羧酶后,来自乳酸乳球菌的Kivd对5个碳原子的α-酮异戊酸表现出了最高的催化活性,从而与来自酿酒酵母的醇脱氢酶ADH2组成了异丁醇合成途径的最后两个步骤。同时,为增强初级代谢流向缬氨酸合成途径的分配,来自枯草芽孢杆菌的乙酰乳酸合酶AlsS被引入合成途径。相比于大肠杆菌自身的乙酰乳酸合酶IlvIH,AlsS对丙酮酸具有更高的亲和力,可驱动丙酮酸优先流向缬氨酸合成途径,削弱异亮氨酸等竞争合成途径对丙酮酸的分流。以AlsS取代IlvIH后,异丁醇产量显著提升了1.7倍。在同步过表达缬氨酸合成途径的ilvCD后,异丁醇的产量突破20 g/L,达到了工业化生产要求。该工作充分借助了大肠杆菌氨基酸合成途径的高活性优势和Ehrlich途径酶的宽底物谱特性,一次性实现了从无到有的异丁醇异源微生物合成和产量的跨越式提升。因氨基酸合成机制在微生物中广泛存在,该途径展现出极佳的物种适配性,已被成功移植入枯草芽孢杆菌[7]、梭菌[6]、钝齿棒杆菌Corynebacterium crenatium[33]、罗氏真养产碱菌[8]等多种常见宿主(表 3)。
|
| 图 2 高级醇的生物合成途径 Fig. 2 Biosynthetic pathways for higher alcohols. Asterisk represents engineered enzymes. |
| |
正丁醇的天然生产者梭状芽孢杆菌通过辅酶A(CoA) 依赖型途径进行正丁醇的合成[3](图 2)。该途径始于两个乙酰CoA分子的缩合,由此生成碳链长度为4的乙酰乙酰CoA,作为正丁醇的碳骨架;乙酰乙酰CoA经一步还原、一步脱水,以及再次还原脱去3位的羰基,生成丁酰CoA;随后再经2步连续的还原反应脱去CoA,并将剩余的醛基还原为羟基,完成正丁醇的合成。该途径是目前应用最为广泛的正丁醇合成途径,已被移植入大肠杆菌[30]、枯草芽孢杆菌[27]、聚球藻[9]等多种底盘中(表 3)。尽管如此,这一CoA依赖型途径依然存在以下天然不足:1) 该途径总共包含4步还原反应,对还原力需求较高;2) 催化还原步骤的原生酶对氧浓度十分敏感,在好氧菌中活性不稳定;3) 途径中的所有反应均为可逆反应,缺乏可作为驱动力的不可逆步骤。因此,该途径在异源宿主中普遍表现不佳,植入大肠杆菌后仅能生成13.9 mg/L的正丁醇。后续优化在一定程度上解决了CoA依赖性途径的先天不足,正丁醇的生产效率得到了显著改善。其中重要的一步是将原生的丁酰CoA脱氢酶(Bcd) 替换为反式烯酰CoA还原酶(Ter)[30]。该酶的优势在于将原有的巴豆酰CoA向丁酰CoA的可逆反应改变为单向不可逆反应。其次,Ter酶能以NADH作为直接的电子供体,摆脱了原生Bcd酶对电子传递黄素蛋白的依赖,这使得整条合成途径中的4步还原反应均以NADH作为唯一的辅因子,从而将正丁醇的合成转化为一条高效回收NADH的途径。Ter酶的以上特点为CoA依赖型途径创造了强劲的驱动力,配合对大肠杆菌底盘菌株NADH消耗途径等代谢反应的优化,正丁醇的产量显著提升至30 g/L,产率达到理论水平的88%[30]。
为彻底消除CoA依赖型途径的不足,James C. Liao教授课题组依照异丁醇合成途径的设计原理,结合氨基酸合成及Ehrlich途径,于同年创建了CoA非依赖型的正丁醇合成途径[4]。该途径的瓶颈在于大肠杆菌只能生成微量的正丁醇前体α-酮戊酸,为提高前体供应水平,该设计将苏氨酸的酮酸前体α-酮丁酸用于α-酮戊酸的合成,以亮氨酸合成途径中宽底物谱的LeuABCD催化α-酮丁酸的碳链延伸,生成的α-酮戊酸经脱羧与还原转化为正丁醇。经过以上优化,正丁醇产量提升了3倍,达到44 mg/L。此外,α-酮戊二酸途径也被应用于正丁醇的合成[46]。相较于天然的CoA依赖型途径,该途径的酶促反应在热力学上更易进行。然而,该途径需要将1分子的磷酸烯醇丙酮酸羧化为草酰乙酸,并使用乙酰CoA为供体将2-羟基戊二酸活化为2-羟基戊二酰CoA。这些步骤需要额外的能量供应,削弱了α-酮戊二酸途径的优势。因此,正丁醇合成途径的深度优化或从头设计仍存在很大的发展空间。
1.3 其他高级醇除正丁醇和异丁醇这两种C4高级醇外,同为C4的2-丁醇,以及具有更多碳原子的2-甲基-1-丁醇(2-methyl-1-butanol,2MB) 和3-甲基-1-丁醇(3-methyl-1-butanol,3MB) 两种C5高级醇、C5以上的4-甲基-1-戊醇(4-methyl-1-pentanol,4MP) 等长链醇也是理想的生物燃料,这类高级醇同样遵从C4高级醇的合成原理[5]。其中2MB和3MB可分别由异亮氨酸和亮氨酸的α-酮酸前体经转化生成(图 2),并与异丁醇共同出现于终产物中。三种高级醇的比例取决于乙酰乳酸合酶的底物偏好性,如鼠伤寒沙门氏菌的乙酰乳酸合酶Ⅱ (IlvGM) 对底物α-酮丁酸更具偏好,可驱动代谢流向异亮氨酸的合成途径转移,适用于2MB的生产[44]。对于碳链长度超过5的长链醇,其合成瓶颈在于大肠杆菌无法天然合成长链的酮酸前体,Zhang等[5]开创性地将亮氨酸合成途径中LeuABCD催化的碳链延伸反应改造为碳链迭代延长的流水线。为了使LeuA获得合成长链酮酸的能力,Zhang等通过理性改造手段扩大了LeuA的底物结合口袋,得到了可以催化C7–C9酮酸合成的一系列LeuA突变体,实现了C6–C8非天然长链醇的微生物合成。此外,C4的2-丁醇具有比其同分异构体更低的细胞毒性和相近的辛烷值,可作为正丁醇的替代品。某些乳酸菌可在厌氧发酵下天然合成这一产物。其合成途径同样起始于乙酰乳酸,后者经脱羧酶和二级醇脱氢酶(sADH) 的催化生成2, 3-丁二醇,再由二醇脱水酶催化生成2-丁酮,最后在醇脱氢酶的作用下转化为2-丁醇[43](图 2)。目前,乳酸菌依然是2-丁醇合成的主要宿主,通过耦联2, 3-丁二醇的合成并强化内源的醇脱氢酶,乳酸菌Lactobacillus diolivorans的2-丁醇产量可达到13.4 g/L[39]。
2 高级醇生产菌株的优化改造高级醇微生物合成途径的构建展示了代谢工程技术和微生物细胞工厂的巨大潜力,为生物能源产业开辟了新的发展阵地。上述早期研究主要实现了由零到一的突破,并在实验室水平取得了产量验证。然而,初代菌株在合成途径效率与底盘细胞适配等方面仍存在不足,生产能力普遍未达到工业化要求,亟待进行精细优化。在后续众多的优化改良中,与高级醇合成最为相关的两点分别为还原力的优化与菌株的溶剂耐受性优化。
2.1 还原力优化高级醇的生物合成需要大量的还原力,如正丁醇的原生CoA依赖型途径需要4个NADH提供还原力,异丁醇、2MB、3MB等分支高级醇的合成途径需要两个NAD(P)H提供还原力。对辅因子的大量需求带来了3点适配性问题,第一是量的适配,即如何优化胞内的辅因子供应以满足合成途径对辅因子数量的需求;第二是种类的适配,即如何将合成途径的辅因子需求类型与细胞所能提供的辅因子类型相统一;第三是分配的平衡,即如何分配辅因子使其兼顾产物合成与细胞生长,实现产量的最大化。因此,辅因子优化是高级醇生产途径优化中的关键问题。
目前的辅因子工程提供了4种还原力优化策略,分别为改变合成途径的辅因子偏好[30]、强化辅因子合成[47]、调节辅因子再生[30, 47]以及利用合成辅因子[48]。其中,改变途径的辅因子偏好可通过酶的替换或酶的工程改造实现,如醇脱氢酶可分为NADPH和NADH两种依赖型,以异源的NADH依赖型醇脱氢酶(如乳酸乳球菌的AdhA) 替换大肠杆菌原生的NADPH依赖型醇脱氢酶,可部分改变异丁醇合成途径的辅因子偏好性,使其得以直接利用糖酵解途径产生的NADH,有助于实现微氧条件下的发酵生产,异丁醇产量有所提升[49]。而对于异丁醇合成途径中仅存的NADPH依赖型还原酶——酮醇酸还原异构酶IlvC,Frances H. Arnold教授团队采用定向进化策略对其NADPH的结合口袋进行了改造,得到了以NADH作为还原力供体的IlvC[14]。这一工程酶的引入将异丁醇的合成途径彻底转化为一条依赖NADH的途径,实现了完全厌氧的异丁醇发酵,并最终达到了100%的理论产率。除了优化辅因子需求类型与供给类型的匹配度,胞内辅因子的供应与再生水平也需要进行适配优化。强化辅因子的合成与调节辅因子的再生可最大化还原力的供应,同时避免还原力过量对细胞的伤害,更重要的是可以为合成途径提供额外的驱动力。实现这一目标的关键在于将产物的合成途径改造为辅因子再生的唯一渠道,在此情况下,胞内积累的还原态辅因子只能通过目标合成途径转变为对应的氧化形态,从而驱动代谢流涌入产物合成方向,推动合成途径的高效运转[30]。该策略已被用于正丁醇合成途径的优化,如过表达甲酸脱氢酶Fdh可满足正丁醇合成途径对NADH的大量需求[50];而切断大肠杆菌的原生NADH消耗途径(ΔadhE ΔldhA Δfrd),NADH依赖的改良版正丁醇合成途径成为了NAD+的唯一再生通路,为正丁醇的厌氧合成提供了有效的驱动力,最终实现了高达10倍的产量提升[30]。
2.2 菌株的耐受性优化尽管实验室条件下的高级醇发酵已突破20 g/L,更高水平的产量浓度依然是打造经济性高级醇产业的持续追求。在众多制约产量提升的因素中,高级醇的细胞毒性是长期困扰放大生产的瓶颈。其致毒浓度远低于工业发酵的产量水平,如超过8 g/L的异丁醇即会减缓大肠杆菌的生长[51],而正丁醇对天然宿主的致毒浓度更是低至2 g/L[52]。随着发酵液中高级醇的持续累积,这一产物自身对细胞工厂的危害将会越发凸显。因此,提升菌株对高级醇的耐受能力已成为后续研究的关注热点。
高级醇对菌体生长与产物合成的抑制作用成因复杂。已发现的致毒途径包括改变细胞膜的脂质组分、破坏细胞膜流动性、降低胞内pH和ATP浓度、阻碍葡萄糖吸收、抑制氨基酸合成、影响呼吸系统等[51]。由此可见,通过缺失或过表达单个功能基因很难实现耐受性提升,理性设计高级醇耐受菌株也变得极为困难,适应性进化法顺势成为了菌株耐受性优化的首选策略。Atsumi等在异丁醇胁迫环境下对异丁醇宿主菌株JCL260进行了适应性进化,经过45轮的连续富集,最终筛得了一株耐受性显著提高的菌株SA481,其在8 g/L异丁醇胁迫下的菌体数较原始菌株提升5倍[10]。全基因组测序与遗传学鉴定明确了5个关键突变基因(acrA、gatY、tnaA、yhbJ和marCRAB),其中YhbJ的失活提高了肽聚糖和脂多糖前体葡萄糖胺-6-磷酸的合成水平,由此增强了细胞膜的合成能力,从而赋予了菌株更强的高级醇耐受性。该研究得到的SA481菌株为进一步揭示菌株耐受高级醇的机制提供了理想的研究对象,有助于开发抗溶剂毒性的底盘菌株。对于正丁醇,目前较多以更具正丁醇耐受性的其他菌种来替代低耐受性的正丁醇天然生产菌种,包括可以耐受3%正丁醇的枯草芽孢杆菌GRSW2-B1、短乳杆菌Lactobacillus brevis,可以耐受4%正丁醇的酿酒酵母YPS128,以及耐受度高达6%的恶臭假单胞菌Pseudomonas putida[52-55]。
此外,在菌株耐受性改良进展缓慢的背景下,发酵方式的优化成为了缓解高级醇毒性的直接有效手段。其中,气体剥离法(Gas stripping) 已被广泛应用于高级醇的工业化生产,成为连续发酵的关键技术。该方法通过向发酵罐中通入氮气等惰性气体,将发酵罐中积累的高级醇实时带出液相并随气流脱离罐体,使发酵液中的高级醇维持在不引发毒性的低浓度水平,从而实现持续稳定的发酵生产。采用该方法,异丁醇的发酵产量突破了早期22 g/L的浓度记录,达到了50 g/L以上的产量水平[31, 56]。
3 利用替代资源生产高级醇初代高级醇的生产普遍以玉米等淀粉类生物质为原料,随着人口、粮食、环境问题的日益凸显,以工业、农业侧线产品及废弃物作为基础原料进行高附加值产品的生产愈发受到重视。在高级醇合成途径取得突破之后,如何将各类废弃物高效地转化为高级醇产品随即成为了该领域的研究热点。其中,木质纤维素、废弃蛋白质、CO2等C1化合物的资源化受到了广泛关注,多项里程碑式成果相继产出。
3.1 基于木质纤维素的高级醇生产木质纤维素是地球上储量最大的可再生资源,通常被当作不可食用的植物残体处理,作为发酵原料具有物料来源广泛、成本低下、避免与人争粮的优势,已取代了玉米等淀粉类原料用于下一代生物燃料的生产。
木质纤维素作为发酵原料的瓶颈是无法被常见的工业微生物直接利用,需要通过物理、化学以及酶解方法将纤维素预先分解为微生物可利用的单糖。该过程工艺难、成本高、收率低,产生的副产物对下游菌株有明显的抑制作用,严重限制了纤维素高级醇的工业化进程。相比之下,构建具有纤维素利用能力的高级醇生产菌株可实现纤维素水解与产物合成的整合,免除了预处理步骤,是更具应用前景的解决方案。这一纤维素的联合生物加工技术(Consolidated bioprocessing,CBP) 可通过两种策略实现。其一是向高级醇生产菌株中引入纤维素分解系统,如源自梭菌、醋弧菌、瘤胃球菌等厌氧微生物的纤维小体[57]。该系统包含纤维素酶亚基和支架亚基两个部分,二者通过各自的锚定结构域(Dockerin) 和粘附结构域(Cohesin) 组装为多酶复合体,并通过支架亚基上的纤维素结合域将底物结合于纤维素酶复合体上。目前,来自解纤维素梭菌的纤维小体已被成功引入丙酮丁醇梭菌Clostridium acetobutylicum[58]、酵母[59-60]等天然醇类生产菌株,为开发具有纤维素转化能力的高级醇微生物细胞工厂奠定了基础。
目前,纤维素降解途径的移植改造仍处于起步阶段,工程菌株转化纤维素的能力普遍低下[61]。为加快工业化进程,将纤维素天然降解菌株改造为高级醇生产底盘的策略随之产生。2011年,Higashide等将异丁醇的合成途径引入解纤维梭菌Clostridium cellulolyticum,首次证明了纤维素直接转化为异丁醇的可行性,实现了660 mg/L的产量水平[6]。后续研究将正丁醇的CoA依赖型合成途径植入具备纤维素降解能力的Clostridium cellulovorans,通过适应性进化与理性改造,成功将正丁醇产量提升至3.47 g/L,实现了产量的跨越式提升[62]。
3.2 基于蛋白质原料的高级醇生产在生物质精炼过程中,葡萄糖、淀粉、纤维素等碳水化合物被微生物转化为目标化学品,而原料中其他难以利用的碳源以及全部菌体被留存于发酵残渣中。同时,种植、畜牧、食品加工等产业也持续产生废弃的生物质。在此类废弃物中,蛋白质是剩余养分的主要组成之一,通常被加工为畜禽饲料。然而,饲料形式的再利用规模有限,仍有数量庞大的蛋白生物质难以实现高附加值的循环利用,产生了严重的资源浪费。此外,作为一类含氮养分,蛋白质中的还原态氮一直缺少经济可行的提取和再利用手段,导致大量氮素被排放到环境当中,加剧了水体富营养化和温室效应等一系列环境问题。而在新批次的生物质原料培育过程中,又需要再次投入化工法生产的氮肥,进一步加大了生物质精炼的总能耗和总污染。因此,实现废弃蛋白质中碳、氮元素的双重循环是生物质精炼领域面临的巨大挑战。
作为高级醇的前体,将蛋白质(氨基酸) 中的碳骨架转化为生物燃料,同时回收氮素转化为氨态氮肥,在理论上可以同步实现碳中性和氮中性的高级醇生产。为达成这一设想,Huo等[13]重构了大肠杆菌的氨同化途径,通过敲除GdhA和GlnA驱动的氨同化反应阻断了菌体对游离氨的再利用,避免了氨基酸分解产物,即氨分子和α-酮酸再度加合为氨基酸的可能,从而促进了α-酮酸的累积。在植入Ehrlich途径后,累积的α-酮酸即刻被单向的酮酸脱羧酶转化为醛,继而还原为高级醇。这一Ehrlich途径的加入迅速消耗了氨基酸分解产生的α-酮酸,打破了原有的氨吸收-氨基酸合成-氨基酸分解平衡,驱动氨基酸的整体代谢向分解与高级醇合成方向转移。通过过表达缬氨酸转氨酶AvtA、亮氨酸脱水酶LeuDH和丝氨酸脱氨酶SdaB又进一步强化了氨基酸特别是分支氨基酸向α-酮酸的转化能力。至此,蛋白质中的分支氨基酸可被直接转化为对应的C4、C5高级醇,而其他种类的氨基酸则可通过各自的转氨、脱氨途径转化为中心代谢物丙酮酸(苏氨酸转化为2MB前体α-酮丁酸),继而汇入高级醇的合成途径。在氨基酸碳骨架的转化过程中,脱去的氨以游离形态积累于发酵液中,可被直接收集作为氮肥,用于培养固碳微藻或能源作物。利用上述系统,源自酵母、微藻等菌体的废弃蛋白质成功转化为高达4.0 g/L的高级醇,取得了56%的理论产率。
在蛋白质微生物精炼原型系统的基础上,后续研究又对该系统进行了完善优化。如以氨响应的σ54依赖型启动子驱动蛋白质向高级醇的转化,赋予了合成途径自激活、自增强的特性,实现了跨越整个菌体生长周期的高级醇持续生产[63];为了简化蛋白质的前处理过程,又将该系统植入具备发达外泌型蛋白酶的枯草芽孢杆菌,实现了“一锅法”蛋白质水解与高级醇合成[7]。近年来,蛋白质的高值利用越发受到重视[64],深入挖掘微生物细胞工厂的蛋白质转化潜力,继续提升高级醇的产量水平,拓展新的高附加值产品是发展绿色生物制造的优选途径。
3.3 基于C1原料的高级醇生产C1原料是指只有一个碳原子的化合物,如一氧化碳(CO)、二氧化碳(CO2)、甲烷(CH4)、甲酸(HCOOH) 等。这类化合物或在自然环境中大量存在,或可以通过简单的方式快速合成,是取之不尽的绿色原料。同时,多数C1化合物又是主要的温室气体,以其为原料进行高级醇的生产将直接减少碳排放,实现碳中性的绿色合成。
通过微生物将CO2直接转化为高级醇是人们追求的终极目标。在当前阶段,该过程普遍依赖于光能自养的蓝细菌,其中聚球藻Synechococcus elongatus PCC 7942是普遍选用的底盘宿主。该菌株通过卡尔文循环(Calvin-Benson-Bassham cycle,CBB cycle) 将CO2固定为3-磷酸甘油醛,再经糖酵解途径将其转化为中心代谢物丙酮酸,继而参与到一系列产物合成当中。通过引入异丁醇合成途径并增加固碳关键酶1, 5-二磷酸核酮糖羧化酶的拷贝数,聚球藻凭借固碳生成了450 mg/L的异丁醇[32];通过植入正丁醇的CoA依赖型合成途径并进行辅因子优化,菌株成功合成了14.5 mg/L的正丁醇[9]。然而,以光合蓝细菌进行高级醇的固碳生产还面临以下问题:1) 光合作用伴随氧气的生成,会对氧敏感的正丁醇合成途径产生毒害;2) 蓝细菌的遗传操作工具有限,难以对菌株实施深度改造;3) CBB循环能耗高,固碳效率低下,限制了高级醇的产量提升。解决氧毒性的方法包括改用对氧不敏感的同工酶、通过蛋白质工程降低天然酶对氧的敏感性、开发除氧发酵工艺、或改用紫色硫细菌等以硫化氢为电子供体的不产氧光合细菌。而近期在重构固碳循环方面的重大突破为解决蓝细菌及CBB途径的内在不足提供了新思路,具备固碳能力的工程化大肠杆菌[65]和酵母[66],以及全新设计的非天然固碳循环[67]为实现高级醇的高效自养合成提供了无限可能。
目前,直接将CO2转化为高级醇的自养生产方式仍处于低产能水平,产物浓度远未达到工业化发酵的基本要求,间接式的CO2生物转化技术由此产生,并显著提升了高级醇的产量水平。该方法将CO2的固定与产物合成分割为两个独立的模块,首先采用高效的化学固定法将CO2转化为甲酸等形式的C1化合物,再以该C1化合物饲喂工程菌株进行下游的高级醇合成[8]。该方法绕开了低效的天然固碳途径,大幅提高了CO2的固定总量。其中电化学方法可将CO2与水分子在阳极端高效地转化为甲酸,而能够耐受电场胁迫的甲基营养型细菌——罗氏真养产碱菌被用于高级醇的生产。该技术实现了CO2电化学固定与下游转化的高度集成,在电催化与生物转化同步进行的“通电微生物”反应器中总共产出了140 mg/L的C4、C5高级醇,而将固碳生成的甲酸作为唯一碳源的独立发酵模式下,则可产出高达1.4 g/L的高级醇燃料。该技术中的电能可由风力、水力、太阳能等可再生能源转化生成,进而驱动CO2的固定,并最终将原本难以存储和利用的天然能源封装于生物燃料之中,极大地方便了能源的存储、运输及利用。这一思路也激发了多项后续研究[68-69],实现了固碳过程能量效率的进一步提升。
4 高级醇生产菌株的筛选高产菌株的选育普遍依赖于对突变库的定向筛选,其中筛选方法的效率是决定成败的关键。与基于目标化合物自身理化特性的传统方法相比,近年来兴起的生物传感器技术在原位、实时筛选中展现出巨大优势,已被应用于多种小分子化合物[15]及复杂天然产物[70]高产菌株的筛选。该系统通常由特定的转录因子及其调控的启动子组成,其中的转录因子能够特异性地结合目标产物,继而开启或关闭对应的启动子。这一调控强度通常与目标化合物浓度呈正相关,当启动子下游连接荧光蛋白编码基因或抗性基因时,即可通过光学信号的改变或菌株的生长水平鉴别出高产菌株。
假单胞菌属的Pseudomonas butanovora含有一套醇诱导型基因表达调控系统,该系统由转录因子BmoR和bmo启动子(Pbmo) 组成,其中BmoR具有与σ54型转录因子相似的结构,包括激活因子交互域、ATP酶域,以及C端的“螺旋-转角-螺旋”DNA结合域。野生型BmoR可以结合C2-C8的直链醇、分支醇及部分二醇,进而激活Pbmo启动子驱动的基因转录[15, 71]。该系统已被应用于高级醇生产菌株的选择(Selection) 与筛查(Screening)。在选择系统中,四环素抗性基因tetA被置于Pbmo下游,只有当菌体累积目标高级醇时才能诱发BmoR的转录激活功能,从而产生TetA以抵御四环素毒性,使菌体生长与高级醇产量相偶联[15];在筛查系统中,筛选标记被替换为荧光蛋白编码基因,菌体的荧光强度作为高级醇产量的指征[12]。上述系统已被成功应用于合成途径基因表达强度的优化和底盘菌株的筛选,分别得到了正丁醇及异丁醇的高产菌株。
高产量是生产菌株筛选的关键指标,然而,基于野生型BmoR-Pbmo的高级醇传感系统只能响应0–40 mmol/L (0–3.0 g/L) 的正丁醇或异丁醇[31, 15],在底物浓度达到40 mmol/L即达到饱和,无法用于更高产菌株的鉴别。为扩展筛选体系的响应范围,Yu等利用随机突变、结构模拟及分子对接技术推测了BmoR的3个底物结合位点,在单点突变的基础上构建了包含N端和C端两个功能域的组合突变,成功将筛选系统对异丁醇的检测上限提升至100 mmol/L,实现了对7.4 g/L以上产量水平的异丁醇高产菌株的筛选[31],证明通过蛋白质工程技术可以改变转录因子对响应底物的结合偏好和结合动力学特征,实现对剂量响应范围的调控。后续可针对BmoR的感应对象进行改造,构建特异性响应不同种类高级醇的专用筛选体系。
5 高级醇的工业化现状全球的高级醇市场正处于高速上升阶段,以丁醇为例,当前异丁醇的全球规模已突破10亿美元,并以6.4%的复合年均增长率持续增长[72];正丁醇的市场规模更是达到50亿美元,复合年均增长率达到5.9%[73]。基于前述的合成途径与菌株优化等技术,以发酵法生产的生物丁醇已成功实现了商业化。美国的Gevo公司通过削弱丙酮酸脱氢酶活性抑制了酵母的产乙醇能力,将其改造为高级醇生产宿主[74],再凭借过表达转录激活因子AFT增强了高级醇合成途径中二羟酸脱水酶的活性[75],构建了高效的异丁醇生产酵母,实现了高达90%理论产率的超高效工业化生产。该公司于2012年建立了世界上第一套商业化生物基异丁醇生产厂房,年产能可达6800万升。拥有异丁醇合成途径专利[76]的Butamax公司也于2013年启动了异丁醇生产厂房的建设,并于2015年与Gevo达成了专利交叉许可协议,共同推动生物基异丁醇的工业化进程。此外,其他生物燃料企业也加大了生物基高级醇的研发投入,美国的Easel Biotechnologies公司开发了两步法生产技术,通过拆分高级醇的合成途径降低了有毒中间产物的累积,大幅提高了合成前体的供应水平以及下游转化反应的驱动力[77];Butalco公司改造了酵母的酮醇酸还原异构酶和3-磷酸甘油醛脱氢酶的辅因子偏好性,实现了异丁醇产量的提升[78]。
6 总结与展望代谢工程的诞生使高级醇的生产方式在过去30年发生了根本性变革,以微生物异源合成为代表的高级醇发酵生产模式克服了化学合成与天然宿主合成的不足,已被证明具有商业可行性。目前,高级醇的工业化发酵普遍以玉米等粮食作物为原料,在全球粮食短缺与碳超排的双重危机下,基于纤维素废弃生物质及非粮作物的“第二代”生物高级醇,乃至由自养微生物直接固碳生成的高级醇有望成为未来重要的绿色能源,亟待实现产量及产率的突破。因此,优良底盘菌株的选育、菌株代谢模式的重构、合成途经与代谢网络的适配、原料碳源向合成途径的重定向,以及配套的原料处理和发酵工艺是生物高级醇未来的研究重点。随着代谢工程、合成生物学、系统生物学、人工智能等技术的进步,高级醇的生物合成即将迎来新一轮技术革新与产业升级,争夺这一领域高地对于推动我国的能源产业升级和生态可持续发展具有重大意义。
| [1] |
Lan EI, Liao JC. Microbial synthesis of n-butanol, isobutanol, and other higher alcohols from diverse resources. Bioresour Technol, 2013, 135: 339-49. DOI:10.1016/j.biortech.2012.09.104
|
| [2] |
Nawab S, Wang N, Ma XY, et al. Genetic engineering of non-native hosts for 1-butanol production and its challenges: a review. Microb Cell Fact, 2020, 19(1): 79. DOI:10.1186/s12934-020-01337-w
|
| [3] |
Atsumi S, Cann AF, Connor MR, et al. Metabolic engineering of Escherichia coli for 1-butanol production. Metab Eng, 2008, 10(6): 305-311. DOI:10.1016/j.ymben.2007.08.003
|
| [4] |
Atsumi S, Hanai T, Liao JC. Non-fermentative pathways for synthesis of branched-chain higher alcohols as biofuels. Nature, 2008, 451(7174): 86-89. DOI:10.1038/nature06450
|
| [5] |
Zhang KC, Sawaya MR, Eisenberg DS, et al. Expanding metabolism for biosynthesis of nonnatural alcohols. Proc Natl Acad Sci USA, 2008, 105(52): 20653-20658. DOI:10.1073/pnas.0807157106
|
| [6] |
Higashide W, Li YC, Yang YF, et al. Metabolic engineering of Clostridium cellulolyticum for production of isobutanol from cellulose. Appl Environ Microbiol, 2011, 77(8): 2727-2733. DOI:10.1128/AEM.02454-10
|
| [7] |
Choi KY, Wernick DG, Tat CA, et al. Consolidated conversion of protein waste into biofuels and ammonia using Bacillus subtilis. Metab Eng, 2014, 23: 53-61. DOI:10.1016/j.ymben.2014.02.007
|
| [8] |
Li H, Opgenorth PH, Wernick DG, et al. Integrated electromicrobial conversion of CO2 to higher alcohols. Science, 2012, 335(6076): 1596. DOI:10.1126/science.1217643
|
| [9] |
Lan EI, Liao JC. Metabolic engineering of cyanobacteria for 1-butanol production from carbon dioxide. Metab Eng, 2011, 13(4): 353-363. DOI:10.1016/j.ymben.2011.04.004
|
| [10] |
Atsumi S, Wu TY, Machado IMP, et al. Evolution, genomic analysis, and reconstruction of isobutanol tolerance in Escherichia coli. Mol Syst Biol, 2010, 6: 449. DOI:10.1038/msb.2010.98
|
| [11] |
Wang JG, Huang CY, Guo K, et al. Converting Escherichia coli MG1655 into a chemical overproducer through inactivating defense system against exogenous DNA. Synth Syst Biotechnol, 2020, 5(4): 333-342. DOI:10.1016/j.synbio.2020.10.005
|
| [12] |
Yu H, Chen ZY, Wang N, et al. Engineering transcription factor BmoR for screening butanol overproducers. Metab Eng, 2019, 56: 28-38. DOI:10.1016/j.ymben.2019.08.015
|
| [13] |
Huo YX, Cho KM, Rivera JG, et al. Conversion of proteins into biofuels by engineering nitrogen flux. Nat Biotechnol, 2011, 29(4): 346-351. DOI:10.1038/nbt.1789
|
| [14] |
Bastian S, Liu X, Meyerowitz JT, et al. Engineered ketol-acid reductoisomerase and alcohol dehydrogenase enable anaerobic 2-methylpropan-1-ol production at theoretical yield in Escherichia coli. Metab Eng, 2011, 13(3): 345-352. DOI:10.1016/j.ymben.2011.02.004
|
| [15] |
Dietrich JA, Shis DL, Alikhani A, et al. Transcription factor-based screens and synthetic selections for microbial small-molecule biosynthesis. ACS Synth Biol, 2013, 2(1): 47-58. DOI:10.1021/sb300091d
|
| [16] |
Atsumi S, Liao JC. Directed evolution of Methanococcus jannaschii citramalate synthase for biosynthesis of 1-propanol and 1-butanol by Escherichia coli. Appl Environ Microbiol, 2008, 74(24): 7802-7808. DOI:10.1128/AEM.02046-08
|
| [17] |
Choi YJ, Park JH, Kim TY, et al. Metabolic engineering of Escherichia coli for the production of 1-propanol. Metab Eng, 2012, 14(5): 477-486. DOI:10.1016/j.ymben.2012.07.006
|
| [18] |
Inokuma K, Liao JC, Okamoto M, et al. Improvement of isopropanol production by metabolically engineered Escherichia coli using gas stripping. J Biosci Bioeng, 2010, 110(6): 696-701. DOI:10.1016/j.jbiosc.2010.07.010
|
| [19] |
Soma Y, Inokuma K, Tanaka T, et al. Direct isopropanol production from cellobiose by engineered Escherichia coli using a synthetic pathway and a cell surface display system. J Biosci Bioeng, 2012, 114(1): 80-85. DOI:10.1016/j.jbiosc.2012.02.019
|
| [20] |
Jojima T, Inui M, Yukawa H. Production of isopropanol by metabolically engineered Escherichia coli. Appl Microbiol Biotechnol, 2008, 77(6): 1219-1224. DOI:10.1007/s00253-007-1246-8
|
| [21] |
Lee J, Jang YS, Choi SJ, et al. Metabolic engineering of Clostridium acetobutylicum ATCC 824 for isopropanol-butanol-ethanol fermentation. Appl Environ Microbiol, 2012, 78(5): 1416-1423. DOI:10.1128/AEM.06382-11
|
| [22] |
Dusséaux S, Croux C, Soucaille P, et al. Metabolic engineering of Clostridium acetobutylicum ATCC 824 for the high-yield production of a biofuel composed of an isopropanol/butanol/ethanol mixture. Metab Eng, 2013, 18: 1-8. DOI:10.1016/j.ymben.2013.03.003
|
| [23] |
Lan EI, Liao JC. ATP drives direct photosynthetic production of 1-butanol in cyanobacteria. Proc Natl Acad Sci USA, 2012, 109(16): 6018-6023. DOI:10.1073/pnas.1200074109
|
| [24] |
Jang YS, Lee JY, Lee J, et al. Enhanced butanol production obtained by reinforcing the direct butanol-forming route in Clostridium acetobutylicum. mBio, 2012, 3(5): e00314-12.
|
| [25] |
Yu MR, Du YM, Jiang WY, et al. Effects of different replicons in conjugative plasmids on transformation efficiency, plasmid stability, gene expression and n-butanol biosynthesis in Clostridium tyrobutyricum. Appl Microbiol Biotechnol, 2012, 93(2): 881-889. DOI:10.1007/s00253-011-3736-y
|
| [26] |
Steen EJ, Chan R, Prasad N, et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microb Cell Fact, 2008, 7: 36. DOI:10.1186/1475-2859-7-36
|
| [27] |
Nielsen DR, Leonard E, Yoon SH, et al. Engineering alternative butanol production platforms in heterologous bacteria. Metab Eng, 2009, 11(4/5): 262-273.
|
| [28] |
Berezina OV, Zakharova NV, Brandt A, et al. Reconstructing the clostridial n-butanol metabolic pathway in Lactobacillus brevis. Appl Microbiol Biotechnol, 2010, 87(2): 635-646. DOI:10.1007/s00253-010-2480-z
|
| [29] |
Köpke M, Held C, Hujer S, et al. Clostridium ljungdahlii represents a microbial production platform based on syngas. Proc Natl Acad Sci USA, 2010, 107(29): 13087-13092. DOI:10.1073/pnas.1004716107
|
| [30] |
Shen CR, Lan EI, Dekishima Y, et al. Driving forces enable high-titer anaerobic 1-butanol synthesis in Escherichia coli. Appl Environ Microbiol, 2011, 77(9): 2905-2915. DOI:10.1128/AEM.03034-10
|
| [31] |
Yu H, Wang N, Huo WB, et al. Establishment of BmoR-based biosensor to screen isobutanol overproducer. Microb Cell Fact, 2019, 18(1): 30. DOI:10.1186/s12934-019-1084-2
|
| [32] |
Atsumi S, Higashide W, Liao JC. Direct photosynthetic recycling of carbon dioxide to isobutyraldehyde. Nat Biotechnol, 2009, 27(12): 1177-1180. DOI:10.1038/nbt.1586
|
| [33] |
Su HF, Lin JF, Wang GW. Metabolic engineering of Corynebacterium crenatium for enhancing production of higher alcohols. Sci Rep, 2016, 6: 39543. DOI:10.1038/srep39543
|
| [34] |
Boock JT, Freedman AJE, Tompsett GA, et al. Engineered microbial biofuel production and recovery under supercritical carbon dioxide. Nat Commun, 2019, 10: 587. DOI:10.1038/s41467-019-08486-6
|
| [35] |
Li SS, Jia XQ, Wen JP. Improved 2-methyl-1-propanol production in an engineered Bacillus subtilis by constructing inducible pathways. Biotechnol Lett, 2012, 34(12): 2253-2258. DOI:10.1007/s10529-012-1041-1
|
| [36] |
Blombach B, Riester T, Wieschalka S, et al. Corynebacterium glutamicum tailored for efficient isobutanol production. Appl Environ Microbiol, 2011, 77(10): 3300-10. DOI:10.1128/AEM.02972-10
|
| [37] |
Lu JN, Brigham CJ, Gai CS, et al. Studies on the production of branched-chain alcohols in engineered Ralstonia eutropha. Appl Microbiol Biotechnol, 2012, 96(1): 283-297. DOI:10.1007/s00253-012-4320-9
|
| [38] |
Matsuda F, Ishii J, Kondo T, et al. Increased isobutanol production in Saccharomyces cerevisiae by eliminating competing pathways and resolving cofactor imbalance. Microb Cell Fact, 2013, 12: 119. DOI:10.1186/1475-2859-12-119
|
| [39] |
Russmayer H, Marx H, Sauer M. Microbial 2-butanol production with Lactobacillus diolivorans. Biotechnol Biofuels, 2019, 12: 262. DOI:10.1186/s13068-019-1594-5
|
| [40] |
Chen Z, Wu Y, Huang JH, et al. Metabolic engineering of Klebsiella pneumoniae for the de novo production of 2-butanol as a potential biofuel. Bioresour Technol, 2015, 197: 260-265. DOI:10.1016/j.biortech.2015.08.086
|
| [41] |
Ghiaci P, Lameiras F, Norbeck J, et al. Production of 2-butanol through meso-2, 3-butanediol consumption in lactic acid bacteria. FEMS Microbiol Lett, 2014, 360(1): 70-75. DOI:10.1111/1574-6968.12590
|
| [42] |
Ghiaci P, Norbeck J, Larsson C. 2-Butanol and butanone production in Saccharomyces cerevisiae through combination of a B12 dependent dehydratase and a secondary alcohol dehydrogenase using a TEV-based expression system. PLoS ONE, 2014, 9(7): e102774. DOI:10.1371/journal.pone.0102774
|
| [43] |
Speranza G, Corti S, Fontana G, et al. Conversion of meso-2, 3-butanediol into 2-butanol by Lactobacilli. stereochemical and enzymatic aspects. J Agric Food Chem, 1997, 45(9): 3476-3480. DOI:10.1021/jf970103+
|
| [44] |
Cann AF, Liao JC. Production of 2-methyl-1-butanol in engineered Escherichia coli. Appl Microbiol Biotechnol, 2008, 81(1): 89-98. DOI:10.1007/s00253-008-1631-y
|
| [45] |
Connor MR, Cann AF, Liao JC. 3-methyl-1-butanol production in Escherichia coli: random mutagenesis and two-phase fermentation. Appl Microbiol Biotechnol, 2010, 86(4): 1155-1164. DOI:10.1007/s00253-009-2401-1
|
| [46] |
Ferreira S, Pereira R, Liu F, et al. Discovery and implementation of a novel pathway for n-butanol production via 2-oxoglutarate. Biotechnol Biofuels, 2019, 12: 230. DOI:10.1186/s13068-019-1565-x
|
| [47] |
Ma C, Ou JF, Xu NN, et al. Rebalancing redox to improve biobutanol production by Clostridium tyrobutyricum. Bioengineering (Basel), 2015, 3(1): 2. DOI:10.3390/bioengineering3010002
|
| [48] |
Du YM, Jiang WY, Yu MR, et al. Metabolic process engineering of Clostridium tyrobutyricum Δack-adhE2 for enhanced n-butanol production from glucose: effects of methyl viologen on NADH availability, flux distribution, and fermentation kinetics. Biotechnol Bioeng, 2015, 112(4): 705-715. DOI:10.1002/bit.25489
|
| [49] |
Atsumi S, Wu TTY, Eckl EM, et al. Engineering the isobutanol biosynthetic pathway in Escherichia coli by comparison of three aldehyde reductase/alcohol dehydrogenase genes. Appl Microbiol Biotechnol, 2010, 85(3): 651-657. DOI:10.1007/s00253-009-2085-6
|
| [50] |
Lim JH, Seo SW, Kim SY, et al. Model-driven rebalancing of the intracellular redox state for optimization of a heterologous n-butanol pathway in Escherichia coli. Metab Eng, 2013, 20: 56-62. DOI:10.1016/j.ymben.2013.09.003
|
| [51] |
Brynildsen MP, Liao JC. An integrated network approach identifies the isobutanol response network of Escherichia coli. Mol Syst Biol, 2009, 5: 277. DOI:10.1038/msb.2009.34
|
| [52] |
Knoshaug EP, Zhang M. Butanol tolerance in a selection of microorganisms. Appl Biochem Biotechnol, 2009, 153(1/3): 13-20.
|
| [53] |
Rühl J, Schmid A, Blank LM. Selected Pseudomonas putida strains able to grow in the presence of high butanol concentrations. Appl Environ Microbiol, 2009, 75(13): 4653-4656. DOI:10.1128/AEM.00225-09
|
| [54] |
Zaki AM, Wimalasena TT, Greetham D. Phenotypic characterisation of Saccharomyces spp. for tolerance to 1-butanol. J Ind Microbiol Biotechnol, 2014, 41(11): 1627-1636. DOI:10.1007/s10295-014-1511-7
|
| [55] |
Kataoka N, Tajima T, Kato J, et al. Development of butanol-tolerant Bacillus subtilis strain GRSW2-B1 as a potential bioproduction host. AMB Express, 2011, 1: 10. DOI:10.1186/2191-0855-1-10
|
| [56] |
Baez A, Cho KM, Liao JC. High-flux isobutanol production using engineered Escherichia coli: a bioreactor study with in situ product removal. Appl Microbiol Biotechnol, 2011, 90(5): 1681-1690. DOI:10.1007/s00253-011-3173-y
|
| [57] |
Artzi L, Bayer EA, Moraïs S. Cellulosomes: bacterial nanomachines for dismantling plant polysaccharides. Nat Rev Microbiol, 2017, 15(2): 83-95. DOI:10.1038/nrmicro.2016.164
|
| [58] |
Willson BJ, Kovács K, Wilding-Steele T, et al. Production of a functional cell wall-anchored minicellulosome by recombinant Clostridium acetobutylicum ATCC 824. Biotechnol Biofuels, 2016, 9: 109. DOI:10.1186/s13068-016-0526-x
|
| [59] |
Fan LH, Zhang ZJ, Mei S, et al. Engineering yeast with bifunctional minicellulosome and cellodextrin pathway for co-utilization of cellulose-mixed sugars. Biotechnol Biofuels, 2016, 9: 137. DOI:10.1186/s13068-016-0554-6
|
| [60] |
Anandharaj M, Lin YJ, Rani RP, et al. Constructing a yeast to express the largest cellulosome complex on the cell surface. Proc Natl Acad Sci USA, 2020, 117(5): 2385-2394. DOI:10.1073/pnas.1916529117
|
| [61] |
Wen ZQ, Li Q, Liu JL, et al. Consolidated bioprocessing for butanol production of cellulolytic Clostridia: development and optimization. Microb Biotechnol, 2020, 13(2): 410-422. DOI:10.1111/1751-7915.13478
|
| [62] |
Wen ZQ, Ledesma-Amaro R, Lin JP, et al. Improved n-butanol production from Clostridium cellulovorans by integrated metabolic and evolutionary engineering. Appl Environ Microbiol, 2019, 85(7): e02560-18.
|
| [63] |
Ma LJ, Guo LW, Yang YP, et al. Protein-based biorefining driven by nitrogen-responsive transcriptional machinery. Biotechnol Biofuels, 2020, 13: 29. DOI:10.1186/s13068-020-1667-5
|
| [64] |
Li SY, Ng IS, Chen PT, et al. Biorefining of protein waste for production of sustainable fuels and chemicals. Biotechnol Biofuels, 2018, 11: 256. DOI:10.1186/s13068-018-1234-5
|
| [65] |
Gleizer S, Ben-Nissan R, Bar-On YM, et al. Conversion of Escherichia coli to generate all biomass carbon from CO2. Cell, 2019, 179(6): 1255-1263. DOI:10.1016/j.cell.2019.11.009
|
| [66] |
Gassler T, Sauer M, Gasser B, et al. The industrial yeast Pichia pastoris is converted from a heterotroph into an autotroph capable of growth on CO2. Nat Biotechnol, 2020, 38(2): 210-216. DOI:10.1038/s41587-019-0363-0
|
| [67] |
Schwander T, Von Borzyskowski LS, Burgener S, et al. A synthetic pathway for the fixation of carbon dioxide in vitro. Science, 2016, 354(6314): 900-904. DOI:10.1126/science.aah5237
|
| [68] |
Liu C, Colón BC, Ziesack M, et al. Water splitting-biosynthetic system with CO2 reduction efficiencies exceeding photosynthesis. Science, 2016, 352(6290): 1210-1213. DOI:10.1126/science.aaf5039
|
| [69] |
Torella JP, Gagliardi CJ, Chen JS, et al. Efficient solar-to-fuels production from a hybrid microbial-water-splitting catalyst system. Proc Natl Acad Sci USA, 2015, 112(8): 2337-2342. DOI:10.1073/pnas.1424872112
|
| [70] |
Siedler S, Stahlhut SG, Malla S, et al. Novel biosensors based on flavonoid-responsive transcriptional regulators introduced into Escherichia coli. Metab Eng, 2014, 21: 2-8. DOI:10.1016/j.ymben.2013.10.011
|
| [71] |
Kurth EG, Doughty DM, Bottomley PJ, et al. Involvement of BmoR and BmoG in n-alkane metabolism in 'Pseudomonas butanovora'. Microbiology, 2008, 154(1): 139-147. DOI:10.1099/mic.0.2007/012724-0
|
| [72] |
Isobutanol market size, growth & analysis, by product, by distribution channel (online, offline), by application (oil & gas, solvents & coatings, chemical intermediate) by region, forecasts to 2017-2027. Reports and Data, 2020. 9, 168.
|
| [73] |
n-butanol market by application (butyl acrylate, butyl acetate, glycol ethers, direct solvents, plasticizers), and region (Asia Pacific, North America, Europe, Middle East & Africa, South America) - global forecast to 2022. MarketsandMarkets, 2018. 4, 110.
|
| [74] |
Feldman RMR, Gunawardena U, Urano J, et al. Yeast organisms for the production of isobutanol: EP, 2446043. 2010-06-22.
|
| [75] |
Dundon CA, Aristidou A, Hawkins A, et al. Methods of increasing dihydroxy acid dehydratase activity to improve production of fuels, chemicals, and amino acids: US, 8017376. 2011-09-13.
|
| [76] |
Donaldson GK, Eliot AC, Flint D, et al. Fermentive production of four carbon alcohols: US, 7851188. 2010-12-14.
|
| [77] |
Huo YX, Cho KM. Two-stage production of higher alcohols: US, 20150225750. 2015-08-13.
|
| [78] |
Festel G, Boles E, Weber C, et al. Fermentative production of isobutanol with yeast: US, 8530226. 2013-9-10.
|
 2021, Vol. 37
2021, Vol. 37




