中国科学院微生物研究所、中国微生物学会主办
文章信息
- 苏纪勇, 姚圆, 刘玉含, 韩秋宇, 张雯露
- Su Jiyong, Yao Yuan, Liu Yuhan, Han Qiuyu, Zhang Wenlu
- 蔗糖磷酸合酶功能、结构与催化机制的研究进展
- Function, structure and catalytic mechanism of sucrose phosphate synthase: a review
- 生物工程学报, 2021, 37(6): 1858-1868
- Chinese Journal of Biotechnology, 2021, 37(6): 1858-1868
- 10.13345/j.cjb.200743
-
文章历史
- Received: November 23, 2020
- Accepted: March 4, 2021
- Published: March 11, 2021
2. 吉林工程技术师范学院 传媒学院,吉林 长春 130052
2. Media Academy, Jilin Engineering Normal University, Changchun 130052, Jilin, China
蔗糖是一种常见的可食用二糖,许多食品中都添加有蔗糖。甘蔗和甜菜是蔗糖的重要植物来源。蔗糖广泛地调节植物的生长、果实的成熟、淀粉和纤维素的合成等生命过程,相关科学问题的解答能够促进农业发展,因此研究植物蔗糖合成具有明显的实际意义。
蔗糖是植物贮藏和运输碳水化合物的重要形式之一[1]。植物通过光合作用固定二氧化碳后,在叶绿体中合成磷酸丙糖类分子(包括3-磷酸甘油醛和二羟丙酮),这些分子可作为合成蔗糖的原料分子。磷酸丙糖类分子从叶绿体输出至细胞质后,细胞质中的酶就开始合成蔗糖[2]。合成蔗糖的关键限速酶是蔗糖磷酸合酶(Sucrose phosphate synthase,SPS),这种酶催化的反应是:UDP-葡萄糖+果糖-6-磷酸→蔗糖-6-磷酸+UDP。另外一种酶—蔗糖磷酸酶(Sucrose phosphate phosphatase,SPP),进一步把蔗糖-6-磷酸上的磷酸根水解形成蔗糖[3]。SPS是本文将要重点介绍的。
1 SPS的进化与分类SPS是一种糖基转移酶。糖基转移酶可以把NDP-单糖分子上的单糖基团转移到另外一种分子之上,包括糖、蛋白、脂类、DNA、RNA等。糖基转移酶分为两种,一种含有1个单独的结构域,也就是糖基转移酶/GT (Glycotransferase)-A型;另外一种含有2个结构域,即GT-B型,其N-端的结构域命名为A结构域(A-domain),C-端的结构域命名为B结构域(B-domain)[4]。SPS的三维结构预测显示SPS属于GT-B型糖基转移酶[5]。
SPS广泛地存在于植物、蓝细菌、藻类和一些化能自养生物之中[6-9],这提示蔗糖是这些生命贮存能量和碳水化合物的一种分子。分子进化分析显示所有的SPS都是由一个共同祖先基因— “SPS-like”基因进化而来的[5]。高等植物SPS有3种SPS基因家族,分别是A、B和C家族[10]。单子叶植物和双子叶植物的SPS序列在进化上存在着一定差异,并且在它们进化过程中发生了SPS基因数量增加等事件[11]。
植物的基因组中存在多种SPS,并可表达于不同的组织和器官。一种烟草SPS参与了在黑暗状态下淀粉降解过程中蔗糖的合成[12];一种大米SPS所控制的蔗糖合成与花粉萌发有关[13];拟南芥的两个主要SPS在叶片中的缺失限制了蔗糖合成和夜间淀粉降解,但不会改变光合作用过程中的碳分配[14]。总之,植物进化出不同的SPS,说明不同的SPS在时空表达和活性上存在差异,从而达到调节植物特定生理功能的目的。
植物中SPS的氨基酸序列普遍较长,这些SPS除了含有催化结构域以外,还含有较长的额外氨基酸序列,这些序列可以受到翻译后修饰,进而调控SPS的活性[10, 15]。蓝细菌中也存在大量的SPS,一般仅含有一个具有催化活性的紧凑结构。研究发现一种鱼腥藻属蓝细菌Anabaena sp. PCC7120的SPS具有最小的催化活性结构[5]。蓝细菌中的SPS普遍缺少额外的氨基酸序列,这表示这些SPS的活性可能不受共价修饰调控,而可能主要受转录水平、翻译水平和酶降解的调控。
2 SPS的酶学研究蔗糖几乎存在于植物的任何部位,包括叶、根、茎、花朵、果实等,这提示这些部位都含有SPS。在蛋白质异源表达技术还没有广泛应用时,研究者直接从菠菜、大豆、西红柿、烟草、甜菜、甘蔗等经济作物中直接提取SPS,其中菠菜SPS是研究最多的SPS。早期提取菠菜SPS的实验步骤仅包括组织破碎和DEAE离子交换层析[16]。后来,研究者发现通过组合使用多种层析方法对植物SPS进行纯化,能够获得纯度较高的SPS[17]。随着蛋白质异源表达技术的成熟,有多种SPS获得了异源表达,并且SPS的纯度得到了一定的提高[17],包括聚球藻Synechocystis sp. strain PCC 6803 SPS[18]、甘蔗Saccharum officinarum SPS[19]、奥氏嗜热盐丝菌Halothermothrix orenii SPS[20]、嗜甲基菌Methylobacillus flagellatus KT SPS[21]、欧李Cerasus humilis SPS[22]以及细长嗜热聚球藻Thermosynechococcus elongatus SPS-TeSPS[23]。
SPS催化的反应是:UDP-葡萄糖+果糖-6-磷酸→蔗糖-6-磷酸+UDP。反应产物是蔗糖-6-磷酸和UDP,因此,可通过检测蔗糖-6-磷酸和UDP的产量来测定SPS的活力。下面介绍几种测定蔗糖-6-磷酸含量的方法:1) 间苯二酚和蔗糖-6-磷酸混合以后,可以呈现颜色反应,在480 nm处有特征吸收峰[24];2) 使用14C标记的UDP-葡萄糖作为底物,产生具有放射性的蔗糖-6-磷酸,进而联合使用HPLC和闪烁计数器可分别测定UDP-葡萄糖和蔗糖-6-磷酸的含量[25];3) 蔗糖-6-磷酸被碱性磷酸酶[16]或者SPP[26]水解以后,形成蔗糖。使用离子交换层析[24]或薄层层析的方法[23]测定蔗糖的含量,也可以用来测定SPS的活性。
SPS催化反应中的UDP含量也可以被测定。目前有许多试剂盒可以检测UDP的含量,检测的原理是:丙酮酸激酶将磷酸烯醇丙酮酸上的磷酸根转移到UDP上形成丙酮酸和UTP;乳酸脱氢酶将NADH上的氢传递给丙酮酸而形成NAD+和乳酸;NADH在340 nm处有吸收峰,使用分光光度计测定NADH减少可间接测定UDP的产生量。另外,丙酮酸激酶将磷酸烯醇丙酮酸上的磷酸根转移到UDP上形成丙酮酸和UTP后,丙酮酸和2, 4-二硝基苯肼可以呈现颜色反应,在420 nm处有特征吸收峰[24]。总之,测定蔗糖-6-磷酸和UDP的含量变化,可以计算SPS的酶活性。
酶活实验显示多种SPS对UDP-葡萄糖的Km值的范围在1.3–77 mmol/L之间,对果糖-6-磷酸的Km值的范围在0.2–9.3 mmol/L之间[27]。不同种属SPS对UDP-葡萄糖和果糖-6-磷酸的Vmax值也具有一定的差异。早期的研究还发现,磷酸根可抑制SPS的活性[16, 28-30],并推测磷酸根通过别构效应抑制SPS的活性[28],但是这种推测没有得到晶体结构学的支持。最近,我们解析了一种SPS的晶体结构[23],发现其催化位点含有一些精氨酸,这些精氨酸可协助酶结合果糖-6-磷酸上的磷酸根。反应体系中如有磷酸根,那么这些磷酸根会与果糖-6-磷酸竞争结合于催化位点,进而影响酶的催化效率,表明磷酸根抑制SPS的活性不是通过别构效应而是通过竞争性抑制作用。
磷酸根抑制SPS的活性具有一定的生理意义。磷酸丙糖类分子从叶绿体中输出至细胞质,并转化为蔗糖后[31-32],释放的磷酸根可抑制SPS的活性。另外,磷酸根是植物生长所需要的一种营养分子,因此植物吸收的磷酸根也会抑制SPS的活性。除了磷酸根以外,蔗糖-6-磷酸也会抑制SPS[16],这说明产物的释放速率或者逆反应会影响SPS的反应速率。总之,磷酸根和蔗糖-6-磷酸是SPS的抑制剂。
SPS也有激活剂。果糖-6-磷酸和1, 5-脱水葡萄糖醇-6-磷酸可以激活菠菜SPS的活性[16]。研究发现果糖-6-磷酸不仅可以结合于催化位点,还可以结合于催化位点之外,通过别构效应上调SPS的活性。另外,葡萄糖-6-磷酸也可以上调菠菜SPS的活性[28]。植物SPS的N-端存在磷酸化位点[33]。植物SPS被磷酸化以后,活性就会受到抑制。葡萄糖-6-磷酸可抑制SPS激酶的活性,而达到上调SPS活性的目的[34-35]。
除了抑制剂和激活剂可以调节SPS的活性以外,植物SPS的活性还有其他调节方式。植物SPS序列中存在与14-3-3蛋白相互结合的区域,14-3-3蛋白结合SPS后明显抑制SPS的活性[36]。另外,对SPS的组氨酸进行化学修饰后发现SPS的活性完全丧失,这说明有一个重要的组氨酸直接参与催化[37]。不同物种SPS的氨基酸序列对比显示,有一个保守的组氨酸可能存在于催化中心[15, 23]。在已解析的H. orenii SPS和TeSPS的晶体结构中,确实有一个组氨酸存在于催化中心[15, 23]。
尽管前期研究揭示了一些SPS的激活剂和抑制剂,但是还有必要继续深入研究其他小分子物质对SPS的影响,特别是代谢物对SPS的激活或者抑制作用。植物SPS是一个分子量较大的酶,除了催化结构域以外还含有其他序列,这表明植物SPS必然会与其他蛋白质相互作用,进而调节其自身的活性,这一问题有待于进一步研究。
3 SPS的生理功能和提高果实蔗糖含量光合作用的最终产物包括淀粉和蔗糖[38]。淀粉是碳水化合物的贮存形式,而蔗糖既是碳水化合物的贮存形式,也是碳水化合物的运输形式。淀粉合成和蔗糖合成存在一定的对立。当植物进行淀粉合成时,蔗糖的合成受到一定程度的抑制;当植物降解淀粉时,蔗糖的合成又受到一定程度的上调。其中,SPS的活性影响了光合作用,而光合作用是碳在植物中如何分配的主要决定因素[39-40]。SPS也是淀粉与蔗糖合成之间碳分配的关键酶[41]。高活性的SPS意味着碳在蔗糖中的分配增加和淀粉形成中的减少[42]。
SPS在植物体内合成蔗糖能够促进植物的生长。玉米SPS在烟草中的过度表达对叶片碳水化合物收支产生重要影响,进而影响整个植株的发育[43]。菠菜SPS在棉花中的表达可以促进蔗糖的合成和提高纤维的长度和质量[44]。拟南芥SPS在烟草中的过表达也提高了蔗糖在烟草中的含量,促进了烟草的生长。转化株的茎高均显著增加,这与植物纤维增长有关,这提示蔗糖含量的增加能够促进植物生长发育[45]。在烟草中过表达玉米SPS,也能够促进蔗糖合成,最终促进生长发育[46]。
果实中蔗糖水平的增加与SPS活性的增加有着密切的关系。SPS在蔗糖积累和高糖分果实的成熟过程中起着关键的作用[47]。甜瓜中SPS表达量的高低决定了蔗糖在果实中的积累[48]。番茄在成熟的最后阶段,SPS的表达量迅速上调,蔗糖在果实中的量快速积累,同时淀粉的含量急剧下降[47, 49]。在香蕉的成熟阶段,SPS的mRNA转录水平和蛋白翻译水平显著提高[50]。甘蔗茎中蔗糖的积累通过上调SPS的表达量和活性实现,同时下调蔗糖转化酶的表达量和活性[51]。总之,果实成熟阶段,SPS的表达量和活性都上调,进而促进蔗糖的合成,增加果实的甜度。
当植物或者蓝细菌受到环境胁迫压力时,会提高SPS的活性和表达量,以促进蔗糖的合成。蔗糖含有多个羟基,能够模仿水的功能,与生物大分子形成氢键等非共价键,以稳定蛋白质和生物膜的结构功能[6]。植物经历四季变化的过程中,特别是从秋季到冬季时,植物SPS的表达量会大大提高,合成出大量蔗糖来应对寒冷[52]。在5 ℃低温情况下,菠菜合成SPS和蔗糖的量都会大大提高[52]。当绿色生命体处于干旱等环境下时,SPS的表达量和活性也会大大上调[53]。当水稻在缺水环境下时,淀粉快速被α-淀粉酶水解,SPS的表达量和活性迅速上调,合成蔗糖来应对干旱[54]。当蓝藻处于高盐环境中时,蔗糖的含量也会大大提高[55]。当拟南芥处于渗透胁迫环境中时,SPS的表达量同样会上调[56]。总之,绿色生命体可以合成蔗糖来应对环境胁迫压力。绿色生命体应对环境胁迫压力的这种方式,正好可用于扩大蔗糖的产量,以利于相关生物能源产业的发展。
4 植物体内SPS活性的调节方式在1971年,De Fekete推测植物SPS的活性可能会受到翻译后修饰调节[57]。1982年的研究发现,大麦叶子在经过光照后,SPS保持在高活性状态;而大麦在黑暗中放置一段时间以后,从叶子中提取的SPS的活性明显降低。大豆、菠菜、豌豆和甜菜的SPS活性也具有类似的现象[58-59]。光照提高SPS的活性后,玉米通过光合作用合成的蔗糖的量也提高[60]。这说明植物SPS的活性受到昼夜明暗节律的调节,也暗示着有一种未知的翻译后修饰调节SPS的活性[58]。进一步研究发现,未知的翻译后修饰不影响SPS的最大活性。动力学特性的改变会强烈改变体内SPS的活性,这说明SPS在酶动力学活性上存在着不同的形式,并且这些形式之间的分布可以迅速改变[32]。后来,Joan Huber等使用32Pi同位素标记的方法证明,在黑暗情况下SPS被磷酸化,活性受到抑制;在光照情况下,SPS发生磷酸化,活性恢复,表明磷酸化和去磷酸化可以调节植物SPS的活性[61]。
Huber等发现PP2A型磷酸酶的抑制剂冈田酸和微囊藻毒素-LR可以抑制SPS的去磷酸化和活性恢复,这就说明至少有一种PP2A型磷酸酶可以使SPS去磷酸化[62],而许多种激酶可以磷酸化SPS[63],并且磷酸化位点有多个[64]。菠菜中有一种激酶可以对SPS上的两个位点进行磷酸化,并且这种激酶的活性受到葡萄糖-6-磷酸的抑制[34]。相反,NO3-可以上调小麦SPS激酶的活性,从而抑制SPS的活性[65]。另外,两种菠菜SNF1-相关蛋白激酶也可以磷酸化SPS[66]。McMichael等发现,第158位丝氨酸(Ser158) 是SPS的主要磷酸化位点,它的可逆磷酸化调节着SPS的活性。后来Hendrik Weiner获得了几种抗体,可以特异性区别Ser158是否发生磷酸化[67]。除了Ser158可以被磷酸化以外,菠菜SPS的Ser424也可以被磷酸化。有意思的是Ser424磷酸化可以激活SPS的活性[68]。虽然植物SPS磷酸化位点及作用已经明确,但是还缺少植物SPS的晶体结构信息。植物SPS结构的解析可以用来解释磷酸化如何影响SPS结合底物和催化过程。
除了翻译后修饰可以调节SPS活性以外,控制SPS磷酸酶的蛋白合成速率也可以调节SPS的活性。环己酰亚胺是一种蛋白合成抑制剂,能够抑制番茄和柔黄花牧豆树合成SPS的磷酸酶,进而影响SPS磷酸化状态变为去磷酸化状态[69-70]。
5 SPS的晶体结构与催化机制菠菜SPS (UniProt登录号:P31928) 是研究最多的SPS,该酶含有1 056个氨基酸。研究菠菜SPS时,一般直接从菠菜组织中纯化SPS,所获得菠菜SPS的质量和纯度可用于酶活测试,但不适于用来进行蛋白质晶体结构学分析。即使使用大肠杆菌异源过表达技术,也难以获得大量高纯度的菠菜SPS[17]。除了菠菜SPS以外,其他植物的SPS的晶体结构也没有得到解析,这就限制了从分子层面解释植物SPS的催化机制及其调控机制。植物SPS除了催化结构域以外还含有额外的调节部位,相反,细菌的SPS仅含有催化结构域[5],没有额外的序列,结构非常紧凑,适用于晶体结构学研究。
第一个晶体结构得到解析的SPS来源于奥氏嗜热盐丝菌Halothermothrix orenii[15]。H. orenii SPS的晶体结构显示,该酶的N-端的A结构域远离C-端的B结构域,这说明H. orenii SPS在晶体中处于开放状态(Open conformation),没有形成催化中心。H. orenii SPS的A结构域结合有果糖- 6-磷酸,B结构域没有结合UDP-葡萄糖。奥氏嗜热盐丝菌不属于绿色生命体,因此不能使用该晶体结构解释绿色生命体SPS的催化机制。另外,H. orenii SPS的催化活性没有得到鉴定。
蓝细菌被认为是植物叶绿体的祖先,因此,蓝细菌SPS的晶体结构较为适于解释植物SPS的催化机制。蓝细菌中普遍含有SPS基因,鱼腥藻属蓝细菌Anabaena sp. PCC7120就含有最小的SPS[5]。细长嗜热聚球藻Thermosynechococcus elongatus是一种能够在57 ℃生存的蓝细菌[71]。这种蓝细菌的基因组中有一个基因(基因编码是:tll1590;UniProt登录号:Q8DIJ5),NCBI CDD预测这个蛋白可能是SPS。这个基因编码的蛋白质序列仅含有452个氨基酸,比菠菜SPS小很多,没有翻译后修饰位点,结构十分紧凑。CAZY (www.cazy.org) 预测TeSPS属于糖基转移酶家族4的成员。嗜热细菌的蛋白一般更加紧凑,没有额外的无规则卷曲,更加适于蛋白质晶体结构学研究。
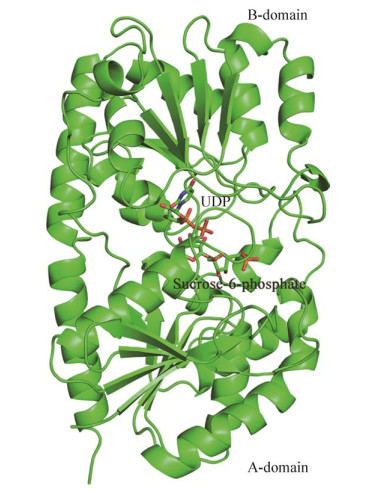
TeSPS能够催化合成蔗糖-6-磷酸[23],但是,蔗糖-6-磷酸由于极性比较大,在薄层层析(Thin layer chromatography,TLC) 上难以展开。加入SPP[26]以后,可以将磷酸从蔗糖-6-磷酸上水解下来,形成的蔗糖可使用TLC检测。最终,TeSPS被证明是有活性的SPS。TeSPS与UDP和蔗糖-6-磷酸可形成共晶体,TeSPS与UDP和蔗糖-6-磷酸共晶体结构得到了解析(图 1)。
晶体结构显示TeSPS的A结构域和B结构域紧紧靠拢,处于关闭状态(Close conformation)。催化中心位于A结构域和B结构域的交界面形成的沟壑里,并且A结构域结合了蔗糖-6-磷酸,B结构域结合了UDP。TeSPS晶体结构和分子动力学模拟显示TeSPS先结合UPD-葡萄糖,再结合果糖-6-磷酸,也就是催化的序列机制。
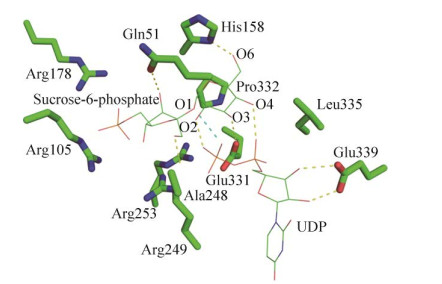
TeSPS晶体结构显示有多个精氨酸位于催化中心周围(图 2),通过离子键稳定蔗糖-6-磷酸上的磷酸根;当然,在TeSPS结合果糖-6-磷酸时,这些精氨酸也可起到稳定果糖-6-磷酸的作用;自由的磷酸根也会通过与这些精氨酸相互作用,影响酶结合果糖-6-磷酸,因此磷酸根是SPS的抑制剂。TeSPS的一级结构中还含有“GPGTP”模块[72],该模块的存在标志着TeSPS属于“保留型”糖基转移酶。所谓“保留型”指糖基转移酶的催化过程不改变糖分子异头碳的构型。
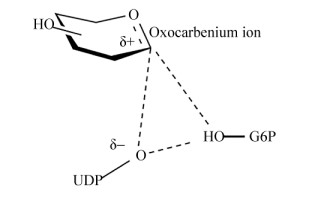
SPS催化的本质是使UDP-葡萄糖分子上葡萄糖与UDP的共价键发生断裂,再使葡萄糖C1与果糖- 6-磷酸的OH2上的氧原子形成共价键。Lee等曾提出这类“保留型”糖基转移酶催化的符合类SNi催化机制。他们的实验结果证实,催化过程存在正面亲核攻击,并且离去基团(UDP-葡萄糖) 和亲核试剂(葡萄糖-6-磷酸) 能够形成一个氢键网络。动力学同位素效应还表明催化过程存在一个高度离解的类氧碳鎓离子样过渡态(图 3)[73],但类氧碳鎓离子是如何形成的还不得而知。
在TeSPS的催化中心(图 2),第331位谷氨酸(Glu331) 位于“GPGTP”模块,Glu331的羧酸侧链可以和蔗糖-6-磷酸的葡萄糖残基上的一个羟基(OH3) 形成氢键。关于同类酶的研究显示,这个位置的酸性氨基酸(谷氨酸或者天冬氨酸) 对于催化来说至关重要,如果把这些氨基酸突变成其他氨基酸,那么酶就会失去催化活性[74-75]。第158位组氨酸(His158) 在催化中心与葡萄糖残基上的另外一个羟基(OH6) 形成氢键。我们把His158突变成成丙氨酸以后,TeSPS也失去了活性[23]。除此之外,葡萄糖残基上的其他3个羟基(OH1、OH2和OH4) 可以和UDP上的2个磷酸根形成氢键。
根据TeSPS与UDP和蔗糖-6-磷酸的共晶体结构,我们对Lee等的类SNi催化机制[73]进行了完善(图 4)。蔗糖-6-磷酸的葡萄糖残基和TeSPS催化中心形成的氢键网络的情形,应该和UDP-葡萄糖的葡萄糖残基结合于TeSPS催化中心时的情形类似。UDP-葡萄糖的葡萄糖残基的羟基上的所有氢原子会与催化中心的His158、Glu331及自身的磷酸根形成氢键网络,这就诱导O1、O2、O3、O4和O6的电荷偏负,并且葡萄糖的呋喃环可能会共享这些负电荷,这会强迫C1带上正电荷以中和这些负电荷,进而达到一种平衡状态,加之葡萄糖C6和OH6在溶液之中活跃度比较高[76-77],OH6与His158形成的氢键并不稳固,如果发生断裂,那么OH6上的O6的电荷就会恢复到中性,那么也意味着呋喃环共享的负电荷就会减少,但是C1带的正电荷没有减少,C1需要寻找一个新的带有负电荷的原子形成共价键。这时,果糖-6-磷酸2号位的氧正好可以与C1形成共价键,果糖-6-磷酸2号位的氢与磷酸根上与葡萄糖连接的氧原子形成共价键,至此TeSPS合成出了蔗糖-6-磷酸。
SPS是植物蔗糖合成的关键限速酶。随着近几十年对SPS不断深入的研究,SPS的翻译后修饰、SPS的抑制剂和激活剂、SPS对植物生长调节等问题渐渐有了较为清晰的答案。但是,为了更深入地研究植物SPS的酶作用机制,需要获得SPS的三维结构,然而目前还未见植物SPS的晶体结构报道。目前,只有两个非植物SPS的晶体结构得到了解析,它们仅有催化结构域而缺少额外的调控序列,不能阐明植物SPS如何受到共价修饰调控,比如Ser158和Ser424的磷酸化如何影响SPS的活性。此外,植物SPS的一级结构较大,提示有其他蛋白可以通过蛋白-蛋白相互作用调控SPS的活性,这方面的研究还比较欠缺。SPS属于糖基转移酶,这类酶底物识别特异性比较差,可对其进行酶进化,也许会获得新颖的糖基转移酶,以用于对其他分子进行糖基化修饰。总之,SPS还有待于继续深入研究。
| [1] |
Stein O, Granot D. An overview of sucrose synthases in plants. Front Plant Sci, 2019, 10: 95. DOI:10.3389/fpls.2019.00095
|
| [2] |
Pallardy S. Physiology of Woody Plants. 3rd Ed. London: Academic Press, 2007: 107-167.
|
| [3] |
Sybesma C. Advances in Photosynthesis Research. Dordrecht: Springer, 1984, 501-504.
|
| [4] |
Lairson LL, Henrissat B, Davies GJ, et al. Glycosyltransferases: structures, functions, and mechanisms. Annu Rev Biochem, 2008, 77: 521-555. DOI:10.1146/annurev.biochem.76.061005.092322
|
| [5] |
Cumino A, Curatti L, Giarrocco L, et al. Sucrose metabolism: Anabaena sucrose-phosphate synthase and sucrose-phosphate phosphatase define minimal functional domains shuffled during evolution. FEBS Lett, 2002, 517(1-3): 19-23. DOI:10.1016/S0014-5793(02)02516-4
|
| [6] |
Lunn JE. Evolution of sucrose synthesis. Plant Physiol, 2002, 128(4): 1490-1500. DOI:10.1104/pp.010898
|
| [7] |
Salerno GL, Curatti L. Origin of sucrose metabolism in higher plants: when, how and why?. Trends Plant Sci, 2003, 8(2): 63-69. DOI:10.1016/S1360-1385(02)00029-8
|
| [8] |
Wind J, Smeekens S, Hanson J. Sucrose: metabolite and signaling molecule. Phytochemistry, 2010, 71(14-15): 1610-1614. DOI:10.1016/j.phytochem.2010.07.007
|
| [9] |
Chain P, Kurtz S, Ohlebusch E, et al. An applications-focused review of comparative genomics tools: capabilities, limitations and future challenges. Brief Bioinform, 2003, 4(2): 105-123. DOI:10.1093/bib/4.2.105
|
| [10] |
Langenkämper G, Fung RWM, Newcomb RD, et al. Sucrose phosphate synthase genes in plants belong to three different families. J Mol Evol, 2002, 54(3): 322-332. DOI:10.1007/s00239-001-0047-4
|
| [11] |
Lutfiyya LL, Xu NF, D'Ordine RL, et al. Phylogenetic and expression analysis of sucrose phosphate synthase isozymes in plants. J Plant Physiol, 2007, 164(7): 923-933. DOI:10.1016/j.jplph.2006.04.014
|
| [12] |
Chen S, Hajirezaei M, Börnke F. Differential expression of sucrose-phosphate synthase isoenzymes in tobacco reflects their functional specialization during dark-governed starch mobilization in source leaves. Plant Physiol, 2005, 139(3): 1163-1174. DOI:10.1104/pp.105.069468
|
| [13] |
Hirose T, Hashida Y, Aoki N, et al. Analysis of gene-disruption mutants of a sucrose phosphate synthase gene in rice, OsSPS1, shows the importance of sucrose synthesis in pollen germination. Plant Sci, 2014, 225: 102-106. DOI:10.1016/j.plantsci.2014.05.018
|
| [14] |
Volkert K, Debast S, Voll LM, et al. Loss of the two major leaf isoforms of sucrose-phosphate synthase in Arabidopsis thaliana limits sucrose synthesis and nocturnal starch degradation but does not alter carbon partitioning during photosynthesis. J Exp Bot, 2014, 65(18): 5217-5229. DOI:10.1093/jxb/eru282
|
| [15] |
Chua TK, Bujnicki JM, Tan TC, et al. The structure of sucrose phosphate synthase from Halothermothrix orenii reveals its mechanism of action and binding mode. Plant Cell, 2008, 20(4): 1059-1072. DOI:10.1105/tpc.107.051193
|
| [16] |
Amir J, Preiss J. Kinetic characterization of spinach leaf sucrose-phosphate synthase. Plant Physiol, 1982, 69(5): 1027-1030. DOI:10.1104/pp.69.5.1027
|
| [17] |
Sonnewald U, Quick WP, MacRae E, et al. Purification, cloning and expression of spinach leaf sucrose-phosphate synthase in Escherichia coli. Planta, 1993, 189(2): 174-181.
|
| [18] |
Curatti L, Folco E, Desplats P, et al. Sucrose-phosphate synthase from Synechocystis sp. strain PCC 6803: identification of the spsA gene and characterization of the enzyme expressed in Escherichia coli. J Bacteriol, 1998, 180(24): 6776-6779. DOI:10.1128/JB.180.24.6776-6779.1998
|
| [19] |
Sawitri WD, Narita H, Ishizaka-Ikeda E, et al. Purification and characterization of recombinant sugarcane sucrose phosphate synthase expressed in E. coli and insect Sf9 cells: an importance of the N-terminal domain for an allosteric regulatory property. J Biochem, 2016, 159(6): 599-607. DOI:10.1093/jb/mvw004
|
| [20] |
Huynh F, Tan TC, Swaminathan K, et al. Expression, purification and preliminary crystallographic analysis of sucrose phosphate synthase (SPS) from Halothermothrix orenii. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2005, 61(Pt 1): 116-117.
|
| [21] |
But SY, Khmelenina VN, Reshetnikov AS, et al. Bifunctional sucrose phosphate synthase/phosphatase is involved in the sucrose biosynthesis by Methylobacillus flagellatus KT. FEMS Microbiol Lett, 2013, 347(1): 43-51. DOI:10.1111/1574-6968.12219
|
| [22] |
Wang J, Du JJ, Mu XP, et al. Cloning and characterization of the Cerasus humilis sucrose phosphate synthase gene (ChSPS1). PLoS ONE, 2017, 12(10): e0186650. DOI:10.1371/journal.pone.0186650
|
| [23] |
Li YY, Yao Y, Yang GS, et al. Co-crystal structure of Thermosynechococcus elongatus sucrose phosphate synthase with UDP and sucrose-6- phosphate provides insight into its mechanism of action involving an oxocarbenium ion and the glycosidic bond. Front Microbiol, 2020, 11: 1050. DOI:10.3389/fmicb.2020.01050
|
| [24] |
Huber SC. Zeitschrift für Pflanzenphysiologie. London: Elsevier GmbH, 1981: 443-450.
|
| [25] |
Salvucci ME, Crafts-Brandner SJ. A high-performance liquid chromatography-based radiometric assay for sucrose-phosphate synthase and other UDP-glucose requiring enzymes. Anal Biochem, 1991, 194(2): 365-368. DOI:10.1016/0003-2697(91)90242-L
|
| [26] |
Fieulaine S, Lunn JE, Borel F, et al. The structure of a cyanobacterial sucrose-phosphatase reveals the sugar tongs that release free sucrose in the cell. Plant Cell, 2005, 17(7): 2049-2058. DOI:10.1105/tpc.105.031229
|
| [27] | |
| [28] |
Doehlert DC, Huber SC. Regulation of spinach leaf sucrose phosphate synthase by glucose-6-phosphate, Inorganic Phosphate, and pH. Plant Physiol, 1983, 73(4): 989-994. DOI:10.1104/pp.73.4.989
|
| [29] |
Doehlert DC, Huber SC. Phosphate inhibition of spinach leaf sucrose phosphate synthase as affected by glucose-6-phosphate and phosphoglucoisomerase. Plant Physiol, 1984, 76(1): 250-253. DOI:10.1104/pp.76.1.250
|
| [30] |
Crafts-Brandner SJ, Salvucci ME. Species and environmental variations in the effect of inorganic phosphate on sucrose-phosphate synthase activity: reliability of assays based upon UDP formation. Plant Physiol, 1989, 91(2): 469-472. DOI:10.1104/pp.91.2.469
|
| [31] |
Stitt M, Wirtz W, Heldt HW. Regulation of sucrose synthesis by cytoplasmic fructosebisphosphatase and sucrose phosphate synthase during photosynthesis in varying light and carbon dioxide. Plant Physiol, 1983, 72(3): 767-774. DOI:10.1104/pp.72.3.767
|
| [32] |
Kerr PS, Kalt-Torres W, Huber SC. Resolution of two molecular forms of sucrose-phosphate synthase from maize, soybean and spinach leaves. Planta, 1987, 170(4): 515-519. DOI:10.1007/BF00402985
|
| [33] |
McMichael Jr RW, Klein RR, Salvucci ME, et al. Identification of the major regulatory phosphorylation site in sucrose-phosphate synthase. Arch Biochem Biophys, 1993, 307(2): 248-252. DOI:10.1006/abbi.1993.1586
|
| [34] |
Huber SC, Huber JL. In vitro phosphorylation and inactivation of spinach leaf sucrose-phosphate synthase by an endogenous protein kinase. Biochim Biophys Acta Mol Cell Res, 1991, 1091(3): 393-400. DOI:10.1016/0167-4889(91)90205-C
|
| [35] |
McMichael RW, Kochansky J, Klein RR, et al. Characterization of the substrate specificity of sucrose-phosphate synthase protein kinase. Arch Biochem Biophys, 1995, 321(1): 71-75. DOI:10.1006/abbi.1995.1369
|
| [36] |
Toroser D, Athwal GS, Huber SC. Site-specific regulatory interaction between spinach leaf sucrose-phosphate synthase and 14-3-3 proteins. FEBS Lett, 1998, 435(1): 110-114. DOI:10.1016/S0014-5793(98)01048-5
|
| [37] |
Sinha AK, Pathre UV, Sane PV. Essential histidyl residues at the active site(s) of sucrose-phosphate synthase from Prosopis juliflora. Biochim Biophys Acta Prot Struct Mol Enzymol, 1998, 1388(2): 397-404. DOI:10.1016/S0167-4838(98)00199-X
|
| [38] |
McClain AM, Sharkey TD. Triose phosphate utilization and beyond: from photosynthesis to end product synthesis. J Exp Bot, 2019, 70(6): 1755-1766. DOI:10.1093/jxb/erz058
|
| [39] |
Galtier N, Foyer CH, Huber J, et al. Effects of elevated sucrose-phosphate synthase activity on photosynthesis, assimilate partitioning, and growth in tomato (Lycopersicon esculentum var UC82B). Plant Physiol, 1993, 101(2): 535-543. DOI:10.1104/pp.101.2.535
|
| [40] |
Worrell AC, Bruneau JM, Summerfelt K, et al. Expression of a maize sucrose phosphate synthase in tomato alters leaf carbohydrate partitioning. Plant Cell, 1991, 3(10): 1121-1130.
|
| [41] |
Rufty TW, Huber SC. Changes in starch formation and activities of sucrose phosphate synthase and cytoplasmic fructose-1, 6-bisphosphatase in response to source-sink alterations. Plant Physiol, 1983, 72(2): 474-480. DOI:10.1104/pp.72.2.474
|
| [42] |
Huber SC, Rufty TW, Kerr PS. Effect of photoperiod on photosynthate partitioning and diurnal rhythms in sucrose phosphate synthase activity in leaves of soybean (Glycine max L.[Merr.]) and tobacco (Nicotiana tabacum L.). Plant Physiol, 1984, 75(4): 1080-1084. DOI:10.1104/pp.75.4.1080
|
| [43] |
Baxter CJ, Foyer CH, Turner J, et al. Elevated sucrose-phosphate synthase activity in transgenic tobacco sustains photosynthesis in older leaves and alters development. J Exp Bot, 2003, 54(389): 1813-1820. DOI:10.1093/jxb/erg196
|
| [44] |
Haigler CH, Singh B, Zhang DS, et al. Transgenic cotton over-producing spinach sucrose phosphate synthase showed enhanced leaf sucrose synthesis and improved fiber quality under controlled environmental conditions. Plant Mol Biol, 2007, 63(6): 815-832. DOI:10.1007/s11103-006-9127-6
|
| [45] |
Park JY, Canam T, Kang KY, et al. Over-expression of an arabidopsis family A sucrose phosphate synthase (SPS) gene alters plant growth and fibre development. Transgenic Res, 2008, 17(2): 181-192. DOI:10.1007/s11248-007-9090-2
|
| [46] |
Seger M, Gebril S, Tabilona J, et al. Impact of concurrent overexpression of cytosolic glutamine synthetase (GS1) and sucrose phosphate synthase (SPS) on growth and development in transgenic tobacco. Planta, 2015, 241(1): 69-81. DOI:10.1007/s00425-014-2165-4
|
| [47] |
Dali N, Michaud D, Yelle S. Evidence for the involvement of sucrose phosphate synthase in the pathway of sugar accumulation in sucrose-accumulating tomato fruits. Plant Physiol, 1992, 99(2): 434-438. DOI:10.1104/pp.99.2.434
|
| [48] |
Hubbard NL, Huber SC, Pharr DM. Sucrose phosphate synthase and acid invertase as determinants of sucrose concentration in developing muskmelon (Cucumis melo L.). Fruits. Plant Physiol, 1989, 91(4): 1527-1534. DOI:10.1104/pp.91.4.1527
|
| [49] |
Miron D, Schaffer AA. Sucrose phosphate synthase, sucrose synthase, and invertase activities in developing fruit of Lycopersicon esculentum Mill. and the sucrose accumulating Lycopersicon hirsutum Humb. and Bonpl. Plant Physiol, 1991, 95(2): 623-627. DOI:10.1104/pp.95.2.623
|
| [50] |
Do Nascimento JRO, Cordenunsi BR, Lajolo FM, et al. Banana sucrose-phosphate synthase gene expression during fruit ripening. Planta, 1997, 203(3): 283-288. DOI:10.1007/s004250050193
|
| [51] |
Zhu YJ, Komor E, Moore PH. Sucrose accumulation in the sugarcane stem is regulated by the difference between the activities of soluble acid invertase and sucrose phosphate synthase. Plant Physiol, 1997, 115(2): 609-616. DOI:10.1104/pp.115.2.609
|
| [52] |
Guy CL, Huber JLA, Huber SC. Sucrose phosphate synthase and sucrose accumulation at low temperature. Plant Physiol, 1992, 100(1): 502-508. DOI:10.1104/pp.100.1.502
|
| [53] |
Hershkovitz N, Oren A, Cohen Y. Accumulation of trehalose and sucrose in cyanobacteria exposed to matric water stress. Appl Environ Microbiol, 1991, 57(3): 645-648. DOI:10.1128/aem.57.3.645-648.1991
|
| [54] |
Yang JC, Zhang JH, Wang ZQ, et al. Activities of starch hydrolytic enzymes and sucrose-phosphate synthase in the stems of rice subjected to water stress during grain filling. J Exp Bot, 2001, 52(364): 2169-2179. DOI:10.1093/jexbot/52.364.2169
|
| [55] |
Liang YJ, Zhang MY, Wang M, et al. Freshwater Cyanobacterium synechococcus elongatus PCC 7942 adapts to an environment with salt stress via ion-induced enzymatic balance of compatible solutes. Appl Environ Microbiol, 2020, 86(7): e02904-19.
|
| [56] |
Solís-Guzmán MG, Argüello-Astorga G, López-Bucio J, et al. Arabidopsis thaliana sucrose phosphate synthase (sps) genes are expressed differentially in organs and tissues, and their transcription is regulated by osmotic stress. Gene Expr Patterns, 2017, 25-26: 92-101. DOI:10.1016/j.gep.2017.06.001
|
| [57] |
De Fekete MA. The regulative properties of UDPglucose: D-fructose-6-phosphate 2-glucosyltransferase (sucrose phosphate synthetase) from Vicia faba cotyledons. Eur J Biochem, 1971, 19(1): 73-80. DOI:10.1111/j.1432-1033.1971.tb01289.x
|
| [58] |
Sicher RC, Kremer DF. Changes of sucrose-phosphate synthase activity in barley primary leaves during light/dark transitions. Plant Physiol, 1984, 76(4): 910-912. DOI:10.1104/pp.76.4.910
|
| [59] |
Vassey TL. Light/dark profiles of sucrose phosphate synthase, sucrose synthase, and acid invertase in leaves of sugar beets. Plant Physiol, 1989, 89(1): 347-351. DOI:10.1104/pp.89.1.347
|
| [60] |
Sicher RC, Kremer DF. Possible control of maize leaf sucrose-phosphate synthase activity by light modulation. Plant Physiol, 1985, 79(3): 695-698. DOI:10.1104/pp.79.3.695
|
| [61] |
Huber JLA, Huber SC, Nielsen TH. Protein phosphorylation as a mechanism for regulation of spinach leaf sucrose-phosphate synthase activity. Arch Biochem Biophys, 1989, 270(2): 681-690. DOI:10.1016/0003-9861(89)90551-1
|
| [62] |
Huber SC, Huber JL. Activation of sucrose-phosphate synthase from darkened spinach leaves by an endogenous protein phosphatase. Arch Biochem Biophys, 1990, 282(2): 421-426. DOI:10.1016/0003-9861(90)90138-O
|
| [63] |
McMichael Jr RW, Bachmann M, Huber SC. Spinach leaf sucrose-phosphate synthase and nitrate reductase are phosphorylated/inactivated by multiple protein kinases in vitro. Plant Physiol, 1995, 108(3): 1077-1082. DOI:10.1104/pp.108.3.1077
|
| [64] |
Huber JL, Huber SC. Site-specific serine phosphorylation of spinach leaf sucrose-phosphate synthase. Biochem J, 1992, 283(3): 877-882. DOI:10.1042/bj2830877
|
| [65] |
Van Quy L, Champigny ML. NO3− Enhances the kinase activity for phosphorylation of Phosphoenolpyruvate carboxylase and sucrose phosphate synthase proteins in wheat leaves: evidence from the effects of mannose and okadaic acid. Plant Physiol, 1992, 99(1): 344-347. DOI:10.1104/pp.99.1.344
|
| [66] |
Sugden C, Donaghy PG, Halford NG, et al. Two SNF1-related protein kinases from spinach leaf phosphorylate and inactivate 3-hydroxy-3-methylglutaryl- coenzyme A reductase, nitrate reductase, and sucrose phosphate synthase in vitro. Plant Physiol, 1999, 120(1): 257-274. DOI:10.1104/pp.120.1.257
|
| [67] |
Hendrik W. Antibodies That Distinguish between the Serine-158 Phospho- and Dephospho-Form of Spinach Leaf Sucrose-Phosphate Synthase. Plant Physiol, 1995, 108(1): 219-225. DOI:10.1104/pp.108.1.219
|
| [68] |
Toroser D, Huber SC. Protein phosphorylation as a mechanism for osmotic-stress activation of sucrose-phosphate synthase in spinach leaves. Plant Physiol, 1997, 114(3): 947-955. DOI:10.1104/pp.114.3.947
|
| [69] |
Jones TL, Ort DR. Circadian regulation of sucrose phosphate synthase activity in tomato by protein phosphatase activity. Plant Physiol, 1997, 113(4): 1167-1175. DOI:10.1104/pp.113.4.1167
|
| [70] |
Sinha AK, Shirke P, Pathre U, et al. Sucrose-phosphate synthase in tree species-light dark regulation involves a component of protein turnover in Prosopis juliflora (SW DC). Biochem Mol Biol Int, 1997, 43(2): 421-431.
|
| [71] |
Yamaoka T, Satoh K, Katoh S. Photosynthetic activities of a thermophilic blue-green alga. Plant Cell Physiol, 1978, 19(6): 943-954. DOI:10.1093/oxfordjournals.pcp.a075684
|
| [72] |
Wrabl JO, Grishin NV. Homology between O-linked GlcNAc transferases and proteins of the glycogen phosphorylase superfamily. J Mol Biol, 2001, 314(3): 365-374. DOI:10.1006/jmbi.2001.5151
|
| [73] |
Lee SS, Hong SY, Errey JC, et al. Mechanistic evidence for a front-side, SNi-type reaction in a retaining glycosyltransferase. Nat Chem Biol, 2011, 7(9): 631-638. DOI:10.1038/nchembio.628
|
| [74] |
Abdian PL, Lellouch AC, Gautier C, et al. Identification of essential amino acids in the bacterial α-mannosyltransferase aceA. J Biol Chem, 2000, 275(51): 40568-40575. DOI:10.1074/jbc.M007496200
|
| [75] |
Cid E, Gomis RR, Geremia RA, et al. Identification of two essential glutamic acid residues in glycogen synthase. J Biol Chem, 2000, 275(43): 33614-33621. DOI:10.1074/jbc.M005358200
|
| [76] |
Su JY, Zhang T, Wang PQ, et al. The water network in galectin-3 ligand binding site guides inhibitor design. Acta Biochim Biophys Sin (Shanghai), 2015, 47(3): 192-198. DOI:10.1093/abbs/gmu132
|
| [77] |
Si YL, Wang Y, Gao J, et al. Crystallization of galectin-8 linker reveals intricate relationship between the N-terminal tail and the linker. Int J Mol Sci, 2016, 17(12): 2088. DOI:10.3390/ijms17122088
|
 2021, Vol. 37
2021, Vol. 37